A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
استخدام الأنابيب الفلورية الرنين نقل الطاقة لدراسة ديناميات البروتين المجمعات في مقياس زمني ميلي ثانية
In This Article
Summary
تفاعلات البروتين البروتين حاسمة بالنسبة للنظم البيولوجية، والدراسات المتعلقة بحركية ملزمة التبصير بديناميات ووظيفة البروتين المجمعات. يمكننا وصف أسلوب التي يوضحها المعلمات الحركية بروتين معقدة باستخدام أسلوب توقف تدفق ونقل الطاقة صدى الأسفار.
Abstract
البروتينات هي عوامل التشغيل الأساسي للنظم البيولوجية، وعادة ما تتفاعل مع غيرها الكلي أو الجزيئات الصغيرة الاضطلاع بوظائفها البيولوجية. يمكن أن تكون مثل هذه التفاعلات ديناميكية للغاية، بمعنى الوحدات الفرعية المتفاعلة المرتبطة باستمرار ونأت بأسعار معينة. بينما قياس تقارب ملزم باستخدام تقنيات مثل الكمية المنسدلة يكشف قوة التفاعل، دراسة حركية ملزم يقدم أفكاراً حول كيفية حمل يحدث التفاعل وكم من الوقت يمكن أن يتواجد كل مجمع. وعلاوة على ذلك، يساعد قياس حركية تفاعل حضور عامل إضافي، مثل عامل تبادل بروتين أو دواء، تكشف عن الآلية التي تنظم التفاعل العامل الآخر، توفير معلومات هامة النهوض بالبحوث الطبية والبيولوجية. هنا، يمكننا وصف بروتوكول لقياس حركية ملزمة البروتين المعقدة التي لديها نسبة عالية من رابطة جوهرية ويمكن أن تنفصل بسرعة بروتين آخر. يستخدم الأسلوب نقل الطاقة صدى الأسفار إلى تقرير تشكيل البروتين المجمع في المختبر، وأنه يمكن رصد رابطة سريعة والانفصال من المجمع في الوقت الحقيقي على فلوريميتير توقف تدفق. باستخدام هذا الفحص، كمياً الثوابت معدل الرابطة وتفكك البروتين المعقدة.
Introduction
في نهاية المطاف تنفذ الأنشطة البيولوجية بالبروتينات، أكثر منها التفاعل مع الآخرين للوظائف البيولوجية المناسبة. استخدام نهج حسابي، ويقدر المبلغ الإجمالي لتفاعلات البروتين البروتين في الإنسان تكون ~ 650,0001، وتعطل هذه التفاعلات غالباً ما يؤدي إلى أمراض2. بسبب أدوارها الأساسية في السيطرة على العمليات الخلوية والعضوي، تطورت أساليب عديدة لدراسة تفاعلات البروتين البروتين، مثل الخميرة-اثنين-الهجين، والتكامل bimolecular الأسفار، سبليت-لوسيفراس التكامل، و الإنزيم co-إيمونوبريسيبيتيشن3. بينما هذه الأساليب جيدة في اكتشاف وتأكيد تفاعلات البروتين البروتين، وعادة غير كمي وهكذا توفر معلومات محدودة عن تقارب بين الشركاء البروتين المتفاعلة. هبوطاً سحب الكمية يمكن استخدامها لقياس تقارب ملزم (مثلاً، تفارق الثابت كد)، ولكن لا قياس حركية للتوثيق، ولا يمكن تطبيقها عند كد منخفضة للغاية نظراً عدم كفاية الإشارات إلى الضجيج نسبة4. مطيافية الرنين (موارد البرنامج الخاصة) السطحية مأكل مثل الطحين يوضحها حركية ملزمة، ولكن يتطلب سطح معين والتثبيت من مادة التفاعل واحد على السطح، والتي يحتمل أن تكون تغيير خاصية الربط من مادة التفاعل5. وعلاوة على ذلك، من الصعب على موارد البرنامج الخاصة لقياس رابطة سريعة و معدلات الانفصال5، وأنه من غير المناسب لاستخدام موارد البرنامج الخاصة لوصف هذا الحدث من تبادل مفارز بروتين في بروتين معقدة. هنا، نحن تصف أسلوب الذي يتيح قياس معدلات البروتين الجمعية المعقدة والتفكيك في مقياس زمني ميلي ثانية. وكان هذا الأسلوب ضروري لتحديد دور جالين-صمامات-Nedd8-دإيسوسياتيد البروتين 1 (Cand1)ك مربع و تبادل6،عامل البروتين7.
وينظم Cand1 ديناميات مربع Skp1•Cul1•F البروتين (SCF) E3 ليجاسيس، التي تنتمي إلى عائلة كبيرة من عصابة سنعمل ubiquitin ligases. سكفس تتألف من كولن Cul1، الذي يربط البروتين المجال الدائري Rbx1، وبروتين و مربع لتبادل، الذي يجند ركائز ويربط Cul1 عن طريق البروتين محول Skp18. ليجاسى E3، SCF يحفز على تصريف أوبيكويتين إلى أن الركيزة، ويتم تنشيطه عندما يتم تجنيد الركيزة بالبروتين و مربع، وعندما يتم تعديل Cul1 بالبروتين مثل أوبيكويتين Nedd89. يربط Cand1 Cul1 غير معدلة، وعند الربط، أنه يعطل كل رابطة البروتين مربع Skp1•F مع Cul1 وعلى تصريف Nedd8 Cul110،11،،من1213. كنتيجة لذلك، Cand1 على ما يبدو مثبط لنشاط صندوق إنقاذ الطفولة في المختبر، ولكن نقص Cand1 في الكائنات الحية بسبب العيوب التي تشير إلى دور إيجابي من Cand1 في تنظيم أنشطة صندوق إنقاذ الطفولة في فيفو14،،من1516 , 17-وأوضح هذه المفارقة أخيرا بدراسة كمية التي كشفت التفاعلات الدينامية بين البروتين Cul1، Cand1، و Skp1•F--مربع. استخدام fluorescence الرنين الطاقة نقل (الحنق) فحوصات الكشف عن تشكيل المجمعات SCF و Cul1•Cand1، رابطة وتفكك معدل الثوابت (كعلى kإيقاف، على التوالي) كانت تقاس على حدة. وكشفت القياسات أن Cand1 و Skp1•F--مربع البروتين شكل ضيق للغاية معقدة مع Cul1، ولكن كالخروج من صندوق إنقاذ الطفولة هو زيادة كبيرة من Cand1 و كالخروج من Cul1•Cand1 هو زيادة هائلة بمربع Skp1•F البروتين6،7. هذه النتائج توفير الدعم الأولى والحاسمة لتحديد دور Cand1 كعامل تبادل بروتين، مما يحفز تشكيل مجمعات SCF الجديدة من خلال إعادة التدوير Cul1 من مجمعات SCF القديمة.
هنا، نحن نقدم إجراءات لتطوير واستخدام مقايسة الحنق لدراسة ديناميات معقدة Cul1•Cand17، ويمكن تطبيق نفس المبدأ على دراسة ديناميات الجزيئات الحيوية المختلفة. بكى عند إحدى الجهات مانحة متحمس مع الطول الموجي المناسب، ويقبلون مع طيف الإثارة تداخل طيف انبعاث المانحة موجوداً ضمن مسافة 10-100. يتم تحويل الدولة متحمس يقبلون، وبالتالي تقليل كثافة المانحين وزيادة كثافة يقبلون18. كفاءة الحنق (ﻫ) يعتمد على دائرة نصف قطرها فورستر (ص0) والمسافة بين المانحين ويقبلون fluorophores (r)، ويتم تعريف بواسطة: E = 0ص6/(ص0 6 + ص6). دائرة نصف قطرها فورستر (ص0) يعتمد على عدد قليل من العوامل، بما في ذلك الاتجاه الزاوي ثنائي القطب، تستخدم التداخل الطيفي زوج يقبلون المانحين، والحل19. لتطبيق التحليل الحنق على فلوريميتير توقف التدفق، الذي يرصد تغير الانبعاثات المانحين في الوقت الحقيقي وتتيح قياسات سريعة كعلى و kإيقاف، من الضروري إثبات كفاءة الحنق التي النتائج في انخفاض كبير في انبعاثات المانحين. ولذلك، تصميم الحنق الكفاءة عن طريق اختيار الزوج المناسب الأصباغ الفلورية ومواقع على البروتينات المستهدفة تعلق الأصباغ مهم وسيتم مناقشتها في هذا البروتوكول.
Protocol
1-تصميم المقايسة الحنق.
- تحميل ملف هيكل Cul1•Cand1 المعقدة من "بنك بيانات البروتين" (الملف 1U6G).
- عرض هيكل Cul1•Cand1 المعقدة في بيمول.
- استخدام دالة القياس تحت قائمة معالج بيمول لتقدير المسافة بين الأحماض الأمينية الأولى من Cand1 وحمض أميني آخر من Cul1 (الشكل 1).
- تحميل عارض أطياف على الإنترنت (انظر الجدول للمواد) وعرض الإثارة وأطياف الانبعاثات 7-أمينو-4-ميثيلكومارين (المجلس الطبي الأسترالي) وفلاش في وقت واحد (الشكل 2). ملاحظة أن المجلس الطبي الأسترالي هو الجهة المانحة الحنق وفلاش يقبلون الحنق.
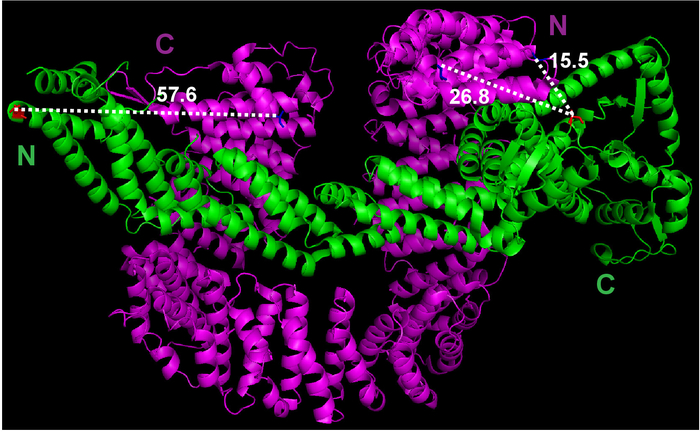
رقم 1: بنية بلورية Cul1•Cand1 وقياس المسافة بين إمكانية تصنيف المواقع. الملف بنية بلورية تحميلها من "مصرف بيانات البروتين" (ملف 1U6G)، وينظر في بيمول. وأجرى قياسات بين ذرات المحدد بيمول. الرجاء انقر هنا لمشاهدة نسخة أكبر من هذا الرقم-
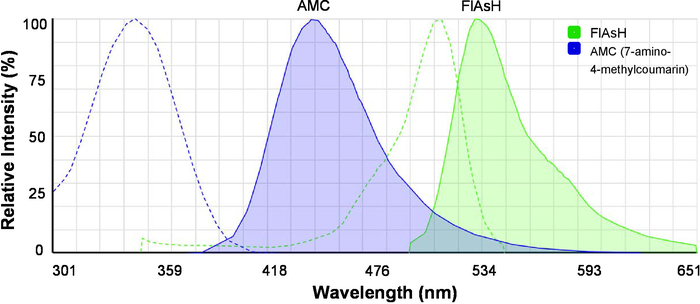
رقم 2: الإثارة وأطياف الانبعاث من الأصباغ الفلورية للحنق. تظهر أطياف المجلس الطبي الأسترالي (7-أمينو-4-ميثيلكومارين) وفلاش. تشير الخطوط المتقطعة إلى أطياف الإثارة، وتشير الخطوط الصلبة إلى أطياف الانبعاث. الصورة تم إنشاؤها أصلاً بواسطة سبيكترافيوير Fluorescence وتعديل لتحسين الوضوح. الرجاء انقر هنا لمشاهدة نسخة أكبر من هذا الرقم-
2-إعداد Cul1 •Rbx1المجلس الطبي الأسترالي، والبروتين المانحين الحنق
- بناء والبلازميدات للتعبير عن •Rbx1سورتاسيCul1 البشرية في الخلايا كولاي . ملاحظة أن والبلازميدات اثنين المشترك معربا عن Cul1•Rbx1 البشرية في الخلايا كولاي موصوفة بالتفصيل في تقرير سابق20.
- إضافة تسلسل الحمض النووي الترميز "لبيتجههههه" (سورتاسي-صاحب6 العلامة) إلى 3 ' نهاية تسلسل Cul1 الترميز عن طريق PCR القياسية وأساليب الاستنساخ21،22.
- التسلسل بلازميد جديد للتأكد من إدراج الجينات دقيقة.
- •Rbx1سورتاسيCul1 إكسبرس المشارك في الخلايا كولاي . الطريقة مستمدة من تقرير سابق20.
- مزيج 100 نانوغرام كل من البلازميدات اثنين مع BL21 (DE3) الخلايا المختصة كيميائيا للتحول المشارك استخدام الحرارة صدمة أسلوب23. تنمو الخلايا في رطل أجار صفيحة تحتوي على 100 ميكروغرام/مل الأمبيسلّين والكلورامفينيكول 34 ميكروغرام/مل عند 37 درجة مئوية بين عشية وضحاها.
- تلقيح 50 مل ثقافة رطل مع المستعمرات تحولت طازجة وتنمو بين عشية وضحاها في 37 درجة مئوية مع الهز 250 دورة في الدقيقة. وهذا يعطي ثقافة كاتب.
- تطعيم 6 قوارير، كل منها 1 لتر متوسطة رطل، مع 5 مل كاتب ثقافة كل وتنمو في 37 درجة مئوية مع 250 دورة في الدقيقة تهتز حتى OD600 ~ 1.0. بارد الثقافة إلى 16 درجة مئوية وإضافة الأيزوبروبيل-β-د-ثيوجالاكتوسيدي (إيبتج) إلى 0.4 مم. الحفاظ الثقافة في 16 درجة مئوية بين عشية وضحاها مع الهز 250 دورة في الدقيقة.
- حصاد الخلايا كولاي عن طريق استخدام الطرد المركزي في 5,000 س ز لمدة 15 دقيقة، وجمع الكريات الخلايا في أنابيب مخروطية 50 مل.
ملاحظة: الكريات خلية يمكن معالجتها لتنقية البروتين أو تجميدها في-80 درجة مئوية قبل الشروع في خطوات تنقية البروتين.
- تنقية •Rbx1سورتاسيCul1 المعقدة. هذا الأسلوب مشتق من تقرير سابق20.
- إضافة 50 مل من تحلل المخزن المؤقت (30 ملم تريس-HCl، 200 ملم كلوريد الصوديوم، 5 ملم DTT، والغليسيرول 10 ٪، 1 قرص من مثبطات البروتياز كوكتيل، درجة الحموضة 7.6) إلى بيليه الخلايا كولاي معربا عن Cul1سورتاسي•Rbx1.
- الخلايا على الجليد مع سونيكاتيون في السعة 50%. بالتناوب بين في ثانية واحدة والثانية 1 إيقاف وتشغيل لمدة 3 دقائق.
- كرر الخطوة 2.3.2 x 2-3.
- نقل الخلية ليستي في أنبوب 50 مل الطرد المركزي وإزالة الأنقاض الخلية باستخدام الطرد المركزي في 25,000 س ز لمدة 45 دقيقة.
- احتضان الخلية واضحة ليساتي مع 5 مل خرز الجلوتاثيون في 4 درجات مئوية عن ح 2.
- الطرد المركزي خليط الخرز ليساتي في 1,500 س ز 2 دقيقة في 4 درجات مئوية. إزالة المادة طافية.
- أغسل حبات مع 5 مل من تحلل العازلة (مع لا مثبطات البروتياز) وإزالة المادة طافية بعد الطرد المركزي في س 1,500 ز لمدة 2 دقيقة في 4 درجات مئوية.
- كرر الخطوة 2.3.7 2 x.
- أضف 3 مل من المخزن المؤقت لتحلل الخرز غسلها ونقل الطين حبة في عمود فارغ.
- إضافة 5 مل من شطف المخزن المؤقت (50 مم تريس-HCl، 200 ملم كلوريد الصوديوم، الجلوتاثيون 10 ملم خفضت، pH 8.0) في العمود. احتضان لمدة 10 دقائق، وجمع النذرة.
- كرر الخطوة 2.3.10 x 3-4.
- إضافة 200 ميليلتر من ثرومبين 5 ملغ/مل (انظر الجدول للمواد) إلى النذرة من الخرز الجلوتاثيون واحتضان بين عشية وضحاها في 4 درجات مئوية.
ملاحظة: يمكن أن يكون مؤقتاً البروتوكول هنا. - تمييع عينة البروتين مع المخزن المؤقت A (25 مم هيبيس، 1 مم DTT، والغليسيرول 5%، الرقم الهيدروجيني 6.5) ثلاثة إضعاف.
- حجته عمود كروماتوغرافيا تبادل الأيونات الموجبة (انظر الجدول للمواد) على نظام فبلك مع مخزن مؤقت أ
- تحميل العينة البروتين على تبادل الأيونات الموجبة متوازن اللوني العمود بمعدل تدفق 0.5 مل/دقيقة.
- الوت البروتين مع تدرج من كلوريد الصوديوم في 40 مل بخلط المخزن المؤقت A و 0 إلى 50% المخزن المؤقت ب (25 مم حبيس، كلوريد الصوديوم م 1، 1 مم DTT، والغليسيرول 5%، الرقم الهيدروجيني 6.5) بمعدل تدفق 1 مل/دقيقة.
- تحقق من البروتين التيد في كسور مختلفة باستخدام مخزونات النشر الاستراتيجي-الصفحة24.
ملاحظة: يمكن أن يكون مؤقتاً البروتوكول هنا. - تجمع النذرة الكسور التي تحتوي على Cul1سورتاسي•Rbx1.
- تركيز العينة •Rbx1سورتاسيCul1 المجمعة إلى 2.5 مل بتمرير المخزن المؤقت عن طريق غشاء أولترافيلتريشن (استقطاع 30 كاتشين).
- إضافة المجلس الطبي الأسترالي إلى ج-محطة Cul1 من خلال وساطة سورتاسي ترانسبيبتيديشن،من2122.
- تغيير المخزن المؤقت في العينة •Rbx1سورتاسيCul1 إلى المخزن المؤقت سورتاسي (50 مم تريس-HCl، 150 مم كلوريد الصوديوم، 10 مم كاكل2، الرقم الهيدروجيني 7.5) باستخدام عمود ديسالتينج (انظر الجدول للمواد).
- حجته عمود desalting مع 25 مل من سورتاسي المخزن المؤقت.
- تحميل 2.5 مل عينة •Rbx1سورتاسيCul1 في العمود. تجاهل في التدفق من خلال.
- الوت العينة مع 3.5 مل من سورتاسي المخزن المؤقت. جمع في التدفق من خلال.
- في 900 ميليلتر من الحل •Rbx1سورتاسيCul1 في المخزن المؤقت سورتاسي، إضافة 100 ميليلتر من 600 ميكرون تنقية سورتاسي الحل و 10 ميليلتر من الببتيد جججالمجلس الطبي الأسترالي 25 مم. احتضان الخليط رد الفعل عند 30 درجة مئوية في ليل الظلام. علما بأن هذه الخطوة سوف تولد Cul1 •Rbx1المجلس الطبي الأسترالي.
تنبيه: الأصباغ الفلورية حساسة للضوء، لذا تجنب تعريضها للضوء المحيط أثناء إعداد البروتين وعينه بقدر الإمكان.
ملاحظة: يمكن أن يكون مؤقتاً البروتوكول هنا. - إضافة 50 ميليلتر من الخرز [اغروس] ني-الإدارة الوطنية للسياحة إلى خليط التفاعل واحتضان في درجة حرارة الغرفة لمدة 30 دقيقة.
- بيليه الخرز [اغروس] ني-الإدارة الوطنية للسياحة بالطرد المركزي في 5,000 س ز 2 دقيقة وجمع المادة طافية.
- توازن حجم استبعاد لوني لعمود (انظر الجدول للمواد) مع المخزن المؤقت (30 ملم تريس-HCl، 100 ملم كلوريد الصوديوم، 1 مم DTT، والغليسيرول 10 ٪) في النظام فبلك.
- تحميل جميع Cul1المجلس الطبي الأسترالي•Rbx1 العينة على حجم الاستبعاد اللوني للعمود. الوت مع 1.5 x عمود حجم المخزن المؤقت.
- تحقق من الكسور النذرة بالحزب الديمقراطي الصربي صفحة24.
- تجمع النذرة الكسور التي تحتوي على Cul1 •Rbx1المجلس الطبي الأسترالي.
- قياس تركيز البروتين باستخدام امتصاص في 280 نيوتن متر مع جهاز المطياف الضوئي.
- قاسمة الحل البروتين ومخزن في-80 درجة مئوية.
ملاحظة: يمكن أن يكون مؤقتاً البروتوكول هنا.
- تغيير المخزن المؤقت في العينة •Rbx1سورتاسيCul1 إلى المخزن المؤقت سورتاسي (50 مم تريس-HCl، 150 مم كلوريد الصوديوم، 10 مم كاكل2، الرقم الهيدروجيني 7.5) باستخدام عمود ديسالتينج (انظر الجدول للمواد).
3-إعداد فلاشCand1، البروتين يقبلون الحنق
ملاحظة: معظم الخطوات في هذا الجزء هي نفس الخطوة 2. يتم وصف الظروف التي تختلف بالتفصيل أدناه.
- بناء بلازميد للتعبير عن Cand1 تيتراسيسالبشرية في الخلايا كولاي .
- إضافة تسلسل الحمض النووي الترميز "ككبجككجسج" (تيتراسيستيني/تيتراسيس العلامة) قبل الأحماض الأمينيةال 15 من Cand1 عن طريق PCR العادية25 (تسلسل التمهيدي: تجكتجتككججكتجكتجكجكاجكجكاتجاكاتككاجكجاكاجاكتتاج؛ كتاكتاجتجتككاتجاتككاج).
- إدراج المنتج بكر ناقل بجيكس-4T-2. التسلسل بلازميد للتأكد من إدراج الجينات دقيقة وفي الإطار.
- التعبير عن تيتراسيسCand1in كولاي الخلايا بنفس الطريقة كخطوة 2-2، إلا أن تتحول بلازميد رشيد الخلايا المختصة.
- تنقية Cand1 تيتراسيسمن الخلايا كولاي .
- الكريات خلية كولاي واستخراج الخرز الجلوتاثيون Cand1using تيتراسيس. هذه الخطوات هي نفس الخطوات 2.3.1–2.3.12.
- تمييع النذرة البروتين من الخرز الجلوتاثيون مع "المخزن المؤقت ج" (50 مم تريس-HCl، 1 مم DTT، والغليسيرول 5%، درجة الحموضة 7.5) بثلاثة إضعاف. حجته على عمود كروماتوغرافيا تبادل شاردة (انظر الجدول للمواد) على نظام فبلك مع "ج المخزن المؤقت" وتحميل العينة البروتين المخفف بمعدل تدفق 0.5 مل/دقيقة.
- الوتي البروتين مع تدرج من كلوريد الصوديوم في 40 مل بخلط "ج المخزن المؤقت" و 0 إلى 50% د المخزن المؤقت (50 مم تريس-HCl، كلوريد الصوديوم م 1، 1 مم DTT، والغليسيرول 5%، درجة الحموضة 7.5) بمعدل تدفق 1 مل/دقيقة. تحقق من البروتين التيد في كسور مختلفة باستخدام مخزونات النشر الاستراتيجي-الصفحة24، وتجمع الكسور التي تحتوي على Cand1 تيتراسيس. لاحظ أن Cand1 تيتراسيساحتفاظ بحجم أكبر من ضريبة السلع والخدمات مجاناً.
- تركيز العينة Cand1 تيتراسيسالمجمعة بتمرير المخزن المؤقت عن طريق غشاء أولترافيلتريشن (استقطاع 30 كاتشين).
- حجته العمود حجم الاستبعاد اللوني مع المخزن المؤقت وضع العلامات (20 مم تريس-HCl، 100 ملم كلوريد الصوديوم، 2 مم تسيب، 1 مم يدتا، والغليسيرول 5%) على نظام فبلك. تحميل عينة Cand1 تيتراسيس(500 ميليلتر في كل مرة) والتحقق من الكسور النذرة بالحزب الديمقراطي الصربي صفحة24.
- تجمع جميع الكسور التي تحتوي على Cand1 تيتراسيسوتركيز البروتين إلى ميكرومتر ~ 40 بتمرير المخزن المؤقت عن طريق غشاء أولترافيلتريشن (استقطاع 30 كاتشين). تقدير تركيز البروتين باستخدام امتصاص في 280 نانومتر. تخزين البروتين ك 50 ميليلتر مختبرين في-80 درجة مئوية.
ملاحظة: يمكن أن يكون مؤقتاً البروتوكول هنا.
- إعداد Cand1 فلاش.
- أضف 1 ميليلتر من الحل فلاش (انظر الجدول للمواد) 50 ميليلتر تيتراسيسCand1 الحل.
- يخلط جيدا واحتضان الخليط في درجة حرارة الغرفة في الظلام ح 1-2 للحصول على فلاشCand1.
ملاحظة: يمكن أن يكون مؤقتاً البروتوكول هنا.
4-إعداد Cand1، البروتين تشيس الحنق
ملاحظة: بروتوكول إعداد البروتين مشابهة إلى الخطوة 3، مع إدخال التعديلات التالية.
- إدراج تسلسل الترميز Cand1 كامل طول الموجه بجي-4T-2.
- تغيير المخزن المؤقت المستخدم في الخطوة 3.3.5 إلى المخزن مؤقت الذي يحتوي على 30 ملم تريس-HCl، 100 ملم كلوريد الصوديوم، 1 مم DTT، والغليسيرول 10%.
- إلغاء الخطوات 3.3.7 و 3.3.8.
5-اختبار وتأكيد التحليل الحنق
- إعداد المخزن المؤقت الحنق التي تحتوي على 30 ملم تريس-HCl، 100 ملم كلوريد الصوديوم، 0.5 مم DTT، أوفالبومين 1 ملغ/مل، درجة الحموضة 7.6، واستخدم في درجة حرارة الغرفة.
- اختبار الحنق بين Cul1 •Rbx1المجلس الطبي الأستراليو فلاشCand1 على فلوريميتير.
- في 300 ميليلتر من المخزن المؤقت لبكي، إضافة Cul1 •Rbx1المجلس الطبي الأسترالي(الحنق المانحة) بتركيز نهائي من 70 نانومتر. نقل الحل إلى ومبومو.
- مكان ومبومو في حامل فلوريميتير عينة. تثير العينة مع 350 نانومتر الإثارة الخفيفة ومسح الإشارات الانبعاثات من 400 نانومتر إلى 600 نانومتر 1 نانومتر زيادات.
- كرر الخطوة 5.2.1 لكن التغيير Cul1 •Rbx1المجلس الطبي الأسترالي فلاشCand1 (الحنق يقبلون). مسح العينة Cand1 فلاشباستخدام نفس الأسلوب كما في الخطوة 5.2.2.
- (اختياري) تثير Cand1 فلاشمع 510 نانومتر الإثارة الخفيفة، ومسح إشارة الانبعاثات من 500 نانومتر إلى 650 نانومتر.
- في 300 ميليلتر الحنق العازلة، إضافة كلا Cul1 •Rbx1المجلس الطبي الأستراليو فلاشCand1 إلى نهائي تركز 70 نانومتر. تحليل العينة بنفس الطريقة كما في الخطوة 5.2.2.
- تأكيد الحنق بين Cul1 •Rbx1المجلس الطبي الأستراليو فلاشCand1 عن طريق إضافة البروتين تشيس (غير المسماة Cand1) (الشكل 3).
- في 300 ميليلتر من المخزن المؤقت لبكي، إضافة 70 نانومتر Cul1المجلس الطبي الأسترالي•Rbx1 و 700 نانومتر Cand1. مسح انبعاث عينة كما هو الحال في الخطوة 5.2.2.
- في 300 ميليلتر من المخزن المؤقت لبكي، إضافة 70 نانومتر فلاشCand1 و 700 نانومتر Cand1. مسح انبعاث عينة كما هو الحال في الخطوة 5.2.2.
- في 300 ميليلتر من المخزن المؤقت لبكي، إضافة 70 نانومتر Cul1المجلس الطبي الأسترالي•Rbx1 و 70 نانومتر Cand1 فلاش، واحتضان العينة في درجة حرارة الغرفة لمدة 5 دقائق. قم بإضافة 700 نانومتر Cand1، ومباشرة بعد الإضافة إلى ذلك، تفحص انبعاث عينة كما هو الحال في الخطوة 5.2.2). لاحظ أن هذه الخطوة مشابهة لخطوة 5.2.5.
- في 300 ميليلتر الحنق العازلة، إضافة تسلسلياً 70 نانومتر Cul1المجلس الطبي الأسترالي•Rbx1، 700 نانومتر Cand1، و 70 نانومتر Cand1 فلاش. احتضان العينة في درجة حرارة الغرفة لمدة 5 دقائق، ومسح انبعاث عينة كما هو الحال في الخطوة 5.2.2. لاحظ أن هذا هو عينة تشيس (الخط الأخضر في الشكل 3).
6-قياس ثابت معدل رابطة (كعلى) من Cul1•Cand1
ملاحظة: تفاصيل التشغيل فلوريميتير توقف تدفق قد وصفت تقرير سابق26.
- إعداد فلوريميتير توقف تدفق للقياس.
- قم بتشغيل فلوريميتير توقف التدفق وفقا لتعليمات الشركة المصنعة.
- مجموعة الإثارة الخفيفة في 350 نانومتر، واستخدام مرشح تمرير فرقة يسمح 450 نانومتر انبعاث الضوء لتمرير وكتل 500-650 نانومتر انبعاث الضوء.
- تبقى الصمامات عينة في منصب شغل ، والاتصال 3 مل حقنه مملوءة بالمياه. أغسل المحاقن عينة اثنين (A و B) مع الماء بتحريك محرك الأقراص حقنه عينة صعودا وهبوطاً عدة مرات. تجاهل جميع المياه المستخدمة في هذه الخطوة.
- تبقى الصمامات عينة في منصب شغل ، والاتصال 3 مل حقنه مليئة بالحنق المخزن المؤقت. أغسل المحاقن عينة اثنين مع المخزن المؤقت الحنق بتحريك محرك الأقراص حقنه عينة صعودا وهبوطاً عدة مرات. تجاهل كل الحنق المخزن المؤقت المستخدم في هذه الخطوة.
- تأخذ قياس تحكم (الشكل 4).
- الاتصال حقنه 3 مل وتحميل حقنه A مع 100 نانومتر Cul1المجلس الطبي الأسترالي•Rbx1 في المخزن المؤقت للحنق. تشغيل صمام نموذج لموقف السيارة .
- توصيل حقنه 3 مل وتحميل "ب المحاقن" مع المخزن المؤقت الحنق. تشغيل صمام نموذج لموقف السيارة .
- استخدم لوحة التحكم تحت الحصول على في البرنامج أن خمس طلقات (مزيج تساوي حجم العينات من المحاقن أ وب المحاقن) في فلوريميتير توقف التدفق دون تسجيل النتائج.
- فتح لوحة التحكم تحت الحصول على البرمجيات، وبرنامج لتسجيل انبعاث Cul1المجلس الطبي الأسترالي أكثر من 60 ثانية. ثم تأخذ رصاصة واحدة.
- كرر الخطوة 6.2.4 2 x.
- تشغيل صمام عينة لمنصب شغل . ب المحاقن الفارغة وتغسل بالحنق المخزن المؤقت.
- قياس لاحظت رابطة معدل ثوابت (كobs) Cul1•Cand1 (الشكل 4 باء).
- الاحتفاظ العينة في حقنه بنفس الخطوة 6.2.1.
- توصيل حقنه 3 مل وهي محملة "حقنه ب" 100 نانومتر Cand1 فلاشفي المخزن المؤقت للحنق. تشغيل صمام نموذج لموقف السيارة .
- استخدام لوحة التحكم تحت الحصول على البرمجيات اتخاذ خمس طلقات دون تسجيل النتائج.
- فتح لوحة التحكم تحت الحصول على البرمجيات، وبرنامج لتسجيل انبعاث Cul1المجلس الطبي الأسترالي أكثر من 60 ثانية. ثم تأخذ رصاصة واحدة.
- كرر الخطوة 6.3.4 2 x.
- ب المحاقن الفارغة وتغسل بالحنق المخزن المؤقت.
- كرر الخطوات 6.3.1–6.3.6 عدة مرات مع التركيزات المتزايدة فلاشCand1 في المخزن المؤقت للحنق.
- تناسب هذا التغيير (النقصان) في نيون إشارات يقاس على مر الزمن من كل طلقة لمنحنى أسي واحدة. وهذا سيعطي كobsفي كل قياس، والوحدة s-1. علما بأن أساس هذا الحساب قد نوقشت أيضا في تقرير سابق27.
- حساب المتوسط والانحراف المعياري ل كبسلكل تركيز Cand1 فلاشالمستخدمة. ارسم متوسط كobsضد Cand1 تركيز (الشكل 4)، ويمثل منحدر الخط في كعلى من Cul1•Cand1، مع وحدة من M s-1 -1.
7-قياس تفارق ثابت معدل (kإيقاف) من Cul1•Cand1 حضور مربع Skp1•F البروتين.
ملاحظة: هذه الخطوة مشابهة إلى الخطوة 6، مع إدخال التعديلات التالية.
- في المحاقن، ظل موقف ملء ، تحميل حلاً من 100 نانومتر Cul1المجلس الطبي الأسترالي•Rbx1 و 100 نانومتر Cand1 فلاشفي المخزن المؤقت للحنق. تشغيل صمام نموذج لموقف "محرك الأقراص".
- في "حقنه ب"، ظل موقف ملء ، تحميل حل Skp1•Skp2 (أعدت في أعقاب تقرير سابق20). تشغيل صمام نموذج لموقف "محرك الأقراص".
- فتح لوحة التحكم تحت الحصول على البرمجيات، وبرنامج لتسجيل انبعاث Cul1المجلس الطبي الأسترالي أكثر من 30 ثانية. ثم تأخذ رصاصة واحدة. زيادة إشارات مضيئة على مر الزمن بعد خلط الحلول من ألف حقنه وحقنه ب (الشكل 5).
النتائج
لاختبار الحنق بين Cul1المجلس الطبي الأسترالي و فلاشCand1، نحن أولاً بتحديد كثافة الانبعاثات من 70 نانومتر Cul1المجلس الطبي الأسترالي (الجهة المانحة) و 70 نانومتر فلاشCand1 (acceptor)، على التوالي (الشكل 3Aخطوط-ج، الأزرق). في كل تحليل، الو...
Discussion
بكى ظاهرة الفيزيائية التي ذات أهمية كبيرة لدراسة وفهم النظم البيولوجية19. نقدم هنا، على بروتوكول لاختبار واستخدام الحنق لدراسة حركية ملزمة من اثنين من البروتينات المتفاعلة. عند تصميم الحنق، يمكننا النظر في ثلاثة عوامل رئيسية: التداخل الطيفي بين المانحين الانبعاثات ويقبلون ا...
Disclosures
الكتاب ليس لها علاقة بالكشف عن.
Acknowledgements
ونحن نشكر شو-أوو "ولاية شأن" (معهد كاليفورنيا للتكنولوجيا) لمناقشة الثاقبة بشأن وضع مقايسة الحنق. الشخص، و Y.Z.، و X.L. ومولت أموال بدء التشغيل من جامعة بوردو إلى Y.Z. ودعم العمل X.L.This جزئيا بمنحه بذور من مركز جامعة بوردو "البيولوجيا النباتية".
Materials
| Name | Company | Catalog Number | Comments |
| Anion exchange chromatography column | GE Healthcare | 17505301 | HiTrap Q FF anion exchange chromatography column |
| Benchtop refrigerated centrifuge | Eppendorf | 2231000511 | |
| BL21 (DE3) Competent Cells | ThermoFisher Scientific | C600003 | |
| Calcium Chloride | Fisher Scientific | C78-500 | |
| Cation exchange chromatography column | GE Healthcare | 17505401 | HiTrap SP Sepharose FF |
| Desalting Column | GE Healthcare | 17085101 | |
| Floor model centrifuge (high speed) | Beckman Coulter | J2-MC | |
| Floor model centrifuge (low speed) | Beckman Coulter | J6-MI | |
| Fluorescence SpectraViewer | ThermoFisher Scientific | https://www.thermofisher.com/us/en/home/life-science/cell-analysis/labeling-chemistry/fluorescence-spectraviewer.html | |
| FluoroMax fluorimeter | HORIBA | FluoroMax-3 | |
| FPLC | GE Healthcare | 29018224 | |
| GGGGAMC peptide | New England Peptide | custom synthesis | |
| Glutathione beads | GE Healthcare | 17075605 | |
| Glycerol | Fisher Scientific | G33-500 | |
| HEPES | Fisher Scientific | BP310-100 | |
| Isopropyl-β-D-thiogalactoside (IPTG) | Fisher Scientific | 15-529-019 | |
| LB Broth | Fisher Scientific | BP1426-500 | |
| Ni-NTA agarose | Qiagen | 30210 | |
| Ovalbumin | MilliporeSigma | A2512 | |
| pGEX-4T-2 vector | GE Healthcare | 28954550 | |
| Protease inhibitor cocktail | MilliporeSigma | 4693132001 | |
| Reduced glutathione | Fisher Scientific | BP25211 | |
| Refrigerated shaker | Eppendorf | M1282-0004 | |
| Rosetta Competent Cells | MilliporeSigma | 70953-3 | |
| Size exclusion chromatography column | GE Healthcare | 28990944 | Superdex 200 10/300 GL column |
| Sodium Chloride (NaCl) | Fisher Scientific | S271-500 | |
| Stopped-flow fluorimeter | Hi-Tech Scientific | SF-61 DX2 | |
| TCEP·HCl | Fisher Scientific | PI20490 | |
| Thrombin | MilliporeSigma | T4648 | |
| Tris Base | Fisher Scientific | BP152-500 | |
| Ultrafiltration membrane | MilliporeSigma | UFC903008 | Amicon Ultra-15 Centrifugal Filter Units, Ultra-15, 30,000 NMWL |
References
- Stumpf, M. P. H., et al. Estimating the size of the human interactome. Proceedings of the National Academy of Sciences of the United States of America. 105 (19), 6959-6964 (2008).
- Kuzmanov, U., Emili, A. Protein-protein interaction networks: probing disease mechanisms using model systems. Genome Medicine. 5 (4), 37 (2013).
- Titeca, K., Lemmens, I., Tavernier, J., Eyckerman, S. Discovering cellular protein-protein interactions: Technological strategies and opportunities. Mass Spectrometry Reviews. , 1-33 (2018).
- Lapetina, S., Gil-Henn, H. A guide to simple, direct, and quantitative in vitro binding assays. Journal of Biological Methods. 4 (1), 62 (2017).
- Zheng, X., Bi, C., Li, Z., Podariu, M., Hage, D. S. Analytical methods for kinetic studies of biological interactions: A review. Journal of Pharmaceutical and Biomedical Analysis. 113, 163-180 (2015).
- Pierce, N. W., et al. Cand1 promotes assembly of new SCF complexes through dynamic exchange of F box proteins. Cell. 153 (1), 206-215 (2013).
- Liu, X., Reitsma, J. M., Mamrosh, J. L., Zhang, Y., Straube, R., Deshaies, R. J. Cand1-Mediated Adaptive Exchange Mechanism Enables Variation in F-Box Protein Expression. Molecular Cell. 69 (5), 773-786 (2018).
- Zheng, N., et al. Structure of the Cul1-Rbx1-Skp1-F boxSkp2 SCF ubiquitin ligase complex. Nature. 416 (6882), 703-709 (2002).
- Kleiger, G., Deshaies, R. Tag Team Ubiquitin Ligases. Cell. 166 (5), 1080-1081 (2016).
- Zheng, J., et al. CAND1 binds to unneddylated CUL1 and regulates the formation of SCF ubiquitin E3 ligase complex. Molecular Cell. 10 (6), 1519-1526 (2002).
- Hwang, J. W., Min, K. W., Tamura, T. A., Yoon, J. B. TIP120A associates with unneddylated cullin 1 and regulates its neddylation. FEBS Letters. 541 (1-3), 102-108 (2003).
- Min, K. W., Hwang, J. W., Lee, J. S., Park, Y., Tamura, T., Yoon, J. B. TIP120A associates with cullins and modulates ubiquitin ligase activity. Journal of Biological Chemistry. 278 (18), 15905-15910 (2003).
- Goldenberg, S. J., et al. Structure of the Cand1-Cul1-Roc1 complex reveals regulatory mechanisms for the assembly of the multisubunit cullin-dependent ubiquitin ligases. Cell. 119 (4), 517-528 (2004).
- Chuang, H. W., Zhang, W., Gray, W. M. Arabidopsis ETA2, an apparent ortholog of the human cullin-interacting protein CAND1, is required for auxin responses mediated by the SCF(TIR1) ubiquitin ligase. Plant Cell. 16 (7), 1883-1897 (2004).
- Feng, S., et al. Arabidopsis CAND1, an Unmodified CUL1-Interacting Protein, Is Involved in Multiple Developmental Pathways Controlled by Ubiquitin/Proteasome-Mediated Protein Degradation. the Plant Cell Online. 16 (7), 1870-1882 (2004).
- Cheng, Y., Dai, X., Zhao, Y. AtCAND1, a HEAT-repeat protein that participates in auxin signaling in Arabidopsis. Plant Physiology. 135 (June), 1020-1026 (2004).
- Lo, S. -. C., Hannink, M. CAND1-Mediated Substrate Adaptor Recycling Is Required for Efficient Repression of Nrf2 by Keap1. Molecular and Cellular Biology. 26 (4), 1235-1244 (2006).
- Okamoto, K., Sako, Y. Recent advances in FRET for the study of protein interactions and dynamics. Current Opinion in Structural Biology. 46, 16-23 (2017).
- Hussain, S. A. An introduction to fluorescence resonance energy transfer (FRET). arXiv preprint. , (2009).
- Li, T., Pavletich, N. P., Schulman, B. A., Zheng, N. High-level expression and purification of recombinant SCF ubiquitin ligases. Methods in Enzymology. 398 (1996), 125-142 (2005).
- Popp, M. W., Antos, J. M., Grotenbreg, G. M., Spooner, E., Ploegh, H. L. Sortagging: A versatile method for protein labeling. Nature Chemical Biology. 3 (11), 707-708 (2007).
- Antos, J. M., Ingram, J., Fang, T., Pishesha, N., Truttmann, M. C., Ploegh, H. L. Site-Specific Protein Labeling via Sortase-Mediated Transpeptidation. Current Protocols in Protein Science. 89, (2017).
- Froger, A., Hall, J. E. Transformation of Plasmid DNA into E. coli Using the Heat Shock Method. Journal of Visualized Experiments. , (2007).
- Simpson, R. J. SDS-PAGE of Proteins. Cold Spring Harbor Protocols. 2006 (1), (2006).
- Kleiger, G., Saha, A., Lewis, S., Kuhlman, B., Deshaies, R. J. Rapid E2-E3 Assembly and Disassembly Enable Processive Ubiquitylation of Cullin-RING Ubiquitin Ligase Substrates. Cell. 139 (5), 957-968 (2009).
- Biro, F. N., Zhai, J., Doucette, C. W., Hingorani, M. M. Application of Stopped-flow Kinetics Methods to Investigate the Mechanism of Action of a DNA Repair Protein. Journal of Visualized Experiments. (37), 2-8 (2010).
- Patel, J. T., Belsham, H. R., Rathbone, A. J., Friel, C. T. Use of Stopped-Flow Fluorescence and Labeled Nucleotides to Analyze the ATP Turnover Cycle of Kinesins. Journal of Visualized Experiments. (92), 1-6 (2014).
- Bajar, B. T., Wang, E. S., Zhang, S., Lin, M. Z., Chu, J. A guide to fluorescent protein FRET pairs. Sensors (Switzerland). 16 (9), 1-24 (2016).
- Chen, A. K., Cheng, Z., Behlke, M. A., Tsourkas, A. Assessing the sensitivity of commercially available fluorophores to the intracellular environment. Analytical Chemistry. 80 (19), 7437-7444 (2008).
- Lin, C. T., Rorabacher, D. B. Mathematical approach for stopped-flow kinetics of fast second-order reactions involving inhomogeneity in the reaction cell. Journal of Physical Chemistry. 78 (3), 305-308 (1974).
- Toseland, C. P., Geeves, M. A. Rapid Reaction Kinetic Techniques. Fluorescent Methods for Molecular Motors. , 49-65 (2014).
- Adams, S. R., et al. New biarsenical ligands and tetracysteine motifs for protein labeling in vitro and in vivo: Synthesis and biological applications. Journal of the American Chemical Society. 124 (21), 6063-6076 (2002).
- Lin, C. W., Ting, A. Y. Transglutaminase-catalyzed site-specific conjugation of small-molecule probes to proteins in vitro and on the surface of living cells. Journal of the American Chemical Society. 128 (14), 4542-4543 (2006).
- Yin, J., et al. Genetically encoded short peptide tag for versatile protein labeling by Sfp phosphopantetheinyl transferase. Proceedings of the National Academy of Sciences of the United States of America. 102 (44), 15815-15820 (2005).
- Diaspro, A., Chirico, G., Usai, C., Ramoino, P., Dobrucki, J. Photobleaching. Handbook Of Biological Confocal Microscopy. , 690-702 (2006).
- Aoki, K., Kamioka, Y., Matsuda, M. Fluorescence resonance energy transfer imaging of cell signaling from in vitro to in vivo: Basis of biosensor construction, live imaging, and image processing. Development, Growth & Differentiation. 55 (4), 515-522 (2013).
- Kilic, S., et al. Single-molecule FRET reveals multiscale chromatin dynamics modulated by HP1α. Nature Communications. 9 (1), (2018).
- Shen, K., Arslan, S., Akopian, D., Ha, T., Shan, S. O. Activated GTPase movement on an RNA scaffold drives co-translational protein targeting. Nature. 492 (7428), 271-275 (2012).
- Bajar, B. T., et al. Improving brightness and photostability of green and red fluorescent proteins for live cell imaging and FRET reporting. Scientific Reports. 6 (February), 1-12 (2016).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved