A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
خط أنابيب المعلوماتية الحيوية لتحليل بدقه وكفاءه الناسخ ميكرورنا في النباتات
* These authors contributed equally
In This Article
Summary
وهناك خط أنابيب المعلوماتية الحيوية ، وهي miRDeep-P2 (miRDP2 لفتره قصيرة) ، مع تحديث معايير ميرنا النبات وخوارزميه إصلاحها ، يمكن بدقه وكفاءه تحليل ميكرورنا النصوص في النباتات ، وخاصه بالنسبة للأنواع مع الجينوم المعقدة والكبيرة.
Abstract
ميكرورناس (miRNAs) هي من 20 إلى 24-النيوكليوتيد (nt) RNAs الصغيرة الذاتية (sRNAs) الموجودة علي نطاق واسع في النباتات والكائنات التي تلعب أدوارا قويه في تنظيم التعبير الجيني علي مستوي ما بعد النقل. وقد استخدمت علي نطاق واسع التسلسل المكتبات sRNA بواسطة أساليب تسلسل الجيل التالي (خ ع) لتحديد وتحليل المدونات ميرنا في العقد الماضي ، مما ادي إلى زيادة سريعة في اكتشاف ميرنا. ومع ذلك ، تنشا تحديين رئيسيين في التعليق التوضيحي الخاص بالنبات بسبب العمق المتزايد لمكتبات sRNA المتسلسلة وكذلك حجم وتعقيد الجينوم النباتي. أولا ، العديد من الأنواع الأخرى من sRNAs ، علي وجه الخصوص ، التداخل القصير RNAs (siRNAs) من مكتبات Srnas ، يتم بشكل خاطئ المشروح كما miRNAs بواسطة العديد من الاداات الحسابية. ثانيا ، فانه يصبح عمليه تستغرق وقتا طويلا لتحليل النسخ ميرنا في الأنواع النباتية مع الجينوم الكبيرة والمعقدة. للتغلب علي هذه التحديات ، قمنا مؤخرا بترقيه miRDeep-P (أداه شعبيه للتحليلات الناسخة ميرنا) إلى miRDeep-P2 (miRDP2 لفتره قصيرة) من خلال توظيف استراتيجية تصفيه جديده ، وإصلاح خوارزميه التهديف ودمج المصنع تحديث حديثا ميرنا معايير التعليق التوضيحي. اختبرنا miRDP2 ضد السكان المتسلسلين sRNA في خمسه مصانع تمثيليه مع زيادة تعقيد الجينوم ، بما في ذلك Arabidopsis والأرز والطماطم والذرة والقمح. وتشير النتائج إلى ان miRDP2 عالج هذه المهام بكفاءة عاليه جدا. الاضافه إلى ذلك ، تتفوق miRDP2 علي أدوات التنبؤ الأخرى فيما يتعلق بالحساسية والدقة. معا, تظهر نتائجنا miRDP2 كاداه سريعة ودقيقه لتحليل النباتات الناسخ ميرنا, التالي أداه مفيده في مساعده المجتمع أفضل التعليق miRNAs في النباتات.
Introduction
واحده من الاكتشافات الأكثر أثاره في العقدين الماضيين في علم الاحياء هو الدور المنتشر للأنواع sRNA في تنظيم وظائف متنوعة من الجينوم1. وعلي وجه الخصوص ، تشكل mirnas فئة هامه من 20 إلى 24-nt srnas في النواة ، وتعمل أساسا في مرحله ما بعد النقل كمنظمات الجينات البارزة في جميع مراحل تطوير دوره الحياة وكذلك في التحفيز والإجهاد الاستجابات2،3. في النباتات ، mirnas تنشا من النصوص الاوليه التي تسمي pri-mirnas ، والتي يتم نسخها بشكل عام من قبل الجيش النيبالي الريبي البلمره الثاني كوحدات الاستنساخ الفردية4،5. معالجتها عن طريق التطور آلات الخلوية المحفوظة (drosha rnase الثالث في الحيوانية ، مثل dicer في النباتات) ، والمؤسسة العامة-mirnas في السلائف الفورية ميرنا ، قبل mirnas ، والتي تحتوي علي متواليات تشكيل الجذعية داخل الجزيئية هياكل حلقه6،7. ثم تتم معالجه ما قبل mirnas في وسيطه مزدوجة تقطعت بهم السبل ، وهي ميرنا الدوبلكس ، تتالف من حبل وظيفية ، ميرنا ناضجه ، والشريك الوظيفي اقل في كثير من الأحيان ، ميرنا *2،8. بعد تحميلها في مجمع إسكات الناجمة عن الجيش الملكي النيبالي (risc) ، يمكن ان تعترف mirnas ناضجه أهدافهم mrna علي أساس التكامل تسلسل ، مما ادي إلى وظيفة التنظيمية السلبية2،8. mirnas يمكن اما زعزعه استقرار النسخ المستهدفة أو منع الترجمة المستهدفة ولكن الطريقة السابقة تهيمن في النباتات8,9.
منذ اكتشاف الصدفة من ميرنا الاولي في الديدان الخيطية caenorhabditis ايليتس10،11، وقد التزمت الكثير من البحوث لتحديد ميرنا وتحليلها الوظيفي ، وخاصه بعد توافر طريقه خ ع. وقد شجع التطبيق الواسع النطاق لأسلوب النظام المذكور علي استخدام الاداات الحسابية التي صممت للتقاط السمة الفريدة لmirnas ، مثل هيكل حلقه الجذعية من السلائف وتراكمها التفضيلي للتسلسل يقرا علي ميرنا ناضجه وميرنا *. ونتيجة لذلك ، حقق الباحثون نجاحا ملحوظا في تحديد miRNAs في الأنواع المختلفة. بناء علي نموذج الاحتمالات الموصوف سابقا12، قمنا بتطوير Mirdeep-P13، والذي كان أول أداه حسابيه لاكتشاف mirdeep المصنع من بيانات خ ع و. كان mirdeep-P تهدف علي وجه التحديد إلى قهر التحديات من النباتات الفك mirdeep يضم أكثر متغير طول السلائف والأسر بارالوجوس كبيره13,14,15. بعد الإفراج عنها, وقد تم تحميل هذا البرنامج آلاف المرات وتستخدم للتعليق علي المدونات ميرنا في أكثر من 40 الأنواع النباتية16. دفعت بواسطة الاداات المستندة إلى خ ع مثل miRDeep-P ، كانت هناك زيادة كبيره في عدد Mirdeep المسجلة في مستودع ميرنا العامة Mirdeep17، حيث يتم استضافتها أكثر من 38,000 ميرنا البنود حاليا (الإفراج عن 22.1) بالمقارنة فقط ~ 500 ميرنا البنود (الإفراج 2.0) في 200818.
ومع ذلك ، نشات تحديين جديدين من التعليق التوضيحي لنبات ميرنا. أولا, نسب عاليه من الإيجابيات كاذبه وقد أثرت بشكل كبير علي نوعيه النباتات ميرنا التعليقات التوضيحية16,19 للأسباب التالية: 1) وقد تم بشكل خاطئ تعليق الطوفان من rnas التداخل الذاتية الداخلية (sirnas) من مكتبات srna التابعة لشركه الطيران الخاصة. 2) بالنسبة للأنواع دون المعلومات المسبقة ميرنا ، من الصعب التخلص من الإيجابيات كاذبه استنادا إلى بيانات خ ع و. باستخدام miRBase كمثال, تايلور وآخرون20 وجدت ثلث مدخلات النبات ميرنا في مستودع العام21 (الإصدار 21) تفتقر إلى أدله داعمه مقنعه وحتى ثلاثه أرباع الأسر ميرنا النبات كانت مشكوك فيها. ثانيا ، يصبح عمليه تستغرق وقتا طويلا للتنبؤ النباتات miRNAs مع الجينوم الكبيرة والمعقدة16. للتغلب علي هذه التحديات ، قمنا بتحديث miRDeep-P عن طريق أضافه استراتيجية تصفيه جديده ، وإصلاح خوارزميه التهديف ودمج معايير جديده لشرح ميرنا النبات ، وأصدرت النسخة الجديدة miRDP2. الاضافه إلى ذلك ، اختبرنا miRDP2 باستخدام مجموعات البيانات الخاصة بالجينات التي تزيد تدريجيا من احجام الجينوم: Arabidopsis والأرز والطماطم والذرة والقمح. بالمقارنة مع الاداات الخمس الأخرى المستخدمة علي نطاق واسع والإصدار القديم ، miRDP2 تحليل هذه البيانات sRNA وتحليلها النسخ ميرنا أسرع مع تحسين الدقة والحساسية.
محتويات حزمه miRDP2
حزمه miRDP2 يتكون من سته مخطوطات بيرل الموثقة التي ينبغي تشغيلها بالتعاقب من قبل النصي باش المعدة. ومن بين النصوص الستة ، ورثت ثلاثه (convert_bowtie_to_blast، filter_alignments pl، و excise_candidate) من mirdeep-P. يتم تعديل البرامج النصية الأخرى من الإصدار الأصلي. وفيما يلي وصف لوظائف البرامج النصية الستة:
preprocess_reads. pl مرشحات الإدخال يقرا ، بما في ذلك القراءات التي هي طويلة جدا أو قصيرة جدا (< 19 nt أو > 25 nt) ، ويقرا مرتبطة مع تسلسل Rfam ncRNA ، فضلا عن يقرا مع RPM (يقرا لكل مليون) اقل من 5. البرنامج النصي ثم يسترد القراءات المرتبطة بتسلسل الناضجة ميرنا المعروفة. الملفات المدخلة هي القراءة الاصليه في شكل FASTA/FASTA و bowtie2 الإخراج من يقرا تعيين لتسلسل ميرنا و ncRNA.
صيغه لحساب RPM كما يلي:
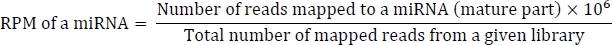
convert_bowtie_to_blast. pl يغير شكل ربطه إلى شكل تحليل الانفجار. تنسيق تحليل الانفجار هو تنسيق جدولي مفصوله مخصصه مشتقه من تنسيق الإخراج البدائية NCBI القياسية.
filter_alignments. pl مرشحات محاذاة التسلسل العميق يقرا إلى الجينوم. وهو يقوم بتصفية المحاذاة الجزئية وكذلك القراءات المتعددة المحاذاة (قطع التردد المحدد من قبل المستخدم). الإدخال الأساسي هو ملف في تنسيق تحليل الانفجار.
excise_candidate. pl يقطع تسلسل السلائف المحتملة من تسلسل مرجعي باستخدام محاذاة يقرا كمبادئ توجيهيه. الإدخال الأساسي هو ملف في تنسيق تحليل الانفجار وملف FASTA. الإخراج هو كل تسلسل السلائف المحتملة في شكل FASTA.
mod-miRDP.pl يحتاج إلى ملفين الإدخال ، ملف التوقيع وهيكل الملف ، والتي يتم تعديلها من خوارزميه Mirdeep-P الاساسيه عن طريق تغيير نظام التهديف مع معلمات محدده المصنع. الملفات المدخلة هي نقطه قوس الهيكل السلائف ويقرا ملف توقيع التوزيع.
وزاره الدفاع-rm_redundant_meet_plant يحتاج إلى ثلاثه ملفات الإدخال: الchromosome_length والسلائف والoriginal_prediction التي تنتجها mod-miRDP.pl. فانه يولد اثنين من ملفات الإخراج, غير زائده عن الحاجة الملف المتوقع والملف المتوقع تصفيتها من قبل تحديثها حديثا معايير ميرنا النبات. يتم وصف تفاصيل حول تنسيق ملف الإخراج في القسم 1.4.
Protocol
1. التركيب والاختبار
- تحميل التبعيات المطلوبة: Bowtie222 و RNAfold23. الحزم المترجمة مستحسنه.
- تحميل Bowtie2 ، أداه لتعيين القراءة ، من موقعها الرئيسي (http://bowtie-bio.sourceforge.net/bowtie2/index.shtml).
- تحميل RNAfold, أداه من حزمه فيينا تستخدم للتنبؤ الهيكل الثانوي RNA, من http://www.tbi.univie.ac.at/~ivo/RNA/.
- قبل تثبيت miRDP2 ، تاكد من تثبيت هذين التبعيتين بشكل صحيح ، وتخصيص ملف بيئة باش (علي سبيل المثال ، .bashrc) لتعيين مسار صحيح لهذين التبعيتين.
ملاحظه: أدوات الرسم الأخرى مثل Bowtie24 هي أيضا مناسبه لmiRDP2; اما Bowtie أو Bowtie2 يمكن استخدامها بعد الإصدار 1-1-3.
- لتحميل حزمه miRDP2 ، انتقل إلى https://sourceforge.net/projects/mirdp2/files/latest_version/ وجلب ملفات قطران.
- قبل تثبيت miRDP2 ، تاكد من ان بيرل في المسار. لتثبيت miRDP2 ، قم باستخراج كافة محتويات ملف قطران الذي تم تحميله إلى مجلد واحد (أسطر الأوامر كما في الشكل 1) ، ثم اضبط مسار المجلد في path.
ملاحظه: يتم المستحسن كمبيوتر أو عقده الحوسبة مع ذاكره الوصول العشوائي 8 غيغابايت علي الأقل و 100 غيغابايت التخزين لتشغيل miRDP2. - اختبار خط أنابيب MiRDP2.
- لاختبار ما إذا كان قد تم تثبيت miRDP2 بشكل صحيح ، استخدم بيانات الاختبار والإخراج المتوقع الموجود في https://sourceforge.net/projects/mirdp2/files/TestData/. تحتوي بيانات الاختبار علي ملف تسلسل GSM منسق واحد وملف جينوم واحد Arabidopsis thaliana .
- نقل كافة الملفات التي تم تنزيلها إلى دليل العمل الحالي:
mv MiRDP2-v* testdata. tar ncRNA_rfam < user_selected_folder ال> الخاصة بها
< cd user_selected_folder > - استخراج الملفات المضغوطة قطران:
tar -xvzf miRDP2-v*
tar -xvzf testdata. tar. gz
tar -xvzf ncRNA_rfam. - بناء الفهرس المرجعي لجينوم Arabidopsis:
bowtie2-البناء -f./testdata/TAIR10_genome./testdata/TAIR10_genome - إنشاء فهرس مرجع ncRNA:
bowtie2-البناء -f./ncRNA_rfam/1.1.3/script/index/rfam_index - تشغيل خط أنابيب miRDP2:
باش ./1.1.3/mirdp2-v1.1.3_pipeline-g./testdata/TAIR10_genome. fa./testdata/tair 10_genome-f./Testdata/gsm2094927.fa-o...
ملاحظه: أوامر Linux المستخدمة في خطوط غامقه ومائله ، مع خيارات سطر الأوامر بخط مائل. * يشير إلى إصدار miRDP2 (الإصدار الحالي هو 1-1-3). يجب ان يستغرق الأمر bowtie2 10 دقائق تقريبا ، ويجب ان ينتهي خط أنابيب miRDP2 خلال عده دقائق
- تحقق من اختبار المخرجات.
- لاحظ ان المجلد المسمي ' GSM2094927-0-10 ' يتم إنشاؤه تلقائيا في < user_selected_folder >، ويحتوي علي كافة الملفات الوسيطة والنتائج.
- تحقق من ان ملف الإخراج المحدد بعلامات جدوله GSM2094927-0-10_filter_P_prediction ، الإخراج النهائي لmirnas المتوقع ، يحتوي علي أعمده تشير إلى معرف الكروموسوم ، اتجاه الحبل ، الممثل يقرا المعرف ، معرف السلائف ، موقع ميرنا ناضجه ، السلائف الموقع ، تسلسل ناضجه ، وتسلسل السلائف. لاحظ ملف السرير الإضافي المشتق من هذا الملف لتسهيل المزيد من التحليل.
- تحقق من الملف "progress_log" ، الذي يوفر معلومات حول الخطوات النهائية ، والملفات "script_log" و "script_err" ، التي تحتوي علي إخراج البرنامج والتحذيرات.
ملاحظه: حاليا ، لقد اختبرنا miRDP2 علي منصات لينكس اثنين ، بما في ذلك CentOS الإصدار 6.5 علي ملقم كتله ، و Cygwin 2.6.0 علي نظام الكمبيوتر ويندوز ، و miRDP2 يجب ان تعمل علي أنظمه مماثله التي تدعم بيرل.
2. تحديد الرواية miRNAs
- قبل تشغيل خط الأنابيب ، تاكد من ان يقرا الإدخال تتم معالجته مسبقا في التنسيق المناسب.
ملاحظه: يمكن ان يقبل الإصدار الجديد 1-1-3 من miRDP2 ملفات التنسيق FASTQ الأصلي كادخالات ، علي الرغم من ان تتم عمليه القراءة التنسيق كما في الإصدارات السابقة.- أولا ، أزاله محولات من 5 ' و 3 ' ينتهي من التسلسل العميق يقرا (إذا كان موجودا).
- ثانيا ، تحليل التسلسل العميق يقرا في شكل FASTA.
- ثالثا ، أزاله التكرار مثل ان يقرا مع تسلسل متطابقة يتم تمثيل مع إدخال FASTA واحد وفريدة من نوعها.
- وأخيرا ، تاكد من ان كافة معرفات FASTA فريدة. يجب ان ينتهي كل معرف تسلسل باستخدام ' _x ' وعدد صحيح ، مع الاشاره إلى رقم النسخة الخاص بالتسلسل الدقيق الذي تم استرداده في مجموعات بيانات التسلسل العميق. أحدي الطرق لضمان معرف FASTA الفريد هو تضمين رقم قيد التشغيل في المعرف. للرجوع اليها ، راجع الملف GSM2094927 في بيانات الاختبار (https://sourceforge.net/projects/mirdp2/files/TestData/).
- راجع ما يلي للحصول علي أمثله القراءات المنسقة بشكل صحيح:
> read0_x29909
في التي
> read1_x36974
المجموعة الانجليزيه
> read2_x32635
الاتحاد الانجليزيه
- إنشاء المؤشرات المرجعية.
- للحصول علي مرجع الجينوم ، لتوفير الوقت ، قم بتنزيل ملفات فهرس Bowtie2 من موقع iGenomes (https://support.illumina.com/sequencing/sequencing_software/igenome.html) إذا تمت فهرسه تسلسلات الجينوم للأنواع ذات الفائدة. والا ، فهرس المستخدمين تسلسل مرجع والاحتفاظ ملف الفهرس لفتره من الوقت حتى يتم الانتهاء من المشروع منذ تسلسل الجينوم قد تحتاج إلى أعاده فهرسه. يتم تضمين تفاصيل حول كيفيه فهرسه مرجع الجينوم في دليل bowtie2 (http://bowtie-bio.sourceforge.net/bowtie2/manual.shtml).
- وهناك حاجه أيضا إلى مؤشر آخر غير ميرنا ncRNA لتصفيه متواليات صاخبه من غيرها من شظايا الحمض الريبي النيبالي غير الترميز. الملف هو مجموعه من متواليات ncRNA الرئيسية من Rfam ، بما في ذلك rRNA ، tRNA ، snRNA ، وشمنا. لبناء هذا الفهرس ، يرجى الرجوع إلى الجزء 1.4 ، كما يجب وضع الفهرس وتسميته بشكل صحيح ، اي < miRDP2_version >/script/index/rfam_index.
- تشغيل miRDP2.
- لاستخدام miRDP2 للكشف عن miRNAs الجديدة من بيانات التسلسل العميق ، قم بتشغيل البرنامج النصي باش في الحزمة لبدء خط أنابيب التحليل (يمكن العثور علي مثال في الخطوة 1.4):
< path_to_miRDP2_folder >/miRDP2 *. * _pipeline باش -ز < genome_file >-i < path_to_index/index_prefix >-f < seq_file >-o < output_folder >
حيث * يشير إلى إصدار خط الأنابيب باش النصي. هناك ثلاثه المعلمات التي يمكن تعديلها: 1) عدد المواقع المختلفة قراءه يمكن تعيين إلى ، 2) رقم عدم تطابق لتشغيل bowtie2 ، و 3) عتبه RPM (يقرا لكل مليون). تعديل هذه باستخدام-L ،-M ، و-R الخيارات ، علي التوالي. ويرد شرح مفصل في الفرع 3.1.
- لاستخدام miRDP2 للكشف عن miRNAs الجديدة من بيانات التسلسل العميق ، قم بتشغيل البرنامج النصي باش في الحزمة لبدء خط أنابيب التحليل (يمكن العثور علي مثال في الخطوة 1.4):
- تحقق من مخرجات miRDP2.
- لاحظ انه سيتم إنشاء مجلد الإخراج تلقائيا ضمن < output_folder > والمسمي ' < seq_file_name >-15-0-10 '; تشير الأرقام الثلاثة الاخيره إلى القيم (الافتراضية في هذه الحالة) للمعلمات 1 و 2 و 3 علي التوالي. يحتوي الملف < seq_file_name > _filter_P_prediction علي معلومات عن miRNAs النهائية المتوقعة تلبيه معايير التعليق التوضيحي ميرنا المصنع المحدثة حديثا. يتم وصف تفاصيل حول تنسيق ملف الإخراج في الجزء 1.4.
3. التعديلات والحذر باستخدام miRDP2
- المعلمات التي يمكن تعديلها
- استخدم الخيار '-L ' لتعيين الحد الأقصى لعدد المواقع التي يمكن تعيين القراءة اليها (المعلمة 1). قراءه التعيين إلى العديد من المواقع من المحتمل ان تقترن التسلسلات تكرار ، وليس من المحتمل ان miRNAs. الاعداد الافتراضي هو 15. لأنواع معينه ، إذا كانت هناك عائلات ميرنا مع العديد من الأعضاء ، يمكن زيادة المعلمة الاولي يدويا للتكيف مع المشهد الجينوم.
- استخدم الخيار '-M ' لتعيين التطابقات المسموح بها لربطه العنق (المعلمة 2). الاعداد الافتراضي هو 0.
- استخدم الخيار '-R ' لتعيين عتبه القراءة التي يحتمل ان تتطابق مع miRNAs الناضجة (المعلمة 3). لتقليل استهلاك الوقت والإيجابيات الزائفة ، يقرا الفلتر بواسطة RPM. فقط يقرا يتجاوز عتبه معينه RPM قد تمثل تسلسل ناضجه من miRNAs بدلا من الضوضاء في الخلفية ، سيتم الاحتفاظ لمزيد من التحليل. الاعداد الافتراضي هو 10 RPM.
- لاحظ ان تغيير هذه المعلمات من المحتمل ان يؤثر علي استهلاك الوقت والأداء. وبصفه عامه ، فان زيادة المعلمتين 1 و 2 وانخفاض المعلمة 3 سيولدان نتيجة اقل صرامة ووقتا أطول والعكس صحيح.
- التكرار و ميرنا *
- لاحظ ان الإخراج miRNAs من miRDP2 قد تختلف عن miRNAs المعروفة. وجدنا ان هذا يرجع أساسا إلى واحد من سببين: عدم تجانس miRNAs ناضجه أو وفره نسبي من ميرنا وميرنا *. وجدنا ان هذا لا يؤثر علي اختيار طول الأمثل من السلائف والتنميط من الجينات ميرنا المعروفة.
النتائج
يتم تطبيق خط أنابيب الشرح ميرنا ، miRDP2 ، الموصوفة هنا إلى 10 العامة srna-seq المكتبات من 5 أنواع النباتات مع زيادة تدريجيا طول الجينوم ، بما في ذلك Arabidopsis thaliana، اوريزا ساتيفا (رايس) ، سولانوم ليكوبرزيكوم (الطماطم) ، زي ميس (الذرة) و triticum استيفوم (الشكل 1ا<...
Discussion
ومع ظهور الرقم المرجعي (خ ع و) ، تم تحديد عدد كبير من الميرنا المكانية من الكمية المتزايدة باستمرار من بيانات تسلسل srna في مختلف الأنواع29و30. في قاعده بيانات المجتمع مركزيه miRBase21, وقد زادت البنود ميرنا المودعة تقريبا 100 مرات في العقد الماضي. ومع ذلك...
Disclosures
وليس لدي المؤلفين ما يفصحون عنه.
Acknowledgements
وقد تم دعم هذا العمل من قبل اكاديميه بكين للعلوم الزراعية والحراجة (KJCX201917 ، KJCX20180425 ، و KJCX20180204) إلى XY والمؤسسة الوطنية للعلوم الطبيعية في الصين (31621001) إلى ليرة لبنانية.
Materials
| Name | Company | Catalog Number | Comments |
| Computer/computing node | N/A | N/A | Perl is required; at least 8 GB RAM and 100 GB storage are recommended |
References
- Ghildiyal, M., Zamore, P. D. Small silencing RNAs: an expanding universe. Nature Reviews Genetics. 10 (2), 94-108 (2009).
- Bartel, D. P. MicroRNAs: target recognition and regulatory functions. Cell. 136 (2), 215-233 (2009).
- Moran, Y., Agron, M., Praher, D., Technau, U. The evolutionary origin of plant and animal microRNAs. Nature Ecology Evolution. 1 (3), 27 (2017).
- Xie, Z., et al. Expression of Arabidopsis MIRNA genes. Plant Physiology. 138 (4), 2145-2154 (2005).
- Zhao, X., Zhang, H., Li, L. Identification and analysis of the proximal promoters of microRNA genes in Arabidopsis. Genomics. 101 (3), 187-194 (2013).
- Bologna, N. G., Mateos, J. L., Bresso, E. G., Palatnik, J. F. A loop-to-base processing mechanism underlies the biogenesis of plant microRNAs miR319 and miR159. EMBO JOURNAL. 28 (23), 3646-3656 (2009).
- Rogers, K., Chen, X. Biogenesis, turnover, and mode of action of plant microRNAs. Plant Cell. 25 (7), 2383-2399 (2013).
- Voinnet, O. Origin, biogenesis, and activity of plant microRNAs. Cell. 136 (4), 669-687 (2009).
- Iwakawa, H. O., Tomari, Y. The Functions of MicroRNAs: mRNA Decay and Translational Repression. Trends in Cell Biology. 25 (11), 651-665 (2015).
- Lee, R. C., Feinbaum, R. L., Ambros, V. The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14. Cell. 75 (5), 843-854 (1993).
- Wightman, B., Ha, I., Ruvkun, G. Posttranscriptional regulation of the heterochronic gene lin-14 by lin-4 mediates temporal pattern formation in C. elegans. Cell. 75 (5), 855-862 (1993).
- Friedlander, M. R., et al. Discovering microRNAs from deep sequencing data using miRDeep. Nature Biotechnology. 26 (4), 407-415 (2008).
- Yang, X., Li, L. miRDeep-P: a computational tool for analyzing the microRNA transcriptome in plants. Bioinformatics. 27 (18), 2614-2615 (2011).
- Meyers, B. C., et al. Criteria for annotation of plant MicroRNAs. Plant Cell. 20 (12), 3186-3190 (2008).
- Yang, X., Zhang, H., Li, L. Global analysis of gene-level microRNA expression in Arabidopsis using deep sequencing data. Genomics. 98 (1), 40-46 (2011).
- Kuang, Z., Wang, Y., Li, L., Yang, X. miRDeep-P2: accurate and fast analysis of the microRNA transcriptome in plants. Bioinformatics. , (2018).
- Kozomara, A., Birgaoanu, M., Griffiths-Jones, S. miRBase: from microRNA sequences to function. Nucleic Acids Research. 47 (1), 155-162 (2019).
- Griffiths-Jones, S., Saini, H. K., van Dongen, S., Enright, A. J. miRBase: tools for microRNA genomics. Nucleic Acids Research. 36, 154-158 (2008).
- Axtell, M. J., Meyers, B. C. Revisiting Criteria for Plant MicroRNA Annotation in the Era of Big Data. Plant Cell. 30 (2), 272-284 (2018).
- Taylor, R. S., Tarver, J. E., Hiscock, S. J., Donoghue, P. C. Evolutionary history of plant microRNAs. Trends in Plant Science. 19 (3), 175-182 (2014).
- Kozomara, A., Griffiths-Jones, S. miRBase: annotating high confidence microRNAs using deep sequencing data. Nucleic Acids Research. 42, 68-73 (2014).
- Langmead, B., Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nature Methods. 9 (4), 357-359 (2012).
- Lorenz, R., et al. ViennaRNA Package 2.0. Algorithms for Molecular Biology. 6, 26 (2011).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biology. 10 (3), 25 (2009).
- An, J., Lai, J., Sajjanhar, A., Lehman, M. L., Nelson, C. C. miRPlant: an integrated tool for identification of plant miRNA from RNA sequencing data. BMC Bioinformatics. 15, 275 (2014).
- Lei, J., Sun, Y. miR-PREFeR: an accurate, fast and easy-to-use plant miRNA prediction tool using small RNA-Seq data. Bioinformatics. 30 (19), 2837-2839 (2014).
- Evers, M., Huttner, M., Dueck, A., Meister, G., Engelmann, J. C. miRA: adaptable novel miRNA identification in plants using small RNA sequencing data. BMC Bioinformatics. 16, 370 (2015).
- Mathelier, A., Carbone, A. MIReNA: finding microRNAs with high accuracy and no learning at genome scale and from deep sequencing data. Bioinformatics. 26 (18), 2226-2234 (2010).
- Zhu, Q. H., et al. A diverse set of microRNAs and microRNA-like small RNAs in developing rice grains. Genome Research. 18 (9), 1456-1465 (2008).
- Fahlgren, N., et al. MicroRNA gene evolution in Arabidopsis lyrata and Arabidopsis thaliana. Plant Cell. 22 (4), 1074-1089 (2010).
- Fromm, B., et al. A Uniform System for the Annotation of Vertebrate microRNA Genes and the Evolution of the Human microRNAome. Annual Review of Genetics. 49, 213-242 (2015).
- Blevins, T., et al. Identification of Pol IV and RDR2-dependent precursors of 24 nt siRNAs guiding de novo DNA methylation in Arabidopsis. Elife. 4, 09591 (2015).
- Zhai, J., et al. A One Precursor One siRNA Model for Pol IV-Dependent siRNA Biogenesis. Cell. 163 (2), 445-455 (2015).
- Werner, S., Wollmann, H., Schneeberger, K., Weigel, D. Structure determinants for accurate processing of miR172a in Arabidopsis thaliana. Current Biology. 20 (1), 42-48 (2010).
- Mateos, J. L., Bologna, N. G., Chorostecki, U., Palatnik, J. F. Identification of microRNA processing determinants by random mutagenesis of Arabidopsis MIR172a precursor. Current Biology. 20 (1), 49-54 (2010).
- Vitsios, D. M., et al. Mirnovo: genome-free prediction of microRNAs from small RNA sequencing data and single-cells using decision forests. Nucleic Acids Research. 45 (21), 177 (2017).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved