Method Article
إجراء سيميومتوميد ChIP-Seq للدراسات اللاجينية على نطاق واسع
In This Article
Erratum Notice
Summary
تصف هذه الورقة بروتوكول ChIP-Seq شبه دقيق الحجم لمناطق الحمض النووي المرتبطة بتعديل معين لهستون (H3K27ac) باستخدام منصة معالج سائل ChIP ، يليه إعداد المكتبة باستخدام العلامة. ويشمل الإجراء تقييم الرقابة لأغراض الجودة والكمية ويمكن اعتماده لتعديلات الهستون الأخرى أو عوامل النسخ.
Abstract
إن التكفير المناعي للكروماتين يليه التسلسل (ChIP-Seq) هو نهج قوي ومستخدم على نطاق واسع لملف الحمض النووي الكروماتين المرتبط بتعديلات محددة لهستون ، مثل H3K27ac ، للمساعدة في تحديد عناصر الحمض النووي التنظيمية لرابطة الدول المستقلة. العملية اليدوية لإكمال ChIP-Seq كثيفة العمالة ، وصعبة من الناحية الفنية ، وغالبا ما تتطلب أعدادا كبيرة من الخلايا (>100،000 خلية). وتساعد الطريقة الموصوفة هنا على التغلب على تلك التحديات. ويرد وصف مفصل لإجراء كامل شبه دقيق H3K27ac ChIP-Seq بما في ذلك تثبيت الخلايا، وقص الكروماتين، وprecipitation، وإعداد مكتبة التسلسل، لدفعة من 48 عينة لإدخالات عدد الخلايا أقل من 100،000 خلية. تقلل المنصة شبه المستقلة من التباين التقني، وتحسن نسب الإشارة إلى الضوضاء، وتقلل بشكل كبير من المخاض. وبالتالي يمكن للنظام خفض التكاليف من خلال السماح بتقليل أحجام التفاعل ، والحد من عدد الكواشف باهظة الثمن مثل الإنزيمات والخرز المغناطيسي والأجسام المضادة والوقت العملي المطلوب. هذه التحسينات على طريقة ChIP-Seq تناسب تماما للدراسات اللاجينية على نطاق واسع من العينات السريرية مع عدد محدود من الخلايا بطريقة قابلة للاستنساخ للغاية.
Introduction
الاستخدام الواسع لمقايسات ChIP-Seq لتحديد شظايا الحمض النووي المرتبطة بتعديلات معينة لهستون يرجع جزئيا إلى قدرته على تحديد عناصر الحمض النووي التنظيمية لرابطة الدول المستقلة ، بما في ذلك القدرة النشطة ، والمروجين ، وكواتم الصوت ، والهتيروماتين ، وغيرها1،2،3،4. وقد أظهرت تحديد المناطق التنظيمية غير الترميز عبر الجينوم نظرة قيمة لفهم أفضل لتنظيم الجينات في الصحة والأمراض4. وقد استخدم العمل السابق من المختبر ChIP-Seq لإظهار أن رابطة الدول المستقلة-العناصر التنظيمية يمكن أن تلعب أدوارا هامة في أنواع الخلايا المختلفة5. وقد استخدمت معامل النسخ (TF) ChIP المقايسات لإظهار الأمراض المرتبطة خطر واحد النيوكليوتيدات تعدد الأشكال6.
استخدام ChIP-Seq مع العينات السريرية البشرية أمر صعب ، ويرجع ذلك أساسا إلى الحد من أعداد الخلايا أو عينة الأنسجة المطلوبة. ونتيجة لذلك، كان هناك جهد متضافر في هذا المجال لتحسين وmicroscale هذه التقنيات ونتيجة لذلك، ظهرت العديد من المقايسات، مثل CUT&TAG5،7،8،9،10،11،12. يستخدم هذا المقايسة نقل إلى tagment وعزل المناطق الجينومية ملزمة بأجسام مضادة محددة9. وقد تمكنت هذه التقنية من تقليل أعداد الخلايا إلى 1000s وفي بعض الحالات إلى خلية واحدة ، ومع ذلك ، فإن استخدام هذه التقنية في البحوث التحويلية والايكريرية قد أظهرت قيودا بسبب متطلبات استخدام الخلايا الحيةلهذهالطريقة 9،12. متطلبات الخلية الحية يجعل العينات السريرية من الصعب من الناحية اللوجستية للتعامل مع ويمكن إدخال آثار دفعية إذا لم تتم معالجة العينات في نفس الوقت. وقد الأمثل الآخرين تقنيات microscaled للخلايا الفورمالديهايد الثابتة، بما في ذلك تطوير ChIPmentation11،والتي يتم تكييفها هنا بطريقة عالية الإنتاجية. استخدام الخلايا الثابتة يسمح العينات ليتم تخزينها حتى جمع ومعالجة لاحقة من جميع العينات معا للحد من الآثار الدفعية.
هنا ، يوصف فحص ChIP-Seq شبه المصغر الذي يقلل من الوقت العملي التجريبي لملف تعديلات الهستون10. تسمح الطريقة شبه الذرية بإجراء فحوصات ChIP-Seq عالية الإنتاجية ، مما يسمح بمعالجة ما يصل إلى 48 عينة بالكامل وجاهزة للتسلسل في أقل من 5 أيام ، مقابل عدد قليل يصل إلى 10000 خلية لكل عينة باستخدام معالج سائل ChIP. يكمل المعالج التكفير المناعي (IP) ويغسل لاحقا بطريقة مستقلة ، مما يساعد على تقليل التباين بين العينات. وتخفض الطريقة شبه الذرية الوقت العملي بأكثر من 15 ساعة ل 48 عينة والتباين التقني، مما يتيح إجراء دراسات لاجينية واسعة النطاق بطريقة قابلة للاستنساخ وسريعة للخلايا الأولية أو المستزرعة. البروتوكول يشرح العملية من البداية إلى النهاية للحصول على جودة عالية ChIP-Seq. إذا لم تتوفر أجهزة معينة، سيظل البروتوكول موردا مفيدا لإعداد تجارب ChIP-Seq وتصويرها يدويا.
تم إجراء الفحص بثلاثة أنواع مختلفة من الخلايا المناعية البشرية الأولية وخط خلية مثقف واحد (HUT78 – ATCC: TIB-161). للوضوح ، تم تقسيم البروتوكول إلى سبعة أقسام: تثبيت الخلايا ، وقص الكروماتين عن طريق سونيكيشن ، والمناعة الآلية للكروماتين ، وإعداد المكتبة عن طريق تصنيف جزء الحمض النووي ، وتضخيم المكتبة ، وتنقية المكتبة ، تليها تحديد كمي للحمض النووي. للحصول على وصفات عازلة يرجى الرجوع إلى الجدول التكميلي 1.
Protocol
مجلس المراجعة المؤسسية التابع لمعهد لا جولا للحساسية وعلم المناعة ( LJI ) بروتوكول IRB رقم SGE-121-0714) وافقت على الدراسة. وتم تجنيد متطوعين أصحاء وتزويدهم بعينات من اللوكافيريس بعد موافقة خطية مستنيرة.
1. تثبيت الخلية
- جعل تركيز تعليق الخلية إلى 1 إلى 2 × 106 خلايا / مل مع متوسطة ثقافة الخلية كاملة في أنبوب 15 مل (<10 مل من التعليق) أو أنبوب 50 مل (10-30 مل من الخلايا). إذا <1 × 106 خلايا، استخدم 0.5 مل من الوسط في أنبوب 1.5 مل.
- دوامة بلطف تعليق الخلية، إضافة 10x الخلية تثبيت العازلة دروبوايز (1:10؛ vol:vol) وتدوير بسرعة منخفضة لمدة 10 دقيقة في درجة حرارة الغرفة (RT).
- وقف رد الفعل عن طريق دوامة بلطف وإضافة 2.5 M الجليسين في نسبة 1:20 (vol:vol). عكس الأنابيب عدة مرات واحتضان على الجليد لمدة 5 دقائق على الأقل.
- قم بتنفيذ الخطوات المتبقية عند درجة حرارة 4 درجات مئوية أو على الجليد. تدور الأنابيب في 800 × ز لمدة 5 دقائق في 4 درجة مئوية والتخلص من supernatant.
- Resuspend بيليه بلطف مع 5 مل من الجليد الباردة 1x PBS واحتضان لمدة 2 دقيقة على الجليد.
- كرر 1.4 و 1.5 مع 1 مل من الجليد البارد 1x PBS ونقل العينة إلى أنبوب 1.5 مل مبردة مسبقا (وصفت للتخزين على المدى الطويل). إذا كان ذلك ممكنا، ينصح بإعداد aliquots.
- تدور الأنابيب في 1200 س ز في 4 درجة مئوية وإزالة أكبر قدر ممكن من supernatant دون إزعاج بيليه الخلية. المفاجئة تجميد بيليه في النيتروجين السائل. يخزن عند -80 درجة مئوية.
تنبيه: خذ الحماية المناسبة عند التعامل مع النيتروجين السائل.
2. الكروماتين القص
ملاحظة: تم تحسين هذا البروتوكول لقص الكروماتين من الكريات مع 0.3 إلى 3 × 106 خلايا في أنابيب ربط منخفضة 0.65 مل.
- إزالة أنبوب عينات مع الكريات الخلية المجمدة من -80 درجة مئوية وتخزينها على الجليد الجاف لتجنب أي ذوبان بيليه قبل إضافة العازلة تحلل. هذه الخطوة حاسمة.
- إضافة 70 ميكرولتر من الطازجة، RT كامل تحلل العازلة إلى بيليه والحفاظ على RT لمدة 1 دقيقة.
- Resuspend بيليه لمدة 1 دقيقة دون إدخال أي فقاعات ومن ثم احتضان تعليق الخلية في RT لمدة 1 دقيقة قبل وضع العينة على الجليد.
- نقل بيليه resuspended في أنبوب ملزم منخفض 0.65 مل والحفاظ على الجليد.
ملاحظة: للحصول على sonication القابلة للاستنساخ، قبل الحارة سونيكاتور عن طريق تشغيله مع أنابيب فارغة فقط لدورات 3-6 قبل sonicating العينات. - ضع العينات في حامل أنبوب سونيكاتور وملء أي ثغرات مع أنابيب التوازن مليئة 70 ميكرولتر من الماء. ترك العينات في حمام الماء لمدة 1 دقيقة قبل بدء sonication.
- تنفيذ سونيكيشن لدورات س (اعتمادا على نوع الخلية) مع 16 ق على / 32 ق قبالة في الدورة الواحدة.
ملاحظة: تتطلب هذه الخطوة تجارب التحقق من الصحة لتحديد العدد الأمثل للدورات الصوتية الفعالة. - بعد كل 3 دورات، وإزالة العينات من sonicator، دوامة بلطف ونبض تدور الأنابيب قبل وضعها مرة أخرى في حامل. تأكد من عدم وجود قطرات صغيرة على السطح الخارجي للأنبوب لأن ذلك يمكن أن يسبب تكوين فقاعة.
- بعد الانتهاء من الدورات اللازمة، تدور العينات في >14،000 × ز لمدة 15 دقيقة في 4 درجة مئوية. نقل supernatant إلى جديد, تبريد مسبق, منخفضة ملزمة 0.65 مل منخفضة ملزمة أنبوب والحفاظ على الجليد.
- Decrosslink جزء صغير من العينات سونيكاتيد للتحقق من كفاءة سونيكيشن.
- نقل 1-7 ميكرولتر من supernatant (ما يعادل حوالي 250 نانوغرام من الكروماتين القص) إلى أنبوب PCR 0.2 مل وجعل حجم يصل إلى 10 ميكرولتر مع العازلة تحلل على المدى القصير في RT.
- أضف 1 ميكرولتر RNase A واحتضن لمدة 30 دقيقة عند 37 درجة مئوية عند 800 دورة في الدقيقة، ثم أضف 1 ميكرولتر بروتيناز K.
- حضانة لمدة 2 ساعة في 55 درجة مئوية مع اهتزاز في 1000 دورة في الدقيقة.
- إزالة 2 ميكرولتر من العينة decrosslinked لقياس كمي الحمض النووي باستخدام المقايسة الكمية الفلورية10 (لا ينصح مطياف الصابون والبروتين المتدهورة يمكن أن تنتج التحيز في النتائج).
- تشغيل العينة المتبقية على هلام agarose 1.2٪ لمدة 1 ساعة في 70 V. وصمة عار مع صبغة حمض النيوكليك (1:20،000) وقراءة هلام باستخدام محول الأشعة فوق البنفسجية.
- إعداد الأسهم الكروماتين aliquots للتخزين (تمييع العينة إلى 25 نانوغرام / μL في 20 ميكرولتر مع العازلة تحلل كاملة). تخزين جميع الكروماتين مقص في -80 درجة مئوية.
3. الآلي ChIP-Seq لتعديل الهستون
ملاحظة: تم تصميم هذا البروتوكول لتشغيل معالج سائل ChIP. على الرغم من أن النظام يمكن استخدام المخازن المؤقتة المخصصة، يتم توفير كافة المخازن المؤقتة مع مجموعة ChIP. شرائح ChIP مع 8 أنابيب المستخدمة في هذا القسم هي محددة لمعالج السائل ChIP.
- نقل 16 عينة aliquots مع 500 نانوغرام من الكروماتين القص في 20 ميكرولتر من -80 درجة مئوية ووضعها على الجليد لإذابة الكروماتين ببطء. مرة واحدة إذابة تماما، دوامة لفترة وجيزة والنبض تدور.
- إعداد الكروماتين
- ماصة 100 ميكرولتر من العازلة TC1 تستكمل مع مثبط 1x بروتيز و20 mM الصوديوم بوتيرات (كامل tC1 العازلة) في اثنين من شرائح ChIP 8 أنبوب.
- نقل 20 ميكرولتر من كل عينة كروماتين في أنبوب مناسب من شرائح ChIP 8 أنبوب تحتوي على 100 ميكرولتر من العازلة tC1 كاملة. غسل أنابيب الكروماتين بإضافة 80 ميكرولتر كامل tC1 العازلة إلى أنابيب الكروماتين ومن ثم نقل مرة أخرى إلى أنبوب المناسبة من شرائح ChIP 8 أنبوب لحجم النهائي من 200 ميكرولتر.
- إعداد الأجسام المضادة
- حساب حجم الأجسام المضادة بحيث 0.5 ميكروغرام من الأجسام المضادة في كل أنبوب.
حجم الأجسام المضادة = (عدد العينات x الأجسام المضادة لكل رد فعل) / تركيز الأجسام المضادة - إضافة المبلغ المحسوب من الأجسام المضادة إلى 500 ميكرولتر من المخزن المؤقت tBW1. دوامة بسرعة ونبض تدور.
- ماصة 70 ميكرولتر من tBW1 في كل من اثنين من شرائح ChIP 8 أنبوب وإضافة 30 ميكرولتر من الأجسام المضادة + tBW1 إلى كل من الأنابيب. وهذا سوف يصل إجمالي حجم في كل من الأنابيب إلى 100 ميكرولتر.
- حساب حجم الأجسام المضادة بحيث 0.5 ميكروغرام من الأجسام المضادة في كل أنبوب.
- إعداد حبة المغناطيسي
- دوامة البروتين حل حبة جيدا. ل0.5 ميكروغرام من الأجسام المضادة، ماصة 5 ميكرولتر من الخرز في مجموعة جديدة من شرائح ChIP 8 أنبوب وتدور النبض.
- املأ الصف الأخير من معالج السائل ChIP بشرائط ChIP 8-tube المسماة والفارغة.
- اتبع مواصفات برنامج ChIP-16-IPure-200D لوضع جميع الشرائط في آلة معالج السائل ChIP. إضافة المخازن المؤقتة في الموضع الصحيح ولكن استخدام tW4 بدلا من المخزن المؤقت tE1.
ملاحظة: تنظيم اليوم بحيث معالج السائل ChIP سيتم تنفيذ ChIP بين عشية وضحاها. سيتم تشغيل البرنامج لحوالي 16 ساعة ل16 عينة. هذا يمثل نهاية اليوم الأول.
4. نقل التكامل بين محولات المكتبة لإعداد المكتبة
- قم بتعيين مixer حراريا مسبقا إلى 37 درجة مئوية و500 دورة في الدقيقة. تبريد مغناطيس لشرائط أنبوب 0.2 مل على الجليد.
- للحصول على 16 عينة، قم بإعداد 440 ميكرولتر من حاجز الوسم على الجليد. ماصة 53 ميكرولتر في شريط واحد جديد 8 أنبوب والحفاظ على الجليد.
- في شريط جديد 0.2 مل 8 أنبوب، إضافة 220 ميكروغرام من العازلة TC1 الباردة والحفاظ على الجليد. يمكن للأنابيب الشريط 8 عقد هذا الحجم ومازال توج.
- قم بإزالة أنبوب الشريط "عينات IP" من آلة معالج السائل ChIP (الصف 12) وسقف الأنابيب قبل غزل النبض. التقاط الخرز باستخدام المغناطيس لشرائط 8 أنبوب لمدة 2 دقيقة وإزالة بعناية supernatant.
- نقل 25 ميكرولتر من العازلة tagmentation إلى الخرز مع قناة متعددة، وإزالة من المغناطيس، وتخلط بلطف حتى الخرز هي متجانسة (حوالي 5 مرات صعودا وهبوطا مع ماصة تعيين إلى 20 ميكرولتر).
- قبعة الأنابيب ومكان في الحرارية قبل ساخنة واحتضان لمدة 3 دقائق. زيادة الوقت سوف يقلل من كفاءة إعداد المكتبة.
- نقل الأنابيب إلى رف معدني مبرد وإضافة 100 ميكرولتر تبريد TC1 العازلة إلى كل عينة. تعيين ماصة متعددة القنوات إلى 80 ميكرولتر وخلط العينة حتى الخرز هي متجانسة، ووقف رد فعل tagmentation.
- ضع العينات مرة أخرى في معالج السائل ChIP والمضي قدما في إجراء الغسيل Washing_for_IP reacts_16_Ipure. تأكد من إجراء الغسيل مرتين مع المخزن المؤقت TC1 ومرتين مع tW4. يجب إكمال elution كما تم وضع علامة تخطيط البرنامج مع tE1 المخزن المؤقت.
- إزالة الارتباط بين الحمض النووي
- قم بإزالة شرائح ChIP 8-tube في الصف الأخير من معالج السائل ChIP وإضافة 2 ميكرولتر RNase A إلى كل عينة.
- قم بتدريج الأنابيب، وتدور النبضة، واخلط الخرز برفق مع ماصة متعددة القنوات حتى يصبح الخليط متجانسا، ثم أعيدي غطاء الأنابيب.
- احتضان العينات في thermomixer لمدة 30 دقيقة في 37 درجة مئوية و 900 دورة في الدقيقة.
- إزالة العينات من thermomixer، إضافة 2 ميكرولتر من بروتيناز K. اتبع نفس الإجراء كما 4.9.2 بعد الإضافة.
- احتضان العينات في thermomixer لمدة 4 ساعات في 55 درجة مئوية و 1250 دورة في الدقيقة ، تليها 65 درجة مئوية في 1000 دورة في الدقيقة بين عشية وضحاها.
ملاحظة: هذه هي نهاية اليوم الثاني.
5. تنقية شظايا الحمض النووي الوسمية
- تسمية ستة عشر أنابيب 1.5 مل مع عدد العينة المناسبة وإضافة 400 ميكرولتر من الحمض النووي العازلة ملزمة من مجموعة تنظيف الحمض النووي لكل منهما.
- إزالة شرائط 8 أنبوب من thermomixer ونبض تدور الشرائط لضمان الاحتفاظ بأي منتج تبخرت. وضع شرائط على مغناطيس 8 قطاع لالتقاط الخرز.
- نقل 100 ميكرولتر من الحمض النووي غير المترابط إلى كل من أنابيب 1.5 مل. أضف 100 ميكرولتر من حاجز ربط الحمض النووي إلى شرائط الأنبوبة 8 لغسل الخرز ثم نقله إلى أنبوب 1.5 مل مناسب.
- دوامة لحوالي 10 ق ونبض تدور أنابيب 1.5 مل.
- تحميل الأعمدة مع 600 ميكرولتر تحتوي على مخزن الحمض النووي ملزمة وعينة ChIP.
- تدور عينات لمدة 20 ق في 10000 س ز وإعادة تحميل العمود مع تدفق من خلال. تدور مرة أخرى مع نفس الشروط وتجاهل تدفق من خلال.
- اغسل الأعمدة مرتين باستخدام مخزن غسيل سعة 200 ميكرولتر (نفس الطرد المركزي الذي تم غسله في الخطوة السابقة) وتخلص من التدفق.
- جفف الأعمدة عن طريق الطرد المركزي لمدة دقيقتين عند 12,000 × ز.
- نقل العمود إلى أنبوب جمع 1.5 مل جديدة وإضافة 9 ميكرولتر الدافئة TE المخزن المؤقت (قبل تسخينها إلى 55 درجة مئوية) مباشرة إلى مصفوفة العمود. السماح للعمود باحتضان لمدة دقيقة واحدة قبل الطرد المركزي لمدة دقيقة واحدة عند 10000 × غرام.
- نقل 9 ميكرولتر من elute إلى مجموعة جديدة مناسبة من شرائط 8 أنبوب.
- أكمل الوضوء مرة أخرى مع 8 ميكرولتر TE المخزن المؤقت كما كان من قبل. نقل elute إلى شرائط 8 أنبوب المناسبة (المجلد النهائي 17 ميكرولتر لكل عينة) والحفاظ على الجليد.
6. تضخيم واختيار حجم العينات المنقى
- الخطوات التالية استخدام qPCR لتحديد عدد الدورات المطلوبة للتضخيم الأمثل (CtD - Ct تحديد)
- قم بإعداد مزيج CtD لكافة العينات بضرب محتويات المخزن المؤقت CtD Mix بعدد العينات.
- الاستغناء عن 3.6 ميكرولتر من مزيج CtD في لوحة qPCR وإضافة 1.4 ميكرولتر من عينات الحمض النووي tagmented (~ 10 ٪ من الحجم الإجمالي). قم بإجراء qPCR التالي: 98 درجة مئوية لمدة 3 دقائق، و72 درجة مئوية لمدة 5 دقائق، و98 درجة مئوية لمدة 30 s، و26 دورة من 98 درجة مئوية لمدة 10 s، و63 درجة مئوية لمدة 30 s، و72 درجة مئوية لمدة 30 s.
- قم بإعداد Amp Mix لجميع العينات بضرب محتويات المخزن المؤقت AMP Mix بعدد العينات. توزيع 14 ميكرولتر من الحمض النووي الوسم في آبار منفصلة من لوحة PCR، ثم إضافة 2.5 ميكرولتر من اثنين من التمهيديات مؤشر التسلسل (25 ميكرومتر) إلى كل عينة (حجم رد الفعل النهائي هو 50 ميكرولتر).
- خلط العينات بواسطة ماصة متعددة القنوات وتنفيذ برنامج التضخيم المستخدمة في CtD مع العدد المناسب من الدورات.
ملاحظة: هذه نقطة توقف جيدة حيث يمكن تخزين العينات مكبرة في -20 درجة مئوية لبضعة أسابيع. ومع ذلك، يمكن الانتهاء من تنقية في نفس اليوم. وبالنسبة ل 48 عينة، استكملت الخطوات 3-6-5 بدفعتين منفصلتين أخريين ثم تضخمت في دفعة واحدة على النحو المبين أدناه. - إجراء ما بعد التضخيم، وتحديد الحجم، وتحديد كمي للحمض النووي الوسمي كما هو موضح أدناه. ويمكن عادة أن يكتمل ذلك ب 48 عينة (يمكن إكمالها بعدد أقل من العينات حسب الرغبة).
- أضف 90 ميكرولتر من الخرز المغنطيسي (نسبة 1:1.8) إلى كل بئر، واخلطها، واسمح لها باحتضانها في RT لمدة دقيقتين.
- التقاط الخرز باستخدام مغناطيس لوحة والتخلص من supernatant. غسل الخرز 3 مرات مع 200 ميكرولتر من الإيثانول الطازج 80٪ دون تعطيل بيليه حبة.
- إزالة أي الإيثانول الزائد مع نصائح 20 ميكرولتر بعد غسل النهائي وترك الخرز لتجف لمدة 10 دقيقة أو حتى تظهر الشقوق في الكريات حبة.
- مع لوحة لا تزال على المغناطيس، إضافة 40 ميكرولتر من الماء قبل الحارة إلى كل بئر. ختم لوحة، دوامة جيدا، ولفترة وجيزة نبض تدور لوحة.
- التقاط الخرز عن طريق وضع لوحة مرة أخرى على المغناطيس ونقل 40 μL elute إلى لوحة "عينة" جديدة. يتم الآن تنقية العينات ، والخطوات التالية تثري شظايا تتراوح بين 200-1000 نقطة أساس.
- خطوة QC الاختيارية: إزالة 4 ميكرولتر من العينات ونقلها إلى لوحة مراقبة الجودة. إضافة 4 ميكرولتر المياه مرة أخرى إلى العينات. يحدد هذا النسبة المئوية للأجزاء الكبيرة.
- أضف 22 ميكرولتر من الخرز المغنطيسي (نسبة 1:0.55) إلى العينات، واخلط بعناية، واحتضنها في RT لمدة دقيقتين.
- مكان على المغناطيس لالتقاط الخرز لمدة 5 دقائق ونقل الطوابير إلى أعمدة 7-12 من لوحة "عينة". إزالة لوحة من المغناطيس وإضافة 30 ميكرولتر من الخرز (النسبة النهائية من 1:1.3). مزيج بعناية والسماح لها بالجلوس في RT لمدة 2 دقيقة.
- التقاط الخرز لمدة 5 دقائق ثم تجاهل supernatant.
- غسل جميع الخرز 3 مرات مع 200 ميكرولتر طازجة 80 ٪ الإيثانول كما هو موضح سابقا (الخطوة 6.6.2 – 6.6.3).
- مرة واحدة الكريات جافة، الحمض النووي elute مع 8 ميكرولتر قبل الاحماء TE العازلة لكل بئر، في حين لا يزال على المغناطيس.
- إزالة لوحة من المغناطيس، ختم، ودوامة جيدا. السماح للوح لاحتضان لمدة 2 دقيقة في RT، نبض تدور، ووضع لوحة مرة أخرى على المغناطيس لمدة 2 دقيقة. نقل supernatant إلى لوحة جديدة (لوحة 2).
- للحصول على أقصى قدر من الاسترداد، كرر الوضوء مع 8 ميكرولتر إضافية من العازلة TE قبل تسخينها. ضع العينات في الآبار المناسبة بحيث تحتوي كل عينة على 16 ميكرولتر من المكتبة النهائية.
ملاحظة: في نهاية هذه الخطوة يجب أن يكون هناك لوحتين (واحدة إذا لم يتم إكمال لوحة QC). سيكون لوحة مراقبة الجودة شظايا مختارة قبل الحجم واللوحة الثانية ينبغي أن يكون 48 بئرا من المكتبة النهائية (16 ميكرولتر المجموع).
7. تحديد حجم المكتبات النهائية وعينات مراقبة الجودة باستخدام مضان المقايسة
- قياس الحمض النووي الكامل باستخدام المقايسة المقايسة الفلورية أو طريقة مماثلة.
- إذا تم الانتهاء من تحديد كمي QC، حدد النسبة المئوية لفقدان العينة التي تم < 1000 نقطة أساس. يجب ألا يكون هناك أكثر من حوالي 20٪ خسارة - إذا كان هناك أكثر من ذلك ، يمكن أن تكون هناك مشكلة مع نسب الخرز المطبقة.
- تحديد حجم شظايا كل عينة، ويفضل استخدام جهاز الكهربائي الشعرية. لحساب تركيز الضرس استخدم المعادلة التالية: [تركيز المكتبة (نانوغرام/ميكرولتر) * 106]/[660 * متوسط حجم الجزء (bp)]).
ملاحظة: المكتبات جاهزة لتجميع (كميات متساوية) وتسلسلها باتباع إجراءات تسلسل الجيل التالي القياسية.
النتائج
كدليل على المفهوم ، تم الانتهاء من ChIP-Seq لستة متبرعين بشريين مع ثلاث مجموعات من أنواع الخلايا المناعية: خلايا CD4 T الساذجة (CD4) ، والخلايا الأحادية الكلاسيكية (MO) والخلايا القاتلة الطبيعية (NK) ، التي أثراها فرز FACS كما هو موضح قبل13. ويتكون الإجراء المسطر من تسعة إجراءات متميزة كما هو ممثل في الشكل 1.
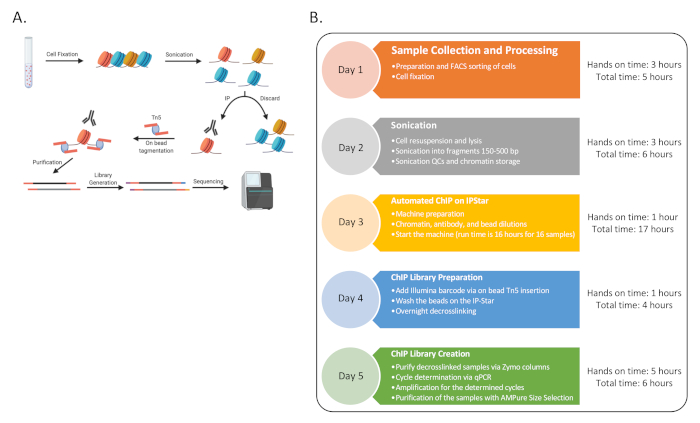
الشكل 1: مخطط انسيابي عام للإجراء. (أ) رسم كاريكاتوري للإجراء العام (تم إنشاؤه في BioRender). (ب) مخطط التدفق لجميع الخطوات الرئيسية للبروتوكول والوقت العملي المقدر وإجمالي الوقت المرتبط بكل يوم. يمكن أن يحدث التسلسل في نهاية اليوم الخامس أو في وقت لاحق مع جولات متعددة. يمكن أيضا أن يكون الجدول الزمني متداخلا على مدار الأسبوع ، حيث يمكن إكمال اليوم التسلسلي 3-4 عدة مرات في الأسبوع لتوليد 48 عينة من ChIP. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
بعد عزل الخلية عن طريق قياس التدفقالخلوي 13، تم طرد الخلايا المفرزة وإصلاح الخلايا وتخزينها على النحو المبين أعلاه. وبمجرد جمع جميع العينات، تم تحليل العينات وإعدادها للقص اللوني على دفعات من 12 كما هو موضح أعلاه. لكل عينة، تم الانتهاء من عدد من الدورات للوصول إلى سونيكيشن الأمثل10. القياس الكمي، وكذلك قياسات حجم جزء الكروماتين المقص أظهرت استنساخ كبير لطريقتنا على ثلاث مجموعات من الخلايا المناعية(الشكل 2A). تم سونيكاتيد الخلايا المناعية البشرية المختلفة على دفعات منفصلة وأسفرت بشكل متسق جدا مع > 70٪ من العينة بين 100-500 نقطة أساس لمدة 14 دورة (16 s ON، 32 s OFF لكل دورة). عند هذه النقطة، اعتبرت عينات مع شظايا كبيرة بعد سونيكيشن (< 70٪ من العينة بين 100 -500 نقطة أساس) كما فشلت. يمكن لهذه العينات إما sonicated لدورات إضافية 1-2 أو تم التخلص منها واستبدالها في وقت لاحق مع خلايا من بيليه آخر. أظهرت طريقتنا أيا من العينات المطلوبة أكثر sonication أو تم القضاء عليها، مما يشير إلى قوة مطلقة من الإجراء.
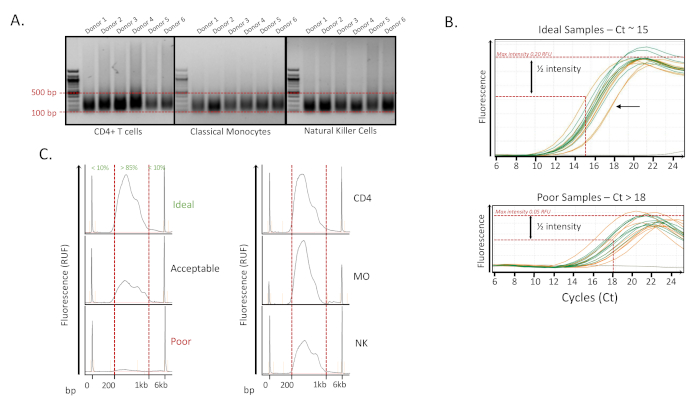
الشكل 2: أمثلة مراقبة الجودة قبل التسلسل. (A) 1.2٪ المواد الهلامية agarose تظهر استنساخ سونيكيشن. عينات Sonication ل 6 متبرعين في ثلاثة أنواع من الخلايا: خلايا CD4 T ساذجة (CD4) ، الخلايا الأحادية الكلاسيكية (MO) ، والخلايا القاتلة الطبيعية (NK). وكانت العينات sonicated لمدة 14 دورة (16 ق على، 32 ق قبالة في الدورة الواحدة). لكل عينة تم تحميل حوالي 200 نانوغرام من الكروماتين decrosslinked على هلام agarose 1٪. واعتبرت عينات جيدة إذا كان أكثر من 70 ٪ من شظايا في حدود 100-500 نقطة أساس. (ب) أعلى -- تحليل qPCR منحنيات التضخيم لتحديد العدد الأمثل للدورات للتضخيم (Ct حيث هناك 1 / 2 الحد الأقصى للكثافة). عينات مثالية لديها Ct من حوالي 15 والتضخيم يمكن أن تكتمل ما يصل إلى 2 دورة أكثر من Ct. قياس السهم هو مثال على مثال سيء حيث Ct هو أكبر من 18. أسفل - يظهر مثال على مجموعة ضعيفة من العينات التي تحتوي على Ct أكبر من 18. وأظهرت هذه العينات أيضا انخفاض كثافة الفلورسينس. (C) اليسار - أظهرت آثار محلل الشظايا الكهربائي توزيع المكتبات التي تم وضع علامات عليها النهائية بعد التضخيم واختيار الحجم. العينات مع أكثر من 85 ٪ من مكتبة جزء تقع ضمن 200-1000 نقطة أساس واعتبرت عينات جيدة. ويعتبر قياس كثافة الذروة من الفلورسينس أيضا كمعلمة مراقبة الجودة الهامة، في الواقع إذا كانت الإشارة منخفضة، العينة من غير المرجح أن تسلسل جيدا. اليمين - يتم عرض أمثلة للعينات الإيجابية في CD4 و MO و NK. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
بعد القياس الكمي، تم تشغيل العينات على معالج سائل ChIP مع الأجسام المضادة H3K27ac، تليها tagmentation مع انزيم Tn5 transposase. لتحديد العدد المناسب لدورات التضخيم بواسطة qPCR، تم استخدام 10٪ من العينات التي تم وضع علامات عليها. لتحديد عدد الدورات لتضخيم العينات، نجد الدورة التي كثافة العينة هو نصف متوسط الحد الأقصى لتحديد دورة(الشكل 2B). ولم يكن أداء العينات التي تزيد قيمها على 18 ct جيدا بعد التسلسل، وبالتالي فإن قيمتها المقطعية تدل على فشل عينة من برنامج تحسين الأداء. هذه العينات عموما أسفرت أيضا عن كمية أقل من الحمض النووي بعد التضخيم. وكانت العينات (000 100 خلية مدخلة) ذات القيمة المقطعية المتساوية أو الأقل من 15 عينة مثالية وكانت العينات بين 15 و18 عينة مقبولة ولكنها أقل اتساقا في تسلسل ما بعد الخلايا. بالنسبة للعينات التي تحتوي على أقل من 100,000 خلية إدخال، تم العثور على قيم Ct عادة بين 15 و 18 ولكنها لم تكن بحاجة إلى أكثر من 18 دورة لتسفر عن منتج كاف للتسلسل.
بعد تضخيم الحمض النووي، تم تنقية المكتبات واختيار حجمها للحصول على توزيع مثالي للحجم، يتراوح بين 200 إلى 1000 نقطة أساس، لتسلسل NextGen. تم الانتهاء من تقييم توزيع الحجم على كل مكتبة لأنه تم الحصول على أفضل بيانات التسلسل عندما تراوحت أكثر من 85٪ من شظايا الحمض النووي بين 200 إلى 1000 نقطة أساس(الشكل 2C). وتجدر الإشارة إلى أنه كما تم تحميل نفس الكمية من الحمض النووي (تقاس قياس كمي مضان) ، لوحظ أن العينات ذات كثافة الفلورية المنخفضة تسلسلها بشكل سيئ بشكل عام(الشكل 2C).
بعد التسلسل، تم تطبيق ضوابط الجودة القياسية على أساس المبادئ التوجيهية ENCODE ChIP-Seq5،14،15.
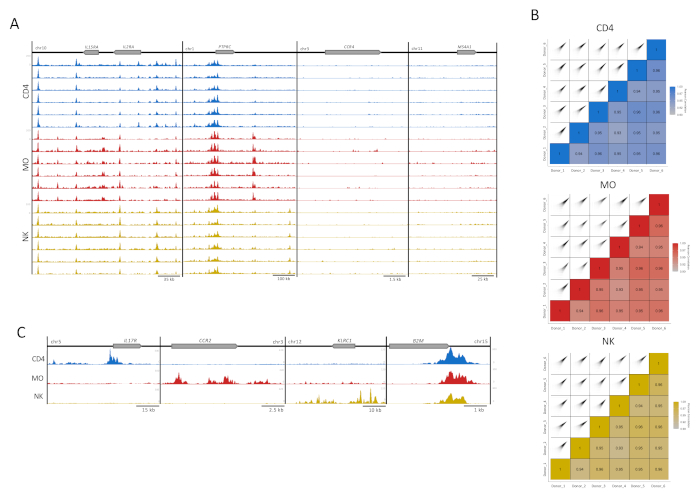
الشكل 3: استنساخ عينات الخلايا المناعية. (أ) مسارات H3K27ac (UCSC Genome Browser، الحد الأقصى للكثافة، وظيفة التنعيم 4، كل ذلك مع محور ص بالتساوي) ل 6 متبرعين (100,000 خلية لكل تكرار) في كل نوع خلية (CD4 و MO و NK). وتظهر أربعة الجراد المثالي، اثنان مع (IL2RA مكان وPTPRC) واثنين من دون تخصيب لH3K27ac (CCR4 و MS4A1). (ب) بيرسون الارتباط بين الجهات المانحة والمؤامرات الارتباط المقابلة المتولدة باستخدام تمديد 300 نقطة أساس ونافذة 500 نقطة أساس داخل حزمة MEDIPS لكل من نوع الخلية يكرر16. (C) دمج ملفات المانحين لكل نوع الخلية التي تظهر المسارات H3K27ac (UCSC الجينوم متصفح أقصى كثافة، وظيفة تنعيم 4) في مناطق نوع الخلية (IL17R لCD4، CCR2 لمو، وKLRC1 لNK) وجين حفظ المنزل B2M، موجودة في جميع أنواع الخلايا. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
لمراقبة الجودة البصرية، تم إعداد مسارات تخصيب H3K27ac للعرض في متصفح الجينوم UCSC. بالنسبة لأربعة مسارات جينية، أظهرت المسارات الفردية لكل عينة جودة رسم خرائط عالية ونسبة إشارة إلى ضوضاء تعكس الاتساق العالي وقوة المقايسة لدينا(الشكل 3A). لا يتم التعبير عن اثنين من loci إلى الميناء الأيسر الجينات المعبر عنها بشكل جيد في هذه الأنواع من الخلايا، في حين أن الجينات في loci اثنين إلى اليمين وخدم كعناصر التحكم الخلفية13 (الشكل 3A). وعلاوة على ذلك، تم استخدام حزمة تحليل MEDIPS كمتغير ما بعد التسلسل لتقييم مؤشر الارتباط بين التكرارات التقنية(الشكل 3B)5،16،17، وتحديد درجة الارتباط لمستوى إثراء القراءات ل 500 سلة bp16. بالنسبة لغالبية المقارنات الثنائية، أظهرت مؤشرات ارتباطات بيرسون أكثر من 90٪ ارتباط يشير إلى مستوى عال من الاتساق بين التكرارات البيولوجية(الشكل 3B). تم دمج التكرارات ذات الارتباط المقبول لزيادة نسبة الإشارة إلى الضوضاء. في حين أظهرت الخلية نوع محدد loci إثراء عالية في الخلايا المناسبة، وأظهرت جين حفظ المنزل (B2M) تعديل الهستون متسقة جدا(الشكل 3C). بالنسبة للتحليل ، فإن دمج المسارات من التكرارات سيزيد من الإثراء ، ويعزز الإشارة المحددة ، بما في ذلك القدرة الهامة الخاصة بنوع الخلية ، ويقلل من التباين بين الأفراد المتأصل في العينات البشرية5.
على الرغم من استخدام 100,000 خلية لهذه الدراسة، إلا أنه كان هناك قابلية عالية للاستنساخ لعدد قليل يصل إلى 10,000 خلية في خط الخلية التائية المثقف البشري (HUT78). أظهر تحليل الارتباط بين مجموعة بيانات ChIP-Seq التي أجريت من عينات تحتوي على أقل من 100,000 خلية قابلية عالية للاستنساخ والارتباط إلى 10,000 خلية(الشكل 4A).
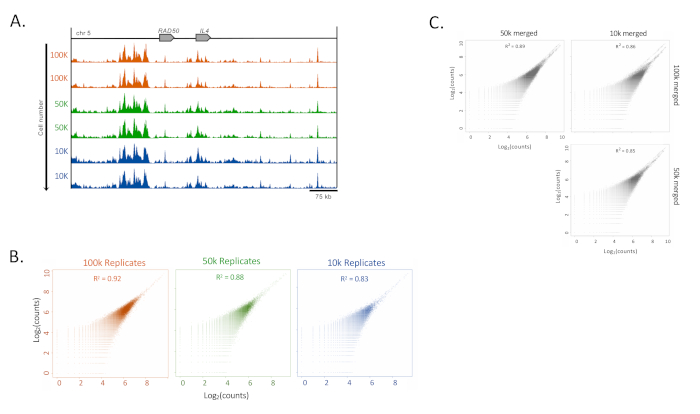
الشكل 4: استنساخ عينات المدخلات المنخفضة. (أ) أمثلة على اتساق H3K27ac ChIP-Seq للخلايا 100،000 وصولا الى 10،000 في خلايا HUT-78 (خط الخلية الليمفاوية تي الخلية). المسارات (UCSC متصفح الجينوم، والحد الأقصى للكثافة، ووظيفة تنعيم 4، مع كل تحجيم على قدم المساواة Y-محور) تظهر الموضع IL4. (ب) بيرسون الارتباطات من replicates باستخدام تمديد 300 نقطة أساس ونافذة 500 نقطة أساس داخل حزمة MEDIPS16. (C) Pearson الارتباطات بين مجموعات أرقام الخلايا المختلفة (100,000, 50,000, و 10,000 خلايا) باستخدام نفس معلمات MEDIPS كما هو الحال في (B)16. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
أظهر تحليل ارتباط بيرسون مؤشر ارتباط مرتفع (83٪ إلى 92٪)، مما يشير إلى الحفاظ على الإشارة في عينات عدد الخلايا المنخفضة. ومع ذلك، كانت هناك خلفية متزايدة كما تم تخفيض أعداد الخلايا وكذلك إسقاط معاملات الارتباط(الشكل 4B). للحفاظ على إشارات خلفية منخفضة، تم دمج التكرارات التقنية، وتم اختبار الارتباط بين المجموعات(الشكل 4C).
| 10X الخلية تثبيت المخزن المؤقت | |
| مركب | التركيز النهائي |
| حل الفورمالديهايد | 11% |
| ناكل | 100 مليون متر |
| EDTA، pH 8.0 | 1 مليون متر |
| EGTA، مؤشر الحموضة 8.0 | 0.5 مليون متر |
| HEPES، pH 7.5 | 50 مليون متر |
| إكمال مخزن تحلل المؤقت | |
| مركب | التركيز النهائي |
| تريس-HCI، pH 8.0 | 50 مليون متر |
| EDTA، pH 8.0 | 10 مليون متر |
| الحزب الديمقراطي الصربي | 0.25% |
| الصوديوم بوتيرات | 20 مليون متر |
| كوكتيل مثبطات بروتياز | 1X |
| مخزن Lysis المؤقت قصير الأجل | |
| مركب | التركيز النهائي |
| تريس-HCI، pH 8.0 | 50 مليون متر |
| EDTA، pH 8.0 | 10 مليون متر |
| الحزب الديمقراطي الصربي | 0.25% |
| مزيج العلامة | |
| مركب | التركيز النهائي |
| تريس-HCI، pH 8.0 | 10 مليون متر |
| MgCl2 | 5 مليون متر |
| N,N-ثنائي ميثيلفورمايد | 10% |
| إنزيم إيلامينا للتوسم | 1:24 فول:vol |
| CtD ميكس | |
| مركب | لكل عينة (ميكرولتر) |
| مؤشر نكستيرا التمهيدي A (25 ميكرومتر) | 0.275 |
| مؤشر نكستيرا التمهيدي B (25 ميكرومتر) | 0.275 |
| 2X كابا HiFi HotStart جاهزة ميكس | 2.75 |
| 1:1000 SYBR صبغة خضراء | 0.11 |
| ROX صبغ السلبي | 0.11 |
| الماء | تعبئة إلى 4 ميكرولتر |
| AMP ميكس | |
| مركب | لكل عينة (ميكرولتر) |
| 2X كابا HiFi HotStart جاهزة ميكس | 27.5 |
| الماء | تعبئة إلى 31 ميكرولتر |
الجدول التكميلي 1: وصفات العازلة.
الجدول التكميلي 2: سبيرمان وبيرسون عينة الارتباطات للمتبرعين 6 وكل نوع الخلية. الرجاء الضغط هنا لتحميل هذا الجدول.
Discussion
الطريقة الموصوفة هنا تتوسع في إجراء ChIPmentation11، الذي ينفذ بروتوكول إعداد مكتبة الوسم قبل تنقية الحمض النووي ، عن طريق أتمتة البروتوكول وmicroscaling. منذ بداية ChIP-Seq ، تم تخفيض أعداد الخلايا المطلوبة بشكل كبير ، من حوالي 20 مليون خلية لهيستون إلى المئات وحتى الخلايا المفردة1و7و10و 12و18و19و20و21. وقد سمحت هذه الأساليب المطورة حديثا لفهم أعمق لكيفية عمل الآليات التنظيمية لرابطة الدول المستقلةفي الخلايا من خلال زيادة الحساسية والسماح لتجمعات الخلايا السريرية النادرة ليتم اختبارها5و6و12و17. على سبيل المثال، واحدة من الإجراءات الأكثر حداثة وشعبية، ودعا CUT &؛ TAG، كما ChIP-Seq قوية وحساسة البديل9. وتنتج نسبة ممتازة إشارة إلى الضوضاء كما يرتبط الإنزيم Tn5 بشكل متناقض إلى البروتين A ويعترف سلسلة FC من الأجسام المضادة ChIP مع خصوصية عالية9. يتم تقليل النشاط الخلفية من Tn5-انزيم كما انزيم ليست وظيفية قبل ربط للأجسام المضادة المستهدفة9. ومع ذلك ، فإن تنفيذ هذه الطريقة في سياق سريري محدود لأنه يتطلب خلايا حية غير ثابتة. أيضا، إزالة شظايا الحمض النووي من نواة هيبوتونيك يمكن أن يكون لها آثار سلبية على الكروماتين كما تتم إزالته من أثناء الفحص. الشرط اللازم للعمل مع الخلايا الطازجة والحية هو مصدر للقضايا لعينات سريرية نادرة والأفواج الكبيرة من العينات، لأن الأفواج الكبيرة يمكن أن يستغرق سنوات عديدة لجمع5. نوع آخر من الأسلوب، قطرة ChIP، يستخدم بأناقة جهاز microfluidics لتوليد tagmention قطرة على أساس قبل معالجة ChIP19. ومع ذلك، فإنه يستخدم جهاز microfluidic متخصصة للغاية، وعلى الرغم من أنه من الممكن لإكمال خلية واحدة ChIP-Seq، فإنه يقتصر أيضا على استخدام الخلايا الحية7،8،9،18،19. أحدث الأساليب التي تعتمد على ChIP-Seq مثل PLAC-Seq أو HiChIP، محاولة لفهم التفاعلات 3 الأبعاد (3D) بين قمم ChIP-Seq22،23. هذه الأساليب ثلاثية الأبعاد مثيرة لأنها تحدد التفاعلات بين رابطة الدول المستقلةأو TF بوساطة عبر الجينوم وفهم أفضل لتنظيم التعبير الجيني في أنواع الخلايا ذات الاهتمام ، في الأنسجة السليمة وفي سياق المرض.
هناك بعض الخطوات الحاسمة للنظر في البروتوكول لتكون ناجحة مثل نوعية الكروماتين سونيكاتيد وجودة الأجسام المضادة. القص كفاءة أمر بالغ الأهمية، إذا لم يتم sonicated الكروماتين بشكل جيد، وكفاءة المقايسة يقلل بشكل كبير24. سونيكيشن هو جانب من جوانب التحدي ChIP- Seq بسبب أرقام الخلايا المطلوبة. على sonicator المستخدمة في البروتوكول ، وخفضت الكفاءة بشكل كبير تحت 300،000 الخلايا. هذا هو جانب من جوانب التحدي في ChIP-Seq كما سونيكاتي تحت هذا المستوى غالبا ما تتطلب تجزئة الأنزيمية، وهو أقل حيادية. ونتيجة لذلك ، سونيكيشن هو عامل محدود رئيسي لChIP - Seq microscaled صحيح. تم اختبار منصات سونيكيشن أخرى ومجموعات متاحة تجاريا لchromatin سونيكاتي، ولكن سونيكاتور المستخدمة هنا كان النتائج الأكثر قوة واستنساخها. ميزة أخرى من sonicator هو عدم الاضطرار إلى شراء أنابيب متخصصة لتشغيل سونيكيشن، مما يقلل من التكاليف عند التعامل مع عدد كبير من العينات. ل sonication الأمثل ، أولا ، من المهم أن ما قبل الحارة sonicator كما هو موضح أعلاه. ثانيا، لlyse بيليه، فمن المستحسن أن يكون طرف ماصة لمس الجزء السفلي من الأنبوب في حين lysing لتفريق الخلايا مع المزيد من القيود المادية. ثالثا، أي تشكيل فقاعة قبل sonication يعوق قدرة العينة أن سونيكاتيد بالتساوي. إذا كان هناك أي فقاعات تشكلت أثناء التحلل ، فمن المهم إزالتها مع ماصة. هذا يمكن أن يكون تحديا دون إزالة الكثير من العينة، ولكن إذا تم الضغط على طرف طفيفة ضد فقاعة يمكن أن توضع ببطء دون فقدان الكثير من العينة. وأخيرا، عند تحديد عدد الدورات، أكمل دورة زمنية حيث تتم إزالة العينة وتنقيتها، ويتم تشغيلها على جل الآغاروز كل ثلاث دورات. تجنب أكثر من / تحت sonication من العينات وهذا يقلل من كفاءة ChIP. إذا كانت العينة تحت sonicated، شظايا كبيرة يمكن أن يكون لها تأثير سلبي على جودة ChIP-Seq24. من ناحية أخرى ، إذا كانت العينة أكثر من sonicated ، هناك خطر من الضياع epitope الهدف في هذه العملية.
جزء أساسي آخر من ChIP-Seq هو جودة الأجسام المضادة. قبل إجراء أي دراسة واسعة النطاق ، من الضروري تحسين الجسم المضاد الذي سيتم استخدامه. والهدف من ذلك هو الحصول على إشارة عالية إلى حد كبير إلى نسبة الضوضاء من المناطق المعروفة من الجينوم وآخر هو استنساخ. إذا كان الجسم المضاد هو سحب الكثير من إشارة الخلفية، قد يكون من المستحسن استخدام مدخلات أكبر أو محاولة الكثير مختلفة / المورد. وهذا سيضيف وقتا قبل البدء في تجربة واسعة النطاق، ولكنه خطوة أساسية. لاختبار الإشارة إلى الضوضاء، يوصى باستخدام qPCR مع المناطق المعروفة بأنها هدف للأجسام المضادة ومنطقة أخرى معروفة بالتغيب. وقد لوحظ تعديلات الهستون هي أكثر قوة وأسهل لتحسين من TFs.
يوفر البروتوكول المذكور أعلاه طريقة قوية لتعديل الهستون عالي الإنتاجية ChIP-Seq بطريقة شبه مستقلة ومصغرة. الأسلوب يحد من مقدار التدريب العملي على الوقت ويزيد من القابلية للاستنساخ على ChIP-Seq اليدوي. الدراسات السابقة التي أنجزت في المختبر تستخدم دليل ChIP على تكرار التقنية وحصلت على متوسط ارتباط سبيرمان من 0.505، ومع ذلك ، مع النظام شبه الموتوم ، وارتباط سبيرمان بين مختلف الجهات المانحة مع متوسط خلايا NK من 0.66(الجدول التكميلي 2). كما تم الانتهاء من ذلك مع حوالي 40٪ أقل التدريب العملي في الوقت المحدد. تم تحسين الطريقة الموضحة هنا لتعديلات الهستون (H3K27ac الموضحة هنا ، ولكن البروتوكول لا ينبغي أن يحتاج إلى أي تعديل للآخرين) وسيتطلب فقط تعديلات طفيفة ليتم تنفيذها ل TF ChIP-Seq. على الرغم من جودة الأجسام المضادة ، فإن التعديل الرئيسي سيكون لوقت سونيكيشن وربما المخازن المؤقتة المستخدمة أثناء IP. عادة، ل TF ChIP المقايسات، قد تعمل هذه الطريقة بشكل أفضل مع شظايا أطول قليلا من الكروماتين (مع مجموعة من حوالي 350-800 نقطة أساس) كما TF:مجمعات الحمض النووي هي على الأرجح أقل قدرة على الحفاظ عليها من خلال سونيكيشنصارمة 6. قد تحتاج المخازن المؤقتة أيضا إلى تغيير إلى مزيج مخصص أو مجموعات أخرى متوفرة في الصناعة ، حيث يمكن أن تتصرف TFs بشكل مختلف عن تعديلات الهستون.
وعلى الرغم من أن المعالج السائل الآلي ل ChIP قد تم اختباره لعدد قليل من الخلايا يصل إلى 10,000 خلية، إلا أنه كان هناك انخفاض ملحوظ في القابلية للاستنساخ بتركيزات الكروماتين المنخفضة. ونتيجة لهذا، لم يوص البروتوكول إلى أقل من 10،000 خلية، مع 100،000 الخلايا هي الظروف المثلى. كما تم الانتهاء من البروتوكول باستخدام مخازن ChIP الصناعية ، والتي كانت نفقات إضافية ولكنها قدمت بيانات ذات جودة أعلى. يمكن تعديل البروتوكول فيما يتعلق بشروط سونيكيشن (طالما يتم الاحتفاظ الكروماتين القص داخل نفس النطاق)، يمكن تخصيص المخازن المؤقتة لprecipitation المناعي (IP؛ قد تكون هناك حاجة إلى التحسين)، أو قد لا يتم استخدام معالج السائل ChIP. وهناك قيود على البروتوكول هو استخدام معالج السائل ChIP، والتي يمكن أن تكون استثمارا مكلفا ويمكن تشغيل 16 عينة فقط في وقت واحد. ويقتصر معالج السائل ChIP على ردود الفعل على نطاق صغير وأرقام الخلايا أكبر من مليون لا ينصح. ومع ذلك، يمكن إكمال البروتوكول بدونه، من خلال إكمال خطوات IP والغسيل يدويا. إذا تم الانتهاء من الملكية الفكرية ويغسل باليد، والوقت لإكمال الفحص سوف تزيد والقابلية للاستنساخ قد تنخفض، ولكن هذا الدليل سوف لا تزال مفيدة تشغيل تجربة عالية الجودة ChIP-Seq. وتجدر الإشارة إلى أنه يمكن تكييف معالجات سائلة أخرى لتشغيل ردود فعل ChIP شبه الفغرة.
وخلاصة الأمر أن الفوائد الرئيسية لهذا النظام هي الطبيعة العالية الإنتاجية، حيث يتم الانتهاء من خطوات الملكية الفكرية والغسيل بشكل مستقل. وعلى هذا النحو، يمكن إكمال جولات متتابعة من تجارب ChIP، مما يسمح بمعالجة ما يصل إلى 48 عينة بالكامل وجاهزة للتسلسل في غضون 5 أيام، مع وقت عملي محدود مقارنة بتجارب ChIP-Seq اليدوية. وثمة فائدة أخرى هي زيادة القابلية للاستنساخ لأن ChIP-Seq يمكن أن يكون من الصعب الحصول على نتائج قابلة للاستنساخ للغاية. وهناك طرق أخرى تتطلب إما خلايا حية، أو أنظمة معقدة للأنابيب الدقيقة، أو العمل الذي يجب إكماله يدويا. يجب تحسين هذا النظام للعينات منخفضة الإدخال (<10,000 خلية)، مما يسمح في نهاية المطاف بتفاعلات ChIP أحادية الخلية. النظام قادر أيضا على التكيف مع أحدث أساليب ChIP ، مثل PLAC - Seq وHiChIP22،23.
Disclosures
وليس لدى صاحبي البلاغ ما يكشفان عنه.
Acknowledgements
نشكر أعضاء مختبر Vijayanand على المساعدة التقنية والمناقشات البناءة والدكتور شارون سكوازو والسيد جيفري بيرجيه من Diagenode للمساعدة التقنية مع آلة معالج السائل sonicator وChIP والبروتوكولات. تم دعم هذا العمل من قبل المعاهد القومية للصحة منح AI108564، R01HL114093، S10RR027366 (BD FACSAria II)، وS10OD016262 (Illumina HiSeq 2500).
Materials
| Name | Company | Catalog Number | Comments |
| 200 µl tube strips (8 tubes/strip) + cap strips | Diagenode | C30020002 | Strip tubes for use on the IP Star; ChIP 8-tube strip |
| AMPure XP for PCR Purification | Beckman Coulter | A63880 | SPRI beads |
| Axygen 0.6 mL MaxyClear Snaplock Microcentrifuge Tube | Corning | MCT-060-C | 0.65 mL low binding tube |
| Bioruptor Pico Sonicator | Diagenode | B01060010 | Sonicator used in the lab but others can be used |
| ChIP DNA Clean & Concentrator (Capped Columns) | Zymo Research | D5205 | DNA clean-up kit |
| Dynabeads Protein A for Immunoprecipitation | ThermoFisher | 10001D | |
| EDTA (0.5 M), pH 8.0, RNase-free | ThermoFisher | AM9260G | |
| EGTA pH 8.0 | Millipore Sigma | E3889-25G | |
| Eppendorf ThermoMixer C | Eppendorf | 2231000667 | |
| Formaldehyde solution | Millipore Sigma | 252549-1L | |
| Glycine | Millipore Sigma | 50046-250G | |
| H3K27ac polyclonal antibody - Premium | Diagenode | C15410196 | |
| HEPES (1 M) pH 7.5 | ThermoFisher | 15630080 | |
| IDT for Illumina Nextera DNA Unique Dual Indexes | Illumina | 20027213 | |
| Illumina Tagment DNA Enzyme and Buffer Small Kit | Illumina | 20034197 | |
| IP-Star Compact Automated System | Diagenode | B03000002 | Automated system for ChIP-Seq studies; ChIP liquid handler |
| KAPA HiFi HotStart ReadyMix | Roche | KK2601 | PCR mix |
| Medium reagent container for SX-8G IP-Star Compact | Diagenode | C30020003 | |
| MgCl2 (magnesium chloride) (25 mM) | ThermoFisher | R0971 | |
| N,N-Dimethylformamide | Millipore Sigma | D4551-250ML | CAUTION - low flash point |
| NaCl (5 M), RNase-free | ThermoFisher | AM9760G | |
| PBS (10X), pH 7.4 | ThermoFisher | 70011044 | |
| PCR Flex-free 8-tube stripes, attached individual optical caps | USA Scientific | 1402-4700 | 8 strip tubes, 0.2 mL 8-tube strip |
| Proteinase Inhibitor Cocktail | Millipore Sigma | P8340 | |
| Proteinase K Solution (20 mg/mL), RNA grade | ThermoFisher | 25530049 | |
| PureLink RNase A (20 mg/mL) | ThermoFisher | 12091021 | |
| Quant-iT PicoGreen dsDNA Reagent | ThermoFisher | P7581 | Used in the flourescence quantification |
| QuantStudio 6 Flex Real-Time PCR System | ThermoFisher | 4485699 | qPCR |
| ROX Reference Dye | ThermoFisher | 12223012 | |
| Sodium butyrate | Millipore Sigma | 303410-100G | |
| SYBR Gold Nucleic Acid Gel Stain (10,000X Concentrate in DMSO) | ThermoFisher | S11494 | nucleic acid dye |
| SYBR Green I Nucleic Acid Gel Stain - 10,000X concentrate in DMSO | ThermoFisher | S7563 | |
| TE Buffer | ThermoFisher | 12090015 | |
| Tips (bulk) | Diagenode | C30040020 | Tips for the IP Star |
| True MicroChIP Kit | Diagenode | C01010130 | Contains all the buffers for the IP; ChIP kit |
| UltraPure 1M Tris-HCI, pH 8.0 | ThermoFisher | 15568025 | |
| UltraPure SDS Solution, 10% | ThermoFisher | 24730020 |
References
- Barski, A., et al. High-resolution profiling of histone methylations in the human genome. Cell. 129 (4), 823-837 (2007).
- Johnson, D., Mortazavi, A., Myers, R., Wold, B. Genome-Wide Mapping of in Vivo Protein-DNA Interactions. Science. 316, 1497-1502 (2007).
- Mikkelsen, T. S., et al. Genome-wide maps of chromatin state in pluripotent and lineage-committed cells. Nature. 448 (7153), 553-560 (2007).
- Furey, T. S. ChIP-seq and beyond: new and improved methodologies to detect and characterize protein-DNA interactions. Nature Reviews Genetics. 13 (12), 840-852 (2012).
- Seumois, G., et al. Epigenomic analysis of primary human T cells reveals enhancers associated with TH2 memory cell differentiation and asthma susceptibility. Nature Immunology. 15 (8), 777-788 (2014).
- Schmiedel, B. J., et al. 17q21 asthma-risk variants switch CTCF binding and regulate IL-2 production by T cells. Nature Communication. 7, 13426 (2016).
- Ai, S., et al. Profiling chromatin states using single-cell itChIP-seq. Nature Cell Biology. 21 (9), 1164-1172 (2019).
- Brind'Amour, J., et al. An ultra-low-input native ChIP-seq protocol for genome-wide profiling of rare cell populations. Nature Communication. 6, 6033 (2015).
- Kaya-Okur, H. S., et al. CUT&Tag for efficient epigenomic profiling of small samples and single cells. Nature Communication. 10 (1), 1930 (1930).
- Youhanna Jankeel, D., Cayford, J., Schmiedel, B. J., Vijayanand, P., Seumois, G. An Integrated and Semiautomated Microscaled Approach to Profile Cis-Regulatory Elements by Histone Modification ChIP-Seq for Large-Scale Epigenetic Studies. Methods Molecular Biology. 1799, 303-326 (2018).
- Schmidl, C., Rendeiro, A. F., Sheffield, N. C., Bock, C. ChIPmentation: fast, robust, low-input ChIP-seq for histones and transcription factors. Nature Methods. 12 (10), 963-965 (2015).
- Skene, P. J., Henikoff, S. An efficient targeted nuclease strategy for high-resolution mapping of DNA binding sites. Elife. 6, 21856 (2017).
- Schmiedel, B. J., et al. Impact of Genetic Polymorphisms on Human Immune Cell Gene Expression. Cell. 175 (6), 1701-1715 (2018).
- Landt, S. G., et al. ChIP-seq guidelines and practices of the ENCODE and modENCODE consortia. Genome Research. 22 (9), 1813-1831 (2012).
- Chen, L., et al. Genetic Drivers of Epigenetic and Transcriptional Variation in Human Immune Cells. Cell. 167 (5), 1398-1414 (2016).
- Lienhard, M., Grimm, C., Morkel, M., Herwig, R., Chavez, L. MEDIPS: genome-wide differential coverage analysis of sequencing data derived from DNA enrichment experiments. Bioinformatics. 30 (2), 284-286 (2014).
- Engel, I., et al. Innate-like functions of natural killer T cell subsets result from highly divergent gene programs. Nature Immunology. 17 (6), 728-739 (2016).
- Grosselin, K., et al. High-throughput single-cell ChIP-seq identifies heterogeneity of chromatin states in breast cancer. Nature Genetics. 51 (6), 1060-1066 (2019).
- Rotem, A., et al. Single-cell ChIP-seq reveals cell subpopulations defined by chromatin state. Nature Biotechnology. 33 (11), 1165-1172 (2015).
- Wang, Q., et al. CoBATCH for High-Throughput Single-Cell Epigenomic Profiling. Molecular Cell. 76 (1), 206-216 (2019).
- Consortium, E. P. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Fang, R., et al. Mapping of long-range chromatin interactions by proximity ligation-assisted ChIP-seq. Cell Research. 26 (12), 1345-1348 (2016).
- Mumbach, M. R., et al. HiChIP: efficient and sensitive analysis of protein-directed genome architecture. Nature Methods. 13 (11), 919-922 (2016).
- Diagenode. The Ultimate Guide for Chromatin Shearing Optimization with Bioruptor Standard and Plus. Diagenode. , (2012).
Erratum
Formal Correction: Erratum: A Semiautomated ChIP-Seq Procedure for Large-scale Epigenetic Studies
Posted by JoVE Editors on 9/14/2020. Citeable Link.
An erratum was issued for: A Semiautomated ChIP-Seq Procedure for Large-scale Epigenetic Studies. An author's name was updated.
The name was corrected from:
Pandurangan Vijayanad
to:
Pandurangan Vijayanand
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved