A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
التحليل الكمي لديناميكيات حافة الخلية أثناء انتشار الخلايا
* These authors contributed equally
In This Article
Summary
في هذا البروتوكول، نقدم الإجراءات التجريبية للخلية التي تنشر المقايسة التي تقوم على المجهر الحي الخلية. نحن نقدم أداة حسابية مفتوحة المصدر للتجزئة غير المتحيزة للخلايا المسماة بالفلورسنت والتحليل الكمي لديناميكيات اللاميلبوديا أثناء انتشار الخلايا.
Abstract
انتشار الخلية هو عملية ديناميكية تعلق فيها الخلية المعلقة في الوسائط على الركيزة وتسطيح نفسها من شكل مدور إلى شكل رقيق ومنتشر. بعد مرفق ركيزة الخلية، تشكل الخلية ورقة رقيقة من اللاميليبوديا المنبثقة من جسم الخلية. في اللاميلبوديا ، يبوليمر أكتين كروي (G-actin) إلى شبكة أكتين خيوط كثيفة (F-actin) تدفع ضد غشاء البلازما ، وبالتالي توفير القوى الميكانيكية اللازمة للخلية للانتشار. وتجدر الإشارة إلى أن اللاعبين الجزيئيين الذين يتحكمون في بلمرة أكتين في اللاميلبوديا ضروريون للعديد من العمليات الخلوية الأخرى ، مثل هجرة الخلايا وداء الغدد الصماء.
منذ انتشار الخلايا تشكل lamellipodia المستمر التي تمتد على محيط الخلية بأكملها والتوسع باستمرار إلى الخارج، أصبحت المقايسات انتشار الخلايا أداة فعالة لتقييم حركية نتوءات اللاميليبوديال. وعلى الرغم من وضع عدة تطبيقات تقنية للتشتيت الذي ينشر الخلية، فإن هناك نقصا في وصف مفصل لسير العمل، الذي سيشمل بروتوكولا تدريجيا وأدوات حسابية لتحليل البيانات. هنا، نقوم بوصف الإجراءات التجريبية للخلية التي تنشر المقايسة وتقديم أداة مفتوحة المصدر للتحليل الكمي وغير المتحيز لديناميكيات حافة الخلية أثناء الانتشار. عندما يقترن التلاعب الدوائي و / أو تقنيات إسكات الجينات ، وهذا البروتوكول قابلة لشاشة واسعة النطاق من اللاعبين الجزيئية التي تنظم نتوءات lamellipodial.
Introduction
نتوءات لاميليبوديال هي هياكل هيكلية خلوية بارزة تشكلت في الجزء الأمامي من الخلية المهاجرة. في lamellipodia، البلمرة من أكتين بمساعدة مجمع Arp2/3 وفورمين يخلق شبكة أكتين المتفرعة سريعة النمو التي تدفع ضد غشاء البلازما1،2. دفع القوة التي ولدتها شبكة actin يدفع جسديا الخلية إلى الأمام1،3،4،5. استنفاد Arp2/3 معقدة أو تعطيل مسارات الإشارات الضرورية لproprorusions lamellipodial غالبا ما يضعف هجرة الخلايا6، 7. علىالرغممن أن هجرة الخلايا اللاميلبوديا ناقصة كما تم الإبلاغ عن 8,9, أهمية lamellipodia في هجرة الخلايا واضح كما استنفاد هذا الهيكل النتوءي يبخل قدرة الخلية على التحرك من خلال البيئات الدقيقة البيولوجية المعقدة6,10.
عائق رئيسي لفهم تنظيم lamellipodia في الخلايا المهاجرة هو التغير الطبيعي في الحركية نتوء lamellipodial، وحجم، وشكل11،12،13،14. وعلاوة على ذلك، أظهرت الدراسات الحديثة أن lamellipodia يحمل السلوكيات النتوءية المعقدة، بما في ذلك النتوءات المتقلبة والدورية والمتسارعة14،15. بالمقارنة مع lamellipodia متغير للغاية من الخلايا المهاجرة6،16، lamellipodia شكلت خلال انتشار الخلية هي أكثر اتساقا12. منذ النشاط النتوءي للخلايا المنتشرة والمهاجرة مدفوعة بتجميعات الجزيئات الكلية المتطابقة ، والتي تشمل شبكة أكتين متفرعة ، وحزم أكتوميوسين انقباضية ، والتصاقات مصفوفة الخلايا القائمة على integrin17،18، فقد تم استخدام خلايا الانتشار على نطاق واسع كنموذج للتحقيق في تنظيم ديناميات اللاميلبوديا.
انتشار الخلية هو عملية ميكانيكية ديناميكية حيث تلتزم الخلية المعلقة أولا بركيزة من خلال التصاقات المستندة إلى integrin17و19و20 ثم تنتشر عن طريق توسيع نتوءات المستندة إلى أكتين21،22،23. خلال مرحلة الانتشار، lamellipodia المنبثقة من جسم الخلية تبرز isotropically وبإصرار مع تراجع ضئيلة أو معدومة أو المماطلة12. بروتوكولات انتشار الخلايا الأكثر استخداما هي المقايسات نقاط النهاية ، حيث يتم إصلاح خلايا الانتشار في أوقات مختلفة بعد طلاء19،24. هذه المقايسات، على الرغم من سريعة وبسيطة، محدودة في قدرتها التشخيصية للكشف عن التغيرات في الميزات الديناميكية للlamellipodia. لتحديد الآليات الجزيئية التي تتحكم في ديناميات اللاميلبوديا ، كانت مجموعة شيتز رائدة في استخدام التحليل الكمي للخلايا الحية المنتشرة وكشفت عن العديد من الخصائص الأساسية لنتوءات حافة الخلية11و12و22. وقد أظهرت هذه الدراسات أن الخلايا الحية نشر المقايسة هو تقنية قوية وقوية في صندوق أدوات مختبر بيولوجيا الخلية. وعلى الرغم من ذلك، فإن البروتوكول التفصيلي والأداة الحسابية مفتوحة المصدر لإجراء فحص للخلايا الحية المنتشرة غير متاحة حاليا لمجتمع بيولوجيا الخلايا. وتحقيقا لهذه الغاية، يحدد بروتوكولنا إجراءات تصوير خلايا الانتشار الحي ويوفر أداة تحليل تلقائي للصور. للتحقق من صحة هذه الطريقة، استخدمنا Arp2/3 تثبيط كعلاج تجريبي وأظهرت أن تثبيط وظيفة مجمع Arp2/3 لم يوقف انتشار الخلايا ولكن تسبب في انخفاض كبير في سرعة نتوء الخلية، فضلا عن استقرار نتوءات حافة الخلية، مما أدى إلى حواف الخلية خشنة. هذه البيانات تثبت أن الجمع بين التصوير بالخلايا الحية وتحليل الصور الآلي هو أداة مفيدة لتحليل ديناميات حافة الخلية وتحديد المكونات الجزيئية التي تنظم lamellipodia.
Protocol
1. البذر الخلية
ملاحظة: تم تنفيذ بروتوكول نشر الخلية الموصوف باستخدام الخلايا الليفية الجنينية الماوس (MEFs) التعبير PH-Akt-GFP (علامة الفلورسنت لبرنامج تطبيق السلام3/PI(3,4)P2). تم إنشاء خط الخلية هذا عن طريق دمج بناء تعبير عن PH-Akt-GFP (أدجين #21218) من خلال تحرير الجينات بوساطة CRISPR. ومع ذلك، يمكن أيضا استخدام علامات الفلورسنت الأخرى التي يتم التعبير عنها بشكل عابر أو متكاملة في الجينوم في هذا المقايسة. للحصول على تجزئة الصورة المثلى، نوصي باستخدام علامات الفلورسنت التي يتم توزيعها بالتساوي في السيتوبلازم، على سبيل المثال، GFP السيتوسوليك.
- ثقافة طبق 10 سم من الخلايا إلى التقاء 90٪.
- بمجرد أن تصل الخلايا إلى التقاء مناسب، ضع غطاء 22 مم × 22 مم (#1.5؛ سمك 0.17 مم) في طبق ثقافة الخلية مقاس 35 ملم. معطف coverlip مع 400 ميكرولتر من فيبروكتين التي تم تخفيفها في برنامج تلفزيوني إلى تركيز النهائي من 2.5 ميكروغرام / مل.
ملاحظة: يتم تحديد عدد الأغطية المطلوبة لإجراء الفحص بواسطة عدد الشروط التجريبية والنسخة المتماثلة التقنية. - ضع طبق 35 مم مع غطاء مغطى بالفيبروكتين في حاضنة 37 درجة مئوية و5٪ CO2 لمدة ساعة واحدة.
- إزالة الطبق مع غطاء من الحاضنة. اسبيرات فيبروكتين وغسل coverslip مع برنامج تلفزيوني عن طريق الأنابيب بلطف حول coverslip مرتين إلى ثلاث مرات.
- يستنشق وسائط ثقافة الخلية من طبق 10 سم من الخلايا ويغسل الطبق مع PBS.
- إضافة 650 ميكرولتر من 0.05٪ تريبسين-EDTA إلى طبق التقاء 90٪ من الخلايا، وإمالة الطبق لتوزيع الإنزيم بالتساوي. ضع الطبق مع التريبسين في الحاضنة لمدة دقيقة واحدة.
- إزالة الطبق مع الخلايا من الحاضنة. إضافة 10 مل من وسائط ثقافة الخلية إلى أنبوب الطرد المركزي 15 مل. إضافة بسرعة آخر 10 مل من وسائل الإعلام في الطبق لإرواء تريبسين.
- ماصة 1 مل من الخلايا المثقبة في أنبوب الطرد المركزي 15 مل من أجل تمييع الخلايا. ماصة محتويات الأنبوب صعودا وهبوطا لضمان توزيع الخلايا حتى داخل وسائل الإعلام. بالنسبة لأنواع الخلايا ذات الميل العالي إلى التجميع، يوصى بتصفية الخلايا من خلال مصفاة الخلايا (حجم شبكة 100 ميكرومتر) لتقليل حدوث تكتل الخلايا.
- من الأنبوب، ماصة 500 - 1000 ميكرولتر من الخلايا المخففة في طبق 35 ملم التي تحتوي على غطاء.
- يهز الطبق بلطف لنشر الخلايا بالتساوي. تأكد من أن الغطاء هو في ~ 10٪ التقاء (~ 50،000 الخلايا / مل) وضبط حجم الخلايا المخففة حسب الحاجة.
ملاحظة: الغرض من وجود خلايا في مثل هذا الالتقاء المنخفض هو التأكد من وجود خلايا مستقطبة 1-2 في كل مجال من مجالات الرؤية التي سيتم استخدامها لتركيز الهدف في جميع أنحاء الخلية انتشار اكتساب. - مرور 1/5 من الخلايا المتبقية في طبق 10 سم في طبق واحد 6 سم لكل حالة علاج. ضع الأطباق المقطعة وطبق 35 مم مع غطاء في الحاضنة بين عشية وضحاها.
ملاحظة: هذه ستكون الخلايا التي سيتم تحليلها لنشر ديناميكيات.
2. حضانة المخدرات واستعادة الخلايا
- إضافة 5 مل من وسائط ثقافة الخلية في كل من اثنين من أنابيب الطرد المركزي 15 مل، و 20 مل من الفينول الأحمر DMEM الحرة في كل من اثنين من أنابيب الطرد المركزي 50 مل.
ملاحظة: يجب أن يتوافق عدد أزواج الأنبوب (15 مل + 50 مل) مع عدد الحالات التجريبية. - لاختبار أهمية Arp2/3 لانتشار الخلايا، ماصة إما المثبط الدوائية من Arp2/3، CK-666، أو علاج التحكم، مثل DMSO، في كل زوج من أنابيب الطرد المركزي تصل إلى التركيز المطلوب.
- أزل الأطباق المقطعة التي 6 سم (انظر الخطوة 1.11) من الحاضنة وخص وسائل الإعلام. غسل الأطباق مع برنامج تلفزيوني دافئ.
- أضف محتويات أنابيب الطرد المركزي CK-666 أو DMSO المكملة ب 15 مل في كل طبق من الأطباق المقطعة. تسمية كل طبق مع العلاج الدوائي الصحيح ووضع الأطباق في الحاضنة لمدة ساعة واحدة.
- إزالة الأطباق من الحاضنة وتوبيخ وسائل الإعلام. غسل الأطباق مع برنامج تلفزيوني دافئ من أجل إزالة تماما جميع وسائل الإعلام الحمراء الفينول المتبقية.
- إضافة 230 ميكرولتر من 0.05٪ تريبسين-EDTA إلى كل طبق 6 سم واحتضان الخلايا لمدة دقيقة واحدة.
ملاحظة: إذا كان ذلك ممكنا، يمكن استبدال التريبسين بحاصر الالتصاق بالخلايا غير البروتيوليكية. - إزالة الأطباق من الحاضنة. لكل علاج, إضافة 5 مل من المخدرات تكملها الفينول الأحمر مجانا DMEM في أنبوب الطرد المركزي 15 مل المعينة باسم "أنبوب B". إضافة 5 مل إضافية من نفس الوسائط في الطبق ذات الصلة لإرواء التريبسين. نقل محتويات الطبق إلى أنبوب الطرد المركزي 15 مل المعينة باسم "أنبوب A".
- نقل 1 مل من الخلايا من الأنبوب A إلى الأنبوب B. كرر لكل علاج.
- ضع الأنبوبين A و B في الحاضنة لمدة 45 دقيقة للسماح للخلايا بالتعافي من التربسين. تخفيف قليلا سقف أنابيب الطرد المركزي قبل وضعها في الحاضنة للسماح ل PENETRANCE CO2.
ملاحظة: قد تختلف مدة وقت الاسترداد لأنواع الخلايا المختلفة. على الرغم من أن التعافي لمدة 45 دقيقة في تجاربنا كان له تأثير لا يذكر على صلاحية الخلية ، إلا أن بعض أنواع الخلايا قد تخضع لأنويكي عندما يتم الاحتفاظ بها في التعليق لفترة طويلة جدا. لذلك، نوصي بتحديد وقت الاسترداد الأمثل تجريبيا. يتيح وقت الاسترداد الأمثل انتشار الخلايا السريعة والمتزامنة دون وجود خلايا ميتة أو أبوبتوتية في العينة.
3. إعداد الغرفة المغناطيسية
- تأكد من تنظيف جميع أجزاء غرفة مغناطيسية 1 Well Chamlide Cell التي يمكنها استيعاب غطاء مربع 22 مم × 22 مم قبل الاستخدام.
- أزل طبق 35 مم مع غطاء الغطاء (انظر الخطوة 1.11) من الحاضنة. يستنشق وسائل الإعلام ثقافة الخلية وغسل coverslip مع برنامج تلفزيوني دافئ.
- إزالة غطاء من طبق 35 ملم باستخدام زوج من ملقط ووضع بلطف coverslip على لوحة أسفل الغرفة المغناطيسية.
- ضع طوقا السيليكون على رأس غطاء الغطاء.
ملاحظة: طوقا السيليكون وضعت بشكل غير صحيح هو السبب الأكثر شيوعا لغرفة المغناطيسي راشح. تأكد من أن طوقا تقع في المسافة البادئة من لوحة أسفل ولا ترتفع إلى ما بعد المسافة البادئة. - إرفاق الجسم الرئيسي على لوحة أسفل.
ملاحظة: القيام بهذا الجزء ببطء شديد. نصيحة جيدة هو الاستمرار على لوحة أسفل بيد واحدة في حين وضع الجسم الرئيسي على القمة. وهذا يضمن أن المغناطيس في الجسم الرئيسي لا ترفع لوحة أسفل لأعلى، والتي يمكن أن تحل محل ولقضاء على coverslip. - إضافة 1 مل من المخدرات تكملها الفينول الأحمر DMEM الحرة إلى الغرفة المغناطيسية. خذ أنسجة خالية من الوبر وداب بعناية الضميمة بين الجسم الرئيسي ولوحة أسفل من أجل التحقق من أي تسرب.
ملاحظة: إذا كان هناك تسرب، بسرعة يستنشق الوسائط ثم انتقل مرة أخرى من الخطوة 3.4. - خفض الغطاء الشفاف على الجسم الرئيسي لإحاطة الغرفة المغناطيسية.
- رش الأنسجة المختبرية أولا بالماء ومسح الجزء السفلي من الغرفة المغناطيسية (غطاء، وليس الجزء المعدني). بعد ذلك، رش نسيج مختبري ثان مع كمية صغيرة من الإيثانول 70٪ ومسح، مع الحرص على عدم كسر غطاء.
4. الحصول على صورة
- سخني حاضنة المرحلة العليا وسخان الهدف إلى 37 درجة مئوية وحددمستوى ثاني أكسيد الكربون في حاضنة المرحلة العليا إلى 5٪.
ملاحظة: إذا لم يتم توصيل حاضنة المرحلة العليا بإمدادات CO2، يجب أن تستكمل وسائط ثقافة الخلية ب 25 mM HEPES للحفاظ على رقم الحموضة الثابت 7.4. - تطبيق كمية كافية من زيت الغمر إلى 60X قبل الحارة، 1.4 N.A. هدف النفط.
ملاحظة: نستخدم هدف الغمر بالنفط 60X و 1.4 N.A. في هذا البروتوكول بسبب مجال الرؤية الكبير بشكل معقول وكفاءة جمع الضوء المتميزة. إذا كان هناك حاجة إلى مجال رؤية أكبر، يمكن استخدام هدف تكبير أقل(على سبيل المثال، 20x) طالما أن نسبة الإشارة إلى الضوضاء للصور أكبر من 2.5. - جلب كل من الغرفة المغناطيسية المكتملة وB الأنبوب (الخطوة 2.9) إلى المجهر confocal. ضع الغرفة المغناطيسية على حاضنة المسرح العلوية.
ملاحظة: ضع الغرفة المغناطيسية برفق على المسرح لتجنب خلق فقاعات في زيت الغمر. - تعيين التركيز على الخلايا الفلورية باستخدام قناة GFP. تأكد من أن حافة الخلية حادة ومحددة جيدا.
- إزالة الغطاء الشفاف للغرفة المغناطيسية وماصة 500 ميكرولتر من الأنبوب B إلى الغرفة المغناطيسية. ضع الغطاء الشفاف مرة أخرى على أعلى الغرفة المغناطيسية.
- لتحديد الخلايا المثالية لتحليل انتشار الخلايا، ابحث عن "هالات" الخلايا التي لم تعلق بعد على غطاء الغطاء ولكنها لم تعد تتدحرج. الخلايا التي هي في المراحل الأولى من مرفق coverslip هي أيضا المرشحين كبيرة، ولكن يجب أن يكون اكتساب الصورة سريعة من أجل التقاط انتشار.
- تكوين الحصول على صورة الفاصل الزمني للقناة الخضراء لتشمل أربعة مجالات للعرض، في الصورة في فترات 6 ثانية.
ملاحظة: نظرا للتغير العالي لسرعة نتوء لاميليبوديا بين أنواع الخلايا المختلفة، يجب تحديد معدل الإطار الأمثل تجريبيا. فترة التصوير من 6 ثوان المستخدمة في تجاربنا هو نقطة انطلاق جيدة لتحليل العديد من الخلايا الظهارية والنسنة. ومع ذلك، قد تتطلب الخلايا التي تنتشر بسرعة كبيرة(مثلالخلايا المناعية) معدل إطار أعلى بكثير (فاصل تصوير أقصر). يضمن معدل الإطار الأمثل لأفلام نشر الخلايا إزاحة 2-5 بكسل لحافة الخلية البارزة بين الإطارات اللاحقة. وبالنظر إلى دقة تركيب المنحنى المستخدم لتحديد هضبة انتشار الخلايا، ينبغي أن يضمن معدل الإطار الأمثل أيضا قياسات 50-100 لإزاحة حافة الخلية خلال مرحلة التوسع السريع لانتشار الخلايا. يجب تعديل عدد مجالات الرؤية اعتمادا على وقت التعريض الضوئي والمسافة بين نقاط الامتلاك وسرعة حركة المرحلة. ينصح المستخدمون بتحديد الحد الأقصى لعدد مجالات العرض التي يمكن الحصول عليها مع معدل الإطار المطلوب. - بعد تحديد مجال رؤية مناسب، احفظ إحداثيات X و Y لمرحلة المجهر. تابع تحديد ثلاثة مجالات أخرى للعرض قريبة نسبيا من بعضها البعض على غطاء. حفظ إحداثيات مرحلة المجهر لكل مجال من مجالات الرؤية المطلوبة.
ملاحظة: يوصى بشدة بتحسين مسار حركة المرحلة بين حقول العرض لتقليل أي حركة عينة غير ضرورية. يمكن تنفيذ هذا التحسين إما يدويا أو تلقائيا. حركة عينة الزائد يبطئ اقتناء وقد يسبب الخلايا لطرح خارج العرض كما أنها تنازلي. - احصل على الصور لمدة 15 دقيقة بمعدل إطار 6 ثوان واحفظ الملفات. إذا كان هناك حاجة إلى المزيد من عمليات الاستحواذ، كرر بدءا من الخطوة 4.6.
5. تحليل منطقة الخلية، ديناميات التعميم والنتوء أثناء انتشار الخلايا
- إعداد الصور لمعالجة البيانات وتحليلها
ملاحظة: يتطلب البرنامج صورة بتنسيق .tiff وحجم بكسل مثل معلمات الإدخال. ويمكن استيفاء كلا الأمرين باستخدام برنامج الاقتناء أو فيجي (في هذا البروتوكول). إذا تم استيفاء هذه المتطلبات، انتقل إلى الخطوة 5.2.- تثبيت أحدث إصدار من تطبيق فيجي (https://imagej.net/Fiji/Downloads).
- افتح صورة الفاصل الزمني باستخدام فيجي.
- نسخ حجم بكسل الصورة عن طريق تحديد خصائص > الصورة. نسخ ولصق حجم البكسل في μm إلى المفكرة / Word.
- لتحليل منطقة انتشار الخلية والتعميم، احفظ صورة الفاصل الزمني ككومة صور tiff. لا يدعم برنامج تحليل البنية المخصصة تنسيقات الملفات الخاصة. حفظ مكدس صورة tiff الخلية الفردية عن طريق تحديد ملف > حفظ كما > Tiff.
- تثبيت IDE بيثون (سبايدر) والحزم اللازمة(PySimpleGUI و tifffile)لمعالجة البيانات وتحليلها.
ملاحظة: تثبيت Python وحزم مطلوب فقط الإعداد الأولي.- سيتم تحليل الأفلام الفاصل الزمني في IDE سبايدر باستخدام سيناريو بيثون مخصص البناء. لتحميل IDE سبايدر، تحميل الموزع أناكوندا (https://www.anaconda.com/products/individual) الذي يتضمن IDE سبايدر ومعظم المكتبات اللازمة وحزم لهذا التحليل.
- تثبيت أناكوندا وإطلاق سبايدر من خلال أناكوندا نافيجيتور.
- في علامة التبويب وحدة تحكم IPython (الموجود في القسم الأيمن السفلي من Spyder) ، نسخ ولصق الأمر التالي: pip install PySimpleGUI واضغط على مفتاح الإدخال. تشغيل هذا الأمر سيتم تثبيت الحزمة المطلوبة لبدء واجهة المستخدم الرسومية (GUI).
- في وحدة التحكم نفسها، نسخ ولصق الأمر التالي: pip تثبيت tifffile واضغط على مفتاح Enter. تشغيل هذا الأمر سيتم تثبيت الحزمة المطلوبة لحفظ الصور كملفات tiff.
- تحميل جميع البرامج النصية بيثون من الملفات التكميلية أو البرامج النصية الأكثر تحديثا من GitHub في: https://github.com/ernestiu/Cell-spreading-analysis.git
- قياس مساحة الخلية وعوامل شكل الخلية أثناء انتشار الخلية
- فتح السيناريو التحليل الرئيسي "cell_spreading_GUI.py" عن طريق تحديد خيار الملف المفتوح في اللوحة العليا من سبايدر أو باستخدام الاختصار Ctrl + O.
- فتح الخلية نشر تحليل واجهة المستخدم الرسومية عن طريق تحديد "تشغيل الملف" في اللوحة العليا أو باستخدام الاختصار F5.
- انقر فوق علامة التبويب منطقة انتشار الخلية ( الشكل3A).
- حدد صورة tiff ليتم تحليلها باستخدام زر الاستعراض.
ملاحظة: يجب أن يكون الملف المحدد ملف tiff. - حدد الدليل الوجهة حيث سيتم حفظ مخرجات البيانات (مثل أقنعة الخلية والقيم).
- تحديد إعدادات إخراج البيانات:
- حفظ أقنعة: حفظ أقنعة الخلية التي تم إنشاؤها أثناء عملية التقسيم.
- تصدير البيانات: تصدير جدول بيانات Excel (.xlsx) يحتوي على كافة بيانات التحليل إلى المجلد الوجهة.
سيتم حفظ منطقة الخلية، نسبة التدوير والنسبة إلى الارتفاع لكافة الخلايا المنتشرة كجداول بيانات Excel في المجلد الوجهة. يتم حساب منطقة الخلية على النحو التالي: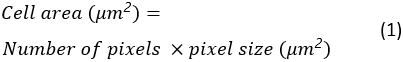
دائرية الخلية هي مقياس لمدى قرب الخلية من خلية مستديرة تماما. يتم حسابها على النحو: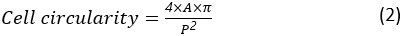
حيث A و P هي منطقة الخلية ومحيط الخلية، على التوالي. تمثل نسبة العرض إلى الارتفاع للخلية مدى استطالة الخلية. يجب أن يكون للخلية المنتشرة نسبة عرض إلى ارتفاع قريبة من 1. يتم حساب نسبة العرض إلى الارتفاع على النحو التالي: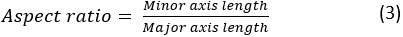
- حفظ الخطوط المحيطة: حفظ صور تراكب محيط حدود الخلية في المجلد الوجهة.
- تحديد إعدادات التقسيم
- إظهار التقسيم: إظهار نتيجة التقسيم في وحدة تحكم Spyder أثناء عملية التحليل.
- أصغر منطقة خلايا (μm2):أدخل القيمة الدنيا لمنطقة الخلية، بما في ذلك قيم المساحة للخلايا في المراحل الأولى من المرفق. لن تعتبر الكائنات ذات المساحة الأصغر من هذه العتبة كخلايا منتشرة. سيؤثر هذا الرقم على عملية التقسيم.
- حدد معلمات الصورة.
- الفاصل الزمني للاستحواذ (s): أدخل تكرار اكتساب الصورة بالثواني.
- حجم البكسل (μm): أدخل حجم البكسل الذي تم تسجيله عند إعداد الصور للتحليل.
- عمق بت الصورة: أدخل عمق بت الكاميرا/الكاشف.
- انقر فوق تشغيل. في حالة ظهور خطأ، ستظهر رسالة خطأ في وحدة تحكم Spyder. وإلا، سيتم عرض عملية تحليل الصورة في وحدة التحكم.
ملاحظة: تظهر الصورة الأولى التي تظهر في قسم وحدة التحكم/المؤامرات (اعتمادا على إعدادات Spyder) كافة الخلايا المحددة في مجال العرض. تشير المربعات الخضراء الموضوعة حول الخلايا إلى انتشار خلايا مناسبة للتجزئة والتحليل. تشير المربعات الرمادية إلى خلايا غير مناسبة للتحليل. سيظهر العدد الإجمالي للخلايا المنتشرة المحددة أيضا في علامة التبويب وحدة التحكم. يرسم البرنامج منطقة الخلية (باللون الأزرق) وتعاميم الخلية (باللون الأحمر) كدالة للوقت. تسمح هذه الرسوم البيانية للمستخدمين بتقييم دقة تجزئة الخلايا. ينتج عن التقسيم الناجح منحنى متزايد بشكل رتيب لمنطقة الخلية. للحصول على منحنى تمثيلي لمنطقة انتشار الخلية، يجب إزالة مرحلة التأخر من الرسم البياني يدويا. تتضمن مرحلة التأخر قياسات لمنطقة الخلية قبل أن تبدأ الخلية في الانتشار. يشار إلى مرحلة التأخر من خلال التقلبات السريعة، كما هو موضح في مؤامرة منطقة الخلية(الشكل 3C الحق).
6. قياس ديناميات حافة الخلية أثناء انتشار الخلية باستخدام kymographs
- قبل تشغيل التحليل، قم بقص الأفلام الخام لنشر الخلايا لإنشاء سلسلة زمنية من خلايا الانتشار الفردية.
- استخدم أداة المستطيل في شريط الأدوات فيجي لتحديد منطقة الاهتمام (ROI) التي تغلف خلية واحدةيدويا. (للتأكد من أن ROI تغليف الخلية المنتشرة بشكل كامل، استخدم دالة التمرير لفحص عائد الاستثمار في كافة النقاط الزمنية.)
- انقر بزر الماوس الأيمن على عائد الاستثمار وحدد مكرر.
- تحقق من المكدس المكرر وانقر فوق موافق.
- افتح برنامج التحليل الرئيسي "cell_spreading_GUI.py" عن طريق تحديد الزر فتح ملف في شريط أدوات Spyder أو باستخدام الاختصار Ctrl + O. إذا تم فتح واجهة المستخدم الرسومية بالفعل، انتقل مباشرة إلى الخطوة 6.3.
- فتح الخلية نشر تحليل واجهة المستخدم الرسومية عن طريق تحديد تشغيل الملف في اللوحة العليا أو باستخدام الاختصار F5 (الشكل 3B).
- انقر فوق علامة التبويب مولد كيموغراف والتحليل.
- استخدم زر الاستعراض لتحديد صورة tiff للتحليل.
ملاحظة: تنسيقات الملفات الخاصة، مثل nd2، lif، zen، غير معتمدة من قبل البرنامج النصي. - حدد المجلد الوجهة لحفظ بيانات الإخراج (أقنعة الخلية والقيم).
- حدد إعدادات الإخراج.
- تصدير البيانات: تصدير جدول بيانات excel (.xlsx) إلى المجلد الوجهة الذي يحتوي على مواضع حافة الخلية النسبية وأحداث التراجع من kymographs.
- حدد معلمات الصورة:
- الفاصل الزمني للاستحواذ (s): أدخل تكرار الحصول على الصورة بالثواني.
- حجم البكسل (μm): أدخل حجم البكسل الذي تم تسجيله عند إعداد الصور للتحليل في الخطوة 5.1.3.
- أصغر منطقة خلايا (μm2):أدخل القيمة الدنيا لمنطقة الخلية، بما في ذلك قيم المساحة للخلايا في المراحل الأولى من المرفق. لن تعتبر الكائنات ذات المساحة الأصغر من هذه العتبة كخلايا منتشرة. سيؤثر هذا الرقم على عملية التقسيم.
- عمق بت الصورة: أدخل عمق بت الكاميرا/الكاشف.
- انقر فوق تشغيل. في حالة ظهور خطأ، ستظهر رسالة خطأ في وحدة تحكم Spyder. وإلا، سيتم عرض ملخص لديناميات النتوءات في وحدة التحكم. سيكون هناك 4 أزواج من تردد التراجع وقياسات سرعة النتوء ، والتي يتم استخراجها من 4 kymographs ولدت من الأجزاء العلوية والسفلية واليسفلية والأيمنة من الخلية. يتم حساب تردد التراجع على النحو التالي:
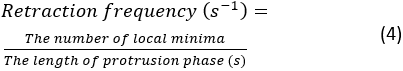
ملاحظة: يوضح هذا الرقم عدد مرات تراجع اللاميليبوديوم على مدار الانتشار. يقاس متوسط سرعة نتوء من قبل المنحدر بين بداية نتوء ونقطة الهضبة على kymograph. سيتم عرض رقم kymograph موجز في وحدة التحكم بعد التقسيم. لحفظ الشكل الموجز، انقر بزر الماوس الأيمن على الشكل واحفظ الصورة.
النتائج
يصف البروتوكول أعلاه الإجراءات التجريبية لتصوير الخلايا الحية للخلايا المنتشرة وأداة حسابية للتحليل الكمي لديناميكيات انتشار الخلايا. يمكن استخدام الأداة الحاسوبية في شكل منخفض أو عالي الإنتاجية لتحديد اللاعبين الجزيئيين الذين ينظمون آلات البلمرة actin على الحافة الرائدة للخلية.
Discussion
يسمح المقايسة المنتشرة للخلية الموصوفة بالتتبع المستمر للتغيرات المورفولوجية(على سبيل المثال ، حجم الخلية وشكلها) وحركات حافة الخلية(أي سرعة النتوء وتردد التراجع) ، وهي ميزات مفقودة في معظم بروتوكولات انتشار الخلايا19،24. في حين أن شائعة الاستخدا?...
Disclosures
وليس لدى صاحبي البلاغ ما يكشفان عنه.
Acknowledgements
تم دعم هذا العمل من خلال جائزة المحقق الجديد لصندوق كونوت ل S.P. ، المؤسسة الكندية للابتكار ، برنامج منح اكتشاف NSERC (المنح RGPIN-2015-05114 و RGPIN-2020-05881) ، صندوق الأبحاث المشتركة لجامعة مانشستر وجامعة تورنتو ، وبرنامج جامعة تورونتو XSeed.
Materials
| Name | Company | Catalog Number | Comments |
| 0.05% Trypsin (0.05%), 0.53 mM EDTA | Wisent Bioproducts | 325-042-CL | |
| 10.0 cm Petri Dish, Polystyrene, TC Treated, Vented | Starstedt | 83.3902 | |
| 15 mL High Clarity PP Centrifuge Tube, Conical Bottom, with Dome Seal Screw Cap, Sterile | Falcon | 352097 | |
| 1-Well Chamlide CMS for 22 mm x 22 mm Coverslip | Quorum Technologies | CM-S22-1 | |
| 35 mm TC-treated Easy-Grip Style Cell Culture Dish | Falcon | 353001 | |
| 50 mL Centrifuge Tube, Transparent, Plug Seal | Nest | 602002 | |
| 6.0 cm Cell Culture Dishes Treated for Increased Cell Attachment, Sterile | VWR | 10861-658 | |
| Arp2/3 Complex Inhibitor I, CK-666 | Millipore Sigma | 182515 | |
| Camera, Prime 95B-25MM | Photometrics | ||
| Dimethyl Sulfoxide, Sterile | BioShop | DMS666 | |
| DMEM, 1x, 4.5 g/L Glucose, with L-Glutamine, Sodium Pyruvate and Phenol Red | Wisent Bioproducts | 319-005 CL | |
| DMEM/F-12, HEPES, No Phenol Red | Gibco | 11039021 | |
| D-PBS, 1X | Wisent Bioproducts | 311-425 CL | |
| Fetal Bovine Serum | Wisent Bioproducts | 080-110 | |
| Fiji Software | ImageJ | ||
| HEPES (1 M) | Gibco | 15630080 | |
| Human Plasma Fibronectin Purified Protein 1 mg | Millipore Sigma | FC010 | |
| Immersion Oil | Cargille | 16241 | |
| L-Glutamine Solution (200 mM) | Wisent Bioproducts | 609-065-EL | |
| MEM Non-Essential Amino Acids Solution (100X) | Gibco | 11140050 | |
| Micro Cover Glasses, Square, No. 11/2 22 x 22 mm | VWR | CA48366-227-1 | |
| Microscope Body, Eclipse Ti2-E | Nikon | ||
| Objective, CFI Plan Apo Lambda 60X Oil | Nikon | MRD01605 | |
| Penicillin-Streptomycin | Sigma | P4333 | |
| Spinning Disk, Crest Light V2 | CrestOptics | ||
| Spyder | Anaconda | ||
| Stage top incubator | Tokai Hit | ||
| Statistics Software, Prism | GraphPad | ||
| Tweezers, Style 2 | Electron Microscopy Sciences | 78326-42 |
References
- Mullins, R. D., Heuser, J. A., Pollard, T. D. The interaction of Arp2/3 complex with actin: Nucleation, high affinity pointed end capping, and formation of branching networks of filaments. Proceedings of the National Academy of Sciences. 95 (11), 6181-6186 (1998).
- Yang, C., Czech, L., Gerboth, S., Kojima, S., Scita, G., Svitkina, T. Novel Roles of Formin mDia2 in Lamellipodia and Filopodia Formation in Motile Cells. PLoS Biology. 5 (11), 317 (2007).
- Mogilner, A., Oster, G. Cell motility driven by actin polymerization. Biophysical Journal. 71 (6), 3030-3045 (1996).
- Mogilner, A., Oster, G. Force Generation by Actin Polymerization II: The Elastic Ratchet and Tethered Filaments. Biophysical Journal. 84 (3), 1591-1605 (2003).
- Pollard, T. D., Borisy, G. G. Cellular Motility Driven by Assembly and Disassembly of Actin Filaments. Cell. 112 (4), 453-465 (2003).
- Wu, C., et al. Arp2/3 is critical for lamellipodia and response to extracellular matrix cues but is dispensable for chemotaxis. Cell. 148 (5), 973-987 (2012).
- Steffen, A., et al. Rac function is crucial for cell migration but is not required for spreading and focal adhesion formation. Journal of cell science. 126, 4572-4588 (2013).
- Gupton, S. L., et al. Cell migration without a lamellipodium. The Journal of Cell Biology. 168 (4), 619-631 (2005).
- Dimchev, V., et al. Induced Arp2/3 Complex Depletion Increases FMNL2/3 Formin Expression and Filopodia Formation. Frontiers in Cell and Developmental Biology. 9, 634708 (2021).
- Leithner, A., et al. Diversified actin protrusions promote environmental exploration but are dispensable for locomotion of leukocytes. Nature cell biology. 18 (11), 1253-1259 (2016).
- Giannone, G., Dubin-Thaler, B. J., Döbereiner, H. -. G., Kieffer, N., Bresnick, A. R., Sheetz, M. P. Periodic Lamellipodial Contractions Correlate with Rearward Actin Waves. Cell. 116 (3), 431-443 (2004).
- Dubin-Thaler, B. J., et al. Quantification of Cell Edge Velocities and Traction Forces Reveals Distinct Motility Modules during Cell Spreading. PLoS ONE. 3 (11), 3735 (2008).
- Suraneni, P., Rubinstein, B., Unruh, J. R., Durnin, M., Hanein, D., Li, R. The Arp2/3 complex is required for lamellipodia extension and directional fibroblast cell migration. The Journal of cell biology. 197 (2), 239-251 (2012).
- Wang, C., et al. Deconvolution of subcellular protrusion heterogeneity and the underlying actin regulator dynamics from live cell imaging. Nature Communications. 9 (1), 1688 (2018).
- Dimchev, G., et al. Lamellipodin tunes cell migration by stabilizing protrusions and promoting adhesion formation. Journal of cell science. 133 (7), 239020 (2020).
- Burnette, D. T., et al. A role for actin arcs in the leading-edge advance of migrating cells. Nature cell biology. 13 (4), 371-381 (2011).
- Yamada, K. M., Kennedy, D. W. Dualistic nature of adhesive protein function: fibronectin and its biologically active peptide fragments can autoinhibit fibronectin function. The Journal of Cell Biology. 99 (1), 29-36 (1984).
- Cai, Y., et al. Nonmuscle Myosin IIA-Dependent Force Inhibits Cell Spreading and Drives F-Actin Flow. Biophysical Journal. 91 (10), 3907-3920 (2006).
- Humphries, M. J. Cell adhesion assays. Molecular Biotechnology. 18 (1), 57-61 (2001).
- Cavalcanti-Adam, E. A., Volberg, T., Micoulet, A., Kessler, H., Geiger, B., Spatz, J. P. Cell Spreading and Focal Adhesion Dynamics Are Regulated by Spacing of Integrin Ligands. Biophysical Journal. 92 (8), 2964-2974 (2007).
- Dubin-Thaler, B. J., Giannone, G., Döbereiner, H. -. G., Sheetz, M. P. Nanometer Analysis of Cell Spreading on Matrix-Coated Surfaces Reveals Two Distinct Cell States and STEPs. Biophysical Journal. 86 (3), 1794-1806 (2004).
- Gauthier, N. C., Fardin, M. A., Roca-Cusachs, P., Sheetz, M. P. Temporary increase in plasma membrane tension coordinates the activation of exocytosis and contraction during cell spreading. Proceedings of the National Academy of Sciences. 108 (35), 14467-14472 (2011).
- Wolfenson, H., Iskratsch, T., Sheetz, M. P. Early Events in Cell Spreading as a Model for Quantitative Analysis of Biomechanical Events. Biophysical Journal. 107 (11), 2508-2514 (2014).
- Guan, J. -. L., Berrier, A. L., LaFlamme, S. E. Cell Migration, Developmental Methods and Protocols. Methods in molecular biology. 294, 55-68 (2004).
- Raucher, D., et al. Phosphatidylinositol 4,5-Bisphosphate Functions as a Second Messenger that Regulates Cytoskeleton-Plasma Membrane Adhesion. Cell. 100 (2), 221-228 (2000).
- Machacek, M., Danuser, G. Morphodynamic Profiling of Protrusion Phenotypes. Biophysical Journal. 90 (4), 1439-1452 (2006).
- Zack, G. W., Rogers, W. E., Latt, S. A. Automatic measurement of sister chromatid exchange frequency. The journal of histochemistry and cytochemistry official journal of the Histochemistry Society. 25 (7), 741-753 (1977).
- Bardsley, W. G., Aplin, J. D. Kinetic analysis of cell spreading. I. Theory and modelling of curves. Journal of cell science. 61, 365-373 (1983).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved