A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
فحص خلوي قائم على المراسل لمراقبة كفاءة الربط
In This Article
Summary
يصف هذا البروتوكول فحصا لمراسل minigene لمراقبة تأثير طفرات موقع 5'-splice على الربط ويطور مثبط U1 snRNA لإنقاذ تثبيط الربط الناجم عن الطفرة. يتم التعبير عن بنى المراسل والمثبط U1 snRNA في خلايا HeLa ، ويتم تحليل الربط عن طريق تمديد التمهيدي أو RT-PCR.
Abstract
أثناء التعبير الجيني ، تتضمن الخطوة الحيوية لربط ما قبل mRNA التعرف الدقيق على مواقع الربط والتجميع الفعال للمجمعات spliceosomal للانضمام إلى exons وإزالة الإنترونات قبل التصدير السيتوبلازمي للحمض النووي الريبوزي المرسال الناضج. يمكن تغيير كفاءة الربط من خلال وجود طفرات في مواقع الربط ، أو تأثير عوامل الربط المتحولة ، أو نشاط العلاجات. هنا ، نصف بروتوكول الفحص الخلوي الذي يمكن تطبيقه لمراقبة كفاءة الربط لأي إكسون معين. يستخدم الفحص مراسلا بلازميديا قابلا للتكيف مشفرا 3-exon / 2-intron minigene ، والذي يمكن التعبير عنه في خلايا الثدييات عن طريق النقل العابر. بعد النقل ، يتم عزل الحمض النووي الريبي الخلوي الكلي ، ويتم تحديد كفاءة ربط exon في mRNA المراسل إما عن طريق تمديد التمهيدي أو تفاعل سلسلة النسخ العكسي والبوليميراز شبه الكمي (RT-PCR). نحن نصف كيف يمكن تحديد تأثير الطفرات المرتبطة بالمرض في موقع الربط 5 ′ من خلال تقديمها في المراسل ؛ وكيف يمكن تحقيق قمع هذه الطفرات عن طريق النقل المشترك مع بناء الحمض النووي الريبي النووي الصغير U1 (snRNA) الذي يحمل طفرات تعويضية في منطقته 5 التي تقترن بمواقع ربط 5 عند تقاطعات exon-intron في ما قبل mRNAs. وبالتالي ، يمكن استخدام المراسل لتصميم جزيئات U1 العلاجية لتحسين التعرف على مواقع الربط المتحورة 5 ′. يمكن أيضا استخدام إدراج المواقع التنظيمية التي تعمل على رابطة الدول المستقلة ، مثل محسن الربط أو تسلسل كاتم الصوت ، في المراسل لدراسة دور U1 snRNP في التنظيم بوساطة عامل ربط بديل محدد. وأخيرا، يمكن احتضان الخلايا التي يعبر عنها المراسل بجزيئات صغيرة لتحديد تأثير العلاجات المحتملة على ربط ما قبل الحمض النووي الريبوزي المرسال التأسيسي أو على الإكسونات التي تحمل مواقع الربط المتحورة 5 ′. بشكل عام ، يمكن تطبيق فحص المراسل لمراقبة كفاءة الربط في مجموعة متنوعة من الظروف لدراسة آليات الربط الأساسية والأمراض المرتبطة بالربط.
Introduction
يعد ربط ما قبل mRNA خطوة معالجة أساسية تزيل الإنترونات غير المشفرة وتربط بدقة إكسونات الترميز لتشكيل mRNA ناضجة. إن التعرف على تسلسلات الإجماع عند تقاطعات exon-intron ، المشار إليها باسم موقع 5 ′ splice وموقع 3 ′ splice ، بواسطة مكونات آلة الربط يبدأ عملية الربط. يتعرف البروتين الريبي النووي الصغير U1 (snRNP) على موقع الربط 5 ′ عن طريق الاقتران الأساسي للحمض النووي الريبي السري U1 بالحمض النووي الريبي السري (snRNA1) قبل mRNA1. ترتبط الطفرات الموروثة وراثيا التي تغير تسلسل موقع 5′-splice بالعديد من الأمراض2,3. من المتوقع أن يؤدي فقدان الاقتران الأساسي ل U1 snRNA مع مواقع الربط 5 ′ المتحولة إلى ربط شاذ ، مما قد يعرض ترجمة النص المصاب للخطر. يتضمن النهج العلاجي المحتمل لتصحيح عيوب الربط قمع الطفرات بواسطة U1 snRNA المعدل الذي يحمل تغيرات النيوكليوتيدات التعويضية في منطقته 5 التي تتطابق مع موقع الربط 5. وقد وجد أن مثل هذه الحمض النووي الريبوزي المرسال U1 المعدل ، والذي يشار إليه أيضا باسم الحمض النووي الريبوزي المرسال U1 المحدد من exon ، فعال في عكس عيوب الربط ، مما يؤدي إلى زيادة التعبير عن البروتين من mRNA4,5,6,7,8 الذي تم إنقاذه.
هنا ، نصف اختبار تكامل U1 snRNP ، وهو فحص ربط خلوي قائم على المراسل يسمح بتقييم تأثير طفرات 5 ′ ss على ربط exon ويمكن استخدامه أيضا لتطوير U1 snRNAs المعدلة لتمكين إنقاذ تضمين exon. كما نقدم بروتوكولات لمراقبة نصوص المراسل المتسلسل عن طريق تمديد التمهيدي و RT-PCR ، ولتحديد التعبير عن U1 snRNAs المعدلة عن طريق تمديد التمهيدي و RT-qPCR.
Protocol
1. الكواشف والمخازن المؤقتة
ملاحظة: يجب إجراء جميع عمليات التعقيم باستخدام مرشحات التفريغ باستخدام غشاء بولي إيثرسلفون (PES) بسعة 0.2 ميكرومتر في خزانة السلامة الأحيائية.
- تحضير الماء الخالي من RNase عن طريق إضافة 1.0 مل من ثنائي إيثيل بيروكربونات (DEPC) إلى 1.0 لتر من الماء منزوع الأيونات ، واخلطه لمدة ساعة واحدة على الأقل في درجة حرارة الغرفة (RT) ، والأوتوكلاف مرتين ، ثم تبرد إلى RT قبل الاستخدام.
- تحضير دولبيكو النسر المعدل المتوسط (DMEM) عن طريق خلط حزمة واحدة من مسحوق DMEM (13.4 غرام)، 3.7 غرام من بيكربونات الصوديوم، 100 مل من مصل البقر الجنيني (FBS)، البنسلين، والستربتومايسين إلى ~ 800 مل من الماء المعقم منزوع الأيونات. يجب أن يكون التركيز النهائي للبنسلين والستربتومايسين في DMEM 50 U / mL و 50 ميكروغرام / مل ، على التوالي. اضبط الرقم الهيدروجيني على 7.4 ثم قم بتكوين الحجم إلى 1.0 لتر بالماء المعقم منزوع الأيونات. تعقيم عن طريق الترشيح وتخزينها في 4 درجات مئوية.
- تحضير 10x الفوسفات المالحة العازلة (10x PBS) عن طريق إضافة 25.6 غرام من هيبتاهيدرات الهيدروجين ثنائي الصوديوم (Na2HPO4· 7H2O) ، 2 غرام من فوسفات ثنائي هيدروجين البوتاسيوم (KH2PO4) ، 2 غرام من كلوريد البوتاسيوم (KCl) ، و 80 غرام من كلوريد الصوديوم (NaCl) إلى 800 مل من الماء منزوع الأيونات. يخلط المزيج ليذوب ويشكل الحجم إلى 1.0 لتر، ويعقم بالترشيح ويخزن على درجة حرارة 4 درجات مئوية.
- تحضير 0.5 مليون ثنائي الإيثيلين حمض رباعي الخليك (EDTA) عن طريق إذابة 186.1 غرام من Na2 • EDTA • 2H2O في ~ 800 مل من الماء منزوع الأيونات. اضبط الرقم الهيدروجيني إلى 8.0 ثم اجعل الحجم يصل إلى 1.0 لتر بالماء المعقم منزوع الأيونات. تعقيم عن طريق الترشيح وتخزينها في 4 درجات مئوية.
- قم بإعداد محلول 1x trypsin-EDTA عن طريق خلط 100 مل من 10x trypsin (2.5٪) ، 2 مل من 0.5 M EDTA ، وإضافة 1x PBS حتى 1.0 L. تعقيم عن طريق الترشيح و aliquot في أنابيب مخروطية 50 مل. يخزن في درجة حرارة 4 درجات مئوية لمدة 1-2 أسابيع أو يتجمد عند -20 درجة مئوية للاستخدام على المدى الطويل.
- قم بتحضير صبغة تحميل 2x formamide DNA / RNA عن طريق خلط 14.4 مل من الفورماميد و 0.6 مل من 0.5 M EDTA للحصول على حجم نهائي من 15 مل. يضاف مسحوق البروموفينول الأزرق والزيلين السيانول إلى تركيز نهائي قدره 0.02٪ ويخزن عند 4 درجات مئوية.
ملاحظة: الفورماميد سام وقابل للتآكل. اقرأ أوراق بيانات سلامة المواد للحصول على توصيات السلامة الإضافية. - قم بإعداد 5x Tris / Borate / EDTA (5x TBE) عن طريق خلط 54.0 جم من قاعدة tris ، و 27.5 جم من حمض البوريك ، و 20 مل من 0.5 M EDTA في ~ 800 مل من الماء منزوع الأيونات. اخلطي المكونات لتذوب المكونات وتصل إلى 1.0 لتر بالماء منزوع الأيونات.
- تحضير محلول الرحلان الكهربائي لليوريا بولي أكريلاميد هلام (urea-PAGE) عن طريق خلط 200 مل من 5x TBE ، و 250 مل من 40٪ 19: 1 مكرر / أكريلاميد ، و 450.5 غرام من اليوريا. ثم يضاف الماء منزوع الأيونات حتى 1.0 لتر. اخلطه حتى تذوب المكونات تماما ، ثم قم بالتعقيم عن طريق الترشيح ، وخزنه على درجة حرارة 4 درجات مئوية في زجاجة زجاجية كهرمانية.
ملاحظة: مكرر / الأكريلاميد سام. اقرأ أوراق بيانات سلامة المواد لمعرفة إجراءات السلامة الإضافية. - تحضير 10٪ من بيرسلفات الأمونيوم (APS) عن طريق إذابة 1 غرام من APS في 10 مل من الماء منزوع الأيونات وتخزينها في 4 درجات مئوية.
2. النقل المشترك لخلايا HeLa مع المراسل وبلازميدات U1 snRNA
ملاحظة: يجب إجراء نقل خلايا هيلا تحت ظروف معقمة في خزانة السلامة البيولوجية. يجب رش السطح الخارجي لجميع المواد بالإيثانول بنسبة 70٪ قبل إدخاله في خزانة السلامة البيولوجية.
- الحفاظ على خلايا هيلا في DMEM في حاضنة 37 درجة مئوية مع 5 ٪ CO2 عن طريق المرور كل 2-3 أيام عندما تكون الخلايا حوالي 80-90 ٪ ملتقية.
- لتمرير خلايا HeLa ، قم بشفط الوسط المستهلك ثم احتضن الخلايا ب 3 مل من 0.25٪ من التربسين الذي يحتوي على 1 mM EDTA عند 37 درجة مئوية لمدة 3 دقائق.
- بعد الحضانة ، أضف 7 مل من DMEM الطازج. انقل تعليق الخلية إلى أنبوب 10 مل ، جهاز طرد مركزي عند 1000 × جم لمدة 5 دقائق.
- أعد تعليق حبيبات الخلايا في 10 مل من DMEM الطازج ، ثم قم بطلاء الخلايا على طبق جديد لزراعة الأنسجة عند التقاء 20٪.
- بالنسبة لعمليات النقل العابرة ، عد خلايا Hela ذات شريحة مقياس الدم النظيفة وقم بإعداد تعليق بكثافة 2.5 × 105 خلية / مل.
- البذور 1.0 مل من 2.5 × 105 خلية / مل تعليق خلية هيلا في كل بئر من لوحة 12 بئر وحضانتها بين عشية وضحاها عند 37 درجة مئوية.
- في اليوم التالي ، استنشق الوسط المستنفد وأضف 0.8 مل من DMEM الطازج مع المصل.
- قم بإعداد الحل الأول عن طريق إضافة 0.2 ميكروغرام من بلازميد مراسل Dup51 أو Dup51p ، و 1.8 ميكروغرام من pcDNA أو pNS6U1 أو pNS6U1-5a plasmid ، و 100 ميكرولتر من وسط النقل إلى أنبوب طرد مركزي دقيق جديد 1.5 مل.
- قم بإعداد مزيج رئيسي من المحلول الثاني لجميع العينات التي سيتم نقلها عن طريق إضافة 100 ميكرولتر من وسط النقل و 4.0 ميكرولتر من كاشف النقل لكل عينة.
- قم بإعداد مزيج النقل عن طريق إضافة 100 ميكرولتر من المحلول II إلى كل أنبوب جهاز طرد مركزي دقيق يحتوي على المحلول الأول.
- دوامة يختلط النقل لمدة 15 ثانية ، وأجهزة الطرد المركزي في جهاز طرد مركزي دقيق على الطاولة عند 3000 × g لمدة 10 ثوان في RT ، ثم يحضن في RT لمدة 5 دقائق.
- أضف كل 200 ميكرولتر من مزيج النقل في بئر واحد من صفيحة خلية HeLa المكونة من 12 بئرا لتحقيق حجم نهائي قدره 1.0 مل لكل بئر واحتضن اللوحة عند 37 درجة مئوية لمدة 48 ساعة.
- بعد الحضانة ، قم باستخراج الحمض النووي الريبي من خلايا HeLa المنقولة باستخدام محلول ثيوسيانات الجوانيدين والفينول المتاحين تجاريا.
ملاحظة: يحتوي هذا الكاشف على الفينول ويجب تنفيذ هذه الخطوة في غطاء الدخان. يوصى باستخدام المياه المعالجة DEPC لإعادة تعليق الحمض النووي الريبي المستخلص.- استنشق الوسط المستهلك وأضف 500 ميكرولتر من الكاشف إلى كل بئر. احتضان في RT لمدة 5 دقائق.
- التجانس عن طريق السحب لأعلى ولأسفل. ثم انقل المحلول إلى أنبوب طرد مركزي دقيق جديد سعة 1.5 مل.
- أضف 100 ميكرولتر من الكلوروفورم والدوامة لمدة 15 ثانية.
- جهاز طرد مركزي عند 12000 × g لمدة 15 دقيقة في RT.
- نقل 200 ميكرولتر من الحمض النووي الريبي الذي يحتوي على طبقة مائية علوية إلى أنبوب طرد مركزي دقيق جديد سعة 1.5 مل.
- أضف 2 ميكروغرام من الجليكوجين و 200 ميكرولتر من الأيزوبروبانول إلى كل عينة من عينات الحمض النووي الريبي. اخلط عن طريق عكس الأنابيب.
- جمع راسب الحمض النووي الريبي عن طريق الطرد المركزي في 12000 × غرام لمدة 10 دقائق عند 4 درجات مئوية.
- قم بإزالة والتخلص من supernatant دون إزعاج بيليه الحمض النووي الريبي.
- اغسل الكريات مرتين بإضافة 1.0 مل من الإيثانول بنسبة 70٪ ، وعكس الأنابيب ، والطرد المركزي كما هو موضح في الخطوة 2.13.7.
- قم بتجفيف الكريات في الهواء لمدة 10-20 دقيقة تقريبا في RT وأعد تعليق الحمض النووي الريبي في 10-20 ميكرولتر من الماء الخالي من RNase.
- أوجد تركيز الحمض النووي الريبي عن طريق قياس الامتصاص عند 260 نانومتر كما هو موضح من قبل Desjardins و Conklin9.
- تابع تمديد التمهيدي أو تخزين عينات الحمض النووي الريبي في -20 درجة مئوية. يمكن تخزين الحمض النووي الريبي المعزول عند -20 درجة مئوية لمدة 6-12 شهرا.
3. 32P وضع العلامات على قلة النوكليوتيدات
ملاحظة: يجب تنفيذ الخطوات التي تنطوي على استخدام أوليغونوكليوتيدات 32P-ATP و 32P الموسومة خلف درع أكريليك من قبل أفراد مدربين بموافقة من الكيانات المؤسسية المعتمدة. يمكن استخدام البروتوكول الموضح أدناه لوضع العلامات على أوليغونوكليوتيدات ، Dup3r و U17-26-R ، وعلامات اليوريا-PAGE. يوصى باستخدام المياه المعالجة DEPC لإعادة تعليق oligonucleotides والخرز الاستبعاد الحجم.
- إلى أنبوب جهاز طرد مركزي دقيق سعة 1.5 مل ، أضف oligonucleotide ، و T4 polynucleotide kinase (T4 PNK) ، و T4 PNK buffer ، و 32P-ATP كما هو موضح في الجدول 1. أضف 32P-ATP الأخير إلى الخليط ؛ هذا مهم.
ملاحظة: لإضافة المحاليل المشعة، يوصى باستخدام أطراف الفلتر. - حضانة في حمام مائي في 37 درجة مئوية لمدة 30 دقيقة.
- في حين أن تفاعلات وضع العلامات تحضن ، أعد تعليق حبات استبعاد الحجم بوزن جزيئي 25 kDa مقطوع عن طريق الدوامة بلطف لمدة ~ 10 ثوان.
ملاحظة: يجب إعداد حبات استبعاد الحجم وفقا لتعليمات الشركة المصنعة وتخزينها كتعليق بنسبة 50٪ في 25٪ من الإيثانول عند 4 درجات مئوية. - قم بإعداد الأعمدة عن طريق نقل 600 ميكرولتر من الخرز المعاد تعليقه إلى عمود صغير يمكن التخلص منه يوضع في أنبوب تجميع 1.5 مل وإعادة مخزون الخرز إلى 4 درجات مئوية.
- جهاز طرد مركزي عند 2000 × g لمدة 1 دقيقة في RT وتخلص من التدفق من خلاله.
- اغسل الخرز بإضافة 300 ميكرولتر من الماء الخالي من RNase إلى العمود.
- كرر الخطوات 3.5. و 3.6. مرتين ونقل العمود الصغير إلى أنبوب طرد مركزي جديد 1.5 مل.
- أضف مزيج تفاعل كيناز إلى عمود خرزة استبعاد الحجم وأجهزة الطرد المركزي عند 5000 × g لمدة 1 دقيقة في RT.
- جمع وحفظ التدفق من خلال، والتي لديها 32P المسمى oligonucleotide، وتجاهل جميع النصائح والأنابيب في صندوق نفايات الاكريليك.
- أضف 20 ميكرولتر من الماء الخالي من RNase لتخفيف أوليغونوكليوتيد 32P المسمى إلى تركيز نهائي قدره 2.5 ميكرومتر.
ملاحظة: قم بتخفيف العلامات الموسومة حسب الحاجة للتحميل على المواد الهلامية المصنوعة من اليوريا بيج. - قم بتخزين oligonucleotide المسمى في صندوق أنبوب دقيق أكريليك عند -20 درجة مئوية أو تابع تحليل تمديد التمهيدي.
4. تحليل نصوص المراسل المتسلسل حسب امتداد التمهيدي
ملاحظة: يوصى بتنظيف الأسطح والمعدات باستخدام كاشف RNase المعطل قبل الاستخدام.
- أضف 2.0 ميكروغرام من الحمض النووي الريبي المستخرج من خلايا هيلا إلى أنابيب منفصلة لأجهزة الطرد المركزي الدقيقة سعة 200 ميكرولتر وأضف ماء خاليا من RNase لتكوين الحجم إلى 6.55 ميكرولتر.
- قم بإعداد المزيج الرئيسي I مع 32P-Dup3r المخفف و dNTPs كما هو موضح في الجدول 2 وأضف 0.9 ميكرولتر من المزيج إلى كل أنبوب PCR يحتوي على عينات RNA.
- احتضن الأنابيب ، أولا عند 65 درجة مئوية لمدة 5 دقائق ثم على الجليد لمدة 1 دقيقة.
- قم بإعداد Master Mix II باستخدام 5x First Strand Buffer و dithiothreitol (DTT) ومثبط RNase والنسخ العكسي كما هو موضح في الجدول 2.
- أضف 2.55 ميكرولتر من المزيج إلى كل أنبوب يحتوي على الحمض النووي الريبي والمزيج الرئيسي 1 ؛ يجب أن يكون الحجم الإجمالي للتفاعل 10 ميكرولتر.
- انقل الأنابيب إلى حمام جاف أو جهاز تدوير حراري واحتضنها ، أولا عند 50 درجة مئوية لمدة 60 دقيقة ثم عند 70 درجة مئوية لمدة 15 دقيقة.
- بعد الحضانة ، أضف 10 ميكرولتر من صبغة تحميل الحمض النووي الريبي فورماميد 2x إلى كل عينة وتخزينها في صندوق أكريليك عند -20 درجة مئوية أو المضي قدما في فصل الشظايا بواسطة urea-PAGE باستخدام هلام بطول 14 سم وتصور صورة هلام كما هو موضح أدناه في الخطوة 8.
- قم بإجراء مسح مكثف لصورة الجل باستخدام برنامج تحليل الصور واستخدم كثافة النطاق للمنتجات المضمنة والمتخطية لحساب النسبة المئوية لتضمين exon 2 كما هو موضح أدناه.

5. تحليل نصوص المراسل المتسلسل بواسطة RT-PCR الفلورسنت
ملاحظة: يستخدم تحليل RT-PCR الموصوف أدناه أجهزة فحص عشوائية لتخليق cDNA ومزيج من أوليغونوكليوتيدات Dup8f و Dup3r غير المصنفة على اسم Cy5 لتضخيم PCR للمنتجات المقطعة.
- لتخليق cDNA ، أضف 2.0 ميكروغرام من الحمض النووي الريبي المستخرج من خلايا Hela المنقولة إلى أنابيب طرد مركزي دقيقة منفصلة سعة 200 ميكرولتر وأضف ماء خاليا من RNase لتكوين الحجم إلى 11.0 ميكرولتر.
- تحضير المزيج الرئيسي الأول ، الذي يحتوي على hexamers عشوائي و dNTP كما هو موضح في الجدول 3 وإضافة 2.0 ميكرولتر من المزيج إلى كل عينة. احتضان ، أولا عند 65 درجة مئوية لمدة 5 دقائق ثم على الجليد لمدة 1 دقيقة.
- قم بإعداد Master Mix II ، الذي يحتوي على المخزن المؤقت الأول للحبلا ، ومثبط RNase ، و DTT ، والنسخ العكسي كما هو موضح في الجدول 3 وأضف 7.0 ميكرولتر من المزيج إلى كل أنبوب يحتوي على الحمض النووي الريبي والمزيج الرئيسي الأول.
- احتفظ بالأنابيب في RT لمدة 10 دقائق ، ثم احتضنها عند 50 درجة مئوية لمدة 60 دقيقة و 70 درجة مئوية لمدة 15 دقيقة.
ملاحظة: يمكن تخزين تفاعلات cDNA المكتملة عند -20 درجة مئوية. - بالنسبة ل PCR ، انقل 1.0 ميكرولتر (100 نانوغرام / ميكرولتر) من كل عينة من عينات cDNA إلى أنابيب PCR جديدة.
- قم بإعداد مزيج رئيسي يتكون من Dup8f و Cy5-Dup3r و dNTPs و Taq buffer و Taq polymerase والماء كما هو موضح في الجدول 4 وأضف 11.5 ميكرولتر من المزيج إلى كل أنبوب يحتوي على cDNA.
- إجراء PCR باستخدام دورة حرارية مع خطوة تمسخ أولية عند 94 درجة مئوية لمدة 3 دقائق ؛ تليها 20 دورة من تمسخ (94 درجة مئوية لمدة 30 ثانية) ، والتلدين (65 درجة مئوية لمدة 30 ثانية) ، والتمديد (72 درجة مئوية لمدة 15 ثانية) ، وخطوة إنهاء عند 72 درجة مئوية لمدة 5 دقائق.
- أضف 12.5 ميكرولتر من صبغة تحميل الحمض النووي فورماميد 2x إلى كل أنبوب وقم بتسخينها عند 95 درجة مئوية لمدة 5 دقائق.
- قم بتخزين تفاعل PCR عند -20 درجة مئوية أو تابع استخدام urea-PAGE كما هو موضح أدناه في الخطوة 8.1-8.4.
- بعد الرحلان الكهربائي ، قم بإزالة الألواح الزجاجية من جهاز الرحلان الكهربائي والمسح الضوئي باستخدام جهاز تصوير التألق لتصور الجل.
- قم بإجراء مسح مكثف لصورة الجل واستخدم كثافة النطاق للمنتجات المضمنة والمنتجات التي تم تخطيها لحساب النسبة المئوية لتضمين exon 2 كما هو موضح في الخطوة 4.8.
6. تحليل التعبير البديل U1 snRNA عن طريق تمديد التمهيدي
- أضف 2.0 ميكروغرام من الحمض النووي الريبي المستخرج من خلايا هيلا إلى أنابيب منفصلة لأجهزة الطرد المركزي الدقيقة سعة 200 ميكرولتر وأضف ماء خاليا من RNase لتكوين الحجم إلى 4.325 ميكرولتر ثم أضف dATP ، كما هو موضح في الجدول 5.
- أضف 10000 نسخة في الدقيقة من أوليغونوكليوتيد 32P-U17-26-R إلى كل أنبوب.
ملاحظة: لإعداد محلول 10000 نسخة في الدقيقة / ميكرولتر من 32P-U17-26-R (من الخطوة 3) ، قم بتخفيف oligonucleotide المسمى (تخفيف 1:20) ، وتحديد cpm في 1.0 μL باستخدام عداد تلألؤ ، وتخفيف المزيد بالماء منزوع الأيونات لإعداد محلول 10000 cpm / μL في أنبوب طرد مركزي دقيق جديد 1.5 مل. - تحضن في 65 درجة مئوية لمدة 5 دقائق ، ثم على الجليد لمدة 1 دقيقة.
- قم بإعداد مزيج رئيسي مع 5x First Strand Buffer و RNase inhibitor و DTT و transcriptase العكسي كما هو موضح في الجدول 5 وأضف 1.8 ميكرولتر من المزيج إلى كل عينة.
- احتضان ، أولا في RT لمدة 10 دقائق ثم عند 42 درجة مئوية لمدة 10 دقائق.
- بعد الحضانة ، أضف 10 ميكرولتر من صبغة تحميل الحمض النووي الريبي فورماميد 2x في كل عينة وتخزينها في صندوق أكريليك عند -20 درجة مئوية أو المضي قدما في فصل الشظايا بواسطة urea-PAGE باستخدام هلام بطول 38 سم (انظر الخطوة 8).
7. تحليل التعبير المتغير U1 snRNA بواسطة RT-qPCR
- قم بتخفيف مخزون cDNA المحضر كما هو موضح أعلاه في الخطوات 5.1 - 5.4 إلى تركيز 0.2 نانوغرام / ميكرولتر.
- ماصة 5.0 ميكرولتر من cDNA المخفف في آبار فردية من لوحة qPCR 96 بئر في ثلاثة أضعاف. أضف الماء منزوع الأيونات بدلا من cDNA للتحكم بدون قالب (NTC).
- قم بإعداد خليطين تمهيديين منفصلين يتألفان من الاشعال الأمامي والخلفي ل U1 و U2 snRNAs ، والماء كما هو موضح في الجدول 6.
ملاحظة: ترد في الجدول 7 تسلسلات لأوليات خاصة ب U1 و U2 snRNA. - أضف 5.0 ميكرولتر من مزيج التمهيدي U1 و U2 snRNA إلى العينة وآبار NTC.
- أضف 10.0 ميكرولتر من مزيج PCR في الوقت الفعلي إلى كل بئر.
- قم بإغلاق الألواح بفيلم بصري ، ثم جهاز طرد مركزي عند 1000 × g لمدة 2 دقيقة في RT لجمع التفاعلات إلى قاع الآبار.
- قم بإجراء qPCR مع خطوة تمسخ أولية عند 95 درجة مئوية لمدة 10 دقائق ، تليها 40 دورة من بروتوكول من خطوتين يتكون من تمسخ (95 درجة مئوية لمدة 15 ثانية) والتلدين / التمديد (62 درجة مئوية لمدة 60 ثانية) أثناء جمع قيم دورة القياس الكمي للعتبة (Cq) للمضخمات المستهدفة.
- قم بإنهاء تفاعل qPCR عن طريق التحقق من وجود ذروة واحدة في منحنى التفكك لتفاعلات snRNA U1 و U2.
- من قيم Cq ، احسب دلتا Cq (ΔCq) ل U1 و U2 snRNAs مقارنة بالتحكم في pcDNA.
- حدد تعبير المتغير U1 snRNA ككمية نسبية (RQ) ل U1 مقارنة ب U2 باستخدام قيمة ΔΔCq كما هو موضح أدناه لجميع العينات.

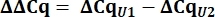
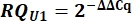
8. إعداد وتشغيل المواد الهلامية Urea-PAGE
ملاحظة: يجب إجراء تجميع الألواح الزجاجية وجهاز تشغيل الجل وفقا لتعليمات الشركة المصنعة. يمكن إجراء صب هلام اليوريا PAGE بنسبة 10٪ وفقا لبروتوكول موصوف مسبقا بواسطة Summer et al.10. الخطوات التي تنطوي على إعداد العلامات والعينات ، وتشغيل وتصور المواد الهلامية موصوفة أدناه. اختياريا ، لمنع الجل من الالتصاق بالألواح الزجاجية ، يمكن طلاء السطح الداخلي بمحلول السيليكون عن طريق إضافة 1 مل من المحلول على السطح ونشره بالتساوي على السطح بأكمله بالأنسجة. بمجرد أن تجف ، يجب غسل الألواح بالماء منزوع الأيونات وتجفيفها مرة أخرى.
تنبيه: مادة الأكريلاميد غير المبلمرة سامة للأعصاب ويجب التعامل معها بوسائل الحماية الموصى بها في ورقة بيانات سلامة المواد.
- قم بإعداد عينات وعلامات تمديد التمهيدي عن طريق التسخين عند 95 درجة مئوية لمدة 5 دقائق ثم الطرد المركزي في جهاز طرد مركزي دقيق على الطاولة عند 3000 × g لمدة 5 ثوان في RT.
- قبل تحميل العلامات والعينات ، اطرد الآبار باستخدام مخزن مؤقت 1x TBE لإزالة اليوريا المستقرة.
- قم بتحميل 10 ميكرولتر / عينة / بئر وقم بتشغيل الجل عند 300-500 فولت لمدة 2-3 ساعات أو حتى يصل سيانول الزيلين إلى القاع.
ملاحظة: يمكن تحميل حوالي 1000 نسخة في الدقيقة من العلامة التي تحمل علامة 32P. - بعد الرحلان الكهربائي ، قم بإزالة الألواح الزجاجية من جهاز الرحلان الكهربائي.
- افصل اللوحتين بعناية بحيث يوضع الجل بشكل مسطح على أي من السطحين الزجاجيين وينقل الجل إلى ورقة تصفية ويغطيه بغلاف بلاستيكي.
- جفف الجل بالمكنسة الكهربائية على ورق الترشيح على درجة حرارة 80 درجة مئوية لمدة 30 دقيقة باستخدام مجفف هلام.
- ضع الجل المجفف في كاسيت تصوير فوسفوري واحتفظ به في RT طوال الليل.
ملاحظة: يجب مسح شاشة الفوسفور باستخدام صندوق ضوء قبل الاستخدام. - لتصور صورة الجل ، قم بإزالة الشاشة والمسح الضوئي باستخدام جهاز تصوير الفوسفور.
النتائج
تم اشتقاق مراسل الربط Dup51 ، وهو جين صغير من ثلاثة إكسون اثنين ، من جين β-globin البشري وتم وصفه سابقا (الشكل 1A) 11,12 . أنشأنا مراسلا متحورا ، Dup51p ، من خلال إدخال طفرات موقع 5'-splice المرتبطة بمتلازمة آشر التي تحدث في exon 3 من جين protocadherin 15 (PCDH15)
Discussion
يمكن تكييف الفحص لتحليل الربط في خطوط الخلايا بخلاف HeLa، ومع ذلك، قد تحتاج العوامل التي تؤثر على كفاءة النقل، مثل التقاء الخلايا وكمية الحمض النووي إلى تحسينها. نسبة بناء المراسل إلى U1 هي معلمة حرجة أخرى قد تحتاج إلى تحديدها اعتمادا على مستويات التعبير التي لوحظت في أنواع الخلايا الأخرى. ج?...
Disclosures
ليس لدى المؤلفين ما يكشفون عنه.
Acknowledgements
تم دعم هذا العمل من خلال الأموال المقدمة إلى S.S. من المعاهد الوطنية للصحة (R21CA170786 و R01GM127464) وجمعية السرطان الأمريكية (منحة البحوث المؤسسية 74-001-34-IRG) وإلى S.S. و W.M. من برنامج شراكة أبحاث الوادي (P1-4009 و VRP77). المحتوى هو مسؤولية المؤلفين وحدهم ولا يمثل بالضرورة وجهات النظر الرسمية للمعاهد الوطنية للصحة.
Materials
| Name | Company | Catalog Number | Comments |
| Reagent Grade Deionized Water | ThermoFisher Scientific | 23-751628 | |
| Diethyl pyrocarbonate (DEPC) | Sigma-Aldrich | D5758-25ML | |
| Dulbecco's Modified Eagle Medium (DMEM) powder packet | Gibco | 12100-046 | |
| Sodium Bicarbonate | ThermoFisher Scientific | S233-500 | |
| Fetal Bovine Serum (FBS), Australian Source, Heat Inactivated | Omega Scientific | FB-22 | |
| Penicillin-Streptomycin (P/S) | Sigma-Aldrich | P4458-100ML | |
| Sodium Hydroxide, Standard Solution 1.0N | Sigma-Aldrich | S2567-16A | |
| Hydrochloric Acid, Certified ACS Plus, 36.5 to 38.0% | ThermoFisher Scientific | A144-500 | |
| Disposable PES Bottle Top Filters | ThermoFisher Scientific | FB12566510 | |
| EDTA Disodium Salt Dihydrate | Amresco | 0105-2.5KG | |
| 2.5% Trypsin (10x), no phenol red | ThermoFisher Scientific | 15090046 | |
| Sodium Chloride | Fisher Bioreagent | BP358-212 | |
| Potassium Chloride | Fisher Bioreagent | BP366-1 | |
| Disodium Hydrogen Phosphate Heptahydrate | Fisher Bioreagent | BP332-1 | |
| Potassium Dihydrogen Phosphate | Fisher Bioreagent | BP362-1 | |
| Transfection medium - Opti-MEM™ I Reduced Serum Medium, no phenol red | ThermoFisher Scientific | 11058021 | |
| Transfection Reagent - Lipofectamine™ 2000 | ThermoFisher Scientific | 13778150 | |
| TRIzol™ Reagent | ThermoFisher Scientific | 15596018 | |
| Chloroform (Approx. 0.75% Ethanol as Preservative/Molecular Biology) | ThermoFisher Scientific | BP1145-1 | |
| Ethanol, Absolute (200 Proof), Molecular Biology Grade, Fisher BioReagents | ThermoFisher Scientific | BP2818-4 | |
| Isopropanol, Molecular Biology Grade, Fisher BioReagents | ThermoFisher Scientific | BP2618-212 | |
| Glycogen (5 mg/ml) | ThermoFisher Scientific | AM9510 | |
| Direct-zol RNA Miniprep Kit | Zymo Research | R2052 | |
| ATP, [γ-32P]- 6000Ci/mmol 150mCi/ml Lead, 1 mCi | PerkinElmer | NEG035C001MC | |
| T4 Polynucleotide Kinase | New England Biolabs | M0201L | |
| Size exclusion beands - Sephadex® G-25 | Sigma-Aldrich | G2580-10G | |
| Size exclusion mini columns | USA Scientific | 1415-0600 | |
| pBR322 DNA-MspI Digest | New England Biolabs | N3032S | |
| Low Molecular Weight Marker, 10-100 nt | Affymetrix | 76410 100 UL | |
| Rnase inactivating reagents - RNaseZAP™ | Sigma-Aldrich | R2020-250ML | |
| dNTP Mix (10 mM ea) | ThermoFisher Scientific | 18427013 | |
| RNaseOUT™ Recombinant Ribonuclease Inhibitor | ThermoFisher Scientific | 10777019 | |
| Reverse Transcriptase - M-MLV Reverse Transcriptase | ThermoFisher Scientific | 28025013 | used for primer extension |
| Taq DNA Polymerase | ThermoFisher Scientific | 10342020 | |
| Random Hexamers (50 µM) | ThermoFisher Scientific | N8080127 | |
| Real time PCR mix - SYBR™ Select Master Mix | ThermoFisher Scientific | 4472903 | |
| SuperScript™ III Reverse Transcriptase | ThermoFisher Scientific | 18080093 | used for cDNA preparation |
| Dithiothreitol (DTT) | ThermoFisher Scientific | 18080093 | |
| 5X First-Strand Buffer | ThermoFisher Scientific | 18080093 | |
| Formamide (≥99.5%) | ThermoFisher Scientific | BP228-100 | Review Material Safety Data Sheets |
| Bromophenol Blue sodium salt | Sigma-Aldrich | 114405-5G | |
| Xylene Cyanol FF | Sigma-Aldrich | 2650-17-1 | |
| Tris Base (White Crystals or Crystalline Powder/Molecular Biology) | ThermoFisher Scientific | BP152-5 | |
| Boric Acid (Crystalline/Electrophoresis) | ThermoFisher Scientific | BP168-500 | |
| Acrylamide: Bis-Acrylamide 19:1 (40% Solution/Electrophoresis) | ThermoFisher Scientific | BP1406-1 | Review Material Safety Data Sheets |
| Urea (Colorless-to-White Crystals or Crystalline Powder/Mol. Biol.) | ThermoFisher Scientific | BP169-212 | |
| Ammonium peroxodisulphate (APS) ≥98%, Pro-Pure, Proteomics Grade | VWR | M133-25G | |
| Sigmacote | Sigma-Aldrich | SL2-100ML | |
| N,N,N',N'-Tetramethylethylenediamine (TEMED) ≥99%, Ultrapure | VWR | 0761-25ML | Review Material Safety Data Sheets |
| Adjustable Slab Gel Systems, Expedeon | VWR | ASG-400 | |
| Vertical Gel Wrap™ Glass Plate Sets, 16.5 x 14.5cm | VWR | NGP-125NR | |
| Vertical Gel Wrap™ Glass Plate Sets, 16.5 x 22.0cm | VWR | NGP-200NR | |
| Vertical Gel Wrap™ Glass Plate Sets, 16.5 x 38.7cm | VWR | NGP-400NR | |
| GE Storage Phosphor Screens | Sigma-Aldrich | GE28-9564 | |
| Typhoon™ FLA 7000 Biomolecular Imager | GE Healthcare | 28-9610-73 AB | |
| Beckman Coulter LS6500 Liquid Scintillation Counter | GMI | 8043-30-1194 | |
| C1000 Touch Thermal Cycler | ThermoFisher Scientific | ||
| QuantStudio 6 Flex Real-Time PCR Systems | ThermoFisher Scientific |
References
- Zhuang, Y., Weiner, A. M. A compensatory base change in U1 snRNA suppresses a 5' splice site mutation. Cell. 46 (6), 827-835 (1986).
- Scotti, M. M., Swanson, M. S. RNA mis-splicing in disease. Nature Review Genetics. 17 (1), 19-32 (2016).
- Ward, A. J., Cooper, T. A. The pathobiology of splicing. Journal of Pathology. 220 (2), 152-163 (2010).
- Scalet, D., et al. Disease-causing variants of the conserved +2T of 5' splice sites can be rescued by engineered U1snRNAs. Human Mutatation. 40 (1), 48-52 (2019).
- Yamazaki, N., et al. Use of modified U1 small nuclear RNA for rescue from exon 7 skipping caused by 5'-splice site mutation of human cathepsin A gene. Gene. 677, 41-48 (2018).
- Yanaizu, M., Sakai, K., Tosaki, Y., Kino, Y., Satoh, J. I. Small nuclear RNA-mediated modulation of splicing reveals a therapeutic strategy for a TREM2 mutation and its post-transcriptional regulation. Science Reports. 8 (1), 6937 (2018).
- Balestra, D., et al. Splicing mutations impairing CDKL5 expression and activity can be efficiently rescued by U1snRNA-based therapy. International Journal of Molecular Sciences. 20 (17), 20174130 (2019).
- Donadon, I., et al. Exon-specific U1 snRNAs improve ELP1 exon 20 definition and rescue ELP1 protein expression in a familial dysautonomia mouse model. Human Molecular Genetics. 27 (14), 2466-2476 (2018).
- Desjardins, P., Conklin, D. NanoDrop microvolume quantitation of nucleic acids. Journal of Visualized Experiments. (45), e2565 (2010).
- Summer, H., Gramer, R., Droge, P. Denaturing urea polyacrylamide gel electrophoresis (Urea PAGE). Journal of Visualized Experiments. (32), e1485 (2009).
- Dominski, Z., Kole, R. Selection of splice sites in pre-mRNAs with short internal exons. Molecular Cell Biology. 11 (12), 6075-6083 (1991).
- Amir-Ahmady, B., Boutz, P. L., Markovtsov, V., Phillips, M. L., Black, D. L. Exon repression by polypyrimidine tract binding protein. RNA. 11 (5), 699-716 (2005).
- Le Guedard-Mereuze, S., et al. Sequence contexts that determine the pathogenicity of base substitutions at position +3 of donor splice-sites. Human Mutation. 30 (9), 1329-1339 (2009).
- Sharma, S., Wongpalee, S. P., Vashisht, A., Wohlschlegel, J. A., Black, D. L. Stem-loop 4 of U1 snRNA is essential for splicing and interacts with the U2 snRNP-specific SF3A1 protein during spliceosome assembly. Genes and Development. 28 (22), 2518-2531 (2014).
- Steitz, J. A., et al. . Functions of the abundant U-snRNPs. Structure and function of major and minor small nuclear ribonucleoprotein particles. , 115-154 (1988).
- Fortes, P., et al. Inhibiting expression of specific genes in mammalian cells with 5' end-mutated U1 small nuclear RNAs targeted to terminal exons of pre-mRNA. Proceedings of the National Academy of Sciences U.S.A. 100 (14), 8264-8269 (2003).
- Roca, X., et al. Widespread recognition of 5' splice sites by noncanonical base-pairing to U1 snRNA involving bulged nucleotides. Genes and Development. 26 (10), 1098-1109 (2012).
- Roca, X., Krainer, A. R. Recognition of atypical 5' splice sites by shifted base-pairing to U1 snRNA. Nature Structural Molecular Biology. 16 (2), 176-182 (2009).
- Taladriz-Sender, A., Campbell, E., Burley, G. A. Splice-switching small molecules: A new therapeutic approach to modulate gene expression. Methods. 167, 134-142 (2019).
- Hamid, F. M., Makeyev, E. V. A mechanism underlying position-specific regulation of alternative splicing. Nucleic Acids Research. 45 (21), 12455-12468 (2017).
- Martelly, W., et al. Synergistic roles for human U1 snRNA stem-loops in pre-mRNA splicing. RNA Biology. , 1-18 (2021).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved