A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
توصيف ثلاثي الأبعاد لمواقع التلامس بين الأعضاء في خلايا الكبد باستخدام المجهر الإلكتروني التسلسلي
In This Article
Summary
بروتوكول بسيط وشامل للحصول على تفاصيل ثلاثية الأبعاد لمواقع ملامسة الغشاء بين العضيات في خلايا الكبد من الكبد أو الخلايا في الأنسجة الأخرى.
Abstract
لطالما اعتبر المجهر الإلكتروني الناقل هو المعيار الذهبي لتصور البنية الفائقة الخلوية. ومع ذلك ، غالبا ما يقتصر التحليل على بعدين ، مما يعوق القدرة على الوصف الكامل للبنية الفائقة ثلاثية الأبعاد (3D) والعلاقة الوظيفية بين العضيات. يصف المجهر الإلكتروني الحجمي (vEM) مجموعة من التقنيات التي تمكن من استجواب البنية الفائقة الخلوية في 3D بدقة متوسطة الحجم ومجهرية ونانوية.
يوفر هذا البروتوكول طريقة سهلة الوصول وقوية للحصول على بيانات vEM باستخدام إرسال القسم التسلسلي EM (TEM) ويغطي الجوانب التقنية لمعالجة العينات من خلال إعادة الإعمار الرقمي 3D في سير عمل واحد ومباشر. لإثبات فائدة هذه التقنية ، يتم تقديم العلاقة فوق الهيكلية 3D بين الشبكة الإندوبلازمية والميتوكوندريا ومواقع الاتصال الخاصة بها في خلايا الكبد الكبدية. تؤدي الاتصالات بين الأعضاء أدوارا حيوية في نقل الأيونات والدهون والمواد المغذية والجزيئات الصغيرة الأخرى بين العضيات. ومع ذلك ، على الرغم من اكتشافها الأولي في خلايا الكبد ، لا يزال هناك الكثير لتعلمه عن ميزاتها الفيزيائية وديناميكياتها ووظائفها.
يمكن أن تعرض جهات الاتصال بين الأعضاء مجموعة من المورفولوجيات ، تختلف في قرب العضيتين من بعضهما البعض (عادة ~ 10-30 نانومتر) ومدى موقع الاتصال (من جهات الاتصال المثقوبة إلى جهات الاتصال الأكبر الشبيهة بالصهريج ثلاثي الأبعاد). يتطلب فحص المخالطين المقربين تصويرا عالي الدقة ، والقسم التسلسلي TEM مناسب تماما لتصور البنية الفائقة 3D للاتصالات بين الأعضاء أثناء تمايز خلايا الكبد ، وكذلك التغييرات في بنية خلايا الكبد المرتبطة بأمراض التمثيل الغذائي.
Introduction
منذ اختراعها في 1930s ، سمحت المجاهر الإلكترونية للباحثين بتصور المكونات الهيكلية للخلايا والأنسجة 1,2. قدمت معظم التحقيقات معلومات ثنائية الأبعاد ، حيث يتطلب بناء نماذج ثلاثية الأبعاد جمع قسم تسلسلي مضني ، والتصوير اليدوي ، والمعالجة السلبية ، والتتبع اليدوي ، وإنشاء وتجميع نماذج ثلاثية الأبعاد من صفائح الزجاج أو البلاستيك أو الستايروفوم 3,4. بعد ما يقرب من 70 عاما ، كان هناك تقدم كبير في العديد من جوانب العملية ، من أداء المجهر ، وجمع القسم التسلسلي ، والتصوير الرقمي الآلي ، والبرامج والأجهزة المتطورة لإعادة الإعمار 3D ، والتصور ، والتحليل إلى النهج البديلة لما يسمى الآن بشكل جماعي حجم EM (vEM). تعتبر تقنيات vEM هذه بشكل عام لتوفير معلومات فوق هيكلية 3D بدقة نانومتر عبر مقاييس ميكرون وتشمل المجهر الإلكتروني للإرسال (TEM) وتقنيات المجهر الإلكتروني الماسح الأحدث (SEM) ؛ انظر المراجعات5،6،7،8.
على سبيل المثال ، يستخدم شعاع الأيونات المركز SEM (FIB-SEM) شعاعا أيونيا مركزا داخل SEM لطحن سطح الكتلة بين عمليات مسح تصوير SEM المتسلسلة لسطح الكتلة ، مما يسمح بالطحن / التصوير الآلي المتكرر لعينة وبناء مجموعة بيانات ثلاثية الأبعاد لإعادة الإعمار 9,10. في المقابل ، يستخدم وجه الكتلة التسلسلي SEM (SBF-SEM) ميكروتوم فائق الميكروتوم داخل SEM لإزالة المواد من وجه الكتلة قبل التصوير 11,12 ، في حين أن التصوير المقطعي للصفيف هو عملية غير مدمرة تتطلب جمع المقاطع التسلسلية ، على أغطية أو رقائق أو شريط ، قبل إعداد سير عمل آلي لتصوير المنطقة ذات الأهمية في الأقسام المتسلسلة في SEM لإنشاء مجموعة بيانات ثلاثية الأبعاد 13 . على غرار التصوير المقطعي بالمصفوفة ، يتطلب القسم التسلسلي TEM (ssTEM) جمع الأقسام المادية قبل التصوير ؛ ومع ذلك ، يتم جمع هذه الأقسام على شبكات TEM وتصويرها في TEM14،15،16. يمكن تمديد ssTEM عن طريق إجراء التصوير المقطعي بالإمالة17،18،19. يوفر التصوير المقطعي التسلسلي للإمالة أفضل دقة في x و y و z ، وعلى الرغم من استخدامه لإعادة بناء الخلايا الكاملة20 ، إلا أنه يمثل تحديا معقولا. يركز هذا البروتوكول على الجوانب العملية ل ssTEM باعتبارها تقنية vEM الأكثر سهولة المتاحة للعديد من مختبرات EM التي قد لا تتمكن حاليا من الوصول إلى أدوات التقسيم المتخصصة أو vEM ولكنها ستستفيد من توليد بيانات 3D vEM.
وقد سبق أن اعتبر الاستئصال التسلسلي للميكروتومي لإعادة بناء 3D تحديا. كان من الصعب قص شرائط مستقيمة بسماكة مقطع ، والقدرة على ترتيب والتقاط شرائط بالحجم الصحيح ، بالترتيب الصحيح ، على الشبكات مع الدعم الكافي ، ولكن بدون قضبان الشبكة التي تحجب مناطق الاهتمام ، والأهم من ذلك ، دون فقدان الأقسام ، لأن سلسلة غير مكتملة قد تمنع إعادة الإعمار 3D الكاملة21. ومع ذلك ، فإن التحسينات التي أدخلت على الميكروتومات الفائقة التجارية ، وسكاكين قطع الماس وتقليمه 22،23 ، وأفلام دعم الإلكترون لوسنت على الشبكات 21،24 ، والمواد اللاصقة للمساعدة في التصاق القسم والحفاظ على الشريط13،21 ليست سوى بعض من التطورات التدريجية على مر السنين التي جعلت هذه التقنية أكثر روتينية في العديد منالمختبرات. بمجرد جمع الأقسام التسلسلية ، يكون التصوير التسلسلي في TEM مباشرا ويمكن أن يوفر صورا EM بأحجام px تحت النانومتر في x و y ، مما يسمح باستجواب عالي الدقة للهياكل دون الخلوية - وهو شرط محتمل للعديد من الأسئلة البحثية. توضح دراسة الحالة المقدمة هنا استخدام ssTEM وإعادة بناء 3D في دراسة اتصالات الخلايا الإندوبلازمية (ER) في خلايا الكبد الكبدية ، حيث لوحظت اتصالات ER-organelle لأول مرة25,26.
في حين أن ER متجاور مع الغلاف النووي ، فإنه يقوم أيضا بإجراء اتصالات وثيقة مع العديد من عضيات الخلايا الأخرى ، بما في ذلك الليزوسومات والميتوكوندريا وقطرات الدهون وغشاء البلازما27. تورطت جهات الاتصال العضوية ER في استقلاب الدهون28 ، وإشارات الفوسفوينوسيتيد والكالسيوم29 ، وتنظيم الالتهام الذاتي ، والاستجابة للإجهاد30,31. اتصالات العضيات ER وغيرها من الاتصالات بين الأعضاء هي هياكل ديناميكية للغاية تستجيب لاحتياجات التمثيل الغذائي الخلوي والإشارات خارج الخلية. وقد تبين أنها تختلف من الناحية المورفولوجية في حجمها وشكلها والمسافات بين أغشية العضيات32,33. ويعتقد أن هذه الاختلافات فوق الهيكلية من المرجح أن تعكس تركيباتها المختلفة للبروتين / الدهون ووظيفتها34,35. ومع ذلك ، لا تزال مهمة صعبة لتحديد جهات الاتصال بين الأعضاء وتحليلها36. وبالتالي ، هناك حاجة إلى بروتوكول موثوق به ولكنه بسيط لفحص وتوصيف الاتصالات بين الأعضاء لإجراء مزيد من التحقيقات.
نظرا لأن جهات اتصال ER organelle يمكن أن تتراوح من 10 إلى 30 نانومتر في الفصل بين الغشاء والغشاء ، فإن المعيار الذهبي لتحديد الهوية كان تاريخيا TEM. كشف قسم رفيع TEM عن توطين نطاق فرعي محدد لبروتينات ER المقيمة في جهات اتصال غشائية متميزة37. تقليديا ، كشف هذا عن اتصالات ER-organelle بدقة nm ولكن في كثير من الأحيان لم يقدم سوى عرض 2D لهذه التفاعلات. ومع ذلك ، تكشف مناهج vEM عن العرض والسياق الهيكلي لمواقع الاتصال هذه في 3D ، مما يتيح إعادة البناء الكامل لجهات الاتصال وتصنيف أكثر دقة لجهات الاتصال (النقطة مقابل الأنبوبي مقابل الشبيه بالصخر) والقياس الكمي38,39. بالإضافة إلى كونها أول نوع من الخلايا حيث لوحظت اتصالات ER-organelle25,26 ، فإن خلايا الكبد لديها نظام واسع من الاتصالات الأخرى بين الأعضاء التي تخدم أدوارا حيوية في هندستها المعمارية وعلم وظائف الأعضاء 28,40. ومع ذلك ، لا يزال التوصيف المورفولوجي الشامل للعضيات ER-organelle وغيرها من الاتصالات بين العضيات في خلايا الكبد غير موجود. وفقا لذلك ، فإن كيفية تشكل الاتصالات بين الأعضاء وإعادة تشكيلها أثناء التجديد والإصلاح لها أهمية خاصة لبيولوجيا خلايا الكبد ووظائف الكبد.
Access restricted. Please log in or start a trial to view this content.
Protocol
تم إيواء جميع الحيوانات وفقا للمبادئ التوجيهية لوزارة الداخلية في المملكة المتحدة ، وتم حصاد الأنسجة وفقا لقانون المملكة المتحدة للحيوانات (الإجراءات العلمية) لعام 1986.
1. تثبيت العينات وإعدادها
- قم بتشريح أنسجة الكبد إلى قطع مناسبة الحجم ، حوالي 8 مم × 8 مم × 3 مم ، وضع القطع في محلول ملحي دافئ مخزن بالفوسفات (PBS ، 37 درجة مئوية).
- حقن درجة حرارة الغرفة (20-25 درجة مئوية) المثبتة (1.5 ٪ غلوتارالدهيد في 1 ٪ السكروز ، 0.1 متر كاكوديلات الصوديوم) في قطع الكبد ونقلها من PBS إلى مثبت لمدة تصل إلى 20 دقيقة في درجة حرارة الغرفة. حافظ دائما على الأنسجة مغمورة في المحاليل لتجنب التجفيف.
ملاحظة: الألدهيدات هي مهيجات قابلة للتآكل ويحتمل أن تكون مسرطنة. كاكوديلات الصوديوم سام إذا تم ابتلاعه أو استنشاقه. يجب التعامل مع جميع المثبتات أثناء ارتداء معدات الحماية الشخصية المناسبة ، ويجب إجراء التجربة في غطاء الدخان. التثبيت الجيد سيؤدي إلى نسيج أكثر صلابة. - قم بإعداد الميكروتوم المهتز باستخدام شفرة وحمام ثلج وصينية عازلة باردة مملوءة ب PBS. قم بتركيب القطعة الأولى من أنسجة الكبد الثابتة على حامل عينة باستخدام غراء سيانو أكريليت ونقل الكتلة إلى الميكروتوم المهتز.
- باتباع توصيات الشركة المصنعة ، اقترب من الأنسجة وقم بتقطيع الكبد الثابت إلى شرائح بسماكة 100 ميكرومتر.
- اجمع الشرائح باستخدام ملعقة أو فرشاة طلاء شعر طبيعية وانقلها إلى طبق مكون من 12 أو 24 بئرا يحتوي على محلول ثلجي بارد (1.5٪ غلوتارالدهيد ، 0.1 مليون كاكوديلات الصوديوم) على الثلج. اترك الشرائح على الثلج حتى يتم تقطيع جميع العينات وتصبح جاهزة للمعالجة بشكل أكبر.
- حدد الشرائح التي تحتوي على المناطق ذات الأهمية لمزيد من المعالجة واغسلها بإثارة لطيفة. قم بإجراء ثلاث غسلات لمدة 5 دقائق مع درجة حرارة الغرفة 0.1 متر كاكوديلات الصوديوم في طبق من 12 أو 24 بئرا ، مما يضمن أن الشرائح تحتوي على مخزن مؤقت كاف للتحرك بحرية.
ملاحظة: بشكل عام، يتم اختيار المناطق ذات الأهمية بناء على السمات التشريحية المتعلقة بالمسألة البيولوجية للدراسة وتسترشد بمناطق الأنسجة التي من المحتمل أن تكون موجودة في السلسلة بأكملها، على سبيل المثال، ليست على حافة القسم، والتي يتم الحفاظ عليها بشكل جيد. - في غطاء الدخان ، استبدل كاكوديلات الصوديوم 0.1 م بأول أكسيد الأوزميوم الطازج بنسبة 1٪ / 1.5٪ من فيروسيانيد البوتاسيوم. ضع الطبق المكون من 12 أو 24 بئرا في حاوية محكمة الإغلاق وانقل الحاوية إلى ثلاجة المواد الكيميائية الخطرة لمدة 1 ساعة.
ملاحظة: الأوزميوم خطير للغاية في حالة الابتلاع والاستنشاق وملامسة الجلد. فيريسيانيد البوتاسيوم هو مهيج وضار عن طريق الاستنشاق وملامسة الجلد. تعامل دائما مع استخدام معدات الحماية الشخصية المناسبة ، وقم بإجراء التجربة في غطاء الدخان. - في غطاء الدخان ، قم بإزالة رابع أكسيد الأوزميوم / فيسيانيد البوتاسيوم إلى زجاجة نفايات الأوزميوم المخصصة ، واغسل العينات لمدة 5 دقائق باستخدام 0.1 مليون كاكوديلات الصوديوم ثلاث مرات. اترك العينات في حاوية مغلقة طوال الليل عند 4 درجات مئوية.
ملاحظة: نقطة توقف مؤقت. يمكن تخزين العينات في كاكوديلات الصوديوم 0.1 M في حاوية مغلقة عند 4 درجات مئوية لأسابيع مع القليل من الضرر للحفظ. تأكد من وجود مخزن مؤقت كاف لمنع التجفيف. - احتضن العينات بحمض التانيك الطازج بنسبة 1٪ في 0.05 M كاكوديلات الصوديوم لمدة 45 دقيقة في الظلام في درجة حرارة الغرفة.
ملاحظة: حمض التانيك هو مهيج ويمكن أن يسبب تلف العين. ارتداء معدات الحماية الشخصية المناسبة وإجراء التجربة في غطاء الدخان. - قم بإجراء ثلاث غسلات لمدة 5 دقائق باستخدام ddH2O قبل الجفاف والتضمين.
2. عينة الجفاف، الراتنج إبون يشبع، وتصاعد
- تحضير راتنج إبون وفقا للنسبة التالية حسب الوزن (انظر الخطوة 2.2). قم بتزيين الميزان بكوب بلاستيكي يمكن التخلص منه سعة 100 مل يحتوي على قضيب تحريك. قطع النهايات من 5 ماصات باستور البلاستيكية التي يمكن التخلص منها واستخدامها لنقل مكونات الراتنج اللزج إلى الدورق.
- أضف بالتتابع 19.2 جم من راتنج-812 و 7.6 جم من DDSA و 13.2 جم من MNA و 0.8 جم من مسرع DMP-30 في الدورق. باستخدام ماصة باستور البلاستيكية النظيفة الخامسة ، امزج مكونات الراتنج جيدا باليد.
ملاحظة: تجنب إدخال الفقاعات ولكن تأكد من الخلط الكافي للراتنج السفلي مع الجزء العلوي لتحقيق تغيير اللون والخلط الخشن للطبقات المكونة. جميع مكونات الراتنج هي مهيجات وضارة عن طريق الاستنشاق وملامسة الجلد. DMP-30 هو تآكل ويمكن أن يسبب تآكل الجلد. ارتداء معدات الحماية الشخصية المناسبة. - ضع الكأس على مقلي مغناطيسي واتركه ليحرك بلطف ، واخلط الراتنج يدويا بشكل دوري.
- اغسل العينات بإثارة لطيفة لمدة 5 دقائق مع 70٪ من الإيثانول ؛ كرر مرة واحدة.
- اغسل العينات بإثارة لطيفة لمدة 5 دقائق مع 90٪ من الإيثانول ؛ كرر مرة واحدة.
- اغسل العينات بإثارة لطيفة لمدة 5 دقائق مع الإيثانول بنسبة 100٪ ؛ كرر مرة واحدة.
- في حين أن العينات في 100٪ يغسل الإيثانول في غطاء الدخان ، قم بإعداد أكسيد البروبيلين (PO) بنسبة 50:50 (v / v): خليط Epon في قارورة زجاجية مع غطاء بلاستيكي مقاوم لأكسيد البروبيلين (PO). قم بالتثبيت بعناية ولكن بشكل آمن على غطاء القارورة الزجاجي ، ومع الحفاظ على الاحتفاظ بكل من الغطاء والقارورة ، رج أو دوامة للخلط.
ملاحظة: أكسيد البروبيلين هو مهيج سام للغاية وقابل للاشتعال يذوب بعض البلاستيك. ارتداء معدات الحماية الشخصية المناسبة وإجراء التجربة في غطاء الدخان. - بعد الخطوة 2.6 ، احتضن العينات باستخدام PO: Epon لمدة 1 ساعة في حاوية مقاومة ل PO (على سبيل المثال ، صواني الألومنيوم أو القوارير الزجاجية) ، مع اهتزاز / إثارة لطيفة في غطاء الدخان.
- في غطاء الدخان ، انقل العينات إلى 100٪ Epon. احتضان لمدة 2 ساعة في درجة حرارة الغرفة في غطاء الدخان مع هزاز / دوران / تحريض. انقل خليط PO:Epon إلى زجاجة نفايات Epon زجاجية مخصصة.
- كرر الخطوة 2.9 مرة واحدة.
- قم بتركيب العينات لتضمينها. اعتمادا على حجم الشرائح والمنطقة محل الاهتمام ، قم بتركيب الشرائح مباشرة على كعوب راتنجية مسبقة البلمرة أو قم بتضمينها بشكل مسطح للتشريح وإعادة تضمينها في وقت لاحق.
ملاحظة: بالنسبة للتضمين المسطح، يمكن استخدام "شريحة المصبوبة" لتضمين العديد من الشرائح في وقت واحد. يمكن استخدام الراتنج المتبقي لملء كبسولات الشعاع وخبزه لصنع كعوب مسبقة البلمرة أو مجمدة للاستخدام لاحقا. - بمجرد تركيبها وتغطيتها بالراتنج الكافي لملء تجويف "المصبوب الشريحة" ، اخبز العينات طوال الليل في فرن 60 درجة مئوية.
ملاحظة: نقطة توقف مؤقت. يمكن تخزين العينات في درجة حرارة الغرفة لسنوات. - لإعادة التضمين، حدد المنطقة ذات الأهمية في شرائح الأنسجة المسطحة المدمجة. باستخدام منشار صائغ ، قم بقطع قطعة الأنسجة ذات الحجم المناسب (1 مم 2 إلى 4 مم 2) وإعادة تضمينها باستخدام الراتنج المحضر ، كما هو الحال في الخطوة2.2 ، على الجزء العلوي من كتلة مسبقة البلمرة واخبزها طوال الليل في فرن 60 درجة مئوية.
ملاحظة: بدلا من ذلك ، يمكن لصق قطعة الأنسجة على كعب أو دبوس مع راتنجات الايبوكسي المكونة من جزأين. اتركيه لضبطه طوال الليل. نقطة توقف محتملة.
3. التشذيب والتقسيم التسلسلي للعينات المضمنة
ملاحظة: التقسيم هو مهارة مكتسبة. يجب أن يكون المستخدمون بارعين في التقسيم الرقيق للغاية قبل محاولة التقسيم التسلسلي. نظرا لأن عناصر التحكم الدقيقة الدقيقة تختلف عبر الشركات المصنعة، فاتبع تعليمات الشركة المصنعة وإرشاداتها.
- مع قفل العينة في محول التشذيب ، استخدم شفرة حلاقة لتقليم الأنسجة المضمنة في الراتنج بعناية لتلبية المعايير التالية (انظر الشكل 1A ، B):
- تأكد من وجود سطح علوي مستو يعرض الأنسجة حول المنطقة محل الاهتمام.
- تأكد من شكل شبه منحرف مع الحواف العلوية والسفلية نظيفة ومتوازية.
- ضمان الأبعاد الإجمالية من 200-500 ميكرومتر في x ، 100-500 ميكرومتر في y.
- تأكد من وجود وجه كتلة غير متماثل ، على سبيل المثال ، زوايا الجانب الأيمن من ~ 90 درجة ، والزاوية العلوية اليسرى منفرجة ، والزاوية السفلية اليسرى حادة.
ملاحظة: يمكن أن تكون السكين المبردة المشذبة أداة بديلة لشفرة الحلاقة. التوصيات الأخرى هي لراحة المستخدم لطلب الأقسام عند التصوير. اختياري: إذا فشلت المقاطع في تكوين شرائط ثابتة، يمكن تطبيق أسمنت تلامس على الحافة الأمامية لوجه الكتلة للمساعدة في تكوين الشريط. شفرات الحلاقة حادة. احرص على الاحتفاظ بشفرة الحلاقة بحيث لا يؤدي الانزلاق العرضي إلى ضرر شخصي.
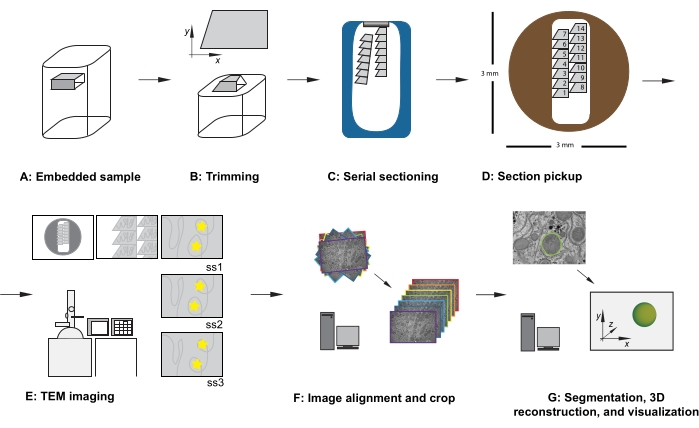
الشكل 1: سير عمل TEM للمقطع التسلسلي . (أ) رسم تخطيطي للعينة في كتلة الراتنج. (ب) تقليم الكتلة لتوليد شكل شبه منحرف مع حواف مناسبة للتقسيم التسلسلي ووجه كتلة غير متماثل لضمان الاتجاه المعروف. (ج) رسم بياني يوضح شرائط المقاطع التسلسلية، العائمة على سطح الماء في قارب السكين الماسي. (د) رسم تخطيطي يوضح تنظيم المقطع والشريط، مع إملاء ترتيب الأقسام، على شبكة فتحة TEM قطرها 3 مم. (ه) التصوير والملاحة TEM. إظهار الشريط وترتيب الأقسام واستخدام "ملصقات النجمة الصفراء" على الشاشة للرجوع إليها على الشاشة لضمان إعادة تصوير نفس المنطقة ذات الاهتمام في الأقسام اللاحقة. (و) محاذاة الصورة واقتصاصها. (ز) التجزئة ، وإعادة الإعمار 3D ، والتصور. اختصار: TEM = المجهر الإلكتروني للإرسال. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
- بمجرد القص ، انقل قوس العينة ، مع تشاك والعينة ، إلى ذراع العينة في الميكروتوم ، مع وضع قوس العينة بحيث يمتد نطاق انتقال القوس من الأعلى إلى الأسفل ؛ تأمين قوس العينة في مكانه.
- ضع السكين الماسي واقفله في حامل السكين ، مما يضمن ضبط زاوية القطع بشكل مناسب على السكين. قفل حامل السكين في المسرح بإحكام.
- مع إضاءة المسرح ، استخدم السكين المتقدم أثناء التحقق باستمرار من العلاقة بين وجه الكتلة وحافة السكين. تقدم السكين بحذر نحو العينة ، مع ضبط الزاوية الجانبية للسكين باستمرار ، وإمالة العينة ، ودوران العينة عن طريق ضبط المقابض ذات الصلة حتى تتم محاذاة الكتلة إلى حافة السكين.
- إيقاف تشغيل إضاءة المسرح ؛ قم بتشغيل إضاءة المسرح ؛ تعيين الجزء العلوي والسفلي من نافذة القطع لذراع العينة ؛ واترك العينة أسفل حافة السكين مباشرة.
- املأ قارب السكين ب ddH2O نظيف وتأكد من أن سطح الماء مستو مع حافة السكين ومقعر قليلا.
- اختياري: اغمس رمشا في 0.1٪ Triton X-100 ثم في ماء قارب السكين لتقليل التوتر السطحي للمياه للمساعدة في تشكيل الكلورو والتقاط الشريط.
- قم بإعداد محطة العمل باستخدام الرموش (رموش ملتصقة بعصا كوكتيل) ، وشبكات فتحات مغلفة بالفورمفار ، وملقط كروس أوفر مكتوب عليه ، وكلوروفورم ، ومحلول Triton X-100 بنسبة 0.1٪ ، وماء مقطر ، وورق تصفية ، وصندوق شبكة مع ملاحظات صندوق الشبكة.
- اضبط سرعة القطع على 1 مم / ثانية وسماكة القطع الأولية على 100 نانومتر وابدأ دورة القطع.
- بعد قطع القسم الأول ، قم بتغيير معلمات القطع إلى سرعة قطع عند 0.8 مم / ثانية وسمك القطع إلى 70 نانومتر ، واستمر في القطع ، مما يسمح للأقسام بتشكيل شريط يتحرك أسفل سطح قارب السكين المملوء بالماء (الشكل 1C).
ملاحظة: من المهم أن تكون على دراية بلون الأقسام التي يتم إنتاجها لأن هذا غالبا ما يكون دليلا أكثر دقة لسمك أقسام الراتنج. عادة ما يكون سمك المقاطع الفضية حوالي 70 نانومتر ، في حين أن الأقسام الرمادية أرق وأقسام الذهب أكثر سمكا. - اسمح للميكروتوم بمواصلة قص المقاطع والشريط لفترة أطول.
ملاحظة: من المهم تجنب الاهتزازات الكبيرة والاضطرابات الجسدية في الغرفة. يمكن أن تتسبب المسودات في تحرك الأقسام على سطح الماء في قارب السكين ، ويمكن أن تتسبب الاهتزازات الفيزيائية في قطع الميكروتوم بشكل غير متساو. - بمجرد جمع أقسام كافية وقبل أن يصل الشريط إلى نهاية القارب ، أوقف القطع (بعد أن تتجاوز العينة حافة السكين مباشرة).
ملاحظة: يعتمد عدد المقاطع المطلوبة على حجم وجه الكتلة وحجم مجموعة البيانات المراد تجميعها. وبالتالي ، من المفيد أن تكون على دراية بالعلاقة بين حجم الكتلة وشبكة الفتحة أثناء خروج أقسام القطع. - باستخدام رمش في كل يد، قم بكسر الشريط برفق إلى شرائط أصغر يمكن أن تتناسب مع طول شبكة الفتحة، مع الحرص على تدوين مواضعها النسبية من داخل العينة.
ملاحظة: إذا كان عرضها المجمع مناسبا، يمكن وضع شرائط متعددة برفق بجانب بعضها البعض والتقاطها معا في شبكة فتحة واحدة. في حالة التقاط شرائط متعددة على شبكة فتحة واحدة، انتبه إلى الترتيب والموضع النسبي للشرائط. على سبيل المثال، ضع دائما الشرائط في العينة على يسار الشريط الموجود بالفعل في العينة (الشكل 1D). - اختياري: باستخدام قضيب قضيب زجاجي ، مرر قطرة من الكلوروفورم فوق الأقسام لتسطيحها.
ملاحظة: الكلوروفورم سام ومهيج. لا تدع الكلوروفورم يلمس سطح الماء أو أقسامه. إذا حدث ذلك عن طريق الخطأ ، فيجب إزالة الماء وغسل السكين قبل العودة إلى التقسيم. يمكن للكلوروفورم إتلاف الأقسام وتحلل الغراء الذي يؤمن الماس في قارب السكين. - باستخدام أول ملقط مرقم ، التقط أول شبكة فتحة فارغة (على الجانب الأيمن من الفتحة ، جانب الفورمفار لأسفل) ، وتراجع بلطف في Triton X-100 ، ثم مرتين في الماء المقطر قبل إزالة الماء الزائد من حافة الملقط باستخدام قطعة من ورق التصفية.
- مع رمش في يد واحدة والملقط في اليد الأخرى ، قم بخفض ما يقرب من 2/3 من شبكة الفتحة المطلية بالفورمفار بلطف في مياه قارب السكين (بعيدا عن الأقسام) ، بحيث يكون جانب الفورمفار متجها لأسفل ، والحافة الطويلة اليمنى للفتحة على سطح الماء وموازية لحافة الماء.
- قم بتحريك الشبكة بلطف في الماء نحو الشرائط بحيث تنجرف الأقسام نحو الشبكة عند ضربة العودة. استمر في القيام بذلك في الفتحات الأصغر والأصغر حجما حتى تصطف الحافة اليمنى للشريط مع الحافة اليمنى للفتحة. ثم ، مع آخر لوح ، أحضر الشبكة برفق لالتقاط الأقسام في شبكة الفتحة.
- اترك الشبكة في الملقط لتجف قبل تخزينها في مربع الشبكة ، مع التعليق عليها بشكل مناسب على الورقة المرجعية لمربع الشبكة.
- كرر الخطوة 3.16 حتى يتم جمع كافة الشرائط، مما يضمن الحفاظ على ترتيب الشرائط.
- إذا كانت هناك حاجة إلى أقسام أخرى ، فقم بسحب السكين 150 نانومتر أو نحو ذلك ، وتحقق من مستوى الماء في القارب ، وأضف المزيد إذا لزم الأمر. ابدأ عملية القطع مرة أخرى، باتباع الخطوات 3.11-3.18.
- بمجرد جمع جميع الأقسام ، تأكد من أن حافة السكين خالية من حطام القسم ، وسحب السكين بعيدا عن وجه الكتلة ، وإزالة السكين وتنظيفه.
4. تلطيخ الشبكة
- بمجرد أن تجف ، قم بتلطيخ الأقسام بسيترات الرصاص من رينولدز إما على بارافيلم على المقعد أو في طبق بتري. ضع عدة كريات من هيدروكسيد الصوديوم تحت غطاء طبق بتري لتوفير بيئة خالية من ثاني أكسيد الكربون. ثم ، بعناية ، بعيدا عن الكريات ، ماصة 40 ميكرولتر قطرات من سيترات الرصاص رينولدز على parafilm ، واحد لكل شبكة.
ملاحظة: لا تلطخ الكثير من الشبكات في وقت واحد. على سبيل المثال ، يجب أن يكون الحد الأقصى 6. حاول ألا تتنفس مباشرة على طبق التلطيخ. يمكن أن يتفاعل ثاني أكسيد الكربون مع سترات الرصاص ويسبب راسبا غير مرغوب فيه على الشبكات. - اقلب كل شبكة (جانب المقطع لأسفل) على قطرة سترات الرصاص واتركها محمية بغطاء طبق بتري لمدة 7 إلى 10 دقائق. في حين أن الشبكات ملطخة ، قم بإعداد قطعة ثانية أكبر من البارافيلم على المقعد مع خمس قطرات 300 ميكرولتر من الماء المقطر لكل شبكة.
- في نهاية حضانة سيترات الرصاص ، انقل كل شبكة إلى قطرة من الماء المقطر لغسلها لمدة 1 دقيقة دون التنفس مباشرة على الشبكات.
- كرر الخطوة 4.3 ما مجموعه خمس مرات.
- باستخدام ملقط كروس أوفر مرقم، التقط الشبكة الأولى، والمس حافة الشبكة لتصفية الورق لإطفاء معظم الماء، واتركه حتى يجف في الملقط (لمدة 20 دقيقة على الأقل). كرر ذلك لكل شبكة.
5. اقتناء التصوير من قبل TEM
ملاحظة: نظرا لأن عناصر تحكم TEM الدقيقة تختلف من جهة مصنعة، فاتبع إرشادات الشركة المصنعة وإرشاداتها. يجب تنفيذ الخطوات التالية من قبل المستخدمين الذين يتقنون بالفعل استخدام TEM.
- قبل التصوير ، قم بإجراء الفحوصات المعتادة ، على سبيل المثال ، محاذاة الشعاع ، واكتساب المراجع ، وعينة eucentricity.
- قم بتحميل الشبكة الأولى من الأقسام التسلسلية بعناية في حامل العينة ، مع الحرص على محاذاة الفتحة (وبالتالي الأقسام) مع المحور الرأسي لمرحلة المجهر.
ملاحظة: هذه الدقة ليست ضرورية ولكنها توفر الوقت أثناء مراحل الحصول على البيانات ومعالجتها في المستقبل. عند إدخال الشبكة (قسم الجانب لأسفل أو الجانب المقطع لأعلى)، احرص على تصوير جميع الشبكات في نفس الاتجاه. - عند التكبير المنخفض، راقب ترتيب الأقسام التسلسلية وموقعها وموضعها (الشكل 1E). انتقل إلى القسم الأوسط من السلسلة على الشبكة.
ملاحظة: اعتمادا على هدف البحث الدقيق ، قد تختلف أساليب التصوير ؛ ومع ذلك ، فإن ما يلي هو نقطة انطلاق مفيدة. يملي شكل الأقسام وعلاقة الشرائط (كما تم التقاطها في الخطوة 3.14) القسم الذي كان أولا والقسم الأخير على الشبكة. - تصفح العينة وحدد منطقة الاهتمام. راقب العينة عند التكبير المطلوب، وفكر في جمع السلسلة بتكبير أقل قليلا، حيث غالبا ما تكون الأقسام غير محاذاة تماما، وقد تحتاج الصور إلى اقتصاصها لاحقا.
- التقط صورا مرجعية بتكبيرات أقل لتقدير سياق المنطقة محل الاهتمام ، وموقعها التقريبي عند تكبيرات مختلفة فيما يتعلق بحدود القسم ، والمعالم البارزة داخل العينة. استخدمها لوضع علامة على المنطقة ذات الاهتمام في أقسام أخرى.
- اختياري: للإشارة إلى الشاشة، استخدم معجون لاصق قابل لإعادة الاستخدام أو ملصقات أو قطعة من ورق جهاز العرض العلوي (OHP)، مثبت على الشاشة، لوضع علامات مؤقتة على الشاشة للسماح بإعادة التصوير الروتيني لنفس ميزات المنطقة ذات الأهمية في مركز الصورة، في جميع أنحاء مجموعة البيانات (انظر النجوم الصفراء في الشكل 1E).
- باستخدام الصور المرجعية ، انتقل إلى المنطقة ذات الاهتمام في القسم الأول من الشبكة ، واحصل على صورة بالتكبير المطلوب.
ملاحظة: عند حفظ الصور، قم بتدوين اسم الملف الأول للصورة الأولى من السلسلة، واستخدم تسميات التسمية المتسلسلة بحيث تتبع كافة أسماء الصور الترتيب التسلسلي للمقاطع التسلسلية. - انتقل إلى القسم التالي وكرر الخطوة 5.7 حتى يتم تصوير جميع الأقسام لتلك المنطقة محل الاهتمام.
6. تصدير الصور وتسجيل محاذاة القسم التسلسلي
- تصدير ملفات الصور التي تنتمي إلى نفس المكدس إلى مجلد واحد. تأكد من فرز المجلد حسب اسم الملف.
ملاحظة: من الناحية المثالية يجب أن يكون للصور نفس اسم الجذر وأن تتبع الترتيب الذي تم الحصول عليها به. - افتح فيجي وانقر على ملف | | الاستيراد تسلسل الصورة.
- انقر على الصورة الأولى من المجلد وانقر فوق فتح. انتظر حتى تظهر نافذة منبثقة من خيارات التسلسل (الشكل 2A).
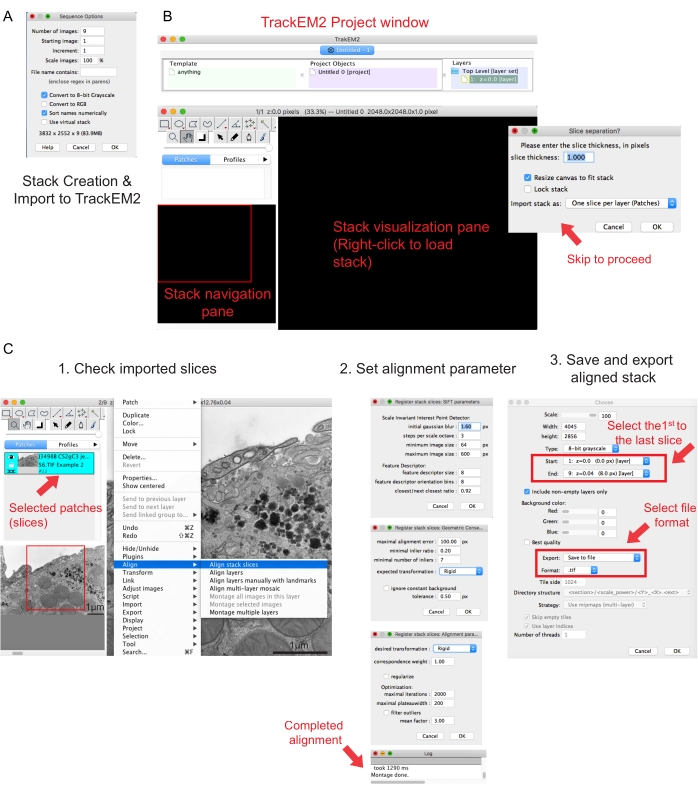
الشكل 2: إنشاء مكدس تسلسلي ومحاذاة مقطع تسلسلي باستخدام فيجي. (أ) لقطة شاشة تعرض خيارات التسلسل عند تحميل الصور لإنشاء مكدس تسلسلي. (B) لقطة شاشة للمكون الإضافي TrackEM2 والنوافذ الرئيسية للمكون الإضافي. اضغط على موافق ( OK) في فصل الشريحة لمتابعة المحاذاة. (C) لقطة شاشة بعد تحميل المكدس التسلسلي بنجاح في جزء المرئيات. ستظهر ثلاث نوافذ متسلسلة لمعلمات المحاذاة بمجرد تحديد محاذاة شرائح المكدس. تصدير المكدس المحاذاة بمجرد اكتمال المحاذاة. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
- انقر فوق فرز الأسماء رقميا | تحويل إلى خيار 8 بت . اضغط على موافق.
ملاحظة: يساعد التحويل إلى 8 بت على استيراد البيانات إلى Amira ويقلل من حجم الملف، مما يسمح بسرعات معالجة أسرع في الخطوات اللاحقة. - تحقق من اكتمال المكدس الذي تم إنشاؤه وتسلسله وتكبيره. احفظ المكدس الذي تم إنشاؤه كملف .tif.
ملاحظة: يجب أن تكون الصور قد تم الحصول عليها بنفس التكبير. - قم بتنفيذ المكون الإضافي TrakEM241. انتقل إلى | الملفات | الجديدة TrackEM2 (فارغ).
ملاحظة: سيطلب المكون الإضافي من المستخدم حفظ ملفات TrackEM2. إذا لزم الأمر، احفظ ملفات TrackEM2 في مجلد الصور. يجب أن تظهر ثلاث نوافذ: نافذة مشروع، ونافذة تنقل مكدس (يسار)، وجزء مرئيات مكدس (الشكل 2B). - انقر بزر الماوس الأيمن للوصول إلى جزء المرئيات الأسود. انقر على استيراد | استيراد مكدس وتحديد المكدس الذي تم إنشاؤه مسبقا.
- انقر فوق موافق لتحميل المكدس إلى نافذة التنقل في المكدس.
ملاحظة: ستظهر نافذة فصل شريحة لطلب علاقة البكسل والبعد. لمحاذاة المكدس فقط، انقر فوق موافق لتخطي هذه الخطوة. - استخدم شريط التمرير للتحقق من جميع شرائح المكدس. ابحث عن الشريحة المحملة، والتي ستظهر كتصحيح في خطة التنقل. حدد التصحيحات التي سيتم تضمينها في المحاذاة التالية.
ملاحظة: ستتحول البقع المحددة إلى اللون الأزرق. - مرر مؤشر الماوس فوق جزء العرض. انقر بزر الماوس الأيمن فوق الصورة، وحدد محاذاة | محاذاة شرائح المكدس (الشكل 2C-1).
- حدد معلمات المحاذاة من خلال مجموعة من ثلاث نوافذ متتابعة.
ملاحظة: بالنسبة لمعظم البيانات، ابدأ بمحاذاة جامدة (تسمح بالدوران والترجمة ولكن ليس التحويل) واحتفظ بالمعلمات الأخرى كافتراضية (الشكل 2C-2). - اسمح بتشغيل المحاذاة حتى يشير سجل القراءة إلى أن المونتاج قد تم.
ملاحظة: يعتمد وقت التشغيل على عدد voxels وسرعة الكمبيوتر. - تحقق من المكدس المحاذي في جزء العرض. اضغط على المفاتيح Alt و - (في الكمبيوتر الشخصي) أو المفاتيح Ctrl و - (في Mac) للحصول على طريقة عرض تصغير للمكدس المحاذاة.
- إذا كنت راضيا عن المكدس المحاذاة، فانقر بزر الماوس الأيمن فوق تصدير | قم بعمل صورة مسطحة لحفظ المكدس المحاذاة.
- حدد الصورة الأولى كبداية للمكدس والصورة الأخيرة كنهاية المكدس، انقر فوق موافق (الشكل 2C-3). احفظ المكدس المحاذي .tif.
ملاحظة: لتقليل حجم الملف، اقتصاص البيانات لتحتوي فقط على المنطقة الضرورية للاهتمام. - إذا لزم الأمر، قم بتنفيذ محاذاة affine على المكدس المحاذاة. افتح المكدس المحاذي في فيجي ، وحدد المكون الإضافي | | التسجيل ستاك ريج.
- اختر خيار affine واضغط على OK. انتظر حتى يكتمل البرنامج.
- احفظ المكدس المحاذي ل affine باسم ملف مختلف.
7. التجزئة وإعادة الإعمار 3D
- افتح أميرة42. انقر فوق ملف | افتح البيانات لتحميل المكدس المحاذاة.
- حدد قياسات voxel في النافذة المنبثقة الجديدة (الشكل 3A).
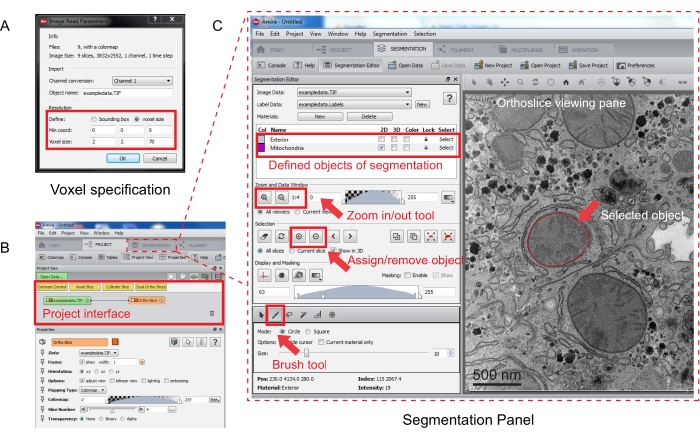
الشكل 3: تجزئة المكدس التسلسلي باستخدام Amira. (A) نافذة منبثقة لتعريف Voxel قبل تحميل مكدس محاذاة. (ب) لقطة شاشة لواجهة المشروع بعد استيراد مكدس. حدد علامة التبويب تجزئة لبدء تتبع الكائن في لوحة محرر التجزئة. (ج) السمات الرئيسية لعلامة التبويب التجزئة. حدد الكائنات للتجزئة في قسم محرر التجزئة من علامة التبويب تجزئة. استخدم وظيفة التكبير/التصغير للمساعدة في تحديد الكائنات. حدد أداة الفرشاة وتتبع حدود الكائن. انقر فوق الرمز + ضمن تحديد لتعيين التتبع. سيظهر كائن معين له حد أحمر في جزء عرض الشريحة التقويمية. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
ملاحظة: ستظهر عقدة مكدس الصور في واجهة المشروع، وستظهر شريحة تقويم العظام في جزء العرض على اليمين (الشكل 3B).
- لبدء التجزئة، حدد علامة التبويب تجزئة (الشكل 3B).
ملاحظة: يوصى بحفظ تقدم التقسيم قبل وأثناء التقسيم. انتقل إلى نموذج | حفظ النموذج كأي ملف .am يناسبك. - انقر جديد في لوحة محرر التقسيم لتعريف كائنات جديدة في قائمة المواد. انقر بزر الماوس الأيمن لتغيير لون الكائن وانقر نقرا مزدوجا لإعادة تسمية الكائن.
- بالنسبة للتجزئة اليدوية، اختر أداة التقسيم أسفل قائمة المواد. حدد أداة الفرشاة الافتراضية لتمييز البيكسلات (الشكل 3C).
ملاحظة: بدلا من ذلك، استخدم أداة الفرشاة لتتبع المخطط التفصيلي للكائن واضغط على Shift + F لتعبئة الكائن. - لتحويل أداة الفرشاة إلى ممحاة، اضغط باستمرار على Ctrl أثناء تحديد البيكسلات للتصحيح. إضافة تعليق توضيحي إلى كل شريحة في المكدس.
- بمجرد التأكيد، قم بتعيين التحديد إلى تسمية بالنقر فوق علامة + . انقر فوق علامة - لإزالة التحديد.
- ارجع إلى واجهة المشروع بمجرد اكتمال التجزئة. ابحث عن عقدة ذات امتداد ".label" متصل بمكدس الصور.
- انقر بزر الماوس الأيمن فوق الملحق ".label" وحدد إنشاء جهاز Surface | تطبيق لإنشاء ملف.surf.
- لعرض نموذج 3D لكائن مجزأ، انقر بزر الماوس الأيمن فوق ملف .surf وحدد طريقة عرض Surface لإنشاء نموذج 3D في جزء العرض.
- حفظ نموذج 3D للتصور أو مزيد من التحليلات الكمية.
Access restricted. Please log in or start a trial to view this content.
النتائج
بالنسبة لهذه التقنية ، يتم اختيار المناطق ذات الأهمية بناء على هدف البحث البيولوجي وتحديدها قبل تقليم وتقسيم الأنسجة المدمجة. وبالمثل ، قد يملي سؤال البحث حجم وجه الكتلة ؛ في هذه الحالة ، تم قص العينة لترك وجه كتلة يبلغ حوالي 0.3 مم × 0.15 مم (الشكل 4A). سمح ذلك بشبكتين من 9 أقسام ?...
Access restricted. Please log in or start a trial to view this content.
Discussion
يتم وصف تقنية vEM التي يمكن الوصول إليها لتصور بنية العضية والتفاعلات في 3D في هذا البروتوكول. يتم تقديم مورفولوجيا الاتصالات بين الأعضاء في خلايا الكبد كدراسة حالة هنا. ومع ذلك ، تم تطبيق هذا النهج أيضا للتحقيق في مجموعة متنوعة من العينات والمجالات البحثية الأخرى ، بما في ذلك التفاعلات بين ?...
Access restricted. Please log in or start a trial to view this content.
Disclosures
وليس لدى المؤلفين أي تضارب في المصالح للإفصاح عنه.
Acknowledgements
ونشكر جوانا هانلي وريبيكا فياديرو وأنيا ستراتمان - إيوانوفسكا على المساعدة التقنية الخبيرة. كما نشكر أعضاء مختبر ستيفان وإيان ج. وايت على المناقشات المفيدة. J.J.B. مدعوم من تمويل MRC لمختبر MRC لبيولوجيا الخلايا الجزيئية في UCL ، رمز الجائزة MC_U12266B. يتم دعم C.J.S. من خلال تمويل MRC لمختبر MRC لوحدة جامعة البيولوجيا الخلوية الجزيئية في UCL ، رمز الجائزة MC_UU_00012/6. يتم تمويل P.G. من قبل مجلس البحوث الأوروبي ، رمز المنحة ERC-2013-StG-337057.
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| 0.22 µm syringe filter | Sarstedt | 83.1826.001 | |
| Aluminum trays | Agar Scientific | AGG3912 | |
| Amira v6 | ThermoFisher | https://www.thermofisher.com | |
| Chloroform | Fisher | C/4960/PB08 | |
| DDSA/Dodecenyl Succinic Anhydride | TAAB | T027 | Epon ingredient |
| Diamond knife | DiaTOME | ultra 45° | |
| DMP-30/2,4,6-tri (Dimethylaminomethyl) phenol | TAAB | D032 | Epon ingredient |
| Dumont Tweezers N5 | Agar Scientific | AGT5293 | |
| Fiji | https://imagej.net/ | ||
| Fiji TrakEM2 plugin | https://imagej.net/ | ||
| Formaldehyde 36% solution | TAAB | F003 | |
| Formvar coated slot grid | Homemade | Alternative: EMS diasum (FF2010-Cu) | |
| Glass bottle with applicator rod | Medisca | 6258 | |
| Glass vials | Fisher Scientific | 15364769 | |
| Gluteraldehyde 25% solution | TAAB | G011 | |
| MNA/Methyl Nadic Anhydride | TAAB | M011 | Epon ingredient |
| Osmium Tetroxide 2% solution | TAAB | O005 | |
| Potassium Ferricyanide | Sigma-Aldrich | P-8131 | |
| Propylene oxide | Fisher Scientific | E/0050/PB08 | |
| Reuseable adhesive | Blue Tack | ||
| Reynolds Lead Citrate | TAAB | L037 | Section stain |
| Sodium Cacodylate | Sigma-Aldrich | C-0250 | to make 0.1 M Caco buffer |
| Super Glue | RS Components | 918-6872 | Cyanoacrylate glue, Step 1.3 |
| TAAB 812 Resin | TAAB | T023 | Epon ingredient |
| Tannic acid | TAAB | T046 | |
| Triton X-100 | Sigma-Aldrich | T9284 | |
| Two part Epoxy Resin | RS Components | 132-605 | Alternative: Step 2.13 |
| Ultramicrotome | Leica | UC7 | |
| Vibrating microtome | Leica | 100 µm thick slices, 0.16 mm/s cutting at 1 mm amplitude . | |
| Weldwood Original Contact cement | DAP | 107 | Contact adhesive: Step 3.1.4 |
References
- Knoll, M., Ruska, E. Das elektronenmikroskop. Zeitschrift für Physik. 78 (5), 318-339 (1932).
- von Ardenne, M. Daselektronen-rastermikroskop. Zeitschrift für Physik. 109 (9), 553-572 (1938).
- Bang, B. H., Bang, F. B. Graphic reconstruction of the third dimension from serial electron microphotographs. Journal of Ultrastructure Research. 1 (2), 138-139 (1957).
- Birch-Andersen, A. Reconstruction of the nuclear sites of Salmonella typhimurium from electron micrographs of serial sections. Journal of General Microbiology. 13 (2), 327-329 (1955).
- Denk, W., Horstmann, H. Serial block-face scanning electron microscopy to reconstruct three-dimensional tissue nanostructure. PLoS Biology. 2 (11), 329(2004).
- Peddie, C. J., Collinson, L. M. Exploring the third dimension: volume electron microscopy comes of age. Micron. 61, 9-19 (2014).
- Titze, B., Genoud, C. Volume scanning electron microscopy for imaging biological ultrastructure. Biology of the Cell. 108 (11), 307-323 (2016).
- Kornfeld, J., Denk, W. Progress and remaining challenges in high-throughput volume electron microscopy. Current Opinion in Neurobiology. 50, 261-267 (2018).
- Heymann, J. A., et al. Site-specific 3D imaging of cells and tissues with a dual beam microscope. Journal of Structural Biology. 155 (1), 63-73 (2006).
- Knott, G., Marchman, H., Wall, D., Lich, B. Serial section scanning electron microscopy of adult brain tissue using focused ion beam milling. Journal of Neuroscience. 28 (12), 2959-2964 (2008).
- Leighton, S. B. SEM images of block faces, cut by a miniature microtome within the SEM - a technical note. Scanning Electron Microscopy. , 73-76 (1981).
- Martone, M. E., Deerinck, T. J., Yamada, N., Bushong, E., Ellisman, M. H. Correlated 3D light and electron microscopy: use of high voltage electron microscopy and electron tomography for imaging large biological structures. Journal of Histotechnology. 23 (3), 261-270 (2000).
- Micheva, K. D., Smith, S. J. Array tomography: a new tool for imaging the molecular architecture and ultrastructure of neural circuits. Neuron. 55 (1), 25-36 (2007).
- Sjostrand, F. S. Ultrastructure of retinal rod synapses of the guinea pig eye as revealed by three-dimensional reconstructions from serial sections. Journal of Ultrastructure Research. 2 (1), 122-170 (1958).
- Ware, R. W. Three-dimensional reconstruction from serial sections. International Review of Cytology. 40, 325(1975).
- Stevens, J. K., Davis, T. L., Friedman, N., Sterling, P. A systematic approach to reconstructing microcircuitry by electron microscopy of serial sections. Cognitive Brain Research. 2 (3), 265-293 (1980).
- Hoppe, W. Three-dimensional electron microscopy. Annual Review of Biophysics. 10, 563-592 (1981).
- Frank, J. Electron tomography: methods for three-dimensional visualization of structures in the cell. , Springer. New York, NY. (2008).
- Baumeister, W. Electron tomography: towards visualizing the molecular organization of the cytoplasm. Current Opinion in Structural Biology. 12 (5), 679-684 (2002).
- Hoog, J. L., Schwartz, C., Noon, A. T., O'Toole, E. T. Organization of interphase microtubules in fission yeast analyzed by electron tomography. Developmental Cell. 12 (3), 349-361 (2007).
- Harris, K. M., Perry, E., Bourne, J., Feinberg, M., Ostroff, L., Hurlburt, J. Uniform serial sectioning for transmission electron microscopy. Journal of Neuroscience. 26 (47), 12101-12103 (2006).
- Jesior, J. C. Use of low-angle diamond knives leads to improved ultrastructural preservation of ultrathin sections. Scanning Microscopy Supplement. 3, 147-152 (1989).
- Studer, D., Gnaegi, H. Minimal compression of ultrathin sections with use of an oscillating diamond knife. Journal of Microscopy. 197, 94-100 (2000).
- Gay, H., Anderson, T. F. Serial sections for electron microscopy. Science. 120 (3130), 1071-1073 (1954).
- Bernhard, W., Rouiller, C. Close topographical relationship between mitochondria and ergastoplasm of liver cells in a definite phase of cellular activity. The Journal of Biophysical and Biochemical Cytology. 2, 73-78 (1956).
- Palade, G. E. An electron microscope study of the mitochondrial structure. The Journal of Histochemistry & Cytochemistry. 1 (4), 188-211 (1953).
- Wu, H., Carvalho, P., Voeltz, G. K. Here, there, and everywhere: The importance of ER membrane contact sites. Science. 361 (6401), (2018).
- Vance, J. E. Inter-organelle membrane contact sites: implications for lipid metabolism. Biology Direct. 15 (1), 24(2020).
- Stefan, C. J. Endoplasmic reticulum-plasma membrane contacts: Principals of phosphoinositide and calcium signaling. Current Opinion in Cell Biology. 63, 125-134 (2020).
- Zaman, M. F., Nenadic, A., Radojicic, A., Rosado, A., Beh, C. T. Sticking with it: ER-PM membrane contact sites as a coordinating nexus for regulating lipids and proteins at the cell cortex. Frontiers in Cell and Developmental Biology. 8, 675(2020).
- van Vliet, A. R., Sassano, M. L., Agostinis, P. The unfolded protein response and membrane contact sites: tethering as a matter of life and death. Contact. 1, 1-15 (2018).
- Cohen, S., Valm, A. M., Lippincott-Schwartz, J. Interacting organelles. Current Opinion in Cell Biology. 53, 84-91 (2018).
- Hariri, H., et al. Lipid droplet biogenesis is spatially coordinated at ER-vacuole contacts under nutritional stress. EMBO Reports. 19 (1), 57-72 (2018).
- Stefan, C. J., Trimble, W. S., Grinstein, S., Drin, G. Membrane dynamics and organelle biogenesis-lipid pipelines and vesicular carriers. BMC Biology. 15 (1), 102(2017).
- Eisenberg-Bord, M., Shai, N., Schuldiner, M., Bohnert, M. A tether is a tether is a tether: tethering at membrane contact sites. Developmental Cell. 39 (4), 395-409 (2016).
- Scorrano, L., De Matteis, M. A., Emr, S., Giordano, F. Coming together to define membrane contact sites. Nature Communications. 10 (1), 1287(2019).
- Lak, B., Li, S., Belevich, I., Sree, S. Specific subdomain localization of ER resident proteins and membrane contact sites resolved by electron microscopy. European Journal of Cell Biology. 100 (7), 151180(2021).
- Collado, J., Kalemanov, M., Campelo, F., Bourgoint, C. Tricalbin-mediated contact sites control ER curvature to maintain plasma membrane integrity. Developmental Cell. 51 (4), 476-487 (2019).
- West, M., Zurek, N., Hoenger, A., Voeltz, G. K. A 3D analysis of yeast ER structure reveals how ER domains are organized by membrane curvature. Journal of Cell Biology. 193 (2), 333-346 (2011).
- Ilacqua, N., Anastasia, I., Raimondi, A., Lemieux, P. A three-organelle complex made by wrappER contacts with peroxisomes and mitochondria responds to liver lipid flux changes. Journal of Cell Science. 135 (5), 259091(2022).
- Cardona, A., Saalfeld, S., Schindelin, J., Arganda-Carreras, I. TrakEM2 software for neural circuit reconstruction. PLoS One. 7 (6), 38011(2012).
- Stalling, D., Westerhoff, M., Hege, H. -C. Amira: A highly interactive system for visual data analysis. The Visualization Handbook. 38, 749-767 (2005).
- Hsieh, T. S., Chen, Y. J., Chang, C. L., Lee, W. R., Liou, J. Cortical actin contributes to spatial organization of ER-PM junctions. Molecular Biology of the Cell. 28 (23), 3171-3180 (2017).
- Anastasia, I., Ilacqua, N., Raimondi, A., Lemieux, P. Mitochondria-rough-ER contacts in the liver regulate systemic lipid homeostasis. Cell Reports. 34 (11), 108873(2021).
- Cattin, A. L., Burden, J. J., Van Emmenis, L., Mackenzie, F. E. Macrophage-Induced Blood Vessels Guide Schwann Cell-Mediated Regeneration of Peripheral Nerves. Cell. 162 (5), 1127-1139 (2015).
- Lopes-da-Silva, M., et al. A GBF1-dependent mechanism for environmentally responsive regulation of ER-Golgi transport. Developmental Cell. 49 (5), 786-801 (2019).
- Banushi, B., Forneris, F., Straatman-Iwanowska, A., Strange, A. Regulation of post-Golgi LH3 trafficking is essential for collagen homeostasis. Nature Communications. 7, 12111(2016).
- Rey, S. A., et al. Ultrastructural and functional fate of recycled vesicles in hippocampal synapses. Nature Communications. 6, 8043(2015).
- Belicova, L., Repnik, U., Delpierre, J., Gralinska, E. Anisotropic expansion of hepatocyte lumina enforced by apical bulkheads. Journal of Cell Biology. 220 (10), 202303003(2021).
- Kizilyaprak, C., Daraspe, J., Humbel, B. M. Focused ion beam scanning electron microscopy in biology. Journal of Microscopy. 254 (3), 109-114 (2014).
- Xu, C. S., Hayworth, K. J., Lu, Z., Grob, P. Enhanced FIB-SEM systems for large-volume 3D imaging. Elife. 6, 1-36 (2017).
- Parlakgül, G., Arruda, A. P., Cagampan, E., Pang, S. High resolution 3D imaging of liver reveals a central role for subcellular architectural organization in metabolism. bioRxiv. , (2020).
- Guerin, C. J., Kremer, A., Borghgraef, P., Lippens, S. Targeted studies using serial block face and focused ion beam scan electron microscopy. The Journal of Visualized Experiments: JoVE. (150), e59480(2019).
- Kremer, A., et al. A workflow for 3D-CLEM investigating liver tissue. Journal of Microscopy. 281 (3), 231-242 (2021).
- Hayat, M. Principles and techniques of electron microscopy: biological applications. , Cambridge University Press. (2000).
- Wisse, E., Braet, F., Duimel, H., Vreuls, C. Fixation methods for electron microscopy of human and other liver. World Journal of Gastroenterology. 16 (23), 2851-2866 (2010).
- Hanley, J., Dhar, D. K., Mazzacuva, F., Fiadeiro, R. Vps33b is crucial for structural and functional hepatocyte polarity. Journal of Hepatology. 66 (5), 1001-1011 (2017).
- Deerinck, T. J., Bushong, E. A., Thor, A., Ellisman, M. H. NCMIR methods for 3D EM: a new protocol for preparation of biological specimens for serial block face scanning electron microscopy. Microscopy. 1, 6-8 (2010).
- Miranda, K., Girard-Dias, W., Attias, M., de Souza, W., Ramos, I. Three dimensional reconstruction by electron microscopy in the life sciences: An introduction for cell and tissue biologists. Molecular Reproduction and Development. 82 (7-8), 530-547 (2015).
- Yamaguchi, M., Chibana, H. A method for obtaining serial ultrathin sections of microorganisms in transmission electron microscopy. The Journal of Visualized Experiments: JoVE. (131), e56235(2018).
- Hall, D. H., Hartwieg, E., Nguyen, K. C. Modern electron microscopy methods for C. elegans. Methods in Cell Biology. 107, 93-149 (2012).
- Hagler, H. K. Ultramicrotomy for biological electron microscopy. Methods in Molecular Biology. 369, 67-96 (2007).
- Arganda-Carreras, I., et al. Consistent and elastic registration of histological sections using vector-spline regularization. Computer vision approaches to medical image analysis, CVAMIA 2006, Lecture Notes in Computer Science. Beichel, R. R., Sonka, M., et al. 4241, Springer Berlin Heidelberg. Berlin, Heidelberg. 85-95 (2006).
- Belevich, I., Joensuu, M., Kumar, D., Vihinen, H., Jokitalo, E. Microscopy image browser: a platform for segmentation and analysis of multidimensional datasets. PLoS Biology. 14 (1), 1002340(2016).
- Fiala, J. C. Reconstruct: a free editor for serial section microscopy. Journal of Microscopy. 218, 52-61 (2005).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD). Journal of Structural Biology. 116 (1), 71-76 (1996).
- Iudin, A., Korir, P. K., Salavert-Torres, J., Kleywegt, G. J., Patwardhan, A. EMPIAR: a public archive for raw electron microscopy image data. Nature Methods. 13 (5), 387-388 (2016).
- Xu, C. S., Pang, S., Shtengel, G., Muller, A. An open-access volume electron microscopy atlas of whole cells and tissues. Nature. 599 (7883), 147-151 (2021).
- Karabag, C., et al. Semantic segmentation of HeLa cells: An objective comparison between one traditional algorithm and four deep-learning architectures. PLoS One. 15 (10), 0230605(2020).
- Heinrich, L., Bennett, D., Ackerman, D., Park, W. Whole-cell organelle segmentation in volume electron microscopy. Nature. 599 (7883), 141-146 (2021).
- Kim, J. S., Greene, M. J., Zlateski, A., Lee, K. Space-time wiring specificity supports direction selectivity in the retina. Nature. 509 (7500), 331-336 (2014).
- Spiers, H., Songhurst, H., Nightingale, L., de Folter, J. Deep learning for automatic segmentation of the nuclear envelope in electron microscopy data, trained with volunteer segmentations. Traffic. 22 (7), 240-253 (2021).
- Hasan, N. M., Gupta, A., Polishchuk, E., Yu, C. H. Molecular events initiating exit of a copper-transporting ATPase ATP7B from the trans-Golgi network. The Journal of Biological Chemistry. 287 (43), 36041-36050 (2012).
- Stoeck, I. K., Lee, J. Y., Tabata, K., Romero-Brey, I. Hepatitis C virus replication depends on endosomal cholesterol homeostasis. The Journal of Virology. 92 (1), 01196(2018).
- Ma, X., Qian, H., Chen, A., Ni, H. M., Ding, W. X. Perspectives on mitochondria-ER and mitochondria-lipid droplet contact in hepatocytes and hepatic lipid metabolism. Cells. 10 (9), 2273(2021).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved