A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
نظام الكشف عن فيروسات الحمض النووي استنادا إلى تقنية RPA-CRISPR/Cas12a-SPM والتعلم العميق
In This Article
Summary
نقدم بروتوكولا يجمع بين تضخيم بوليميراز المؤتلف ونظام CRISPR / Cas12a للكشف عن تتبع فيروسات الحمض النووي ويبني مجهرا محمولا للهواتف الذكية مع تصنيف مدعوم بالذكاء الاصطناعي للكشف عن فيروس الحمض النووي في نقطة الرعاية.
Abstract
لقد أبلغنا عن نظام سريع وسهل التنفيذ وحساس للغاية ومحدد التسلسل ونقطة رعاية (POC) للكشف عن فيروسات الحمض النووي ، والذي يجمع بين تضخيم بوليميراز الاتحاد (RPA) ونظام CRISPR / Cas12a للكشف عن تتبع فيروسات الحمض النووي. يتم تضخيم الحمض النووي المستهدف والتعرف عليه بواسطة RPA و CRISPR / Cas12a بشكل منفصل ، مما يؤدي إلى نشاط الانقسام الجانبي ل Cas12a الذي يشق مراسل الحمض النووي المسمى بالفلوروفور ويعمم التألق. للكشف عن POC ، تم تصميم الفحص المجهري المحمول للهواتف الذكية لالتقاط صور الفلورسنت. إلى جانب ذلك ، يتم نشر نماذج التعلم العميق للتصنيف الثنائي للعينات الإيجابية أو السلبية ، وتحقيق دقة عالية ، داخل النظام. تم اختبار فيروس الضفدع 3 (FV3 ، أجناس رانافيروس ، عائلة Iridoviridae) كمثال على نظام الكشف عن POC لفيروس الحمض النووي ، ويمكن أن تصل حدود الكشف (LoD) إلى 10 صباحا في غضون 40 دقيقة. بدون مشغلين ماهرين وأدوات ضخمة ، يظهر RPA-CRISPR / Cas12a-SPM المحمول والمصغر مع التصنيف بمساعدة الذكاء الاصطناعي (الذكاء الاصطناعي) إمكانات كبيرة للكشف عن فيروس الحمض النووي POC ويمكن أن يساعد في منع انتشار مثل هذه الفيروسات.
Introduction
في السنوات الأخيرة ، حدثت أوبئة الأمراض المعدية التي تسببها فيروسات مختلفة بشكل متكرر ، بما في ذلك وباء مرض فيروس الإيبولا (EVD) في عامي 20141 و 20182 ، ومتلازمة الشرق الأوسط التنفسية (MERS) في عام 20153 ، ووباء مرض فيروس زيكا في عام 20154 ، ومرض فيروس كورونا 2019 (COVID-19) الناجم عن فيروس كورونا 2 المسبب للمتلازمة التنفسية الحادة الوخيمة (SARS-CoV-2)5 واستمرار جدري الناجم عن فيروس جدري (MKPV) في عام 20226. هذه الفاشيات المفاجئة للأمراض المعدية الوبائية تسبب عددا كبيرا من الوفيات وتجلب خسائر اقتصادية ضخمة واضطرابات اجتماعية. هناك حاجة ماسة إلى نظام كشف سريع ودقيق لتشخيص العدوى بسرعة ومنع انتشار الفيروس.
في الآونة الأخيرة ، اكتسبت التكرارات العنقودية القصيرة المتباعدة بانتظام (CRISPR) والبروتينات المرتبطة بكريسبر (Cas) اهتماما عالميا وأظهرت نتائج واعدة في الكشف عن الحمض النووي7،8،9،10،11،12،13،14،15. يرتبط بروتين كريسبر / كاس 12 أ ، الذي يسترشد ب CRISPR RNA (crRNA) ، بالحمض النووي المستهدف ويقطعه. يؤدي هذا النشاط إلى إطلاق الحمض النووي أحادي الشريط غير المحدد (ssDNA) ، والمعروف باسم الانقسام العابر ، ويمكن استخدامه لتعزيز إشارة الكشف للكشف عن الحمض النووي. بعض طرق الكشف التقليدية مثل تفاعل البوليميراز المتسلسل (PCR) ، وتفاعل البوليميراز المتسلسل الكمي في الوقت الفعلي (qPCR) ، ومقايسة الممتز المناعي المرتبط بالإنزيم (ELISA) معقدة وتستغرق وقتا طويلا ومكلفة للكشف عن نقطة الرعاية (POC). نجح عملنا السابق في تطوير نظام كشف آلي ومتكامل وفعال من حيث التكلفة لفيروس حمى الخنازير الأفريقية (ASFV) يعتمد على تقنية CRISPR / Cas12a. في هذا النظام ، حققنا حدا للكشف يبلغ 1 pM في إطار زمني 2 ساعة دون الحاجة إلى التضخيم. يتم الجمع بين نظام CRISPR / Cas12a وتضخيم البوليميراز المؤتلف (RPA) لتحسين الحساسية والنوعية للكشف عن الحمض النووي النزرة. بالمقارنة مع تقنيات التضخيم متساوية الحرارة الأخرى ، فإن RPA بسيط في التصميم ومريح في التشغيل نظرا لأنه يتمتع بوقت رد فعل أقصر بدون معدات متطورة للتحكم في درجة الحرارة.
للكشف عن POC لمسببات الأمراض ، تم تطوير أدوات مثل الفحص المجهري للهواتف الذكية (SPM) أو مقياس الفلور المحمول أو شرائط التدفق الجانبي لقراءات النتائج16،17،18. يلتقط SPM الصور من خلال الكاميرا ويحملها إلى بعض تطبيقات الهاتف المحمول لتحليل البيانات بسرعة. يصنع هذا الفحص المجهري نظاما محمولا ورخيصا ومصغرا لاكتساب الإشارات بحساسية عالية وقد أظهر مزايا في الكشف عن مسببات الأمراض مثل H5N1 وفيروس زيكا و SARS-CoV-219,20. لذلك ، نقوم ببناء SPM محمول لالتقاط إشارات التألق الناتجة عن اكتشاف RPA-CRISPR / Cas12a لفيروس الحمض النووي المستهدف. سيتم شق مسبار مراسل ssDNA الذي يربط بين الفلوروفور والمروي عندما يتعرف CRISPR / Cas12a على فيروس الحمض النووي المستهدف ، ويمكن التقاط التألق المنبعث من الفلوروفور بواسطة SPM.
مقارنة بالبرامج الاحترافية المستخدمة عادة للحصول على معلومات النتائج من الصور الفلورية من SPM21 ، يستخدم بعض الخبراء التعلم الآلي والتعلم العميق لتحديد تركيزات الحمض النووي للفيروس بعد الحصول على صور مضان22 ، وهو ما يستغرق وقتا أطول. عندما يتعلق الأمر بتصنيف الصور الطبية ، غالبا ما تستخدم الشبكات العصبية التقليدية (CNNs) لتعلم الميزات من الصور المنقطة الخام بطريقة شاملة23،24،25،26. تم تطبيق نماذج التعلم العميق الشائعة المستندة إلى CNN مثل AlexNet و DenseNet-121 و EfficientNet-B7 بنجاح في هذا المجال27,28. ومع ذلك ، قد يكون الحصول على مجموعات بيانات كبيرة في مجالات محددة أمرا صعبا ، مما يستلزم نقل التعلم29,30. يقوم هذا النهج بتدريب نموذج التعلم العميق مسبقا باستخدام مجموعة بيانات كبيرة ، ويتم استخدام النموذج المدرب مسبقا كنقطة انطلاق لمهمة جديدة مع مجموعة بيانات صغيرة. يمكن أن تقلل هذه التقنية من الحاجة إلى مجموعات البيانات الكبيرة ، ومكافحة الإفراط في التجهيز ، وتقليل وقت التدريب31. هنا ، نستخدم نماذج التعلم العميق مع نقل التعلم للتصنيف الثنائي للصور الفلورية للعينات الموجبة والسالبة.
في هذه الطريقة ، نجمع بين RPA ونظام CRISPR / Cas12a للكشف عن تتبع فيروسات الحمض النووي. يتم تضخيم الحمض النووي المستهدف والتعرف عليه بواسطة RPA و CRISPR / Cas12a بشكل منفصل ، مما يؤدي إلى نشاط الانقسام الجانبي ل Cas12a الذي يشق مراسل الحمض النووي المسمى بالفلوروفور ويعمم التألق. نحن نبني SPM محمول لالتقاط صور الفلورسنت للكشف عن POC وتطوير نماذج التعلم العميق للتصنيف الثنائي. يظهر الرسم التخطيطي لنظام الكشف عن POC المدمج في الشكل 1. بدون مشغلين ماهرين وأدوات ضخمة، تظهر تقنية RPA-CRISPR/Cas12a-SPM المزودة بتصنيف بمساعدة الذكاء الاصطناعي (الذكاء الاصطناعي) إمكانات كبيرة للكشف عن فيروس الحمض النووي ل POC.
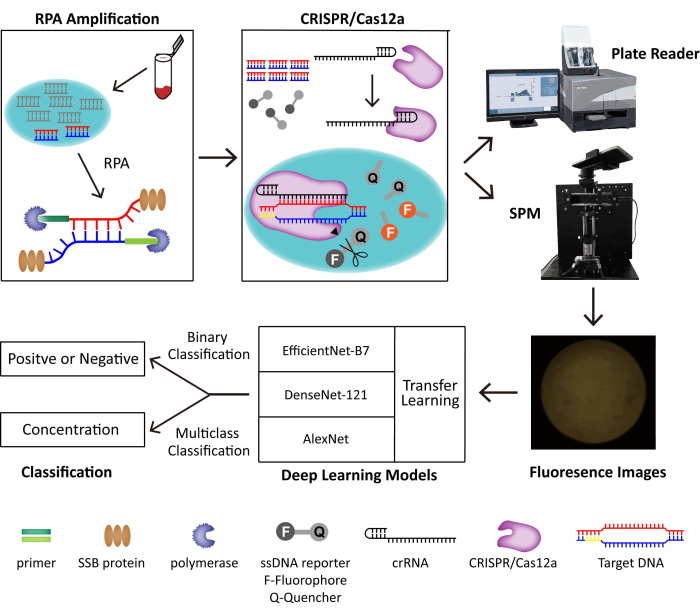
الشكل 1: رسم تخطيطي لنظام الكشف RPA-CRISPR/Cas12-SPM إلى جانب التصنيف الذكاء الاصطناعي للصور المجمعة. يتم إطلاق الأحماض النووية للعينات المشتقة من بواسطة PINDBK. يتم تضخيم الحمض النووي المستهدف للفيروس والتعرف عليه على وجه التحديد بواسطة نظام RPA-CRISPR / Cas12a. ترتبط CRISPR / Cas12a مع crRNA والروابط المعقدة Cas12a-crRNA مع الحمض النووي المستهدف ، مما يؤدي إلى الانقسام الجانبي ل CRISPR / Cas12a على تحقيقات مراسل ssDNA. يتم إطلاق الفلوروفور على المراسل ، ويتم الكشف عن التألق بواسطة قارئ لوحة تجاري أو SPM الذي نبنيه. يتم استخدام ثلاثة نماذج مختلفة للتعلم العميق ، بما في ذلك AlexNet و DenseNet-121 و EfficientNet-B7 مع نقل التعلم ، لتصنيف الصور الفلورية. أعيد استخدام هذا الرقم بإذن من Lei et al.35. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
Protocol
1. تجهيز العينات
- خذ فيروس الضفدع 3 (FV3 ، أجناس رانافيروس ، عائلة Iridoviridae) ، وهو فيروس DNA مزدوج تقطعت بهم السبل. حدد جين القفيصة الرئيسية (mcp) كهدف للكشف عن FV3 لأنه محفوظ للغاية وعادة ما يعتبر هدفا للكشف عن فيروس رانا. ويبين الجدول 1 التسلسل المستهدف المحدد.
ملاحظة: يؤخذ فيروس الضفدع 3 كمثال في هذا البروتوكول. - لإعداد شظايا الحمض النووي المستهدفة ، استخدم شظايا الحمض النووي لجين mcp من FV3 وفيروس الطحال المعدية ونخر الكلى (ISKNV ، فيروس رانا آخر).
ملاحظة: في هذه الدراسة ، تم الحصول على شظايا الحمض النووي المستهدفة تجاريا من شركة مدرجة في جدول المواد. وهي تعتبر الهدف والسيطرة على الكشف اللاحق.
| اسم | تسلسل | ||
| FV3 MCP | NTS: 5 '... gtaacccggctttcGGGCAGCAGTTTCGGTCGGCGTtcccaggtcg... 3' (240 نقطة أساس) | ||
| TS: 5 '... ccgacctgggaACGCCGACCGAAACTGCTGCCCtgctgcccgaaagc... 3' (240 نقطة أساس) | |||
| ISKNV MCP | NTS: 5 '... ggccatgccaatttTGGGCAGGAGTTTAGTGTGACGgtggcgaggg... 3' (231 نقطة أساس) | ||
| TS: 5 '... ccctcgccaccgtcACACTAAACTCCTGCCCAAAATtggcatggcc... 3' (231 نقطة أساس) | |||
الجدول 1: تسلسل الهدف المحدد في هذه الطريقة.
2. تفاعل تقنية RPA
- تصميم وتوليف أزواج RPA التمهيدية للتسلسل الهدف. يتم وصف تسلسل أزواج التمهيدي RPA في الجدول 2.
- قم بإعداد المخزن المؤقت لتفاعل RPA 5x (الجدول 3) و MgCl2 (100 mM).
- امزج إنزيمات RPA الرئيسية الأربعة (UvsX و UvsY و GP32 وبروتين Bsu) في المخزن المؤقت لتفاعل RPA 1x ، جنبا إلى جنب مع البادئات المصممة مسبقا ، كما هو مفصل في الجدول 4.
- دوامة الخليط جيدا.
- أضف 1 ميكرولتر من الهدف الذي تم الحصول عليه من الخطوة 1 لكل تفاعل RPA ، واخلطه جيدا بواسطة الدوامة مرة أخرى.
- أضف 7 ميكرولتر من MgCl2 (100 mM) لبدء التفاعل. الحجم النهائي لكل تفاعل RPA هو 50 ميكرولتر.
- قم بإجراء الفحص عند 37 درجة مئوية لمدة 30 دقيقة.
- يمكن تخزين منتج RPA في درجة حرارة 4 درجات مئوية لبضعة أيام. مع تدهور منتجات تقنية RPA، استخدمها لمزيد من الاكتشاف في أسرع وقت ممكن للحصول على نتائج تشخيص أفضل.
- [اختياري] إجراء الحمض النووي هلام الكهربائي.
- أخرج 5 ميكرولتر من منتجات RPA ، وأضف حجما مناسبا من المخزن المؤقت لتحميل الحمض النووي 6x ، وقم بإجراء الرحلان الكهربائي لهلام الحمض النووي في المخزن المؤقت Tris-acetate EDTA (TAE).
- قم بتشغيل الكهربائي تحت 120 فولت لمدة 20 دقيقة تقريبا حتى تصل نطاقات المخزن المؤقت للتحميل إلى قاع الجل.
- حدد ما إذا كان التسلسل المستهدف قد تم تضخيمه بنجاح من العينة من خلال مقارنة حجم نطاقات العينة بنطاقات العلامة.
| اسم | تسلسل |
| RPA التمهيدي F | ATGTCTTCTTACTGGTTCAGGTATCACA |
| RPA التمهيدي R | GGCGTTGAGGATGTAATCCCCCGACCTGGG |
الجدول 2: بادئات RPA المستخدمة في هذه الطريقة.
| مكون | التركيز الأصلي | جَمْع |
| بيج 20,000 | - | 114 ملغ |
| ATP | 100 مللي متر | 125 ميكرولتر |
| dNTPs | 25 مللي متر | 48 ميكرولتر |
| تريس حمض الهيدروكلوريك | 1 م | 125 ميكرولتر |
| دي تي تي | 1 م | 125 ميكرولتر |
| فوسفوكرياتين | 1 م | 250 ميكرولتر |
| الكرياتين كيناز | 10 ميكروغرام / ميكرولتر | 50 ميكرولتر |
| ddH2O | - | 277 ميكرولتر |
| الحجم الكلي | - | 1 مل |
الجدول 3: تكوين المخزن المؤقت لتفاعل RPA 5x (الرقم الهيدروجيني 7.5).
| مكون | التركيز الأصلي | جَمْع |
| 5x مخزن مؤقت لتفاعل RPA | - | 10 ميكرولتر |
| بروتين UvsX | 5 ملغ/مل | 2.6 ميكرولتر |
| بروتين UvsY | 5 ملغ/مل | 0.9 ميكرولتر |
| بروتين GP32 | 5 ملغ/مل | 2.54 ميكرولتر |
| بروتين Bsu | 5 ملغ/مل | 0.88 ميكرولتر |
| التمهيدي إلى الأمام | 100 ميكرومتر | 0.25 ميكرولتر |
| التمهيدي المعكوس | 100 ميكرومتر | 0.25 ميكرولتر |
| ddH2O | - | 24.58 ميكرولتر |
| هدف | - | 1 ميكرولتر |
| *مغكلوريد 2 | 100 مللي متر | 7 ميكرولتر |
| الحجم الكلي | 50 ميكرولتر | |
| * يجب إضافة MgCl2 أخيرا لبدء تفاعل RPA. | ||
الجدول 4: تكوين تفاعل RPA.
3. اكتشاف كريسبر/كاس 12 أ بدون SPM
- استخدم crRNAs (جدول المواد) للتسلسل المستهدف ومسبار مراسل ssDNA الذي يربط بين الفلوروفور والمروي ل CRISPR / Cas12a. هنا ، يرتبط كربوكسي رباعي ميثيل رودامين (TAMRA) باعتباره الفلوروفور في نهايات 5 'من مجسات مراسل ssDNA و Black Hole Quencher-2 (BHQ2) كمطفأ في نهايات 3'. يتم وصف التسلسل التفصيلي لمراسل crRNA و ssDNA في الجدول 5.
- تحضير بروتين بكتيريا Lachnospiraceae Cas12a (LbCas12a) مع 10x CRISPR / Cas12a مخزن مؤقت.
- قم بإذابة 1 ميكرولتر من منتج تفاعل RPA من القسم 2 في 1x مخزن مؤقت لتفاعل CRISPR / Cas12a مع معقدات LbCas12a-crRNA ومسبار مراسل ssDNA 500 نانومتر في حجم تفاعل 100 ميكرولتر.
- بعد خلط LbCas12a و crRNA ، اترك الخليط لمدة 5 دقائق على الأقل لتشكيل مركب وظيفي. بعد الحضانة ، أضف مكونات أخرى إلى خليط التفاعل وقم بإجراء التفاعل بالكامل عند 37 درجة مئوية. الحجم النهائي لكل تفاعل كريسبر / كاس 12 أ هو 100 ميكرولتر. ويرد في الجدول 6 وصف للتركيزات التفصيلية لكل مكون في كل تفاعل كريسبر/كاس12أ.
- قم بإجراء تفاعل الكشف CRISPR / Cas12a سعة 100 ميكرولتر عند 37 درجة مئوية لمدة 30 دقيقة.
- افحص إشارات التألق باستمرار بواسطة قارئ صفيحة مجهرية بطول موجة إثارة يبلغ 535 نانومتر وطول موجة انبعاث يبلغ 595 نانومتر مع كسب 60.
ملاحظة: يعتمد اكتشاف الأطوال الموجية المختلفة لضوء الإثارة والانبعاث على اختيار الفلوروفور والمروي في مجسات مراسل ssDNA المصممة مسبقا. - بالنسبة لهذه البيانات التي تم جمعها ، قسم قيمة التحكم على قياس العينات الإيجابية لتطبيع جميع البيانات ، ثم دمجها لتحليل اختبار t لعينتين.
| اسم | تسلسل | |
| LbCas12a crRNA ل FV3 | uaauuucuacuaaguguagauGGGCAGCAGTTTTCGGTCGGCGT | |
| مراسل ssDNA | /5TAMRA/TTATT/3BHQ2 | |
الجدول 5: تسلسل مراسل CRISPR / Cas12a crRNA و ssDNA المستخدم في هذه الطريقة.
| مكون | التركيز الأصلي | جَمْع |
| نيبوفر r2.1 | - | 10 ميكرولتر |
| Lba Cas12a (Cpf1) | 10 ميكرومتر | 0.5 ميكرولتر |
| crRNA | 10 ميكرومتر | 0.625 ميكرولتر |
| انتظر لمدة 5 دقائق على الأقل للسماح لمركب LbCas12a / crRNA بالجمع. | ||
| مراسل الحمض النووي | 100 ميكرومتر | 0.5 ميكرولتر |
| ddH2O | - | 87.375 ميكرولتر |
| هدف | - | 1 ميكرولتر |
| الحجم الكلي | - | 100 ميكرولتر |
الجدول 6: تكوين تفاعل كريسبر / كاس 12 أ.
4. إعداد SPM
- بالنسبة لمسار الإثارة ، اضبط شعاع الليزر ليمر عبر مرشحات الكثافة المحايدة (ND) لتخفيف شدة الليزر.
- توليد شعاع متوازي من عدسة شبه كروية (يشار إليها فيما يلي باسم L1) وعكسها بواسطة مرآة ثنائية اللون (DM).
- وجه الضوء إلى الشريحة الزجاجية حيث يتم وضع العينة من خلال الهدف (20x) لإلقاء الضوء وإثارة مضان العينة. تتيح مرحلة العينة ضبطا دقيقا للمستوى البؤري ، وتوجيه الحزمة إلى المستوى البؤري الخلفي للهدف. تشكل الخطوات المذكورة أعلاه مسار الإثارة لأداة SPM.
- بالنسبة لمسار الانبعاث ، ضع عدسة خارجية (يشار إليها فيما يلي باسم L2) لتشكيل صورة وسيطة على الجانب الآخر من الهدف. الهدف يضيء العينة في وقت واحد ويجمع إشارة الانبعاث.
- سجل إشارة التألق من العينة باستخدام الهاتف الذكي الموضوع في نهاية مسار الانبعاث. استخدم دعامة ثابتة لتجنب الاهتزاز.
- اضبط مرشح تمرير النطاق بين L1 وكاميرا الهاتف الذكي لتصفية الضوء المتحمس مع السماح فقط للضوء المنبعث من العينة بالوصول إلى الكاميرا ، مما يمكنه تحسين الكشف.
- قم بتثبيت إعداد SPM على لوح تجارب للنشر المحمول.
ملاحظة: يوضح الشكل 2 المظهر التخطيطي والفيزيائي لجهاز SPM للكشف عن التألق بناء على تفاعل RPA-CRISPR / Cas12a . يتم اكتشاف CRISPR / Cas12a على الشريحة الزجاجية المعالجة مسبقا الموضحة في الخطوة التالية.
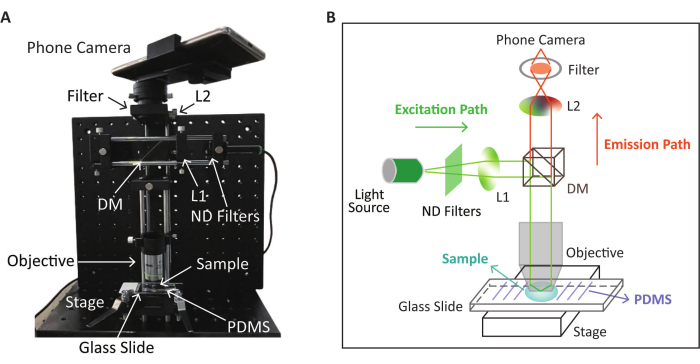
الشكل 2: المظهر التخطيطي والفيزيائي لجهاز SPM المستخدم للكشف عن التألق. (أ) المظهر المادي لجهاز SPM لجمع الصور الفلورية بعد تفاعل RPA-CRISPR/Cas12a. (B) رسم تخطيطي لجهاز SPM للكشف عن التألق بناء على تفاعل RPA-CRISPR / Cas12a. تم تعديل هذا الرقم (تعديل موضع الصورة ولونها) بإذن من Lei et al.35. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
5. معالجة الشريحة الزجاجية للكشف مع SPM
- تحضير بوليديميثيل سيلوكسان (PDMS) عن طريق خلط القاعدة وعامل المعالجة بنسبة 10: 1 ، متبوعا بالخبز على لوح تسخين عند 80 درجة مئوية لمدة 2 ساعة.
- معالجة كل من PDMS والشريحة الزجاجية (الطول: 75 مم ؛ الارتفاع: 50 مم) مع علاج بلازما الأكسجين لمدة 120 ثانية ، ثم اضغط عليهما معا.
- اخبز الزجاج / PDMS على حرارة 95 درجة مئوية لمدة 2 ساعة ؛ يتم ختمه بشكل دائم بواسطة رابطة Si-O-Si. يتمتع PDMS بشفافية عالية ولا يوجد مضان تلقائي ، مما يساعد على اكتشاف SPM. تتم معالجة الشريحة الزجاجية و PMDS وفقا ل He et al.32
6. اكتشاف كريسبر / كاس 12 أ مع SPM
- استخدم crRNAs (جدول المواد) للتسلسل المستهدف ومسبار مراسل ssDNA الذي يربط بين الفلوروفور والمروي ل CRISPR / Cas12a. في هذه الطريقة ، يرتبط كربوكسي رباعي ميثيل رودامين (TAMRA) باعتباره الفلوروفور في نهايات 5 'من تحقيقات مراسل ssDNA و Black Hole Quencher-2 (BHQ2) كمطفأ في نهايات 3. يتم وصف التسلسل التفصيلي لمراسل crRNA و ssDNA في الجدول 5.
- تحضير بكتيريا Lachnospiraceae Cas12a (LbCas12a) البروتين مع 10x كريسبر / كاس12a تفاعل العازلة.
- قم بإذابة 1 ميكرولتر من منتج تفاعل RPA من القسم 2 في 1x مخزن مؤقت لتفاعل CRISPR / Cas12a مع معقدات LbCas12a-crRNA ومسبار مراسل ssDNA 500 نانومتر في حجم تفاعل 100 ميكرولتر.
- بعد خلط LbCas12a و crRNA ، اترك الخليط لمدة 5 دقائق على الأقل لتشكيل مركب وظيفي. بعد الحضانة، أضف مكونات أخرى إلى خليط التفاعل وأكمل التفاعل عند 37 درجة مئوية. الحجم النهائي لكل تفاعل كريسبر / كاس 12 أ هو 100 ميكرولتر. ويرد في الجدول 6 وصف للتركيزات التفصيلية لكل مكون في كل تفاعل كريسبر/كاس12أ.
- قم بإجراء تفاعل الكشف CRISPR / Cas12a سعة 100 ميكرولتر على الشريحة الزجاجية المعالجة مسبقا وقم بتغطيتها بغطاء سليب. احتضان الشريحة الزجاجية مع رد فعل في RT لمدة 10 دقائق.
- قم بقياس إشارات التألق بواسطة SPM. ضع الشريحة الزجاجية مع تفاعل الكشف على مرحلة SPM ، وحافظ على مسافة مناسبة ، واضبط البعد البؤري والوضوح ، ثم ابحث عن مجال رؤية التفاعل وركزه لالتقاط صورة.
ملاحظة: يجب الحصول على منحنى قياسي أولا بحيث يمكن تحجيم البيانات من العينات إلى نطاق التركيز التقريبي. يتم استخدام تركيزات مختلفة من الأهداف المنقاة ، بما في ذلك 10 نانومتر ، 1 نانومتر ، 100 ب م ، و 10 ب م ، فيروس آخر كتحكم سلبي (قبل RPA).
7. مجموعة البيانات وزيادة البيانات
- اجمع صور التألق من مقايسة الكشف في القسم 6 كمجموعات بيانات. كرر ما لا يقل عن ثلاثة اكتشافات متوازية لكل عينة لضمان توازي البيانات.
- يمكن الموافقة على بعض الطرق المناسبة لتحقيق توازي أعلى. على سبيل المثال ، عند جمع صور التألق من كل عينة ، ركز يدويا وابحث عن حقل أكثر سطوعا نسبيا لالتقاط الصور. في الوقت نفسه ، قم بتصوير كل عينة للوصول إلى إشارة التألق في أكثر من خمسة مواقع مختلفة.
- قم بقياس متوسط القيمة الرمادية لكل صورة والانحراف المعياري لمتوسط القيمة الرمادية في مجموعة تركيز بواسطة ImageJ.
- قم بتعيين نطاق شدة [الوسيط - الانحراف المعياري ، الوسيط + الانحراف المعياري] لتنظيف البيانات.
ملاحظة: في الصور التي تم الحصول عليها في الخطوات المبكرة ، قد تكون هناك صور ذات اختلافات كبيرة ، لذلك من الضروري فحص الصور. إذا كانت كثافة الصور خارج الحد المحدد ، فيجب اعتبارها قيم متطرفة واستبعادها. - قم بتسمية الصور للهدف المنقى بتركيزات متزايدة مع 0-6 بترتيب تصاعدي ، على التوالي.
- لتعزيز متانة النظام ومنع الإفراط في التركيب ، قم بتنفيذ تقنيات زيادة الصورة مثل التقليب الأفقي والتقليب الرأسي والضوضاء العشوائية من خلال وظائف التحويل في Python. يساعد هذا في إدخال اختلافات في مجموعة البيانات.
8. نقل التعلم
- كشبكة العمود الفقري ، اعتمد نموذج التعلم العميق AlexNet33 للتصنيف.
- لتلبية قيود النموذج الذي تم تدريبه مسبقا في القسم 7 ، أعد تشكيل الصور المدخلة إلى 224 بكسل × 224 بكسل × 3 قنوات (ارتفاع وعرض 224 بكسل وعمق 3 قنوات لقنوات الألوان الأحمر والأخضر والأزرق) من خلال وظائف التحويل في Python.
ملاحظة: هذه الخطوة هي معالجة مسبقة شائعة للبيانات غير المتجانسة في نقل التعلم ، بما في ذلك التحويل. - استخدم شبكة أساسية مدربة مسبقا مع مجموعة بيانات ImageNet لاستخراج الميزات مع الاستفادة من أوزان الطبقات المخفية الوسيطة التي تم تعلمها.
- في سياق وظيفة تصنيف التألق ، استبدل الطبقة النهائية المتصلة بالكامل من الشبكة العصبية ، والتي تضمنت في الأصل 1000 خلية عصبية لمهمة ImageNet ، بطبقة متصلة بالكامل إما مع 2 أو 7 خلايا عصبية.
- قم بتقييم أداء نموذج تدريب الإعداد باستخدام سلسلة من المقاييس ، بما في ذلك مصفوفة الارتباك والدقة والدقة والاستدعاء ودرجة F1 بناء على Lawton و Viriri34.
النتائج
تركز هذه الطريقة على نظام كشف سريع وسهل التنفيذ وحساس للغاية ونقطة رعاية (POC) لفيروسات الحمض النووي. يعد تصميم أزواج التمهيدي لتفاعل RPA وتصميم crRNA لتفاعل CRISPR / Cas12a من الأجزاء الأساسية لأنهما سيؤثران على كفاءة تفاعل RPA-CRISPR / Cas12a ويؤثران على الكشف والتصنيف اللاحقين.
ف...
Discussion
في هذه الطريقة ، نقوم بتطوير نظام سريع وسهل التنفيذ وحساس للغاية ومحدد التسلسل وللكشف عن فيروس الحمض النووي POC بمساعدة الذكاء الاصطناعي. بعد الحصول على العينات ، يتم تطبيق RPA لتضخيم تسلسل الهدف ، ومن ثم يمكن ل CRISPR / Cas12a التعرف على الحمض النووي المستهدف وإطلاق التألق ، مما يؤدي إلى توسيع إشار...
Disclosures
ليس لدى المؤلفين ما يكشفون عنه.
Acknowledgements
يتم دعم هذا العمل من قبل المؤسسة الوطنية للعلوم الطبيعية في الصين 31970752 ، العلوم والتكنولوجيا ، لجنة الابتكار في بلدية شنتشن JCYJ20190809180003689 ، JSGG20200225150707332 ، JSGG20191129110812708 ، WDZC20200820173710001 ؛ التمويل المفتوح لمختبر خليج شنتشن ، SZBL2020090501004 ؛ مؤسسة الصين لعلوم ما بعد الدكتوراه 2020M680023 ؛ والإدارة العامة للجمارك في جمهورية الصين الشعبية 2021HK007.
Materials
| Name | Company | Catalog Number | Comments |
| 20x amplification | OLYMPUS | OPLN20X | |
| 532 nm green laser | Thorlabs | PL201 | with 0.9 mW output power |
| 535 nm cutoff wavelength | chrome | AT535 | |
| 6x DNA loading buffer | Thermo scientific | R0611 | |
| 96-well black microplate | Corning Incorporated | 3603 | Black with flat clear bottom |
| Aspherical lens | Lubang | N/A | |
| Bandpass filter | SEMROCK | FF01-542/27-25 | |
| Bsu DNA Polymerase | ATG Biotechnology | M103 | Large Fragment |
| crRNA | Sangon Biotech | N/A | |
| DNA fragments | Sangon Biotech | N/A | |
| Dichroic holders | Ruicage | N/A | |
| Dichroic mirror | SEMROCK | FF555-Di03-25x36 | with a cutoff wavelength of 535 nm |
| E.Z.N.A Gel Extraction Kit | Omega Biotek | D2500-02 | |
| EnGen Lba Cas12a (Cpf1) | New England Biolabs (Beijing) LTD | M0653T | |
| Filter holders | Ruicage | N/A | |
| Fluorophore-ssDNA-Quencher reporter probes | Sangon Biotech | N/A | TAMRA (carboxy tetramethylrhodamine) as the fluorophore at the 5 ends; BHQ2 (Black Hole Quencher-2) as the quencher at the 3 ends |
| GP32 | ATG Biotechnology | M104 | |
| ImageJ | Open-source | Version 1.53t 24 | Downloaded from https://imagej.nih.gov/ij/ |
| Microplate reader | SPARK, TECAN | N/A | |
| Multi-Block thermal Cycler PCR instrument | LongGene | N/A | |
| NanoDrop 2000/2000c Spectrophotometers | Thermo Scientific | ND-2000 | |
| NEBuffer r2.1 | New England Biolabs (Beijing) LTD | B6002S | 10x CRISPR/Cas12a Reaction buffer |
| Oxygen plasma treatment | Electro-Technic Products | N/A | |
| Pathogen Inactivate, Nucleic acid extraction-free, Direct-to-PCR Buffer with Proteinase K (PINDBK) | Ebio | PINDBK -25mL | |
| PCR primer pairs | Sangon Biotech | N/A | |
| PDMS | Dow Corning | Sylgard 184 | |
| RPA primer pairs | Sangon Biotech | N/A | |
| Smartphone | Huawei | Mate10 | |
| Translation stages | Ruicage | N/A | |
| Transmitted neutral density filters | Thorlabs | ND40A | |
| Triplet achromatic lenses | Thorlabs | TRH127-020-A | |
| UvsX | ATG Biotechnology | M105 | |
| UvsY | ATG Biotechnology | M106 |
References
- Gire, S. K., et al. Genomic surveillance elucidates Ebola virus origin and transmission during the 2014 outbreak. Science. 345 (6202), 1369-1372 (2014).
- The Ebola Outbreak Epidemiology Team. Outbreak of Ebola virus disease in the Democratic Republic of the Congo, April-May 2018: an epidemiological study. Lancet. 392 (10143), 213-221 (2018).
- Zumla, A., Hui, D. S., Perlman, S. Middle East respiratory syndrome. Lancet. 386 (9997), 995-1007 (2015).
- Plourde, A. R., Bloch, E. M. A literature review of Zika virus. Emerg Infect Dis. 22 (7), 1185-1192 (2016).
- Yuan, X., et al. Current and perspective diagnostic techniques for COVID-19. ACS Infect Dis. 6 (8), 1998-2016 (2020).
- Minhaj, F. S., et al. Monkeypox outbreak - nine states, May 2022. MMWR Morb Mortal Wkly Rep. 71 (23), 764-769 (2022).
- Bao, M., et al. Challenges and opportunities for clustered regularly interspaced short palindromic repeats based molecular biosensing. ACS Sens. 6 (7), 2497-2522 (2021).
- Broughton, J. P., et al. CRISPR-Cas12-based detection of SARS-CoV-2. Nat Biotechnol. 38 (7), 870-874 (2020).
- Chen, J. S., et al. CRISPR-Cas12a target binding unleashes indiscriminate single-stranded DNase activity. Science. 360 (6387), 436-439 (2018).
- Gootenberg, J. S., et al. Nucleic acid detection with CRISPR-Cas13a/C2c2. Science. 356 (6336), 438-442 (2017).
- Kellner, M. J., Koob, J. G., Gootenberg, J. S., Abudayyeh, O. O., Zhang, F. SHERLOCK: nucleic acid detection with CRISPR nucleases. Nat Protoc. 14 (10), 2986-3012 (2019).
- Mukama, O., et al. An ultrasensitive and specific point-of-care CRISPR/Cas12 based lateral flow biosensor for the rapid detection of nucleic acids. Biosens Bioelectron. 159, 112143 (2020).
- Schwank, G., et al. Functional repair of CFTR by CRISPR/Cas9 in intestinal stem cell organoids of cystic fibrosis patients. Cell Stem Cell. 13 (6), 653-658 (2013).
- Yin, L., Man, S., Ye, S., Liu, G., Ma, L. CRISPR-Cas based virus detection: Recent advances and perspectives. Biosens Bioelectron. 193, 113541 (2021).
- Dronina, J., Bubniene, U. S., Ramanavicius, A. The application of DNA polymerases and Cas9 as representative of DNA-modifying enzymes group in DNA sensor design (review). Biosens Bioelectron. 175, 112867 (2021).
- Fozouni, P., et al. Amplification-free detection of SARS-CoV-2 with CRISPR-Cas13a and mobile phone microscopy. Cell. 184 (2), 323-333.e9 (2021).
- Kumar, M., et al. FnCas9-based CRISPR diagnostic for rapid and accurate detection of major SARS-CoV-2 variants on a paper strip. eLife. 10, e67130 (2021).
- Lee, R. A., et al. Ultrasensitive CRISPR-based diagnostic for field-applicable detection of Plasmodium species in symptomatic and asymptomatic malaria. Proc Natl Acad Sci U S A. 117 (41), 25722-25731 (2020).
- Ganguli, A., et al. Hands-free smartphone-based diagnostics for simultaneous detection of Zika, Chikungunya, and Dengue at point-of-care. Biomed Microdevices. 19 (4), 73 (2017).
- Yeo, S. J., et al. Smartphone-based fluorescent diagnostic system for highly pathogenic H5N1 viruses. Theranostics. 6 (2), 231-242 (2016).
- von Chamier, L., et al. Democratising deep learning for microscopy with ZeroCostDL4Mic. Nat Commun. 12 (1), 2276 (2021).
- Shiaelis, N., et al. Virus detection and identification in minutes using single-particle imaging and deep learning. ACS Nano. 17 (1), 697-710 (2020).
- Liu, Y., et al. Mixed-UNet: Refined class activation mapping for weakly-supervised semantic segmentation with multi-scale inference. Front. Comput. Sci. 4, 1036934 (2022).
- Lawrimore, J., Doshi, A., Walker, B., Bloom, K. AI-assisted forward modeling of biological structures. Front Cell Dev Biol. 7, 279 (2019).
- Yang, Y., Hu, Y., Zhang, X., Wang, S. Two-stage selective ensemble of CNN via deep tree training for medical image classification. IEEE Trans Cybern. 52 (9), 9194-9207 (2022).
- Zhang, R., et al. RCMNet: A deep learning model assists CAR-T therapy for leukemia. Comput Biol Med. 150, 106084 (2022).
- Xie, Y., et al. Stroke prediction from electrocardiograms by deep neural network. Multimed Tools Appl. 80, 17291-17297 (2021).
- Wang, J., Zhu, H., Wang, S., Zhang, Y. -. D. A review of deep learning on medical image analysis. Mobile Netw Appl. 26, 351-380 (2021).
- Artoni, P., et al. Deep learning of spontaneous arousal fluctuations detects early cholinergic defects across neurodevelopmental mouse models and patients. Proc Natl Acad Sci U S A. 117 (38), 23298-23303 (2020).
- Li, J., et al. DeepLearnMOR: a deep-learning framework for fluorescence image-based classification of organelle morphology. Plant Physiol. 186 (4), 1786-1799 (2021).
- Yosinski, J., Clune, J., Bengio, Y., Lipson, H. How transferable are features in deep neural networks. Proceedings of the 27th International Conference on Neural Information Processing Systems. 2, 3320-3328 (2014).
- He, Q., et al. High-throughput and all-solution phase African Swine Fever Virus (ASFV) detection using CRISPR-Cas12a and fluorescence based point-of-care system. Biosens Bioelectron. 154, 112068 (2020).
- Krizhevsky, A., Sutskever, I., Hinton, G. E. ImageNet classification with deep convolutional neural networks. Commun. ACM. 60 (6), 84-90 (2017).
- Lawton, S., Viriri, S. Detection of COVID-19 from CT lung scans using transfer learning. Comput Intell Neurosci. 2021, 5527923 (2021).
- Lei, Z., et al. Detection of frog virus 3 by integrating RPA-CRISPR/Cas12a-SPM with deep learning. ACS Omega. 8 (36), 32555-32564 (2023).
- Chen, Z., Huang, J., Zhang, F., Zhou, Y., Huang, H. Detection of shrimp hemocyte iridescent virus by recombinase polymerase amplification assay. Mol Cell Probes. 49, 101475 (2020).
- Fu, X., Sun, J., Ye, Y., Zhang, Y., Sun, X. A rapid and ultrasensitive dual detection platform based on Cas12a for simultaneous detection of virulence and resistance genes of drug-resistant Salmonella. Biosens Bioelectron. 195, 113682 (2022).
- Habimana, J. D., et al. Mechanistic insights of CRISPR/Cas nucleases for programmable targeting and early-stage diagnosis: A review. Biosens Bioelectron. 203, 114033 (2022).
- Liang, Y., Lin, H., Zou, L., Deng, X., Tang, S. Rapid detection and tracking of Omicron variant of SARS-CoV-2 using CRISPR-Cas12a-based assay. Biosens Bioelectron. 205, 114098 (2022).
- Sivaraman, D., Biswas, P., Cella, L. N., Yates, M. V., Chen, W. Detecting RNA viruses in living mammalian cells by fluorescence microscopy. Trends Biotechnol. 29 (7), 307-313 (2011).
- Wang, I. H., Burckhardt, C. J., Yakimovich, A., Greber, U. F. Imaging, tracking and computational analyses of virus entry and egress with the cytoskeleton. Viruses. 10 (4), 166 (2018).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved