طريقة LipidUNet القائمة على التعلم الآلي لتوصيف وقياس رواسب الدهون باستخدام ظهارة صبغة الشبكية المشتقة من iPSC
In This Article
Summary
أمراض العين التنكسية التي تؤثر على طبقة ظهارة الصباغ في شبكية العين لها أصول أحادية المنشأ ومتعددة الجينات. تم تطوير العديد من نماذج الأمراض وتطبيق برمجي ، LipidUNet ، لدراسة آليات المرض ، وكذلك التدخلات العلاجية المحتملة.
Abstract
ظهارة الشبكية الصبغية (RPE) هي طبقة أحادية من الخلايا السداسية الموجودة في الجزء الخلفي من العين. يوفر التغذية والدعم للمستقبلات الضوئية والشعيرات الدموية المشيمية ، ويؤدي البلعمة للأجزاء الخارجية للمستقبلات الضوئية (POS) ، ويفرز السيتوكينات بطريقة مستقطبة للحفاظ على توازن الشبكية الخارجية. يؤدي RPE المختل وظيفيا ، الناجم عن الطفرات والشيخوخة والعوامل البيئية ، إلى تنكس طبقات الشبكية الأخرى ويسبب فقدان البصر. السمة الظاهرية المميزة لتدهور RPE هي الرواسب الغنية بالدهون داخل الخلايا وشبه الخلوية. هذه الرواسب هي نمط ظاهري شائع عبر الأمراض التنكسية الشبكية المختلفة. لإعادة إنتاج النمط الظاهري لرواسب الدهون لتنكس الشبكية أحادي المنشأ في المختبر ، تم إنشاء RPE المستحث المشتق من الخلايا الجذعية متعددة القدرات (iRPE) من الخلايا الليفية للمرضى. تم تغذية خطوط الخلايا المتولدة من المرضى الذين يعانون من مرض Stargardt ومرض تنكس الشبكية المتأخر (L-ORD) بنقاط البيع لمدة 7 أيام لتكرار الوظيفة الفسيولوجية RPE ، والتي تسببت في علم الأمراض الناجم عن البلعمة POS في هذه الأمراض. لإنشاء نموذج للتنكس البقعي المرتبط بالعمر (AMD) ، وهو مرض متعدد الجينات مرتبط بتنشيط مكمل بديل ، تم تحدي iRPE مع السموم التأقانية التكميلية البديلة. تم توصيف رواسب الدهون داخل الخلايا وشبه الخلوية باستخدام أحمر النيل ، البورون ديبيروميثين (BODIPY) ، والبروتين الشحمي E (APOE). لتحديد كثافة رواسب الدهون ، تم تطوير برنامج قائم على التعلم الآلي ، LipidUNet. تم تدريب البرنامج على صور الإسقاط القصوى الكثافة ل iRPE على أسطح الثقافة. في المستقبل ، سيتم تدريبه على تحليل الصور ثلاثية الأبعاد (3D) وتحديد حجم قطرات الدهون. سيكون برنامج LipidUNet موردا قيما لاكتشاف الأدوية التي تقلل من تراكم الدهون في نماذج الأمراض.
Introduction
ظهارة الشبكية الصبغية (RPE) هي طبقة أحادية من الخلايا تقع في الجزء الخلفي من العين بجوار المستقبلات الضوئية للشبكية. يلعب RPE دورا حيويا في الحفاظ على الرؤية السليمة من خلال توفير الدعم الأيضي والهيكلي للمستقبلات الضوئية. تتميز خلايا RPE السليمة بمورفولوجيا سداسية مميزة. وهي متصلة بواسطة تقاطعات ضيقة ، والتي تسمح ل RPE بالعمل كحاجز بين choriocapillaris الموجود على جانبها القاعدي والمستقبلات الضوئية الموجودة قميا. للحفاظ على النظام البيئي للشبكية ، يقوم RPE بنقل المستقلبات الرئيسية ، على سبيل المثال ، الجلوكوز ، إلى المستقبلات الضوئية بطريقة تقلل من استهلاك الجلوكوز في RPE1. بسبب هذا القيد ، يعتمد RPE على مستقلبات أخرى للحفاظ على احتياجاتهم الأيضية ، بما في ذلك الأحماض الدهنية ، والتي يحولها RPE إلى كيتونات من خلال أكسدة β2. بالنظر إلى ميل RPE إلى استخدام الأحماض الدهنية ، والتي من المحتمل أن يتم إعادة تدويرها من هضم الجزء الخارجي لمستقبلات الضوء (POS) ، كمصدر للطاقة ، فإن التغييرات الضارة في مسارات معالجة الدهون في RPE غالبا ما تؤدي إلى أمراض الشبكية التنكسية أحادية المنشأ ومتعددة الجينات3.
كما تم ربط التنكس البقعي المرتبط بالعمر (AMD) ، وهو مرض تنكسي متعدد الجينات في العين يسبب تنكس RPE ، بالالتهام الذاتي الشاذ واستقلاب الدهون في طبقة RPE الأحادية. يؤدي فشل طبقة أحادية RPE المختلة وظيفيا في معالجة نقاط البيع وأداء وظائف حرجة أخرى إلى رواسب خارج الخلية (RPE) تسمى الرواسب الخطية القاعدية (BLinD) الموجودة بين RPE وغشاء Bruch - وهي السمة المميزة لأمراض AMD. تشمل المكونات الرئيسية ل BLinD البروتينات الدهنية ، وأكثرها وفرة هو صميم البروتين الشحمي E (APOE)4. يمكن أن يؤدي تراكم الطبقات الرقيقة من BLinD إلى دروسين ناعم ، والذي يعرف بأنه أحد الأعراض السريرية ل AMD 5,6.
أظهرت عدة مجموعات أن الخلايا الجذعية المشتقة في نماذج الأمراض المختبرية التي تسبب اختلال وظيفي في RPE تتميز بتراكم الدهون دون RPE7،8،9. Hallam et al. (2017) ولدت RPE المستحثة المشتقة من الخلايا الجذعية متعددة القدرات (iRPE) من المرضى الذين يعانون من مخاطر عالية للإصابة ب AMD بسبب تعدد الأشكال في جين CFH. أظهر iRPE تراكم drusen ، كما هو موضح بواسطة APOE ، وتراكمت رواسب RPE عالية الخطورة أكبر من iRPE الناتجة عن المرضى منخفضيالخطورة 10.
لإنشاء نموذج في المختبر يلخص السمات الخلوية المميزة ل AMD ، مثل قطرات الدهون وترسب drusen ، تم إنشاء خطوط iRPE المتولدة من عينات دم المريض باستخدام بروتوكول موجه تنمويا تم نشره مسبقا11. تم إخضاع iRPE لمصل بشري متمم (CC-HS) ، وهو محلول يحتوي على السموم التأقانية التي تحاكي أحد الأسباب المحتملة ل AMD: زيادة إشارات المتممة البديلة8. تم قياس الترسب الخلوي وشبه الخلوي الناتج لرواسب الدهون باستخدام علامات الدهون والبروتين الدهني شائعة الاستخدام ، APOE ، Nile Red ، و BODIPY. من خلال هذه العلامات ، تبين أن الإشارات التكميلية المنشطة عبر CC-HS أدت إلى تفاقم تراكم الدهون في خلايا iRPE8.
لتطوير نموذج مرض تنكسي أحادي المنشأ في الشبكية ، تم تطوير خطوط iRPE من المرضى الذين يعانون من مرض Stargardt ، وهو مرض ناتج عن طفرات في جين ABCA4 في RPE. لقد ثبت سابقا أنه عندما يتم التخلص من ABCA4 ، يتراكم A2E lipofuscin ، وهو رواسب داخل الخلايا معروفة باحتوائها على مستويات عالية من الدهون الفوسفاتية ومنتجات بيروكسيد الدهون المعتمدة على الضوء ، داخل RPE12. تم تطوير خطوط خروج المغلوب ABCA4 جنبا إلى جنب مع خطوط المريض ، وكلاهما تعرض لتغذية نقاط البيع. أظهر Stargardt iRPE علم الأمراض الناجم عن البلعمة POS ، مما يدل على زيادة تراكم الدهون كميا بواسطة تلطيخ BODIPY. وخضعت مركبات RPE المستمدة من ABCA4 KO iPSCs لمعاملة CC-HS؛ أظهر القياس الكمي لإشارة BODIPY خللا في معالجة الدهون في نموذج مرض Stargardt أيضا9.
نظرا لانتشار هذه الأمراض والحاجة إلى علاجات فعالة ، إلى جانب نماذج الأمراض ذات الصلة الموضحة أعلاه ، هناك حاجة إلى إنشاء طرق قوية لتحديد فعالية العلاجات المحتملة. لتحديد رواسب الدهون بطريقة موضوعية وآلية وموحدة ، تم إنشاء برنامج قائم على التعلم الآلي ، LipidUNet ، بحيث ، عند إقرانه بأدوات تحليل القناع ، يمكن تحديد ترسب الدهون بسرعة وفعالية باستخدام العلامات الشائعة Nile Red و BODIPY و APOE. يمكن بعد ذلك تحليل الإحصائيات الموجزة التي تم الحصول عليها باستخدام خط أنابيب التحليل هذا وعرضها بيانيا ، مما يسمح بمقارنة سهلة لظروف العلاج. يظهر الرسم التخطيطي للبروتوكول في الشكل 1.
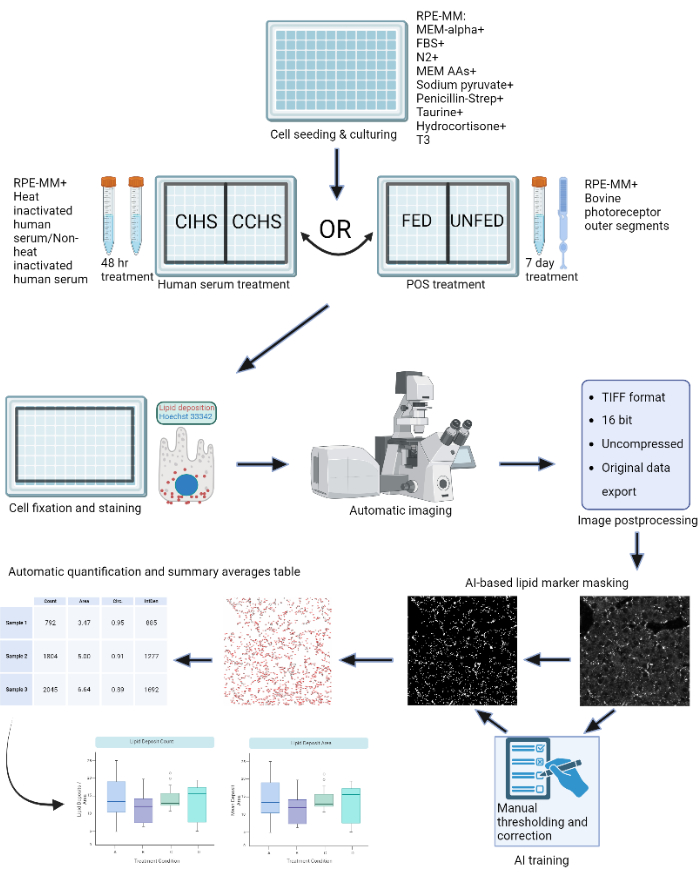
الشكل 1: رسم تخطيطي للبروتوكول: تزرع خلايا RPE على صفيحة من 96 بئرا ويتم تحديها بمصل بشري نشط أو شرائح خارجية بقري منقاة لنمذجة أنواع مختلفة من تنكس الشبكية في المختبر. يتم تثبيت خلايا RPE وتلطيخها لرواسب البروتين الدهني مع النيل الأحمر ، BODIPY ، و APOE. يتم استخدام مجهر متحد البؤر للحصول على مداخن Z من جزيئات الدهون ذات العلامات الفلورية ، والتي تتم معالجتها لاحقا في إسقاطات كثافة قصوى 2D. تم تدريب خوارزمية التعلم الآلي على التعرف على جزيئات البروتين الدهني وتقسيمها بشكل صحيح. يتم إنشاء جداول موجزة تحتوي على عدد الجسيمات ومقاييس الأشكال المختلفة ويمكن استخدامها للتخطيط اللاحق والتحليل الإحصائي. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
Protocol
تلتزم جميع خطوات البروتوكول بالمبادئ التوجيهية التي وضعتها لجنة أخلاقيات البحوث البشرية التابعة للمعاهد الوطنية للصحة. تمت الموافقة على عمل الخلايا الجذعية وجمع عينات المرضى من قبل مجلس المراجعة المؤسسية لعلم الأعصاب المشترك (CNS IRB) التابع لمكتب حماية البحوث البشرية (OHRP) ، المعاهد الوطنية للصحة ، وفقا لإرشادات 45 CFR 46 لحكومة الولايات المتحدة. تم جمع عينات المرضى باستخدام نموذج الموافقة المعتمد من CNS IRB وفقا للمعايير التي حددها إعلان هلسنكي بموجب رقم البروتوكول NCT01432847 (https://clinicaltrials.gov/ct2/show/NCT01432847?cond=NCT01432847&draw=2&rank=1).
1. جيل iRPE
- قم بإنشاء iRPE من iPSC المشتق من دم المريض باتباع البروتوكول المنشور بواسطة Sharma et al. ، 202211 (الشكل 2 والشكل 3).
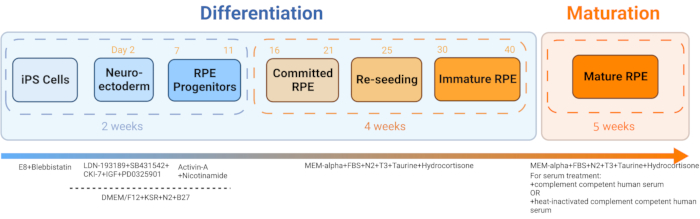
الشكل 2: رسم تخطيطي لتمايز ونضج iRPE. لتوليد iRPE ، تم اتباع بروتوكول تمايز ثابت ، وسمح للخلايا بالنضوج لمدة 5 أسابيع. تعمل زراعة الخلايا الناتجة كنموذج في المختبر يمكن التلاعب به بعلاجات مختلفة لتقليد خلل RPE في أمراض مثل AMD ومرض Stargardt. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
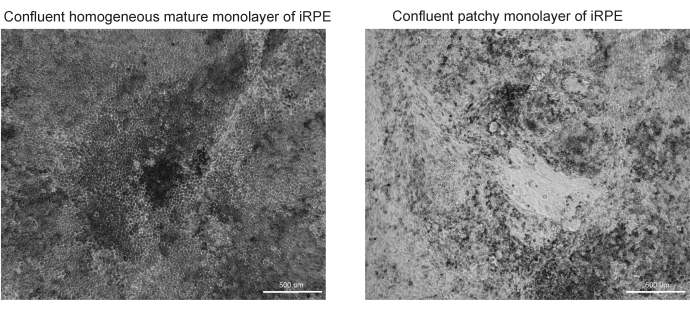
الشكل 3: صور تمثيلية لتمايز ونضج RPE الناجح وغير الناجح. يتم عرض صورتين ساطعتين بتكبير 10x ل TJP1 RPE في اليوم 42 من بروتوكول iRPE. (أ) سيظهر التمايز والنضج الناجح RPE متقاربا مع تصبغ ومورفولوجيا متعددة الأضلاع. (ب) التمايز والنضج غير الناجحين سيظهران تجمعات من الخلايا الميتة، كما هو موضح هنا. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
2. إعداد وسائط صيانة RPE (RPE-MM)
- قم بإذابة مكمل N2 عند 4 درجات مئوية طوال الليل. قم بإذابة جميع الكواشف الأخرى في درجة حرارة الغرفة (RT).
- في ظل ظروف معقمة ، أضف الكواشف المدرجة في الجدول 1 في عوامل التخفيف المدرجة ، وفقا للبروتوكول الذي وضعه Sharma et al. ، 202211.
- امزج الوسائط جيدا وقم بترشيحها باستخدام وحدة ترشيح 0.22 ميكرومتر.
ملاحظة: الوسائط مناسبة للاستخدام في غضون 2 أسابيع إذا تم تخزينها في 4 درجات مئوية.
3. 96 بئر لوحة البذر
- قم بإذابة حصة من الفيترونيكتين في RT لمدة 3-5 دقائق أو حتى يذوب الجليد تماما.
- قم بتخفيف الفيترونيكتين باستخدام محلول ملحي مخزن بالفوسفات من Dulbecco (DPBS) للحصول على محلول العمل المطلوب باستخدام تخفيف 1: 200 (vitronectin: DPBS). للحصول على لوحة 96 بئرا ، قم بتغطية كل بئر ب 200 ميكرولتر من محلول العمل.
- الجمع بين مثبط ROCK المذاب (Y-27632 ثنائي هيدروكلوريد) مع RPE-MM عند تخفيف 1: 1000 لتحقيق تركيز نهائي قدره 10 ميكرومتر. هذه هي وسائط الطلاء لخلايا RPE.
- قم بإذابة قارورة iRPE باستخدام نظام إذابة الخلايا الآلي وانقل تعليق خلية iRPE إلى أنبوب سعة 50 مل.
- تمييع تعليق الخلية مع وسائط الطلاء في تخفيف 1:10. جهاز طرد مركزي الأنبوب عند 400 × جم لمدة 5 دقائق.
- استنشاق بعناية طاف وإعادة تعليق الخلايا في 10 مل من وسائط الطلاء.
- امزج 400 ميكرولتر من وسائط الطلاء مع 100 ميكرولتر من محلول الخلية المعاد تعليقه لعد الخلايا. استخدم هذه القسمة لتحديد تركيز الخلية القابل للحياة لتعليق الخلية باستخدام عداد صلاحية الخلية.
- قم بتخفيف تعليق الخلية باستخدام وسائط الطلاء إلى تركيز نهائي يبلغ 60000 خلية / مل.
- قم بشفط محلول طلاء vitronectin تماما من لوحة 96 بئرا وتوزيع 200 ميكرولتر من تعليق الخلية في كل بئر. سيكون هناك ما يقرب من 12000 خلية / بئر أو ~ 200 خلية / مم2.
- احتضان ألواح الخلايا المصنفة لمدة 48 ساعة عند 37 درجة مئوية و 5٪ CO2. بعد 48 ساعة، قم بتغيير الوسائط إلى RPE-MM بدون ملحق مثبطات ROCK. قم بتغيير الوسائط كل 2-3 أيام خلال فترة النضج التي تبلغ 5 أسابيع.
4. نماذج المرض في المختبر
- مكمل علاج المصل البشري المختص (CC-HS)
- قم بإذابة المصل البشري المكمل عند 4 درجات مئوية طوال الليل.
- تحضير CC-HS واستكمال وسائط المصل البشري غير الكفء (CI-HS).
- لتحضير وسائط CC-HS بنسبة 5٪ ، امزج المصل البشري المكمل المذاب مع RPE-MM بتخفيف 1:20. قم بتصفية المحلول من خلال مرشح وسائط 0.22 ميكرومتر قبل الاستخدام.
- لتحضير 5٪ مكمل لوسائط المصل البشري غير الكفء (CI-HS) ، قم أولا بتسخين CC-HS في حمام مائي بدرجة حرارة 57 درجة مئوية لمدة 30 دقيقة ثم اخلطه مع وسائط الاستزراع بتخفيف 1:20. قم بتصفية المحلول من خلال مرشح وسائط 0.22 ميكرومتر قبل الاستخدام.
- يعالج المصل الخلايا ب 200 ميكرولتر من وسائط CC-HS 5٪ أو 5٪ CI-HS لوقت حضانة إجمالي يبلغ 48 ساعة ، مما ينعش الوسائط بعد 24 ساعة.
- اغسل الخلايا باستخدام 1x DPBS وقم بتثبيتها باستخدام 4٪ بارافورمالدهيد لمدة 20 دقيقة في RT. اغسل مرة أخرى باستخدام 1x DPBS وقم بتخزين العينات عند 4 درجات مئوية ، مغمورة في 200 ميكرولتر من DPBS.
- اختياري: إذا رغبت في ذلك ، قم بتحليل الخلايا من اللوحة لإظهار ترسب الدهون الفرعية RPE فقط.
- لتحلل الخلايا وترك رواسب الدهون فقط ، قم بإزالة الوسائط وإضافة 200 ميكرولتر من الماء منزوع الأيونات إلى كل بئر.
- احتضان لمدة 10-15 دقيقة ، ماصة صعودا وهبوطا حتى تتم إزالة الخلايا. اغسل مرة أخرى ب 200 ميكرولتر من الماء منزوع الأيونات وقم بإصلاح الخلايا على الفور باستخدام 4٪ بارافورمالدهيد.
- تأكيد فعالية إزالة الخلايا مع تلطيخ النووي باستخدام Hoechst. أضف Hoechst في تخفيف 1: 2000 إلى محلول DPBS واحد يحتوي على 1٪ ألبومين مصل بقري (BSA) ، 0.5٪ توين 20 ، و 0.5٪ Triton-X 100. احتضان في RT لمدة 1 ساعة في الظلام. بعد ذلك ، يغسل مع 1x DPBS.
- معالجة الجزء الخارجي للمستقبلات الضوئية (POS) على iRPE
- إعداد نقاط البيع
- قم بإزالة أنبوب الحبيبات POS من تخزين -80 درجة مئوية وقم بإذابة الثلج طوال الليل عند 4 درجات مئوية في دلو ثلج مغطى.
- تحضير محلول غسيل POS عن طريق خلط 10 جم من السكروز في 40 مل من H 2 O منزوع الأيونات المزدوج (ddH2O).
- يسخن المزيج على حرارة 40-50 درجة مئوية مع التحريك برفق لمدة 15 دقيقة. أضف 840 مجم من بيكربونات الصوديوم إلى الخليط وحركه أثناء التسخين لمدة 10 دقائق.
- اضبط الحجم الإجمالي للمخزن المؤقت لغسيل نقاط البيع إلى 100 مل باستخدام ddH2O واضبط الرقم الهيدروجيني للمحلول على 8.3 مع 1 N HCl أو 1 N NaOH ، حسب الحاجة. قم بتصفية محلول الغسيل باستخدام مرشح 0.22 ميكرومتر.
ملاحظة: يمكن إيقاف البروتوكول مؤقتا هنا ؛ يمكن تخزين المخزن المؤقت لغسيل نقاط البيع عند 4 درجات مئوية طوال الليل. - بمجرد إذابتها ، قم بتعليق الحبيبات في 15 مل من المخزن المؤقت لغسيل نقاط البيع. كن لطيفا أثناء تعليق الحبيبات لضمان سلامة نقاط البيع. أجهزة الطرد المركزي تعليق نقاط البيع عند 600 × جم عند 4 درجات مئوية لمدة 20 دقيقة ثم نضح المادة الطافية.
- أعد تعليق حبيبات نقاط البيع في 10 مل من المخزن المؤقت لغسيل نقاط البيع.
- قم بإزالة حصة 100 ميكرولتر من المخزن المؤقت لغسيل POS + POS (محلول POS) وقم بتخفيفه في 400 ميكرولتر من 1x DPBS. انشر 50 ميكرولتر من محلول نقاط البيع المخفف على صفيحة أجار الدم وصفيحة أغاروز للتحقق من الملوثات البكتيرية والفطرية. قم بإعداد عناصر تحكم إيجابية لكل منها واحتضان جميع الألواح لمدة 48 ساعة عند 37 درجة مئوية.
- قم بإجراء مقايسة qPCR عن طريق إضافة 1 ميكرولتر من محلول نقاط البيع إلى بئر الكشف لاختبار الميكوبلازما. لتضخيم شظايا الحمض النووي ، قم بإجراء 40 دورة من التمسخ (95 درجة مئوية ، 15 ثانية) ، والتلدين والاستطالة (60 درجة مئوية ، 1 دقيقة). البادئات الأمامية والخلفية للكشف عن الميكوبلازما في عينة نقاط البيع هي كما يلي:
التمهيدي الأمامي: GGA TTA GAT ACC CTG GTA GTC CAC G
التمهيدي العكسي: CGT CAA TTC CTT TAA GTT TCA CTC TTG GC - قم بقياس تركيز نقاط البيع باستخدام محلل الخلية والقسمة حسب الحاجة. بالنسبة لبئر واحد مكون من 96 لوحة بئر مع خلايا RPE ، يكفي 3 × 106 نقاط بيع. النسبة المطلوبة هي 10 خلية POS / RPE. قم بتخزين الحصص عند 80 درجة مئوية لاستخدامها في المستقبل.
- إضافة نقاط البيع إلى الخلايا
- قم بإذابة قوارير نقاط البيع في حمام جليدي.
- امزج الكمية المحسوبة من نقاط البيع المحضرة مع RPE-MM وعالج الخلايا بنقاط البيع مرة واحدة يوميا لمدة 7 أيام.
ملاحظة: تحضير محلول نقاط البيع طازجا يوميا. - اغسل الخلايا باستخدام 1x DPBS ثم ثبتها بنسبة 4٪ بارافورمالدهيد لمدة 20 دقيقة في RT. اغسل باستخدام DPBS مرة أخرى وقم بتخزين العينات عند 4 درجات مئوية ، مغمورة في 200 ميكرولتر من DPBS.
- إعداد نقاط البيع
5. تلطيخ لرواسب RPE الفرعية
- بروتوكول تلطيخ النيل الأحمر
- بعد تثبيت PFA ، اغسل العينات 3 مرات باستخدام 1x DPBS.
ملاحظة: إذا لم يتم استخدامه على الفور ، يمكن إيقاف البروتوكول مؤقتا هنا ، ولكن يجب تخزين العينات في محلول 1x DPBS + 0.02٪ أزيد الصوديوم عند 4 درجات مئوية. - لتحضير محلول مرق النيل الأحمر، قم بإذابة مسحوق النيل الأحمر في الأسيتون بتركيز 3 ملغ/مل. احتضان لمدة 15 دقيقة في RT مع الخلط الدوري. قم بتصفية المحاليل باستخدام مرشح 0.22 ميكرومتر مرة أو مرتين حسب مستوى الراسب المتبقي في المحلول.
ملاحظة: حماية محلول المخزون من الضوء. - لإعداد حل العمل ، قم بتخفيف حل المخزون بنسبة 1: 500 في 1x DPBS. أضف 200 ميكرولتر من محلول العمل إلى العينة لمدة 30 دقيقة في RT على شاكر واحميه من الضوء.
- اغسل 3 مرات باستخدام 1x PBS وقم بتخزين العينات في درجة حرارة 4 درجات مئوية ، مغمورة في 200 ميكرولتر من DPBS.
ملاحظة: في حالة إجراء تجربة على الآبار المرسلة بدلا من لوحة 96 بئرا ، يمكن تركيب العينات على شريحة بها وسائط تثبيت ، ومغطاة بغطاء زجاجي ، ومختومة بطلاء أظافر شفاف. يجب توخي الحذر لتركيب العينة بحيث تكون الخلايا متجهة لأعلى.
- بعد تثبيت PFA ، اغسل العينات 3 مرات باستخدام 1x DPBS.
- بروتوكول تلطيخ BODIPY
- بالنسبة لمحلول المخزون ، قم بإذابة BODIPY في ثنائي ميثيل سلفوكسيد اللامائي (DMSO) للوصول إلى تركيز مخزون يبلغ 3.8 mM.
- بالنسبة للعينات الثابتة PFA ، قم بتخفيف مخزون BODIPY عند 1: 300 في 1x DPBS. أضف 200 ميكرولتر إلى الخلايا واحتضانها طوال الليل على الروك في RT.
- اغسل 3 مرات باستخدام 1x DPBS وقم بتخزين العينات في درجة حرارة 4 درجات مئوية ، مغمورة في 200 ميكرولتر من DPBS.
ملاحظة: في حالة إجراء تجربة على الآبار المرسلة بدلا من لوحة 96 بئرا ، يمكن تركيب العينات على شريحة بها وسائط تثبيت ، ومغطاة بغطاء زجاجي ، ومختومة بطلاء أظافر شفاف. يجب توخي الحذر لتركيب العينة بحيث تكون الخلايا متجهة لأعلى.
- بروتوكول APOE المناعي
- اجمع 1x DPBS مع 1٪ ألبومين مصل بقري (BSA) و 0.5٪ Tween 20 و 0.5٪ Triton-X 100 لإنشاء محلول عازل.
- بالنسبة للعينات الثابتة PFA ، قم بحظر العينة واختراقها في 200 ميكرولتر من المحلول العازل لمدة 1 ساعة عند RT.
- أضف الجسم المضاد الأولي APOE المخفف عند 1: 100 في المحلول العازل واحتضانه طوال الليل في RT.
- في اليوم التالي ، اغسل العينات 3 مرات باستخدام 1x DPBS.
- أضف جسما مضادا ثانويا عند تخفيف 1: 1000 في المحلول العازل وأضف 200 ميكرولتر من المحلول إلى الخلايا لمدة 1 ساعة في RT.
- اغسل 3 مرات باستخدام 1x DPBS وقم بتخزين العينات عند 4 درجات مئوية ، مغمورة في 200 ميكرولتر من DPBS.
ملاحظة: في حالة إجراء تجربة على الآبار المرسلة بدلا من لوحة 96 بئرا ، يمكن تركيب العينات على شريحة بها وسائط تثبيت (Fluoromount) ، ومغطاة بغطاء زجاجي ، ومختومة بطلاء أظافر شفاف. يجب توخي الحذر لتركيب العينة بحيث تكون الخلايا متجهة لأعلى.
6. أتمتة الصور ومعالجتها
- المسح الآلي للصور
ملاحظة: تم استخدام مجهر المسح المقلوب متحد البؤر Zeiss LSM 800 وبرنامج ZEN 3.2 (الإصدار الأزرق) في هذه الدراسة. تأكد من تسخين اللوحة المكونة من 96 بئرا إلى RT لمدة لا تقل عن 60 دقيقة قبل التصوير لتجنب انحراف المستوى البؤري أثناء الفحص بسبب تغير معامل الانكسار للوسط مع تغير درجة الحرارة.- باستخدام مجهر متحد البؤر وهدف 40x ، قم بإنشاء ملف تعريف مسح مع قنوات الفلورسنت المناسبة لعلامة الدهون المستخدمة وأي أجسام مضادة إضافية.
- استخدم خانة الاختيار اللوحات لإعداد أتمتة الصور. لمعايرة لوحة 96 بئرا ، تأكد من إدخال قياسات حامل العينة الصحيحة وتحديدها. ثم ، انقر فوق معايرة زر لمعايرة اللوحة وفقا للتعليمات ، الأمر الذي يتطلب استخدام هدف 10x.
- اختر طريقة عرض الإعداد المتقدم لتحديد الآبار المناسبة وإضافة 3 نقاط تصوير مختلفة بالقرب من مركز البئر باستخدام وظيفة المواضع . يمكن القيام بذلك يدويا ضمن علامة التبويب الفرعية الموضع أو بشكل عشوائي باستخدام علامة التبويب إعداد الموضع وتحديد الإعداد بواسطة شركة الاتصالات. كرر لجميع الآبار من نفس تلطيخ.
- للحصول على التركيز البؤري الأمثل وتحديد موضع مكدس Z أثناء التشغيل التلقائي، انتقل إلى علامة التبويب استراتيجية التركيز لتحديد استخدام قيم Focus Surface/Z المعرفة بواسطة إعداد اللوحات. يمكن أن تستخدم الطرق البديلة استراتيجيات تركيز أخرى ، ولكن يوصى باستخدام هذا الإعداد للحصول على نتائج أكثر اتساقا.
- ضمن علامة التبويب التجانب، انقر فوق التحقق من المواضع وقم بتعيين مستوى Z المركزي يدويا لكل موضع. ستطلب الإعدادات الموجودة في علامة التبويب الفرعية "خيارات" الحصول على الصور ، لذا تحقق من ذلك قبل البدء في الصورة. للحصول على صور بالترتيب الذي تم تحديد المواضع به، قم بإلغاء تحديد خانات الاختيار مناطق/مواضع التجانب وآبار حاملة/حاوية. حدد تقسيم المشاهد إلى ملفات منفصلة لسهولة معالجة الصور.
- تأكد من تعيين علامة التبويب Z-Stack إلى المركز، وإدخال نطاق لتفضيلات المستخدم، وتحديد الزر الأمثل لتعيين الفاصل الزمني للشريحة.
- بعد تحسين علامات تبويب وضع الاستحواذ والقنوات واستراتيجية التركيز و Z-Stack و Tiles ، ابدأ التجربة.
- معالجة الصور
- باستخدام طريقة معالجة الصور الدفعية، قم بإنشاء أقصى إسقاطات لكل مكدس Z باستخدام طريقة عمق التركيز الممتد .
- باستخدام طريقة معالجة الصور الدفعية، قم بتصدير الحد الأقصى لملفات الإسقاط كصور TIFF 16 بت. اضبط الضغط على لا شيء وتأكد من فحص البيانات الأصلية . يجب أن تكون الصورة الناتجة عبارة عن TIFF بتدرج رمادي أقصى للإسقاط لقناة التألق التي يتم التعبير عن علامة الدهون عليها.
7. التجزئة والقياس الكمي
ملاحظة: تم تدريب برنامج LipidUNet على صور 40x من لوحة 96 بئر. يوصى بشدة باستخدام الصور التي تم الحصول عليها باستخدام هدف 40x.
- تثبيت برنامج LipidUNet. يمكن تنزيل LipidUNet من مستودع GitHub التالي: https://github.com/RPEGoogleMap/LipidUNet
- حدد صور TIFF التي تمثل إما Nile Red أو Bodipy أو APOE وانقلها إلى مجلد باسم imgs داخل دليل يسمى إما Nile_Red أو Bodipy أو APOE ، اعتمادا على الطريقة المستخدمة.
ملاحظة: يجب استخدام اصطلاحات التسمية الدقيقة لبرنامج LipidUNet للتعرف على الدلائل. - افتح برنامج LipidUNet (الشكل 4).
- في علامة التبويب توقع في البرنامج ، حدد الدليل ذي الصلة (Nile_Red أو Bodipy أو APOE) بالنقر فوق علامة الحذف والانتقال إلى الدليل المسمى. تأكد من أن برنامج LipidUNet قد حدد الصور بشكل صحيح عن طريق التحقق من إدخال الفئة.
- حدد عتبة احتمال للخوارزمية بين 0.01 و 0.99. ستؤدي القيمة الأعلى إلى القضاء على المزيد من الإيجابيات الخاطئة ولكنها قد تسبب المزيد من السلبيات الخاطئة ، وقد تؤدي القيم المنخفضة إلى تقديم المزيد من الإيجابيات الخاطئة مع التخلص من المزيد من السلبيات الخاطئة. القيمة 0.65 هي القيمة الافتراضية ويوصى بها.
- انقر على توقع.
ملاحظة: سيقوم البرنامج بالتكرار من خلال جميع الصور تلقائيا وإنشاء مجلد جديد يسمى predicted_masks في الدليل المحدد. - استخدم أداة تحليل القناع للتكرار من خلال الأقنعة المولدة وتوفير عدد كمي لرواسب الدهون العتبة من صور القناع .
- تحليل بيانات الأعداد التي تم إنشاؤها لمقارنة ظروف العلاج.
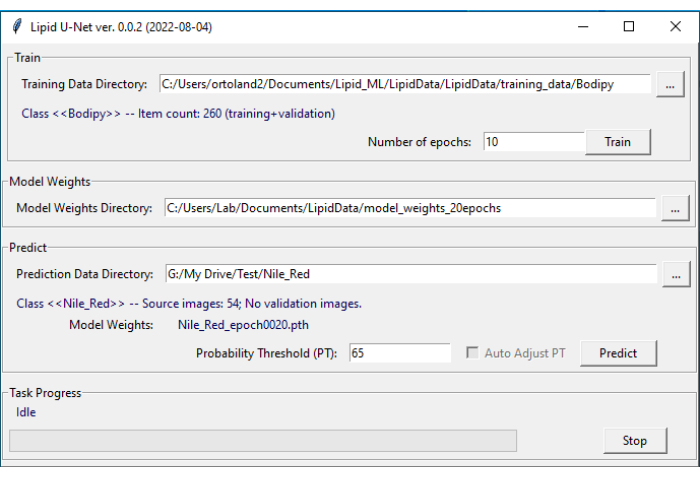
الشكل 4: واجهة مستخدم LipidUNet. يحتوي برنامج LipidUNet على أقسام مختلفة لاختيارها لدليل بيانات التدريب ، حيث تم تحديد صور رواسب الدهون بشكل صحيح ؛ دليل أوزان النموذج ، الذي يتم إنتاجه من بيانات التدريب ؛ ودليل بيانات التنبؤ الذي سيقوم المستخدم بإدخال صوره للتقسيم. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
النتائج
يوفر هذا البروتوكول سير عمل لتحديد رواسب الدهون الملطخة ب Nile Red و BODIPY و APOE. يمكن للبرنامج المطور تحديد رواسب الدهون وقياسها تلقائيا ويعمل بشكل أفضل عندما يتم تحسين البروتوكول المحدد. تم تضمين أمثلة على RPE المتمايز بنجاح (الشكل 3A) و RPE ضعيف التمايز (الشكل 3B) ، حيث تؤثر جودة نموذج الخلية بشكل كبير على جودة تجزئة الصورة المناسبة.
تم تحديد اثنتين من العلامات الثلاث الموصوفة في البروتوكول ، النيل الأحمر و BODIPY ، كنقاط دائرية صغيرة ساطعة بشكل واضح في صور الفلورسنت (الشكل 5 والشكل 6). وستكون الصورة "الإيجابية" من البروتوكول بمثابة تحديد مناسب لهذه الرواسب المتميزة (الشكل 5A-D والشكل 5E-H). قد تظهر النتيجة "السلبية" تقسيما غير صحيح للصورة عن طريق الخطأ في مضان الخلفية كرواسب ، إما بسبب تلطيخ ضعيف (الشكل 6A-C والشكل 6D-F) أو بسبب كثافة الخلفية العالية (الشكل 6G-I).
تحتوي رواسب APOE على مجموعة متنوعة من الأحجام والأشكال ، وتظهر بيضاوية أو غير منتظمة بدلا من الرواسب الدائرية لأحمر النيل و BODIPY. هذه الرواسب هي أيضا أقل نقطا ، ويمكن أن تختلف شدة الإشارة بين الرواسب بسبب الاختلافات في نفاذية العينة. سيحدد التحديد الصحيح كل رواسب ، بما في ذلك تلك الأقل تشبعا (الشكل 5I-L) ، في حين أن التجزئة غير الصحيحة لن تلتقط هذه الرواسب (الشكل 6J-L). لذلك ، من المهم تحسين طرق التلوين والتصوير لتجنب الاختلاف الجذري. تتمثل إحدى طرق القيام بذلك في الانتباه بعناية إلى خطوات نفاذية العينة أثناء التلوين المناعي. لتحسين إشارة الفلورسنت ، يمكن تحلل الخلايا قبل التثبيت والتلطيخ المناعي ل APOE ، مما يؤدي إلى تشبع متساو وتجزئة أفضل لرواسب APOE.
كما تتوفر صور مجزأة للخلايا الناضجة على منصة استزراع بخلاف صفيحة بئر 96. تم تشغيل برنامج LipidUNet على صور الخلايا المزروعة على transwell ، وبينما يتم عتبة رواسب الدهون ، وكذلك المسام في غشاء transwell (الشكل 6M-O). بسبب التشابه في الشكل والحجم ، فإن برنامج LipidUNet في شكله الحالي سوف يخفي كل من رواسب الدهون والمسام العابرة بشكل عشوائي.
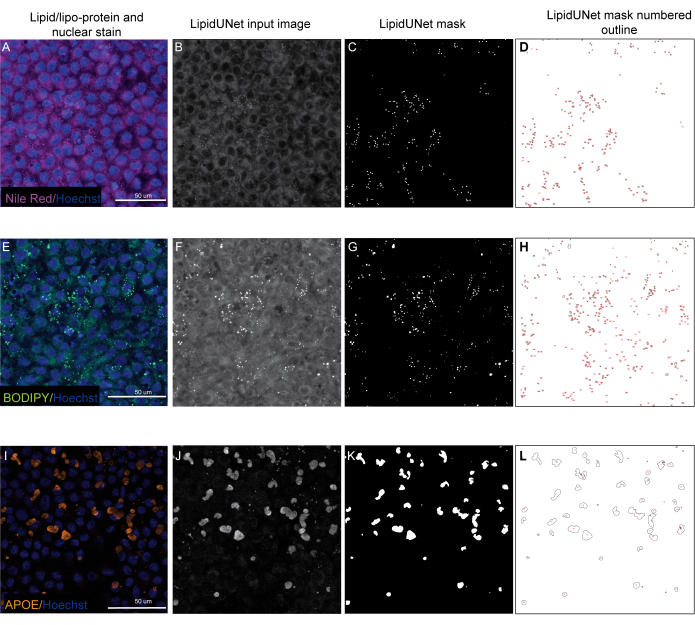
الشكل 5: النتائج التمثيلية. (A ، E ، I) 96 RPE مطلية جيدا ملطخة بتلطيخ Hoechst النووي (الأزرق) وإما أحمر النيل (أرجواني) أو BODIPY (أخضر) أو APOE (برتقالي) وهي أقصى إسقاطات كثافة لمكدس Z. (ب، و، ي) صور إدخال التدرج الرمادي لبرنامج LipidUNet بعد معالجة الصور. (ج، ز، ك) أقنعة تم إنشاؤها بواسطة LipidUNet ، حيث يتم تحديد جميع الرواسب بشكل صحيح. (د، ح، ل) الخطوط العريضة لكل جسيم مقنع مرقمة. تسمح هذه التسميات بتوصيل كل جسيم في الصورة بإدخال في الانتشار مع البيانات الأولية. (A-D) يظهر تلطيخ النيل الأحمر ، والبرنامج قادر على التعرف على الرواسب على الخلفية بدقة على الرغم من ضعف الإشارة. (E-H) يظهر تباينا قويا بين إشارة BODIPY والخلفية ، وهو أمر مثالي. يحدد LipidUNet بشكل صحيح كل إيداع في الصورة. (I-L) يظهر إشارة APOE قوية ويمثل تباين تشبع الإشارة الذي غالبا ما يرى مع هذه البقعة. ومع ذلك ، فإن تجزئة الصورة قادرة على تحديد حدود كل إيداع APOE. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
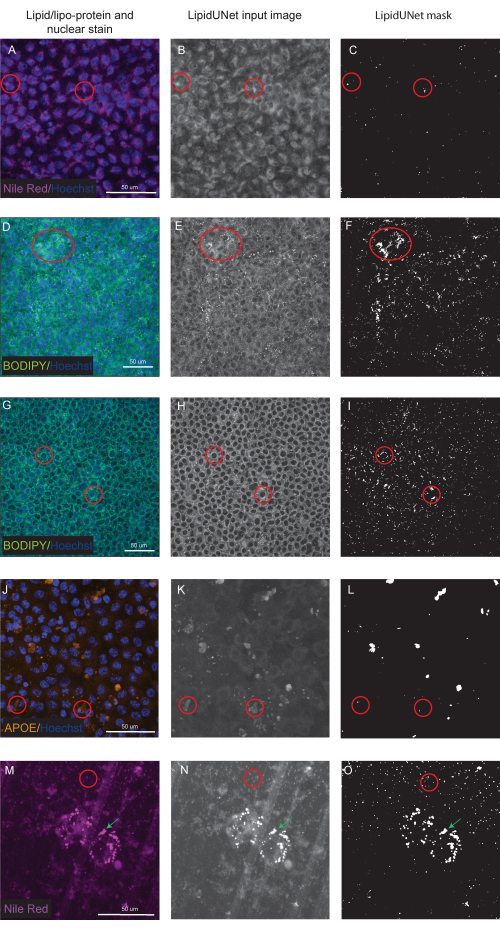
الشكل 6: النتائج دون المستوى الأمثل. (A ، D ، G ، J ، M) 96 RPE مطلي جيدا ملطخة بتلطيخ Hoechst النووي (أزرق) وإما أحمر النيل (أرجواني) أو BODIPY (أخضر) أو APOE (برتقالي) وهي أقصى إسقاطات كثافة لمكدس Z. (ب، ه، ح، ك، ن) صور إدخال التدرج الرمادي لبرنامج LipidUNet بعد معالجة الصور. (ج، و، ط، ل، س) الأقنعة غير الصحيحة التي تم إنشاؤها بواسطة LipidUNet. تشير الدوائر الحمراء إلى المكان الذي حدد فيه البرنامج بشكل غير صحيح رواسب الدهون. (أ-ج) معالجة Nile Red غير صحيحة لأن البرنامج قد حدد تلطيخ الخلفية على أنه إيداع. يمكن أن يحدث هذا في كثير من الأحيان عندما تكون هناك خلفية عالية ولكن القليل من رواسب الدهون في الصورة. يتم عرض مثالين على تلطيخ BODIPY: صورة ذات جودة رديئة بسبب تلطيخ BODIPY الضعيف (D-F) و (G - I) إشارة BODIPY قوية ذات خلفية عالية. في كلتا الحالتين ، لا يستطيع البرنامج التمييز بين رواسب الدهون الدائرية الصغيرة من الحلقة الدائرية الخلفية المحيطة بالنواة. في حين ينبغي تحسين تلطيخ والتصوير لتجنب هذه الأخطاء، يتم تحسين أحدث إصدار من LipidUNet إلى حد كبير لهذه الصور. (ي-ل) تجزئة APOE غير صحيحة. نظرا لأن الرواسب أكثر تنوعا في الحجم وتشبع الإشارة ، يواجه البرنامج صعوبة في التعرف على بعض الرواسب. (م-س) RPE المصنف على ترانسويل وملطخ باللون الأحمر النيل. تظهر هنا شريحة من مكدس Z مع كل من رواسب الدهون الحمراء النيلية ومسام الساقطة. البرنامج غير قادر على التمييز بين الاثنين ، كما هو موضح في الدائرة الحمراء التي تحتوي على مسام transwell والسهم الأخضر الذي يشير إلى رواسب النيل الأحمر. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.

الشكل 7: مقارنة أداة القناع. (أ ، ب ، ج) تم تحديد RPE 96 مطلي جيدا مع كميات متغيرة من ترسب الدهون مع أحمر النيل (أحمر). يتم إخفاء الصور باستخدام ثلاث طرق إخفاء شائعة مختلفة ، Find Maxima و Max Entropy و Renyi Entropy ، ومقارنتها بالقناع الذي تم إنشاؤه بواسطة LipidUNet. الصورة الأصلية مصحوبة بإحصاء يدوي لرواسب الدهون ، بينما تعرض الأقنعة الأعداد المتوقعة حسب كل طريقة تجزئة. تم حساب متوسط معدل الخطأ لكل طريقة تجزئة باستخدام الصيغة التالية: يعني[(|العدد المتوقع - العد اليدوي | / العد اليدوي) × 100]. يحدد القناع الذي تم إنشاؤه بواسطة LipidUNet بشكل أكثر دقة رواسب الدهون عبر الصور ذات الترسيب المتغير عند مقارنته بطرق التقنيع الأخرى (متوسط معدلات الخطأ: 23٪ LipidUnet ، 1164٪ Find Maxima ، 851٪ Max Entropy ، 203٪ Renyi Entropy). يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
| مكون | رقم القط | الأسهم Conc. | النهائي Conc. | مل |
| MEM ألفا | 12571-063 | غير متوفر | 500 | |
| ملحق N2 | 17502-048 | غير متوفر | 1% | 5 |
| FBS معطل حراريا | SH30071.03 | غير متوفر | 5% | 25 |
| نميم نيا | 11140-050 | 10 مللي متر | 0.01 مللي متر | 5 |
| بيروفات الصوديوم | 11360-070 | 100 مللي متر | 1 مللي متر | 5 |
| البنسلين والستربتومايسين | 15140-122 | 10000u / مل | 100 وحدة / مل | 5 |
| توراين | T4571 | 50 ملغ / مل | 250 ميكروغرام / مل | 2.5 |
| الهيدروكورتيزون | H6909 | 18.1 مجم / لتر | 20 ميكروغرام / لتر | 0.553 |
| تي 3 | T5516 | 20 ميكروغرام / لتر | 0.013 ميكروغرام / لتر | 0.33 |
| الحجم الكلي ، مل | 548.383 | |||
الجدول 1: تكوين كاشف RPE-MM. قائمة بالكواشف والتركيزات المثلى ل RPE-MM.
Discussion
يوفر هذا البروتوكول طريقة لتسمية وتصوير وقياس رواسب الدهون بكفاءة في نماذج الأمراض أحادية المنشأ ومتعددة الجينات في المختبر لأمراض العين التنكسية. يمكن تطبيق البرنامج القائم على الذكاء الاصطناعي ، LipidUNet ، على ثلاث علامات دهنية شائعة ، APOE و Nile Red و BODIPY ، ويوفر طريقة سريعة وتلقائية للتحليل تسمح بأن يكون القياس الكمي قياسيا وغير متحيز.
القيد الرئيسي ل LipidUNet هو حقيقة أن مجموعة بيانات التدريب ل الذكاء الاصطناعي اقتصرت على صور تكبير 40x للخلايا المزروعة في لوحة 96 بئرا. نتيجة لمجموعة صور التدريب ، يقتصر LipidUNet ، في شكله الحالي ، على تحليل صور التكبير 40x. يمكن استخدام البرنامج لتحليل صور 40x للخلايا المزروعة على أسطح استزراع أخرى إلى جانب لوحة بئر 96 ، ولكن يجب توخي الحذر لفحص أقنعة الإخراج التي تم إنشاؤها للتحقق من العتبة الدقيقة بواسطة البرنامج. ستكون هناك حاجة إلى المزيد من مجموعات الصور (بتكبيرات مختلفة) لتوسيع نطاق العينات / الصور التي يمكنها تحليلها.
يحتوي البروتوكول على عدة خطوات حاسمة. في خطوة علامة الدهون ، يجب على المستخدم التأكد من أن مركب الملصقات الذي اختاره (BODIPY ، APOE ، Nile Red) قد قام بتسمية عينته بشكل فعال. غالبا ما تكون خلايا RPE الناضجة مصطبغة بشدة ، مما قد يضعف إشارة الفلورسنت للتلطيخ المناعي للأجسام المضادة. عندما تكون إشارة مضان ضعيفة أو عندما يكون هناك الكثير من تلطيخ الخلفية ، لا يمكن ل LipidUNet تمييز قطرات الدهون بدقة. لسبب مماثل ، يجب استخدام إعدادات الاستحواذ المحددة بشكل صحيح لخطوة التصوير التلقائي للبروتوكول. إذا كانت الصور التي تم الحصول عليها ذات جودة رديئة ، فسوف تكافح LipidUNet لإخفاء الصور بشكل صحيح ، وبالتالي ، سيكون القياس الكمي غير دقيق (الشكل 6A-L). وأخيرا، تعد المعالجة اللاحقة للصور خطوة مهمة، حيث أن LipidUNet لديها متطلبات محددة للبرنامج للعمل.
عند مقارنتها بسير العمل لتحليل الدهون التي تستخدم العتبة اليدوية ، أو التقنيات التي تتضمن العتبة التلقائية في برامج مثل فيجي ، تقدم LipidUNet تقسيما غير متحيز وموثوقا عبر الصور ذات ترسب الدهون المتغير ، كما ينعكس في معدل خطأ صغير في تحديد جزيئات الدهون (الشكل 7). يسمح البرنامج للمستخدم بإدخال صور تدريب إضافية ، مما يسمح بتحليل مجموعات الصور بخلاف تلك التي تستخدم هدف تكبير 40x أو حتى تلك التي تستخدم علامة دهون مختلفة ، كما هو موضح في البروتوكول. في المستقبل ، سيتم تدريب البرنامج على تحليل صور 3D بحيث يكون القياس الكمي لحجم رواسب الدهون ممكنا. تنتشر أمراض العين التنكسية التي تنطوي على ترسب الدهون كمساهم رئيسي في علم الأمراض ، ومن المتوقع أن تزداد الحالات مع توسع عدد السكان المسنين13. ستسمح نماذج الأمراض الدقيقة وأدوات التحليل الفعالة ، كما أوضحنا في هذا البروتوكول ، بتطوير تدخلات علاجية جديدة.
Disclosures
لا إفصاحات.
Acknowledgements
نشكر مركز الأنسجة في المعهد الوطني للعيون (NEI) على استخدام نظام زايس متحد البؤر. تم دعم هذا العمل من قبل أموال NEI IRP (رقم المنحة ZIA EY000533-04).
Materials
| Name | Company | Catalog Number | Comments |
| 0.22 µm Steriflip filter system | EMD Millipore | SCGP00525 | |
| 1x Dulbecco's Phosphate Buffered Saline | Gibco | 14190-144 | |
| 3,3',5-Triiodo-L-thyronine | Sigma | T5516 | |
| Albumin Bovine, Fraction V | MP Biomedical | 160069 | |
| Alexa Fluor 555 rabbit anti-goat IgG (H+L) | Invitrogen | A21431 | APOE secondary antibody |
| APOE primary antibody | Millipore Sigma | AB947 | |
| BODIPY 493/503 | Invitrogen | D3922 | Protect from light |
| Complement competent human serum | Millipore Sigma | S1-LITER | |
| CTS N2 Supplement | Life Technologies | A13707-01 | |
| Fetal Bovine Serum | Hyclone | SH30071.03 | |
| Fluoromount-G | SouthernBiotech | 0100-01 | Slide mounting media |
| Glass Cover Slips #1 1/2 22 mm x 22 mm | Electron Microscopy Sciences | 72204-01 | |
| Glass Microscope Slide 25 mm x 75 mm- 1.2 mm Thick | Electron Microscopy Sciences | 71870-01 | |
| Hydrocortisone | Sigma | H0396 | |
| MEM Alpha | Life Technologies | 12571-063 | |
| MEM non-essential Amino Acids | Life Technologies | 11140 | |
| Nile Red | Sigma | 72485-100MG | Protect from light |
| Paraformaldehyde 16% Solution, EM Grade | Electron Microscopy Sciences | 15710 | |
| Penicillin-Strep | Life Technologies | 15140-148 | |
| Phosphate Buffered Saline 10x | Gibco | 70011-044 | |
| Rod Outer Segments (OS) | InVision Bioresources | 98740 | |
| Sodium bicarbonate | Sigma Aldrich | S5761 | |
| Sodium Pyruvate | Life Technologies | 11360-070 | |
| Sucrose | Sigma Aldrich | S1888 | |
| SYBR Green Master Mix | Bio-Rad | 1725274 | |
| Taurine | Sigma | T0625 | |
| Triton X-100 | Sigma | 9002-93-1 | |
| Tween 20 Ultrapure | Affymetrix | 9005-64-5 | |
| Vitronectin | Life Technologies | A14701SA | |
| Y-27632 dihydrochloride | R&D Systems | 1254 |
References
- Kanow, M. A., et al. Biochemical adaptations of the retina and retinal pigment epithelium support a metabolic ecosystem in the vertebrate eye. eLife. 6, e28899 (2017).
- Adijanto, J., et al. The retinal pigment epithelium utilizes fatty acids for ketogenesis. The Journal of Biological Chemistry. 289 (30), 20570-20582 (2014).
- Farnoodian, M., et al. Cell-autonomous lipid-handling defects in Stargardt iPSC-derived retinal pigment epithelium cells. Stem Cell Reports. 17 (11), 2438-2450 (2022).
- Curcio, C. A. Soft drusen in age-related macular degeneration: Biology and targeting via the oil spill strategies. Investigative Ophthalmology & Visual Science. 59 (4), AMD160-AMD181 (2018).
- Cankova, Z., Huang, J. D., Kruth, H. S., Johnson, M. Passage of low-density lipoproteins through Bruch's membrane and choroid. Experimental Eye Research. 93 (6), 947-955 (2011).
- Curcio, C. A., et al. Esterified and unesterified cholesterol in drusen and basal deposits of eyes with age-related maculopathy. Experimental Eye Research. 81 (6), 731-741 (2005).
- Miyagishima, K. J., et al. AMPK modulation ameliorates dominant disease phenotypes of CTRP5 variant in retinal degeneration. Communications Biology. 4 (1), 1360 (2021).
- Sharma, R., et al. Epithelial phenotype restoring drugs suppress macular degeneration phenotypes in an iPSC model. Nature Communications. 12 (1), 7293 (2021).
- Farnoodian, M., et al. Cell-autonomous lipid-handling defects in Stargardt iPSC-derived retinal pigment epithelium cells. Stem Cell Reports. 17 (11), 2438-2450 (2022).
- Hallam, D., et al. An induced pluripotent stem cell patient specific model of complement factor H (Y402H) polymorphism displays characteristic features of age-related macular degeneration and indicates a beneficial role for UV light exposure. Stem Cells (Dayton, Ohio). 35 (11), 2305-2320 (2017).
- Sharma, R., Bose, D., Montford, J., Ortolan, D., Bharti, K. Triphasic developmentally guided protocol to generate retinal pigment epithelium from induced pluripotent stem cells. STAR Protocols. 3 (3), 101582 (2022).
- Issa, P. C., Barnard, A. R., Herrmann, P., Washington, I., MacLaren, R. E. Rescue of the Stargardt phenotype in Abca4 knockout mice through inhibition of vitamin A dimerization. Proceedings of the National Academy of Sciences of the United States of America. 112 (27), 8415-8420 (2015).
- GBD 2019 Blindness and Vision Impairment Collaborators. Blindness and Vision Impairment Collaborators. Causes of blindness and vision impairment in 2020 and trends over 30 years, and prevalence of avoidable blindness in relation to VISION 2020: the Right to Sight: an analysis for the Global Burden of Disease Study. Lancet Global Health. 9 (2), e144-e160 (2021).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved