دراسة ديناميكا نسخ العضلات على مقاييس أحادية الجزيء في ذبابة الفاكهة
In This Article
Summary
ذبابة الفاكهة هي نموذج راسخ لدراسة الجزيئات الرئيسية التي تنظم تكوين العضلات. ومع ذلك ، فإن الطرق الحالية غير كافية لتحديد ديناميكيات نسخ mRNA والتوزيع المكاني داخل الخلايا المخلية. لمعالجة هذا القيد ، قمنا بتحسين طريقة تهجين مضان الحمض النووي الريبي في الموقع مما يسمح باكتشاف وقياس mRNAs على نطاق جزيء واحد.
Abstract
العضلات الهيكلية هي خلايا تخليقية كبيرة تتكون من العديد من الألياف العضلية المجمعة التي تنتج قوى وتمكن من حركة الجسم. ذبابة الفاكهة هي نموذج كلاسيكي لدراسة بيولوجيا العضلات. أدى الجمع بين كل من علم الوراثة ذبابة الفاكهة ونهج omics المتقدمة إلى تحديد الجزيئات المحفوظة الرئيسية التي تنظم تكوين العضلات وتجديدها. ومع ذلك ، لا يمكن تقييم ديناميكيات النسخ لهذه الجزيئات والتوزيع المكاني للحمض النووي الريبي الرسول داخل المخلوي بالطرق التقليدية. هنا قمنا بتحسين طريقة مضان الحمض النووي الريبي أحادي الجزيء في الموقع (smFISH) لتمكين اكتشاف وقياس جزيئات mRNA الفردية داخل عضلات الطيران البالغة وخلاياها الجذعية العضلية. كدليل على المفهوم ، قمنا بتحليل تعبير mRNA وتوزيع عاملين للنسخ المحفوظ التطوري ، Mef2 و Zfh1 / Zeb. لقد أوضحنا أن هذه الطريقة يمكنها اكتشاف جزيئات mRNA المفردة وتحديدها بكفاءة لكل من النسخ في الخلايا السليفة العضلية والعضلات البالغة والخلايا الجذعية العضلية.
Introduction
تتكون العضلات الهيكلية البالغة من ألياف عضلية متمايزة متعددة النوى تولد خواصها المقلصة حركات. يعتمد نمو العضلات وصيانتها وتجديدها على أسلاف العضلات والخلايا الجذعية العضلية (MuSCs) التي يتم تحديدها أثناء التطور الجنيني1. يتم التحكم في البرنامج العضلي بدقة من خلال مجموعة من عوامل النسخ العضلية الأساسية (TFs) (على سبيل المثال ، Pax3 / 7 و MYOD و Mef2 و ZEB) 2،3،4. يعد فك رموز الآليات الجزيئية التي تنظم بيولوجيا العضلات أمرا مهما لتوضيح الأسئلة الأساسية المتعلقة بعلم العضلات وللاستخدام العلاجي المحتمل في علاج الأمراض التنكسية العضلية.
ذبابة الفاكهة لها تاريخ طويل كنموذج وراثي لدراسة تكوين العضلات5. وقد ظهر مؤخرا كنموذج جديد للتحقيق في تجديد العضلات6،7،8. يتم الحفاظ على بنية العضلات والبرامج العضلية الأساسية بشكل كبير بين الذباب والثدييات. على سبيل المثال ، TFs Mef2 و Zfh1 / ZEB لها وظيفة محفوظة في تنظيم نمو العضلات وتجديدها3،9،10،11. تتشكل عضلات ذبابة الفاكهة البالغة ، مثل عضلات الطيران غير المباشرة (IFMs) ، من مجموعة محددة من MuSCs ، والمعروفة باسم أسلاف العضلات البالغة (AMPs) 12. يتم تحديد AMPs هذه أثناء التطور الجنيني وترتبط بالهياكل الظهارية مثل أقراص الجناح والساق. طوال المراحل الجنينية واليرقية ، تظل AMPs غير متمايزة حتى التحول عندما تنخرط في التمايز والاندماج لتشكيل IFMs13,14. يتم التعبير عن TF Spalt Major (spalt ، salm) أثناء تطوير عضلات الطيران وهو ضروري لتحديد هويتها الهيكلية15. Mef2 هو TF مهم آخر ضروري لتكوين العضلات البالغة16,17. يتم التعبير عنها في AMPs ويتم الحفاظ عليها في IFMsللبالغين 10,18. في حين أن غالبية AMPs تتمايز إلى عضلات وظيفية ، فإن مجموعة فرعية تفلت من التمايز وتشكل MuSCsالبالغة 11. على غرار الفقاريات ، فإن TF Zfh1 / ZEB مطلوب لمنع التمايز المبكر ل MuSCs البالغة والحفاظ على جذعها 9,10.
ثبت أن ديناميكيات التعبير الجيني تنظم العمليات البيولوجية المختلفة للعضلات 19,20. مكن ظهور تقنيات تسلسل الحمض النووي الريبي أحادية الخلية والنوى المفردة عالية الإنتاجية من استكشاف شامل لديناميكيات النسخ هذه21,22. أحد القيود الملحوظة لهذه الأساليب هو عدم قدرتها على توفير التوزيع المكاني لجزيئات mRNA في ألياف العضلات متعددة النوى. يمكن التحقق من هذه الميزات عن طريق التهجين الفلوري أحادي الجزيء في الموقع (smFISH) الذي يكشف عن نوعين من أجسام mRNA: 1. جزيئات mRNA الفردية المنتشرة في السيتوبلازم وتمثل الحمض النووي الريبي الناضج و 2. تتوافق بؤرتان نوويتان ساطعتان كحد أقصى مع النسخة الوليدة وتكشف عن الأليلات النشطة نسخيا23. لذلك ، فإن smFISH هي طريقة مفضلة لقياس جزيء mRNA الفردي ، والتحقيق في توزيعها المكاني وتقديم لقطات لديناميكيات النسخ الجيني.
تعتمد طريقة smFISH على مجموعة من مجسات قليل النوكليوتيد الفلورية القصيرة المصممة خصيصا لتكون مكملة للنسخة المستهدفة. عند التلدين ، فإنه يولد مصادر نقطية عالية الكثافة تسمح باكتشاف mRNA على مستوى جزيء واحد باستخدام المجهر متحد البؤر24. يتم تطبيق هذه الطريقة بشكل متزايد على مجموعة واسعة من أنواع الخلايا ، بما في ذلك أنسجة عضلات الثدييات19,20. ومع ذلك ، في ذبابة الفاكهة مثل النماذج الحيوانية الأخرى ، فإن معظم المعرفة حول التعبير الجيني للعضلات البالغة مستمدة من المقايسات الجزيئية السائبة ، والتي تفتقر إلى المعلومات الكمية حول الموقع المكاني لجزيئات mRNA. هنا قمنا بتحسين طريقة لأداء smFISH على خلايا سلائف العضلات وعضلات ذبابة الفاكهة البالغة23,25. يتضمن هذا البروتوكول خط أنابيب تحليل مؤتمت بالكامل لتجزئة النوى وعد وتوطين mRNA.
Protocol
1. قرص الجناح وتشريح العضلات البالغة وإعدادها
ملاحظة: قم بتنظيف مقعد التشريح وجميع أدوات التشريح بمحلول مثبط RNAse (جدول المواد). يجب ارتداء قفازات المختبر أثناء الإجراء التجريبي بأكمله.
- قرص الجناح
- ضع اليرقات المرحلية L3 في محلول ملحي بارد 1x مخزن بالفوسفات (PBS) (الشكل 1 أ).
- شطف اليرقات 3x مع 1x PBS والاحتفاظ بها في هذا الحل لمدة 5 دقائق.
- باستخدام زوجين من الملقط الحاد ، أمسك اليرقة من الجزء الأمامي واسحب بقية أنسجة الجسم وتخلص منها (حوالي 2/3).
- امسك كل من أول 1/3 من اليرقة بزوج واحد من الملقط وخطافات الفم مع الزوج الآخر. اسحب خطافات الفم داخل اليرقة حتى تنقلب بالكامل.
ملاحظة: تشريح ما يقرب من 30 يرقة لكل تجربة ، لضمان حجم عينة مناسب للتحليل. - لاحظ أن أقراص الجناح ترتبط ارتباطا وثيقا بالفرعين الأساسيين للقصبة الهوائية ، اللذين يجريان على طول كل جانب من اليرقة.
- قم بإزالة جميع مكونات اليرقات الأخرى (أقراص الساق والدماغ) للحصول على جثة يرقية مقلوبة نظيفة مع زوج من أقراص الجناح متصلة بالقصبة الهوائية (الشكل 1 ب).
- ضع العينات في أنبوب طرد مركزي سعة 1 مل وقم بتثبيتها في 4٪ بارافورمالدهايد (PFA) مخفف في 1x PBS لمدة 45 دقيقة في درجة حرارة الغرفة تحت التحريض.
ملاحظة: يتم تحقيق تقليب العينة بواسطة محرك متذبذب (جدول المواد). - اغسل العينات 3 × 5 دقائق بإيثانول 70٪ (EtOH ، مخفف في ماء خال من الحمض النووي الريبي) وقم بتخزينها في نفس محلول الغسيل عند 4 °C لمدة يومين على الأقل وحتى 1 أسبوع.
- عضلات الطيران غير المباشرة
- تخدير الذباب البالغ على وسادة ذبابة CO2 وإزالة الرؤوس والبطن والساقين والأجنحة.
- ضع الصدر المشريح في 1x PBS بارد واستمر في القيام بذلك حتى يتم تشريح جميع العينات (الشكل 1 د).
- بادئة الصدر في 4٪ PFA ، (1٪ Triton) لمدة 20 دقيقة في درجة حرارة الغرفة تحت التحريض.
- شطف العينات 3 × 5 دقائق مع 1x محلول الغسيل PBT (1٪ Triton في 1x PBS).
- ضع الصدر على شريط على الوجهين (جدول المواد) على شريحة زجاجية وقسمها بشفرة ميكروتوم حادة لإنتاج صدرين نصفيين (الشكل 1E ، F).
- ثبت العينات في 4٪ PFA (1٪ Triton) لمدة 45 دقيقة في درجة حرارة الغرفة تحت التحريض.
- اغسل 2 × 20 دقيقة بمحلول غسيل 1x PBT (1٪ Triton ، 1x PBS) في درجة حرارة الغرفة تحت التحريض.
- ضع الصدر النصفي في 1x PBS بارد واستخدم الملقط لعزل IFMs برفق عن نصف الصدر (الشكل 1G).
ملاحظة: لتحقيق ذلك ، من المهم إزالة بشرة بأكبر قدر ممكن من الدقة. - لتخلل العينات ، قم بتخزين IFMs المعزولة في محلول EtOH بنسبة 70٪ لمدة 2 أيام على الأقل وحتى أسبوع 1 عند 4 °C.
ملاحظة: عزل IFMs من حوالي 10 صدريات لكل تجربة لضمان حجم عينة مناسب للتحليل.
2. التهجين ، تلطيخ المناعة ، والتركيب
ملاحظة: يتم تطبيق إجراء تهجين مماثل لكل من أقراص الجناح وعينات IFMs. من الأهمية بمكان عزل أقراص الجناح عن جثث اليرقات ووضعها في 200 ميكرولتر من المخزن المؤقت A قبل بدء التهجين.
- أعد تعليق المجسات في 400 ميكرولتر من المخزن المؤقت TE (10 mM Tris-HCl ، 1 mM EDTA ، الرقم الهيدروجيني 8.0) للحصول على محلول مخزون نهائي يبلغ 12.5 ميكرومتر.
- نقل العينات إلى أنبوب طرد مركزي سعة 0.5 مل يحتوي على Buffer A (جدول المواد).
ملاحظة: من خلال تقليل حجم الحاوية ، يصبح من الأسهل تصور العينات وتجنب فقدانها أثناء عملية الغسيل. - اغسل العينات 2 × 20 دقيقة باستخدام Buffer A.
- نضح المخزن المؤقت A ، واستبدله ب 400 ميكرولتر من مخزن التهجين (جدول المواد) ، وقم بتهجين العينات مسبقا لمدة 30 دقيقة عند 37 درجة مئوية في خلاط حراري (جدول المواد).
- قم بتخفيف المجسات إلى تركيز نهائي يبلغ 125 نانومتر لأقراص الجناح و 200 نانومتر للعضلات البالغة ، في مخزن التهجين وأضف الجسم المضاد الأساسي.
ملاحظة: يختلف تخفيف الأجسام المضادة اعتمادا على الجسم المضاد المحدد المستخدم (جدول المواد). إضافة الأجسام المضادة اختياري. قد يختار المستخدمون التخلي عن تلطيخ البروتين وبدلا من ذلك يستخدمون بروتوكول smFISH مشابها لا يتضمن إضافة الأجسام المضادة. - نضح المخزن المؤقت للتهجين واستبدله ب 100 ميكرولتر من محلول التهجين الذي يحتوي على المسبار والجسم المضاد.
- احتضان العينات في الظلام عند 37 درجة مئوية لمدة 16 ساعة على الأقل تحت 300 دورة في الدقيقة في خلاط حراري.
- قم بتسخين حصصة من المخزن المؤقت أ عند 37 درجة مئوية.
- اغسل العينات لمدة 3 × 10 دقائق في الخلاط الحراري عند 37 درجة مئوية تحت التقليب 300 دورة في الدقيقة.
- أثناء الغسيل الأخير ، قم بتخفيف الجسم المضاد الثانوي (1/200) و DAPI (1/10000) في المخزن المؤقت A واحتضان العينات في هذا المحلول عند 37 درجة مئوية لمدة ساعة واحدة على الأقل.
- اغسل العينات لمدة 3 × 20 دقيقة باستخدام Buffer B (جدول المواد).
- انقل العينات بعناية على شريحة مجهرية ، وامسح المخزن المؤقت B المتبقي ، وأضف 30 ميكرولتر من وسيط التثبيت ، وقم بتغطيتها بغطاء 18 × 18 مم. استخدم طلاء الأظافر لإغلاق التحضير.
- قم بتخزين العينات لمدة تصل إلى 1 أسبوع عند 4 درجات مئوية.
ملاحظة: ومع ذلك ، يوصى بإجراء التصوير في أسرع وقت ممكن لتجنب تدهور إشارة المسبار.
3. التصوير
- التقط الصور باستخدام وضع اكتساب xyz باستخدام مجهر متحد البؤر مزود بأهداف غمر الزيت 40x و 63x (جدول المواد). يتم الكشف عن إشارة smFISH بواسطة كاشف HyD عبر وضع عد الفوتون.
- حدد موقع العينات باستخدام إشارة DAPI ومصباح الأشعة فوق البنفسجية.
- لتصوير أسلاف العضلات المرتبطة بقرص الجناح و IFMs (الشكل 2 والشكل 3) ، قم بتطبيق الإعداد التالي لخطوط الليزر ومرشحات الانبعاثات: حدد DAPI (الإثارة (على سبيل المثال) 405 نانومتر ، الانبعاث (em.) 450 نانومتر) ، Alexa Fluor 488 (على سبيل المثال 496 نانومتر ، em. 519 نانومتر) ، و Quasar 670 لمجسات smFISH (على سبيل المثال 647 نانومتر ، em. 670 نانومتر).
- لتصوير MuSCs البالغة (الشكل 2D) ، قم بتطبيق الإعداد التالي لخطوط الليزر ومرشحات الانبعاثات: حدد DAPI (على سبيل المثال 405 نانومتر ، em. 450 نانومتر) ، Alexa Fluor 488 (على سبيل المثال 496 نانومتر ، em. 519 نانومتر) ، أليكسا فلور 555 (على سبيل المثال 555 نانومتر ، em. 565 نانومتر) ، و Quasar 670 لتحقيقات smFISH (على سبيل المثال 647 نانومتر ، em. 670 نانومتر).
ملاحظة: بالنسبة للتصوير ، يمكن تكييف الأطوال الموجية وفقا للأصباغ المرتبطة بالأجسام المضادة والمجسات الثانوية. - احفظ الصور متحدة البؤر ك . ملفات TIFF.
4. تحليل ما بعد التصوير
- للبدء ، قم بتشغيل ImageJ وانتقل إلى قائمة المكونات الإضافية . من هناك ، حدد وحدات الماكرو | تحرير لفتح التعليمات البرمجية المصدر لوحدات الماكرو.
ملاحظة: سيمكن هذا من ضبط المعلمات حسب الحاجة ، وإذا لزم الأمر ، قم بتعديل دليل المجلد لكل قناة داخل التعليمات البرمجية المصدر للماكرو. - لاتباع هذا البروتوكول وتحديد بقع Mef2 smFISH في اليرقات ، استخدم الإعدادات التالية: Log_radius = 3.0 و Log_quality = 20.0. نوى موجبة Segement Mef2 مع طمس = 2 ؛ nucleus_scale_parameter = 30 ؛ nucleus_threshold = -8.0 ؛ nucleus_size = 300.
- في ألياف العضلات ، استخدم المعلمات التالية: Log_radius = 2.5 ؛ Log_quality = 60.0 ؛ طمس = 2 ؛ nucleus_scale_parameter = 100 ؛ nucleus_threshold = 0.0 ؛ nucleus_size = 300.
- لبدء الماكرو ، ما عليك سوى تنفيذ أمر التشغيل وانتظر حتى يقوم الماكرو بتحميل جميع الصور تلقائيا من المجلد المحدد وتحديدها بطريقة متسلسلة.
ملاحظة: يرجى ملاحظة أن مدة هذه العملية قد تختلف من بضع ثوان إلى عدة ساعات ، اعتمادا على حجم البيانات التي يتم تحليلها. - ابحث عن النتائج المعروضة في ملفين جديدين .csv: file_FISH_results و file_nuclei_results. يوضح الأول عدد بقع النسخ ومتوسط شدتها والحد الأقصى. والثاني يعطي كلا من إجمالي عدد النوى وعدد البقع داخل كل نواة.
النتائج
في هذا البروتوكول ، استخدمنا مجسات منتجة تجاريا تستهدف Mef2 و zfh1 mRNA. لقد صممنا المجسات مع مصمم مجسات FISH الخاص بالشركة المصنعة (جدول المواد). تستهدف مجموعة Mef2 الإكسونات المشتركة بين جميع أشكال الحمض النووي الريبي Mef2 (من exon 3 إلى exon 10). تستهدف مجموعة zfh1 الإكسون الثالث المشترك لكل من أشكال zfh1-RB و zfh1-RE 10. يتم اقتران كلا المجسين بصبغة الفلورسنت Quasar 670.
لقد أنشأنا ماكرو داخليا متوافقا مع برنامج ImageJ لتحليل بيانات .tif الأولية تلقائيا. يسمح الماكرو بتجزئة 3D للنوى بواسطة Cellpose26 باستخدام المكون الإضافي Fiji BIOP والقياس الكمي للبقعة النسخية بواسطة Laplacian of Gaussian كما هو مطبق في المكون الإضافي Trackmate27. يستخدم المكون الإضافي MorpholibJ28 للمعالجة اللاحقة (الملف التكميلي 1).
كخطوة أولى ، أجرينا smFISH على أقراص وهمية للجناح ، حيث من المعروف أن Mef2 و zfh1 يتم التعبير عنهما في AMPs. تظهر هذه البيانات أن كلا النصين تم اكتشافهما بشكل موحد في مجموعة AMPs الملطخة إما بالأجسام المضادة Mef2 أو Zfh1 (الشكل 2A ، B). يمكن الكشف عن مواقع النسخ النشطة (TS) وتمييزها عن mRNA الناضجة بواسطة smFISH لأنها تميل إلى أن تكون أكبر ولديها إشارات أكثر كثافة من النسخ السيتوبلازمية الفردية أو النسخ النووية الناضجة. باستمرار ، تميز التكبيرات الأعلى ل AMPs بين بؤر TS و mRNA الناضجة المنتشرة في السيتوبلازم (الشكل 2A '، B') ، مما يؤكد حساسية بروتوكول smFISH هذا. ومع ذلك ، تجدر الإشارة إلى أننا لاحظنا TS نشطا واحدا لكل نواة لكل من zfh1 و Mef2 (الشكل 2A '، B'). تؤكد هذه البيانات أيضا دقة تصميم المسبار نظرا لأن أنماط النسخ Mef2 و zfh1 في AMPs تتزامن مع اكتشاف البروتينات الخاصة بكل منها (الشكل 2).
في الخطوة الثانية ، قمنا بفحص موقع النسخ وتوزيع Mef2 و zfh1 mRNAs في IFMs البالغة المتمايزة والخلايا الجذعية المرتبطة بها (الشكل 2C ، D). توضح هذه البيانات بوضوح Mef2 TSs في نوى العضلات المخلوية وتوزيع Mef2 mRNA في جميع أنحاء السيتوبلازم (الشكل 2C ، C). يشير Zfh1 على وجه التحديد إلى عدد سكان MuSCs10,11. باستخدام بروتوكول smFISH هذا ، نجحنا في اكتشاف نسخ zfh1 في MuSCs المميزة بتعبير Zfh1-Gal4 > GFP. يوضح التكبير العالي اكتشاف كل من zfh1 TS و mRNAs السيتوبلازمية المفردة (الشكل 2D).
أخيرا ، استخدمنا ماكرو ImageJ المصمم داخليا لتحليل ديناميكيات النسخ Mef2 والتوزيع المكاني كميا. يمكن لخط الأنابيب الحسابي اكتشاف وتقسيم بقع Mef2 ونوى العضلات بكفاءة (الشكل 3A-C). ومع ذلك ، نظرا لمشكلات نسبة الإشارة إلى الضوضاء ، قد لا يتم اكتشاف بعض البقع في حالات معينة (الشكل 3A '، B'). استخدمنا هذه الطريقة الآلية للتحقق مما إذا كانت وفرة Mef2 mRNA تختلف بين خلايا سلائف العضلات (AMPs) و IFMs البالغة المتباينة. يظهر تحليلنا أن عدد Mef2 mRNA لكل نواة في IFMs البالغة و AMPs لا يختلف اختلافا كبيرا (الشكل 3D).
للحصول على نظرة ثاقبة للتوزيع المكاني لنصوص Mef2 في عضلات IFM البالغة ، قمنا بقياس جزء بقع السيتوبلازم مقابل Mef2 mRNA النووية (الشكل 3E ، F). باستخدام برنامجنا ، قمنا بحساب كل من العدد الإجمالي لبقع Mef2 mRNA وعدد بقع Mef2 mRNA المتداخلة مع تلطيخ النوى (Mef2). بطرح mRNA المرتبط بالنوى من إجمالي عدد mRNA يوفر عدد بقع mRNA السيتوبلازمية. تكشف النتائج التي توصلنا إليها أن ما يقرب من 92٪ من Mef2 mRNA موجود في السيتوبلازم ، بينما يرتبط 8٪ المتبقية بنوى العضلات ، كما هو موضح في الشكل 3E ، F.
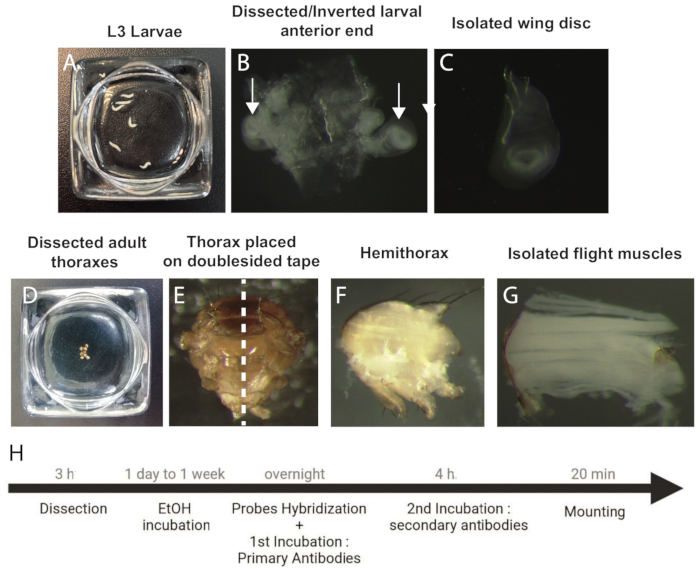
الشكل 1: إجراء تشريح وإعداد أقراص الجناح وعينات IFM ل smFISH. (أ) اليرقات المرحلية L3. ب: الطرف الأمامي لليرقة المقطعة والمقلوبة. تشير الأسهم إلى أقراص الجناح. ج: قرص الجناح المعزول؛ د: الصدر البالغ المقطع. (ه) الصدر البالغ موجه على شريط مزدوج الجوانب قبل التقسيم (خط منقط). (و) النزف الصدري للبالغين. (ز) IFMs المعزولة. (ح) تدفق عمل بروتوكول smFISH. الاختصارات: IFMs = عضلات الطيران غير المباشرة. smFISH = مضان الحمض النووي الريبي أحادي الجزيء في التهجين في الموقع ؛ EtOH = الإيثانول. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
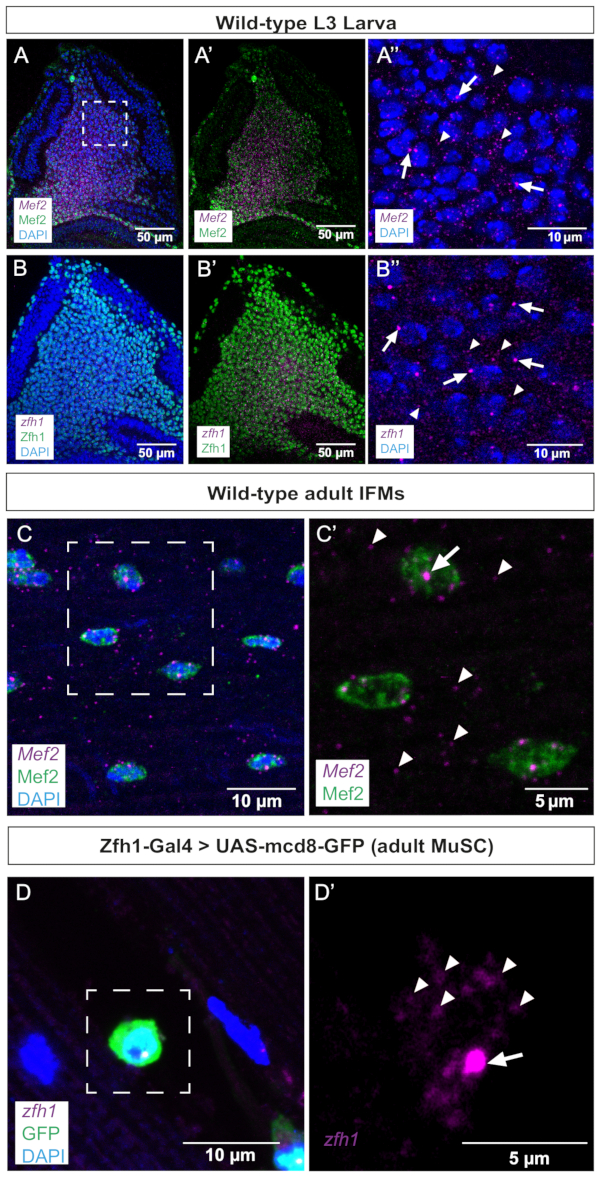
الشكل 2: صور تمثيلية ل Mef2 و zfh1 smFISH في أقراص الجناح و IFMs البالغة. (A ، B) تم الكشف عن Mef2 و zfh1 mRNAs (أرجواني) بشكل موحد في AMPs وتتمركز بشكل مشترك مع (A ، A') Mef2 و (B ، B') بروتينات Zfh1 (خضراء) ، على التوالي. (أ'',ب'') تكبير أعلى من AMPs. (ج) تم الكشف عن Mef2 mRNAs وبروتين Mef2 في IFMs البالغين. (ج') تكبير أعلى للمنطقة المحاصرة في C. (د) Zfh1-Gal4 >تعبير UAS-mCD8GFP (أخضر) يسمي MuSCs للبالغين. زفه1 تم الكشف عن الحمض النووي الريبي في MuSCs البالغين. (د') تكبير أعلى للمنطقة المحاصرة في D. يشار إلى مواقع بدء النسخ Zfh1 و mRNAs المفردة بالأسهم ورؤوس الأسهم ، على التوالي. دابي تلطيخ (الأزرق). أشرطة المقياس = 50 ميكرومتر (أ ، ب ، أ ، ب) ، 10 ميكرومتر (أ "، ب "، ج ، د) ، 5 ميكرومتر (ج ، د). الاختصارات: IFMs = عضلات الطيران غير المباشرة. smFISH = مضان الحمض النووي الريبي أحادي الجزيء في التهجين في الموقع ؛ AMPs = أسلاف العضلات البالغة. MuSCs = الخلايا الجذعية العضلية. DAPI = 4 '، 6-دياميدينو -2-فينيليندول ؛ GFP = بروتين الفلورسنت الأخضر. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
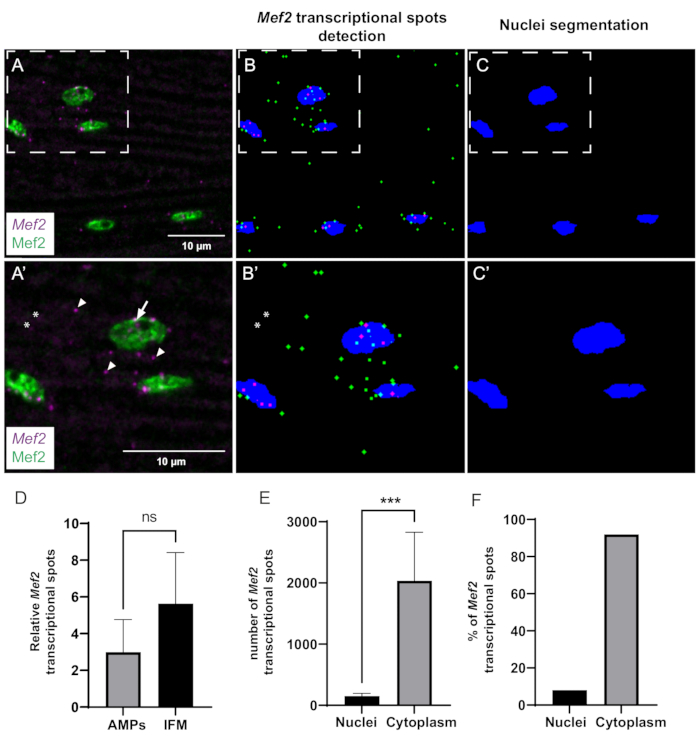
الشكل 3: القياس الكمي لنسخ Mef2 وتراكم mRNA السيتوبلازمي بواسطة smFISH. (أ) توزيع نسخ Mef2 (أرجواني) وبروتين Mef2 (أخضر) في IFMs البالغة من النوع البري. (أ') تكبير أعلى للمنطقة المحاصرة في A. يشار إلى مواقع بدء النسخ و mRNAs المفردة بالأسهم ورؤوس الأسهم ، على التوالي. (ب، ج) العثور التلقائي على بقع Mef2 والتجزئة النووية. يشار إلى السيتوبلازم والنووي Mef2 mRNAs باللون الأخضر والأرجواني ، على التوالي. (ب',ج') تكبير أعلى للمناطق المحاصرة في B و C. تشير العلامات النجمية إلى البقع التي لا يتم حسابها بواسطة الماكرو. (د) مثال على القياس الكمي الموضعي Mef2 في AMPs و IFMs البالغة ، بالنسبة إلى العدد الإجمالي للنوى. (p = 0,0785 ، اختبار t للطالب ، أقراص الجناح (n = 4) ، IFMs (n = 11)). (ه) مثال على التحديد الكمي للتوزيع الفوري Mef2 في IFMs البالغة. (*** ص< 0.0007 ، اختبار t للطالب ، ن = 5). (F) النسبة المئوية لبقع Mef2 mRNA المكتشفة داخل وخارج النوى (n = 5). قضبان المقياس = 10 ميكرومتر (A ، A'). الاختصارات: IFMs = عضلات الطيران غير المباشرة. smFISH = مضان الحمض النووي الريبي أحادي الجزيء في التهجين في الموقع ؛ AMPs = أسلاف العضلات البالغة. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
الملف التكميلي 1: الماكرو المستخدم للمعالجة اللاحقة. الرجاء الضغط هنا لتنزيل هذا الملف.
Discussion
اكتسب تطبيق طريقة smFISH شعبية في الآونة الأخيرة ، مع امتداد استخدامها إلى أنواع مختلفة من الخلايا والكائنات الحية النموذجية. ومع ذلك ، في حالة ذبابة الفاكهة ، فإن غالبية المعرفة حول التعبير الجيني للعضلات البالغة مستمدة من المقايسات الجزيئية السائبة ، والتي تفشل في توفير معلومات كمية عن الموقع المكاني الدقيق لجزيئات mRNA. لمعالجة هذه الفجوة ، قمنا بتحسين طريقة لأداء smFISH على خلايا سلائف العضلات وعضلات ذبابة الفاكهة البالغة. تم تكييف هذا النهج من البروتوكول23 المنشور سابقا وتم تحسينه لأنسجة عضلات ذبابة الفاكهة .
العقبة الرئيسية في الحصول على صور smFISH عالية الجودة هي سمك عضلات البالغين ، مما يعيق الاختراق الأمثل للمجسات. لذلك ، من الأهمية بمكان فصل العضلات البالغة عن الأنسجة الأخرى للحيوان وتنفيذ عملية التهجين بإثارة خفيفة. هذه الخطوة بالذات تضمن نفاذية فعالة للأنسجة.
جانب آخر مهم هو أن نسبة الإشارة إلى الضوضاء يمكن أن تتسبب في فشل خط الأنابيب الحسابي في اكتشاف بعض بقع mRNA. وجدنا أنه يمكن تحقيق زيادة نسبة الإشارة إلى الضوضاء من خلال إيجاد التركيز الأمثل للمجسات. قد يختلف التخفيف الأمثل اعتمادا على تكوين كل مجموعة مسبار ، بما في ذلك الصبغة المترافقة وتكوين قليل النوكليوتيد. نوصي بتجربة تخفيفات مختلفة. أسفر التخفيف النهائي البالغ 200 نانومتر عن أفضل نسبة إشارة إلى ضوضاء للأنسجة البالغة في هذه التجارب.
باستخدام طريقة smFISH ، من الممكن تحديد عدد الحمض النووي الريبي المركب حديثا في بؤر TS. عندما يتم نسخ الجين بنشاط ، يتم إنتاج العديد من الحمض النووي الريبي الوليد بشكل متزامن في TS. نتيجة لذلك ، ستتجاوز شدة TS كثافة الحمض النووي الريبي السيتوبلازمي الناضج ، وهذه الميزة ، جنبا إلى جنب مع توطينها النووي ، يمكن أن تميز TS عن الحمض النووي الريبي السيتوبلازمي الفردي. في سياق بيولوجيا العضلات ، يعد اكتشاف TS والقياس الكمي مهمين بشكل خاص لتحديد تزامن نسخ جين معين بين نوى العضلات داخل نفس المخلية. ومع ذلك ، لم يتم تصميم خط الأنابيب الحسابي هذا للتمييز بين إشارات mRNA السيتوبلازمية و TS. كبديل ، نقترح الجمع بين طريقة smFISH هذه وأدوات تحليل smFISH الراسخة الأخرى ، مثل BayFISH أو FISH-quant29,30. وقد ثبت أن هذه الأدوات تسهل التجزئة الآلية وحسابات شدة التألق لمجاميع الحمض النووي الريبي بدقة ملحوظة.
أخيرا ، بينما يكتشف smFISH جزيئات mRNA بدقة مكانية عالية ، فإنه يقتصر على تحليل عدد صغير من mRNA في نفس الوقت. تتيح الطرق متعددة المقاييس مثل merFISH (التألق متعدد الأخطاء القوي في الموقع التهجين) التحليل المتزامن لعدد كبير من mRNA31 المختلفة. من خلال الجمع بين مجسات قليل النوكليوتيد ورموز تصحيح الأخطاء ، تسهل هذه الطريقة اكتشاف مئات أنواع الحمض النووي الريبي في خلايا مفردة.
Disclosures
ليس لدى المؤلفين أي تضارب في المصالح للكشف عنه.
Acknowledgements
نشكر آلان فنسنت على قراءته النقدية للمخطوطة. نشكر روث ليمان على توفير الجسم المضاد Zfh1. نحن نعترف بمساعدة ستيفاني دوتيرتر وكزافييه بينسون للفحص المجهري متحد البؤر في منصة التصوير بالرنين المغناطيسي. يتم دعم EL من قبل زمالة الدكتوراه من وزارة التعليم العالي والبحث العلمي. يتم دعم NA من قبل AFM-Telethon Ph.D. زمالة 23846. يتم تمويل TP من خلال منحة مبادرة Chan Zuckerberg DAF إلى TP (2019-198009). يتم دعم HB من قبل CNRS ، وبرنامج Atip Avenir ، ومنحة الترامبولين AFM-Telethon 23108.
Materials
| Name | Company | Catalog Number | Comments |
| Confocal microscope SP8 | Leica | SP8 DMI 6000 | |
| DAPI | ThermoFisher | 62248 | |
| Double-sided tape | Tesa | 57910-00000 | |
| Dumont #55 Forceps | Fine Science Tools | 11255-20 | |
| Formaldehyde 16% | Euromedex | EM-15710 | |
| Mef2 antibody | DHSB | NA | |
| PBS 10x | Euromedex | ET330 | |
| RNaseZAP | Sigma-Aldrich | R2020 | |
| Stellaris probes | LGC | NA | |
| Stellaris RNA FISH Hybridization Buffer | LGC | SMF-HB1-10 | |
| Stellaris RNA FISH Wash Buffer A | LGC | SMF-WA1-60 | |
| Stellaris RNA FISH Wash Buffer B | LGC | SMF-WB1-20 | |
| Swann-Morton Blades | Fisher scientific | 0210 | |
| ThermoMixer | Eppendorf | 5382000015 | |
| Triton X-100 | Euromedex | 2000 | |
| UAS-mCD8::GFP line | Bloomington | RRID:BDSC_5137 | |
| Ultrapure water | Sigma-Aldrich | 95284 | |
| Watch Glass, Square, 1 5/8 in | Carolina | 742300 | |
| Zfh1 antibody | Gifted by Ruth Leahman | ||
| Zfh1-Gal4 | Bloomington | BDSC: 49924, FLYB: FBtp0059625 |
References
- Relaix, F., et al. Perspectives on skeletal muscle stem cells. Nature Communications. 12 (1), 692 (2021).
- Chal, J., Pourquie, O. Making muscle: skeletal myogenesis in vivo and in vitro. Development. 144 (12), 2104-2122 (2017).
- Taylor, M. V., Hughes, S. M. Mef2 and the skeletal muscle differentiation program. Seminars in Cell and Developmental Biology. 72, 33-44 (2017).
- Siles, L., et al. ZEB1 imposes a temporary stage-dependent inhibition of muscle gene expression and differentiation via CtBP-mediated transcriptional repression. Molecular and Cellular Biology. 33 (7), 1368-1382 (2013).
- Weitkunat, M., Schnorrer, F. A guide to study Drosophila muscle biology. Methods. 68 (1), 2-14 (2014).
- Boukhatmi, H. Drosophila, an integrative model to study the features of musclestem cells in development and regeneration. Cells. 10 (8), 2112 (2021).
- Bothe, I., Baylies, M. K. Drosophila myogenesis. Current Biology. 26 (17), R786-R791 (2016).
- Catalani, E., et al. RACK1 is evolutionary conserved in satellite stem cell activation and adult skeletal muscle regeneration. Cell Death Discovery. 8 (1), 459 (2022).
- Siles, L., Ninfali, C., Cortes, M., Darling, D. S., Postigo, A. ZEB1 protects skeletal muscle from damage and is required for its regeneration. Nature Communications. 10 (1), 1364 (2019).
- Boukhatmi, H., Bray, S. A population of adult satellite-like cells in Drosophila is maintained through a switch in RNA-isoforms. Elife. 7, e35954 (2018).
- Chaturvedi, D., Reichert, H., Gunage, R. D., VijayRaghavan, K. Identification and functional characterization of muscle satellite cells in Drosophila. Elife. 6, e30107 (2017).
- Gunage, R. D., Reichert, H., VijayRaghavan, K. Identification of a new stem cell population that generates Drosophila flight muscles. Elife. 3, e03126 (2014).
- Figeac, N., Daczewska, M., Marcelle, C., Jagla, K. Muscle stem cells and model systems for their investigation. Developmental Dynamics. 236 (12), 3332-3342 (2007).
- Aradhya, R., Zmojdzian, M., Da Ponte, J. P., Jagla, K. Muscle niche-driven insulin-Notch-Myc cascade reactivates dormant adult muscle precursors in Drosophila. Elife. 4, e08497 (2015).
- Schonbauer, C., et al. Spalt mediates an evolutionarily conserved switch to fibrillar muscle fate in insects. Nature. 479 (7373), 406-409 (2011).
- Bryantsev, A. L., Baker, P. W., Lovato, T. L., Jaramillo, M. S., Cripps, R. M. Differential requirements for Myocyte Enhancer Factor-2 during adult myogenesis in Drosophila. Developmental Biology. 361 (2), 191-207 (2012).
- Soler, C., Han, J., Taylor, M. V. The conserved transcription factor Mef2 has multiple roles in adult Drosophila musculature formation. Development. 139 (7), 1270-1275 (2012).
- Soler, C., Taylor, M. V. The Him gene inhibits the development of Drosophila flight muscles during metamorphosis. Mechanisms of Development. 126 (7), 595-603 (2009).
- Pinheiro, H., et al. mRNA distribution in skeletal muscle is associated with mRNA size. Journal of Cell Science. 134 (14), jcs256388 (2021).
- Denes, L. T., Kelley, C. P., Wang, E. T. Microtubule-based transport is essential to distribute RNA and nascent protein in skeletal muscle. Nature Communications. 12 (1), 6079 (2021).
- Li, H., et al. Fly Cell Atlas: A single-nucleus transcriptomic atlas of the adult fruit fly. Science. 375 (6584), eabk2432 (2022).
- Santos, M. D., et al. Extraction and sequencing of single nuclei from murine skeletal muscles. STAR Protocols. 2 (3), 100694 (2021).
- Raj, A., van den Bogaard, P., Rifkin, S. A., van Oudenaarden, A., Tyagi, S. Imaging individual mRNA molecules using multiple singly labeled probes. Nature Methods. 5 (10), 877-879 (2008).
- Femino, A. M., Fay, F. S., Fogarty, K., Singer, R. H. Visualization of single RNA transcripts in situ. Science. 280 (5363), 585-590 (1998).
- Raj, A., Tyagi, S. Detection of individual endogenous RNA transcripts in situ using multiple singly labeled probes. Methods in Enzymology. 472, 365-386 (2010).
- Stringer, C., Wang, T., Michaelos, M., Pachitariu, M. Cellpose: a generalist algorithm for cellular segmentation. Nature Methods. 18 (1), 100-106 (2021).
- Tinevez, J. Y., et al. TrackMate: An open and extensible platform for single-particle tracking. Methods. 115, 80-90 (2017).
- Legland, D., Arganda-Carreras, I., Andrey, P. MorphoLibJ: integrated library and plugins for mathematical morphology with ImageJ. Bioinformatics. 32 (22), 3532-3534 (2016).
- Mueller, F., et al. FISH-quant: automatic counting of transcripts in 3D FISH images. Nature Methods. 10 (4), 277-278 (2013).
- Gomez-Schiavon, M., Chen, L. F., West, A. E., Buchler, N. E. BayFish: Bayesian inference of transcription dynamics from population snapshots of single-molecule RNA FISH in single cells. Genome Biology. 18 (1), 164 (2017).
- Chen, K. H., Boettiger, A. N., Moffitt, J. R., Wang, S., Zhuang, X. RNA imaging. Spatially resolved, highly multiplexed RNA profiling in single cells. Science. 348 (6233), aaa6090 (2015).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved