Method Article
PyDesigner v1.0: تنفيذ Pythonic لخط أنابيب DESIGNER للتصوير بالرنين المغناطيسي بالانتشار
* These authors contributed equally
In This Article
Summary
نقدم هنا PyDesigner ، وهو خط أنابيب معالجة للتصوير بالرنين المغناطيسي المنتشر (dMRI) المستند إلى Python قادر على تصحيح القطع الأثرية النموذجية للرنين المغناطيسي الديناميكي (dti) وإنتاج تصوير موتر الانتشار (DTI) ، وتصوير التفرط المنتشر (DKI) ، وتصوير الكرة الليفية (FBI) ، ونمذجة البنية المجهرية (سلامة المادة البيضاء [WMTI] والمادة البيضاء الكروية الليفية [FBWM]) ، ومخرجات الtractography.
Abstract
PyDesigner عبارة عن حزمة برامج تستند إلى Python تعتمد على معلمة Diffusion الأصلية EStImation مع خط أنابيب Gibbs and NoisE Removal (DESIGNER) (Dv1) للمعالجة المسبقة للرنين المغناطيسي الرقمي المغناطيسي وتقدير الموتر. يتم توفير هذا البرنامج بشكل مفتوح للأبحاث غير التجارية ولا يجوز استخدامه للرعاية السريرية. يجمع PyDesigner بين أدوات من FSL و MRtrix3 لإجراء إزالة الضوضاء ، وتصحيح رنين جيبس ، وتصحيح حركة التيار الدوامي ، وإخفاء الدماغ ، وتنعيم الصورة ، وتصحيح انحياز Rician لتحسين تقدير مقاييس الانتشار المتعددة. يمكن استخدامه عبر الأنظمة الأساسية على أنظمة التشغيل Windows و Mac و Linux لاشتقاق المقاييس شائعة الاستخدام بدقة من مجموعات بيانات DKI و DTI و WMTI و FBI و FBWM بالإضافة إلى ملفات ODFs و .fib للجراعة. كما أنه محايد لتنسيق الملف ، حيث يقبل المدخلات في شكل تنسيق .nii و .nii.gz و .mif و dicom. سهل الاستخدام وسهل التثبيت ، يقوم هذا البرنامج أيضا بإخراج مقاييس مراقبة الجودة التي توضح الرسوم البيانية لنسبة الإشارة إلى الضوضاء ، والفوكسيل الخارج ، وحركة الرأس لتقييم سلامة البيانات. بالإضافة إلى ذلك ، يدعم خط أنابيب معالجة dMRI معالجة مجموعة بيانات متعددة لوقت الصدى ويتميز بتخصيص خط الأنابيب ، مما يسمح للمستخدم بتحديد العمليات المستخدمة والمخرجات التي يتم إنتاجها لتلبية مجموعة متنوعة من احتياجات المستخدم.
Introduction
يتم تطبيق التصوير بالرنين المغناطيسي للانتشار (dMRI) على نطاق واسع للدراسة غير الغازية لخصائص البنية المجهرية في الدماغ. بينما تم اقتراح العديد من طرق التصوير بالرنين المغناطيسي المنخفض ، فإن طريقتين شائعتين الاستخدام هما التصوير الموتر المنتشر (DTI) وتصوير التفرط المنتشر (DKI). ترتبط هذه التقنيات ارتباطا وثيقا ، حيث يعد DKI امتدادا ل DTI الذي يتضمن القياس الكمي لعدم الانتشارالغوسي 1. يوفر كلاهما مجموعة متنوعة من تدابير الانتشار العددي ويمكنان من بناء تخطيط الجر من ألياف المادة البيضاء. من المزايا المهمة ل DTI و DKI أن لديهما أساسا متينا في فيزياء الانتشار بحيث لا تعتمد صلاحيتهما على افتراضات مفصلة فيما يتعلق بالبنية المجهرية للأنسجة2،3. يسمح ذلك بتطبيق DTI و DKI في جميع أنحاء الدماغ والجسم للمشاركين من أي عمر وحالة مرضية.
نظرا لأن الصور الأولية المرجحة بالانتشار (DWIs) تتحلل بسبب عوامل متعددة ، بما في ذلك ضوضاء الإشارة والحركة ورنين جيبس وتشويه التيار الدوامي ، يجب استخدام المعالجة المسبقة قبل حساب أي كميات انتشار4. المعالجة المسبقة ل DWIs هي مجال نشط للبحث تم تطويره الآن بشكل كبير. ومع ذلك، فإن الجمع بين خطوات المعالجة الضرورية في مسار واحد يعطي نتائج متسقة يمثل تحديا لأنه يجب تعديل العديد من الإعدادات المحددة من قبل المستخدم اعتمادا على تفاصيل اكتساب dMRI ولأن الترتيب الذي يتم به تنفيذ خطوات المعالجة المسبقة يؤثر على النتيجة. لهذا السبب ، تم اقتراح خط أنابيب معلمة الانتشار EStImation مع إزالة Gibbs و NoisE (DESIGNER ، GitHub: NYU-DiffusionMRI / DESIGNER) في البداية في عام 2016 لتحسين وتوحيد وتبسيط المعالجة المسبقة ل DWIs5. يعتمد DESIGNER Dv1 على أدوات البرامج المضمنة في FSL و MRtrix3 و MATLAB و Python لإنشاء عملية DWI سلسة وكاملة - عملية تشمل تصحيح الصورة من خلال المعالجة المسبقة وتقدير توتر الانتشار / التفرط5. باستخدام علامات التحكم لتبديل خطوات المعالجة المسبقة أو إيقاف تشغيلها ، يمكن إجراء تصحيحات DWI بشكل انتقائي. العمليات المسبقة ل DESIGNER بترتيب معين - (ط) تحليل المكون الرئيسي Marchenko-Pastur (MP-PCA) تقليل الضوضاء6 ، (ii) تصحيح رنين Gibbs7 ، (iii) تصحيح تشويه التصوير المستوي للصدى (EPI)8 ، تصحيح التيار الدوامي9 ، تصحيح الحركة10 ، والاستبدال الخارجي11 ، (رابعا) تصحيح مجال التحيز B1 ، (تثم) توليد قناع الدماغ ، (سادسا) التنعيم ، (سابع) تصحيح انحياز ضوضاء Rician ، و (viii) تطبيع b0. تعمل المعالجة المسبقة بهذا الترتيب المحدد على تحسين كل من الدقة ونسبة الإشارة إلى الضوضاء الفعالة (SNR)5. تجدر الإشارة إلى أن كل خطوة من خطوات PyDesigner اختيارية ويمكن استخدامها أو تخطيها بناء على تفضيلات المستخدم. قد لا يكون التنعيم ، على سبيل المثال ، خطوة معالجة مسبقة ضرورية لبعض مجموعات البيانات. على الرغم من أنه يساعد في التخفيف من أخطاء التصفية ، فقد لا تكون هناك حاجة إليه لمجموعات البيانات فائقة الجودة. على هذا النحو ، يمكن للمستخدمين اختيار استخدام الخطوات اللازمة لبياناتهم فقط.
يعد تنفيذ DESIGNER عبر الأنظمة الأساسية أمرا صعبا بسبب الاختلافات في أنظمة التشغيل وإعدادات البيئة. على وجه الخصوص ، حقيقة أن DESIGNER Dv1 مكتوب بشكل أساسي في MATLAB يخلق مشكلات كبيرة في قابلية النقل تنشأ عن متطلبات التكوين المعقدة اللازمة لتمكين واجهة Python-MATLAB. علاوة على ذلك ، فإن المجموعات المختلفة من إصدارات MATLAB و Python والتبعية تضر بقابلية تكرار خط الأنابيب هذا. لهذا السبب ، قمنا بتطوير PyDesigner ، وهو قائم بالكامل على Python. لا يسمح خط أنابيب معالجة dMRI هذا بالمعالجة المسبقة السلسة فحسب ، بل يسمح أيضا بدمج خط الأنابيب في حاوية Docker ، مما يعزز بشكل كبير قابلية النقل والتكرار. بالإضافة إلى ذلك ، من خلال استبدال كود MATLAB ، يتجنب PyDesigner جميع رسوم الترخيص ويحسن إمكانية الوصول.
يعمل خط أنابيب معالجة dMRI هذا على زيادة نهج التحدث الحر الذي قدمته DESIGNER ، ويضيف العديد من الميزات الجديدة ، ويتضمن أدوات من FSL و MRtrix3 لإجراء المعالجة المسبقة. تم استخدام مكتبات Python الرياضية القياسية مثل Numpy12 و SciPy13 و CVXPY14،15 لاستبدال أجزاء MATLAB من DESIGNER بكود Python. يتم توفير هذا البرنامج بشكل مفتوح ومتاح على Github16. الهدف من هذه الورقة ليس التحقق من صحة برنامجنا أو مقارنته بالبرامج المماثلة ، بل إعطاء المستخدمين دليلا تفصيليا لمعالجة بياناتهم باستخدام PyDesigner إذا اختاروا القيام بذلك.
Protocol
تم جمع جميع البيانات المستخدمة لتطوير البرنامج واختباره بموجب بروتوكول دراسة معتمد من مجلس المراجعة المؤسسية.
ملاحظة: تم إنشاء PyDesigner على نظام التشغيل MacOS 10.14 ويتطلب نظام Windows الفرعي لنظام التشغيل Linux (WSL) للتشغيل على Windows. يمكن استخدام نفس الأوامر لأنظمة Linux / Mac.
1. توافق نظام التشغيل
- بالنسبة لأنظمة Windows ، اتبع الارتباط لتثبيت WSL17 ثم انتقل إلى الخطوة 2.6.
- بالنسبة للأنظمة المستندة إلى Unix (مثل Linux و Mac) ، انتقل إلى القسم 2.
ملاحظة: يمكن للأنظمة المستندة إلى Unix تثبيت PyDesigner وتبعياته في الأصل.
2. تثبيت FSL
ملاحظة: تم إجراء جميع اختبارات PyDesigner باستخدام الإصدار 6.0.2 من FSL، ويوصى باستخدام الإصدار 6.0.2 أو أعلى.
- انتقل إلى FSL wiki18 وحدد تنزيل/تثبيت. بالنسبة لمستخدمي Windows، انتقل إلى الخطوة 2.6.
- أختر تنزيل FSL ضمن عنوان تثبيت FSL. ثم حدد FSL Install Software License ، واقرأ الاتفاقية ، وحدد موافق.
- املأ الحقول المطلوبة وحدد تسجيل.
- اختر رابط تنزيل FSL (الرابط الأول على الصفحة). ثم حدد رابط تعليمات التثبيت (الرابط الثاني في الصفحة). اتبع هذه التعليمات لتنزيل FSL لكل نظام تشغيل.
ملاحظة: يستغرق تثبيت FSL حوالي 10 دقائق. - للتحقق من التثبيت الناجح، أدخل ما يلي في نافذة طرفية جديدة:
$ flirt -version. يجب أن يؤدي هذا إلى إرجاع رقم الإصدار. انتقل إلى الخطوة 3. - بالنسبة لمستخدمي Windows، اتبع دليل تثبيت WSL ل FSL19.
3. إنشاء بيئة Conda
- قم بتثبيت حزمة conda لإدارة بيئات Python. يمكن للمستخدمين اختيار إما Miniconda أو Anaconda.
- قم بتنزيل Miniconda / Anaconda20. ينتقل مستخدمو Mac و Linux إلى الخطوة 3.2.
- بالنسبة لنظام التشغيل Windows، قم بإعداد conda داخل WSL21.
ملاحظة: الإرشادات التالية خاصة ب Miniconda ، لكن نفس الأوامر تعمل مع Anaconda. على الرغم من أن conda لم يعد يدعم Python 3.7 ، إلا أنه لا يزال من الممكن إنشاء بيئة conda باستخدام Python 3.7 ، مما يسمح بتنفيذ هذا البروتوكول بدقة كما هو موضح.
- أدخل ما يلي في المحطة الطرفية لإنشاء بيئة conda:
$ conda create -n mri python=3.7. استخدم بيئة conda هذه لجميع الأنشطة المتعلقة ب PyDesigner.- عند مطالبتك باستخدام
Proceed ([y]/n)?Entery.
ملاحظة: -n العلامة: أستخدم هذا للإشارة إلى اسم بيئة conda. في الفيديو ، تسمى بيئة conda بالرنين المغناطيسي.
ملاحظة:python=يوصى بالإصدار 3.7 ، حيث تم إنشاء PyDesigner واختباره على هذا الإصدار.
- عند مطالبتك باستخدام
4. قم بتثبيت MRtrix3
- قم بتنشيط بيئة conda عن طريق إدخال الأمر التالي:
$ conda activate mri - افتح صفحة التنزيل الخاصة ب MRtrix322 واتبع تعليمات التنزيل لنظام التشغيل المناسب. سيطلب رمز التثبيت تأكيد بعض التبعيات. أدخل
yلمتابعة عملية التنزيل.- اطبع وثائق MRtrix3 لتأكيد التثبيت عن طريق إدخال ما يلي في الجهاز الطرفي:
$ mrinfo -h
- اطبع وثائق MRtrix3 لتأكيد التثبيت عن طريق إدخال ما يلي في الجهاز الطرفي:
5. تثبيت PyDesigner
- افتح نافذة طرفية جديدة وقم بتنشيط بيئة conda عن طريق إدخال ما يلي في المحطة الطرفية:
$ conda activate mri - قم بتشغيل الأمر:
$ pip install PyDesigner-DWI - للتحقق من التثبيت الناجح، اطبع رقم الإصدار عن طريق إدخال ما يلي في الجهاز الطرفي:
$ pydesigner -v- إذا ظهر الخطأ "لم يتم العثور على الوحدة النمطية"، فيرجى الاطلاع على قسم استكشاف الأخطاء وإصلاحها في نهاية البروتوكول (القسم 10)
ملاحظة: سيقوم $ pydesigner -h بطباعة وثائق التعليمات التي تشرح الأوامر وبناء الجملة للبرنامج. يمكن أيضا تنزيل PyDesigner عبر Github. انظر تعليمات التثبيت على الموقع23.
- إذا ظهر الخطأ "لم يتم العثور على الوحدة النمطية"، فيرجى الاطلاع على قسم استكشاف الأخطاء وإصلاحها في نهاية البروتوكول (القسم 10)
6. إعداد البيانات
ملاحظة: يعمل PyDesigner مع أنواع ملفات متعددة مثل .nii و .nii.gz و .mif و DICOM. في الفيديو ، يتم فرز DICOMs وتحويلها إلى NIfTI قبل استخدام مسار معالجة dMRI هذا. من السهل التعامل مع ملفات NIfTI لأنها غير محددة الهوية ، وهناك ملف واحد لكل تسلسل.
- لفرز DICOMs ، استخدم أداة DicomSort24.
ملاحظة: يسمح بأي أداة فرز DICOM أخرى متاحة تنجز نفس المهمة. - بعد فرز DICOMs ، قم بتحويلها إلى ملفات NIfTI باستخدام dcm2niix25.
- قم بتثبيت dcm2niix عن طريق إدخال الأمر التالي في المحطة الطرفية:
$ sudo apt installdcm2niix - بمجرد التنزيل ، اكتب ما يلي في الجهاز للتحقق من التثبيت عن طريق طباعة وثائق dcm2nii:
$ dcm2niix -h
- قم بتثبيت dcm2niix عن طريق إدخال الأمر التالي في المحطة الطرفية:
- لتشغيل dcm2niix ، أدخل الأمر التالي في المحطة الطرفية:
$ dcm2niix -f %s_%p_%d -o pathtosavefolder/ pathtorawdicoms/
ملاحظة: -f: لتحديد بنية اسم الملف لملف الإخراج ، اتبع هذه العلامة بأي بنية اسم ملف مطلوبة. يستخدم الفيديو: -f ٪s_٪p_٪d . ٪s و٪p و٪d هي علامات للسلسلة ورقم البروتوكول والوصف، على التوالي. ستعمل أي بنية ملف. المخرجات المتوقعة من dcm2niix هي ملفات .bval و .bvec و .json و .nii. إذا تم الحصول على بيانات TOPUP ، فستكون هناك ملفات .json و .nii TOPUP.
7. استخدام PyDesigner الأساسي
ملاحظة: يمكن أن يستغرق PyDesigner ما يصل إلى 1.5 ساعة لمجموعة بيانات واحدة اعتمادا على معلمات المعالجة وإمكانيات الكمبيوتر.
- لتشغيل المعالجة القياسية، استخدم العلامة
-sأو-standard. سيقوم هذا الأمر بتشغيل مسار المعالجة المسبقة الموصى به بالترتيب التالي: تقليل الضوضاء6 ، وتصحيح رنين جيبس7 ، وتصحيح تشويه EPI8 ، وتصحيح حركة التيار الدوامي9 ، وإنشاء أقنعة الدماغ والسائل النخاعي (CSF) ، والتنعيم ، وتصحيح التحيز Rician ، والكشف عن القيمالمتطرفة 11 ، وتقدير الموتر ، وإنشاء خرائط بارامترية. - لتشغيل الأمر PyDesigner الأساسي ، قم أولا بتنشيط بيئة conda عن طريق إدخال الأمر التالي في المحطة الطرفية:
$ conda activate mri - اطبع وثائق التعليمات الخاصة بالبرنامج عن طريق إدخال الأمر التالي:
$ pydesigner -h. تشرح هذه الوثائق جميع الأوامر وبناء الجملة الممكنة ل PyDesigner. - استخدم المثال التالي لهيكلة الأمر.
$ pydesigner -s --verbose --rpe_pairs 1 -o ./output_folder/ ./input_folder/input1.nii ./input_folder/input2.nii - لاحظ العلامات المهمة التالية:
--verboseاستخدم هذا لعرض وحدة تحكم PyDesigner أثناء معالجة البيانات (غير مطلوب.):--rpe pairs #استخدم هذا للسماح للمستخدمين الذين لديهم تسلسل TOPUP بتسريع عملية تصحيح تشوهEPI 8 باستخدام TOPUP.: يشير # إلى عدد وحدات تخزين B0 من تسلسل TOPUP التي سيتم استخدامها. يؤدي استخدام #=1 إلى منع المبالغة في تقدير حقل التشويه ويقلل من الوقت اللازم لإنشاء هذا الحقل. استخدم المستخدمون بشكل روتيني #=1 بنتائج موثوقة. عند استخدام #=1 ، سيقوم PyDesigner افتراضيا بأخذ المجلد الأول.
8. استخدام PyDesigner المتقدم: تخصيص البرنامج وفقا للاحتياجات الفردية للمستخدمين
ملاحظة: عند تخصيص PyDesigner، احذف العلم -s ؛ هذه العلامة هي أمر المعالجة المسبقة التلقائية.
- قم بتنشيط بيئة conda عن طريق إدخال الأمر التالي في المحطة الطرفية:
$ conda activate mri - استشر موقع البرنامج23 للحصول على قائمة بالأعلام. تسمح كل علامة للمستخدم بتشغيل كل جزء من PyDesigner على حدة.
- هيكلة الأمر. ابدأ بعلامات كل تسلسل يرغب المستخدم في تنفيذه. بعد ذلك ، قم بتوجيه PyDesigner إلى ملفات الإدخال والإخراج. على سبيل المثال:
$ pydesigner flag1 flag2 flag3 -o ./output_folder/ ./input_folder/input1.nii - مثال 1: تشغيل تركيب الموتر بمفرده (بدون معالجة مسبقة إضافية).
- قم بتنشيط بيئة conda عن طريق إدخال الأمر التالي في المحطة الطرفية:
$ conda activate mri - راجع موقع PyDesigner23 للحصول على قائمة بالأعلام. يستخدم هذا المثال
-m. -mأضف هذه العلامة للحد من ملاءمة الموتر داخل فوكسل الدماغ فقط.: هذا يسرع تركيب الموتر + الكشف عن القيم المتطرفة.- استخدم المثال التالي لهيكلة الأمر:
$ pydesigner -m -o ./output_folder/ ./input_folder/input1.nii
ملاحظة: إدخال تركيب الموتر هو ملف .nii معالج مسبقا.
- قم بتنشيط بيئة conda عن طريق إدخال الأمر التالي في المحطة الطرفية:
- مثال 2 تشغيل تقليل الضوضاء بمفرده (بدون معالجة مسبقة إضافية).
- قم بتنشيط بيئة conda عن طريق إدخال ما يلي في المحطة الطرفية:
$ conda activate mri - راجع موقع PyDesigner23 للحصول على قائمة بالأعلام. يستخدم هذا المثال
-n. - -n: أضف هذه العلامة لإكمال خطوة تقليل الضوضاء. أدخل ملف DWI الذي تمت معالجته مسبقا المسمى DWI_preproccessed.nii ، والذي يوجد في مجلد إخراج كل موضوع.
- استخدم المثال التالي لهيكلة الأمر:
$ pydesigner -n -o ./output_folder/ ./input_folder/input1.nii
- قم بتنشيط بيئة conda عن طريق إدخال ما يلي في المحطة الطرفية:
9. تحديثات PyDesigner المستقبلية
ملاحظة: يمكن العثور على التحديثات على الموقع23.
- لتحديث PyDesigner، أدخل ما يلي في نافذة طرفية جديدة:
$ pip install --upgrade PyDesigner-DWI
ملاحظة: إذا تم تثبيت PyDesigner من خلال Github16، فيجب أيضا تحديثه باستخدام git.
10. استكشاف أخطاء التثبيت وإصلاحها
- عند تنزيل PyDesigner، سيظهر رمز الخطأ "لم يتم العثور على الوحدة النمطية" إذا كانت هناك وحدة Python مفقودة.
- قم بإصلاح وحدة نمطية مفقودة عن طريق إدخال ما يلي في الوحدة الطرفية:
$ pip install nameofmodule - ثم كرر أمر التثبيت الأول عن طريق إدخال ما يلي في المحطة الطرفية:
$ pip install PyDesigner-DWI - يجب الآن تثبيت PyDesigner بالكامل. يرجى الرجوع إلى الخطوة 5.3. في حالة حدوث نفس الخطأ، كرر هذه العملية حتى يتم تثبيت جميع وحدات Python النمطية المفقودة.
- قم بإصلاح وحدة نمطية مفقودة عن طريق إدخال ما يلي في الوحدة الطرفية:
11. استكشاف أخطاء الإخراج وإصلاحها
- في حالة حدوث أي مشاكل، راجع ملف log_command.json لتحديد أي أخطاء في المعالجة المسبقة. راجع الملفات الوسيطة لتحديد الخطأ المحدد.
- للمساعدة في استكشاف الأخطاء وإصلاحها، أرسل طلبا على صفحة GitHub16.
12. تشغيل PyDesigner مع مثال على مجموعة البيانات
- قم بتنزيل مجموعة بيانات المثال من OpenNeuro هنا26.
ملاحظة: تحتوي مجموعة البيانات النموذجية هذه على كل من البيانات الأولية والمعالجة مسبقا. الغرض من البيانات الأولية هو الإدخال لمعالجة مثال PyDesigner. تتكون البيانات المعالجة مسبقا من المخرجات التي تم إنشاؤها بعد معالجة البيانات الأولية باستخدام مسار معالجة dMRI على iMac OS v12.4. يمكن للمستخدمين تنزيل البيانات الأولية ومعالجة هذه البيانات باستخدام PyDesigner ومقارنة مخرجاتهم بالمخرجات النموذجية المقدمة للتأكد من تشغيل PyDesigner بشكل صحيح. يتم تضمين البيانات الأولية في المجلد الرئيسي (/PyDesigner Example Dataset/sub-01) ويتم تضمين البيانات المعالجة مسبقا في مجلد المشتقات (/PyDesigner Example Dataset/derivatives/sub-01). ما لم ينص على خلاف ذلك، سيتم تنزيل مجموعة بيانات OpenNeuro في مجلد المستخدم الافتراضي. - افتح محطة طرفية جديدة على Mac / Linux أو WSL على Windows.
ملاحظة: لتشغيل مسار معالجة PyDesigner القياسي، اتبع مثال بنية الأوامر أدناه. أدخل الأوامر التالية أدناه في المحطة الطرفية/WSL. في هذا المثال، حيث يشير "./user_download_folder/" إلى مسار مجلد البيانات المثالي. سيكون هذا المسار خاصا بمكان تخزين بيانات المثال على كمبيوتر المستخدمين الفرديين. - لإنشاء مجلد جديد للمخرجات ، أدخل ما يلي في المحطة الطرفية:
$ mkdir ./user_download_folder/ ds004945-download /PyDesigner_Outputs - لتنشيط بيئة conda ، أدخل ما يلي في المحطة الطرفية:
$ conda activate mri - لتشغيل PyDesigner على مجموعة البيانات المثالية، أدخل ما يلي في المحطة الطرفية:
$ pydesigner - o ./user_download_folder/ds004945-download/PyDesigner_Outputs -s ./user_download_folder/ds004945-download/sub-01/dwi/sub-01_dwi.nii
ملاحظة: في هذا المثال، يشير ./user_download_folder/ إلى مسار مجلد البيانات المثالي. سيكون هذا المسار خاصا بجهاز الكمبيوتر الخاص بكل مستخدم. تتبع الأوامر الواردة في الخطوات من 12.3 إلى 12.5 بنية الخطوة 8.3. يستخدم هذا الأمر علامة -s ، علامة المعالجة القياسية ل PyDesigner ، والتي تقوم بتشغيل خطوات المعالجة التالية بهذا الترتيب: تقليل الضوضاء6 ، وتصحيح رنين Gibbs7 ، وعدم التشويه8،9 ، وإخفاء الدماغ ، والتنعيم ، وتصحيح التحيز Rician. - إذا تم تثبيت البرنامج بشكل صحيح ، ملء جميع المخرجات في مجلد PyDesigner_Outputs الذي تم إنشاؤه حديثا. قارن ملفات المعالجة المسبقة التي ينتجها PyDesigner مع مثال مجلد مجموعة البيانات الذي تمت معالجته مسبقا الموجود في مجلد "المشتقات" الذي تم تنزيله من OpenNeuro.
- راجع قسم النتائج التمثيلية لمقارنة خرائطها المقاييس بتلك الموجودة في نتائج مجموعة البيانات المثالية.
النتائج
يطبق برنامج PyDesigner خطوات متعددة لتصحيح الصور على بيانات الانتشار الأولية ويولد مخرجات تستخدم لتحسين الدقة على الملفات الأولية عند إجراء التحليل. تم التحقق من صحة كل خطوة متاحة في خط الأنابيب مسبقا من خلال المنشورات التي تمت مراجعتها من قبلالأقران 5،6،7،8،9،10،11 كما تمت مناقشتها في المقدمة. يمكن استخدام مخرجات البرنامج في تحليلات مثل ملفات تعريف الtractography ، ومصفوفات الاتصال ، والتحليلات الحكيمة ، وتحليلات العائد على الاستثمار ، وتحليلات fODF ، و TBSS ، والتحليلات القائمة على المثبتات.
يسرد موقع البرنامج23 جميع ملفات الإخراج التي تم إنشاؤها أثناء مسار المعالجة المسبقة. بعد تشغيل كل مطالبة، ستقوم وحدة التحكم بإخراج وصف لجميع العمليات المكتملة. هناك 3 أنواع من ملفات الإخراج: معالجة الملفات والمقاييس ومراقبة الجودة. يظهر هيكل دليل الإخراج في الشكل 1. تتوفر هذه الملفات عند استخدام المعالجة المسبقة القياسية (راجع القسم 7 من البروتوكول). إذا كان المستخدم يحتاج إلى استخدام أكثر تقدما (راجع القسم 8 من البروتوكول)، فستعتمد ملفات الإخراج المتاحة على العمليات التي تم إكمالها.
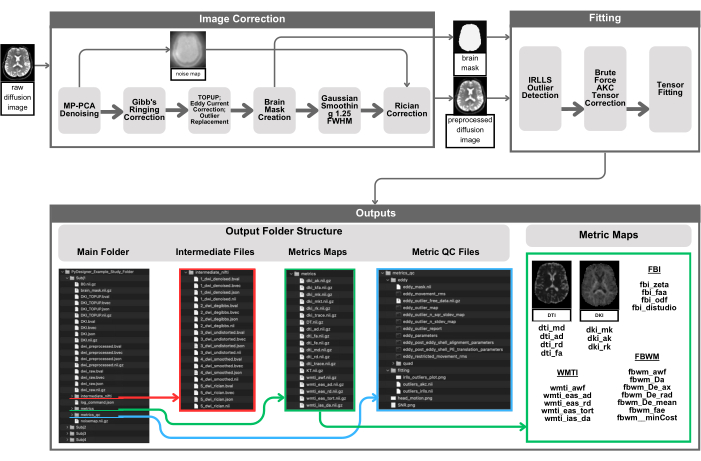
الشكل 1: التمثيل المرئي لخط أنابيب PyDesigner. تبدأ المعالجة المسبقة بتوفير إدخال 4D DWI إلى PyDesigner (أعلى اليسار) ، والذي يخضع بعد ذلك لتقليل الضوضاء MP-PCA لإنتاج 4D DWI غير ضوضائي وخريطة ضوضاء ثلاثية الأبعاد. ثم يخضع 4D DWI الخاضع للضوضاء لتصحيح رنين Gibbs ، وتصحيح انحياز Rician ، و TOPUP ، وتصحيح التيار الدوامي ، والتصحيح الخارجي. ثم يتم حساب قناع الدماغ للخطوات اللاحقة ، والكشف عن القيم المتطرفة ، وتركيب الموتر لتسريع العمليات الحسابية عن طريق إجرائها داخل قناع الدماغ فقط. يمكن العثور على المخرجات في الدلائل الفرعية في مجلد معالجة الموضوع الرئيسي - intermediate_nifti والمقاييس metrics_qc. لاحظ أن لقطات الشاشة لمخرجات PyDesigner لا يقصد بها أن تكون قائمة شاملة لجميع المخرجات الممكنة ، بل تقديم مثال مرئي تخطيطي لما يمكن أن يتوقعه المستخدمون. ستختلف المخرجات، سواء المخرجات النهائية أو الملفات الوسيطة، بناء على بيانات إدخال المستخدم وأعلام المعالجة المستخدمة. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
تجهيز
يتم استخدام ملفات المعالجة أثناء مسار PyDesigner ويتم تخزينها في دليل الإخراج الجذر. لكل خطوة معالجة مسبقة ، يتم حفظ ملفات DWI الوسيطة في مجلد الإخراج "intermediate_nifti" كما هو موضح في الشكل 1. يجب الرجوع إلى هذه الملفات عند معالجة أي مشكلات تتعلق بالمعالجة أو المخرجات لتقييم كل خطوة من خطوات المسار على حدة.
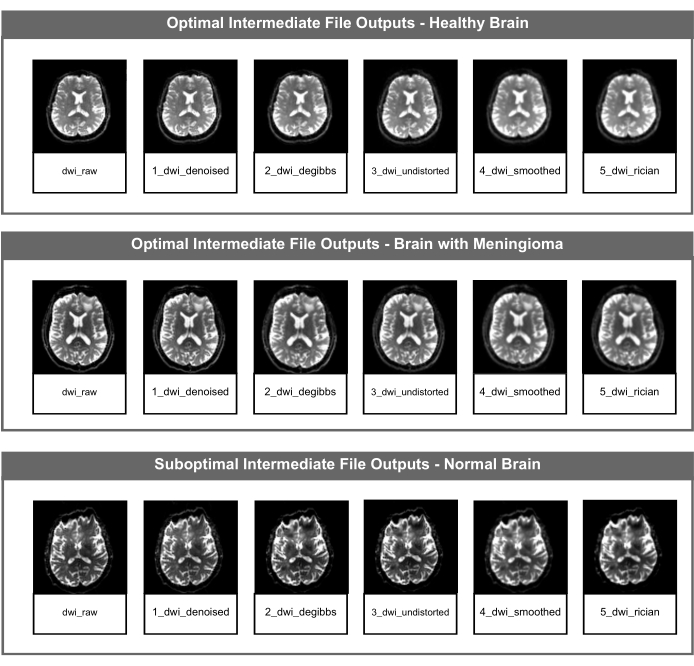
الشكل 2: ملفات DWI NifTI الوسيطة المثلى ودون المستوى الأمثل. يوضح الشكل ملف NIfTI الوسيط لكل خطوة تصحيح صورة من مسارات PyDesigner. الصف العلوي هو مثال على مخرجات الملفات الوسيطة المثلى باستخدام بيانات من دماغ بالغ سليم ، والصف الأوسط هو مثال على مخرجات الملفات الوسيطة المثلى باستخدام بيانات من الدماغ المرضي (الورم السحائي) ، ويعرض الصف السفلي مخرجات ملف وسيطة دون المستوى الأمثل باستخدام بيانات من دماغ بالغ سليم مع قطعة أثرية قابلة للحساسية لا علاقة لها ببنية الدماغ أو صحته. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
المقاييس
يحتوي هذا المجلد على جميع الخرائط المعلمية التي يحسبها PyDesigner (انظر الشكل 1). يتضمن ذلك الخرائط البارامترية ل DTI / DKI ، والتصوير الكروي الليفي (FBI) / المادة البيضاء الكرة الليفية (FBWM) ، ومقاييس سلامة مسار المادة البيضاء (WMTI) (الجدول 1) 16.

الجدول 1: النطاق المتوقع للقيم لمقاييس DTI/DKI ومكتب التحقيقات الفيدرالي/FBWM. يتضمن الجدول قائمة بمقاييس DTI و DKI و FBI و FBWM القوية التي تم إنشاؤها بواسطة PyDesigner ونطاقات قيمتها المتوقعة. يتم أيضا سرد التسلسلات المطلوبة والقيم b (s / mm2) لاشتقاق كل مقياس. مقاييس DTI المدرجة هي FA و MD و AD و RD. مقاييس DKI المدرجة هي MK و AK و RK و KFA. مقياس مكتب التحقيقات الفيدرالي المدرج هو إدارة الطيران الفيدرالية. مقاييس FBWM المدرجة هي AWF و DA و DE_AX و DE_RAD و FAE.
يمكن للمستخدمين إجراء مراقبة الجودة المرئية والقيمية (QC) لمتوسط الانتشار (MD) ، وتباين الخواص الجزئي (FA) ، ومقاييس التفرط المتوسط (MK) لتحديد النتائج دون المستوى الأمثل. إذا كانت هذه المقاييس دون المستوى الأمثل وفقا للمعايير الموضحة أدناه ، فيجب على المستخدمين إلقاء نظرة على كل ملف وسيط موضح في الشكل 2 لتحديد خطوة المعالجة المسبقة التي لم تنجح.
يتم استخدام مراقبة الجودة المرئية لتحديد النتائج دون المستوى الأمثل (على سبيل المثال ، مشكلات تركيب الموتر ، والقطع الأثرية). نوصي باستخدام ImageJ لمراقبة الجودة المرئية لضمان عدم إجراء أي معالجة للصورة عبر الإعدادات الافتراضية للبرنامج. يظهر الصف العلوي من الشكل 3 خرائط مترية نموذجية ل MD و FA و MK باستخدام عتبة معقولة بيولوجيا تبلغ 0-3 ميكرومتر2 / مللي ثانية ، 0-1 ميكرومتر2 / مللي ثانية ، و 0-2 ميكرومتر2 / مللي ثانية ، على التوالي (الشكل 3 [الصف العلوي]). يجب أن تحتوي خريطة MD على أعلى القيم في البطينين وقيم عالية في المادة الرمادية القشرية (الشكل 3 أ [الصف العلوي]). يجب أن تحتوي خريطة FA على مسارات واضحة للمادة البيضاء واضحة في جميع أنحاء الدماغ (الشكل 3 ب [الصف العلوي]). يجب أن تحتوي خريطة MK على قيم عالية في WM وقيم أقل في المادة الرمادية و CSF (الشكل 3C [الصف العلوي]). الشكل 3D [الصف العلوي] هو مثال على خريطة مترية بها مشكلات في تركيب الموتر ، مما ينتج عنه مجموعات من voxels ذات القيمة الصفرية في حالة حدوث أي مشكلات، راجع ملف log_command.json للعثور على أي أخطاء في المعالجة المسبقة. راجع الملفات الوسيطة لتحديد الخطأ المحدد. للمساعدة في استكشاف الأخطاء وإصلاحها، أرسل طلبا على صفحة PyDesigner GitHub.
تستخدم مراقبة جودة القيمة لتحديد ما إذا كانت فوكسل خريطة مترية معينة موحدة نسبيا بين الموضوعات لكل مجموعة بيانات. يعتمد نطاق القيمة المتوقع لكل خريطة ومجموعة بيانات على البيانات ومعلمات PyDesigner. في مجموعة بيانات الاختبار الخاصة بنا ، كان لدينا قمم متسقة في نطاقات 8000 إلى 10,000 و 2500 إلى 4000 و 5000 إلى 13,000 ل MD و FA و MK ، على التوالي ، باستخدام تجميع الرسم البياني الافتراضي FSLeyes. يقدم الصف السفلي من الشكل 3 أمثلة على تباين الرسم البياني. يحتوي الجدول 1 على قيم المحور x المتوقعة لهذه المقاييس. تعتبر الفوكسيل الأعلى أو الأقل من سمات مجموعة البيانات أو تشير إلى مشكلة في الأداة أو المعالجة المسبقة (الشكل 3D [الصف السفلي]).
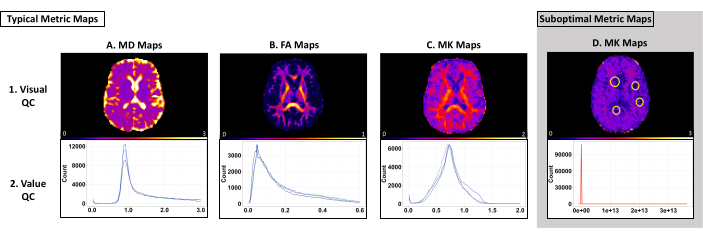
الشكل 3: أمثلة على الخرائط المترية والرسوم البيانية من PyDesigner بنتائج مثالية وغير مثالية. يعرض الصف العلوي أمثلة على خرائط MD وFA وMK أحادية الموضوع المستخدمة لمراقبة الجودة المرئية. يعرض الصف السفلي الرسوم البيانية متعددة الموضوعات المستخدمة لمراقبة الجودة للقيمة. (أ-ج) أمثلة نموذجية للخرائط المترية والرسوم البيانية التي تجتاز مراقبة الجودة المرئية والقيمية. يمثل كل خط أزرق على الرسوم البيانية لكل نوع مقياس مجموعة بيانات فردية. لاحظ أن كل مجموعة بيانات تتبع منحنى مماثلا وتقع ضمن نطاق مماثل. (د) مثال على خريطة مترية لا تجتاز مراقبة الجودة المرئية أو القيمية. لاحظ كيف يظهر الخط الأحمر على الرسم البياني منحنى يختلف عن منحنى A-C. ترجع الفوكسلات الصفرية المحاطة بدائرة على هذه الخريطة المترية إلى مشكلات تركيب الموتر أثناء المعالجة المسبقة (اللوحة D ، الصف العلوي). هذا الرسم البياني هو مثال على فوكسل أعلى أو أقل بشكل عام في مجموعة بيانات أكثر من المتوقع (اللوحة D ، الصف السفلي). الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
مراقبة الجودة
بمجرد أن يقوم PyDesigner بمعالجة البيانات ، يجب استخدام المجلد metrics_qc (انظر الشكل 1) لتحديد مجموعات البيانات دون المستوى الأمثل. لكل مجموعة بيانات ، يقوم PyDesigner بإخراج ثلاث مخططات تستخدم لمراقبة الجودة.
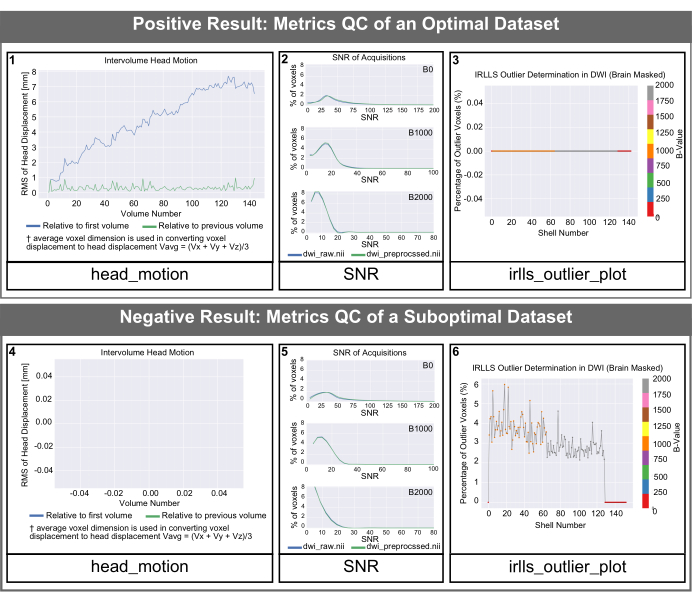
الشكل 4: الرسوم البيانية لمراقبة الجودة التي تم إنشاؤها بواسطة PyDesigner لمجموعات البيانات المثلى ودون المستوى الأمثل. SNR ، وحركة الرأس بين المجلدات ، والرسوم البيانية البعيدة التي تم إنشاؤها بواسطة PyDesigner. يمثل كلا الصفين بيانات من دماغ بالغ سليم. الصف العلوي هو مثال على الرسوم البيانية لمراقبة الجودة للحصول على مجموعة بيانات مثالية. يعرض الصف السفلي مخرجات مراقبة الجودة لمجموعة بيانات دون المستوى الأمثل مع قطعة أثرية للحساسية لا علاقة لها ببنية الدماغ أو صحته. يرجى ملاحظة أن حجم نص التسميات في المخرجات الافتراضية من PyDesigner أصغر مما سيظهر في هذا الشكل. لقد قمنا بزيادة حجم النص في هذا الشكل لسهولة القراءة. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
تظهر الرسوم البيانية head_motion في الشكل 4 إزاحة الرأس بالنسبة إلى المجلد الأول والحجم السابق. كما هو موضح في الشكل 4 (اللوحة 1) ، عادة ما يكون إزاحة الرأس صغيرا ويقوم PyDesigner بضبط هذه القطع الأثرية للحركة في خط أنابيب المعالجة القياسي باستخدام برنامج FSL Eddy جنبا إلى جنب مع TOPUP للحركة وتصحيح الحركة الحاليةالدوامة 9. بالنسبة لمجموعات البيانات دون المستوى الأمثل، قد يظهر الرسم البياني head_motion فارغا، كما هو موضح في الشكل 4 (اللوحة 4). يشير هذا إلى أن تصحيح الحركة الحالية الدوامة لم ينجح ، وبالتالي لم يتمكن PyDesigner من إخراج رسم بياني. يمكن العثور على ملفات سجل التصحيح الحالي Eddy في المجلد الفرعي الدوامة داخل المجلد metrics_qc (انظر الشكل 1). يعرض الرسم البياني لنسبة الإشارة إلى الضوضاء (SNR) 3 مخططات. كل مخطط لقيمة b مختلفة ويعرض كلا من البيانات المعالجة مسبقا والبيانات الأولية. للحصول على مجموعة بيانات مثالية ، يجب أن تكون ذروة SNR للبيانات الأولية ≥5 (الشكل 4 [اللوحة 2]). سيكون لمجموعات البيانات دون المستوى الأمثل ذروة SNR للبيانات الأولية تبلغ ≤3 (الشكل 4 [اللوحة 5]). من الناحية المثالية ، يجب أن يرى المستخدمون أن ذروة SNR لجميع قيم b تزداد بشكل طفيف ولكن ليس بشكل كبير. تم العثور على المخطط الخارجي في مجلد التركيب داخل metrics_qc ويظهر النسبة المئوية للقيم المتطرفة في مجموعة البيانات (الشكل 4 [اللوحة 3 و 6]). يجب أن تحتوي مجموعة البيانات الجيدة على نسبة منخفضة من القيم المتطرفة ، وعادة ما تكون أقل من 5٪ (الشكل 4 [اللوحة 3]). ستحتوي مجموعة البيانات دون المستوى الأمثل على نسبة كبيرة من القيم المتطرفة ، كما هو موضح في الشكل 4 (اللوحة 6).
أمثلة على نتائج مجموعة البيانات
بمجرد انتهاء PyDesigner من معالجة مجموعة بيانات المثال ، يجب تضمين جميع المخرجات في المجلد "PyDesigner_Outputs". يمكن مقارنة هذه المخرجات بتلك الموجودة في مجلد "المشتقات" المعبأ مع مجموعة البيانات النموذجية التي تم تنزيلها من OpenNeuro (تمت معالجتها على MacOS 12.4). إذا كان البرنامج يعمل بشكل صحيح ، فستكون بنية ملف "PyDesigner_Outputs" و "المشتقات" هي نفسها تماما. وبالمثل ، يجب أن تتطابق SNR وحركة الرأس والمخططات الخارجية الموجودة في المجلد الفرعي "metrics_qc" مع تلك الموجودة في الشكل 5 أ. يمكن مقارنة الخرائط المترية (الموجودة في المجلد الفرعي "المقاييس") عبر برامج التصوير مثل FSLeyes و MRIcron و ImageJ وما إلى ذلك. يمكن رؤية الرسوم البيانية لقيم FA و MD و MK من المقاييس المعالجة مسبقا في الشكل 5 ب. لاحظ أن جميع الرسوم البيانية المترية الموضحة في الشكل 5 ب يتم قياسها بناء على مقاييس قيمة القياس المقترحة في الجدول 1.
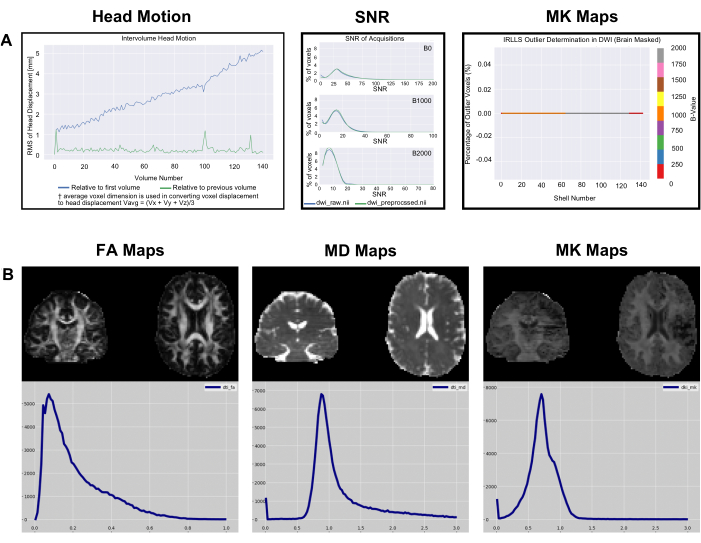
الشكل 5: مثال على مخططات قياس البيانات وقياس الجودة المتري. (أ) SNR ، وحركة الرأس بين وحدات التخزين ، والرسوم البيانية الخارجية التي تم إنشاؤها بواسطة PyDesigner لمثال البيانات التي تم تنزيلها من OpenNeuro. لاحظ أنه تمت زيادة حجم النص على المخططات في هذا الشكل لسهولة القراءة. (ب) يقوم مقياس الموضوع الفردي بتعيين الرسوم البيانية لعدد قيم فوكسل ل FA و MD و MK لنفس مجموعة البيانات ، والتي يتم تصورها من خلال FSLeyes v6.0. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
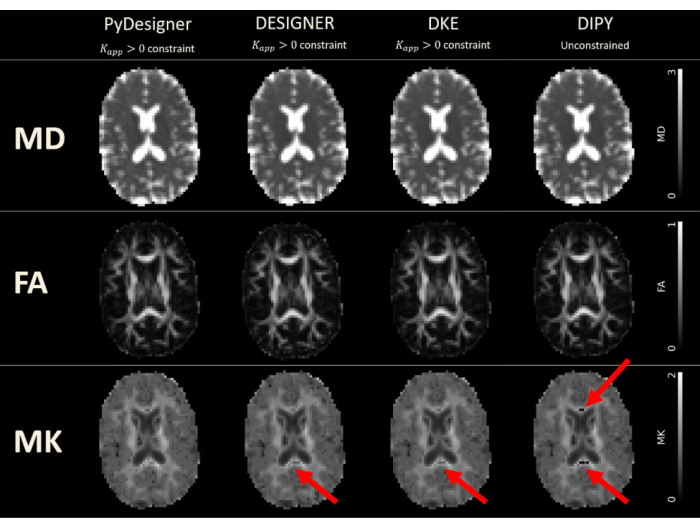
الشكل 6: خرائط DTI و DKI المشتقة من PyDesigner و DESIGNER و DKE و DIPY. تم إجراء تركيب الموتر باستخدام قيود Kapp > 0 في PyDesigner و DESIGNER5 و Diffusional Kurtosis Estimator (DKE) 27 ، بينما تم استخدام التركيب غير المقيد في Diffusion Imaging في Python (DIPY) 28 بسبب قيود البرنامج. وحدات MD هي ميكرومتر مربع لكل مللي ثانية (ميكرومتر2 / مللي ثانية) ، في حين أن المقاييس الأخرى بلا أبعاد. يمكن رؤية المتسربين بسبب مشكلة عامة من تصحيح رنين Gibbs في خرائط MK التي تنتجها DESIGNER و DKE و DIPY. تم استنساخ هذا الرقم بإذن من Dhiman et al.29. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
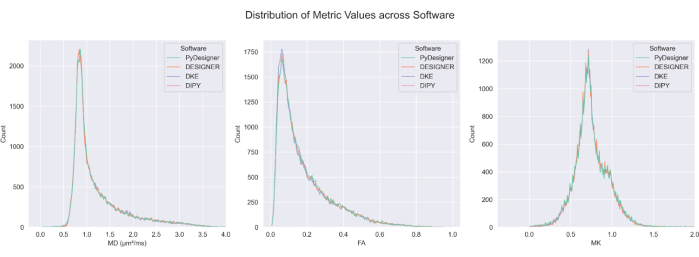
الشكل 7: مقارنة بين FA و MD و MK عبر خطوط الأنابيب. يتشابه توزيع القيم المحسوبة ل FA و MD و MK من PyDesigner و DESIGNER5 و DKE27 و DIPY28 في الأدمغة المستبعدة من CSF عبر معظم voxels. حساب الخريطة المترية عبر الأساليب قابل للمقارنة. تم استنساخ هذا الرقم بإذن من Dhiman et al.29. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
Discussion
كان الدافع الأساسي لتطوير PyDesigner هو تنفيذ العناصر الرئيسية ل DESIGNER مع استبدال جميع كود MATLAB ب Python ، مما يسمح بمزيد من قابلية النقل وإمكانية الوصول. ينتج عن PyDesigner و DESIGNER Dv1 مخرجات متطابقة تقريبا29. ومع ذلك ، هناك بعض الخيارات الإضافية والإعدادات الافتراضية وإصلاحات الأخطاء الطفيفة المضمنة في PyDesigner. تصف وثائق PyDesignerعبر الإنترنت 16 هذه بالتفصيل.
ينتج عن PyDesigner أيضا نتائج مماثلة لمقدر التفرط المنتشر (DKE) 27 والتصوير المنتشر في Python (DIPY) 28 أدوات تحليل DKI29 (انظر الشكل 6 والشكل 7) ولكن الخرائط التي تم إنشاؤها بناء على dMRI المعالجة مسبقا من DESIGNER تعتبر أكثر دقة نظرا لخوارزمية التركيب كما هو موضح من قبل Ades-Aron et al.5. انظر الشكل 6 لمقارنة الخرائط المترية MD و FA و MK من PyDesigner و DESIGNER5 و DKE27 و DIPY28. يوضح الشكل 7 مقارنة الرسوم البيانية MD و FA و MK من كل خط أنابيب. يؤدي الجمع بين تركيب الموتر المقيد والكشف عن القيم المتطرفة وتصحيح معامل التفرط الواضح إلى تركيب موتر أكثر قوة ودقة ، كما هو موضح في PyDesigner و DESIGNER5.
تتمثل إحدى مزايا PyDesigner على DESIGNER Dv1 في أنه متوفر عبر حاوية NeuroDock Docker30 ، مما يعزز بشكل كبير قابلية النقل ويبسط التثبيت. تعمل هذه الحاوية عبر جميع الأنظمة الأساسية الرئيسية لنظام التشغيل المتوافقة مع Docker، بما في ذلك Windows وMac OS وتوزيعات Linux المختلفة. استنادا إلى ملاحظات المستخدمين، سيتضمن الإصدار 2.0 من PyDesigner تحديثات لحاوية PyDesigner Docker. ستعمل التحسينات التي أدخلها الإصدار الجديد من PyDesigner وإدخال Dockerfile متعدد المراحل على حل جميع المشكلات الحالية التي يواجهها المستخدمون. إذا كان المستخدمون يواجهون مشكلات في التثبيت، فإننا نوصي بإرسال الأسئلة إلى صفحة مناقشة PyDesigner16. تتيح تقنية الحاويات من Docker أيضا النشر المباشر إلى المجموعات عالية الأداء (HPCs) لمعالجة DWIs المجمعة بسرعة على المجموعات المحلية المتوافقة مع Docker.
يتضمن PyDesigner أيضا حسابات النمذجة المجهرية التي تتجاوز DKI ، بما في ذلك WMTI3 و FBI و FBWM. بالنسبة إلى WMTI ، تكون مجموعة بيانات DKI القياسية كافية ، ويتم حساب معلمات البنية المجهرية المرتبطة بها افتراضيا. ومع ذلك ، يجب التأكيد على أن صلاحية WMTI تقتصر على مناطق المادة البيضاء ذات FA المرتفع (أي FA ≥ 0.4). بعض مقاييس WMTI لها دقة محدودة بسبب افتراض المحاذاة المتوازية للمحاور في أي فوكسل31 معين. FBI32،33،34 هي طريقة dMRI مميزة قابلة للتطبيق في جميع أنحاء المادة البيضاء الدماغية ، والتي تتطلب قيمة b عالية (على سبيل المثال ، b ≥ 4000 s / mm2) وبيانات dMRI التي تم أخذ عينات منها بحد أدنى 64 اتجاه تشفير الانتشار (جنبا إلى جنب مع بيانات b = 0). المخرجات الرئيسية لمكتب التحقيقات الفيدرالي هي وظيفة كثافة اتجاه الألياف (fODF) لكل فوكسيل للمادة البيضاء ، والتي يمكن استخدامها في تخطيط الجراكية للمادة البيضاء وتعمل كمدخل ل FBWM ، بالإضافة إلى تباين الخواص الجزئي داخل المحور المحوري (FAA). يستخدم FBWM بيانات dMRI من كل من DKI و FBI لتقدير نفس المعلمات مثل WMTI ولكن بدقة محسنة ، ويمكن تطبيقها في جميع أنحاء المادة البيضاء بغض النظر عن قيمة FA. وبالتالي ، إذا كانت هذه البيانات الإضافية متاحة ، تفضيل تقديرات FBWM على تلك الواردة في WMTI35. كما هو الحال مع مكتب التحقيقات الفيدرالي ، تم التحقق من صحة FBWM فقط في المادة البيضاء الدماغية للبالغين.
بالإضافة إلى الثوابت الدورانية التي يوفرها PyDesigner (اتجاه RAS) ، يتم إنتاج ملفات .fib الخاصة ب DSIstudio (اتجاه LPS) بشكل منفصل ل DTI و DKI و FBI. تحتوي ملفات .fib على معلومات اتجاهية ODF لإنشاء ملفات تعريف الtractography لكل طريقة. يمكن استخدام ملفات تعريف الtractography الناتجة لإنشاء مصفوفات الاتصال على مستوى الموضوع والمجموعة. تحتوي ملفات DKI و FBI .fib على معلومات متعددة الاتجاهات تنقل معلومات الألياف المتقاطعة غير المتوفرة مع DTI. علاوة على ذلك ، داخل كل ملف .fib ، تم تضمين الثوابت الدورانية المختلفة لكل طريقة ، والتي يمكن استخدامها في مجموعات مختلفة كمعايير للبذر والأداء وإيقاف tractography. يمكن العثور على مزيد من التفاصيل حول DSIstudio على موقع الويبالخاص بهم 36.
ميزة أخرى بارزة ل PyDesigner هي الإدخال متعدد الملفات ، والذي يسمح له بالتعامل مع مدخلات الملفات المختلفة - NifTi (.nii) ، NifTi المضغوط (.nii.gz) ، DICOM (.dcm) ، وتنسيق ملف MRtrix (.mif). يمكن ل PyDesigner تحديد معلومات الاكتساب تلقائيا من البيانات الوصفية للرأس بغض النظر عن تنسيق الإدخال وإجراء التصحيحات وفقا لذلك ، وبالتالي دعم نهج عدم التدخل. بغض النظر عن الاختلافات في البروتوكولات ، يمكن استخدام نفس الأمر (انظر أعلاه) لمعالجة مجموعة متنوعة من DWIs. وبالتالي يوفر هذا البرنامج الوقت والجهد عن طريق تقليل خطوات وأوامر المعالجة المسبقة اليدوية. في تحديث تم إصداره مؤخرا (v1.0-RC10) ، تم تحسين ذلك من خلال إدخال التوافق لمجموعات بيانات متعددة لوقت الصدى (متعدد TE). يسمح هذا ل PyDesigner بتشغيل خطوات المعالجة المسبقة للصور ، والتي تكون مستقلة إلى حد كبير عن TE ، على DWI متعدد TE لإنتاج صورة بأقل قدر من التشويش والتحف. ثم يتم إجراء حسابات الموتر المعتمدة على TE على كل TE على حدة لإنتاج مقاييس الانتشار أو التفرط.
نلاحظ أن PyDesigner v1.0 لا يمثل نقطة نهاية في تصميم مسارات المعالجة المسبقة للصور. يعد تطوير أدوات معالجة الصور والتحقق من صحتها مجالا نشطا للبحث. على وجه الخصوص ، أثناء إعداد هذه المخطوطة ، تم اقتراح تطورات جديدة في تقليل ضوضاء الصور وإزالة رنين جيبس ، مما أدى إلى إصدار خط أنابيب Designer المنقح ، Designer Dv237 ، والذي يتضمن تقليل الضوضاء المحسنة وتصحيح القطع الأثرية Gibbs للتصوير المغناطيسي الرقمي الرقمي التي تم الحصول عليها من خلال عمليات الاستحواذ الجزئية على فورييه35 ، وهي متوفرة على DESIGNER Github38. وبالمثل ، يعد الامتثال التنظيمي ل BIDS39 جانبا مهما من جوانب أي خط أنابيب لتحليل التصوير بالرنين المغناطيسي. يسمح تنسيق BIDS بمشاركة البيانات بشكل أكثر كفاءة وتنفيذ خط الأنابيب من خلال إنشاء هيكل محدد لجميع أنواع بيانات التصوير بالرنين المغناطيسي. على الرغم من أن PyDesigner ليس متوافقا مع BIDS حاليا، إلا أن الإصدار القادم من PyDesigner سيتضمن مخرجات متوافقة مع BIDS. بالإضافة إلى ذلك ، تمت كتابة PyDesigner في الأصل باستخدام الإصدار 3.7 من Python. في وقت هذا المنشور ، يعتبر Python 3.7 الآن قديما. ومع ذلك ، فإن المعلومات الواردة في هذه المخطوطة مستقلة عن إصدار بايثون. أي أن الإجراءات الموضحة هنا والتي تعد وظائف معالجة مسبقة مهمة (على سبيل المثال ، تقليل الضوضاء ، وتصحيح رنين جيبس ، وما إلى ذلك) ستتبع نفس سير العمل التشغيلي والمفاهيمي بغض النظر عن التغييرات في إجراءات التثبيت. الأهم من ذلك ، كما هو الحال مع جميع البرامج المستندة إلى Python ، سيتم تحديث إصدارات PyDesigner المستقبلية إلى إصدار جديد من Python. نحن نسعى جاهدين للتحقق من صحة هذه التطورات ودمجها بشكل أكبر بمرور الوقت.
للحصول على أحدث إصدار من PyDesigner ، بما في ذلك أي وثائق جديدة ذات صلة بتحديثات إصدار Python ، يتم تشجيع القراء على الرجوع إلى موقع الويب قبل البدء في تحليل جديد والمشاركة في منتدى المناقشة حيث يمكن إرسال الأسئلة المتعلقة ب PyDesigner16. يطلق على تطبيق Docker لقابلية النقل اسم NeuroDock30 ، والذي يحتوي على PyDesigner وتبعياته لتمكين المعالجة عبر مجموعة واسعة من الأنظمة الأساسية.
Disclosures
اي.
Acknowledgements
نحن ممتنون للمناقشات المفيدة مع أوليفيا هورن ودانيال لينش وغراهام وارنر.
تم دعم البحث الوارد في هذا المنشور ، جزئيا ، من خلال منح المعاهد الوطنية للصحة R01AG054159 و R01AG057602 و R01AG055132 و R01DC014021 و R01NS110347 و R21DA050085 و F31NS108623 و P20GM109040 و P50DC000422 و T32GM008716 و T32DC014435. المحتوى هو مسؤولية المؤلفين وحدهم ولا يمثل بالضرورة الآراء الرسمية للمعاهد الوطنية للصحة. وقدمت مؤسسة ليتوين تمويلا إضافيا.
Materials
| Name | Company | Catalog Number | Comments |
| Python version 3.7 or above | Python Software Foundation | https://www.python.org/ | |
| FMRIB Software Library (FSL) verison 6.0.2 or above | University of Oxford Centre for Integrative Neuroimaging | https://fsl.fmrib.ox.ac.uk/fsl/fslwiki/ | |
| MRtrix3 version 3.0_RC3 or above | numerous contributors | https://www.mrtrix.org/ | |
| Anaconda | Anaconda | https://anaconda.org/ | |
| Computer | Apple | Mac OS 10.14 | Built on Mac OS 10.14; tested on Mac OS 12.4, Mac OS 13, Windows 11 via WSL |
References
- Jensen, J. H., Helpern, J. A. MRI quantification of non-Gaussian water diffusion by kurtosis analysis. NMR Biomed. 23 (7), 698-710 (2010).
- Basser, P. J. Relationships between diffusion tensor and q-space MRI. Magn Reson Med. 47 (2), 392-397 (2002).
- Fieremans, E., Jensen, J. H., Helpern, J. A. White matter characterization with diffusional kurtosis imaging. Neuroimage. 58 (1), 177-188 (2011).
- Le Bihan, D., Poupon, C., Amadon, A., Lethimonnier, F. Artifacts and pitfalls in diffusion MRI. J Magn Reson Imaging. 24 (3), 478-488 (2006).
- Ades-Aron, B., et al. Evaluation of the accuracy and precision of the diffusion parameter EStImation with Gibbs and NoisE removal pipeline. Neuroimage. 183, 532-543 (2018).
- Veraart, J., Novikov, D. S., Christiaens, D., Ades-aron, B., Sijbers, J., Fieremans, E. Denoising of diffusion MRI using random matrix theory. Neuroimage. 142, 394-406 (2016).
- Kellner, E., Dhital, B., Kiselev, V. G., Reisert, M. Gibbs-ringing artifact removal based on local subvoxel-shifts. Magn Reson Med. 76 (5), 1574-1581 (2016).
- Holland, D., Kuperman, J. M., Dale, A. M. Efficient correction of inhomogeneous static magnetic field-induced distortion in Echo Planar Imaging. Neuroimage. 50 (1), 175-183 (2010).
- Andersson, J. L. R., Sotiropoulos, S. N. An integrated approach to correction for off-resonance effects and subject movement in diffusion MR imaging. Neuroimage. 125, 1063-1078 (2016).
- Godenschweger, F., et al. Motion correction in MRI of the brain. Phys Med Biol. 61 (5), 32-56 (2016).
- Andersson, J. L. R., Graham, M. S., Zsoldos, E., Sotiropoulos, S. N. Incorporating outlier detection and replacement into a non-parametric framework for movement and distortion correction of diffusion MR images. Neuroimage. 141, 556-572 (2016).
- Harris, C. R., et al. Array programming with NumPy. Nature. 585 (7825), 357-362 (2020).
- Virtanen, P., et al. SciPy 1.0: fundamental algorithms for scientific computing in Python. Nat Methods. 17 (3), 261-272 (2020).
- Agrawal, A., Verschueren, R., Diamond, S., Boyd, S. A rewriting system for convex optimization problems. J Control Decis. 5 (1), 42-60 (2018).
- Diamond, S., Boyd, S. CVXPY: A Python-embedded modeling language for convex optimization. , Available from: http://arxiv.org/abs/1603.00943 (2016).
- PyDesigner. , Available from: https://github.com/muscbridge/PyDesigner (2023).
- How to install Linux on Windows with WSL. , Available from: https://learn.microsoft.com/en-us/windows/wsl/install (2023).
- FSL Wiki. , Available from: https://fsl.fmrib.ox.ac.uk/fsl/fslwiki (2024).
- Fsl Instillation. , Available from: https://fsl.fmrib.ox.ac.uk/fsl/fslwiki/FslInstallation/Windows (2024).
- Installing conda. , Available from: https://conda.io/projects/conda/en/stable/user-guide/install/download.html (2024).
- Steps to install Anaconda on Windows Ubuntu terminal. , Available from: https://gist.github.com/kauffmanes/5e74916617f9993bc3479f401dfec7da (2024).
- Install MRtrix3. , Available from: https://www.mrtrix.org (2024).
- PyDesigner Read the Docs. , Available from: https://pydesigner.readthedocs.io/en/latest/ (2024).
- dicomSort. , Available from: https://github.com/muscbridgelab/dicomSort (2024).
- dcm2niix. , Available from: https://github.com/rordenlab/dcm2niix (2024).
- PyDesigner Example Dataset. , Available from: https://openneuro.org/datasets/ds004945 (2024).
- Tabesh, A., Jensen, J. H., Ardekani, B. A., Helpern, J. A. Estimation of tensors and tensor-derived measures in diffusional kurtosis imaging. Magn Reson Med. 65 (3), 823-836 (2011).
- Garyfallidis, E., et al. Dipy, a library for the analysis of diffusion MRI data. Front Neuroinform. 8, 8(2014).
- Dhiman, S., et al. PyDesigner: A Pythonic implementation of the DESIGNER pipeline for diffusion tensor and diffusional Kurtosis imaging. bioRxiv. , (2021).
- Neurodock. , Available from: https://hub.docker.com/r/dmri/neurodock (2024).
- Jelescu, I. O., et al. One diffusion acquisition and different white matter models: How does microstructure change in human early development based on WMTI and NODDI. Neuroimage. 107, 242-256 (2015).
- Jensen, J. H., Russell Glenn, G., Helpern, J. A. Fiber ball imaging. Neuroimage. 124, 824-833 (2016).
- Moss, H. G., Jensen, J. H. High fidelity fiber orientation density functions from fiber ball imaging. NMR Biomed. 35 (1), e4613(2022).
- Moss, H. G., McKinnon, E. T., Glenn, G. R., Helpern, J. A., Jensen, J. H. Optimization of data acquisition and analysis for fiber ball imaging. Neuroimage. 200, 690-703 (2019).
- McKinnon, E. T., Helpern, J. A., Jensen, J. H. Modeling white matter microstructure with fiber ball imaging. Neuroimage. 176, 11-21 (2018).
- DSIstudio. , Available from: https://dsi-studio.labsolver.org (2024).
- Chen, J., et al. Optimization and validation of the DESIGNER dMRI preprocessing pipeline in white matter aging. ArXiv. , arXiv:2305.14445v2 (2024).
- DESIGNER. , Available from: https://github.com/NYU-DiffusionMRI/DESIGNER (2024).
- Gorgolewski, K. J., et al. The brain imaging data structure, a format for organizing and describing outputs of neuroimaging experiments. Sci Data. 3, 160044(2016).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved