A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
تطبيق تحليل العوامل متعددة الأوميك غير الخاضعة للإشراف للكشف عن أنماط الاختلاف والعمليات الجزيئية المرتبطة بأمراض القلب والأوعية الدموية
In This Article
Summary
نقدم سير عمل مرن وقابل للتمديد قائم على Jupyter-lab للتحليل غير الخاضع للإشراف لمجموعات البيانات المعقدة متعددة الأوميكس التي تجمع بين خطوات المعالجة المسبقة المختلفة ، وتقدير نموذج تحليل العوامل متعددة الأوميكس ، والعديد من التحليلات النهائية.
Abstract
عادة ما تكون آليات المرض معقدة ويحكمها تفاعل العديد من العمليات الجزيئية المتميزة. تعد مجموعات البيانات المعقدة والمتعددة الأبعاد موردا قيما لتوليد المزيد من الأفكار حول تلك العمليات ، ولكن تحليل مجموعات البيانات هذه يمكن أن يكون صعبا بسبب الأبعاد العالية الناتجة ، على سبيل المثال ، عن ظروف مرضية مختلفة ، ونقاط زمنية ، وأوميكس تلتقط العملية بدقة مختلفة.
هنا ، نعرض نهجا لتحليل واستكشاف مجموعة بيانات multiomics المعقدة هذه بطريقة غير خاضعة للإشراف من خلال تطبيق تحليل عامل متعدد الأوميكس (MOFA) على مجموعة بيانات تم إنشاؤها من عينات الدم التي تلتقط الاستجابة المناعية في متلازمات الشريان التاجي الحادة والمزمنة. تتكون مجموعة البيانات من عدة مقايسات بدقة مختلفة ، بما في ذلك بيانات السيتوكين على مستوى العينة ، وبروتينات البلازما ، و العدلات الأولية ، وبيانات RNA-seq أحادية الخلية (scRNA-seq). يضاف المزيد من التعقيد من خلال قياس عدة نقاط زمنية مختلفة لكل مريض والعديد من المجموعات الفرعية للمرضى.
يحدد سير عمل التحليل كيفية دمج البيانات وتحليلها في عدة خطوات: (1) المعالجة المسبقة للبيانات ومواءمتها ، (2) تقدير نموذج MOFA ، (3) التحليل النهائي. توضح الخطوة 1 كيفية معالجة ميزات أنواع البيانات المختلفة ، وتصفية الميزات منخفضة الجودة ، وتطبيعها لتنسيق توزيعاتها لمزيد من التحليل. توضح الخطوة 2 كيفية تطبيق نموذج MOFA واستكشاف مصادر التباين الرئيسية داخل مجموعة البيانات عبر جميع omics والميزات. تقدم الخطوة 3 عدة استراتيجيات لتحليل المصب للأنماط التي تم التقاطها ، وربطها بظروف المرض والعمليات الجزيئية المحتملة التي تحكم تلك الحالات.
بشكل عام ، نقدم سير عمل لاستكشاف البيانات غير الخاضعة للإشراف لمجموعات البيانات المعقدة متعددة الأوميكس لتمكين تحديد محاور الاختلاف الرئيسية المكونة من ميزات جزيئية مختلفة يمكن تطبيقها أيضا على سياقات أخرى ومجموعات بيانات متعددة الأوميكس (بما في ذلك المقايسات الأخرى كما هو موضح في حالة الاستخدام المثالي).
Introduction
عادة ما تكون آليات المرض معقدة ويحكمها تفاعل العديد من العمليات الجزيئية المتميزة. إن فك رموز الآليات الجزيئية المعقدة التي تؤدي إلى أمراض معينة أو تحكم تطور المرض هي مهمة ذات أهمية طبية عالية لأنها قد تكشف عن رؤى جديدة لفهم الأمراض وعلاجها.
تمكن التطورات التكنولوجية الحديثة من قياس تلك العمليات على دقة أعلى (على سبيل المثال ، على مستوى الخلية الواحدة) وعلى طبقات بيولوجية مختلفة (على سبيل المثال ، الحمض النووي ، mRNA ، إمكانية الوصول إلى الكروماتين ، مثيلة الحمض النووي ، البروتينات) في نفس الوقت. وهذا يؤدي إلى زيادة توليد مجموعات البيانات البيولوجية الكبيرة متعددة الأبعاد ، والتي يمكن تحليلها بشكل مشترك لتوليد المزيد من الأفكار حول العمليات الأساسية. وفي الوقت نفسه، يظل الجمع بين مصادر البيانات المختلفة وتحليلها بطريقة ذات مغزى بيولوجي مهمة صعبة1.
تشكل الحدود التكنولوجية المختلفة والضوضاء ونطاقات التباين بين الأوميكس المختلفة تحديا واحدا. على سبيل المثال ، بيانات تسلسل الحمض النووي الريبي أحادي الخلية (scRNA-seq) متفرقة للغاية وغالبا ما تتأثر بتأثيرات تقنية أو دفعية كبيرة. بالإضافة إلى ذلك ، غالبا ما تكون مساحة الميزة كبيرة جدا ، وتتراوح عبر عدة آلاف من الجينات أو البروتينات المقاسة ، في حين أن أحجام العينات محدودة. ويزداد الأمر تعقيدا بسبب التصاميم المعقدة ، والتي قد تشمل العديد من الحالات المرضية والعوامل المربكة والنقاط الزمنية والقرارات. على سبيل المثال ، في حالة الاستخدام المقدمة ، كانت أنواع البيانات المختلفة متاحة إما على مستوى الخلية الواحدة أو العينة (المجمعة). إلى جانب ذلك ، قد تكون البيانات غير كاملة ، وقد لا تكون جميع القياسات متاحة لجميع الموضوعات التي تم تحليلها.
بسبب هذه التحديات ، لا يزال يتم تحليل omics المختلفة والميزات المضمنة بشكل منفصل فقط2 على الرغم من أن إجراء تحليل متكامل لا يمكن أن يوفر فقط صورة كاملة للعملية ولكن يمكن أيضا تعويض الضوضاء البيولوجية والتقنية من omic واحد بواسطة omics 3,4 أخرى. تم اقتراح عدة طرق مختلفة لإجراء تحليل متكامل للبيانات متعددة الأوميكس ، بما في ذلك طرق بايزي ، والطرق القائمة على الشبكة5،6 ، والتعلم العميق متعدد الوسائط7 ، وطرق تقليل الأبعاد عبر عامل المصفوفة8،9. بالنسبة للأخيرة ، أظهرت نتائج دراسة مرجعية كبيرة10 أن طريقة MOFA9 (تحليل العوامل متعددة الأوميك) هي واحدة من الأدوات الأكثر ملاءمة عندما يجب ربط البيانات بالتعليقات التوضيحية السريرية.
خاصة في البيئات المعقدة ، تعد طرق تحليل عوامل المصفوفة غير الخاضعة للإشراف نهجا مفيدا لتقليل التعقيد واستخراج الإشارات المشتركة والتكميلية من مصادر وميزات البيانات المختلفة. من خلال تحليل الفضاء المعقد إلى تمثيلات كامنة منخفضة الرتبة ، يمكن استكشاف مصادر التباين الرئيسية داخل البيانات بسرعة وربطها بالمتغيرات المشتركة المعروفة. في حالة مشاركة نفس نمط الاختلاف عبر ميزات متعددة (على سبيل المثال ، الجينات أو البروتينات) ، قد يتم تجميع ذلك إلى عوامل قليلة أثناء تقليل الضوضاء. يمكن استخدام التنظيم لزيادة تناثر معاملات النموذج ، مما يجعل النهج مناسبا تماما في الإعدادات التي تكون فيها مساحة المعلم كبيرة بينما يكون عدد العينات محدودا9.
يقدم هذا البروتوكول سير عمل تحليل مرن يستخدم نموذج MOFA لعرض كيفية استكشاف مجموعة بيانات معقدة متعددة الأوميكس بسرعة واستخلاص أنماط الاختلاف الرئيسية التي تميز مجموعة البيانات هذه. يتكون سير العمل من ثلاث خطوات رئيسية. في الخطوة الأولى ، المعالجة المسبقة للبيانات ومواءمتها ، يتم تقديم استراتيجيات مختلفة للمعالجة المسبقة للبيانات بناء على أنواع بيانات الإدخال المختلفة (scRNA-seq ، البروتينات ، السيتوكين ، البيانات السريرية). يوضح البروتوكول كيفية معالجة ميزات مجموعات بيانات الإدخال المختلفة ، وتصفية الميزات منخفضة الجودة ، وتطبيعها لتنسيق توزيعاتها. نوضح أيضا كيف يمكن أن تؤثر قرارات المعالجة المسبقة هذه على النتائج النهائية. في الخطوة الثانية ، يتم تطبيق نموذج MOFA على البيانات ، ويمكن استخدام تحليل التباين الناتج لتقييم تكامل مجموعات البيانات المختلفة. توضح الخطوة الثالثة كيفية ربط العوامل التي تم التقاطها بالمتغيرات المشتركة والكشف عن البرامج الجزيئية التي تحدد تلك العوامل. من خلال سير العمل المقدم ، تمكنا من استخراج العديد من العوامل الكامنة المرتبطة بالمتغيرات السريرية المشتركة في مجموعة بيانات للمرضى الذين يعانون من متلازمات الشريان التاجي وتحديد برامج المناعة متعددة الخلايا الأساسية المحتملة من مشروعسابق 11. سنستخدم مجموعة البيانات هذه هنا ، ولكن يمكن بسهولة تطبيق البروتوكول على سياقات أخرى ، بما في ذلك omics الأخرى.
تتكون مجموعة البيانات من عينات من المرضى الذين يعانون من متلازمات الشريان التاجي المزمنة المستقرة (CCS) ، ومتلازمات الشريان التاجي الحادة (ACS) ، ومجموعة مراقبة مع الشريان التاجي الصحي (غير CCS) (الشكل 1). يحدث ACS بسبب تمزق البلاك في CCS الموجود مسبقا ، مما يؤدي إلى اضطراب حاد في تدفق الدم إلى عضلة القلب وإصابة إقفارية لاحقة للقلب. تسبب هذه الإصابة استجابة التهابية من قبل الجهاز المناعي تليها مرحلة تعويضية ، والتي تستمر حتى عدة أيام بعد الحدث الحاد12. لتكون قادرة على توصيف هذه الاستجابة المناعية لمرضى ACS ، تم أخذ عينات الدم في أربع نقاط زمنية مختلفة: الحادة (TP1). بعد إعادة الاستقناء (14 [± 8] ح) (TP2) ؛ 60 [± 12] ساعة في وقت لاحق (TP3) ؛ قبل التفريغ (6.5 [±1.5] يوما) (TP4) (الشكل 1 أ). بالنسبة ل CCS والمرضى الذين يعانون من الشريان التاجي الصحي ، كانت نقطة زمنية واحدة فقط متاحة- (TP0). لجميع المرضى والنقاط الزمنية تم قياس فحوصات مختلفة بناء على عينات الدم: العلامات السريرية للالتهاب (الكرياتين كيناز (CK) ، CK-MB ، Troponin ، C-reactive protein (CRP)) ، scRNA-seq لخلايا الدم أحادية النواة المحيطية (PBMCs) ، تحليل السيتوكين ، بروتينات البلازما وبيانات prime-seq13 من العدلات.
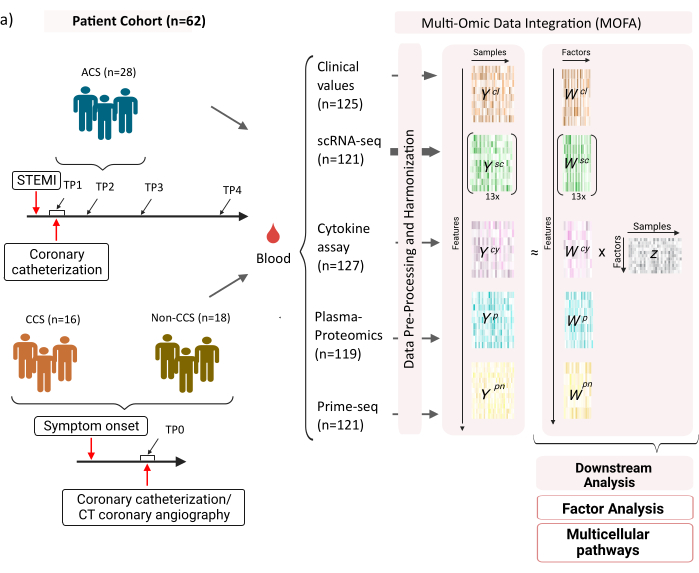
الشكل 1: مجموعة بيانات الإدخال متعددة الأوميك لاحتشاء عضلة القلب. مجموعة بيانات المدخلات: تتضمن البيانات التي تم تحليلها عينات دم من المرضى (ن = 62) الذين يعانون من متلازمة الشريان التاجي الحادة (ACS) ، ومتلازمات الشريان التاجي المزمنة (CCS) ، والمرضى الذين يعانون من الشريان التاجي الصحي (غير CCS). بالنسبة لمرضى ACS ، تم تضمين عينات الدم في أربع نقاط زمنية مختلفة (TP1-4) ، لمرضى CCS وغير مرضى CCS في نقطة زمنية واحدة (TP0). يتم التعامل مع كل مجموعة من المرضى والنقاط الزمنية كعينة منفصلة في التحليل. تم قياس مقايسات أوميك مختلفة على العينات: اختبارات الدم السريرية (ن = 125) ، scRNA-seq (ن = 121) ، بروتينات البلازما (ن = 119) ، مقايسة السيتوكين (ن = 127) و العدلات الأولية (ن = 121). بعد ذلك ، تم تطبيق البروتوكول الموصوف لدمج البيانات عبر جميع omics واستكشافها باستخدام نموذج MOFA والمزيد من التحليل النهائي (تحليل العوامل ، إثراء المسار). يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
كمدخل لسير العمل كما هو موضح هنا ، نأخذ الأعداد الأولية من بيانات scRNA-seq بعد المعالجة باستخدام cellranger ومراقبة الجودة (QC) كما هو موضح ، على سبيل المثال ، في البرنامج التعليمي للمعالجة المسبقة14 الهزيلة. بالنسبة للتعليق التوضيحي من نوع الخلية ، استخدمنا خط أنابيب Azimuth15 الآلي. ثم يتم تجميع الأعداد على مستوى العينة لكل نوع خلية عن طريق أخذ المتوسط عبر جميع الخلايا لكل عينة ونوع الخلية (تجميع شبه مجمع). يتم تضمين البروتينات البلازمية كشدة طبيعية ومتوسطة التركيز ، وبالنسبة للعدلات ، نأخذ عدد إكسون المعرف الجزيئي الفريد UMI (UMI) من التسلسل الأولي. على السيتوكين والقيم السريرية ، لم يتم تطبيق أي معالجة مسبقة سابقة. مزيد من التفاصيل حول توليد البيانات (التجريبية) موضحة في المخطوطة11 المقابلة. نظرا لأن النتائج المقدمة هنا تستند إلى استخدام التعليق التوضيحي الآلي للسمت لأنواع الخلايا في بيانات scRNA-seq مقارنة بالاستراتيجية القائمة على العلامات التي تم استخدامها في المنشور المشار إليه ، فإن النتائج المقدمة هنا متشابهة ولكنها ليست بالضبط نفس النتائج المعروضة في المنشور. في المخطوطة ، يمكن إظهار أن استراتيجية التعليقات التوضيحية من نوع الخلية لا تغير الأنماط الرئيسية والتفسيرات البيولوجية للتحليل ولكن قد تختلف التغييرات الصغيرة في القيم الدقيقة الناتجة عن النموذج. بشكل عام ، كانت بيانات الإدخال عبارة عن مجموعة بيانات معقدة متعددة الأبعاد بما في ذلك نقاط زمنية ومستويات قياس مختلفة (خلايا مفردة مقابل السائبة) لأكثر من 10000 ميزة مختلفة (الجينات والبروتينات والقيم السريرية). وقد تبين أن استراتيجية المعالجة المسبقة الصارمة وتنسيق البيانات التي يتبعها تحليل وزارة الخارجية هي أداة مفيدة وسريعة لاستكشاف البيانات واستخراج برنامج المناعة ذي الصلة. يتم التعامل مع كل نقطة زمنية ومجموعة المرضى كعينة مستقلة في تحليل وزارة الخارجية. يعتبر كل نوع من أنواع البيانات ونوع الخلية وجهة نظر منفصلة في تحليل وزارة الخارجية.
يوفر هذا البروتوكول إرشادات لإعداد بيانات الإدخال لسير العمل ، وتنفيذ خطوات سير العمل المختلفة ، وتخصيص التكوينات ، وتفسير الأرقام الناتجة ، وضبط التكوينات بشكل متكرر بناء على التفسيرات. يتم تقديم نظرة عامة على الخطوات المختلفة للبروتوكول ، ومجموعات بيانات الإدخال المطلوبة في كل خطوة ، والأرقام ومجموعات البيانات الناتجة من خلال نظرة عامة على سير العمل الفني (الشكل 2).
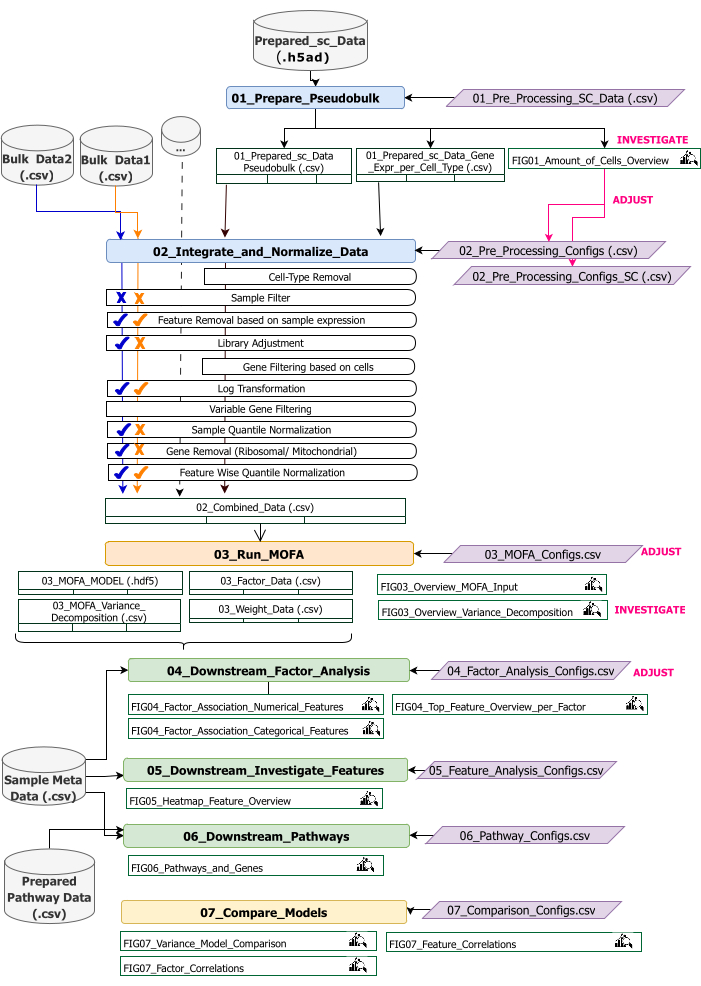
الشكل 2: نظرة عامة على سير العمل الفني. مخطط سير العمل لتحليل مجموعة بيانات متعددة الأوميكس. يتم تمييز العناصر المختلفة بألوان ورموز مختلفة. دفاتر Jupyter التي تنتمي إلى خطوة المعالجة المسبقة للبيانات وتنسيقها (1) ملونة باللون الأزرق. دفاتر Jupyter التي تنتمي إلى خطوة "نموذج MOFA" (2) ملونة باللون البرتقالي. دفاتر Jupyter التي تنتمي إلى خطوة "تحليل المصب" (3) ملونة باللون الأخضر. دفتر ملاحظات Jupyter واحد لاستخدامه لمقارنة النتائج ملون باللون الأصفر. يتم تمييز ملفات التكوين حيث يمكن تعديل معلمات تنفيذ سير العمل باللون الأرجواني. تتم الإشارة إلى مجموعات بيانات الإدخال المطلوبة لتشغيل سير العمل برمز مجموعة البيانات ويتم تمييزها باللون الرمادي. تتم الإشارة إلى جميع مخرجات الشكل التي يتم إنشاؤها أثناء تنفيذ سير العمل بواسطة رمز العدسة المكبرة. تتم الإشارة إلى مجموعات البيانات التي تم إنشاؤها أثناء تنفيذ سير العمل كجداول. بشكل عام ، يتم تنفيذ سير العمل بالتتابع: (1) تتكون المعالجة المسبقة للبيانات وتنسيقها من خطوتين: الجيل الأول من جدول شبه مضخم يعتمد على بيانات إدخال scRNA-seq (01_Prepare_Pseudobulk) والتكامل والتطبيع اللاحق لهذه البيانات مع جميع المدخلات الأخرى على مستوى العينة (المجمعة) (02_Integrate_and_Normalize_Data). ضمن هذه الخطوة عبر ملفات التكوين ، من الممكن تكوين لكل مجموعة بيانات على حدة أي من خطوات المعالجة المسبقة والتطبيع المشار إليها (على سبيل المثال ، مرشح العينة) يجب تطبيقها. (2) "نموذج MOFA": يقوم بتشغيل نموذج MOFA على الإدخال الذي تم إنشاؤه للخطوة الأولى مع التكوينات المحددة في ملف التكوين (03_MOFA_configs.csv) (3) "تحليل المصب": يتكون من ثلاثة دفاتر ملاحظات مختلفة يمكن تشغيلها بشكل مستقل عن بعضها البعض لتوليد رؤى حول نتائج MOFA التي تم إنشاؤها وربطها بعينة البيانات الوصفية (المتغيرات المشتركة) المقدمة كمدخلات عبر ملف "نموذج Data.csv الوصفية". (4) "مقارنة النموذج": هي خطوة صغيرة منفصلة يمكن استخدامها لمقارنة النماذج المختلفة التي تم إنشاؤها في الخطوة 2. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
يتكون سير العمل من عدة دفاتر ملاحظات Jupyter مكتوبة بلغة R و Python (المعرفة بلغة R و Python غير مطلوبة لتشغيل سير العمل ولكنها قد تكون مفيدة في حالة ظهور أخطاء). في خطوات مختلفة من البروتوكول ، يتم تغيير المعلمات عبر ملفات التكوين (ملفات ".csv" التي تحتوي على البادئة اللاحقة "_Configs" في الاسم). ضمن البروتوكول ، نحدد فقط المعلمات التي تحتاج إلى تغيير بدءا من التكوين الافتراضي.
يمكن أيضا تغيير العديد من المعلمات الأخرى ، على سبيل المثال لتخصيص المعالجة المسبقة. يتم تقديم توثيق لهذه المعلمات والتفسيرات في الملف "Documentation_Config_Parameter" ، والذي يتم تضمينه في المستودع الذي تم تنزيله.
Protocol
1. الاستعدادات: الإعداد الفني والتثبيت
ملاحظة: لتشغيل هذا البرنامج ، قم بتثبيت wget و git و Apptainer مسبقا على الجهاز. يتم إعطاء دليل لتثبيت Apptainer على أنظمة مختلفة (Linux و Windows و Mac) هنا: https://apptainer.org/docs/admin/main/installation.html. يمكن العثور على معلومات التثبيت على git هنا: https://git-scm.com/book/en/v2/Getting-Started-Installing-Git. اعتمادا على حجم مجموعات بيانات الإدخال المختلفة ، يوصى بتشغيل سير العمل على جهاز مناسب (16 وحدة معالجة مركزية ، ذاكرة 64 جيجابايت). يمكن إجراء اختبار دخان مع بيانات المثال المقدمة على الجهاز المحلي. وترد التعليمات والنواتج المتوقعة من تشغيل البروتوكول على بيانات المثال في الملف التكميلي 1. ارجع إلى ملف الفيديو التكميلي 1 لمعرفة الخطوات المهمة للبروتوكول التي يتم تنفيذها على مجموعة البيانات الموضحة أعلاه.
- افتح وحدة التحكم واختر أو أنشئ مجلدا حيث سيتم تخزين جميع التعليمات البرمجية للتحليل والمخرجات. انتقل إلى المجلد عن طريق كتابة الأمر: cd path_to_folder في الجهاز.
- قم بتنزيل أو استنساخ مستودع التعليمات البرمجية من Github (https://github.com/heiniglab/mofa_workflow) أو عن طريق كتابة git clone https://github.com/heiniglab/mofa_workflow.git في نافذة المحطة الطرفية.
- قم بتنزيل الصورة التي تحتوي على جميع عمليات التثبيت المطلوبة من Zenodo عن طريق كتابة wget https://zenodo.org/records/11192947/files/mofa_image.sif في نافذة المحطة الطرفية.
- قم بإنشاء مجلد يتم فيه تخزين جميع بيانات النتيجة عن طريق كتابة نتائج mkdir في نافذة المحطة الطرفية.
- قم بإنشاء مجلد تتم فيه إضافة جميع بيانات الإدخال التي سيتم استخدامها في التحليل عن طريق كتابة mkdir input_data في نافذة المحطة الطرفية.
- قم بتنفيذ الحاوية التي ستبدأ جلسة JupyterLab عن طريق كتابة الأمر التالي في المحطة الطرفية: apptainer run mofa_image.sif. انسخ عنوان URL الذي يتم إرجاعه بواسطة الأمر إلى المتصفح ، والذي سيفتح جلسة Jupyter-lab (يمكن العثور على مزيد من المعلومات حول Jupyter-lab في وثائق البرنامج16).
ملاحظة: عند تنفيذ سير العمل محليا على جهاز كمبيوتر محمول ، يوصى باستخدام الأمر apptainer exec mofa_image.sif jupyter-lab بدلا من ذلك ، والذي سيعيد عنوان مضيف محلي مباشرة. في حالة تنفيذ الحاوية داخل بيئة حوسبة مجمعة ، قد يكون من الضروري إعداد إعادة توجيه المنفذ ، والذي يمكن القيام به عبر ssh.
2. التهيئة وإعداد البيانات
- في جلسة Jupyter-Lab ، استخدم قائمة التنقل على الجانب الأيسر. انتقل إلى مجلد input_data بالنقر المزدوج فوق input_data.
- انسخ جميع مجموعات البيانات التي سيتم استخدامها كمدخلات للتحليل إلى دليل input_data باستخدام السحب والإفلات. اسحب الملف من المجلد حيث يوجد حاليا وقم بإفلاته في جلسة Jupyter-lab في المنطقة الموجودة أسفل المجلد input_data.
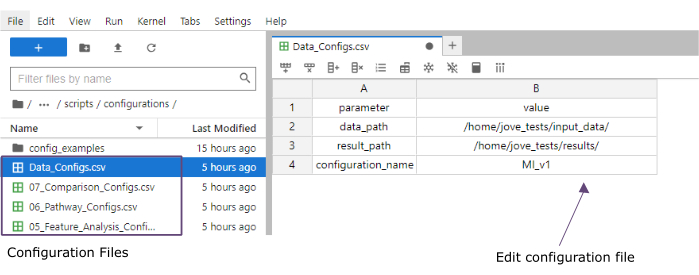
ملاحظة: يجب أن تكون جميع مجموعات البيانات إما بتنسيق .csv أو .h5ad (في حالة البيانات أحادية الخلية). يجب أن تحتوي جميع ملفات .csv على عمود sample_id مطابق (يجب استخدام معرفات متطابقة عبر مجموعات البيانات). سيتم استخدام جميع الأعمدة الأخرى كميزات. ضمن ملف h5ad- ، يجب أن يحتوي التعليق التوضيحي للخلية على معرفين يحددان sample_id و cluster_id. سيتم استخدامها للتجميع والمطابقة. يجب تحويل مجموعات بيانات Omic بتنسيقات أخرى إلى تنسيق .csv المحدد قبل الاستخدام (الشكل 3). يمكن تحويل مجموعات بيانات scRNA-seq الواردة بتنسيق .h5seurat إلى .h5ad لتنفيذ دفتر Jupyter: 00_Data_Conversion.ipynb. - انتقل إلى مجلد التكوينات بالنقر فوق رمز المجلد ثم النقر المزدوج فوق المجلدات mofa_workflow والبرامج النصية والتكوينات. داخل المجلد ، افتح الملف Data_configs.csv بالنقر المزدوج عليه.
- في عمود القيمة ، أضف المسارات إلى مجلدات مجلدي input_data (data_path) والنتائج (result_path). أضف اسما ستتم إضافته كامتداد ملف إلى جميع الملفات المحفوظة في عمود القيمة الخاص configuration_name (يستخدم هذا البروتوكول MI_v1 [احتشاء عضلة القلب الإصدار 1]) (الشكل 4).
- احفظ التغييرات بالنقر فوق ملف > حفظ ملف CSV في القائمة في الأعلى.
- استخدم قائمة التنقل على الجانب الأيسر للانتقال إلى مجلد البرامج النصية بالنقر فوق البرامج النصية. افتح دفتر ملاحظات التهيئة بالنقر المزدوج فوق 00_Configuration_Update.ipynb. قم بتنفيذ البرنامج النصي بالنقر فوق إعادة تشغيل kernel وتشغيل جميع الخلايا زر في الأعلى ، والنقر فوق إعادة التشغيل في النافذة المنبثقة (الشكل 5).
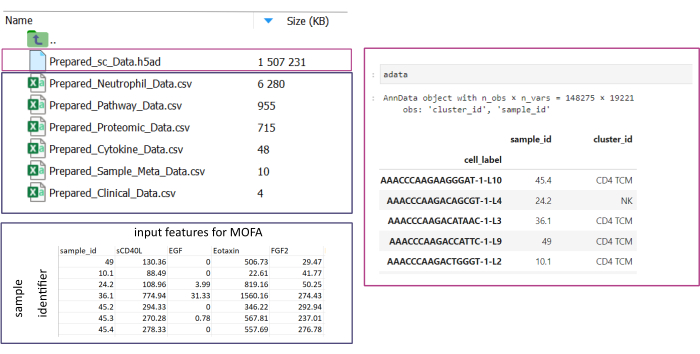
الشكل 3: إدخال البيانات وإعدادها. لتنفيذ سير العمل ، يجب تخزين جميع البيانات في مجلد input_data محدد. لكل مجموعة بيانات إدخال يجب توفير ملف منفصل. يجب إعطاء بيانات الخلية الواحدة بتنسيق .h5ad تحتوي على تعليق توضيحي للخلية على cluster_id (ينتج ، على سبيل المثال ، من خطوات التعليق التوضيحي السابقة لنوع الخلية) وعمود sample_id (يحدد بشكل فريد كل عينة منفصلة يجب تحليلها). يجب تقديم جميع مجموعات بيانات المدخلات الأخرى بتنسيق ".csv" ، بما في ذلك عمود واحد يحدد sample_id (المطابقة للعمود المقابل لبيانات الخلية الواحدة) والميزات التي سيتم استخدامها في تحليل وزارة الخارجية في جميع الأعمدة الأخرى. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
الشكل 4: ملفات تكوين مختبر Jupyter. أثناء تنفيذ سير العمل ، يتم تحديد التغييرات في المعلمات (على سبيل المثال ، ضبط خيارات التصفية وما إلى ذلك) عبر ملفات التكوين ".csv". داخل المستودع المستنسخ ، يتم تضمين ملفات التكوين الافتراضية لكل خطوة. يمكن تحريرها مباشرة في وحدة تحكم jupyter-lab ، كما هو الحال في جدول البيانات. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
الشكل 5: نصوص دفاتر Jupyter. يتكون سير العمل الكامل من سلسلة من دفاتر ملاحظات Jupyter التي سيتم تنفيذها بالتتابع بعد تعديل ملفات التكوين المقابلة. بالنقر المزدوج على دفتر Jupyter على الجانب الأيسر ، سيتم فتح الملف المقابل على الجانب الأيمن. يمكن بدء التنفيذ الكامل للملف مع تمييز الزر في الأعلى. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
3. المعالجة المسبقة للبيانات ومواءمتها
- المعالجة المسبقة - تحويل بيانات sc إلى حجم كاذب.
ملاحظة: يجب تنفيذ هذه الخطوة فقط إذا تم استخدام بيانات خلية واحدة في التحليل.- استخدم قائمة التنقل على الجانب الأيسر للانتقال إلى مجلد التكوينات بالنقر المزدوج فوق التكوين. افتح الملف 01_Preprocessing_SC_Data.csv بالنقر المزدوج. تحقق من القيم المملوءة تلقائيا في الملف، وإذا لزم الأمر، اضبط القيم الموجودة في العمود data_name لتتوافق مع أسماء ملفات مجموعات البيانات أحادية الخلية في المجلد input_data الذي سيتم استخدامه للتحليل.
ملاحظة: بشكل افتراضي، ستتم إضافة كافة أسماء ملفات .h5ad في مجلد بيانات الإدخال إلى ملف التكوين في البرنامج النصي للتهيئة. إذا كان لا ينبغي استخدام بعض مجموعات البيانات للتحليل ، فيمكن إزالتها هنا. - احفظ التغييرات التي تم إجراؤها بالنقر فوق ملف > حفظ ملف CSV في القائمة الموجودة في الأعلى.
- استخدم قائمة التنقل على الجانب الأيسر للانتقال إلى مجلد البرامج النصية بالنقر فوق البرامج النصية. افتح دفتر الملاحظات 01_Prepare_Pseudobulk.ipynb بالنقر المزدوج عليه. قم بتنفيذ البرنامج النصي بالنقر فوق إعادة تشغيل النواة وتشغيل جميع الخلايا زر في الأعلى ، والنقر فوق إعادة التشغيل في النافذة المنبثقة.
- استخدم قائمة التنقل على الجانب الأيسر للانتقال إلى مجلد الأشكال بالنقر المزدوج أولا على الأشكال ثم على 01_figures. افتح FIG01_Amount_of_Cells_overview المؤامرة التي تم إنشاؤها حديثا بالنقر المزدوج عليها.
ملاحظة: قد يستغرق تنفيذ دفتر الملاحظات عدة دقائق. عند تنفيذ دفتر الملاحظات بنجاح ، ستظهر نافذة منبثقة وسيتم تحديث FIG01_Amount_of_Cells_Overview الملف بواسطة دفتر الملاحظات أو إنشاؤه حديثا. يمكن أن يشير العمود آخر تعديل إلى وقت إنشاء الملف لتقييم ما إذا كان ملفا جديدا أم قديما. - تحقق من المخطط لتحديد مجموعات نوع الخلية التي تحتوي على عدد منخفض جدا من الخلايا لكل عينة. دون أسماء أولئك الذين cluster_ids استبعادهم في الخطوات اللاحقة (الشكل 6).
- استخدم قائمة التنقل على الجانب الأيسر للانتقال مرة أخرى إلى مجلد التكوينات بالنقر فوق ... ثم النقر المزدوج على التكوينات. افتح الملف 02_Preprocessing_Configs_SC.csv بالنقر المزدوج عليه.
- تحقق من القيم في العمودين configuration_name و data_name واضبطها إذا لزم الأمر.
ملاحظة: ضمن البرنامج النصي للتهيئة، يتم ملء هذه القيم مسبقا بكافة أسماء ملفات .h5ad في مجلد بيانات الإدخال والقيمة configuration_name التي تم تعيينها داخل ملف Data_Configs.csv مسبقا. في حالة استبعاد الملفات من التحليل أو استخدام امتداد آخر لأسماء الملفات ، يمكن تعديل ذلك هنا. - اضبط القيمة في العمود cell_type_exclusion وأضف كل cluster_id التي تم تحديدها لاستبعادها في الخطوة السابقة مفصولة ب '،'.
- احفظ التغييرات بالنقر فوق ملف > حفظ ملف CSV في شريط التنقل في الأعلى.
- استخدم قائمة التنقل على الجانب الأيسر للانتقال إلى مجلد التكوينات بالنقر المزدوج فوق التكوين. افتح الملف 01_Preprocessing_SC_Data.csv بالنقر المزدوج. تحقق من القيم المملوءة تلقائيا في الملف، وإذا لزم الأمر، اضبط القيم الموجودة في العمود data_name لتتوافق مع أسماء ملفات مجموعات البيانات أحادية الخلية في المجلد input_data الذي سيتم استخدامه للتحليل.
- المعالجة المسبقة - تنسيق ودمج مصادر بيانات omics الأخرى.
- افتح 02_Preprocessing_Configs.csv الملف بالنقر المزدوج فوقه واضبط تكوين المعالجة المسبقة لكل مجموعة من مجموعات البيانات التي سيتم تضمينها وتخزينها في المجلد data_input (صف واحد لكل مجموعة بيانات).
- تحقق من القيم في العمودين configuration_name و data_name واضبطها إذا لزم الأمر.
- اضبط المعلمات الأخرى في الأعمدة وفقا لذلك ، اعتمادا على خطوات المعالجة المسبقة التي يجب تطبيقها.
ملاحظة: تتم إضافة القيم الافتراضية لكل مجموعة بيانات موجودة داخل المجلد input_dataset ولكنها ليست خاصة بأنواع البيانات الفردية للبيانات. لذلك ، ستكون التعديلات ضرورية. يتم تقديم توثيق مفصل للمعلمات في ملف Documentation_Config_Parameter.doc . - احفظ التغييرات بالنقر فوق ملف > حفظ ملف CSV.
- استخدم قائمة التنقل على الجانب الأيسر للانتقال إلى مجلد البرامج النصية بالنقر فوق البرامج النصية. افتح دفتر الملاحظات 02_Integrate_and_Normalized_Data_Sources.ipynb بالنقر المزدوج عليه. قم بتنفيذ البرنامج النصي بالنقر فوق إعادة تشغيل النواة وتشغيل جميع الخلايا زر في الأعلى ، والنقر فوق إعادة التشغيل في النافذة المنبثقة.
- استخدم قائمة التنقل على الجانب الأيسر للانتقال إلى مجلد 02_results الذي تم إنشاؤه بالنقر فوق رمز المجلد ثم النقر المزدوج فوق النتائج و 02_results. تحقق مما إذا كان يتضمن الملف 02_Combined_data_'configuration_name'_Integrated.csv الذي يحتوي على ملف إدخال البيانات المدمج الذي تمت معالجته مسبقا.
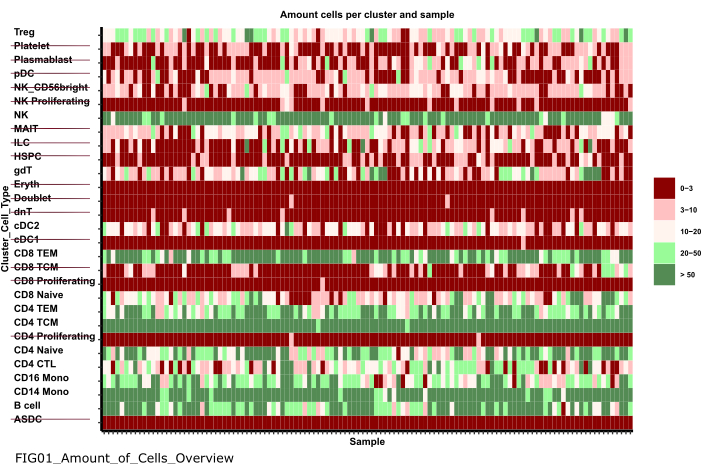
الشكل 6: المعالجة المسبقة للبيانات ومواءمتها. أحد مخرجات الخطوة "01_Prepare_Pseudobulk" هو الحبكة "Fig01_Amount_of_Cells_Overview". هنا ، لكل cluster_id (يشير المحور y إلى نوع الخلية من خطوات التعليقات التوضيحية السابقة لنوع الخلية) ، يتم إعطاء عدد الخلايا لكل عينة ('sample_id'). ضمن النتائج المقدمة ، يتم استبعاد أنواع الخلايا التي تحتوي على كمية منخفضة من الخلايا لكل عينة من التحليل اللاحق (المشار إليه بخط يتوسطه خط). يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
4. تشغيل وزارة الخارجية
- في Jupyter-Lab ، استخدم قائمة التنقل على الجانب الأيسر للانتقال إلى مجلد التكوينات بالنقر فوق رمز المجلد ثم النقر المزدوج فوق mofa_workflow ، متبوعا بالنقر المزدوج على البرامج النصية والتكوينات. افتح الملف 03_MOFA_Configs.csv بالنقر المزدوج عليه.
- تحقق من إدخالات العمودين configuration_name و mofa_result_name واضبط الإدخالات إذا كان يجب استخدام أسماء بديلة.
ملاحظة: سيتم إلحاق mofa_result_name كملحق ملف لجميع ملفات النتائج التي تم إنشاؤها بناء على وزارة الخارجية. يمكن أن يكون هذا مختلفا عن القيمة configuration_name حيث قد يتم تشغيل إعدادات MOFA مختلفة بنفس بيانات الإدخال (يستخدم هذا البروتوكول MI_v1_MOFA). - أدخل مقدار العوامل التي يجب تقديرها في نموذج MOFA (عمود amount_of_factors ) وحدد ما إذا كان يجب تطبيق الترجيح والقياس (weighting_of_views وأعمدة scale_views ) عن طريق ضبط القيم في الملف.
- احفظ التغييرات بالنقر فوق ملف > حفظ ملف CSV.
- استخدم قائمة التنقل على الجانب الأيسر للانتقال إلى مجلد البرامج النصية بالنقر فوق "البرامج النصية". افتح دفتر الملاحظات 03_Run_MOFA.ipynb بالنقر المزدوج على الملف. قم بتنفيذ البرنامج النصي بالنقر فوق الزر إعادة تشغيل kernel وتشغيل جميع الخلايا في الجزء العلوي والنقر فوق إعادة التشغيل في النافذة المنبثقة.
- انتقل إلى مجلد 03_figures بالنقر المزدوج على الأشكال ثم 03_figures. افتح mofa_result_name المخطط الذي تم إنشاؤه FIG03_Overview_Variance_Decomposition_ وتحقق من نتيجة النموذج (الشكل 7 أ).
- استخدم قائمة التنقل على الجانب الأيسر للانتقال إلى مجلد 03_results الذي تم إنشاؤه بالنقر فوق رمز المجلد ثم النقر المزدوج فوق النتائج و 03_results. تحقق مما إذا كان يتضمن ملف قيمة عامل العينة 03_Factor_Data_'mofa_result_name'.csv وملف وزن عامل الميزة 03_Weight_Data_'mofa_result_name'.csv.
5. تحليل المصب
- تفسير العامل.
- استخدم قائمة التنقل على الجانب الأيسر للانتقال إلى مجلد input_data بالنقر فوق رمز المجلد ، متبوعا بالنقر المزدوج فوق input_data.
- قم بإعداد ملف .csv (Prepared_Sample_Meta_Data.csv) يحتوي على جميع البيانات الوصفية (المتغيرات المشتركة) للعينات التي سيتم تحليلها بالاقتران مع العوامل التي تم إنشاؤها. نسخ الملف إلى المجلد input_data باستخدام السحب والإفلات إسقاط الملف في نظرة عامة على المجلد input_data .
ملاحظة: يجب أن يحتوي على العمود sample_id لمطابقته مع البيانات المستخدمة مسبقا وأعمدة أخرى لكل ميزة يجب تحليلها. - في Jupyter-Lab ، استخدم قائمة التنقل على اليسار للانتقال مرة أخرى إلى مجلد التكوينات بالنقر فوق رمز المجلد ثم النقر المزدوج فوق mofa_workflow ، متبوعا بالبرامج النصية والتكوين. افتح الملف 04_Factor_Analysis.csv بالنقر المزدوج عليه.
- تحقق من أن إدخالات configuration_name و mofa_result_name تحتوي على أسماء التكوين ونتائج MOFA التي سيتم تحليلها في البرنامج النصي وتعديلها إذا لزم الأمر.
- في العمود numeric_covariates ، أضف اسم جميع الأعمدة الرقمية في ملف Prepared_Sample_Meta_Data.csv الذي سيتم التحقيق فيه فيما يتعلق بعوامل MOFA مفصولة بفاصلة (يستخدم هذا البروتوكول CRP ، CK).
- في عمود categorical_covariates ، أضف اسم جميع الأعمدة الفئوية في ملف Prepared_Sample_Meta_Data.csv الذي سيتم التحقيق فيه فيما يتعلق بعوامل MOFA مفصولة بفاصلة (يستخدم هذا البروتوكول القياس).
- احفظ التغييرات بالنقر فوق ملف > حفظ ملف CSV.
- استخدم قائمة التنقل على اليسار للانتقال إلى مجلد "البرامج النصية" بالنقر فوق البرامج النصية. افتح دفتر الملاحظات 04_Downstream_Factor_Analysis.ipynb بالنقر المزدوج عليه. قم بتنفيذ البرنامج النصي بالنقر فوق الزر إعادة تشغيل kernel وتشغيل جميع الخلايا في الجزء العلوي والنقر فوق إعادة التشغيل في النافذة المنبثقة.
- استخدم قائمة التنقل الموجودة على اليسار للانتقال إلى مجلد 04_figures بالنقر المزدوج على الأشكال ثم 04_figures. افتح المؤامرات التي تم إنشاؤها بالنقر المزدوج عليها وتحقق من عوامل الأنماط والارتباطات المثيرة للاهتمام: FIG04_Factor_Association_with_numeric_features_
'mofa_result_name.pdf (الشكل 7 ب). FIG04_Factor_Association_
with_categorical_features_'mofa_result_name.pdf (الشكل 7C). FIG04_Top_Feature_Overview_per_Factor _'mofa_result_name.pdf (الشكل 8 أ).
- تحليل الميزات
- استخدم قائمة التنقل الموجودة على اليسار للانتقال مرة أخرى إلى مجلد التكوينات بالنقر فوق ... ثم النقر المزدوج على التكوينات. افتح الملف 05_Feature_Analysis_Configs.csv بالنقر المزدوج عليه.
- تحقق من أن إدخالات العمودين configuration_name و mofa_result_name تتوافق مع أسماء التكوين ونتائج MOFA التي تم إنشاؤها والتي سيتم استخدامها لتحليل المصب وتعديلها إذا لزم الأمر.
- في عمود العامل ، أضف العامل الذي سيتم رسم أهم الميزات له داخل البرنامج النصي التالي.
- في العمود faceting_variable ، أضف اسم عمود لعمود فئوي في Prepared_Sample_Meta_Data .csv والذي سيتم استخدامه لتجميع العينات في المخطط (يستخدم هذا البروتوكول القياس)
- احفظ التغييرات بالنقر فوق ملف > حفظ ملف CSV.
- استخدم قائمة التنقل الموجودة على اليسار للانتقال إلى مجلد البرامج النصية بالنقر فوق البرامج النصية. افتح دفتر الملاحظات 05_Downstream_Investigate_Features_Heatmap.ipynb بالنقر المزدوج عليه. قم بتنفيذ البرنامج النصي بالنقر فوق إعادة تشغيل النواة وتشغيل جميع الخلايا زر في الأعلى ، والنقر فوق إعادة التشغيل في النافذة المنبثقة.
- استخدم قائمة التنقل الموجودة على اليسار للانتقال إلى مجلد 05_figures بالنقر المزدوج أولا على الأشكال ثم على 05_figures. افتح وتحقق من المؤامرة التي تم إنشاؤها FIG05_Heatmap_Feature_Overview__ "mofa_result_name".pdf بالنقر المزدوج على الملف (الشكل 8 ب).
ملاحظة: اعتمادا على مقدار الميزات التي سيتم عرضها في المخطط ، قد يكون من الضروري ضبط المعلمات plot_width plot_height داخل 05_Feature_Analysis_Configs.csv وإعادة تشغيل البرنامج النصي للتأكد من أن كل شيء يناسب المؤامرة.
- تحليل المسار
- استخدم قائمة التنقل على الجانب الأيسر للانتقال إلى مجلد input_data بالنقر فوق رمز المجلد ، متبوعا بالنقر المزدوج فوق input_data.
- قم بإعداد ملف .csv (Prepared_Pathway_Data.csv) يحتوي على قائمة المسارات التي سيتم اختبارها للتخصيب. نسخ الملف إلى المجلد input_data باستخدام السحب والإفلات إسقاط الملف في نظرة عامة على المجلد input_data.
ملاحظة: يجب أن يحتوي على ثلاثة أعمدة: ID (معرف فريد للمسار) ، الجين (الجينات التي يعطيها اسم الجين (SYMBOL) الذي ينتمي إلى المسار ، صف واحد لكل جين) ، pathway_name (اسم / وصف نصي للمسارات). - في جلسة Jupyter-Lab ، استخدم قائمة التنقل على اليسار للانتقال إلى مجلد التكوينات بالنقر فوق ... ثم النقر المزدوج على التكوينات. افتح الملف 06_Pathway_Configs.csv بالنقر المزدوج عليه.
- تحقق من إدخال عمود mofa_result_name وتأكد من أنه يتوافق مع اسم نتيجة MOFA التي تم إنشاؤها والتي سيتم استخدامها لحساب إثراء المسار.
- تحقق من الإدخال في عمود الأنواع وقم بإزالة الإدخالات الموجودة داخل عمود الأنواع التي لا تحتوي على ميزات تطابق عمود الجينات في ملف Prepared_Pathway_Data.csv .
ملاحظة: بشكل افتراضي يتم إضافة كافة طرق العرض المختلفة التي تم استخدامها داخل نموذج MOFA إلى هذا الملف أثناء تنفيذ سير العمل. في حالة وجود طرق عرض لا تحتوي على ميزات تتطابق مع مسار واحد على الأقل ، فيجب إزالتها ؛ خلاف ذلك ، سوف يفشل التنفيذ. مثال على ذلك هو أن ملف المسار يحتوي فقط على تعليقات توضيحية للمسار للجينات ، ولكن هناك طريقة عرض تحتوي على أسماء البروتين. - احفظ التغييرات بالنقر فوق ملف > حفظ ملف CSV.
- استخدم قائمة التنقل للانتقال إلى مجلد البرامج النصية بالنقر فوق البرامج النصية. افتح دفتر الملاحظات 06_Downstream_Pathways.ipynb بالنقر المزدوج عليه. قم بتنفيذ البرنامج النصي بالنقر فوق إعادة تشغيل kernel وتشغيل جميع الخلايا زر في الجزء العلوي ، والنقر فوق إعادة التشغيل في النافذة المنبثقة.
- استخدم قائمة التنقل الموجودة على اليسار للانتقال إلى مجلد 06_figures بالنقر المزدوج أولا على الأشكال ثم 06_figures. افتح FIG06_Pathways_and_Genes_ المؤامرة التي تم إنشاؤها mofa_result_name بالنقر المزدوج عليها وتحقق من المسارات المرئية (الشكل 8C).
ملاحظة: يمكن تكوين كيفية تحديد المسارات المرئية عبر ملف التكوين. لمزيد من التفاصيل ، راجع وثائق المعلمات. - استخدم قائمة التنقل على الجانب الأيسر للانتقال إلى مجلد 06_results الذي تم إنشاؤه بالنقر فوق رمز المجلد ثم النقر المزدوج فوق النتائج و 06_results. تحقق مما إذا كان يتضمن الملف بما في ذلك المسارات المخصبة 06_Pathway_enrichment__'mofa_result_name'.
6. مقارنة التكوينات والإصدارات المختلفة (الشكل التكميلي 1 ، الشكل التكميلي 2 ، الشكل التكميلي 3 ، الشكل التكميلي 4)
- لمقارنة تأثير استخدام معلمات / تكوينات مختلفة خلال سير العمل ، أعد تشغيل الأقسام من 3 إلى 5 ، وتعديل المعلمات في ملفات التكوين واستخدام معرفات configuration_name و mofa_result_name مختلفة.
ملاحظة: سيتم تخزين النتائج الجديدة بهذه الأسماء لاستخدامها في مقارنة عمليات التشغيل المختلفة. - في Jupyter-Lab ، استخدم قائمة التنقل على اليسار للانتقال إلى مجلد التكوينات . افتح الملف 07_Comparison_Configs.csv بالنقر المزدوج عليه.
- في العمود mofa_result_name ، أضف أسماء جميع عمليات تشغيل MOFA السابقة التي ستتم مقارنتها (صف واحد لكل اسم / تكوين ، على سبيل المثال ، MI_v1_MOFA ، MI_v2_MOFA).
- في العمود compare_factors ، أضف العوامل التي ستتم مقارنتها بين النماذج. بشكل افتراضي ، هو العامل 1 ، العامل 2 ، العامل 3. (الشكل التكميلي 2 ألف).
ملاحظة: في هذا البرنامج النصي ، ستتم مقارنة قيم الميزات والعوامل للنماذج المختلفة من خلال ربطها. يعمل هذا فقط مع الطرز التي تستند إلى نفس العينات (المشار إليها ب sample_id) ونفس مجموعة الميزات. في حالة عدم تطابق العينات أو الميزات بين الإصدارات المقارنة ، سيتم استبعادها من المقارنة. - احفظ التغييرات بالنقر فوق ملف > حفظ ملف CSV.
- استخدم القائمة الموجودة على اليسار للانتقال إلى مجلد البرامج النصية بالنقر فوق البرامج النصية. افتح دفتر الملاحظات 07_Compare_Models.ipynb بالنقر المزدوج عليه. قم بتنفيذ البرنامج النصي بالنقر فوق الزر إعادة تشغيل kernel وتشغيل جميع الخلايا والنقر فوق إعادة التشغيل في النافذة المنبثقة.
- استخدم القائمة الموجودة على اليسار للانتقال إلى مجلد 06_figures بالنقر المزدوج أولا على الأشكال ثم 06_figures. افتح المؤامرات التي تم إنشاؤها بالنقر المزدوج على الملفات لتحليل تشابه الإصدارات المختلفة:
FIG07_Variance_Model_Comparison.pdf (الشكل التكميلي 2 باء)
FIG07_Factor_Correlations.pdf
(الشكل التكميلي 2 جيم)
FIG07_Feature_Correlations.pdf
(الشكل التكميلي 3 ج)
7. توسيع سير العمل: إضافة معلمات وتكوينات أخرى
ملاحظة: إلى جانب المعلمات القابلة للتكوين حاليا في ملفات التكوين، قد يتم تضمين تعديلات أخرى في التعليمات البرمجية أو معلمات أخرى. على سبيل المثال ، يقدم نموذج MOFA نفسه العديد من معلمات التدريب الأخرى17 التي يمكن تعديلها مباشرة في الكود أو جعلها قابلة للتعديل عبر ملفات التكوين. سيحدد القسم التالي من البروتوكول مثالا على كيفية القيام بذلك لمعايير التدريب النموذجية الإضافية لوزارة الخارجية. لهذا الجزء ، مطلوب معرفة برمجة R.
- في Jupyter-Lab ، استخدم قائمة التنقل على اليسار للانتقال إلى مجلد البرامج النصية . افتح دفتر الملاحظات 03_Run_MOFA.ipynb بالنقر المزدوج عليه.
- انقر فوق علامة التبويب جدول المحتويات على الجانب الأيسر ثم انتقل إلى القسم الفرعي 4.3 تعيين خيارات تدريب وزارة الخارجية وتشغيل نموذج التدريب بالنقر فوقه. قم بالتمرير لأسفل لرؤية الإخراج المطبوع لنموذج MOFA للمعلمات القابلة للتكوين في دفتر الملاحظات.
- ضمن حلقة R for في الكود أسفل العنوان ، يتم تعيين جميع بيانات MOFA والنموذج وخيارات التدريب. أسفل السطر model_opts دولار num_factors = mofa_configs دولار amount_of_factors [i] ، أضف سطرا آخر بالرمز أدناه
model_opts $ الاحتمالات ['data_type'] = 'poisson'.
ملاحظة: سيؤدي هذا إلى تغيير التوزيع الذي يتخذه النموذج كإدخال للعرض المحدد بواسطة الاسم data_type لكافة عمليات تشغيل MOFA. عند تحديد poisson لنوع بيانات ، سيتم تشغيل النموذج فقط عندما تكون ميزات نوع البيانات هذا أعدادا صحيحة (على سبيل المثال ، أعداد القراءة من RNA-seq). للحصول على مزيد من المعلومات حول بيانات وزارة الخارجية والتدريب وخيارات النموذج ، يمكن للمرء أيضا الرجوع إلى دروس ووثائق وزارة الخارجية17. - احفظ التغييرات في دفتر الملاحظات بالنقر فوق الزر حفظ في الأعلى.
- لتسليم معلمات جديدة عبر ملفات تكوين .csv ، استخدم التنقل على الجانب الأيسر للانتقال إلى مجلد التكوينات بالنقر المزدوج فوق التكوينات وفتح 03_MOFA_Configs.csv الملف بالنقر المزدوج.
- أضف عمودا جديدا يحدد اسم المعلمة ، على سبيل المثال ، number_iterations وأدخل قيمة ، على سبيل المثال ، 1000. احفظ التغييرات بالنقر فوق ملف > حفظ ملف CSV.
- استخدم قائمة التنقل للتنقل في مجلد البرامج النصية بالنقر فوق البرامج النصية. افتح دفتر الملاحظات 03_Run_Mofa.ipynb بالنقر المزدوج عليه. انقر فوق علامة التبويب جدول المحتويات على الجانب الأيسر ثم انتقل إلى القسم الفرعي 4.3 تعيين خيارات تدريب وزارة الخارجية وتشغيل نموذج التدريب بالنقر فوقه.
- استبدل السطر train_opts$maxiter = 50000 ب train_opts$maxiter = mofa_configs$column_name[i] (عندما يكون اسم العمود المضاف number_of_iterations يكون train_opts$maxiter = mofa_configs$number_of_iterations[i]).
ملاحظة: تتم قراءة ملف التكوين 03_MOFA_Configs.csv في دفتر الملاحظات هذا في بداية دفتر الملاحظات هذا (القسم الفرعي: تكوينات ومعلمات المتطلبات الأساسية) ك mofa_config data.frame في جلسة العمل ، وبالتالي ، في هذا السطر من التعليمات البرمجية ، تتم الإشارة إلى هذا الكائن والعمود الذي تم إنشاؤه حديثا المقابل. نظرا لأنه يمكن تشغيل تكوينات متعددة في نفس الوقت ، يحدد i صف data.frame حيث يتم تشغيل تقدير النموذج في حلقة for عبر جميع الصفوف المختلفة في ملف .csv . مبدأ القراءة في ملف التكوين في بداية دفتر الملاحظات في قسم "تكوينات ومعلمات المتطلبات الأساسية" هو نفسه لجميع دفاتر الملاحظات ، ويمكن إجراء المزيد من التعديلات على هذا النحو. - احفظ التغييرات في دفتر الملاحظات بالنقر فوق الزر حفظ .
النتائج
بعد التنفيذ الناجح لسير العمل ، يتم إنشاء العديد من الجداول والأشكال كما هو موضح في الشكل 2. يتم وضع الأشكال في مجلد / الأشكال (الشكل 6 ، الشكل 7 ، الشكل 8 ، الشكل التكميلي 1 ، الشكل التكم?...
Discussion
باستخدام البروتوكول المحدد ، يتم تقديم سير عمل معياري وقابل للتمديد قائم على دفتر ملاحظات Jupyter يمكن استخدامه لاستكشاف مجموعة بيانات معقدة متعددة الأوميكس بسرعة. تتكون الأجزاء الرئيسية من تدفق العمل من جزء المعالجة المسبقة وتنسيق البيانات (تقديم خطوات قياسية مختلفة لتص...
Disclosures
يعلن أصحاب البلاغ عدم وجود تضارب في المصالح.
Acknowledgements
يتم دعم C.L. من قبل جمعية هيلمهولتز في إطار مدرسة الأبحاث المشتركة "مدرسة ميونيخ لعلوم البيانات - MUDS".
Materials
| Name | Company | Catalog Number | Comments |
| Apptainer | NA | NA | https://apptainer.org/docs/admin/main/installation.html |
| Compute server or workstation or cloud (Linux, Mac or Windows environment). Depending on the size of the different input datasets we recommend running the workflow on a suitable machine (in our setting we use: 16 CPU, 64GB Memory) | Any manufacturer | 16 CPU, 64GB Memory | Large Memory is only required for the processing of the raw single cell data. After preprocessing the later analysis steps can also be performed on regular desktop or laptop computers |
| git | NA | NA | https://git-scm.com/book/en/v2/Getting-Started-Installing-Git |
| GitHub | GitHub | NA | https://github.com/heiniglab/mofa_workflow |
References
- Lähnemann, D., et al. Eleven grand challenges in single-cell data science. Genome Biol. 21 (1), 31 (2020).
- Colomé-Tatché, M., Theis, F. J. Statistical single cell multi-omics integration. Curr Opin Syst Biol. 7, 54-59 (2018).
- Hawe, J., Theis, F., Heinig, M. Inferring interaction networks from multi-omics data. Front Genet. 10, 535 (2019).
- Hawe, J. S., et al. Network reconstruction for trans acting genetic loci using multi-omics data and prior information. Genome Med. 14 (1), 125 (2022).
- Koh, H. W. L., Fermin, D., Vogel, C., Choi, K. P., Ewing, R. M., Choi, H. iOmicsPASS: network-based integration of multiomics data for predictive subnetwork discovery. NPJ Syst Biol Appl. 5, 22 (2019).
- Ogris, C., Hu, Y., Arloth, J., Müller, N. S. Versatile knowledge guided network inference method for prioritizing key regulatory factors in multi-omics data. Sci Rep. 11, 6806 (2021).
- Lee, C., vander Schaar, M. A variational information bottleneck approach to multi-omics data integration. Proceedings of The 24th International Conference on Artificial Intelligence and Statistics. 130, 1513-1521 (2021).
- Singh, A., et al. DIABLO: an integrative approach for identifying key molecular drivers from multi-omics assays. Bioinformatics. 35 (17), 3055-3062 (2019).
- Argelaguet, R., et al. Multi-omics factor analysis-a framework for unsupervised integration of multi-omics data sets. Mol Syst Biol. 14 (6), e8124 (2018).
- Cantini, L., et al. Benchmarking joint multi-omics dimensionality reduction approaches for the study of cancer. Nature Commun. 12 (1), 124 (2021).
- Pekayvaz, K., et al. Multiomic analyses uncover immunological signatures in acute and chronic coronary syndromes. Nature Medicine. 30 (6), 1696-1710 (2024).
- Swirski, F. K., Nahrendorf, M. Cardioimmunology: the immune system in cardiac homeostasis and disease. Nat Rev Immunol. 18 (12), 733-744 (2018).
- Janjic, A., et al. Prime-seq, efficient and powerful bulk RNA sequencing. Genome Biol. 23 (1), 88 (2022).
- Wolf, F. A., Angerer, P., Theis, F. J. SCANPY: large-scale single-cell gene expression data analysis. Genome Biol. 19 (1), 15 (2018).
- Cao, Y., et al. Integrated analysis of multimodal single-cell data with structural similarity. Nucleic Acids Res. 50 (21), e121 (2022).
- . Get Started - JupyterLab 4.1.0a4 documentation Available from: https://jupyterlab.readthedocs.io/en/latest/getting_started/overview.html (2024)
- . MOFA2: training a model in R Available from: https://raw.githack.com/bioFAM/MOFA2_tutorials/master/R_tutorials/getting_started_R.html (2020)
- De Silva, D., et al. Robust T cell activation requires an eIF3-driven burst in T cell receptor translation. eLife. 10, e74272 (2021).
- Li, G., Liang, X., Lotze, M. HMGB1: The central cytokine for all lymphoid cells. Front Immunol. 4, 68 (2013).
- Jassal, B., et al. The reactome pathway knowledgebase. Nucleic Acids Res. 48 (D1), D498-D503 (2020).
- Argelaguet, R., et al. MOFA+: a statistical framework for comprehensive integration of multimodal single-cell data. Genome Biol. 21 (1), 111 (2020).
- Velten, B., et al. Identifying temporal and spatial patterns of variation from multimodal data using MEFISTO. Nat Methods. 19 (2), 179-186 (2022).
- Qoku, A., Buettner, F. Encoding domain knowledge in multi-view latent variable models: A Bayesian approach with structured sparsity. Proceedings of The 26th International Conference on Artificial Intelligence and Statistics. 206, 11545-11562 (2022).
- Multi-Omics Factor Analysis. MOFA Available from: https://biofam.github.io/MOFA2/ (2024)
- Mitchel, J., et al. Tensor decomposition reveals coordinated multicellular patterns of transcriptional variation that distinguish and stratify disease individuals. bioRxiv. , (2023).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved