A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
استخراج الحمض النووي الريبي أحادي الأنبوب أحادي لمقارنة مستويات النسخ النسبية عبر qRT-PCR في نموذج Hypsibius بطيئات المشية
In This Article
Summary
يقدم هذا العمل طريقة لاستخراج الحمض النووي الريبي السريع وطريقة مقارنة مستوى النسخ لتحليل التعبير الجيني في بطيئات المشية Hypsibius exemplaris. باستخدام التحلل الفيزيائي ، تتطلب هذه الطريقة عالية الإنتاجية بطيئات المشية واحدة كمادة أولية وتؤدي إلى إنتاج قوي ل (كدنا) لتفاعل البوليميراز المتسلسل للنسخ العكسي الكمي (qRT-PCR).
Abstract
بطيئات المشية Hypsibius exemplaris هو كائن حي نموذجي ناشئ يشتهر بقدرته على البقاء على قيد الحياة في الظروف البيئية المتطرفة. لاستكشاف الآليات الجزيئية والأساس الجيني لمثل هذا التحمل المتطرف ، تعتمد العديد من الدراسات على تسلسل الحمض النووي الريبي (RNA-seq) ، والذي يمكن إجراؤه على مجموعات تتراوح من مجموعات كبيرة إلى فردية. يتم استخدام تفاعل البوليميراز المتسلسل للنسخ العكسي (RT-PCR) وتداخل الحمض النووي الريبي (RNAi) لاحقا لتأكيد نتائج تسلسل الحمض النووي الريبي وتقييم المتطلبات الجينية للجينات المرشحة ، على التوالي. تتطلب مثل هذه الدراسات طريقة فعالة ودقيقة وبأسعار معقولة لاستخراج الحمض النووي الريبي وقياس مستويات النسخ النسبية بواسطة RT-PCR الكمي (qRT-PCR). يقدم هذا العمل طريقة فعالة لاستخراج الحمض النووي الريبي أحادي الأنبوب أحادي الأنبوب (STST) لا تعزل الحمض النووي الريبي بشكل موثوق عن المشية الفردية فحسب ، بل تقلل أيضا من الوقت والتكلفة المطلوبين لكل عملية استخراج. تنتج طريقة استخراج الحمض النووي الريبي هذه كميات من (كدنا) يمكن استخدامها لتضخيم واكتشاف نصوص متعددة عن طريق تفاعل البوليميراز المتسلسل الكمي (qRT-PCR). يتم التحقق من صحة الطريقة من خلال تحليل التغيرات الديناميكية في التعبير عن الجينات التي تشفر بروتينين منظمين بالصدمة الحرارية ، بروتين الصدمة الحرارية 70 β2 (HSP70 β2) وبروتين الصدمة الحرارية 90α (HSP90α) ، مما يجعل من الممكن تقييم مستويات التعبير النسبية لديهم لدى الأفراد المعرضين للحرارة باستخدام qRT-PCR. تكمل STST بشكل فعال طرق استخراج الحمض النووي الريبي السائبة والمفردة الحالية ، مما يسمح بفحص سريع وبأسعار معقولة لمستويات النسخ الفردية بطيئات المشية بواسطة qRT-PCR.
Introduction
بطيئات المشية هي صغيرة متعددة الخلايا تشتهر بقدرتها على البقاء على قيد الحياة في الظروف القاسية المميتة لمعظم أشكال الحياةالأخرى 1. على سبيل المثال ، يمكن لهذه البقاء على قيد الحياة ما يقرب من 1000 مرة من جرعة الإشعاع المؤين المميتة للبشر2،3،4،5،6،7،8،9،10 ، الجفاف شبه الكامل11،12،13،14،15 ، التجميد في حالة عدم وجود إضافة المواد الواقية من التبريد16،17،18 ، وفي حالتها المجففة ، حتى فراغ الفضاء19،20. نظرا لقدرتها الفريدة على البقاء في البيئات القاسية ، أصبحت هذه نماذج أساسية لفهم التحمل المتطرف في الكائنات المعقدة متعددة الخلايا1،21،22،23.
ظل التلاعب الجيني المستقر لهذه الرائعة ، بما في ذلك التحول الجيني وتعديل الجينات الجرثومية ، بعيد المنال حتى وقت قريب24،25. على هذا النحو ، يتم إجراء معظم التجارب للكشف عن الآليات الجزيئية للتحمل المتطرف من خلال التنميط النسخي عبر تسلسل الحمض النووي الريبي. توجد العديد من مجموعات بيانات تسلسل الحمض النووي الريبي القيمة والغنية بالمعلومات لبطيئات المشية في ظل ظروف قاسية مختلفة ، تتراوح من الإشعاع8،9،26،27،28 ، والإجهاد الحراري29 ، وإجهاد التجميد12 ، والجفاف27،30،31،32،33. استخدمت بعض هذه الدراسات طرق استخراج وتنقية الحمض النووي الريبي السائبة لإلقاء الضوء على فهمنا الجزيئي للتحمل الأقصى ومع ذلك ، فإن الاستخراج السائب لنصوص الحمض النووي الريبي من العديد من يمنع تحليل التباين في التعبير الجيني بين الأفراد ، وبالتالي يفقد الثراء المحتمل لمجموعات البيانات الأكثر دقة. الأهم من ذلك ، غالبا ما تحلل هذه الدراسات مجموعات غير متجانسة من التي تشمل كلا من التي تنجو من الضغوطات البيئية وتلك التي لا تنجو منها. على هذا النحو ، يتم الخلط بين هذه الدراسات من خلال متوسط بيانات التعبير من حالات استجابة متعددة وربما مختلفة بشكل كبير. لمعالجة هذه المشكلة ، طور Arakawa et al. ، 201634 خط أنابيب أنيق منخفض المدخلات للحمض النووي الريبي يطبق مجموعة استخراج الحمض النووي الريبي متبوعة بخطوة تضخيم PCR خطية باستخدام34،35،36 أو30،37،38مفردا كمدخلات. كانت هذه الدراسات أساسية لفهمنا للتحمل الرشي المشي22. ومن المثير للاهتمام ، أن هذا البروتوكول قد تم تطبيقه أيضا على qRT-PCR باستخدام سبعة كمادةأولية 24.
في معظم الكائنات الحية النموذجية ، بعد تحديد الأهداف المحتملة عبر تسلسل الحمض النووي الريبي ، يتم بعد ذلك إجراء qRT-PCR لتأكيد التغييرات النسخية التي تم تحديدها بواسطة تسلسل الحمض النووي الريبي وتقييم المسار الزمني للتعبير عن الجينات المرشحة بطريقة عالية الدقة. لاختبار وظيفة الجينات المحددة ، غالبا ما يتبع مثل هذه الدراسات ضربة قاضية بوساطة RNAi للأهداف الجزيئية39،40 وتحليل القدرة المتطرفة12،41. عادة ما يتم تأكيد فعالية كل ضربة قاضية من RNAi بواسطة qRT-PCR من خلال المراقبة المباشرة للانخفاض في وفرة النسخ. ومع ذلك ، فإن RNAi هي عملية كثيفة العمالة في بطيئات المشية حيث يجب تسليم كل dsRNA عن طريق الحقن المجهري اليدوي للأفراد39،40. نظرا لطبيعة الإنتاجية المنخفضة لهذه الاستراتيجية ، فإن طريقة استخراج الحمض النووي الريبي السريع ومنخفضة التكلفة التي تم تكييفها مع qRT-PCR من مفردة ستكون ذات قيمة عالية لأبحاث بطيئات المشية. على الرغم من أن الطرق السابقة قد تم تطويرها لاستخراج الحمض النووي الريبي من بطيئات المشية المفردة ، إلا أن هذه البروتوكولات لم تجمع بين استخراجها مع qRT-PCR ، وبدلا من ذلك تعتمد على الطرق القائمة على الكثافةالضوئية 12،40،41. بدافع من هذه التحديات ، سعينا إلى تطوير بروتوكول ينتج الحمض النووي الريبي بشكل موثوق من حيث الكمية والنوعية والذي يمكن استخدامه ل qRT-PCR من H. exemplaris الفردي.
مقتبس من بروتوكول استخراج الحمض النووي الريبي أحادي الذي تم تطويره ل Caenorhabditis elegans42 ، تم تحسين STST ل H. exemplaris. تتكون طريقة الاستخراج من ست خطوات سريعة للتجميد والذوبان ، مما يؤدي إلى تعطيل البشرة جسديا ، مما يسمح باستخراج الحمض النووي الريبي وتخليق الحمض النووي (كدنا) اللاحق. تقلل طريقة STST من وقت الاستخراج بأكثر من 24 ضعفا مقارنة بطرق استخراج الحمض النووي الريبي السائبة ، كما هو موضح من قبل Boothby ، 201843 ، وبنسبة 30٪ مقارنة بمجموعات استخراج الحمض النووي الريبي بطيئات المشية المفردة ، كما هو موضح بواسطة Arakawa et al. ، 201634. علاوة على ذلك ، تم تقليل عدد تفاعلات العينة والمجرب من 5 إلى 1 فقط مقارنة بمستحضرات مجموعة استخراج الحمض النووي الريبي ، مما يقلل من خطر التلوث بالريبونولياز الخارجية. عند الاستعلام عن الجينات المعبر عنها بشكل كبير ، تنتج طريقة STST (كدنا) كافيا ل 25 تفاعلا كميا RT-PCR لكل بطيئات المشية ، مما يتطلب فقط 1 ميكرولتر من إجمالي حجم 25 ميكرولتر (كدنا) لكل تفاعل. ومع ذلك ، يجب تحديد تركيزات القالب تجريبيا للنصوص المنخفضة الوفرة.
تم تقييم فعالية طريقة STST لتحليل التغيرات الديناميكية في التعبير الجيني من خلال التحقيق في التعبير التفاضلي للجينات التي تشفر بروتين الصدمة الحرارية -90α (HSP90α) وبروتين الصدمة الحرارية 70β2 (HSP70β2) استجابة لصدمة حرارية قصيرة المدى عند 35 درجة مئوية لمدة 20 دقيقة. يتم تنظيم كل من HSP70β2 و HSP90α في معظم الكائنات حقيقية النواة بسرعة بعد التعرض للصدمة الحرارية على المدى القصير (20 دقيقة) 42. التحليل في H. exemplaris كشف أن كلا من الحمض النووي الريبي المشفر HSP70β2 و HSP90α المستخرج من بطيئات المشية المفردة المعالجة حراريا أظهر زيادات ذات دلالة إحصائية في التعبير بعد التعرض للحرارة على المدى القصير. توضح هذه النتائج أنه يمكن استخدام بروتوكول STST لتحليل التغيرات الديناميكية في التعبير الجيني في الفردية بمرور الوقت.
يجب أن تكمل طريقة استخراج STST الطرق التجريبية الحالية مثل RNA-seq من خلال تسهيل استخراج الحمض النووي الريبي السريع وغير المكلف والمقارنة اللاحقة لمستويات النسخ بواسطة qRT-PCR. ستكون هذه الطريقة ذات قيمة أيضا لتقييم كفاءة واختراق الحمض النووي الريبي في الأفراد المحقونين يدويا من الناحية الكمية أكثر من الكثافة البصرية وحدها. أخيرا ، نظرا لتراكبها الجلدية وخصائصها الفيزيائية المتشابهة ، فمن المحتمل أن تكون هذه الطريقة فعالة أيضا في تحليل التعبير الجيني في أنواع بطيئات المشيةالأخرى 44.
Access restricted. Please log in or start a trial to view this content.
Protocol
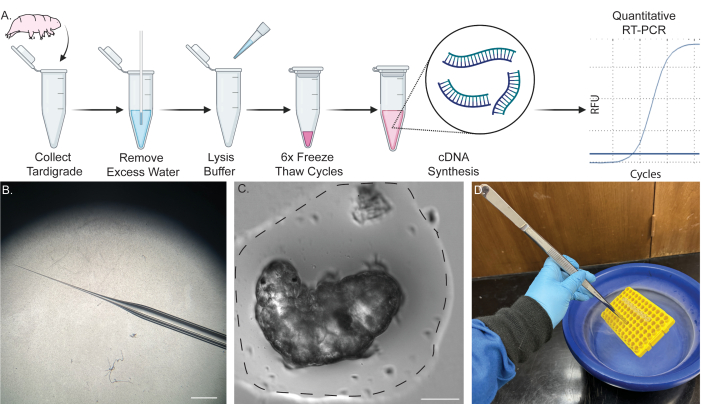
الشكل 1: خط أنابيب أحادي الأنبوب لاستخراج الحمض النووي الريبي من بطيئات مشية واحدة. (أ) مخطط يوضح بروتوكول استخراج الحمض النووي الريبي من بطيئات المشية واحدة ، بما في ذلك ست دورات تجميد وذوبان الجليد وتخليق (كدنا) اللاحق. يمكن استخدام العينات لاحقا لاختبار RT-PCR و qRT-PCR. (ب) صورة للتفتق بالماصة الدقيقة المستخدمة لإزالة الماء. شريط المقياس: 2 مم. (C) صورة ميدانية ساطعة لبطيئات المشية في حجم صغير من الماء (خط منقط). إن إزالة معظم المياه إلى الحد الموضح مطلوبة للاستخراج الناجح وتمنع تخفيف محلول التحلل. شريط المقياس: 50 ميكرومتر. (د) صورة توضح غمر العينات في النيتروجين السائل باستخدام ملقط طويل لتجميد العينات بسرعة وإذابة الثلج بأمان. تم إنشاء بعض المحتوى في BioRender. كيرك ، م. (2022) BioRender.com/d93s511 الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
ملاحظة: يوضح الشكل 1 أ مخططا للإجراء. للحصول على إجراءات مفصلة لزراعة بطيئات المشية والطحالب ، راجع التقارير المنشورةسابقا 45،46،47.
1. تعقيم مياه الينابيع
- صب 2 لتر من مياه الينابيع من إبريق ماء سعة 5 جالون (انظر جدول المواد للحصول على التفاصيل) في زجاجة زجاجية آمنة للأوتوكلاف سعة 2 لتر.
- ضع الغطاء على الزجاجة الآمنة للأوتوكلاف وأغلقها بكمية صغيرة من شريط الأوتوكلاف. لا تشد الزجاجة. ضع الغطاء في الأعلى.
- قم بتعقيم مياه الينابيع لمدة 50 دقيقة في دورة رطبة بدون خطوة تجفيف.
- اترك الماء يصل إلى درجة حرارة الغرفة (RT) ، وأغلق الغطاء بإحكام قبل تخزينه في RT.
2. سحب الماصة الدقيقة الزجاجية (مع مجتذب الماصة)
- قم بتثبيت ماصة زجاجية دقيقة (OD: 1 مم، المعرف: 0.58 مم، الطول: 10 سم) على مجتذب ماصة دقيقة. تجنب ملامسة خيوط التسخين ، لأن ذلك سيغير شكل الماصة ويتلف الفتيل.
- حدد سحب الماصة تجريبيا لكل مجتذب خيوط وماصة. ومع ذلك ، لتكون بمثابة نقطة انطلاق للتحسين ، استخدم 78 درجة مئوية وخطوة سحب واحدة بوزن سحب يبلغ 182.2 جم.
- اسمح للفتيل بالتسخين والجاذبية لفصل الماصة الدقيقة الزجاجية إلى ماصتين زجاجيتين بنقاط حادة (الشكل 1 ب).
- قم بتخزين هذه الماصات الدقيقة الزجاجية المسحوبة في طبق بتري مغلق مقاس 100 مم مع الشمع أو الطين لتثبيتها في مكانها ومنع الأطراف الحادة من الانكسار.
3. سحب الماصة الدقيقة الزجاجية (بدون مجتذب الماصة)
- أشعل موقد بنسن أو أي مصدر لهب آخر يتم التحكم فيه على إعداد منخفض.
- خذ ماصة زجاجية بطرف واحد في كل يد.
- أمسك مركز الماصة الدقيقة الزجاجية فوق اللهب حتى يبدأ الزجاج في الذوبان. ثم افصل الطرفين بسرعة. سيؤدي ذلك إلى إنشاء طرفين حادين دقيقين للغاية.
- كسر الحافة برفق باستخدام زوج من الملقط الدقيق المعقم.
- قم بتخزين هذه الماصات الدقيقة الزجاجية المسحوبة في طبق بتري مغلق مقاس 100 مم مع الشمع أو الطين لتثبيتها في مكانها ومنع الأطراف الحادة من الانكسار.
4. استخراج الحمض النووي الريبي
- احصل على 0.5 لتر من النيتروجين السائل في حاوية آمنة للتبريد.
تنبيه: النيتروجين السائل مبرد وقد يسبب حروقا إذا تعرض للجلد أو العينين. عند المناولة ، استخدم الملابس الواقية ، ونظارات الرش ، وقفازات النتريل ، وقفازات التبريد ، ومعطف المختبر ، والأحذية المغلقة. تأكد من أن الحاوية آمنة للنيتروجين السائل قبل نقل السائل. قد يكون استخدام حمام جليدي جاف بالإيثانول لهذه الخطوة ممكنا أيضا. - اصنع مزيجا رئيسيا لتخليق (كدنا): محلول 10 ميكرولتر يحتوي على 1 ميكرولتر من التمهيدي السداسي العشوائي ، و 2 ميكرولتر من DNase ، و 4 ميكرولتر من 5x RT Buffer ، و 1 ميكرولتر من مزيج الإنزيم ، و 1 ميكرولتر من H2O ، و 1 ميكرولتر من 10 ملي مولار dNTPs. قم بتخزين هذا الحل على الثلج.
- تحضير محلول تحلل بطيئات المشية (5 ملي مولار تريس (الرقم الهيدروجيني = 8) ، 0.5٪ (حجم / حجم) منظف 1 ، 0.5٪ (حجم / حجم) منظف 2 ، 0.25 ملي مولار EDTA في ماء معقم خال من النوكلياز).
ملاحظة: يمكن تخزين هذا المحلول على سطح الطاولة لمدة 6 أشهر. ومع ذلك ، حافظ على العقم وتجنب المصادر المحتملة الملوثة بالحمض النووي الريبي. - Aliquot ما يكفي من المخزن المؤقت للتحلل للاستخراج (2 ميكرولتر / بطيئات المشية).
- أضف مثبط RNAse إلى محلول المخزن المؤقت لتحلل بطيئات المشية إلى تركيز نهائي يبلغ 4 U / μL.
- قم بتدوير المحلول في RT على جهاز طرد مركزي أعلى الطاولة بسرعة 2000 × جم لمدة 5 ثوان قبل تخزين المحلول على الجليد.
- قم بإزالة أكبر عدد ممكن من بطيئات المشية حسب الحاجة للتجربة من المزرعة باستخدام ماصة P1000 معقمة ذات رؤوس مرشح وضعها في طبق بتري معقم مقاس 35 مم.
ملاحظة: يمكن معالجة أي عدد من بطيئات المشية بهذه الطريقة. عادة ، تتم معالجة ثلاث بطيئات المشية لكل حالة للاستخراج. - اغسل بطيئات المشية ثلاث مرات باستخدام 1 مل من مياه الينابيع المعقمة المعقمة وماصة P1000 المعقمة ذات الرؤوس المرشحية. يساعد ماصتها ببطء لأعلى ولأسفل على إزالة ملوثات الطحالب.
- باستخدام مجهر تشريح بتكبير 25x إلى 50x، انقل بطيئات المشية واحدة من هذه المزرعة المغسولة إلى طبق بتري معقم جديد مقاس 35 مم باستخدام ماصة P10 معقمة ذات رؤوس مرشح.
- استخدم ماصة P200 معقمة ذات رؤوس مرشح لغسل بطيئات المشية المفردة في 100 ميكرولتر من الماء المعقم الخالي من النوكلياز.
ملاحظة: تستخدم خطوة الغسيل هذه لإزالة الملوثات بشكل أكبر ، بما في ذلك الريبونوكليازات. - انقل بطيئات المشية المغسولة إلى قاع أنبوب PCR نظيف ومعقم في 1-2 ميكرولتر من الماء المعقم الخالي من النوكلياز باستخدام ماصة P10 معقمة ذات رؤوس مرشح معقمة ، مع التأكد بعناية من عدم التصاق بالمشية بجانب الحافة.
- تصور بطيئات المشية تحت مجهر تشريح بتكبير 25x.
- لتسهيل إزالة الماء من الماء ، اكسر طرف الماصة الدقيقة الزجاجية المسحوبة برفق خارج الأنبوب. تأكد من أن التجويف كبير بما يكفي لسحب الماء ولكن ليس بطيئات المشية.
- باستخدام الحركة الشعرية لماصة زجاجية مسحوبة ، قم بإزالة الماء حتى يحيط بالحيوان فقاعة صغيرة من الماء يبلغ قطرها طولين تقريبا.
- راقب عملية إزالة المياه عبر نطاق التشريح للتأكد من أن مستوى الماء مناسب وأن بطيية المشية تظل رطبة.
ملاحظة: يقدم الشكل 1C مثالا على كمية المياه المراد إزالتها. هذه خطوة حاسمة. ستحيط فقاعة صغيرة من الماء بالمشية لمنعها من الجفاف ، ولكن يجب إزالة أكبر قدر ممكن من الماء الزائد لمنع تخفيف عازلة التحلل. للحصول على مثال على مستويات المياه المتبقية ، يرجى الرجوع إلى الشكل 1C. - مباشرة بعد إزالة الماء ، أضف 2 ميكرولتر من محلول التحلل المشي إلى قاع الأنبوب ، ودوامة لفترة وجيزة ، وقم بالطرد المركزي للأنبوب عند RT لمدة 5 ثوان عند 2000 × جم على جهاز طرد مركزي منضدي.
- ضع العينات التي تحتوي على بطيئات المشية على الفور في رف أنبوب PCR وتأكد من تثبيتها بإحكام بواسطة الرف.
- أمسك الرف باستخدام زوج من الملقط الخشن الطويل واغمس الرف الذي يحتوي على العينات برفق في النيتروجين السائل حتى يتجمد تماما (الشكل 1 د).
- قم بإزالة الرف من النيتروجين السائل وضعه على الفور على الثلج. اسمح للعينة بالذوبان (يستغرق ~ 45 ثانية إلى 1 دقيقة). راقب العينة كل 15 ثانية عن طريق إزالتها من الجليد وفحصها بشكل واضح. بمجرد أن تصبح العينة شفافة بشكل واضح ، انتقل إلى الخطوة التالية.
- كرر الخطوات 4.18-4.19 خمس مرات أخرى. مطلوب ما مجموعه ست دورات تجميد وذوبان الجليد لتحقيق أقصى قدر من التحلل والاستخراج (الشكل 2 أ ، ب).
- بمجرد اكتمال التجميد والذوبان ، ضع عينات على الجليد وانتقل على الفور إلى الخطوة التالية. لا تجمد العينات في هذه المرحلة للتخزين ، لأن هذا سيقلل من الحمض النووي الريبي المتاح لإعداد (كدنا).
5. تخليق (كدنا)
- أضف 2 ميكرولتر من المزيج الرئيسي لتخليق (كدنا) إلى أنبوب تفاعل البوليميراز المتسلسل الذي يحتوي على محللة بطيئات المشية. قم بنقر الأنبوب لفترة وجيزة وقم بتدويره عند RT عند 2000 × جم لمدة 5 ثوان باستخدام جهاز طرد مركزي منضدي قبل استبدال العينات على الجليد.
- ضع العينات في جهاز تدوير حراري واحتضانها عند 25 درجة مئوية لمدة 10 دقائق إلى بادئات صلبة ، عند 55 درجة مئوية لمدة 30 دقيقة لإجراء النسخ العكسي ، وأخيرا ، الإنزيمات المعطلة للحرارة عند 85 درجة مئوية لمدة 5 دقائق.
- بعد الحضانة ، ضع الأنبوب على الفور على الجليد وقم بتخفيف العينة إلى حجم إجمالي يبلغ 25 ميكرولتر عن طريق إضافة 21 ميكرولتر من الماء المعقم الخالي من النوكلياز. بالنسبة للنصوص ذات الأرقام المنخفضة ، قم بتغيير خطوة التخفيف هذه على النحو المحدد تجريبيا.
6. qPCR
- حدد درجة حرارة التلدين لمجموعة التمهيدي باستخدام إجمالي الحمض النووي الريبي المحضر من كميات أكبر من بطيئات المشية ، على سبيل المثال ، طريقة الاستخراج السائبة المعروضة في بوثبي ، 201843.
- قم بتشغيل تدرج درجة حرارة PCR لتحديد درجة حرارة التلدين المثلى قبل تشغيل qRT-PCR (للاطلاع على جميع إعدادات تفاعل البوليميراز المتسلسل المستخدمة في هذا البروتوكول، يرجى الرجوع إلى الجدول 1 والجدول 2).
- قم بإذابة أنبوب واحد من مزيج صبغة المؤشر الفائق على الثلج وعزلها عن الضوء. ضع لوحة qPCR مكونة من 96 بئرا على الجليد وضع 5 ميكرولتر من المزيج الفائق ، و 2 ميكرولتر من الماء ، و 1 ميكرولتر من كل برايمر (10 ميكرومتر) ، و 1 ميكرولتر من منتج (كدنا) في عدد الآبار المطلوبة.
- أغلق لوحة PCR بختم اللوحة وقم بتشغيل qRT-PCR باستخدام درجة حرارة تلدين مناسبة لمجموعة التمهيدي (لجميع إعدادات qRT-PCR المستخدمة في هذه الورقة، راجع الجدول 3).
7. القياس الكمي وتفسير النتائج
- قارن النتائج كميا بواحد أو أكثر من جينات التدبير المنزلي للتحكم ، والتي من المتوقع أن يكون تعبيرها ثابتا خلال الظروف المفروضة. في هذه الدراسة ، تم استخدام جين الأكتين.
- احصل على قيم Ct أو عتبة الدورة لكل بئر وقارنها بقيم Ct لتفاعلات جينات التدبير المنزلي للتحكم. احسب تغير الطية في التعبير الجيني باستخدام المعادلة التالية:
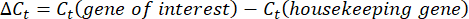
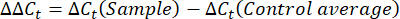
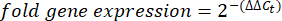
ملاحظة: يتم رسم التعبير الجيني الطي لكل نسخة وبطيئات المشية ك 2- (ΔΔCt) 48. - للحصول على تقدير تقريبي لرقم النص من قيمة Ct ، استخدم المعادلة التالية:
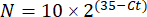
حيث N هو عدد النصوص ، و 2 هي كفاءة تفاعل البوليميراز المتسلسل المفترضة أو زيادة أضعاف التألق لكل دورة من تفاعل البوليميراز المتسلسل48.
Access restricted. Please log in or start a trial to view this content.
النتائج
تطوير وتحسين استخراج الحمض النووي الريبي أحادي المشية
من خلال تكييف البروتوكول من Ly et al. ، 201542 لاستخراج الحمض النووي الريبي في بطيئات المشية ، تم تحسين نظام STST لزيادة كمية ونوعية المستحضر (الشكل 1 أ). تم إجراء RT-PCR لنصوص الأكتين ، وتح...
Access restricted. Please log in or start a trial to view this content.
Discussion
تقدم هذه الدراسة طريقة فعالة لاستخراج الحمض النووي الريبي ل qRT-PCR أحادي المشية. كشفت المقارنة المباشرة لمنهجية STST بمجموعة استخراج الحمض النووي الريبي المشي المفردة الحالية أن استخراج الحمض النووي الريبي STST ينتج كميات أعلى بمقدار >200 ضعف من نصوص الحمض النووي الريبي الأكت?...
Access restricted. Please log in or start a trial to view this content.
Disclosures
يعلن المؤلفون عدم وجود تضارب في المصالح للإفصاح عنهم.
Acknowledgements
نريد أن نعرب عن تقديرنا لزمالة المعاهد الوطنية للصحة روث كيرششتاين # 5F32AG081056-02 وزمالة إريت فيشر لما بعد الدكتوراه ، التي دعمت الدكتورة مولي جيه كيرك ، وزمالة عائلة كرو ، التي دعمت تشومينغ شو ، ومنحة مجلس الشيوخ الأكاديمي بجامعة كاليفورنيا ، سانتا باربرا ، ومنح المعاهد الوطنية للصحة #R01GM143771 و #2R01HD081266 ، التي دعمت هذه الجهود البحثية. يقر المؤلفون أيضا باستخدام مختبر الهياكل النانوية البيولوجية داخل معهد كاليفورنيا للأنظمة النانوية ، بدعم من جامعة كاليفورنيا ، سانتا باربرا ، وجامعة كاليفورنيا ، مكتب الرئيس.
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| 10 µL Premium Barrier Tips Low Binding, Racked, Sterile | Genesee Scientific | 23-401 | Refered to as Sterile Filter-Tipped P 10 Pipette Tips |
| 1000 µL Premium Pipet Tips, Low Binding, Racked, Sterile | Genesee Scientific | 23-165RS | Refered to as Sterile Filter-Tipped P 1000 Pipette Tips |
| 200 µL Premium Barrier Tips Low Binding, Racked, Sterile | Genesee Scientific | 23-412 | Refered to as Sterile Filter-Tipped P 200 Pipette Tips |
| 4 Star Straight Strong Medium Point Tweezer | Excelta | 00-SA-DC | Refered to as Long forceps |
| 96-Well PCR Rack with Lid Assorted, 5 Racks/Unit | Genesee Scientific | 27-202A | Refered to as PCR Rack |
| Andwin Scientific 3M LEAD FREE AUTOCLAVE TAPE 1" | Thermo Fisher Scientific | NC0802040 | Refered to as Autoclave Tape |
| Autoclave Tape | Thermo Fisher Scientific | AB1170 | Refered to as PCR Plate Seals |
| Benchling v8 | Benchling | N/A | Refered to as Benchling |
| BioRadHard-Shell 96-Well PCR Plate | BioRad | HSS9641 | Refered to as PCR Plate |
| BULWARK FR Lab Coat: | Grainger | 26CF64 | Refered to as Lab Coat |
| C1000 Touch Bio-rad Thermocycler | BioRad | 1851148 | Refered to as Thermocycler |
| C1000 Touch Bio-rad Thermocycler with CFX Optics Module | BioRad | 1845097 | Refered to as qPCR thermocycler |
| Chloroccoccum hypnosporum. | Carolina | 152091 | Refered to as Algae |
| Corning PYREX Reusable Media Storage Bottles | Thermo Fisher Scientific | 06-414-1E | Refered to as 2 L Autoclave-safe Glass Bottle |
| Daigger & Company Vortex-Genie 2 Laboratory Mixer | Thermo Fisher Scientific | 3030A | Refered to as Vortexer |
| Direct-zol Micro Prep | Zymo Research | R2060 | Refered to as RNA extraction kit |
| Dumont 5 Biology Tweezers | Fine Science Tools | 11254-20 | Refered to as Fine Forceps |
| EDTA | Fisher Scientific | S311-500 | Refered to as EDTA |
| FIJI v 2.14.0/1.54f | ImageJ, | N/A | Refered to as FIJI/ImageJ |
| Filament for pippette Puller | Tritech Research | PC-10H | Refered to as Filament |
| Fisherbrand Economy Impact Goggles | Fisher Scientific | 19-181-501 | Refered to as Splash Goggles |
| Glass Micropipette O.D. 1mm ID 0.58, Length 10 cm | TriTech Research | GD-1 | Reffered to as glass micropipette |
| Hypsibius exemplaris Z151 Strain | Carolina | 133960 | Refered to as Tardigrades or H. exemplaris |
| Liquid Nitrogen Dewar 1 L | Agar Scientific | AGB7475 | Refered to as Cryo-safe container |
| Maxima H Minus First Strand cDNA Synthesis Kit | Thermo Fisher Scientific | K1651 | Refered to as cDNA Synthesis Master Mix |
| Narishige Dual-Stage Glass Micropipette Puller | Tritech Research | PC-10 | Refered to as micropipette puller |
| Nitrile Gloves | Fisher Scientific | 17-000-314 | Refered to as Nitrile Gloves |
| PETRI DISH, PS, 35/10 mm, WITH VENTS | Grenier | 627102 | Refered to as 35 mm Petri dish |
| PIPETMAN P10, 1–10 µL, Metal Ejector | Gilson | F144055M | Refered to as P 10 Pipette |
| PIPETMAN P1000, 100–1000 µL, Metal Ejector | Gilson | F144059M | Refered to as P 1000 Pipette |
| PIPETMAN P200, 20–200 µL, Metal Ejector | Gilson | F144058M | Refered to as P 200 Pipette |
| Pound This 4-Color Modeling Clay | American Science Surplus | 96517P001 | Refered to as Clay |
| Prism v10.0 | GraphPad | N/A | Refered to a Prism |
| RNAse-Free, 8 Strip 0.2 mL PCR Tubes with caps | Invitrogen | AM12230 | Refered to as Sterile PCR Tube |
| RNasin Ribonuclease Inhibitor | Promega | N2111 | Refered to as RNAse inhibitor |
| Spring water | Nestle Pure Life | 44221229 | Refered to as Spring Water |
| SsoAdvanced Universal SYBR Green Supermix | BIO RAD | 1725271 | Refered to as Indicator Dye Super mix |
| Stereo-Microscope System w/optics and illumination | TriTech Research | SMT1 | Refered to as Dissecting Microscope |
| Supertek Scientific Tirrill Burners | Thermo Fisher Scientific | S09572B | Refered to as Bunsen Burner |
| Table Top Centrifuge | Qualitron | DW-41-115-NEW | Refered to as Table Top Centrifuge |
| Tempshield Cryo-Gloves | Fisher Scientific | 11-394-305 | Refered to as Cryo Gloves |
| Thermo Scientific Nunc Petri Dishes | Thermo Fisher Scientific | 08-757-099 | Refered to as 100 mm Petri dish |
| Tris base | Fisher Scientific | T395-500 | Refered to as Tris or Tris Base |
| Triton X-100 | Fluka | 93443 | Refered to as Detergent 1 |
| TWEEN 20 | Sigma aldrich | P1379-500 | Refered to as Detergent 2 |
| Water - PCR/RT-PCR certified, nuclease-free | Growcells | PCPW-0500 | Refered to as Sterile Nuclease Free Water |
References
- Møbjerg, N., Neves, R. C. New insights into survival strategies of tardigrades. Comp Biochem Physiol Part A Mol Integr Physiol. 254, 110890(2021).
- Jönsson, K. I., Harms-Ringdahl, M., Torudd, J. Radiation tolerance in the eutardigrade Richtersius coronifer. Int J Radiat Biol. 81 (9), 649-656 (2005).
- Horikawa, D. D., et al. Radiation tolerance in the tardigrade Milnesium tardigradum. Int J Radiat Biol. 82 (12), 843-848 (2006).
- Bruckbauer, S. T., Cox, M. M. Experimental evolution of extremophile resistance to ionizing radiation. Trends Genet. 37 (9), 830-845 (2021).
- Jönsson, K. I., Hygum, T. L., Andersen, K. N., Clausen, L. K. B., Møbjerg, N. Tolerance to gamma radiation in the marine heterotardigrade, Echiniscoides sigismundi. PLoS One. 11 (12), e0168884(2016).
- Jönsson, K. I. Radiation tolerance in tardigrades: Current knowledge and potential applications in medicine. Cancers (Basel). 11 (9), 1333(2019).
- Yoshida, Y., et al. RNA sequencing data for gamma radiation response in the extremotolerant tardigrade Ramazzottius varieornatus. Data Brief. 36, 107111(2021).
- Clark-Hachtel, C. M., et al. The tardigrade Hypsibius exemplaris dramatically upregulates DNA repair pathway genes in response to ionizing radiation. Curr Biol. 34 (9), 1819-1830.e6 (2024).
- Anoud, M., et al. Comparative transcriptomics reveal a novel tardigrade specific DNA binding protein induced in response to ionizing radiation. Elife. 13, RP92621(2024).
- Jönsson, K. I., Schill, R. O. Induction of Hsp70 by desiccation, ionising radiation and heat-shock in the eutardigrade Richtersius coronifer. Comp Biochem Physiol B Biochem Mol Biol. 146 (4), 456-460 (2007).
- Boothby, T. C. Desiccation of Hypsibius exemplaris. Cold Spring Harb Protoc. 2018 (11), 871-873 (2018).
- Boothby, T. C., et al. Tardigrades use intrinsically disordered proteins to survive desiccation. Mol Cell. 65 (6), 975-984.e5 (2017).
- Horikawa, D. D., Higashi, S. Desiccation tolerance of the tardigrade Milnesium tardigradum collected in Sapporo, Japan, and Bogor, Indonesia. Zoolog Sci. 21 (8), 813-816 (2004).
- Halberg, K. A., Jørgensen, A., Møbjerg, N. Desiccation tolerance in the tardigrade Richtersius coronifer relies on muscle mediated structural reorganization. PLoS One. 8 (12), e3330(2013).
- Sørensen-Hygum, T. L., Stuart, R. M., Jørgensen, A., Møbjerg, N. Modelling extreme desiccation tolerance in a marine tardigrade. Sci Rep. 8 (1), 11495(2018).
- Lyons, A. M., Roberts, K. T., Williams, C. M. Survival of tardigrades (Hypsibius exemplaris) to subzero temperatures depends on exposure intensity, duration, and ice-nucleation - as shown by large-scale mortality dye-based assays. bioRxiv. , (2024).
- Møbjerg, A., et al. Extreme freeze-tolerance in cryophilic tardigrades relies on controlled ice formation but does not involve significant change in transcription. Comp Biochem Physiol Part A Mol Integr Physiol. 271, 111245(2022).
- Tsujimoto, M., Imura, S., Kanda, H. Recovery and reproduction of an Antarctic tardigrade retrieved from a moss sample frozen for over 30 years. Cryobiology. 72 (1), 78-81 (2016).
- Jönsson, K. I. Tardigrades as a potential model organism in space research. Astrobiology. 7 (5), 757-766 (2007).
- Jönsson, K. I., Rabbow, E., Schill, R. O., Harms-Ringdahl, M., Rettberg, P. Tardigrades survive exposure to space in low Earth orbit. Curr Biol. 18 (17), R729-R731 (2008).
- Kasianchuk, N., Rzymski, P., Kaczmarek, Ł The biomedical potential of tardigrade proteins: A review. Biomed Pharmacother. 158, 113983(2023).
- Arakawa, K. Examples of extreme survival: Tardigrade genomics and molecular anhydrobiology. Annu Rev Anim Biosci. 10 (1), 519-542 (2022).
- Hvidepil, L. K. B., Møbjerg, N. New insights into osmobiosis and chemobiosis in tardigrades. Front Physiol. 14, 1274522(2023).
- Tanaka, S., Aoki, K., Arakawa, K. In vivo expression vector derived from anhydrobiotic tardigrade genome enables live imaging in Eutardigrada. Proc Natl Acad Sci U S A. 120 (5), e2216739120(2023).
- Kondo, K., Tanaka, A., Kunieda, T. Single-step generation of homozygous knockout/knock-in individuals in an extremotolerant parthenogenetic tardigrade using DIPA-CRISPR. PloS Genet. 20 (6), e1011298(2024).
- Yoshida, Y., Hirayama, A., Arakawa, K. Transcriptome analysis of the tardigrade Hypsibius exemplaris exposed to the DNA-damaging agent bleomycin. bioRxiv. , (2024).
- Yoshida, Y., et al. Time-series transcriptomic screening of factors contributing to the cross-tolerance to UV radiation and anhydrobiosis in tardigrades. BMC Genomics. 23 (1), 405(2022).
- Yoshida, Y., et al. RNA sequencing data for gamma radiation response in the extremotolerant tardigrade Ramazzottius varieornatus. Data Brief. 36, 107111(2021).
- Neves, R. C., et al. Differential expression profiling of heat stressed tardigrades reveals major shift in the transcriptome. Comp Biochem Physiol Part A Mol Integr Physiol. 267, 111143(2022).
- Yoshida, Y., et al. Comparative genomics of the tardigrades Hypsibius dujardini and Ramazzottius varieornatus. PLoS Biol. 15 (7), e2002266(2017).
- Wang, C., Grohme, M. A., Mali, B., Schill, R. O., Frohme, M. Towards decrypting cryptobiosis - analyzing anhydrobiosis in the tardigrade Milnesium tardigradum using transcriptome sequencing. PLoS One. 9 (3), e92663(2014).
- Mali, B., et al. Transcriptome survey of the anhydrobiotic tardigrade Milnesium tardigradum in comparison with Hypsibius dujardini and Richtersius coronifer. BMC Genomics. 11 (1), 168(2010).
- Förster, F., et al. Transcriptome analysis in tardigrade species reveals specific molecular pathways for stress adaptations. Bioinform Biol Insights. 6, 69-96 (2012).
- Arakawa, K., Yoshida, Y., Tomita, M. Genome sequencing of a single tardigrade Hypsibius dujardini individual. Sci Data. 3 (1), 160063(2016).
- Arakawa, K. Transcriptome assembly of Richtersius coronifer with annotated BLAST result against Ramazzottius varieornatus. Figshare. Dataset. , (2019).
- Yoshida, Y., Konno, S., Nishino, R., Murai, Y., Tomita, M., Arakawa, K. Ultralow input genome sequencing library preparation from a single tardigrade specimen. J Vis Exp. (137), (2018).
- Murai, Y., et al. Multiomics study of a heterotardigrade, Echiniscus testudo, suggests convergent evolution of anhydrobiosis-related proteins in Tardigrada. bioRxiv. , (2020).
- Yoshida, Y., Sugiura, K., Tomita, M., Matsumoto, M., Arakawa, K. Comparison of the transcriptomes of two tardigrades with different hatching coordination. BMC Dev Biol. 19 (1), 24(2019).
- Tenlen, J. R. Microinjection of dsRNA in tardigrades. Cold Spring Harb Protoc. 2018 (11), (2018).
- Tenlen, J. R., McCaskill, S., Goldstein, B. RNA interference can be used to disrupt gene function in tardigrades. Dev Genes Evol. 223 (3), 171-181 (2013).
- Giovannini, I., et al. Production of reactive oxygen species and involvement of bioprotectants during anhydrobiosis in the tardigrade Paramacrobiotus spatialis. Sci Rep. 12 (1), 15888(2022).
- Ly, K., Reid, S. J., Snell, R. G. Rapid RNA analysis of individual Caenorhabditis elegans. MethodsX. 2, 59-63 (2015).
- Boothby, T. C. Total RNA extraction from tardigrades. Cold Spring Harb Protoc. 2018 (11), 905-907 (2018).
- Czerneková, M., Vinopal, S. The tardigrade cuticle. Limnol Rev. 21 (3), 127-146 (2021).
- Goldstein, B. Hypsibius dujardini. collection notes and culture protocol from Bob McNuff. , At http://tardigrades.bio.unc.edu/protocols/CollectionCulture.pdf (2007).
- McNuff, R. Laboratory culture of Hypsibius exemplaris. Cold Spring Harb Protoc. 2018 (11), 867-870 (2018).
- Gabriel, W. N., et al. The tardigrade Hypsibius dujardini, a new model for studying the evolution of development. Dev Biol. 312 (2), 545-559 (2007).
- Ruiz-Villalba, A., Ruijter, J. M., van den Hoff, M. J. B. Use and misuse of cq in qPCR data analysis and reporting. Life (Basel). 11 (6), 508(2021).
- Antonov, J., et al. Reliable gene expression measurements from degraded RNA by quantitative real-time PCR depend on short amplicons and a proper normalization. Lab Invest. 85 (8), 1040-1050 (2005).
- Toussaint, J., et al. Improvement of the clinical applicability of the genomic grade index through a qRT-PCR test performed on frozen and formalin-fixed paraffin-embedded tissues. BMC Genomics. 10, 424(2009).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved