Method Article
هندسة البروتين بواسطة عرض سطح الخميرة
In This Article
Summary
يصف هذا البروتوكول الخطوات الأساسية لإجراء حملات اختيار عرض سطح الخميرة لإثراء متغيرات البروتين المرتبطة بمستضد ذي أهمية.
Abstract
تتيح هندسة البروتين تحسين الوظائف الحالية لبروتين معين أو توليد وظائف جديدة. واحدة من أكثر الأدوات استخداما وتنوعا في مجال هندسة البروتين هي عرض سطح الخميرة ، حيث يتم التعبير عن مجموعة من البروتينات العشوائية على سطح الخميرة. يتيح ارتباط النمط الظاهري (على سبيل المثال ، ارتباط البروتين المعروض بالخميرة بالمستضد محل الاهتمام) والنمط الجيني (ترميز البلازميد لمتغير البروتين) اختيار هذه المكتبة للخصائص المرغوبة والتسلسل اللاحق للمتغيرات المخصبة. من خلال الجمع بين اختيار الخرزة المغناطيسية والفرز الخلوي للتدفق ، يمكن اختيار متغيرات البروتين ذات الارتباط المعزز بمستضد الهدف وإثرائها. والجدير بالذكر أنه بالإضافة إلى نضوج التقارب ، يمكن أيضا تحقيق الارتباط بالهدف دون أي تقارب ملزم أولي. هنا ، نقدم بروتوكولا خطوة بخطوة يغطي جميع الأجزاء الأساسية لحملة اختيار عرض سطح الخميرة ويقدم أمثلة على نتائج عرض سطح الخميرة النموذجية. نوضح أن عرض سطح الخميرة هو طريقة قوية وقابلة للتطبيق على نطاق واسع يمكن إنشاؤها في أي مختبر للبيولوجيا الجزيئية مع إمكانية الوصول إلى قياس التدفق الخلوي.
Introduction
يعد عرض سطح الخميرة أحد التقنيات الرئيسية في مجال هندسة البروتين. يتيح اختيار متغيرات البروتين ذات الخصائص المرغوبة مثل تحسين التقارب أو الاستقرار. تم تقديمها لأول مرة في عام 19971 ، وهي واحدة من أكثر تقنيات العرض استخداما إلى جانب عرض العاثيات2،3 ، وعرض الريبوسوم4 ، وعرض خلايا الثدييات5،6،7. يتم عرض البروتين محل الاهتمام (POI) على سطح خلايا الخميرة عن طريق دمجها لتثبيت البروتينات. تتوفر مجموعة من بروتينات المرساة المختلفة ، والأكثر شيوعا ، يتم دمج POI في الطرف C لبروتين تزاوج الخميرة التراص Aga2p1،8. بالإضافة إلى ذلك ، عادة ما يكون POI محاطا بعلامتين ، مثل علامة الهيماجلوتينين (HA-tag) وعلامة c-myc ، والتي تتيح اكتشاف مستوى العرض باستخدام الأجسام المضادة المسماة بالفلورسنت وقياس التدفق الخلوي (الشكل 1 أ). تتضمن حملات اختيار الخميرة النموذجية مزيجا من اختيارات الخرزة المغناطيسية والفرز الخلوي للتدفق. تتيح اختيارات الخرزة التعامل مع أعداد الخلايا الكبيرة وإثراء متغيرات البروتين المرتبطة بالمستضد المستهدف أيضا مع تقاربات منخفضة لأن التفاعلات متعددة التكافؤ مع الخرز المحمل بالمستضد تؤدي إلى تأثيرات شغف ، وبالتالي تمنع فقدان المتغيرات منخفضة التقارب (الشكل 1 ب). يوفر تحليل واختيار التدفق الخلوي ميزة تصور ارتباط متغيرات POI المعروضة بالمستضد المسمى. وبالتالي ، يمكن فرز مجموعات الربط وزراعتها ، مما يؤدي إلى إثراء متغيرات البروتين بالخصائص المرغوبة خلال عدة جولات فرز. علاوة على ذلك ، يمكن إجراء جولات إضافية من الطفرات العشوائية لزيادة التنوع ، وبالتالي احتمال العثور على طفرات إضافية تساهم في تقارب و / أو استقرار البروتين.
يقدم عرض سطح الخميرة مزايا معينة ، مثل (أ) آلية التعبير حقيقيات النواة ، مما يتيح طي البروتين المؤكسد بالإضافة إلى تعديلات ما بعد الترجمة حقيقية النواة (مثل N-glycosylation) ، (ب) تطبيع التعبير بسبب الكشف عن علامتي الببتيد المحاطتين بالبروتين ، (ج) الفحص البصري لتقدم الاختيار عن طريق قياس التدفق الخلوي (على سبيل المثال ، النسبة المئوية لخلايا الارتباط وشدة الارتباط) و (د) إمكانية تحليل طفرات البروتين الفردية على الخميرة (على سبيل المثال ، تحليل الثبات الحراري وكذلك التقارب) ، مما يوفر بديلا موفرا للوقت للتعبير عن البروتين الشاق والتنقية9. في الواقع ، أظهرت كل من الصلات (قيم KD) وكذلك الثبات (قيم T50) للبروتينات المعروضة على سطح الخميرة ارتباطات جيدة مع البيانات التي تم الحصول عليها باستخدام الطرق الفيزيائية الحيوية والبروتينات القابلة للذوبان9،10،11،12. تم استخدام عرض سطح الخميرة لهندسة مجموعة متنوعة من البروتينات ، على سبيل المثال ، شظايا الأجسام المضادة13،14،15،16 ، أو مجال الفبرونيكتينمن النوع الثالث17،18 ، أو rcSso7d19،20 ، أو knottins21. وبالمثل ، تم إجراء بحث مكثف لتحسين تصميمات مكتبة الخميرة عن طريق تغيير المواضع العشوائية بالإضافة إلى استخدام كودون الأحماض الأمينية17،22،23. أثبت عرض سطح الخميرة نجاحه في هندسة الاستقرار14،15،24،25 ، والتقارب18،26،27 ، والنشاط الأنزيمي28،29،30،31 ، والتعبير عن البروتين32. بالإضافة إلى ذلك ، تم إنجاز تطبيقات أكثر تعقيدا مثل الربط الشرطي في وجود أو عدم وجود جزيء صغير باستخدام عرض سطح الخميرة20.
في هذا البروتوكول ، نصف جميع الخطوات الأساسية لحملة اختيار مع عرض سطح الخميرة مع مثال مكتبة G4 (بناء على مجال الفبرونيكتينمن النوع الثالث العاشر ، Fn3) المختار ضد البروتين البشري المرتبط بالريتينول 4 (hRBP4) في وجود الجزيء الصغير A112020. تم إجراء هذا الاختيار لإنتاج تفاعل بين البروتين والبروتين يعتمد على جزيء صغير يمكن استخدامه كمحول جزيئي. وتجدر الإشارة إلى أنه في حين أن الأساليب البديلة ممكنة مع عرض سطح الخميرة ، فإن اختيارات الخميرة النموذجية عادة ما تهدف إلى الارتباط بمستضد مستهدف دون أي تقارب ربط سابق. نحن نغطي جميع خطوات حملة اختيار الخميرة ، بما في ذلك زراعة مكتبة الخميرة ، واختيارات الخرز ، وفرز مقياس التدفق الخلوي ، ونضج التقارب عن طريق تفاعل البوليميراز المتسلسل المعرض للخطأ (epPCR). لذلك ، يكمل هذا البروتوكول بروتوكولات عرض سطح الخميرةالسابقة 33،34 ويمكن استخدامه كأساس لاختيارات عرض سطح الخميرة (الشكل 1) مع أي مكتبة خميرة معينة ومستضد مستهدف مفضل.
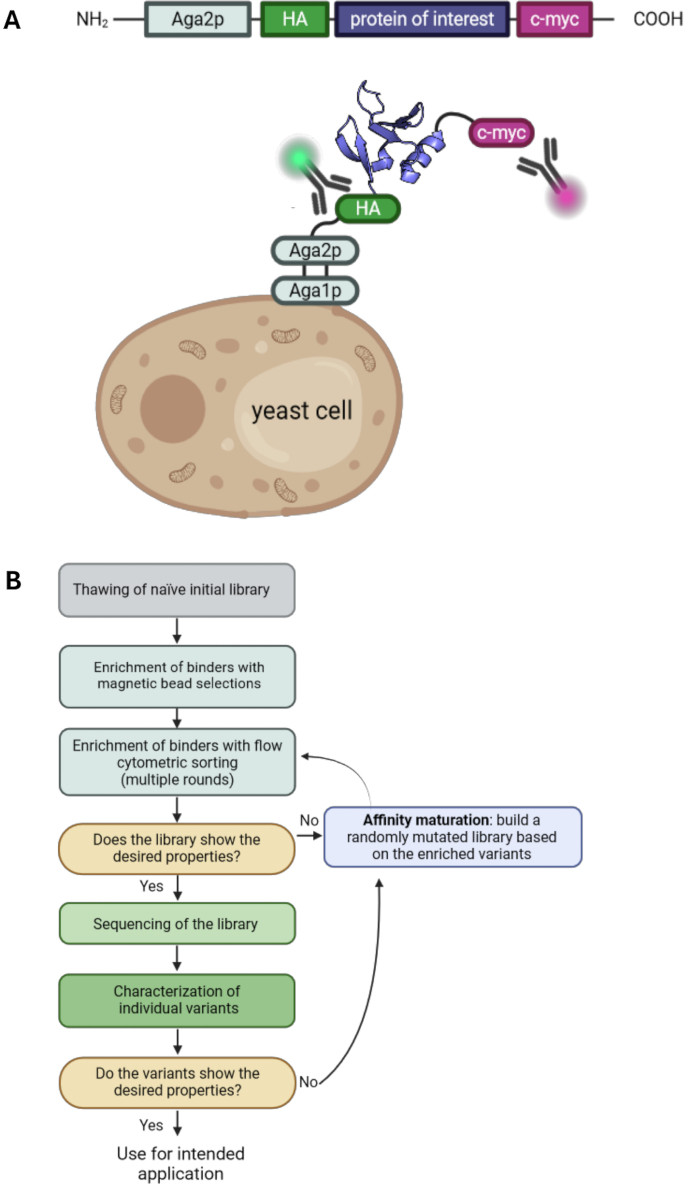
الشكل 1: مبدأ عرض سطح الخميرة وسير عمل نموذجي لاختيارات عرض سطح الخميرة. (أ) يتم استنساخ نقطة الاهتمام في متجه عرض سطح الخميرة وعادة ما يحيط به HA الطرفي N- وعلامة c-myc-terminal C. يتم دمج البناء مع بروتين تزاوج الخميرة Aga2p لعرضه على السطح. البروتين الموضح هو الموثق المصمم هندسيا "RS3" من معرف PDB: 6QBA20. (ب) مخطط انسيابي يوضح سير العمل النموذجي لحملات اختيار عرض سطح الخميرة ، والتي تجمع بين إثراء متغيرات البروتين مع الخصائص المرغوبة عن طريق اختيارات الخرزة والفرز الخلوي للتدفق ، بالإضافة إلى epPCR لنضج التقارب. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
Protocol
1. إذابة وزراعة مكتبات الخميرة
- قم بإذابة كميات كافية من مكتبة الخميرة المجمدة في RT وتخفيفها على الفور في SD-CAA (انظر الجدول 1) إلى OD600 من حوالي 1 (هذا يتوافق مع 1 × 107 خلايا / مل).
ملاحظة: تأكد من تغطية تنوع المكتبة بمقدار 10 أضعاف على الأقل. وبالتالي ، إذا كان حجم المكتبة يمثل 2.5 × 108 نسخ (هذا هو تنوع مكتبة G4 المستخدمة في هذا المثال) ، فقم بتلقيح ما لا يقل عن 2.5 × 109 خلايا في 250 مل من SD-CAA للوصول إلى OD600 من 1. يمكن أيضا بدء هذا البروتوكول من المزارع السائلة (خلايا الخميرة التي تحولت حديثا باستخدام مكتبة). - تقييم عدد الخلايا القابلة للحياة.
- خذ 100 ميكرولتر من معلق الخميرة هذا (عند OD600 من 1) وقم بإجراء سلسلة تخفيف 1:10 مع 900 ميكرولتر من وسط SD-CAA في أنابيب الطرد المركزي الدقيقة.
- قم بإعداد تخفيفات تصل إلى 10-5 ولوحة 100 ميكرولتر من التخفيفات 10-3 و 10-4 و 10-5 على لوحات SD-CAA (الجدول 1). احتضان الألواح عند 30 درجة مئوية لمدة 2-3 أيام. تأكد من أن عدد الخلايا القابلة للحياة بعد الذوبان يتجاوز تنوع المكتبة بمقدار 10 أضعاف على الأقل.
- وفي الوقت نفسه ، استمر في تعليق الخلية المتبقي وقم بزراعة ثقافة الخميرة التي تحتوي على مكتبة G4 في قوارير زجاجية طوال الليل عند 30 درجة مئوية أثناء الاهتزاز (180 دورة في الدقيقة). بعد الحضانة الليلية ، استمر في القسم 2 (تحريض تعبير البروتين على سطح الخميرة).
- بعد 2-3 أيام من الحضانة ، عد المستعمرات على لوحات SD-CAA عن طريق الفحص البصري. عند حساب العدد النهائي للخلايا القابلة للحياة بعد الذوبان ، ضع في اعتبارك عامل التخفيف ونسبة الطلاء (أي 100 ميكرولتر) مقابل حجم الثقافة الأصلي (أي 250 مل).
خلايا قابلة للحياة / مل = عدد المستعمرات على اللوحة × عامل التخفيف
الخلايا القابلة للحياة (الإجمالي) = (خلايا قابلة للحياة / مل) × حجم الثقافة
| متوسط/عازل | مكون | التركيز [جم / لتر] | تعليقات/وصف | |||
| SD-CAA | د-الجلوكوز | 20 | قم بإذابة جميع مكونات الوسائط في 1000 مل ddH2O والترشيح المعقم باستخدام مرشحات معقمة 0.22 ميكرومتر يمكن التخلص منها. | |||
| قاعدة نيتروجين الخميرة | 6.7 | |||||
| أحماض الكاسمينو | 5 | |||||
| حامض الستريك أحادي الهيدرات | 7.4 | |||||
| ثلاثي سترات الصوديوم ثنائي هيدرات | 10.83 | |||||
| SG-CAA | د-جالاكتوز | 20 | قم بإذابة جميع مكونات الوسائط في 1000 مل ddH2O والترشيح المعقم باستخدام مرشحات معقمة 0.22 ميكرومتر يمكن التخلص منها. | |||
| د-الجلوكوز | 2 | |||||
| قاعدة نيتروجين الخميرة | 6.7 | |||||
| أحماض الكازامينو | 5 | |||||
| ثنائي الصوديوم فوسفات الهيدروجين هيبتاهيدرات | 10.2 | |||||
| أحادي هيدرات فوسفات ثنائي هيدروجين الصوديوم | 8.56 | |||||
| لوحات SD-CAA | السوربيتول | 182 | قم بإذابة السوربيتول ، وسباع هيبتاهيدرات فوسفات الهيدروجين ثنائي الصوديوم ، وأحادي هيدرات فوسفات ثنائي هيدروجين الصوديوم وأجار أجار في 900 مل ddH2O والأوتوكلاف. قم بإذابة المكونات المتبقية وتعقيمها في 100 مل ddH2O وأضفها عندما تكون الوسائط المعقمة فاترة. | |||
| ثنائي الصوديوم فوسفات الهيدروجين هيبتاهيدرات | 10.2 | |||||
| أحادي هيدرات فوسفات ثنائي هيدروجين الصوديوم | 7.44 | |||||
| أجار أجار | 15 | |||||
| د-الجلوكوز | 20 | |||||
| قاعدة نيتروجين الخميرة | 6.7 | |||||
| أحماض الكازامينو | 5 | |||||
| YPD | الببتون | 20 | قم بإعداد مخزون 10x D-glucose (200 جم / لتر) ومرشح معقم باستخدام مرشحات معقمة 0.22 ميكرومتر يمكن التخلص منها. قم بإذابة مستخلص الببتون والخميرة في 900 مل ddH2O والأوتوكلاف. عندما يكون فاترا ، أضف 100 مل 10x D-glucose. | |||
| مستخلص الخميرة | 10 | |||||
| د-الجلوكوز | 20 | |||||
| لوحات YPD | الببتون | 20 | قم بإعداد مخزون 10x D-glucose (200 جم / لتر) ومرشح معقم باستخدام مرشحات معقمة 0.22 ميكرومتر يمكن التخلص منها. قم بإذابة البيبتون وخلاصة الخميرة وأجار أجار في 900 مل ddH2O والأوتوكلاف. عندما يكون فاترا ، أضف 100 مل 10x D-glucose. | |||
| مستخلص الخميرة | 10 | |||||
| د-الجلوكوز | 20 | |||||
| أجار أجار | 15 | |||||
| بي بي إس إيه | جيش صرب البوسنه | 1 | قم بإذابة BSA في PBS والترشيح المعقم باستخدام مرشحات معقمة 0.22 ميكرومتر يمكن التخلص منها. | |||
الجدول 1: تكوين الوسائط والعازلة.
2. تحريض تعبير البروتين على سطح الخميرة
- بعد الحضانة بين عشية وضحاها ، تصل مزارع الخميرة عادة إلىOD 600 بين 2-20. قم بقياس OD600 بمقياس ضوئي لتحديد كثافة ثقافة الخميرة.
ملاحظة: قياسات OD خطية فقط في حدود 0.1-1. لذلك ، قم بإجراء التخفيفات (عادة 1:20 و 1:50) في SD-CAA لتحديد OD600.- قم بتخفيف الخلايا في SD-CAA إلى OD600 من 1 وتأكد من تغطية التنوع 10x (على سبيل المثال ، يتم استخدام 2.5 × 109 خلايا لتخفيف مكتبة G4). استمر في الحضانة عند 30 درجة مئوية أثناء الاهتزاز للسماح للخلايا بالوصول إلى مرحلة النمو الأسي.
- تحضير مخزونات الجلسرين (تغطي التنوع 50x) من مكتبات الخميرة (في SD-CAA التي تحتوي على 15٪ جلسرين) من الثقافة الليلية. لهذا الغرض ، قم بالطرد المركزي العدد المطلوب من خلايا الخميرة (2000 × جم ، 3 دقائق ، 4 درجات مئوية) ، وتخلص من المادة الطافية ، وأعد تعليق حبيبات الخلية في 1 مل من وسط SD-CAA مكمل بنسبة 15٪ من الجلسرين. قم بتجميد مخزون الجلسرين على الفور عند -80 درجة مئوية.
ملاحظة: تزداد صلاحية خلايا الخميرة إذا نمت مزارع الخميرة إلى مرحلة ثابتة قبل التجميد. في جولات الاختيار اللاحقة ، عندما يكون تنوع المكتبات أقل ، يكفي عادة استخدام 700 ميكرولتر من ثقافة الخميرة وخلطها مع 300 ميكرولتر من SD-CAA مكمل بنسبة 50٪ جلسرين. هنا ، تظهر مزارع الخميرة عادة قيم OD600 من 15 في المرحلة الثابتة ، ولكن قد تعتمد القيمة الفعلية على مقياس الضوء المستخدم. وبالتالي ، يمكن تحديد المرحلة الثابتة عن طريق قياسات OD600 المتكررة. وإذا لم تزد قيمة OD600 خلال فترة حضانة مدتها 2 ساعة، يتم الوصول إلى مرحلة ثابتة.
- جهاز طرد مركزي حجم مناسب من الخلايا لتغطية التنوع 10x عند 2000 × جم لمدة 5 دقائق ، 4-6 ساعات بعد تخفيف ثقافة الخميرة (قيم OD600 النموذجية بين 4-6) ، وتخلص من المادة الطافية.
- أعد تعليق حبيبات الخلية في SG-CAA (انظر الجدول 1) لإنتاج OD600 من 1 ونقلها إلى قارورة زجاجية جديدة.
ملاحظة: يؤدي إعادة التعليق في وسط يحتوي على الجالاكتوز إلى التعبير عن البروتين على السطح بواسطة المحفز المحفز للجالاكتوز. - احتضان مزارع الخميرة طوال الليل عند 20 درجة مئوية أثناء الاهتزاز عند 180 دورة في الدقيقة للحث على التعبير السطحي عن نقطة الاهتمام. بعد الحث بين عشية وضحاها ، ستصل مزارع الخميرة عادة إلى قيمOD 600 بين 2-5 ؛ إذا تم رفع درجة حرارة الحث إلى 37 درجة مئوية ، فتوقع قيم OD600 من 4-10.
ملاحظة: يؤدي خفض درجة الحرارة إلى 20 درجة مئوية إلى إبطاء نمو الخلايا وعادة ما ينتج عنه مستويات عرض أفضل. ومع ذلك ، قد يكون الحث عند 37 درجة مئوية مفيدا من أجل زيادة ضغط الاختيار نحو الطفرات المستقرة35.
3. الجولة الأولى لاختيار الخرزة لمكتبات الخميرة (اختيار إيجابي)
ملاحظة: يتضمن إجراء اختيار الخرزة القياسي 6 خطوات (الجدول 2).
| يوم | درج | |
| 0 | ثقافة بين عشية وضحاها | |
| 1 | تحريض تعبير البروتين على سطح خلايا الخميرة | |
| 2 | اختيار الخرزة الأولى مع 1 اختيار إيجابي | |
| 3 | إزالة الخرزات ، المرور ، تحريض التعبير البروتيني على سطح خلايا الخميرة وتجميد المكتبة | |
| 4 | اختيار الخرزة الثانية مع 3 اختيار سلبي و 1 إيجابي | |
| 5 | إزالة الخرزات وتجميد المكتبة | |
الجدول 2: الجدول الزمني النموذجي لإجراء اختيارات الخرز لمكتبة الخميرة.
- تحضير الخرز
- قم بإعداد الخرزات (انظر جدول المواد) لاختيار الخرزة الأولى: لكل أنبوب ، أعد تعليق 10 ميكرولتر من الخرز المغناطيسي الموثق للبيوتين (4 × 105 حبات / ميكرولتر) في 990 ميكرولتر من PBSA (الجدول 1) للغسيل ، ضع الأنبوب على رف مغناطيسي لمدة دقيقتين مع فتح الغطاء. قم بإزالة المادة الطافية بعناية. كرر خطوة الغسيل مع 1 مل PBSA.
ملاحظة: إذا كانت هناك حاجة إلى أنابيب متعددة من الخرز بنفس المستضد الحيوي ، فيمكن تحضيرها في أنبوب واحد في هذه الخطوة للحصول على محلول موحد من حبة المستضد. في هذه الحالة ، يجب زيادة كمية المستضد وفقا لذلك في الخطوة التالية. - أعد تعليق الخرزات بحجم إجمالي قدره 1 مل من PBSA مع 6.7-33 pmoles من مستضد البيوتينيل (هنا: 6.86 ميكرولتر من hRBP4 بتركيز 0.125 مجم / مل) في أنبوب طرد مركزي دقيق سعة 1.5 مل.
ملاحظة: في حين أن 6.7 pmoles من المستضد كافية ، يجب استخدام 33 pmoles إذا كان المستضد غير محدد. إذا تم إجراء الاختيار لمستضد مرتبط بدواء جزيئي صغير أو ترابط ، كما في هذه الحالة ، يجب أن يكون هذا الدواء / الترابط موجودا أثناء الاختيار ويجب إضافته في هذه الخطوة (هنا: 5 ميكرومتر A1120). - احتضن عند 4 درجات مئوية لمدة 2 ساعة مع هز أو دوران لطيف.
ملاحظة: يمكن تحضير الخلايا خلال هذه الخطوة. - بعد الحضانة وقبل إضافتها إلى الخلايا ، ضع الأنبوب على رف مغناطيسي لمدة دقيقتين مع فتح الغطاء. قم بإزالة المادة الطافية واغسل الخرزات المحملة بالمستضد ب 1 مل من PBSA كما هو موضح أعلاه. كرر خطوة الغسيل.
ملاحظة: إذا تم اختيار مستضد مرتبط بدواء أو رابط ، فيجب أن يحتوي مخزن الغسيل المخزن المؤقت على هذا الدواء أو الترابط الزائد (هنا: 5 ميكرومتر A1120). - أعد تعليق الخرزات المحملة بالمستضد في 50 ميكرولتر من PBSA (في وجود الدواء أو الترابط ، إن أمكن).
ملاحظة: إذا تم تحضير نفس الخرز المحمل بالمستضد لأنابيب متعددة ، فاضبط مستوى الصوت وفقا لذلك (بالنسبة لمكتبة G4 ، فإن الحجم النهائي هنا هو 100 ميكرولتر ينتج حبات لأبوبين).
- قم بإعداد الخرزات (انظر جدول المواد) لاختيار الخرزة الأولى: لكل أنبوب ، أعد تعليق 10 ميكرولتر من الخرز المغناطيسي الموثق للبيوتين (4 × 105 حبات / ميكرولتر) في 990 ميكرولتر من PBSA (الجدول 1) للغسيل ، ضع الأنبوب على رف مغناطيسي لمدة دقيقتين مع فتح الغطاء. قم بإزالة المادة الطافية بعناية. كرر خطوة الغسيل مع 1 مل PBSA.
- تحضير خلايا الخميرة
- قم بقياس OD600 من ثقافة الخميرة المستحثة كما هو موضح أعلاه.
- بيليه خلايا كافية لتغطية 10 أضعاف تنوع المكتبة (2000 × جم ، 10 دقائق ، 4 درجات مئوية). بالنسبة لمكتبة G4 ، استخدم 2.5 × 109 خلايا للطرد المركزي. قم بإزالة المادة الطافية واغسل الخلايا ب 10 مل من PBSA.
- جهاز الطرد المركزي مرة أخرى (2000 × جم ، 10 دقائق ، 4 درجات مئوية) وإعادة تعليق الخلايا إلى تركيز 1.25 × 109 خلايا / مل في الحجم المناسب من PBSA (هنا: 2 مل) ونقلها إلى أنبوب الطرد المركزي الدقيق.
ملاحظة: يعتمد حجم PBSA على عدد الخلايا. - جهاز طرد مركزي (2000 × جم ، 3 دقائق ، 4 درجات مئوية) ، ويعيد تعليقه في 950 ميكرولتر من PBSA.
- حضانة خلايا الخميرة بخرز محمل بالمستضد
- أضف 50 ميكرولتر من الخرز المحمل بالمستضد إلى 950 ميكرولتر من تعليق الخلية واحتضنه لمدة ساعتين عند 4 درجات مئوية أثناء الاهتزاز أو الدوران.
- بعد الحضانة ، ضع محلول حبة مستضد الخلية على رف مغناطيسي مع فتح الغطاء. سائل الماصة من الغطاء إلى الأنبوب لإخضاع ثقافة الخميرة بأكملها للاختيار المغناطيسي. انتظر لمدة دقيقتين قبل التخلص بعناية من الخلايا غير المنضومة. يغسل على الفور ب 1 مل من PBSA.
ملاحظة: إذا تم إجراء الاختيارات في وجود دواء / ترابط - في هذا المثال A1120 - فيجب أن يكون هذا موجودا في PBSA. - بعد خطوة الغسيل باستخدام PBSA ، أعد تعليق الخلايا بسرعة في 1 مل من SD-CAA لمنعها من الجفاف.
- أضف 200 مل من SD-CAA وقم بإجراء عمليات تخفيف تسلسلية كما هو موضح أعلاه. لوحة 100 ميكرولتر على ألواح SD-CAA واحتضانها لمدة 2-3 أيام عند 30 درجة مئوية لتحديد الحد الأقصى للتنوع الجديد. هنا ، تنوع مكتبة G4 بعد تحديد الخرزة الأول هو 1.25 × 106 خلايا.
ملاحظة: نظرا لأن المكتبة الأصلية قد تم أخذ عينات زائدة بمقدار 10 أضعاف في جولة التحديد هذه ، فمن المعقول افتراض أن معظم المستنسخة ستكون موجودة عدة مرات في المجموعة المخصبة بعد التحديد. نتيجة لذلك ، سيكون التنوع الحقيقي أقل ، ويجب اعتبار هذا الرقم كقيمة قصوى. عادة ، يتم تقليل التنوع بمقدار 100 مرة على الأقل خلال جولة الاختيار الأولى هذه. احتضان المزرعة المتبقية (200 مل) عند 30 درجة مئوية مع الرج (180 دورة في الدقيقة) لمدة 16 ساعة على الأقل. يمكن إيقاف البروتوكول مؤقتا بعد الحضانة الليلية عن طريق وضع الخلايا عند 4 درجات مئوية.
4. إزالة الخرزات وزراعتها قبل جولة اختيار الخرزة التالية
- قم بقياس OD600 كما هو موضح أعلاه. بيليه خلايا كافية لتغطية التنوع بمقدار 10x على الأقل (2000 × جم ، 3 دقائق ، RT). تخلص من المادة الطافية وأعد تعليق الخلايا في 1 مل من SD-CAA.
- انقله إلى أنبوب طرد مركزي دقيق وضعه على رف مغناطيسي لمدة دقيقتين مع فتح الغطاء. انقل الخلايا غير المنضمة إلى أنبوب طرد مركزي دقيق جديد وكرر عملية الفصل.
- قم بتخفيف الخلايا غير المنضمة في 100 مل من SD-CAA واحتضانها عند 30 درجة مئوية مع الاهتزاز عند 180 دورة في الدقيقة حتى يتم الوصول إلى OD600 من 3-4.
ملاحظة: إذا كان OD600 أقل من 4 قبل إزالة الخرزات ، فيمكن تحفيز الخلايا مباشرة بعد ذلك دون حضانة. - عندما يتم الوصول إلى OD600 من 3-4 ، قم بالطرد المركزي بعدد كاف من الخلايا لتغطية التنوع على الأقل 10x (2000 × جم ، 3 دقائق ، RT) وإعادة التعليق في SG-CAA إلى OD600 من 1 لتحريض التعبير عن البروتين واحتضانه عند 20 درجة مئوية أثناء الاهتزاز عند 180 دورة في الدقيقة بين عشية وضحاها.
- عندما تصل زراعة الخميرة المتبقية SD-CAA إلى مرحلة النمو الثابتة (OD600 فوق 15) ، قم بحفر خلايا كافية لتغطية التنوع 50x (2000 × جم ، 3 دقائق ، RT). تجميد الخلايا عن طريق الطرد المركزي (2000 × جم ، 3 دقائق) ، متبوعا بإعادة تعليق حبيبات الخلية في SD-CAA التي تحتوي على 15٪ جلسرين وتخزينها عند -80 درجة مئوية.
5. جولة اختيار الخرزة الثانية مع 3 اختيار سلبي و 1 اختيار إيجابي
- تحضير الخلايا وحبات المستضد كما هو موضح أعلاه. قم بإعداد محلول من الخرز العارية (بدون إضافة المستضد) للتحديدات السلبية.
ملاحظة: نظرا لأنه يتم تنفيذ 3 تحديدات سلبية ، يمكن تحضير الخرزات معا لجميع الجولات الثلاث. - بعد الغسيل ، قم بتعليق حبات المستضد في 50 ميكرولتر من PBSA وأعد تعليق الخرزات العارية في 150 ميكرولتر من PBSA (إذا تم إعدادها لجميع الاختيارات السلبية الثلاثة).
- للاختيار السلبي الأول ، أضف 50 ميكرولتر من الخرزات العارية المغسولة إلى 950 ميكرولتر من الخلايا المغسولة في PBSA واحتضانها لمدة 1.5 ساعة عند 4 درجات مئوية.
- بعد الحضانة ، ضع الأنابيب التي تحتوي على معلقات خلايا الخرزة العارية على رف مغناطيسي مع فتح الغطاء. ماصة أي سائل في الغطاء في الأنبوب وانتظر لمدة 2 دقيقة. انقل الخلايا غير المرتبطة إلى أنبوب طرد مركزي دقيق جديد وأضف 50 ميكرولتر من الخرز العارية المغسولة.
- كرر الإجراء لجولتين أخريين من التحديد السلبي. وفي الوقت نفسه ، قم بإعداد حبات محملة بالمستضد للاختيار الإيجابي.
- بعد 3 جولات اختيار سلبية ، أضف 50 ميكرولتر من محلول الخرزة المحمل بالمستضد إلى الخلايا واحتضنه لمدة ساعتين عند 4 درجات مئوية.
- ضع الخلايا التي تحتوي على الخرزات المحملة بالمستضد على رف مغناطيسي مع فتح الغطاء ، وقم بإدخال أي سائل موجود في الغطاء في الأنبوب. انتظر لمدة دقيقتين قبل التخلص من الخلايا غير المنضومة.
- قم بتنفيذ جميع الخطوات المتبقية كما هو موضح في اختيار حبة المستضد الأولى. الاختلاف الوحيد في الاختيار الإيجابي الأول (الخطوة 3.3.2) هو عدد خطوات الغسيل: بدلا من الغسيل مرة واحدة باستخدام PBSA ، اغسل تعليق خلية الخرزة مرتين باستخدام PBSA لزيادة صرامة الاختيار.
ملاحظة: عادة ، يتم تقليل التنوع مرة أخرى بمقدار 10x-100x. كان الحد الأقصى لتنوع مكتبة G4 بعد اختيار الخرزة الثاني 3.45 × 105 خلايا. كما هو مذكور أعلاه ، من المتوقع أن يكون التنوع الفعلي أقل بسبب الإفراط في أخذ عينات من المكتبة.
6. اختيار المكتبات عن طريق الفرز الخلوي للتدفق
- بعد تحريض التعبير السطحي بين عشية وضحاها في SG-CAA، قم بقياس OD600 كما هو موضح أعلاه. بيليه خلايا كافية لتغطية 10 أضعاف التنوع (2000 × جم ، 5 دقائق ، RT). تخلص من المادة الطافية.
- أعد تعليق الحبيبات في PBSA وانقلها إلى أنابيب أجهزة الطرد المركزي الدقيقة. استخدم 3 × 107 خلايا للتلوين لكل أنبوب. قم بإعداد أكبر عدد ممكن من الأنابيب حسب الحاجة ، اعتمادا على التنوع. قم بإعداد أنبوب تحكم واحد للتلطيخ بدون المستضد.
ملاحظة: على سبيل المثال ، إذا كان يجب فرز 30 × 106 خلايا ، فقم بفرز 300 × 106 خلايا لتحقيق حبيبات خلية مرئية ومنع فقدان الخلايا في الخطوات اللاحقة. قم بإعادة التعليق في 5 مل من PBSA ونقل 3 إلى 4 حصص من 500 ميكرولتر لكل منها إلى أنابيب أجهزة الطرد المركزي الدقيقة. قم بإعداد خلايا أكثر بمقدار 3 أضعاف تقريبا من العدد الذي يجب فرزه لأن جزءا صغيرا من الخلايا سيفقد أثناء عملية التلوين. إذا تم إجراء الاختيار في وجود دواء / ترابط ، فقم بإعداد تحكم إضافي باستخدام المستضد فقط ولكن بدون الدواء / الترابط. - جهاز الطرد المركزي مرة أخرى (2000 × جم ، 5 دقائق ، RT) ، يعلق الحبيبات في 200 ميكرولتر من PBSA المحتوي على المستضد (hRBP4 مع 5 ميكرومتر A1120) ويحتضن لمدة ساعة واحدة عند 4 درجات مئوية.
ملاحظة: يجب تنفيذ جميع الخطوات من هذه النقطة فصاعدا على الجليد والكواشف الباردة. لمنع استنفاد الأجسام المضادة أو المستضد ، يجب إضافة جميع الكواشف بزيادة متكافئة مقارنة بالعدد الإجمالي للبروتينات المعروضة في الخميرة في الأنبوب. خلاف ذلك ، قد يكون هناك خطر الحد من ارتباط المتغيرات المعروضة بالخميرة بسبب التوافر المحدود للمستضدات و / أو الأجسام المضادة. عادة ، يتم استخدام 5 × 104 جزيئات لكل خلية خميرة للحساب ، وينصح بإضافة ما لا يقل عن 10 أضعاف فائض الستاكيومتر من المستضد والأجسام المضادة. إذا كان المستضد محدودا للغاية ، فيمكن تقليل الفائض إلى 5 أضعاف. يتم تقديم تفسير أكثر تفصيلا ، بما في ذلك أمثلة لحسابات تجنب استنفاد الترابط ، في بروتوكولسابق 9. - بعد الحضانة ، خلايا الطرد المركزي (2000 × جم ، 5 دقائق ، 4 درجات مئوية) وتغسل ب 1 مل من PBSA (في حالة وجود دواء / ترابط أثناء الاختيارات - مثل A1120 في هذه الحالة - يجب أن يكون هذا موجودا في مخزن الغسيل).
- أعد تعليق الخلايا في 100 ميكرولتر من PBSA البارد الذي يحتوي على الأجسام المضادة لعرض تلطيخ واكتشاف المستضد المرتبط واحتضانه لمدة 30 دقيقة عند 4 درجات مئوية.
- استخدم الجسم المضاد المضاد ل c-myc (التخفيف النهائي 1:100) للكشف عن نقطة الاهتمام المعروضة بكامل الطول. استخدم الجسم المضاد المضاد ل HA (التخفيف النهائي 1:50) للكشف عن الكمية الإجمالية للبروتينات المعروضة. استخدم الجسم المضاد Penta-His (التخفيف النهائي 1:20) أو الستربتافيدين (التخفيف النهائي 1:200) لتحديد خلايا الخميرة المرتبطة بالمستضد القابل للذوبان الذي يحتوي إما على علامة 6x HIS أو البيوتين ، على التوالي.
- قم بالتبديل بين كواشف الكشف بين جولات الفرز لمنع إثراء متغيرات البروتين التي ترتبط بالكاشف الثانوي.
ملاحظة: يمكن استخدام مجموعات مختلفة من الفلوروفور ، لكن Alexa Fluor 488 و Alexa Fluor 647 هما خياران جيدان لأنهما لا يتطلبان تعويضا في قياس التدفق الخلوي. لمنع استنفاد الأجسام المضادة ، يجب إضافته بزيادة متكافئة مقارنة بالعدد الإجمالي للبروتينات المعروضة في الخميرة في الأنبوب. عادة ، تستند الحسابات إلى 5 × 104 جزيئات لكل خلية خميرة. ويرد شرح أكثر تفصيلا في البروتوكولاتالسابقة 9.
- بعد الحضانة ، خلايا الطرد المركزي (2000 × جم ، 5 دقائق ، 4 درجات مئوية) وإضافة 1 مل من PBSA (التي تحتوي على الدواء / الترابط ، لمكتبة G4 المختارة من hRBP4 5 ميكرومتر من A1120) إلى الحبيبات وأجهزة الطرد المركزي مرة أخرى. قم بإزالة معظم المادة الطافية واحتفظ ب 20-30 ميكرولتر فقط لمنع الجفاف من الحبيبات.
- أعد تعليق الحبيبات في PBSA البارد فقط قبل الفرز والفرز لخلايا الخميرة التي تعرض Fn3 جزيئات بالخصائص المرغوبة (أي الارتباط ب hRBP4 في وجود A1120).
ملاحظة: تم تصوير استراتيجية البوابات في الشكل 2 أ. - قم بفرز الخلايا مباشرة إلى وسيط SD-CAA ، وأضف المزيد من وسيط SD-CAA بعد الفرز ، واحتضن عند 30 درجة مئوية مع الاهتزاز عند 180 دورة في الدقيقة.
ملاحظة: في حالة زيادة الحجم بشكل كبير بعد الفرز ، يجب إجراء خطوة طرد مركزي (2000 × جم ، 5 دقائق ، RT) لإزالة سائل الغمد الزائد ، ويتم إعادة تعليق الخلايا في وسط SD-CAA جديد. اكتب عدد الخلايا التي تم فرزها في الأنبوب الجديد ، حيث يصبح هذا الحد الأقصى الجديد للتنوع للجولة التالية من الفرز. كما هو مذكور أعلاه ، سيكون التنوع الفعلي للمكتبة أقل بسبب الإفراط في أخذ عينات من المكتبة. - كرر أنواع التدفق الخلوي لعدة جولات لرؤية إثراء متغيرات البروتين الملزمة في مكتبة الخميرة (الشكل 2 ب).
ملاحظة: قد يكون من المفيد مقارنة المكتبات جنبا إلى جنب في تجربة التدفق الخلوي التحليلي. في جولات الاختيار اللاحقة ، يمكن أن يكون خفض تركيز المستضد مفيدا في التمييز بين روابط التقارب المنخفضة والعالية والفرز خصيصا لمتغيرات البروتين التي تظهر إشارة ربط أعلى للهدف بتركيزات منخفضة (الشكل 2 ج). في جولات الاختيار اللاحقة ، من الممكن أيضا الفرز لزيادة الثبات الحراري. لهذا الغرض ، تم إجراء طفرات عشوائية للمكتبة بواسطة epPCR ، كما هو موضح في القسم 7. قم بإعداد خلايا مشابهة لجولات الفرز الخلوي الأخرى عن طريق الحث وتحديد OD600 وغسل الخلايا. ضع خلايا الخميرة في PBSA على الجليد لمدة 10 دقائق ، متبوعة بحضانة لمدة 10 دقائق في جهاز تدوير حراري للصدمة الحرارية عند درجة حرارة مرتفعة. بعد ذلك ، ضعها مرة أخرى على الجليد لمدة 10 دقائق واستمر في بروتوكول التلوين. سوف تقاوم المتغيرات المستقرة التمسخ الحراري ، وبالتالي تظهر التزاما ثابتا بعد حضانة الحرارة. يمكن اختبار درجات حرارة مختلفة بحثا عن صدمة حرارية ، اعتمادا على الثبات الحراري الأولي لبروتين النوع البري. عادة ما تكون درجات الحرارة التي تتراوح من 40-80 درجة مئوية مناسبة. ومع ذلك ، فوق 48 درجة مئوية ، ستنخفض صلاحية خلايا الخميرة بشكل كبير. لذلك ، من الضروري استخراج البلازميدات بعد فرز مكتبة الخميرة المصابة بالصدمة الحرارية ، كما تمت مناقشته في الخطوة 7.1. في هذه الحالة ، تحتاج خلايا EBY100 إلى التحويل حديثا باستخدام متغيرات غنية قبل الانتقال إلى جولة الفرزالتالية 36. يمكن العثور على بروتوكول أكثر تفصيلا لزيادة الثبات الحراري في مكان آخر37. إذا كان تمسخ البروتين المعروض قابلا للعكس ، فلن تعمل هذه الطريقة. بدلا من ذلك ، قم بتحفيز التعبير عن البروتين عند 37 درجة مئوية بدلا من 20 درجة مئوية في اليوم السابق للفرز والفرز لمتغيرات البروتين التي لا تزال تظهر عرضا فعالا كامل الطول.
7. نضوج التقارب مع epPCR لإدخال طفرات عشوائية
ملاحظة: يمكن إجراء نضوج التقارب باستخدام epPCR إما قبل جولة الفرز الخلوي الأولى للتدفق أو بين جولات الفرز الخلوي للتدفق. لاختيار مكتبة G4 مع hRBP4 في وجود A1120 ، تم إجراء نضج التقارب قبل الجولة الأولى من الفرز الخلوي للتدفق. يعتمد هذا أيضا على حجم المكتبة بعد اختيار الخرزة وإشارة الربط التي يمكن اكتشافها باستخدام قياس التدفق الخلوي. على وجه الخصوص ، في الحالات التي لا تكون فيها الصلات بعد اختيارات الخرز كافية للحصول على إشارة في تجارب قياس التدفق الخلوي (لأن المستضد ينفصل بسرعة أثناء خطوات الغسيل) ، يمكن أن يولد epPCR متغيرات محسنة يمكن اكتشافها لاحقا واختيارها عبر قياس التدفق الخلوي.
- قم بإجراء استخراج الحمض النووي للخميرة المصغرة من مكتبة الخميرة المخصبة باستخدام مجموعة البلازميد التجارية miniprep II (انظر جدول المواد) مع التعديلات التالية:
- قم بقياس كثافة الخلايا للثقافة الليلية ، وتخفيفها إلىOD 600 من 0.2 في الصباح ، واحتضانها عند 30 درجة مئوية مع الرج.
- عندما يصل OD600 إلى 0.6 ، قم بحصاد 3 مل من الثقافة عن طريق الطرد المركزي للخلايا عند 600 × جم لمدة دقيقتين.
- أضف 200 ميكرولتر من المحلول 1 إلى الحبيبات. أضف 6 ميكرولتر من Zymolyase. أعد تعليق الحبيبات عن طريق النقر أو الدوامة الخفيفة واحتضانها عند 37 درجة مئوية لمدة 3 ساعات.
- أضف 200 ميكرولتر من المحلول 2 واخلطه برفق. أضف 400 ميكرولتر من المحلول 3 واخلطه برفق.
- جهاز طرد مركزي بأقصى سرعة لمدة 3 دقائق.
- انقل المادة الطافية إلى عمود الدوران I ، وجهاز الطرد المركزي بأقصى سرعة لمدة 30 ثانية ، وتخلص من التدفق.
- يغسل ب 550 ميكرولتر من محلول الغسيل الذي يحتوي على الإيثانول وأجهزة الطرد المركزي بأقصى سرعة لمدة 2 دقيقة.
- انقل العمود إلى أنبوب طرد مركزي دقيق نظيف وجهاز طرد مركزي بأقصى سرعة لمدة دقيقتين لإزالة عازلة الغسيل المتبقية.
- انقل العمود إلى أنبوب طرد مركزي دقيق جديد ، وأضف 10 ميكرولتر من الماء الخالي من النوكلياز ، وقم بالدوران لمدة دقيقة واحدة بأقصى سرعة لتصفية الحمض النووي.
- قم بإجراء تفاعل البوليميراز المتسلسل الأولي لتضخيم قالب epPCR.
- تضخيم الحمض النووي عن طريق تفاعل البوليميراز المتسلسل باستخدام التلدين البادئاتي على السطح الخارجي للإدخال على متجه عرض سطح الخميرة.
ملاحظة: نظرا لأن الكثير من الحمض النووي للخميرة الجينومية يتم تقويته جنبا إلى جنب مع البلازميد المثير للاهتمام أثناء إعداد الخميرة الصغيرة ، يوصى بهذه الخطوة للحصول على جزء نظيف من الحمض النووي لاستخدامه كقالب ل epPCR. - بالنسبة إلى pCTCON2 ، استخدم الاشعال fwd 5'- GGCTCTGGTGGAGGCGGTAGCGGAGGCGGGGCGGAG
GGTCGGCTAGC و rev 5'- CTATTACAAGTCC
TCTTCAGAAATAAGCTTTTGTTCGGATCC مع مكونات التفاعل الموضحة في الجدول 3 وظروف ركوب الدراجات الموضحة في الجدول 4. - قم بتنقية تفاعل البوليميراز المتسلسل باستخدام مجموعة تنظيف تفاعل البوليميراز المتسلسل والحمض النووي. قم بإزالة 20 ميكرولتر من H2O الخالي من النوكلياز.
- تضخيم الحمض النووي عن طريق تفاعل البوليميراز المتسلسل باستخدام التلدين البادئاتي على السطح الخارجي للإدخال على متجه عرض سطح الخميرة.
- قم بإجراء epPCR لإدخال طفرات عشوائية.
- قم بإجراء epPCR مع نظائر النيوكليوتيدات مع المكونات المشار إليها (الجدول 5) والشروط (الجدول 6).
- قم بتشغيل منتج epPCR بالكامل على جل agarose 1٪ جاهز عند 120 فولت لمدة 45 دقيقة. قم بتضمين علامة واترك مسارا واحدا بين منتج epPCR والسلم. استئصال النطاق المطلوب المقابل للحجم المتوقع للإدخال ، وتنقيته باستخدام مجموعة استخراج الهلام ، وقياس تركيز الحمض النووي.
- تضخيم الحمض النووي العشوائي بواسطة تفاعل البوليميراز المتسلسل (PCR).
- تضخيم 50 نانوغرام من الحمض النووي باستخدام بوليميراز Q5. قم بإعداد 2 × 100 ميكرولتر من تفاعل تفاعل البوليميراز المتسلسل لكل تثقيب كهربائي (الجدول 7) مع الشروط المشار إليها (الجدول 8). استخدم نفس البادئات التي تم استخدامها في أول تفاعل البوليميراز المتسلسل (PCR).
ملاحظة: هناك حاجة إلى كميات كبيرة من الحمض النووي لتحويل الخميرة إلى ثقب كهربائي. - قم بتحليل 5 ميكرولتر من منتج PCR على جل الاغاروز 1٪ ، وقم بتشغيله بجهد 120 فولت لمدة 45 دقيقة.
- تضخيم 50 نانوغرام من الحمض النووي باستخدام بوليميراز Q5. قم بإعداد 2 × 100 ميكرولتر من تفاعل تفاعل البوليميراز المتسلسل لكل تثقيب كهربائي (الجدول 7) مع الشروط المشار إليها (الجدول 8). استخدم نفس البادئات التي تم استخدامها في أول تفاعل البوليميراز المتسلسل (PCR).
- تنقية الحمض النووي المضخم بهطول الإيثانول.
- تجمع 2 × 100 ميكرولتر معا. أضف 20 ميكرولتر (10٪ من الحجم) من أسيتات الصوديوم المعقمة المفلترة 3 M ، درجة الحموضة 5.2.
ملاحظة: يمكن تحضير هذا المخزن المؤقت مسبقا وتخزينه لبضعة أشهر في RT. - أضف 440 ميكرولتر من الإيثانول بنسبة 100٪ (أو حجم 2 × على الأقل) واحتضنه في RT لمدة دقيقتين.
- جهاز طرد مركزي عند 20,000 × جم لمدة 5 دقائق عند RT. قم بإزالة المادة الطافية ، وأضف 500 ميكرولتر من 70٪ من الإيثانول واخلطها لفترة وجيزة.
- جهاز طرد مركزي عند 20,000 × جم لمدة 5 دقائق عند RT. قم بإزالة المادة الطافية ، وأضف 500 ميكرولتر من الإيثانول بنسبة 100٪ واخلطها لفترة وجيزة.
- جهاز طرد مركزي عند 20,000 × جم لمدة 5 دقائق في RT. قم بإزالة المادة الطافية واترك حبيبات الحمض النووي تجف تحت غطاء التدفق الرقائقي حتى يتبخر كل الإيثانول.
ملاحظة: استخدم كتلة حرارية عند 48 درجة مئوية لتسريع هذه العملية. قد لا تكون حبيبات الحمض النووي مرئية في جميع الأوقات. - قم بإذابة حبيبات الحمض النووي المجففة في 10 ميكرولتر من H2O الخالي من النوكلياز.
ملاحظة: يجب إذابة الحمض النووي بأقل حجم ممكن. لا تقيس تركيز الحمض النووي.
- تجمع 2 × 100 ميكرولتر معا. أضف 20 ميكرولتر (10٪ من الحجم) من أسيتات الصوديوم المعقمة المفلترة 3 M ، درجة الحموضة 5.2.
| الحجم [μL] | التركيز النهائي | |
| 5x Q5 محسن | 10 | 1x |
| 5x المخزن المؤقت Q5 | 10 | 1x |
| برايمر fwd 10 ميكرومتر | 2.5 | 0.5 ميكرومتر |
| التمهيدي rev 10 ميكرومتر | 2.5 | 0.5 ميكرومتر |
| dNTPs 10 ملي مولار | 1 | 200 ميكرومتر |
| البوليميراز Q5 | 0.5 | 20 وحدة / مل |
| الحمض النووي من الخميرة miniprep | 10 | |
| H2O خالي من النوكلياز | 13.5 |
الجدول 3: شروط الخطوة الأولى PCR لتضخيم جينات POI من الخميرة المعزولة.
| درج | درجة الحرارة | الوقت |
| التمسخ الأولي | 98 درجة مئوية | 30 ثانية |
| 25 دورة | 98 درجة مئوية | 10 ثانية |
| 72 درجة مئوية | 30 ثانية | |
| 72 درجة مئوية | 30 ثانية | |
| التمديد النهائي | 72 درجة مئوية | 2 دقيقة |
| مسك | 4 درجة مئوية |
الجدول 4: ظروف ركوب الدراجاتلتفاعل البوليميراز المتسلسل للخطوة الأولى لتضخيم جينات POI من الخميرة الصغيرة المعزولة.
| الحجم [μL] | التركيز النهائي | |
| H2O خالي من النوكلياز | حتى 50 | |
| 10x Thermopol عازلة | 5 | 1x |
| Primer_fwd (10 ميكرومتر) | 2.5 | 0.5 ميكرومتر |
| Primer_rev (10 ميكرومتر) | 2.5 | 0.5 ميكرومتر |
| dNTPs (10 ملليمتر) | 1 | 200 ميكرومتر |
| 8-أوكسو-dGTP (100 ميكرومتر) | 1 | 2 ميكرومتر |
| dPTP (100 ميكرومتر) | 1 | 2 ميكرومتر |
| منتج PCR من 1st PCR | XX | 50 نانوغرام |
| بوليميراز الحمض النووي Taq | 0.5 | 0.05 وحدة / ميكرولتر |
الجدول 5: شروط epPCR الذي يتم إجراؤه بعد تضخيم الحمض النووي POI باستخدام PCRالخطوة 1 .
| درج | درجة الحرارة | الوقت |
| التمسخ الأولي | 94 درجة مئوية | 30 ثانية |
| 15 دورة | 94 درجة مئوية | 45 ثانية |
| 60 درجة مئوية | 30 ثانية | |
| 72 درجة مئوية | 1 دقيقة | |
| التمديد النهائي | 72 درجة مئوية | 10 دقائق |
| مسك | 4 درجة مئوية |
الجدول 6: ظروف ركوب الدراجات لجهاز epPCR.
| الحجم [μL] | التركيز النهائي | |
| 5x Q5 محسن | 20 | 1x |
| 5x المخزن المؤقت Q5 | 20 | 1x |
| برايمر fwd 10 ميكرومتر | 5 | 0.5 ميكرومتر |
| التمهيدي rev 10 ميكرومتر | 5 | 0.5 ميكرومتر |
| dNTPs 10 ملي مولار | 1 | 200 ميكرومتر |
| البوليميراز Q5 | 1 | 20 وحدة / مل |
| 50 نانوغرام الحمض النووي | XX | |
| ddH20 | حتى 100 |
الجدول 7: شروط الخطوة الثانية من تفاعل البوليميراز المتسلسل لتضخيم منتج epPCR قبل التثقيب الكهربائي لخلايا EBY100.
| درج | درجة الحرارة | الوقت |
| التمسخ الأولي | 98 درجة مئوية | 30 ثانية |
| 25 دورة | 98 درجة مئوية | 10 ثانية |
| 72 درجة مئوية | 30 ثانية | |
| 72 درجة مئوية | 30 ثانية | |
| التمديد النهائي | 72 درجة مئوية | 2 دقيقة |
| مسك | 4 درجة مئوية |
الجدول 8: شروط ركوب الدراجاتلتفاعل البوليميراز المتسلسل من الخطوة الثانية لتضخيم منتج epPCR.
8. الخطية لناقل عرض الخميرة للتثقيب الكهربائي
- خطي متجه عرض سطح الخميرة pCTCON2 (بلازميد Addgene # 41843).
- قم بإجراء ملخص واسع النطاق واستخدم هذا الناقل الخطي المهضوم لجميع عمليات التثقيب الكهربائي الإضافية.
- هضم 2 × 200 ميكروغرام من الحمض النووي البلازميد pCTCON2 مع Sal I لمدة 24 ساعة عند 37 درجة مئوية للخطية الأولية (الجدول 9).
- ملخص مع NheI و BamHI لمدة 24 ساعة عند 37 درجة مئوية لاستئصال الملحق (الجدول 10).
- أخيرا ، أضف 5 ميكرولتر من كل إنزيم إلى الأنبوب (Sal I ، NheI ، BamHI) واحتضنه مرة أخرى لمدة 24 ساعة عند 37 درجة مئوية.
- قم بتشغيل المتجه المهضوم على هلام الاغاروز التحضيري. استئصال الأربطة وتنقيتها باستخدام مجموعة استخراج الجل. قم بإزالة المتجه في 20 ميكرولتر ، وقم بقياس تركيز الحمض النووي وضبطه على 2 ميكروغرام / ميكرولتر تقريبا.
ملاحظة: يضمن هضم البلازميد لفترة طويلة انقساما كافيا للإدخال وسيؤدي إلى خلفية أقل من المتجه بعد التثقيب الكهربائي.
| دنا | 200 ميكروغرام |
| 10x CutSmartBuffer | 50 ميكرولتر |
| سال آي إتش إف (نب إي) | 30 ميكرولتر (60 وحدة واحدة) |
| ح2س | ما يصل إلى 500 ميكرولتر |
الجدول 9: شروط الخطوة الأولى من الملخص الواسع النطاق لمتجه عرض سطح الخميرة pCTCON2.
| pCTCON2 (هضم Sal) | 500 ميكرولتر |
| 10x CutSmartBuffer | 37.5 ميكرولتر |
| NheI-HF (NEB) | 15 ميكرولتر (30 وحدة واحدة) |
| BamHI-HF (NEB) | 15 ميكرولتر (30 وحدة واحدة) |
| ح2س | حتى 875 ميكرولتر |
الجدول 10: شروط الخطوة الثانية من الملخص واسع النطاق لمتجه عرض سطح الخميرة pCTCON2.
9. التثقيب الكهربائي ل EBY100 مع الحمض النووي العشوائي والناقل الخطي
- قم بإعداد خلايا الخميرة EBY100 (ATCC ، انظر جدول المواد) عن طريق ضربها من مخزون الجلسرين قبل 3 أيام من التثقيب الكهربائي. نظرا لأن خلايا EBY100 غير المحولة لا تحتوي على متجه عرض سطح الخميرة ، استخدم لوحات YPD للوسائط الكاملة (الجدول 1). احتضان عند 30 درجة مئوية.
- قبل يوم واحد من التثقيب الكهربائي ، قم بتلقيح مستعمرة واحدة من EBY100 في 30 مل من وسط YPD المحضر حديثا (الجدول 1). احتضن طوال الليل عند 30 درجة مئوية مع الاهتزاز عند 180 دورة في الدقيقة.
ملاحظة: يجب ألا يزيد عمر وسيط YPD عن شهر واحد. - في صباح اليوم التالي ، قم بتخفيف استزراع EBY100 بين عشية وضحاها إلى OD600 من 0.2 مل في 100 مل من وسط YPD واحتضانه عند 30 درجة مئوية أثناء الاهتزاز عند 180 دورة في الدقيقة.
ملاحظة: حجم ثقافة 50 مل يكفي ل2 التثقيب الكهربائي. عادة ما يتم تحضير 100 مل للحصول على خلايا كافية للعناصر التحكم (الخلايا فقط والخلايا بالإضافة إلى الناقل). - عند الوصول إلى OD600 من 1.3-1.5 ، قم بتكبيل الخلايا في أنابيب سعة 50 مل عند 2000 × جم لمدة 3 دقائق وقم بإزالة المادة الطافية.
- أعد تعليق الحبيبات في 25 مل من أسيتات الليثيوم المصفاة المعقمة 100 ملي مولار (انظر جدول المواد). أضف DTT المعقم المحضر حديثا (1 م) (انظر جدول المواد) إلى التركيز النهائي البالغ 10 مليمتر.
ملاحظة: يجب أن يكون حجم أسيتات الليثيوم نصف الحجم الأصلي ل YPD. قم بتقليص حجمها أو تصغيرها إذا لزم الأمر. يمكن تحضير أسيتات الليثيوم (100 مليمتر) مسبقا وتخزينها لبضعة أشهر في RT ، ولكن يجب تحضير مخزون 1M DTT قبل الاستخدام مباشرة وحفظه على الجليد. - احتضان الخلايا عند 30 درجة مئوية أثناء الرج لمدة 10 دقائق.
- جهاز طرد مركزي عند 2000 × جم لمدة 3 دقائق ، وتخلص من المادة الطافية ، وضع الخلايا على الجليد.
ملاحظة: يجب تنفيذ جميع الخطوات التالية على الجليد ومع الكواشف المبردة ، والكوفات ، وأنابيب الطرد المركزي الدقيقة. - أعد تعليق الحبيبات في 25 مل من أجهزة الطرد المركزي H2O. المعقمة الباردة عند 2000 × جم لمدة 3 دقائق.
ملاحظة: يجب أن يكون حجم H2O المستخدم نصف وحدة تخزين YPD الأصلية. - يعلق في 350 ميكرولتر من H2O.
ملاحظة: يجب أن يكون الحجم الإجمالي بعد إعادة التعليق 500 ميكرولتر. أضف المزيد من H2O إذا لزم الأمر. - قم بإعداد مكتبة الحمض النووي بالتوازي: ضع أنابيب الطرد المركزي الدقيقة التي تحتوي على إدخالات منقاة بالإيثانول على الجليد وأضف 4 ميكروغرام من ناقل pCTCON2 الخطي.
- أضف 250 ميكرولتر من الخلايا إلى الحمض النووي واخلطها برفق عن طريق القص. انقله إلى كوفيت كهربائي مبرد مسبقا مقاس 2 مم واتركه على الثلج حتى التثقيب الكهربائي.
ملاحظة: أضف عناصر التحكم (الخلايا فقط بدون الحمض النووي والخلايا ذات المتجه الخطي فقط بدون إدراج) لتحديد نجاح التثقيب الكهربائي. - التثقيب الكهربائي باستخدام كوفيتات التثقيب الكهربائي 2 مم (انظر جدول المواد) بالشروط التالية: بروتوكول الموجة المربعة ، نبضة واحدة ، 500 فولت ، مدة النبضة 15 مللي ثانية.
ملاحظة: التدلي النموذجي هو 3٪ - 9٪. يمكن استخدام المنافذ الكهربائية الأخرى ، ولكن تم تحسين الشروط المشار إليها للأداة المستخدمة في هذه الدراسة. - أنقذ الخلايا ب 1 مل من YPD الدافئ مسبقا ، ونقلها إلى أنبوب طرد مركزي دقيق مسخن مسبقا ، واحتضانها دون اهتزاز لمدة ساعة واحدة عند 30 درجة مئوية.
ملاحظة: ضع كتلة التسخين بجوار المثقب الكهربائي لضمان بقاء أنابيب الاسترداد في درجة الحرارة المناسبة. - لوحة 100 ميكرولتر من 10-3 ، 10-4 ، 10-5 ، و 10-6 تخفيفات من الخلايا على ألواح SD-CAA (انظر الجدول 1) وتسمح بنمو المحولات عند 30 درجة مئوية.
ملاحظة: يتم الحصول على تنوع المكتبة النظرية عن طريق حساب عدد المحولات إلى حجم الثقافة الأصلي ، هنا 1 مل. عادة ما ينتج عن بروتوكول التحويل هذا ما يقرب من 107-10 8 خلايا تحويلية. - قم بتكبيل الخلايا المتبقية عند 2000 × جم لمدة 3 دقائق ، وأعد تعليقها في 200 مل SD-CAA ، واحتضانها عند 30 درجة مئوية مع الرج عند 180 دورة في الدقيقة بين عشية وضحاها. يمكن إما تمرير الخلايا المحولة التي تحتوي على مكتبة epPCR العشوائية في SD-CAA وتحفيزها للتعبير السطحي في SG-CAA في اليوم التالي أو تخزينها عند 4 درجات مئوية حتى الاستخدام الآخر. تجميد مخزون الجلسرين في المكتبة بعد epPCR بعد تحديد التنوع. نظرا لأنه في اليوم التالي ، سيظل جزء من الخلايا عبارة عن خلايا غير متحولة تم نقلها في إجراء التحويل دون الحصول على بلازميد ، فمن المستحسن تمرير ما لا يقل عن 20 ضعفا من تنوع المكتبة في خطوة التمرير الأولى للتعويض عن هذا التأثير.
10. تسلسل مكتبات الخميرة بعد عدة جولات اختيار
- استخرج الحمض النووي للخميرة عن طريق إجراء تحضير صغير للخميرة كما هو موضح أعلاه.
- التثقيب الكهربائي التجاري بكتريا قولونية 10-بيتا الخلايا ذات الكفاءة الكهربائية (انظر جدول المواد) مع الحمض النووي المعزول.
- ضع كوفيت التثقيب الكهربائي (1 مم ، انظر جدول المواد) وأنابيب الطرد المركزي الدقيقة على الجليد. أنابيب الطرد المركزي الدقيقة الدافئة مسبقا مع 950 ميكرولتر من وسط النمو عند 37 درجة مئوية لاستعادة الخلايا بعد التثقيب الكهربائي.
- قم بإذابة الخلايا على الثلج وتخلط عن طريق تحريك الأنبوب برفق. انقل 50 ميكرولتر من الخلايا إلى أنبوب طرد مركزي دقيق مبرد. أضف 1 ميكرولتر من الحمض النووي المصغر للخميرة.
- انقل مزيج الحمض النووي للخلية بعناية إلى كوفيت كهربائي مبرد.
ملاحظة: تجنب الفقاعات وتأكد من ترسب الخلايا في قاع الكوفيت. - التثقية الكهربائية بجهاز التثقيب الكهربائي باستخدام الشروط التالية: 2.0 كيلو فولت ؛ 200 Ω; 25 ميكرو فهرنهايت.
ملاحظة: ثوابت الوقت النموذجية هي 4.8-5.1 مللي ثانية. يمكن استخدام المنافذ الكهربائية الأخرى ، ولكن تم تحسين الشروط المشار إليها للأداة المستخدمة في هذه الدراسة. - أضف على الفور 950 ميكرولتر من وسط النمو الدافئ مسبقا 37 درجة مئوية (انظر جدول المواد) إلى الكوفيت ، واخلطه برفق ، ثم انقله مرة أخرى إلى أنابيب الطرد المركزي الدقيقة الدافئة.
ملاحظة: ضع كتلة التسخين بجوار المثقب الكهربائي لضمان بقاء أنابيب الاسترداد في درجة الحرارة المناسبة. - احتضن لمدة 40 دقيقة عند 37 درجة مئوية و 300 دورة في الدقيقة. لوحة 10 ميكرولتر ، 100 ميكرولتر ، وبقية الخلايا (بعد الطرد المركزي) على لوحة LB انتقائية مسخنة مسبقا (انظر الجدول 1). احتضان الألواح طوال الليل عند 37 درجة مئوية.
ملاحظة: بالنسبة لناقل pCTCON2 ، يتم استخدام الأمبيسلين في اختيار المضادات الحيوية. - قم بإعداد لوحة نسخ مع LB أجار والمضاد الحيوي المطلوب (هنا يستخدم الأمبيسلين): ماصة 200 ميكرولتر من LB أجار مع 100 ميكروغرام / مل أمبيسلين في كل بئر من صفيحة 96 بئر واتركها تتصلب.
- قم بإعداد لوحة تسلسل 96 بئر مع 50 ميكرولتر من LB مع 100 ميكروغرام / مل أمبيسلين لكل بئر.
- اختر مستعمرة واحدة من الألواح ، وقم أولا بتلقيح لوحة النسخ ونفس البئر من لوحة التسلسل.
ملاحظة: تضمن لوحة النسخ إمكانية الحصول على البلازميدات المطلوبة بسهولة عن طريق تلقيح مزرعة سائلة وإجراء عملية تحضير صغيرة. - احتضن لوحة النسخ طوال الليل عند 37 درجة مئوية ثم قم بتخزينها في 4 درجات مئوية.
- احتضان لوحة التسلسل لمدة 3-4 ساعات عند 37 درجة مئوية أثناء الرج (180 دورة في الدقيقة) مغطاة بورق بلاستيكي أو ألومنيوم.
- أغلق لوحة التسلسل وأرسلها للتسلسل مع 500 ميكرولتر من 10 ميكرومتر pCTCON2 التمهيدي (5'-CGTTTGTCAGTATTGCGGTTCTC).
- لتحليل التسلسلات ، قم بترجمة تسلسل الحمض النووي الذي تم الحصول عليه إلى تسلسلات الأحماض الأمينية باستخدام برنامج مناسب من الاختيار ، على سبيل المثال ، EMBOSS Transeq38. بعد ذلك ، استخدم محاذاة التسلسل المتعدد (MUSCLE) 39 أو أداة محاذاة مماثلة لمحاذاة التسلسلات المتغيرة مع بعضها البعض ومع بروتين النمط البري الأبوي.
ملاحظة: من خلال إجراء تسلسل لمكتبات جولات الاختيار المختلفة (على سبيل المثال، بعد جولة سابقة من الانتقاء، وكذلك بعد جولات إضافية من الانتقاء بتركيزات مستضد أقل لإثراء المتغيرات عالية التقارب)، من الممكن تحديد المتغيرات المختلفة. يعتمد هذا بشكل كبير على صرامة البوابات المطبقة أثناء جولات الفرز ، وبالتالي تنوع المكتبات.
النتائج
تم اختيار مكتبة G4 مقابل المستضد hRBP4 المرتبط بعقار الجزيء الصغير A1120. تم إجراء تلطيخ المكتبات لفرز التدفق الخلوي كما هو موضح في الطريقة 6 ، وتظهر استراتيجية البوابات المطبقة في الشكل 2 أ. تضمنت البوابة الأولية جميع الخلايا بناء على مورفولوجيا الخلية ، وأظهرت البوابة الثانية (الرسم البياني ل FSC-Width) استراتيجية بوابة صارمة تم تطبيقها لتحديد الخلايا المفردة وإزالة مجاميع الخلايا. أظهرت البوابة الثالثة والأخيرة عرض متغيرات البروتين (المحور السيني) مقابل ارتباط المستضد (المحور الصادي). تم فرز خلايا الخميرة التي تظهر إشارات العرض والربط. الأهم من ذلك ، تم تعيين بوابة الفرز بطريقة صارمة لإثراء مجالات الربط بإشارة ربط عالية وبالتالي تقارب عالي. أسفر هذا الاختيار الصارم عن إثراء عرض خلايا الخميرة التي ترتبط على وجه التحديد بالمستضد المستهدف طوال حملة الاختيار (الشكل 2 ب). في جولات الفرز الخلوي اللاحقة ، انخفض تركيز المستضد بمقدار 10 أضعاف (من 100 نانومتر إلى 10 نانومتر). لذلك ، تم تقليل إشارة الربط الإجمالية ، ولا تزال المواد المجلدة ذات التقارب العالي فقط قابلة للاكتشاف والفرز (الشكل 2 ج).
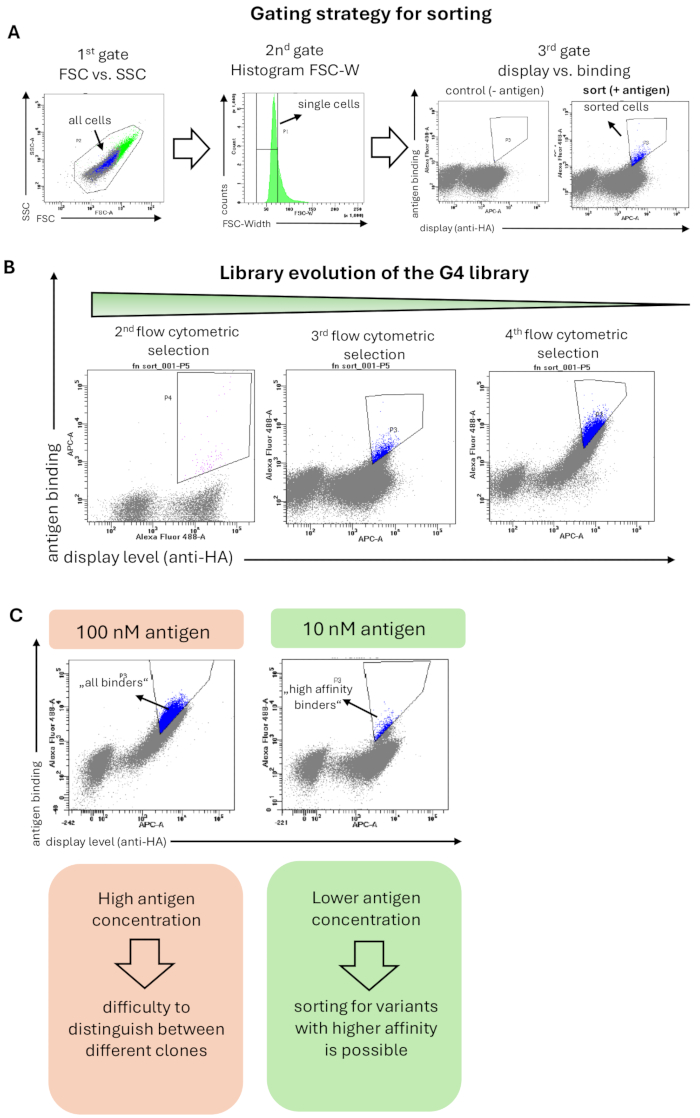
الشكل 2: النتائج التمثيلية من اختيار عرض سطح الخميرة لمكتبة G4 المستندة إلى Fn3 للارتباط بالمستضد (hRBP4 في وجود A1120). (أ) استراتيجية البوابات العامة لفرز مكتبات الخميرة. البوابة الأولى (FSC مقابل SSC) هي تحديد جميع خلايا الخميرة واستبعاد أحداث التشتت. تهدف البوابة الثانية (الرسم البياني ل FSC-W) إلى إزالة مجاميع الخلايا وتحديد خلايا الخميرة المفردة فقط. ترسم البوابة الثالثة مستوى عرض السطح (اكتشاف علامة HA- أو c-myc) مقابل الارتباط بالمستضد (هنا hRBP4 في وجود 5 ميكرومتر A1120 ، تم اكتشافه بواسطة الجسم المضاد له). بالإضافة إلى ذلك ، كانت المكتبة ملطخة بالأجسام المضادة الثانوية فقط (بدون مستضد) ، حيث لا يتوقع ارتباط المستضد. يتم تمييز الخلايا التي تم فرزها باللون الأزرق. (ب) تطور مكتبة G4 خلال 3 جولات من الفرز الخلوي للتدفق. يمكن ملاحظة إثراء السكان الملزمين مع كل جولة اختيار. (ج) يتيح استخدام تركيزات مستضد أقل اختيار متغيرات البروتين ذات التقارب الأكبر تجاه المستضد المستهدف. عند تقليل تركيز المستضد (هنا hRBP4) بمقدار 10 أضعاف ، تظهر أقطار مختلفة ، مما يشير إلى وجود نسخ ذات تقارب أعلى (خلايا مصنفة ، أزرق) أو أقل. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
Discussion
تطور عرض سطح الخميرة كإحدى الطرق الرئيسية المستخدمة في هندسة البروتين. على الرغم من أنه يستخدم بشكل شائع لهندسة التقارب1،18،40،41 ، والتعبير / الاستقرار24،27،42،43 والنشاط28،44 ، إلا أنه يستخدم أيضا مثل رسم خرائط الحاتمة45،46 أو توصيف الطفرات الفردية على سطح خلايا الخميرة9 ممكنة أيضا. في هذا البروتوكول ، نقدم الخطوات الأساسية لبدء حملة اختيار عرض سطح الخميرة ، بما في ذلك الاختيار بالخرز المغناطيسي وعن طريق الفرز الخلوي للتدفق بالإضافة إلى تنويع مكتبة الخميرة بواسطة epPCR لنضج التقارب.
أحد المتطلبات الأساسية لاختيارات عرض سطح الخميرة التقليدية هو توافر البروتين القابل للذوبان بجودة كافية. بدءا من بروتين مستهدف مطوي جيدا مع نقاء عال وحالة قلة القلة المحددة (على سبيل المثال ، يجب أن يكون البروتين الأحادي موجودا فقط كمونومر) يوفر أعلى معدل نجاح لاختيار متغير البروتين المرتبط بالمستضد المستهدف ذو التقارب العالي. بديل للبروتينات المستهدفة التي يصعب التعبير عنها هو الاختيارات القائمة على الخلايا ، والتي تقدم استراتيجية معقولة للتحايل على هذا القيد47. ومع ذلك ، فإن عرض سطح الخميرة يوفر العديد من المزايا ، مثل إمكانية توصيف متغيرات البروتين الناتجة مباشرة على سطح الخميرة دون الحاجة إلى إجراء استنساخ شاق ويستغرق وقتا طويلا ، والتعبير في شكل قابل للذوبان ، وتنقية البروتين. يمكن تحليل كل من تقارب واستقرار المتغيرات مباشرة على سطح الخميرة9.
في هذا البروتوكول ، نوضح كيف تم اختيار مكتبة G4 لمتغيرات البروتين ، وبشكل أكثر تحديدا من المجالالعاشر من النوع الثالث من الفيبرونكتين البشري ، للارتباط بالمستضد hRBP4 في وجود الجزيء الصغير A1120. أدى الجمع بين اختيارات الخرز والفرز الخلوي للتدفق إلى إثراء المتغيرات ، مما أظهر زيادة في الارتباط بالمستضد المستهدف خلال جولات الاختيار (الشكل 2 ب). أظهرنا أن استخدام تركيزات أقل من المستضد يتيح اختيار متغيرات البروتين عالية التقارب (الشكل 2 ج). عادة ما تكون الصلات التي يمكن تحقيقها من خلال تحديدات عرض الخميرة في نطاق nanomolar أو حتى picomolar18. تعتمد الصلات النهائية على المستضد المستهدف ، وعدد جولات الاختيار ونضج التقارب ، وسقالة الربط المستخدمة ، واستراتيجية البوابات المطبقة. لم يتم تغطية توصيف متغيرات البروتين الفردية في هذا البروتوكول ولكن تم شرحه بالتفصيل في عملنا السابق9. على الرغم من أن عرض الخميرة تم استخدامه في الأصل لهندسة شظايا الأجسام المضادة مثل scFvs1،40 ، فقد تم استخدام الطريقة على نطاق واسع للبروتينات غير القائمة على الأجسام المضادة وكذلك10.
باختصار ، يعد عرض سطح الخميرة أداة قوية لهندسة البروتين تتيح توليد متغيرات البروتين ذات الخصائص الجديدة أو المحسنة ، مثل الارتباط بأي بروتين مستهدف تقريبا و / أو زيادة الاستقرار.
Disclosures
تتلقى MWT تمويلا من Miltenyi Biotec. جميع المؤلفين مخترعون في طلبات براءات الاختراع للتقنيات والبروتينات الهندسية التي تم تطويرها باستخدام عرض سطح الخميرة.
Acknowledgements
تم دعم هذا العمل من قبل الصندوق النمساوي للعلوم (FWF Project W1224 - برنامج الدكتوراه حول التكنولوجيا الجزيئية الحيوية للبروتينات - BioToP ومشروع FWF ESP 465-B) ، والوزارة الفيدرالية للشؤون الرقمية والاقتصادية في النمسا ، والمؤسسة الوطنية للبحث والتكنولوجيا والتنمية في النمسا إلى الجمعية المسيحية لأبحاث دوبلر (مختبر دوبلر المسيحي للجيل القادم من الخلايا التائية CAR) ، ومن خلال التبرعات الخاصة لمعهد سانت آنا لأبحاث سرطان الأطفال (فيينا ، النمسا). حصلت E.S. على زمالة DOC من الأكاديمية النمساوية للعلوم في معهد سانت آنا لأبحاث سرطان الأطفال. تم إنشاء الأرقام باستخدام BioRender.com.
Materials
| Name | Company | Catalog Number | Comments |
| 10-beta electrocompetent E. coli | NEB | C3020K | |
| Agar-Agar, Kobe I | Carl Roth | 5210.5 | |
| Ampicillin sodium salt | Carl Roth | K029.2 | |
| Anti-c-myc antibody, clone 9E10, AF488 | Invitrogen | MA1-980-A488 (Thermo Fisher) | |
| Anti-c-myc antibody, clone 9E10, AF647 | Invitrogen | MA1-980-A647 (Thermo Fisher) | |
| Anti-HA antibody, clone 16B12, AF488 | BioLegend | 901509 (Biozym) | |
| Anti-HA antibody, clone 16B12, AF647 | BioLegend | 682404 (Biozym) | |
| BamHI-HF | NEB | R3136S | |
| Bovine serum albumin, cold ethanol fraction | Sigma-Aldrich | A4503 | |
| Citric acid monohydrate | Sigma-Aldrich | C1909 | |
| D-Galactose | Carl Roth | 4987.2 | |
| D-Glucose | Sigma-Aldrich | G8270 | |
| Difco yeast nitrogen base | Becton Dickinson (BD) | 291940 | |
| Di-Sodium hydrogen phosphate heptahydrate | Carl Roth | X987.3 | |
| DL-Dithiothreitol | Sigma-Aldrich | D0632 | |
| D-Sorbitol | Carl Roth | 6213.1 | |
| Dulbecco’s phosphate buffered saline (10x) | Thermo Scientific | 14190169 | |
| Dynabeads Biotin Binder | Invitrogen | 11047 (Fisher Scientific) | |
| DynaMag-2 Magnet | Thermo Fisher | 12321D | |
| EBY100 | ATCC | MYA-4941 | |
| Electroporation cuvette 1 mm (for E.coli) | VWR | 732-1135 | |
| Electroporation cuvette 2 mm (for yeast) | VWR | 732-1136 | |
| Ethanol absolute | MERCK | 1070172511 | |
| GeneMorph II Random Mutagenesis Kit | Agilent Technologies | 200550 | |
| Gibco Bacto Casamino Acids | Becton Dickinson (BD) | 223120 | |
| Glycerol | AppliChem | 131339.1211 | |
| LE agarose | Biozym | 840004 | |
| Lithium acetate dihydrate | Sigma-Aldrich | L4158 | |
| Monarch DNA Gel Extraction Kit | NEB | T1020S | |
| Monarch PCR & DNA Cleanup Kit | NEB | T1030S | |
| Multifuge 1S-R | Heraeus | ||
| NheI-HF | NEB | R3131S | |
| Outgrowth medium | NEB | B9035S | |
| pCTCON2 | Addgene | #41843 | |
| Penicillin G sodium salt | Sigma-Aldrich | P3032 | |
| Penta-His antibody, AF488 | Qiagen | 35310 | |
| Penta-His antibody, AF647 | Qiagen | 35370 | |
| Peptone ex casein tryptically digested | Carl Roth | 8986.3 | |
| Q5 High-Fidelity DNA Polymerase | NEB | M0491S | |
| SaII-HF | NEB | R3138S | |
| Sodium acetate | Sigma-Aldrich | S8750-1KG | |
| Sodium chloride | Carl Roth | 3957.2 | |
| Sodium dihydrogen phosphate monohydrate | Carl Roth | K300.2 | |
| Steritop threaded bottle top filter | MERCK | S2GPT01RE | |
| Streptavidin, AF488 | Invitrogen | S32354 (Thermo Fisher) | |
| Streptavidin, AF647 | Invitrogen | S32357 (Thermo Fisher) | |
| Streptomycin sulfate | Sigma-Aldrich | S6501 | |
| Tri-Sodium citrate dihydrate | Carl Roth | 4088.1 | |
| UV-Vis spectrophotometer | Agilent | 8453 | |
| Yeast extract, micro-granulated | Carl Roth | 2904.4 | |
| Zymoprep Yeast Plasmid Miniprep II | Zymo Research | D2004 |
References
- Boder, E. T., Wittrup, K. D. Yeast surface display for screening combinatorial polypeptide libraries. Nat Biotechnol. 15 (6), 553-557 (1997).
- Scott, J. K., Smith, G. P. Searching for peptide ligands with an epitope library. Science. 249 (4967), 386-390 (1990).
- Smith, G. P. Filamentous fusion phage: novel expression vectors that display cloned antigens on the virion surface. Science. 228 (4705), 1315-1317 (1985).
- Schaffitzel, C., Hanes, J., Jermutus, L., Pluckthun, A. Ribosome display: an in vitro method for selection and evolution of antibodies from libraries. J Immunol Methods. 231 (1-2), 119-135 (1999).
- Ho, M., Nagata, S., Pastan, I. Isolation of anti-CD22 Fv with high affinity by Fv display on human cells. Proc Natl Acad Sci U S A. 103 (25), 9637-9642 (2006).
- Wagner, A., et al. Identification of activating mutations in the transmembrane and extracellular domains of EGFR. Biochemistry. 61 (19), 2049-2062 (2022).
- Wagner, A., et al. PhosphoFlowSeq - A high-throughput kinase activity assay for screening drug resistance mutations in EGFR. J Mol Biol. 433 (22), 167210(2021).
- Cherf, G. M., Cochran, J. R. Applications of yeast surface display for protein engineering. Methods Mol Biol. 1319, 155-175 (2015).
- Zajc, C. U., Teufl, M., Traxlmayr, M. W. Affinity and stability analysis of yeast displayed proteins. Methods Mol Biol. 2491, 155-173 (2022).
- Gai, S. A., Wittrup, K. D. Yeast surface display for protein engineering and characterization. Curr Opin Struct Biol. 17 (4), 467-473 (2007).
- Orr, B. A., Carr, L. M., Wittrup, K. D., Roy, E. J., Kranz, D. M. Rapid method for measuring ScFv thermal stability by yeast surface display. Biotechnol Prog. 19 (2), 631-638 (2003).
- Teufl, M., Zajc, C. U., Traxlmayr, M. W. Engineering strategies to overcome the stability-function trade-off in proteins. ACS Synth Biol. 11 (3), 1030-1039 (2022).
- Wozniak-Knopp, G., et al. Introducing antigen-binding sites in structural loops of immunoglobulin constant domains: Fc fragments with engineered HER2/neu-binding sites and antibody properties. Protein Eng Des Sel. 23 (4), 289-297 (2010).
- Traxlmayr, M. W., et al. Directed evolution of stabilized IgG1-Fc scaffolds by application of strong heat shock to libraries displayed on yeast. Biochim Biophys Acta. 1824 (4), 542-549 (2012).
- Traxlmayr, M. W., et al. Directed evolution of Her2/neu-binding IgG1-Fc for improved stability and resistance to aggregation by using yeast surface display. Protein Eng Des Sel. 26 (4), 255-265 (2013).
- Hasenhindl, C., et al. Stability assessment on a library scale: a rapid method for the evaluation of the commutability and insertion of residues in C-terminal loops of the CH3 domains of IgG1-Fc. Protein Eng Des Sel. 26 (10), 675-682 (2013).
- Koide, A., Wojcik, J., Gilbreth, R. N., Hoey, R. J., Koide, S. Teaching an old scaffold new tricks: monobodies constructed using alternative surfaces of the FN3 scaffold. J Mol Biol. 415 (2), 393-405 (2012).
- Hackel, B. J., Kapila, A., Wittrup, K. D. Picomolar affinity fibronectin domains engineered utilizing loop length diversity, recursive mutagenesis, and loop shuffling. J Mol Biol. 381 (5), 1238-1252 (2008).
- Traxlmayr, M. W., et al. Strong enrichment of aromatic residues in binding sites from a charge-neutralized hyperthermostable Sso7d scaffold library. J Biol Chem. 291 (43), 22496-22508 (2016).
- Zajc, C. U., et al. A conformation-specific ON-switch for controlling CAR T cells with an orally available drug. Proc Natl Acad Sci U S A. 117 (26), 14926-14935 (2020).
- Moore, S. J., Cochran, J. R. Engineering knottins as novel binding agents. Methods Enzymol. 503, 223-251 (2012).
- Hackel, B. J., Ackerman, M. E., Howland, S. W., Wittrup, K. D. Stability and CDR composition biases enrich binder functionality landscapes. J Mol Biol. 401 (1), 84-96 (2010).
- Hackel, B. J., Wittrup, K. D. The full amino acid repertoire is superior to serine/tyrosine for selection of high affinity immunoglobulin G binders from the fibronectin scaffold. Protein Eng Des Sel. 23 (4), 211-219 (2010).
- Laurent, E., et al. Directed evolution of stabilized monomeric CD19 for monovalent CAR interaction studies and monitoring of CAR-T cell patients. ACS Synth Biol. 10 (5), 1184-1198 (2021).
- Julian, M. C., et al. Co-evolution of affinity and stability of grafted amyloid-motif domain antibodies. Protein Eng Des Sel. 28 (10), 339-350 (2015).
- Cochran, J. R., Kim, Y. S., Lippow, S. M., Rao, B., Wittrup, K. D. Improved mutants from directed evolution are biased to orthologous substitutions. Protein Eng Des Sel. 19 (6), 245-253 (2006).
- Kieke, M. C., et al. High affinity T cell receptors from yeast display libraries block T cell activation by superantigens. J Mol Biol. 307 (5), 1305-1315 (2001).
- Lipovsek, D., et al. Selection of horseradish peroxidase variants with enhanced enantioselectivity by yeast surface display. Chem Biol. 14 (10), 1176-1185 (2007).
- Fushimi, T., et al. Mutant firefly luciferases with improved specific activity and dATP discrimination constructed by yeast cell surface engineering. Appl Microbiol Biotechnol. 97 (9), 4003-4011 (2013).
- Lam, S. S., et al. Directed evolution of APEX2 for electron microscopy and proximity labeling. Nat Methods. 12 (1), 51-54 (2015).
- Park, S. W., Lee, D. S., Kim, Y. S. Engineering a U-box of E3 ligase E4B through yeast surface display-based functional screening generates a variant with enhanced ubiquitin ligase activity. Biochem Biophys Res Commun. 612, 147-153 (2022).
- Schutz, M., et al. Directed evolution of G protein-coupled receptors in yeast for higher functional production in eukaryotic expression hosts. Sci Rep. 6, 21508(2016).
- Angelini, A., et al. Protein engineering and selection using yeast surface display. Methods Mol Biol. 1319, 3-36 (2015).
- Chen, T. F., de Picciotto, S., Hackel, B. J., Wittrup, K. D. Engineering fibronectin-based binding proteins by yeast surface display. Methods Enzymol. 523, 303-326 (2013).
- Shusta, E. V., Holler, P. D., Kieke, M. C., Kranz, D. M., Wittrup, K. D. Directed evolution of a stable scaffold for T-cell receptor engineering. Nat Biotechnol. 18 (7), 754-759 (2000).
- Traxlmayr, M. W., Obinger, C. Directed evolution of proteins for increased stability and expression using yeast display. Arch Biochem Biophys. 526 (2), 174-180 (2012).
- Traxlmayr, M. W., Shusta, E. V. Directed evolution of protein thermal stability using yeast surface display. Methods Mol Biol. 1575, 45-65 (2017).
- Madeira, F., et al. The EMBL-EBI Job Dispatcher sequence analysis tools framework in 2024. Nucleic Acids Res. 52 (W1), W521-W525 (2024).
- Edgar, R. C. MUSCLE: multiple sequence alignment with high accuracy and high throughput. Nucleic Acids Res. 32 (5), 1792-1797 (2004).
- Chao, G., et al. Isolating and engineering human antibodies using yeast surface display. Nat Protoc. 1 (2), 755-768 (2006).
- VanAntwerp, J. J., Wittrup, K. D. Fine affinity discrimination by yeast surface display and flow cytometry. Biotechnol Prog. 16 (1), 31-37 (2000).
- McConnell, A. D., et al. A general approach to antibody thermostabilization. MAbs. 6 (5), 1274-1282 (2014).
- Pavoor, T. V., Wheasler, J. A., Kamat, V., Shusta, E. V. An enhanced approach for engineering thermally stable proteins using yeast display. Protein Eng Des Sel. 25 (10), 625-630 (2012).
- Chen, I., Dorr, B. M., Liu, D. R. A general strategy for the evolution of bond-forming enzymes using yeast display. Proc Natl Acad Sci U S A. 108 (28), 11399-11404 (2011).
- Traxlmayr, M. W., et al. Construction of a stability landscape of the CH3 domain of human IgG1 by combining directed evolution with high throughput sequencing. J Mol Biol. 423 (3), 397-412 (2012).
- Chao, G., Cochran, J. R., Wittrup, K. D. Fine epitope mapping of anti-epidermal growth factor receptor antibodies through random mutagenesis and yeast surface display. J Mol Biol. 342 (2), 539-550 (2004).
- Stern, L. A., et al. Cellular-based selections aid yeast-display discovery of genuine cell-binding ligands: Targeting oncology vascular biomarker CD276. ACS Comb Sci. 21 (3), 207-222 (2019).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved