Method Article
嗨 - C:一个方法研究基因组的三维架构。
* 这些作者具有相同的贡献
Erratum Notice
摘要
的HI - C的方法,允许不带偏见,全基因组鉴定(1)染色质相互作用。您好- C的夫妇接近结扎和大规模并行测序。由此产生的数据可以用来研究基因组架构在多尺度的初步结果确定,如染色体领土,染色质开放式和封闭式的隔离,在megabase规模和染色质结构的特点。
摘要
染色体的三维折叠compartmentalizes的基因组,并能带来遥远的功能元素,如推动者和促进剂紧密的空间接近2-6。破译染色体组织和基因组活动之间的关系,将有助于了解基因的转录和复制过程,像。然而,很少有人知道如何染色体倍。显微镜无法辨别大量的基因位点同时进行,或在高分辨率。迄今为止,使用染色体构象捕获(3C)和其后续的适应化修改的染色体相互作用的检测所需的目标位点的选择,使全基因组的研究不可能7-10。
我们开发的Hi - C,3C延伸,是能够找出一个不带偏见,全基因组时尚的长程相互作用。在HI - C的细胞固定与甲醛,造成约束共价DNA -蛋白质交联的方式彼此相互作用的位点。当随后用限制性内切酶的DNA是支离破碎,这些位点保持联系。生物素标记的残留物是成立5'突出英寸下一步,平端结扎是有利于交联的DNA片段之间的结扎事件的稀释条件下进行填写。这在结扎产品的全基因组库的结果,相应的对原本在细胞核中相互接近的片段。交界处的网站,每个结扎产品是用生物素标记。图书馆是剪切,路口拉链霉亲珠子。纯化路口,随后可以使用一种高通量序列分析,导致在交互片段的目录。
直接接触所产生的矩阵分析,揭示基因组组织的许多功能,如染色体领土的存在和优惠的小型基因丰富的染色体协会。相关分析,可应用于接触矩阵,表明人类基因组是分隔成两个隔间:较低的密密麻麻的车厢,开放,平易近人,和积极的染色质和更密集的车厢载有封闭,交通不便,和非活动染色质区域。最后,合奏的接触矩阵的分析,再加上理论推导和计算机模拟,发现megabase规模的Hi - C的揭示与分球的构象一致的功能。
研究方案
这种方法是使用利伯曼-艾登等人 ,科学326,289-293(2009)中报告的研究。
一,交联,消化,DNA末端标记,和钝结扎
- 嗨 - C开始交联的细胞,这是一个所有3C为基础的方法之间的共同点。首先,增长2 × 10 7和2.5 × 10 7哺乳动物细胞之间,无论是贴壁或悬浮液中,交联和细胞。 (细胞的交联的详细信息,请参阅:11
- 在550μLlysisbuffer的细胞裂解(500μL10毫米的Tris - HCl pH值8.0,10 mM氯化钠,0.2%IGEPAL CA - 630和50μL蛋白酶抑制剂)使用均质机。在5000转的旋转染色和两次沉淀用500μL1X NEBuffer 2。
- 染色质重悬在1X NEBuffer 2,分装成5管编号,并添加1X NEBuffer 2到终体积为362μL。添加38μL1%SDS,仔细混合,在65℃孵育10分钟。广场上冰后立即孵化管。
- 淬火的SDS加入44μLTriton X - 100的的混合仔细。精华加入400 HindIII和孵化单元,在37 ° C过夜旋转时的染色质。
- 接下来的步骤是HI - C的具体的,包括标记的DNA与生物素和执行交联片段平端结扎结束。这一步将允许结扎路口后得到净化。管1不应进行生物素一步,而应单独存放,并作为一个3C的控制,以确保消化,并结扎条件最佳。
- 为了填补限制性片段出挑,在余下的4管与生物素标记的DNA结束,加入1.5μL10 mM的dATP的,1.5μL10毫米dGTP,1.5μL10毫米dTTP的37.5微升0.4毫米生物素- 14 - dCTP,和10μL5U/μl的Klenow管2-5。仔细混合和孵育45分钟,在37 ° C。
- 管置于冰上。以灭活酶,添加86μL10%SDS管1-5。管孵育65℃,整整30分钟,他们随即置于冰上。
- 结扎是极稀条件下进行,以有利于结扎事件之间的交联片段。上冰工作,增加7.61毫升结扎组合[745μL10%TRITON X - 100,745μL10X连接缓冲液(500毫米的Tris - HCl pH值7.5,MgCl 2的 100毫米,100毫米数码地面电视),80μL10 mg / ml的BSA 80μL100毫米(ATP)和5.96毫升水]五个编号为15毫升管。每个消化的染色质的混合物转移到相应的15毫升管。
- 对于常规3C结扎,添加10μL1U/μlT4 DNA连接管1。生硬的高端HI - C连接,添加50μL1U/μlT4 DNA连接管2-5。反相管混合孵育4小时5管16 ° C。
- 跨界通道逆转,每管加入50μL10 mg / ml的蛋白酶K和孵化管一夜之间在65 ° C,蛋白质是退化每管加50μL10 mg / ml的蛋白酶K,第二天继续的潜伏期在65 °另外2个小时的彗星。
- 将反应混合物冷却至室温,将它们传输到五个50毫升锥形管。在这些管子执行苯酚抽提纯化的DNA。添加10毫升苯酚的pH值8.0和2分钟的旋涡。管在3,500 rpm转速旋转10分钟,并仔细尽可能多的水相尽可能转移到一个新的50毫升管。
- 重复使用苯酚的pH值8.0:氯仿(1:1)提取,并用乙醇沉淀DNA。 (DNA纯化的详细信息,请参阅 :11
- 乙醇离心沉淀DNA后,溶解DNA沉淀中的每个450μL1X TE(10毫米的Tris - HCl,1 mM EDTA的pH值8.0)。 DNA混合物转移到1.7 mL离心管。
- 做2酚:氯仿抽提进行另一轮的净化。添加500μL酚pH值8.0:氯仿(1:1)和1分钟的旋涡。在14000转5分钟,离心管和水相转移到新管。在第二次提取,沉淀加入0.1倍体积的NaOAc,100%的乙醇和孵化30分钟的2倍量在-80 ° C的DNA
- 纺纱沉淀DNA后,用70%乙醇洗的每个DNA沉淀,每25μL1X TE重悬DNA沉淀。任何RNA的降解可能是由目前的1毫克/毫升核糖核酸酶每管加入1μL,孵育30分钟,37管° C。游泳池管2-5的Hi - C的含量,仍保持单独作为一个3C控制管1。
- 现在是一个很好的机会,研究的Hi - C的标记和连接效率。这些控件的Hi - C库是否会获得成功的优秀指标。
- 要检查的质量和数量的图书馆,运行2μl和6μL分装在0.8%琼脂糖凝胶3C的Hi - C库1:10稀释。 (见图2a)
- 嗨 - C标记和Hi - C连接效率的是由PCR消化实验验证。成功的填充和结扎了的HindIII网站(AAGCTT)创建一个限制性内切酶NheI(GCTAGC)的网站。产品,从附近的两个限制性片段组成的一个特别结扎是用PCR扩增(3C 11为模板0.2μL,每个库,PCR产物,其后的HindIII,NheI或两者消化。运行后2%的凝胶样品3C和高- C连接事件的相对数,可估计量化切割和未切割频段的强度(见图2b)。
- 某些碎片不会被结扎,以避免拉他们下来后,删除从这些unligated结束,用T4 DNA聚合酶的核酸外切酶活性的生物素。
- T4 DNA聚合酶的核酸外切酶活性与非结扎DNA末端生物素- 14 - dCTP被删除。混合1μL10 mg / ml的BSA,10μL10倍NEBuffer 2,1μL10毫米的dATP,1μL10毫米dGTP和5个单位的总体积100μLT4 DNA聚合酶的5微克的Hi - C库,并培育混合物在12 ° C为2小时。如果可能的话,多个5微克反应。
- 加入2μL0.5 M EDTA pH 8.0的反应是停止。
- 为了净化的DNA,苯酚的pH值8.0:氯仿(1:1)提取乙醇沉淀。
- 丢弃上清液,重悬,并在总体积100μl水汇集的DNA颗粒。
二。剪切和尺寸的选择
- 为了使生物素化的DNA适合于高通量测序,DNA必须剪切Covaris S2的文书300-500碱基对(占空比5 5,强度,周期/脉冲200,4个周期的时间为60秒)的大小。
- 要修复剪切的DNA末端,加14μL10X连接缓冲液,14μL2.5 mM的dNTP混合液,5μLT4 DNA聚合酶,T4多聚核苷酸激酶5μL,1μL的Klenow DNA聚合酶1μl水。孵育30分钟在室温。
- 孵化后,用Qiagen公司MinElute列净化的DNA,根据制造商的建议。洗脱的DNA,15μL1X三低EDTA(TLE:10毫米的Tris pH值8.0,0.1毫米EDTA)的两倍。然后,将一个dATP的最终修复DNA的3'末端加入5μL10倍NEBuffer2,10μL1毫米的dATP,2μl水和3μL的Klenow(出)。 30分钟的反应在37 ° C。
- 灭活Klenow片段,反应孵育20分钟在65 ° C和随后在冰上冷却的反应。使用speedvac,减少反应体积20μL。
- 接下来,在1.5%琼脂糖凝胶负载的DNA与1X TAE为3.5小时运行在80 - 90V。使用SYBR绿色凝胶染色后,可视化的DNA上DarkReader。消费DNA片段之间的300和500个碱基对,它们净化用Qiagen公司的凝胶萃取试剂盒,采用2-4列取决于凝胶的重量。 50μL1X TLE的洗脱DNA。
- 从Qiaquick列合并洗脱液,并带来最终体积1X TLE的300μL。最后,确定使用的量子比特的荧光QUANT - IT测定DNA浓度的DNA计算的总金额。
三。生物素下拉配对末端测序
- 在这个协议的部分,结扎路口纯化的DNA池,相互作用的配对末端测序的染色质片段的高效识别。在DNA LoBind管上执行所有后续步骤。
- 准备素珠下拉洗涤150μL与400μL吐温缓冲液(结核病:5毫米的Tris - HCl pH值8.0,0.5毫米EDTA,1 M氯化钠,0.05%吐温)悬浮磁链亲和素珠两次。
这些问题和未来的清洗包括五个步骤:- 添加缓冲区的珠子
- 混合物转移到一个新的管
- 旋转样品在室温下3分钟
- 使用磁粉集中回收珠
- 去除上清
- 重悬于300μL2X没有吐温缓冲液(2X非关税壁垒:10毫米的Tris - HCl pH值8.0,1 mM的EDTA,2 M氯化钠)的珠子和300μL的Hi - C的DNA结合。允许在室温下孵育15分钟旋转的混合物结合的生物素标记的Hi - C的DNA链亲和素珠。
- 回收与磁性粒子concentr的约束链霉亲和素珠的DNAator,去除上清。在400μL1X非关税壁垒(5毫米的Tris - HCl pH值8.0,0.5毫米EDTA,1 M氯化钠),100μL1X连接缓冲,清洗珠。在50μL1X结扎缓冲液重悬珠的混合物转移到一个新的管。
- 要准备Illumina的配对末端测序的DNA,采取总额的DNA,作为生物素拉下来,这是前面计算步骤2.6中输入,并除以20,估计量的Hi - C的DNA被拆掉,并结扎。添加6 Illumina的每微克的Hi - C的DNA可为结扎配对结束适配器picomoles。使用1200个单位的T4 DNA连接酶的DNA结扎的适配器。在室温下孵育2小时。
- 删除非结扎配对完适配器,通过回收的Hi - C的DNA绑定珠,洗衣机400μL1X结核病珠两次。
- 200μL1X非关税壁垒200μL和50μL1X NEBuffer 2,清洗珠。最后一次洗涤后,重悬在50μL1X NEBuffer 2的珠子,并转移到一个新的管。
- 要确定所需的周期数,以产生足够的PCR产物测序,成立6,9,12或15个周期的四个测试PCR反应。 (PCR扩增的详细信息,请参见:12上运行5%的聚丙烯酰胺凝胶和SYBR绿色染色的PCR反应,确保杂散乐队和涂抹在400存在的情况下,确定最佳的循环数 - 。 600个碱基对,这是结扎后的适配器剪切产品的长度。
- 健全的Hi - C语言库绑定的链霉亲和素珠在一个大型的PCR,PCR循环的最佳的其余部分。从单独的井池的PCR产物回收的珠子。保持1%的大型PCR产物分开上运行的一种凝胶,剩余的PCR产物纯化与1.8倍体积Ampure珠根据制造商的建议。
- 与50μL1X TLE的缓冲液洗脱的DNA,并比较Ampure珠纯化的PCR产物的1%至5%的聚丙烯酰胺凝胶上原有的PCR产物的1%等分,确保成功的PCR引物去除。
- 我们还建议克隆1μL的Hi - C库,并确定使用Sanger测序克隆约100的产品。这将使你评估的alignable HI - C在读取PCR混合物的相对数。对于典型的结果,见图3B。
- 序列与Illumina的配对末端测序的高C库将每个独立使用MAQ(http://maq.sourceforge.net/),以确定相互作用的染色质片段的结束。
四。代表的Hi - C结果
- 结果如下预期的Hi - C协议的执行技术上和质量控制标准,可以考虑。
- 质量控制的步骤应该表明,3C和Hi - C的库运行比较紧带大于10 KB。涂抹的DNA表明连接效率差。通常情况下,连接效率是轻微的Hi - C库相比,一个3C的模板(见图2a)。
- 嗨 - C标记和连接效率可估计使用3C引物产生的PCR产物的消化。 3C路口削减HindIII和NheI。为喜- C的路口的情况正好相反。这PCR技术的消化试验表明,70%的高- C的扩增产物削减NheI,而不是由HindIII位,确认有效的结扎路口标志(见图2b)。
- 测序读取分析表明,染色体内,并interchromosomal的相互作用,由蓝色和红色的线条表示读取,对齐显着的HindIII酶切位点相比,随机生成的读取,以绿色显示(参见图3a)。
- 在一个成功的实验中,55%的alignable阅读对代表interchromosomal的相互作用。百分之十五,代表之间的片段相距不到20 KB和30%的染色体内相互作用是染色体内读对,相距20多KB(参见图3B)。这种分布可能是采样的高通量测序之前,作为质量控制的一种形式;桑格约100个克隆的克隆和测序通常是足够的。
- 染色质的相互作用可以作为热图,其中X和Y轴代表在基因组顺序的位点和每个像素代表观察到它们之间的相互作用的数量可视化。通常情况下,非常接近线性基因组彼此的DNA片段的倾向,经常互相交流。这是作为一个突出的对角线的染色体内heatmaps(参见图4A)。
- 下面的结果显示的数据进行分析,以揭示基因组组织各级的不同的方式。绘制接触probabilitŸ与基因组的距离(见图5a)显示,接触的概率随着功能基因组的距离,最终达到一个高原。染色体内的相互作用,在实线显示,在每一个距离,丰富了相对interchromosomal相互作用,虚线表示。这直接暗示染色体领土的存在。
- 计算的染色体之间的所有对interchromosomal接触观察/预期的数字揭示尤其是对染色体之间的优惠的关联。丰富的小型基因染色体优先互相交流,鲜红的颜色(见图5b)表示。
- 也可以单独染色体检查。原料热图可以调整使用预期的热图,占基因组位点对之间的距离,在观察/预期的热图。然后,相关矩阵,可以产生关联的行和列观察/预计heatmaps。使用相关分析,这是表明,人类基因组中分离到两个车厢。在相关heatmaps格子图案(见图4A - D)说明了这一点。 (的Hi - C的数据分析的详细信息, 请参阅: 1。
- 使用的Hi - C数据,新的见解,获得了染色质折叠的megabase规模。古典模型表明聚合物凝结成一个平衡球的染色包。作为距离的函数绘制的接触几率说明与基因组的距离,其斜率大约是-1(图6a)的功法,接触的概率尺度。这不是一个平衡球的行为是一致的,但不匹配的期望,为另一种结构称为一个分球(参见图6B)。
- 在这里,两个球状结构。着色对应一个端点的距离,从蓝色青色,绿色,黄色,橙色和红色(参见图6C顶部)。与平衡球,分球缺乏纠葛。在分球,沿等高线附近的位点,往往是在3D附近,领先的单色块的存在(参见图6C中间)。这样的块都没有发现在平衡球(参见图6C底部)。
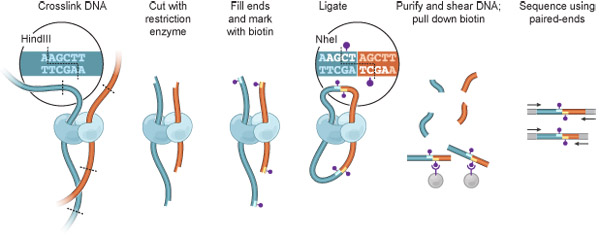
图1高- C的概述。细胞是交叉与甲醛,在空间相邻的染色质分部(DNA片段:深蓝色,红色;蛋白质,它可以调解这种相互作用,在淡蓝色和青色)之间的共价键链接。染色质的消化与限制性内切酶(在这里,HindIII位;限制站点:虚线,见插图)。由此产生的粘性末端核苷酸,其中之一是生物素(紫点)。结扎极稀利于分子内结扎事件的条件下执行;丢失的HindIII网站NheI网站创建(插图)。 DNA纯化和剪切,并使用链霉亲和素磁珠分离生物素标记的路口。交互片段配对末端测序鉴定。

图2。C库的质量控制。 (一)一个3C的控制和一个Hi - C库的数量不断增加,解决了0.8%琼脂糖凝胶。这两个库执行作为一个相当紧迫的波段大于10 KB。典型的Hi - C库连接效率比什么是观察在3C模板略有下降,表明涂片中的Hi - C的车道。 (二)PCR消化控制。使用标准的3C PCR条件扩增交界处附近的两个片段组成,是一个结扎。可区别于那些在传统的3C生产结扎部位消化的Hi - C连接产品。嗨 - C路口削减NheI,不HindIII位; 3C路口的情况正好相反。 NheI削减70%的高- C的扩增,确认结扎交界处的高效率标识。两个重复进行,以确保可靠的量化。

图3的Hi - C读的质量控制。 (一)读片段对应的染色体内(蓝色)和interchromosomal(红色)的相互作用对齐明显的HindIII酶切位点相比,随机生成的读取(绿色)。染色体内读interchromosomal读,曲线迅速下降的HindIII站点增加的距离,直到高原〜500 bp的距离达到。与此对应的最大片段大小,测序。 (二)通常情况下,55%的alignable阅读对代表interchromosomal的相互作用。百分之十五的代表之间的碎片小于20 kb的染色体内的相互作用除了和30%的染色体内读对,相距20多KB。这种分布可能是采样的高通量测序之前,作为质量控制的一种形式;桑格约100个克隆的克隆和测序通常是足够的。

图4相关分析表明,原子核是分隔成两个隔间。 (一)热图对应于14号染色体上的染色体内相互作用。每个像素代表一个1 MB的位点和一个1 MB的轨迹之间的所有交互;强度对应的读取总数(范围:0-200读取)。刻度显示,每10 MB。热图展品下部结构在激烈的对角线的形式和大块的星座。 (第14号染色体是acrocentric;短臂不显示)使用的Hi - C的数据集来计算一个位点对一个给定的基因距离的平均接触的概率,期望矩阵产生(二)相应的会是什么观察,如果没有长期范围内的结构。这两个矩阵的智商是观察/预期的矩阵(c)凡耗损是在蓝色和红色浓缩范围:0.2(蓝色),5(红色)]所示。块模式变得更加明显。相关矩阵(四)说明的相关性研究[范围:-1(蓝色)到1(红色)]之间每双沿14号染色体位点的染色体内的互动状况。醒目的格纹图案表明存在两个车厢内的染色体。

图5。染色体领域的存在和组织。 (一)接触几率下降了1号染色体上的基因距离的功能,最终达到一个高原〜90MB(蓝色)。 interchromosomal接触水平(黑色虚线)不同染色体不同,对1号染色体上的基因位点是最有可能与10号染色体上的基因位点(绿色破折号)和最有可能与21号染色体上的基因位点(红色虚线)交互交互的。 Interchromosomal相互作用耗尽相对染色体内的相互作用。 (二)观察/预计所有对染色体之间的interchromosomal接触。红色表示浓缩,和蓝色表示枯竭[范围:0.5(蓝色),2(红色)]。小的,丰富的基因的染色体往往彼此交互。

图6。染色质的本地包装一个分球的行为是一致的。 (一)联系的概率,作为功能基因组的距离平均在整个基因组(蓝色)。一个突出的功法缩放是500KB和7MB(阴影区域)与斜率为-1.08(适合在青色)之间。 (二)接触的距离为平衡(红色)和分球(蓝色)的功能的概率的仿真结果。分形球的斜率非常接近-1(青色),证实了我们的新的理论预测 1 。一个平衡球的斜率是-3 / 2,前理论的预期相匹配。斜坡形球非常相似的Hi - C结果中观察到的斜坡,而斜坡平衡球是没有看到的Hi - C数据。 (三) 顶:展开的聚合物链,长4000单体。着色对应一个端点的距离,从蓝色青色,绿色,黄色,橙色和红色。中东 :我们的合奏得出一个分球的典型例子。分形球缺乏纠葛。沿等高线附近的位点,往往是在3D附近,导致大型单色块上表面和截面底部明显的存在:一个平衡球。结构高度纠缠,沿等高线(类似的颜色)附近的位点,不必在3D附近。
讨论
我们提出了一个研究基因组的3维结构,映射在一个不带偏见,全基因组的方式的染色质相互作用的方法。最关键的实验步骤设置这项技术,除了以往的工作 - 交联片段的限制,生物素标记的核苷酸掺入结束前平端结扎。执行这一步成功使深结扎路口测序,并给出了高- C的范围和权力。
读的次数将最终确定的相互作用图谱的分辨率。在这里,人类基因组1 MB的互动地图是提出使用〜30万alignable读取。为了增加了N倍的“目的”的决议,读的次数必须增加 N 2倍。
的Hi - C的技术,可随时与其他技术相结合,如后库代杂交捕获(目标基因组的特定部位)和结扎后的染色质免疫沉淀(检查染色质环境与特定的蛋白质有关地区),。
披露声明
致谢
我们感谢讨论和代码A. Kosmrlj; AP艾登,XR宝,M.布伦纳,D.首映礼,W.高斯帕,A.贾费尔,A.尼科夫,A. Miele公司,G. Giannoukos,C. Nusbaum,AJM Walhout属木,和光泽利多维奇进行讨论;和L.加夫尼和B黄与可视化的帮助。
一个房利美和约翰赫兹基金会研究生奖学金,国防科学与工程学院研究生奖学金,美国国家科学基金会研究生奖学金,国家空间生物医学研究所,并授予没有支持。 T32 HG002295由国家人类基因组研究所(NHGRI)致发光(EL); i2b2(整合生物学和床头信息),美国国立卫生研究院支持中心布里格姆与妇女医院(林)生物医学计算;没有批。 HG003143在NHGRI,和一个凯克基金会杰出青年学者奖(JD)。原材料和映射的Hi - C序列数据已存放在GEO数据库( www.ncbi.nlm.nih.gov/geo/ ),加入没有。 GSE18199。额外的可视化是在提供 http://hic.umassmed.edu 。
材料
| Name | Company | Catalog Number | Comments |
| Protease inhibitors | Sigma-Aldrich | P8340-5ml | Step 1.2 |
| biotin-14-dCTP | Invitrogen | 19518-018 | Step 1.6 |
| Klenow | New England Biolabs | M0210 | Steps 1.6 and 2.2 |
| T4 DNA ligase | Invitrogen | 15224 | Step 1.9 |
| T4 DNA polymerase | New England Biolabs | M0203 | Steps 1.17 and 2.2 |
| 10x ligation buffer | New England Biolabs | B0202 | Steps 2.2 and 3.4 |
| T4 PNK | New England Biolabs | M0201 | Step 2.2 |
| Klenow (exo-) | New England Biolabs | M0212 | Step 2.3 |
| Dynabeads MyOne Streptavin C1 Beads | Invitrogen | 650.01 | Step 3.2 |
| T4 DNA ligase HC | Enzymatics | L603-HC-L | Step 3.5 |
| Phusion HF mastermix | New England Biolabs | F531 | Step 3.8 |
| Ampure beads | Beckman Coulter Inc. | A2915 | Step 3.9 |
参考文献
- Lieberman-Aiden, E., Van Berkum, N. L., Williams, L., Imakaev, M., Ragoczy, T., Telling, A., Amit, I., Lajoie, B. R., Sabo, P. J., Dorschner, M. O., Sandstrom, R., Bernstein, B., Bender, M. A., Groudine, M., Gnirke, A., Stamatoyannopoulos, J., Mirny, L. A., Lander, E. S., Dekker, J. Comprehensive mapping of long-range interactions reveals folding principles of the human genome. Science. 326, 289-293 (2009).
- Kosak, S. T., Groudine, M. Form follows function: the genomic organization of cellular differentiation. Genes and Dev. 18, 1371-1384 (2004).
- Misteli, T. Beyond the sequence: cellular organization of genome function. Cell. 128, 787-800 (2007).
- Dekker, J. Gene Regulation in the Third Dimension. Science. 319, 1793-1794 (2008).
- Cremer, T., Cremer, C. Chromosome territories, nuclear architecture and gene regulation in mammalian cells. Nat Rev Genet. 2, 292-301 (2001).
- Sexton, T., Schober, H., Fraser, P., Gasser, S. M. Gene regulation through nuclear organization. Nat Struct and Mol Biol. 14, 1049-1055 (2007).
- Dekker, J., Rippe, K., Dekker, M., Kleckner, N. Capturing Chromosome Conformation. Science. 295, 1306-1311 (2002).
- Zhao, Z., Tavoosidana, G., Sjölinder, M., Göndör, A., Mariano, P., Wang, S., Kanduri, C., Lezcano, M., Sandhu, K. S., Singh, U., Pant, V., Tiwari, V., Kurukuti, S., Ohlsson, R. Circular chromosome conformation capture (4C) uncovers extensive networks of epigenetically regulated intra- and interchromosomal interactions. Nat Genet. 38, 1341-1347 (2006).
- Simonis, M., Klous, P., Splinter, E., Moshkin, Y., Willemsen, R., de Wit, E., van Steensel, B., de Laat, W. Nuclear organization of active and inactive chromatin domains uncovered by chromosome conformation capture-on-chip (4C) uncovers extensive networks of epigenetically regulated intra- and interchromosomal interactions. Nat Genet. 38, 1348-1354 (2006).
- Dostie, J., Richmond, T. A., Arnaout, R. A., Selzer, R. R., Lee, W. L., Honan, T. A., Rubio, E. D., Krumm, A., Lamb, J., Nusbaum, C., Green, R. D., Dekker, J. Chromosome Conformation Capture Carbon Copy (5C): A massively parallel solution for mapping interactions between genomic elements. Genome Res. 16, 1299-1309 (2006).
- Miele, A., Dekker, J. Mapping Cis- and Trans Chromatin Interaction Networks Using Chromosome Conformation Capture (3C). Methods Mol Biol. 464, 105-121 (2009).
- Maccallum, I., Przybylski, D., Gnerre, S., Burton, J., Shlyakhter, I., Gnirke, A., Malek, J., McKernan, K., Ranade, S., Shea, T. P., Williams, L., Young, S., Nusbaum, C., Jaffe, D. B. ALLPATHS 2: small genomes assembled accurately and with high continuity from short paired reads. Genome Biol . 10, R103-R103 (2009).
Erratum
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。