需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
DNA的稳定同位素探测(DNA的SIP)
摘要
DNA的稳定同位素探测,识别和特性的微生物能够利用特定的基板活跃的社区是一个独立的种植方法。基板同化富集重同位素的信息纳入到微生物生物量的标记原子。密度梯度超速离心检索下游分子生物学分析的DNA标记。
摘要
DNA稳定同位素探测(脱氧核糖核酸SIP的)是一个强大的技术,确定积极融入细胞生物量的微生物特别是碳基体和养分。因此,培养独立的技术已分配的代谢功能多样的社区居住的陆地和水生环境的广泛的一个重要方法。与稳定同位素标记化合物的环境样品的潜伏期后,提取核酸是密度梯度离心和随后的梯度分馏分离不同密度的核酸。氯化铯的DNA纯化检索随后的分子特性(如指纹识别,基因芯片,克隆库,宏基因组学)标签和不知名的DNA。这朱庇特的视频协议提供视觉密度梯度超速离心,梯度分馏和回收标记的DNA协议一步一步的解释。该协议还包括样品的SIP数据,并强调重要的提示和警告说,必须考虑,以确保成功的DNA - SIP的分析。
研究方案
1。试剂的制备
DNA - SIP的要求,应在实际过程提前准备试剂的使用。本节列出每种试剂的准备方向,并从以前的SIP协议进行修改。
- 氯化铯(CSCL)的解决方案,准备SIP的梯度逐渐溶解603.0克,中海集运(DDH 2 O)的蒸馏水和去离子水到终体积为500 mL -准备一个7.163中号铯的解决方案。小心不超过500毫升!略有边搅拌变暖的解决方案将有助于溶解所有的CSCL。分装在密封分装的最终解决方案。在我们的实验室中,一个共同的存储的做法是准备在100毫升分装125毫升的血清瓶,然后再与丁基橡胶瓶塞卷曲密封。密封分装,可以无限期地储存在室温(20℃)。的密封有助于防止蒸发和铯的“壳”形成。由重达一式三份100μL分装,或使用数字折射仪(如赖克特AR200),经过精心校准铯解决方案确定的解决方案密度。一旦校准成功,赖克特AR200是一致的,并提供准确的读数几年。在室温(20℃),这种解决方案的最终密度一般为1.88-1.89克毫升-1。密度略有不同每一个新的股票准备时间。
- 准备与溴化乙锭(ETBR)的梯度组合与250毫升的无菌DDH 2 O水250克中海集运-氯化铯的解决方案。分装成单独的血清瓶,已如1.1中所述的丁基橡胶密封压接密封的这一解决方案。
- 渐变缓冲 - 联合收割机50毫升的1 M Tris -盐酸,3.75克氯化钾和0.5 M EDTA 1毫升至400毫升的水。溶解氯化钾,然后添加DDH 2 O到500毫升。过滤,消毒和高压灭菌。最终解决方案的0.1 M Tris,0.1 M的氯化钾和1 mM EDTA的。
- 聚乙二醇(PEG)解决方案-准备在无菌DDH 2 O水溶解聚乙二醇6000和46.8克的氯化钠150克到500毫升(30%PEG,1.6 M氯化钠)的总体积的PEG解决方案。高压灭菌。
注意:此方法分为两个阶段,分离与高压灭菌。使该解决方案能够得到适当的混合,发生这种情况时,包括在蒸压瓶搅拌棒。 - TE缓冲液- 10毫米的Tris - HCL(pH值8.0)和1毫米EDTA(pH 8.0)的解决方案,准备在无菌DDH 2 O水使用蒸压股票的解决方案1中号的Tris - HCl(pH值8.0)和0.5 M EDTA ( pH值8.0)。过滤消毒和高压灭菌。
- 70%的乙醇-结合高纯度乙醇350毫升,150毫升无菌DDH 2 O的水。
2。样品孵化和DNA提取
样本DNA - SIP的孵化,通常培养与重同位素碳(13 C)基板。潜伏期和条件(如营养补充,湿度,光线)会有所不同孵育样品类型和底物的性质而定。 DNA - SIP的实验已经成功地使用各种单碳化合物 2,3,多碳化合物 4,5,6, 使用 7,8或标记的氮氧 9 。然而,使用15 N或18 O标记化合物的缺点是下降的物理分离标记的核酸,主要是由于较少的氮和氧原子在DNA和RNA相对碳原子的存在。
一个DNA的SIP实验的关键控制本机(如12 C)的衬底上建立一个相同的潜伏期。这个孵化提供了后续的比较,以确保没有任何明显的标签核酸神器离心或G + C含量在DNA 分离 10的密度差异。同样重要的是保持冷冻样品材料“轻”和“重”的DNA进行比较,值得包括无衬底控制评估整个SIP孵化背景的人口变化。
- 孵育环境样品中含有标记底物的缩影(图1)。根据我们的经验,我们发现,每克样品中的碳13之间5-500μmol的最低注册将适用于含有高生物量,如土壤样品1样品。 1-100μmol纳入碳13 C每公升含有土壤生物量小于的水样,可能会产生一个检测的重同位素特征1。碳修订的金额,将全部纳入生物质能和补充营养此外同化的要求的碳的比例取决于被分析的样品的特点和目标Ø感兴趣的rganisms的。一个单套样品孵化指引将适用于所有样品。重要的是,SIP的孵化用的底物浓度,最好是尽可能接近原位通常遇到的浓度;实验偏差可能的富集培养条件 10的结果。
- 样品的稳定同位素标记底物孵育,DNA提取采用严格的提取协议(PCR或插入克隆小)或一个值得信赖的高分子量克隆(如大型插入宏基因组学)的酶裂解的缩影。的RNA的合作提取,一般不影响分析,因此可用于协议以及DNA,RNA的产量。提取的DNA超速离心法将不剪片段〜50 KB对1短。
- 量化提取的DNA之前设置的CsCl梯度离心管。量化DNA用分光光度计(如Nanodrop 2000年),如果提取协议的产量只有的DNA(例如基于列的套件)。另外,量化,用琼脂糖凝胶电泳。
3。准备梯度超速离心解决方案
此过程涉及到将DNA超速离心机管。有一个以上的类型,所以确切的协议会有所不同,将取决于制造商的指示管和转子。这就是说,我们建议使用一个垂直的井转子,以确保最大的轻型和重型DNA可能分离。我们用一个贝克曼 - 库尔特VTI 65.2转子持有5.1毫升QuickSeal Polyallomer管的16口井,该协议将提供的步骤和考虑这些条件。
- 使用步骤2.3中确定的DNA含量,计算提取的DNA,是需要提供0.5微克所需的音量 - 超速离心机管在5微克的DNA。
- 梯度缓冲液(见第1.3步)和4.8毫升〜6毫升到15毫升一次性无菌管总量的7.163中号铯结合提取的DNA(0.5 - 5微克)。请注意,铯解决方案密度可以有所不同,即使在相同摩尔(见步骤1.1)。可以使用下面的公式确定,需要产生一个适当的混合比例的梯度缓冲/ DNA混合物的体积:
梯度缓冲液和DNA溶液的体积(毫升 )=(铯原液密度-理想的最终密度)的X铯原液量x 1.52
指定的铯4.80毫升原液量。理想的最终密度应该是1.725克毫升-1。在步骤1.1确定股票的解决方案密度。
还要注意的是中海集运和梯度缓冲/ DNA的相对量将导致在总体积大于5.1毫升。准备比最大卷容量(大于5.1毫升)的超速离心机管更大的卷,将确保有足够的解决方案,完全填补管。 - 混合反相10倍。 DNA是稳定的,在室温中铯。
4。创建一个ETBR控制梯度(可选)
由于ETBR嵌入染料与DNA复合物的紫外光下可见,控制梯度含ETBR是有益的,因为他们提供即时的梯度形成的视觉确认之前,分馏样品管(如图1)。列入控制ETBR和两个12的C - DNA和13 C - DNA(或14 N - DNA和15 N - DNA)的混合管,使管内离心完成后,立即带形成可视化。这一点很重要,因为在离心或不正确的编程运行条件破裂的管,可以导致失败的形成梯度。与DNA结合,ETBR降低密度的DNA,并作为一个结果,不同的协议是遵循梯度准备。请注意其他核酸的污渍可以被用来代替ETBR 11,但该协议将需要与其他荧光团的优化。
- 控制梯度需要的基因组DNA的两卷:完全稳定同位素标记和无标签之一。我们通常使用或者苜蓿根瘤菌培养在含有13 C或12 C葡萄糖作为唯一碳源,或在13 C或12 C型甲烷作为我们的控制存在培养Methylococcus荚膜应变浴媒体。
- 结合5〜10微克数量12的C - DNA梯度缓冲区和13个 C - DNA到1.00毫升15毫升一次性螺丝帽筒的最终体积。
- 添加1.00克固体CSCL同一管。混合颠倒。
- 添加110μL10毫克毫升-1 ETBR的解决方案,1克毫升 -1铯原液步骤4.2中使用相同的螺丝帽筒和4.3毫升。最终的解决方案密度近似原铯原液。
- 还需要一个额外的“空白”控制的解决方案,包含ETBR抗衡步骤4.4中创建的解决方案。结合1.00毫升梯度缓冲液,中海集运1.00克,10毫克毫升-1 ETBR解决方案和1克毫升-1铯在一个单独的15毫升的带螺旋帽的管和颠倒混合原液4.3毫升110μL。
5。超速离心法
- 使用一个灯泡和巴斯德吸管,认真填写梯度解决方案准备步骤3.2(或步骤4.4准备ETBR控制梯度)超速离心机管。小心地使用巴斯德吸管管的解决方案。标签上管肩管具有优良的永久性标记。注意:确保基地的管颈管完全充满。不够填补管可能爆裂,在离心。
- 当所有所需的管充满样品溶液,记录各管的精确质量。对管和平衡他们在0-10毫克。为了平衡,找到近配对,添加或删除微量的解决方案,直到他们的平衡,保持密切尽可能管脖子基地的解决方案级别。注意,称重管,我们使用一个倒15毫升的螺丝帽的管作为平衡管人已减少了一半。
- 密封管,用一个“管礼帽”,根据制造商的指示。
- 检查管件是密封的反相他们正确运用适度的压力。称量管再次检查,他们仍然是平衡密封后在0-10毫克。
- 仔细检查每个转子,以确保井干净,无杂物或灰尘可能在离心管穿刺。
- 相反的另一种平衡双管插入转子。记录每个样品的转子位置,因为离心过程中可能会导致被损坏或删除的标记标签。仔细密封制造商表示转子井。
- 装入超速离心机转子。关闭超速离心机门,适用于真空。如果使用VTI 65.2转子,设置转速为44100 RPM(〜177000 XG AV),温度在20 ° C和离心时间为36-40小时。选择真空,最大加速度,和关闭制动(保证不减速扰乱的梯度)。请注意,关闭制动,将添加一个额外的运行时间1-2小时。还要注意的是运行时间较短,可能无法实现足够的乐队决议。龙离心运行的建议,因为它们会导致更大的分辨率不同的核酸带。
- 离心过程完成后,立即仔细拆下转子。避免任何倾斜或转子碰撞,轻轻取出转子管,管内,以避免干扰梯度。在极少数情况下,在运行过程中会爆管。如果是这样,有一个机会,在其他管的梯度没有形成正确。如果被列入控制梯度,仔细检查紫外光照射下,该管,确认形成梯度。如果还没有形成梯度控制管,这是最好的,重复所有步骤5。请注意ETBR控制管和空白对照,可在黑暗中存储和重用六个月。小心仔细清洁转子根据制造商的指示,一旦爆管已被删除。不要使用金属刷或研磨清洁剂清洁转子井,以避免划伤转子井!转子特定的刷子和清洗液,就可以买到从贝克曼。
6。梯度分馏
目前用于恢复从超速离心机管的DNA有两种方法:分馏和针提取。该协议将只描述提取DNA的过程中,利用分馏技术。这是因为大多数的SIP实验,标记的DNA不能与ETBR可视,而必须通过比较多个样品管相当于轻,重馏分中检测到。强烈建议注射泵,检索等密度梯度超速离心机管分数。我们使用一个BSP模型输液泵(布伦特里科学公司)。也可用于低流量蠕动泵或HPLC泵。
- 填充60毫升无菌注射器,无菌DDH 2 O含有足够提供一个深蓝色的溴酚蓝染料。将注射泵的输油臂注射器。将泵管,装上一个23号1针,并打开泵, 直到一些DDH 2 O有针。注意, 在这DDH 2 O电源的任何气泡会产生负面影响的分馏过程结束通过。
- 修复的离心管钳位立场。确保钳足够紧,以防止流离失所管,但不这样管的压力会导致中海集运的解决方案时,管刺穿的释放。皮尔斯管沿管缝的底部使用一个新的23计1“针。为了获得最佳效果,皮尔斯在控制,快速,自信地管,这是很难做好的,实践前几次这是第一次试图与样品管。
- 对于每个样本,准备12无菌1.5毫升离心管,使用说明样品数和分数(1-12;重到轻)的标签。使用连接到泵管(步骤6.1),皮尔斯管上管肩顶沿缝,针。收集渐变的解决方案,使用离心管。试管底部,皮尔斯在一个快速和可控制的方式管。实践事先必须非常小心,使用控制拉的议案,以防止通过管和成手指强行针!使用以前校准泵率在12分钟内(425μL分钟-1),将产生12 × 425μL分数。
- 使用数字折射仪(如赖克特AR200;建议)或检查从一个渐变的分数的密度分析天平,以确认正确的梯度形成。您将需要使用这个测试〜50μL的样品。我们常常包括纯在一个管文化的DNA(准备ETBR控制梯度)作为一个分馏的控制和使用密度测定。预计的密度范围从1.690-1.760克毫升-1,与中位数的〜1.725 克毫升-1密度,。
7。 DNA沉淀
- 从所有的分数,先加入20微克的线性聚丙烯酰胺作为载体沉淀的沉淀DNA。混合颠倒。新增2卷的PEG溶液(见第1步)和颠倒混合。如果糖原作为本议定书降水的载体,一个载体(如糖原或线性聚丙烯酰胺)的降水梯度派别的DNA定量复苏是至关重要的,但谨慎的注意,应使用。糖原的筹备工作已被证明与细菌核酸的污染和污染,可以很容易混淆解释SIP 梯度分数12。
- 保留在室温下2小时管使DNA沉淀。如果需要的话,可以留管在室温下过夜。
- 13,000克离心30分钟,与所面临的一个一致的管方向,在转子向外管背面。小心吸弃上清。一个颗粒应该是可见的,但可以看到在这个阶段是非常困难的。一个明亮的光源(如台灯)下工作,以协助在可视化的颗粒。
- 用500μL70%的乙醇洗涤沉淀。 13,000克离心10分钟。小心吸弃上清。颗粒通常会更可见这一步,但会更容易地从管壁游离于。
- 允许沉淀在室温下干燥15分钟。
- 每个颗粒暂停50μLTE缓冲液(见第1.5步)。琼脂糖凝胶上运行5μL每一个分数,根据标准的实验室协议。
8。分数表征
用来描述梯度分数评估一个SIP孵化的成功的方法,将不同的实验室和设备的可用性而定。使用针对16S rRNA基因指纹法是一种常用方法和末端限制性片段长度多态性(T - RFLP),变性梯度凝胶电泳等方法(DGGE)是适当的(图1)。上文所述的协议,预计光分数9-11(〜1.705-1.720克毫升-1)和沉重的DNA指纹的DNA内分数5-8(〜1.720-1.735克毫升 -1 )。与稳定同位素孵育样品组分5-8相关的独特的指纹,但不能与本地基板孵育控制的特别标记底物的代谢与特定微生物提供了有力证据。如果没有足够的标记的DNA仍然对一些应用程序(杂交,宏基因组学),可使用多重置换扩增产生更大数量的13-15,但是这可以引入扩增 14,16嵌合体。
9。结果
典型的DNA - SIP的结果将显示一个标签和不知名的DNA形成梯度超速离心法分离。理想的情况下,彻底解决了超高分子量的遗传物质从不知名的材料(如13 C,15 N)的一定会实现。分辨率可直观地观察ETBR控制管带的形成见证。个人梯度分数中检索基因组的DNA的浓度,也可能是用来确定适当的梯度的形成。
我们对于这个协议,包括使用两个纯培养(图2)核酸进行梯度离心代表性的结果。这里包括分馏梯度准备使用从 S中提取的基因组DNA 根瘤菌 (ATCC 1021),和13的C -标记M。荚膜 STR。浴。以下的超速离心法,分馏和DNA恢复,标签和不知名的基因组DNA单独成各自的梯度组分具有不同的密度(图2A)。重同位素标记的DNA,可以观察到分数4-5,而不知名的DNA发现高浓度的分数9-10。从每个分数的DNA特征与变性梯度凝胶电泳17和PCR扩增产生相应梯度(图2B)中的两个生物体的离散带型的产品。组分的密度范围从〜1.580 - 1.759克毫升 -1,他们在降低密度由左到右的顺序显示。
虽然分离纯13的C -和12 C - DNA可显着(图2),环境样品孵化可能更难以解释。举例来说,我们培养苔原从雷索卢特湾(加拿大努纳武特地区的土壤为12 C 或 13 C型标记的葡萄糖为14天的时间内15 ° C。)梯度的一小部分DNA纯化琼脂糖凝胶电泳表明,基因组DNA,跨分数7-10“抹黑”两个12的C - 13 C型孵化(图3A,3C,分别)。在这种情况下,从特定的微生物类群的生物量的13的C -浓缩只能确定一个方法,如16S rRNA基因DGGE技术。 12 C葡萄糖孵育土壤DNA产生了类似的模式,但在所有梯度分数(图3B)13 C -葡萄糖孵育样品DGGE指纹是唯一分数5-8(图3D )。特别感兴趣的是保守带箭头指示。这占主导地位的“phylotype”是所有梯度分数,但较重的DNA获得的分数从13 C -葡萄糖孵育土壤的变化相一致。随后的DNA测序这个乐队和/或克隆库分析会确认这个特殊的16S rRNA基因的身份和指导随后的宏基因组或种植为基础的方法。
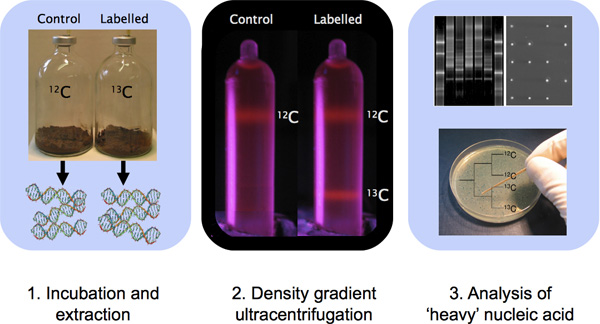
图1。大纲涉及样品孵育,DNA提取,CsCl密度梯度离心和与DNA分子技术特性的一个DNA - SIP的实验。
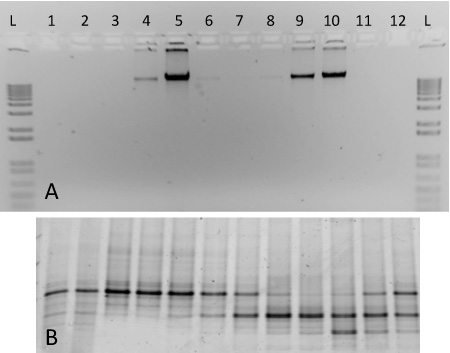
图2为SIP梯度分馏预期的结果,其中包括两种纯培养的DNA。 (一)DNA梯度分数1-12等分1%琼脂糖凝胶上运行,从一个包含13 C 标记 M.的梯度荚膜应变浴(分数4-6)和12个 C型标记S。根瘤菌 (分数8-10)。 1 KB的阶梯,是进行比较(二)PCR扩增的DNA相同的分数10%的变性梯度凝胶电泳凝胶上运行。指纹图谱揭示分数5和9之间的明显差异,例如,。
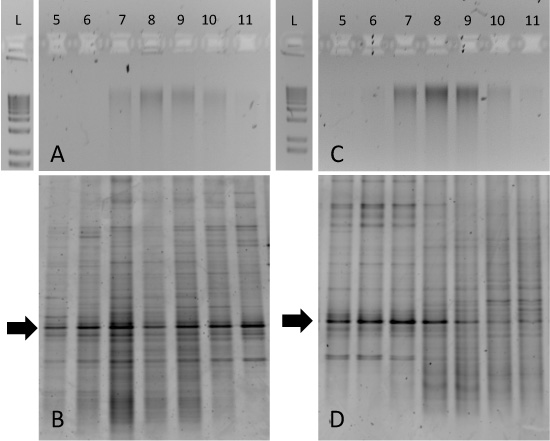
图3:从土壤样品孵化SIP梯度分馏的预期结果。 1%琼脂糖凝胶上运行两个12的C -葡萄糖修订土壤(A)和13 C -葡萄糖修订土壤(三)从梯度分数等分和比较,包括1 - KB的阶梯。通讯DGGE指纹,每个样品(二)和(D)。指纹揭示分数13分数5-8(四)修订的C -葡萄糖样品中富集的特定的细菌类群。
讨论
正确的设计是稳定同位素探测实验,获得上述背景不知名社区标记的DNA至关重要。样品孵育时间,底物浓度,培养条件(如养分,土壤水分含量),跨期和复制有关的注意事项进行了讨论别处 10,18,我们建议读者查阅这些出版物时,设计一个SIP孵化。目前的协议,这是值得评论从SIP梯度数据的解释有关的额外考虑。由于离心过程的性质,它是另外重要的,包括纯文化和母语的底物孵育样?...
披露声明
致谢
这项工作是支持的战略项目和发现资助自然科学和加拿大工程研究理事会(NSERC)JDN。
材料
| Name | Company | Catalog Number | Comments | |
| Bromophenol Blue | Reagent | Fisher Scientific | BP115-25 | |
| Cesium chloride | Reagent | Fisher Scientific | BP210-500 | |
| Ethanol, reagent grade | Reagent | Sigma-Aldrich | 652261 | |
| Ethidium bromide | Reagent | Sigma-Aldrich | E1510 | |
| Hydrochloric acid | Reagent | Fisher Scientific | 351285212 | |
| Linear polyacrylamide | Reagent | Applichem | A6587 | |
| Polyethylene Glycol 6000 | Reagent | VWR international | CAPX1286L-4 | |
| Potassium Chloride | Reagent | Fisher Scientific | AC42409-0010 | |
| Sodium Chloride | Reagent | Fisher Scientific | S2711 | |
| Sodium Hydroxide pellets | Reagent | Fisher Scientific | S3181 | |
| Tris base | Reagent | Fisher Scientific | BP1521 | |
| Dark Reader | Equipment | Clare Chemical | DR46B | |
| Microcentrifuge | Equipment | Eppendorf | 5424 000.410 | |
| Nanodrop 2000 | Equipment | Fisher Scientific | 361013650 | |
| Infusion pump | Equipment | Braintree Scientific, Inc. | N/A | Model Number: BSP See www.braintreesci.com for ordering details. |
| Tube sealer | Equipment | Beckman Coulter Inc. | 358312 | |
| Ultracentrifuge | Equipment | Beckman Coulter Inc. | ||
| Ultracentrifuge rotor | Equipment | Beckman Coulter Inc. | 362754 | |
| Ultraviolet light source | Equipment | UVP Inc. | 95-0017-09 | Any UV source will suffice |
| Ultraviolet light face shield | Equipment | Fisher Scientific | 114051C | |
| Butyl rubber stoppers, gray | Material | Sigma-Aldrich | 27232 | |
| Centrifuge tubes | Material | Beckman Coulter Inc. | 342412 | |
| Hypodermic needle, 23 gauge, 2” length | Material | BD Biosciences | 305145 | |
| Microfuge tubes, 1.5 mL | Material | DiaMed | AD151-N500 | |
| Open center seals, 20 mm diameter | Material | Sigma-Aldrich | 27230-U | |
| Pasteur pipettes, glass | Material | Fisher Scientific | 13-678-6C | |
| Pipet tips | Material | DiaMed | BPS340-1000 | Catalogue number is for 200 μl tips. 10 or 20 μl tips may be purchased from the same source |
| Pump tubing 1.5 mm bore x 1.5 mm wall | Material | Appleton Woods | ||
| Screw-cap tubes, 15 mL | Material | DiaMed | AD15MLP-S | |
| Serum vials, 125 mL volume | Material | Sigma-Aldrich | Z114014 | |
| Syringe, 60 mL | Material | BD Biosciences | 309653 |
参考文献
- Neufeld, J. D. DNA stable-isotope probing. Nat. Protocols. 2, 860-866 (2007).
- Neufeld, J. D., Boden, R., Moussard, H., Schäfer, H., Murrell, J. C. Substrate-specific clades of active marine methylotrophs associated with a phytoplankton bloom in a temperate coastal environment. Appl. Environ. Microbiol. 74, 7321-7328 (2009).
- Nercessian, O., Noyes, E., Kalyuzhnaya, M. G., Lidstrom, M. E., Chistoserdova, L. Bacterial populations active in metabolism of C1 compounds in the sediment of Lake Washington, a freshwater lake. Appl. Environ. Microbiol. 71, 6885-6899 (2005).
- Padmanabhan, P. Respiration of 13C-labelled substrates added to soil in the field and subsequent 16S rRNA gene analysis of 13C-labelled soil DNA. Appl. Environ. Microbiol. 69, 1614-1622 (2003).
- Bernard, L. Dynamics and identification of soil microbial populations actively assimilating carbon from 13C-labelled wheat residue as estimated by DNA- and RNA-SIP techniques. Environ. Microbiol. 9, 752-764 (2007).
- Haichar, e. l. Z. a. h. a. r., F, . Identification of cellulolytic bacteria in soil by stable isotope probing. Environ. Microbiol. 9, 625-634 (2007).
- Addison, S., McDonald, I., Lloyd-Jones, G. Stable isotope probing: Technical considerations when resolving 15N-labelled RNA in gradients. J. Microbiol. Meth. 80, 70-75 (2009).
- Buckley, D. H., Huangyutitham, V., Hsu, S. -. F., Nelson, T. A. Stable isotope probing with 15N achieved by disentangling the effects of genome G + C content and isotope enrichment on DNA density. Appl. Environ. Microbiol. 73, 3189-3195 (2007).
- Schwartz, E. Characterization of growing microorganisms in soil by stable isotope probing with H218O. Appl. Environ. Microbiol. 73, 2541-2546 (2007).
- Neufeld, J. D., Dumont, M. G., Vohra, J., Murrell, J. C. Methodological considerations for the use of stable isotope probing in microbial ecology. Microb. Ecol. 53, 435-442 (2007).
- Martineau, C., Whyte, L., Greer, C. Development of a SYBR safe technique for the sensitive detection of DNA in cesium chloride density gradients for stable isotope probing assays. J. Microbiol. Meth. 73, 199-202 (2008).
- Bartram, A. K., Poon, C., Neufeld, J. D. Nucleic acid contamination of glycogen used in nucleic acid precipitation and assessment of linear polyacrylamide as an alternative co-precipitant. Biotechniques. 47, 1019-1022 (2009).
- Chen, Y. Revealing the uncultivated majority: combining DNA stable-isotope probing, multiple displacement amplification and metagenomic analyses of uncultivated Methylocystis in acidic peatlands. Environ. Microbiol. 10, 2609-2622 (2008).
- Neufeld, J. D., Chen, Y., Dumont, M. G., Murrell, J. C. Marine methylotrophs revealed by stable-isotope probing, multiple displacement amplification and metagenomics. Environ. Microbiol. 10, 1526-1535 (2008).
- Kalyuzhnaya, M. High-resolution metagenomics targets specific functional types in complex microbial communities. Nat. Biotechnol. 26, 1029-1034 (2008).
- Binga, E. K., Lasken, R. S., Neufeld, J. D. Something from (almost) nothing: the impact of multiple displacement amplification on microbial ecology. ISME J. 2, 233-241 (2008).
- Green, S. J., Leigh, M. B., Neufeld, J. D., Timmis, K. N. . Microbiology of Hydrocarbon and Lipid Microbiology. , 4137-4158 (2010).
- Neufeld, J. D., Wagner, M., Murrell, J. C. Who eats what, where and when? Isotope-labelling experiments are coming of age. ISME J. 1, 103-110 (2007).
- Gallagher, E., McGuinness, L., Phelps, C., Young, L. Y., Kerkhof, L. J. DNA shortens the incubation time needed to detect benzoate-utilizing denitrifying bacteria by stable-isotope probing. Appl. Environ. Microbiol. 71, 5192-5196 .
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。