Method Article
RNAi筛选参与宿主因素牛痘病毒感染使用果蝇细胞
摘要
在病毒感染的新型主机所涉及的因素可以识别通过细胞为基础的的全基因组功能RNAi筛选的损失。一果蝇细胞培养模型的易用性和效率的RNAi由于这种方法特别适合。在这里,我们展示了这种技术使用牛痘病毒为例。
摘要
病毒性病原体是一个重大的公众健康构成威胁;病毒不仅能引起人类疾病的天然流行病,但他们在生物恐怖袭击的潜在用途也是一个问题。一个更好地了解细胞因子的影响感染,将有利于发展急需的疗法。 RNA干扰(RNAi)再加上几个生物的完整基因组测序技术的最新进展,导致全基因组,细胞为基础的损失的功能屏幕优化。果蝇细胞由于基因组规模屏幕特别适合在这个系统中1 RNAi的易用性和效率。重要的是,多种病毒可以感染果蝇细胞,包括哺乳动物病毒的医疗和农业的重要性2,3,4数量。前RNAi技术在果蝇中发现屏幕上有主机,是病毒感染的各个步骤,包括入境,翻译和RNA 的复制5所必需的因素。此外,许多在果蝇细胞培养的病毒复制所需的细胞因子也限制在与这些病毒4,6,7,8,9感染的人体细胞。因此,增选在病毒感染的宿主因素的识别抗病毒疗法的新目标。在这里,我们提出了一种高通量RNAi筛选,以确定参与病毒感染的细胞因子, 使用牛痘病毒为例的广义协议。
研究方案
第1部分:在384孔板的RNAi
- 筛选所用试剂的标准化是必不可少的。我们测试个别地段施耐德的媒体,胎牛血清,和染色试剂。然后,我们分装和使用整个屏幕的同一地段。
- 对于任何检测,这是最好的测试几种不同的细胞系,以确定哪些是最适合你的生物读出的。我们通常使用的S2 - DRSC线。
- 果蝇细胞成长在25 ° C在完成施耐德的媒体。
- 施耐德公司的媒体
- 10%热灭活胎牛血清
- 1X大号-谷氨酰胺
- 1X笔/链球菌抗生素
- 果蝇细胞半贴壁和代无胰蛋白酶关闭保温瓶的吹打细胞和稀释1:10到一个新瓶中的细胞每四天。细胞应在实验阶段日志。
- 通过使用多孔板所有液体自动液体处理板到板和每天的日常变异最小化。我们使用一个WellMate(矩阵)。
- 准备WellMate管。
- 喷雾用70%乙醇的发动机罩和WellMate。
- 从包装和喷雾用70%乙醇管。
- 在配药WellMate的位置插入管卡带。
- 总理管25毫升70%乙醇。
- 在乙醇中消毒15分钟,然后用另一乙醇25 ml总理管。
- 通过吸用50毫升无菌PBS冲洗管道。
- 打跑瓶和计数细胞。颗粒细胞,使你有25%以上(300xg,5分钟)。根据测定的长度,每孔接种细胞的数量必须优化。进行自动图像分析时,一个单一的单层无细胞聚集,是理想的图像分割。
- 1.7x10 6细胞/ ml颗粒细胞重悬于无血清施耐德的媒体。
- 我们执行的250ng/well浓度与市售的dsRNA库预分装在384孔板的屏幕。每块板的几口井空的控件,这是手动添加。我们用双链RNA的荧光素酶作为阴性对照。我们使用对主题(dIAP)双链RNA作为一个强大的RNAi控制。击倒,这种戏剧性的细胞死亡的抗凋亡因子结果,是一个快速直观的方式来评估敲质量。最后,我们使用一个双链RNA对病毒的记者,在这种情况下的β- galacosidase,作为阳性对照,验证,我们可以使用我们的检测读出抑制病毒的感染。如果发现成板的dsRNA在细胞播种,分装成的井,以便更容易可视化的角落1μL的双链RNA时,再离心到井底300xg,(1分)的双链RNA,然后加入细胞。
- 使用WellMate添加以上的准备,以每孔10μL,细胞。
- 自旋离心300xg,1分钟到板的细胞。
- 在25 °彗星孵化器孵育45分钟。
- 加入20微升完成施耐德的媒体和离心机300xg,1分钟。
- 放置在加湿的特百惠容器板内衬水浸泡过的纸巾。 384卷,是小;因此,蒸发生物学,这反过来又可以导致在边缘井读出的artifactual变化上可以有很大的影响。为了克服这个问题,板应保持在高湿度。
- 孵育3天,让强大的击倒。有没有必要改变媒体或在这段时间内打开加湿容器。
第2部分:与牛痘病毒感染
- 消毒多渠道Quatricide 15分钟的吸气,然后用清水冲洗70%的乙醇。
- 准备WellMate如上所述。
- 完整的媒体1:5稀释血清中的自由媒体,准备用2%FBS感染施耐德的媒体。
- 通过0.8微米的注射器过滤去除细胞碎片,或用纯化的病毒过滤器的原油病毒库存。
- 稀释牛痘病毒在2%血清施耐德的媒体获得的感染复数(MOI)2。
- 删除PBS WellMate油管,并用稀释的病毒的主要油管,直到无气泡。
- 使用消毒多渠道吸引器和真空歧管,轻轻地清除双链RNA 治疗的果蝇细胞板的媒体。
- 免除到使用的WellMate板,每孔30μL病毒。
- 在300xg离心板1分钟。
- 消毒WellMate管。总理与25毫升10%bleaCH,等待10分钟,并与另外25毫升10%的漂白粉总理。
- 其次是50毫升70%乙醇50 mL水总理油管。返回其包装的油管。
- 返回加湿特百惠容器板。在25 ° C培养48小时。
第3部分:染色板
- 固定板15微升4%甲醛/ PBS 10分钟。液体被删除,使用多渠道吸引器和真空多方面的每一步。一旦板块已经固定,无菌设备和试剂已不再需要。
- 删除修复和使用WellMate加30μLPBST(PBS + 0.1%的Triton - X)。孵育10分钟,然后重复。操作WellMate如上所述,除管不需要进行消毒。
- 为了检测牛痘感染,我们使用了β-半乳糖苷酶的记者,早/晚痘苗病毒启动子驱动。
- 10分钟,取出块中的液体,并培育的细胞(2%BSA PBST)。
- 稀块中的主要抗体,除去液体,并加入15μL抗体,每口井重复使用多通道移液器(矩阵)。
- 离心板(300xg,1分钟),盖有一个明确的贴纸,4℃过夜孵育。
- 删除的主要抗体,并使用WellMate PBST 3次,每次10分钟,洗井。
- 稀释荧光标记二抗(FITC)和核染色块中,并加入15μL井使用多渠道重复移液器。
- 孵育板在室温下1小时,由轻保护。
- PBST 3次,每次10分钟使用的WellMate洗井。
- 密封板明确的贴纸,盖,并将其存储在4 ° C至三个星期。
第4部分:成像和图像分析
- 使用20X放大倍率的自动显微镜图像板。
- 共核(DAPI)和感染的细胞(FITC)捕捉的图像。优化的曝光时间分别为每个通道,以最大限度地提高动态范围。
- ž堆栈的图像,除了间隔1微米,即预期的焦点平面的上方和下方的飞机跨越微调自动对焦功能。一旦最佳焦平面的位置,相应地调整自动对焦设置。
- 整个板块图像,捕捉的Hoechst通道(DAPI)和病毒的通道(FITC)每口井至少有三个图像。选择从几个不同的区域内,以及土地,以捕捉到的图像代表以及作为一个整体。
- 使用MetaXpress软件写日记来分割图像,并使用Count核模块计算出的细胞为核染色(细胞总数)和感染(FITC)为每个板块阳性的总数。
- 计算%的感染(100 * FITC阳性/总细胞)和日志转换。
- 要确定候选人,我们使用基于每块板的中位数和四分位距的一个功能强大的Z评分。这种方法是对异常和非对称数据,从而提供了更大的功率确定,即使弱或中度命中10。对于每个板块,中位数是从数转换的数据中减去。然后把结果值分为四分位距(75 个百分位值和25 个百分位之间的差额)的0.74倍。 0.74的因素是用来近似标准正态分布。
- 强大的Z分数是衡量标准偏差距离该板块的中位数。例如,一个强大的Z - score模型2%以及感染是从板块中位数或P <0.05〜2标准偏差。
- 分析细胞数量,以确定细胞毒性候选人。核计数,计算功能强大的Z评分。与一个强大的Z <重复屏幕-2候选人被认为是细胞毒性。
- 井无细胞毒性%> 2或<重复屏幕-2(P <0.0025)感染的一个强大的Z得分,被认为是从主屏幕上的潜在候选人。
- 积极的候选人是使用独立的试剂验证。 dsRNAs对从其他地区的mRNA的兴趣的基因产生和测试以上。
- 可以考虑进一步研究这些基因的两个独立的dsRNAs有相同的表型。
第5部分:代表性的图像和解释的感染率
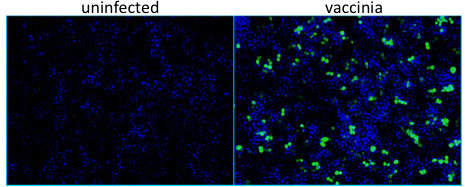
图1未感染井不显示任何染色牛痘病毒蛋白,而感染以及有代表性的包含痘苗病毒表达β-半乳糖苷酶(βgal)蛋白作为免疫荧光显微镜测量,染色阳性的细胞。病毒=绿色,细胞核=蓝色。
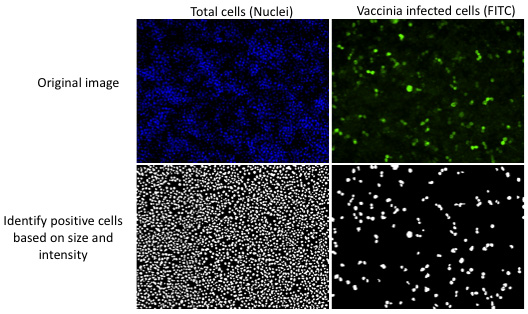
图2。自动图像分析软件量化感染在每个图像使用由用户设置的参数。在一个形象代表,总数计数的细胞核和细胞表达病毒抗原的数量,大小和强度高于当地的背景染色染色的基础上。

图3。击倒线程(dIAP)作为阳性对照为强劲的RNAi靶基因的枯竭。这种抗凋亡因子击倒导致戏剧性的细胞死亡。核=蓝色。
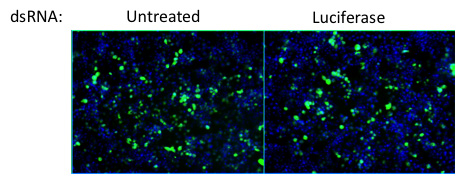
图4。击倒的萤光素酶作为阴性对照为双链RNA对感染的治疗效果。荧光素酶的消耗相对左未经处理的细胞没有感染的效果。病毒=绿色,细胞核=蓝色。

图5。βgal蛋白质击倒作为一个在减少感染的阳性对照,自检测使用βgal的蛋白水平,作为一个读感染。消耗在感染细胞的百分比下降的βgal结果。病毒=绿色,细胞核=蓝色。
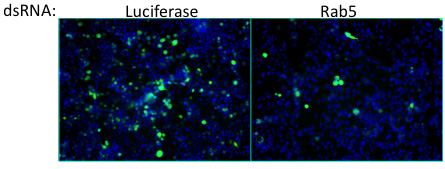
图6。击倒Rab5细胞因子在感染细胞的百分比下降的结果。由于Rab5是已知参与内吞作用,这一因素可能有助于牛痘病毒进入。这代表了从屏幕上的例子离群。病毒=绿色,细胞核=蓝色。
讨论
在果蝇的全基因组的RNAi筛选提供了一个强大和有效的方法,研究病毒感染的细胞成分。一些哺乳动物病毒感染的果蝇细胞,它可以用来识别主机的固有免疫反应的组成部分和主机病毒的复制,可能代表新的治疗目标所需的蛋白质因子。执行屏幕前,必须精心优化检测类型的细胞,细胞数量和感染程度,,必须得到很好的控制,包括正反两方面的病毒和细胞的目标11。之间的阳性和阴性对照,以确保足够的分离是很重要的事先筛选,最大限度地提高检测的动态范围。已执行的画面后,考生可以分成功能类别,以确定哪个主机机制有助于感染。对于哺乳动物,如牛痘病毒,通过RNAi的哺乳动物同源的贡献应确定二次分析的一部分。两者合计,这些强大的筛选方法将让我们获得洞察到其中的病毒与宿主细胞相互作用的复杂机制。
披露声明
致谢
支持这项工作是由NIAID的(R01AI074951,U54AI057168)和宾夕法尼亚大学基因组到SC前沿研究所资助,由美国国立卫生研究院T32HG000046到TSM;从美国国立卫生研究院T32GM07229和T32AI007324劳资关系组。
材料
| Name | Company | Catalog Number | Comments |
| Cell culture | |||
| Schneider’s media | GIBCO, by Life Technologies | 11720 | |
| GlutaMAX 100X | GIBCO, by Life Technologies | 35050 | |
| Penicillin/streptomycin (pen/strep) | GIBCO, by Life Technologies | 15140 | |
| Fetal Bovine Serum (FBS) | Sigma-Aldrich | F6178 | |
| Screening | |||
| Genome-wide dsRNA library | Ambion | ||
| 384 well plates, black with clear bottom | Corning | 3712 | |
| Aluminum seals | Corning | 6569 | |
| Clear seals | Denville Scientific | B1212-4 | |
| Well Mate | Matlab | 201-10001 | |
| 24 pin manifold | Drummond Scientific | 3-000-101 | |
| H–chst 33342 | Sigma-Aldrich | B2261 | |
| Fluorescently-labeled secondary antibodies | Invitrogen | ||
| ImageXpressMicro automated microscope | Molecular Devices | ||
| MetaXpress image analysis software | Molecular Devices |
参考文献
- Ramadan, N., Flockhart, I., Booker, M., Perrimon, N., Mathey-Prevot, B. Design and implementation of high-throughput RNAi screens in cultured Drosophila cells. Nat Protoc. 2, 2245-2264 (2007).
- Chotkowski, H. L. West Nile virus infection of Drosophila melanogaster induces a protective RNAi response. Virology. 377, 197-206 (2008).
- Blondel, D., Petitjean, A. M., Dezelee, S., Wyers, F. Vesicular stomatitis virus in Drosophila melanogaster cells: regulation of viral transcription and replication. J Virol. 62, 277-284 (1988).
- Sessions, O. M. Discovery of insect and human dengue virus host factors. Nature. 458, 1047-1050 (2009).
- Cherry, S. Genomic RNAi screening in Drosophila S2 cells: what have we learned about host-pathogen interactions? Curr Opin Microbiol. 11, 262-270 (2008).
- Hao, L. Drosophila RNAi screen identifies host genes important for influenza virus replication. Nature. 454, 890-893 (2008).
- Cherry, S. COPI activity coupled with fatty acid biosynthesis is required for viral replication. PLoS Pathog. 2, e102-e102 (2006).
- Cherry, S. Genome-wide RNAi screen reveals a specific sensitivity of IRES-containing RNA viruses to host translation inhibition. Genes Dev. 19, 445-452 (2005).
- Moser, T. S., Jones, R. G., Thompson, C. B., Coyne, C. B., Cherry, S. A Kinome RNAi Screen Identified AMPK as Promoting Poxvirus Entry through the Control of Actin Dynamics. PLoS Pathog. 6, e1000954-e1000954 (2010).
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A Simple Statistical Parameter for Use in Evaluation and Validation of High Throughput Screening Assays. J Biomol Screen. 4, 67-73 (1999).
- Echeverri, C. J., Perrimon, N. High-throughput RNAi screening in cultured cells: a user's guide. Nat Rev Genet. 7, 373-384 (2006).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。