需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
生产 C。线虫 galK选择标记
摘要
能力生产的转基因线虫使用fosmids进行基因组DNA作为保留所有原生的调控元件,是特别有吸引力。描述的是一个简单和强大的过程,通过生产转基因的重组工程 galK选择标记。
摘要
转基因动物的建立是广泛使用在 C 线虫的研究,包括研究有关规例及利益或代串联亲和纯化(TAP)的标签的特定基因的版本,以方便其净化的基因表达模式的使用GFP融合蛋白。典型的转基因是通过配售上游启动子的GFP报告基因或cDNA利益的产生,这往往产生一个代表性的表达模式。然而,基因调控的关键要素,如控制在3'非编码区或替代促销员元素,,可以错过这种方法。进一步只有一个单一的拼接变异体,通常可以通过,这意味着研究。相比之下,使用fosmid DNA克隆的蠕虫病毒基因组进行DNA的可能包括大多数,如果不是在基因调控在体内 ,它允许更大的能力,捕捉到真正的表达模式和时序所涉及的所有元素。为了方便使用fosmid DNA的转基因代,我们描述了一个E 。 大肠杆菌基于重组工程的程序,绿色荧光蛋白,TAP标签,或其他感兴趣的序列插入到任何基因的位置。该过程使用都在重组工程的正面和负面的选择步骤,高效率地获得所需的修改结果 galK基因作为选择标记。此外,包含galK基因两侧常用的绿色荧光蛋白的同源性武器和TAP融合基因的质粒可减少50%的成本寡核苷酸时产生的GFP或TAP融合蛋白。这些质粒使用R6K复制起点,这就排除了需要进行广泛的PCR产物纯化。最后,我们还展示了技术集成允许fosmid直接注射或轰击成蠕虫产生转基因动物的fosmid骨干UNC - 119标记。这个视频演示,通过使用这种方法重组工程生成转基因有关手续。
研究方案
概述
许多转基因在转基因C.代线虫包括启动子序列,也许是基因的cDNA克隆到一个由博士安迪消防1实验室生成的向量。虽然这些转基因往往与生产GFP报告基因的cDNA表达所需的模式方面的成功,这些转基因缺乏备用的推动者,增强子元件,和3'非翻译区(UTR)元素发挥重要的作用,在控制在体内的基因表达。例如,无论是DAF - 12和FAH - 1基因位于近端启动子,启动子只错过了构造 3,4,5之外的重要的增强子元件。此外,许多转基因构造使用的UNC - 54 3'UTR防止适当的microRNA 基因的调控6,7,8。因此,产生与蠕虫病毒的基因组DNA的大段转基因将被用于捕获所有的促销员,剪接变异体,并控制元素的3'UTR的理想选择。最近,一个C。线虫 fosmid库由〜40 kb的基因组DNA的地区,几乎涵盖了所有的基因组已经建成。使用蠕虫病毒基因组DNA进行,这些fosmid DNA克隆的结果,在更大的能力,捕捉到真正的2,8,9,10,11特定基因的表达模式和定时。
然而,与大区域的基因组DNA合作带来了很大的困难,如在使用标准的分子生物学技术12的实际挑战。为了克服这些限制,通过在大肠杆菌中同源重组技术来修改fosmids或细菌人工染色体大肠杆菌已被开发,被称为重组工程12,13。重组工程允许无缝绿色荧光蛋白,串联亲和纯化(TAP)的标签,或其他感兴趣的序列插入到任何位置的基因进行了C.线虫 fosmid克隆2,10,14。两侧由50个基点地区的同源性大肠杆菌特别改装的目标网站和目标DNA PCR产物之间发生同源重组大肠杆菌菌株。
最近,我们描述了一个两阶段的过程,修改的C其中包括插入galK基因在所需的位置,然后替换所需的序列 2基因的重组工程线虫fosmids。 galK基因作为一个支持和反对通过使用选择性生长培养基15的选择,因为它可以有效的选择标记的过程中这两个步骤。在第一阶段fosmid修改,galK基因通过同源重组插入所需的位置,确定能够利用半乳糖作为碳源2,15正选择正确修改fosmids。在第二阶段,该galK基因被替换所需的顺序,通过阴性选择反对通过使用有毒的半乳糖衍生deoxygalactose杀死galK +细菌2,15 galK基因识别和正确修改fosmids。 galK优势的正面和负面的选择步骤使用一个单一的基因的能力,而不是其他标志物,在获得高效率2,15的需要修改的每一步单独的基因,结果。
为了促进这项技术的应用为 C 。 线虫研究,我们提出了几个可用的资源的变化。首先,绿色荧光蛋白和TAP标签通常用于生成蠕虫病毒转基因,所以我们建立了地区的同源性在50个基点,这些标签将每个pMOD4 galK - G和pMOD4 galK GT质粒的galK基因源2。这些地区允许的fosmid修改,从而节省了需要为了一个比较昂贵的寡核苷酸的第二套两个阶段使用的寡核苷酸的单套。其次,使用这些质粒R6K这就排除了消化父质粒或广泛的PCR产物纯化的需要,是不是在为重组工程中使用的细菌能够复制父质粒的复制起点,只能复制特殊菌株,如EC100 2 ,16(见表1和表2)。最后,产生转基因C的常见方式线虫是通过使用选择通过转基因蠕虫的UNC - 119突变17救援基因枪法。为了使fosmids轰击兼容,我们开发的pLoxP UNC - 119可用于整合fosmid骨干2 UNC - 119标记的质粒。
一寡设计
随着重组工程所需的序列可以在任何网站内的基因插入。常见的部位是在5'端或3'末端功能域,剪接变异体的翻译后修饰,如裂解蛋白酶。的pMOD4 GFP质粒由本实验室创建可用于在任何网站中插入一个标志,标签的GFP质粒包括一个启动密码子和缺乏3'终止密码子(图1)。相比之下,TAP标签的具体版本为5'和3'融合,由于用于裂解纯化 18,19期间的TEV 。
- 计划内的基因标记插入网站。考虑插入部位时,考虑替代促销员,功能域,替代切片,和翻译后修饰。可用于不同的插入位点标记,一个或一些特定基因亚型。确定50个基点地区的插入点的上游和下游。
- 设计的寡核苷酸(见表1)。为100纳米尺度 - 凝胶纯化的寡核苷酸,或从综合DNA技术Ultramer寡核苷酸,可用于程序。
要执行galK重组工程,你需要设计所需的站点进行修改,这些引物的3'端侧翼一个地区的50个基点的同源性 galK引物结合 galK卡带,这是pMOD4 galK - G和pMOD4 galK - GT(图1)。将是向前寡核苷酸5' ------- 50个基点的同源性------- CCTGTTGACAATTAATCATCGGCA - 3'和5' ------- 50个基点的互补链上的同源性反向 - ------ TCAGCACTGTCCTGCTCCT - 3“。
要执行galK与GFP的重组工程,你需要设计50 bp的同源性pMOD4 galK - G或pMOD4 galK GT引物侧翼所需的站点进行修改(图1)的一个地区。一定要保持在框架的融合蛋白。如果需要的话,可以被丢弃的ATG。这些引物的3'端绑定到绿色荧光蛋白基因同源性的区域,侧翼galK卡带。注意:GFP的第一个和最后一个密码子强调证明阅读框。前锋寡将5' ------- 50 bp的同源性------- ATG GATTACAAGGACGATGACGATAAGATGAG -3'3'和反向5' ------- 50个基点同源的互补链------- 民航局 AGCTTGTGGGCTTTTGTATAG - 3“
要执行galK长期的C - TAP重组工程,你需要pMOD4 galK - GT的引物设计与50 bp的同源性区侧翼所需的站点进行修改(图1)。一定要保持在框架的融合蛋白。这些引物的3'端结合地区TAP的同源性,侧翼galK卡带。注意:水龙头的第一个和最后一个密码子强调证明阅读框。前锋寡将5' ------- 50 bp的同源性------ GAAAAGAGAAGATGGAAAAAG ATG - -3'和反向5' ------- 50个基点的互补链上的同源性- ------ GGT的 TGACTTCCCCGC -3“
要执行galK长期的N - TAP重组工程,你需要pMOD4 galK - GT的引物设计与50 bp的同源性区侧翼所需的站点进行修改(图1)。一定要保持在框架的融合蛋白。这些引物的3'端结合地区TAP的同源性,侧翼galK卡带。注意:水龙头的第一个和最后一个密码子强调证明阅读框。前锋寡将5' ------- 50 bp的同源性------ GCAGGCCTTGCGC ATG - -3'和反向5' ------- 50个基点的互补链上的同源性- ------ AAG TGCCCCGGAGGATGAGATTTTCT -3“ - 生成一个侧翼寡核苷酸在以后的步骤,以及进行PCR测序的fosmid。这些都是标准的PCR寡核苷酸,应结合〜100 bp的插入位点的上游和下游。
二。转让Fosmid SW016细菌
三 fosmids 线虫 fosmid库提供的EPI300的菌株(F - MCRAΔ(MRR - hsdRMS mcrBC)φ80dlacZΔM15ΔlacX74recA1 endA1 araD139Δ(ARA,LEU)7697嘎鲁galKλRPSL nupG trfA托纳)(震中生物技术,麦迪逊,威斯康星),它允许fosmid表达上述每个细胞单拷贝在纯化过程中提高(见表2)DNA产量增加。重组工程,将需要转移的SW106菌株(MCRAΔ(MRR - hsdRMS mcrBC)ΔlacX74deoR endA1 araD139Δ(ARA,LEU)7697 RPSL recA1 nupGφ80dlacZΔM15[λc1857(CRO bioA)<>春节 fosmid (CRO bioA)<> ARAC PBAD的CreΔgalK)(NCI,冯检基)菌株进行λred同源重组基因控制温度敏感的λ阻遏和阿拉伯糖诱导的Cre重组(见表2)15 。
- 订购fosmid克隆感兴趣的基因从GeneService(GOI)(英国剑桥)使用WormbASE作为指导。选择克隆时,我们选择那些有印尼政府序列的中心。排除邻近基因可能是可取的,但可能难以找到。
- 印尼政府文化fosmid克隆在LB含37 12.5μg/ mL氯霉素° C。
- 增长的fosmid过夜培养1.5毫升,和小型预习fosmid DNA使用震中fosmid制备试剂盒(震中生物技术,麦迪逊,威斯康星州)。我们按照指令,其中包括增加在前面的步骤Riboshredder组合中所描述的备用协议。
- 确定分光光度计fosmid DNA浓度。
- 准备在14毫升单元帽筒增长12.5μg/ mL氯霉素LB培养基,在32 ° C过夜培养的SW106 5毫升electrocompetent SW106细胞
- 2升容量瓶中氯霉素1毫升到100毫升的LB接种。一个外径600 0.6-0.8增长SW106细菌。不热休克。
- 在5分钟5000xg离心沉淀,悬浮颗粒,温和的震荡,并加入50 mL冰冷10%甘油。重复此清洗步骤一次。
- 沉淀SW106离心,吸出所有的,但每个上清〜500μL
- 重悬浮颗粒,温和的震荡。冻结100μL分装在液态氮或干冰,并储存在-80 ° C供日后使用。
- 转换成electrocompetent SW106细胞,通过电穿孔〜50 fosmid的DNA,用1350伏在0.1厘米的差距比色皿的Eppendorf 2510 electroporator吴细菌fosmid的DNA。
- 恢复1小时1毫升LB培养基,在32 ° C的细菌
- 氯霉素(12.5微克/毫升)和孵化的LB平板在32 ° C过夜板等分。
- 验证菌落PCR印尼政府的存在。一个5毫升的LB培养过夜增长12.5μg/ mL氯霉素在32 ° C一个标准的侧翼寡核苷酸的PCR反应中加入0.5μL文化,并增加最初的95℃孵育5分钟裂解细菌进行PCR之前。
- 甘油股票准备长期储存。
三。通过重组工程galK基因插入
fosmid修改的第一阶段,galK基因通过同源重组插入的fosmid,并选择正确修改fosmids增长最小的媒体含有半乳糖作为唯一的碳源(图2A) 。 SW106细菌生长缓慢上最小的媒体和3-5天才能看到的殖民地。
- 准备MOPS最小媒体板块,含0.2%的半乳糖。 MOPS最小媒体Teknova公司(霍利斯特,加利福尼亚州)(产品编号M2106),但不包括血糖。
MOPS最小媒体与半乳糖0.2%(1升)
高压灭菌15克在870毫升的水琼脂
冷却至55 ° C,并添加:100毫升 最小10X MOPS媒体 5毫升 0.2 mg / ml的D -生物素(无菌过滤) 4.5毫升 10毫克/毫升L -亮氨酸(1%,加热,然后冷却下来,无菌过滤) 10毫升 20%的半乳糖(灭菌) 1毫升 12.5 mg / ml的氯霉素在乙醇 2.55毫升 20%NH 4 CL 10毫升 0.132中号二元磷酸钾 - PCR扩增pMOD4 galK - G或pMOD4 galK GT磁带使用上述设计的引物。我们使用Phusion(New England Biolabs公司,伊普斯维奇,MA)或GoTaq(Promega公司,麦迪逊,威斯康星州)。
- 凝胶净化产生的乐队。量化凝胶或Nanodrop分光光度计产量。此PCR产物,是准备步骤3.14。
- 接种SW106细胞的fosmid DNA含有氯霉素(12.5μg/μL的)于5 mL LB培养过夜。增长32 ° C。
- 设置摇晃水浴至42 ° C至热身用无菌的持有人250 ml容量瓶中。使用一个惊天水浴是获取高效率的的关键。
- 加入1毫升至100毫升和2升容量瓶中氯霉素的LB培养过夜。成长为一个外径0.6-0.8。这通常需要3-4个小时。
- SW106细胞50毫升转移到250 ml容量瓶中和热休克42℃,整整20分钟。在100转的摇晃水浴离开非诱导控制在32 ° C其余的细菌。
- 清凉冰10分钟的诱导和未诱导细菌。
- 转让〜5000xg两个无菌离心管和沉淀的样品为5分钟。
- 倒掉上清液,并在1毫升冰冷的10%甘油温和的震荡(即设置3-4)重悬沉淀。
- 当重悬,添加49毫升的冰冷10%甘油,和颗粒〜5000的样本XG为5分钟。
- 再次重复步骤3.9,3.10和3.11。
- 反相管取出上清,在剩余的液体中悬浮颗粒(约500μL,每个)。分装成100μL的样品,冻结干冰,并储存在-80 ° C这些都是好几个星期到几个月。 (我们通常会停止在这里,并执行电翌日)。
- 150 PCR产物,在1350伏的Eppendorf 2510 electroporator设置使用0.1厘米的差距试管吴Electroporate诱导和未诱导SW106细胞。
- 恢复1毫升LB培养基,在14毫升猎鹰管的细菌。在32 ° C孵育4.5小时。
- 颗粒的细菌在离心13200 RPM为15秒。细菌悬浮在M9的,然后洗两次删除任何丰富的培养基(见下文为配方)。
- M9培养基(1升)
- 6克NA 2 HPO 4
- 3G KH 2 PO 4
- 1克的NH 4 CL
- 0.5克氯化钠
- 高压灭菌器
- 第二次洗涤后,取上清液被删除,颗粒悬浮在1毫升M9的电镀前连续稀释到最小媒体MOPS M9(100μL,100μL1:10稀释,加入100μL1:100)。
- 孵化3-5天在32 °的孵化器中的C。注:为真正的阳性生长缓慢,请耐心等待。
- 辅以数一个到麦康凯琼脂指示板(BD#281810)几个殖民地,用1%的半乳糖和12.5μg/ mL的氯霉素。所有的殖民地后出现的最后一步, 应 galK +,但以获得摆脱任何 galK -污染物,重要的是要获得单一的,明亮的粉红色菌落,然后进行第二步。 galK -殖民地将白/无色galK +菌将是明亮的红色/粉红色由于pH值的变化,发酵半乳糖在32℃孵育过夜后° C。
- 挑选单个菌落,接种增长的一个5毫升LB +氯霉素隔夜文化在32 ° C。
- 确认插入的galK基因,通过PCR的正确使用两侧的寡核苷酸的位置。加入0.5μL的文化,以一个标准的PCR反应和提高最初的95℃孵育5分钟裂解细菌。 PCR产物应upshifted大小因的galK基因的存在。
- 准备存储甘油股票。
四。 galK更换标签序列通过重组工程
在这个阶段galK基因替换所需的标记序列和正确修改fosmids选择对galK基因选择模拟deoxygalactose有毒半乳糖(狗)(图2B)。
- 准备MOPS最小媒体板,含0.2%deoxygalactose(狗)和0.2%的甘油。 MOPS最小媒体Teknova公司(霍利斯特,加利福尼亚州)(目录#M2106),但不使用包括葡萄糖。
MOPS最小媒体0.2%的狗和甘油(1升)
高压灭菌15克在860毫升的水琼脂
冷却至55 ° C,并添加:100毫升 最小10X MOPS媒体 5毫升 0.2 mg / ml的D -生物素(无菌过滤) 4.5毫升 10毫克/毫升L -亮氨酸(1%,加热,然后冷却下来,无菌过滤) 10毫升 20%deoxygalactose(无菌过滤) 10毫升 20%甘油(灭菌) 1毫升 12.5 mg / ml的氯霉素在乙醇 2.55毫升 20%NH 4 CL 10毫升 0.132中号二元磷酸钾 - PCR扩增,pBS1761 pMOD4绿色荧光蛋白(长期的N - TAP)的,或pBS1479(C -长期TAP)的标签使用相同的在第一轮使用,或使用较短的GFP或点击特定的寡核苷酸(步骤1.2的内部序列寡核苷酸的片段)。如果你正在多个构造,它是特别有用,使用较短的寡核苷酸,可以为所有的构造使用相同的PCR产物。
- 凝胶纯化PCR产物和措施,通过凝胶或分光的浓度。
- 产生诱导和未诱导主管SW106携带galK基因插入以下步骤3.4-3.13以上的fosmid。
- Electroporate〜100 PCR产物,在1350伏的Eppendorf 2510 electroporator设置使用0.1厘米的差距试管吴诱导和未诱导的SW106细胞。
- 在一个14毫升的管理单元帽筒和32℃摇床4.5小时孵育1毫升LB培养基中恢复。
- 清洗和稀释步骤3.16和3.17的细菌。 MOPS最小的媒体含有0.2%的2 - 脱氧-半乳糖(狗)和0.2%的甘油板的板细菌。
- 在32 ° C孵育3天。
- 四殖民地被用来制造5毫升12.5μg/ mL氯霉素的LB过夜培养。这些菌落PCR用于上述确认插入录像带。我们同时使用较短的GFP /点选特定的寡核苷酸和侧翼的寡核苷酸,以证明正确的插入和正确的网站。 GFP是〜800 bp和TAP是〜550基点, 而 galK 1.4 kb的。
- 准备一个甘油。
五, 增加CRE - loxP位的UNC - 119基因重组
修改fosmids产生转基因动物的一个常见的手段是通过利用基因枪法。这种技术使用DNA包覆的金颗粒引入 C. fosmid的DNA 线虫 。转基因动物通常是查明通过UNC - 119与UNC - 119基因突变的救援。在这一步,UNC - 119基因添加CRE - loxP位重组与pLoxP在独联体fosmid骨干UNC - 119质粒(图2C)。
- 准备主管SW106细菌进行步骤4.9使用步骤2.5-2.9修改fosmid。不要诱导在42 ° C。
- Electroporate与50纳克。 UNC - 119 pLoxP从使用0.1厘米的空隙比色皿,在1350伏的Eppendorf 2510 electroporator的小预习。
- 收回含有1个小时的0.1%,阿拉伯糖LB细菌在32 ° C
- 含50μg/mL氨苄青霉素和12.5μg/ mL氯霉素的LB板的板等分。孵育在32 ° C过夜。选择到fosmid pLoxP UNC - 119的整合。
- 生长在含50μg/mL氨苄青霉素和12.5μg/ mL氯霉素的LB培养过夜。使用0.5μL进行PCR验证的UNC - 119(5' - CAAATCCGTGACCTCGACAC - 3')和UNC - 119 R(5' - CACAGTTGTTTCTCGAATTTGG - 3')寡核苷酸(见表1)UNC - 119基因的存在。
- 制作一个最终fosmid的甘油。
六。大型Fosmid准备
为了便于获得轰击需要fosmid DNA的数量较多,在fosmid是EPI300细菌转移到了这一步。这种应变的能力,增加fosmid套数增加在DNA的制备收益。
- 在含氨苄青霉素和氯霉素LB 5.5步将从一个5毫升过夜培养的细菌在32 ° C。使用震中fosmid制备试剂盒,从1.5毫升文化隔离的fosmid。
- Electroporate〜50毫微克到0.1厘米的空隙比色皿,在1350伏的Eppendorf 2510 electroporator设置的EPI300细菌。 EPI300细菌就可以买到从震中生物技术(麦迪逊,威斯康星州)。
- 恢复磅为1小时,细菌在37 ° C。含有50μg/ mL和12.5μg/ mL氯霉素的LB琼脂板等分。
- 生长和诱导EPI300细菌含有使用附带的使用说明书的修改fosmid。 50毫升的诱导培养> 10微克纯化fosmid DNA。净化与震中fosmid准备套件fosmid。
七。轰击
- 使用10微克。 fosmid DNA轰击DP38蠕虫病毒(A. D. Hochbaum,弗格森和A.费舍尔,朱庇特,出版中)。
八。代表性的成果
fosmids通过重组工程稳健和成功率> 90%的负选择步骤修改定期观察2。该协议还需要1〜2个星期才能完成这使得转基因较快的准备。该协议也已试图通过与其他实验室成功20。
| 寡糖 | 序列 |
| 长期的C - TAP F | ATGGAAAAGAGAAGATGGAAAAAG |
| C -长期的TAP ř | GGTTGACTTCCCCGC |
| 标志- GFP的F | ATGGATTACAAGGACGATGACGATAAGATGAG |
| 标志的绿色荧光蛋白ř | CAAAGCTTGTGGGCTTTTGTATAG |
| 长期的N - TAP F | ATGGCAGGCCTTGCGC |
| N -长期的TAP ř | AAGTGCCCCGGAGGATGAGATTTTCT |
| galK F | CCTGTTGACAATTAATCATCGGCA |
| galK ř | TCAGCACTGTCCTGCTCCT |
| UNC - 119 F | CAAATCCGTGACCTCGACAC |
| UNC - 119 ř | CACAGTTGTTTCTCGAATTTGG |
表1。寡核苷酸进行PCR。
| 质粒 | 来源 | 可在 |
| Fosmid克隆 | Geneservice有限公司 | Geneservice |
| pGalK | 15 | NCI,冯检基 |
| pMOD4 - RT - G | 2 | Addgene |
| pMOD4 - galK - G | ||
| pMOD4 - galK - GT | ||
| pLoxP - UNC - 119 | ||
| pMOD4 - GFP | ||
| 细菌 | ||
| SW106 | 15 | NCI,冯检基 |
| EPI300 | 震中生物技术 | 震中 |
| EC100D PIR - 116 | ||
表2。应变和载体的可用性。
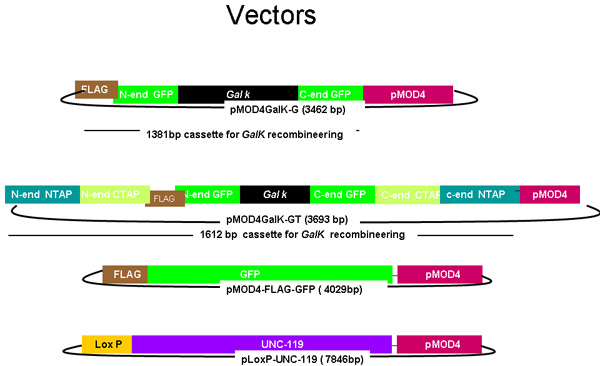
图1。
图pMOD4 - galK - G和pMOD4 galK - GT,pMOD4绿色荧光蛋白,pLoxP - UNC - 119
pMOD4 - galk - G质粒由PMOD 4 galK - GT galK由两侧由50个相同的5'和3'末端的标志(棕色)- GFP(绿色)的核苷酸地区galK卡带(黑色)两侧的FLAG - GFP的同源性区域和50个核苷酸地区相同的5'和3'N端和C端TAP(蓝色和橙色,分别)结束的录像带。 pMOD4 - FLAG - GFP由5'标志,标签和pLoxP UNC - 119的UNC - 119的基因组序列中包含一个loxP位网站质粒(紫色)组成的完整的GFP卡带。所有质粒利用的R6K基于pMOD4(红色)的骨干,这是无法复制在SW106。
图2。
galK重组工程过程概述
图2A - 2C显示的步骤和时间参与重组工程使用galK卡带的独立数字。这些都是相同的数字,在图2d合并,但分别提供清晰和易于阅读。两个步骤的过程涉及的50个基点地区的同源性,本录像带更换了FLAG - GFP或TAP - GFP的标志或TAP(图2A) 两侧的galK卡带插入利益fosmid是第一次修改(图2B)。后来用于产生转基因动物的UNC - 119标记插入LoxP位网站上fosmid骨干(图2C) 。
2D图显示了一个galK重组工程程序的合并数字。
概要描述的过程包括在上述合并图2A - 2C的每一步所需的时间galK重组工程。
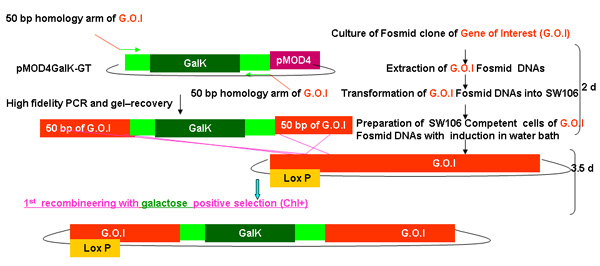
图2a。galk插入galk重组工程。
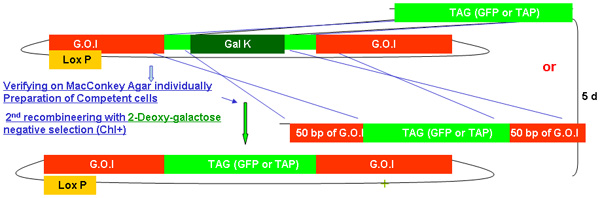
图2b。galk重组工程中的标签(GFP / TAP)插入。
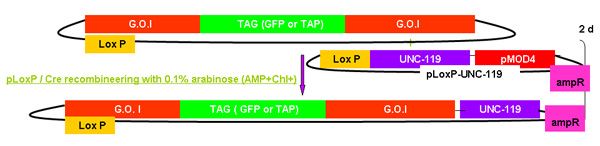
图2c。此外UNC - 119。
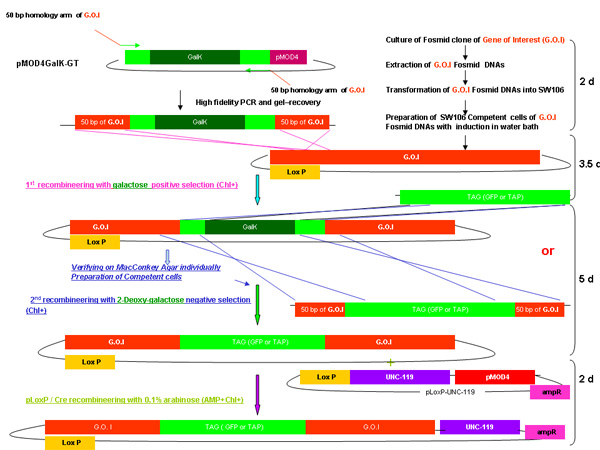
图2d。galk重组工程的合并概述。
讨论
转基因从fosmids一代提供保留所有本机的启动子,剪接变异体,和3'非编码区调控元件的利益。这可能会导致一个转基因这是原生的表达模式,或建设功能的基因,当其他方法失败5反射的建设。由此产生的转基因可以进行各种包括GFP或自来水标记的抗原表位的标签。
转基因的建设涉及到三个步骤都进行SW106细菌染色 15 。首先,galK基因融入在fosmid所需?...
披露声明
致谢
作者想感谢纳什与发展中国家的技术帮助林赛。这项工作是由美国国立卫生研究院授予AG028977 ALF的,从美国匹兹堡大学的一个OAIC匹兹堡大学(AG024827),种子基金的试点项目授予。
材料
| Name | Company | Catalog Number | Comments |
| FosmidMAX kit | Epicentre Biotechnologies | FMAX046 | |
| GoTaq | Promega Corp. | M7122 | |
| MOPS Media | TEKnova, Inc. | M2120 | |
| 0.132 M Potassium phosphate solution | TEKnova, Inc. | M2102 | |
| D-galactose | Sigma-Aldrich | G0750 | |
| 2-deoxygalactose | Sigma-Aldrich | D4407 | |
| Biotin | Sigma-Aldrich | B4639 | |
| Leucine | Sigma-Aldrich | L8000 | |
| NH4Cl | Sigma-Aldrich | A9434 | |
| Phusion DNA polymerase | New England Biolabs | F-530S | |
| MacConkey agar base | BD Biosciences | 281810 | |
| Arabinose | Sigma-Aldrich | A3131 | |
| Chloramphenicol | Sigma-Aldrich | C1919 | |
| Sodium phosphate dibasic | Sigma-Aldrich | S5136 | |
| Potassium phosphate monobasic | Sigma-Aldrich | P5655 | |
| Sodium chloride | Sigma-Aldrich | S5886 | |
| Glycerol | Sigma-Aldrich | G2025 | |
| Bacto Agar | BD Biosciences | 214010 |
参考文献
- Mello, C., Fire, A. DNA transformation. Methods Cell Biol. 48, 451-482 (1995).
- Zhang, Y., Nash, L., Fisher, A. L. A simplified, robust, and streamlined procedure for the production of C. elegans transgenes via recombineering. BMC Dev Biol. 8, 119-119 (2008).
- Antebi, A., Yeh, W. H., Tait, D., Hedgecock, E. M., Riddle, D. L. daf-12 encodes a nuclear receptor that regulates the dauer diapause and developmental age in C. elegans. Genes and Development. 14, 1512-1527 (2000).
- Snow, M. I., Larsen, P. L. Structure and expression of daf-12: a nuclear hormone receptor with three isoforms that are involved in development and aging in Caenorhabditis elegans. Biochim. Biophys. Acta. 1494, 104-116 (2000).
- Fisher, A. L., Page, K. E., Lithgow, G. J., Nash, L. The Caenorhabditis elegans K10C2.4 Gene Encodes a Member of the Fumarylacetoacetate Hydrolase Family. A CAENORHABDITIS ELEGANS MODEL OF TYPE I TYROSINEMIA. J Biol.Chem. 283, 9127-9135 (2008).
- Wightman, B., Ha, I., Ruvkun, G. Posttranscriptional regulation of the heterochronic gene lin-14 by lin-4 mediates temporal pattern formation in C. elegans. Cell. 75, 855-862 (1993).
- Lehrbach, N. J. LIN-28 and the poly(U) polymerase PUP-2 regulate let-7 microRNA processing in Caenorhabditis elegans. Nat Struct Mol Biol. 16, 1016-1020 (2009).
- Tursun, B., Cochella, L., Carrera, I., Hobert, O. A toolkit and robust pipeline for the generation of fosmid-based reporter genes in C. elegans. PLoS One. 4, e4625-e4625 (2009).
- Bamps, S., Hope, I. A. Large-scale gene expression pattern analysis, in situ, in Caenorhabditis elegans. Brief. Funct. Genomic. Proteomic. , (2008).
- Dolphin, C. T., Hope, I. A. Caenorhabditis elegans reporter fusion genes generated by seamless modification of large genomic DNA clones. Nucleic Acids Res. 34, e72-e72 (2006).
- Sarov, M. A recombineering pipeline for functional genomics applied to Caenorhabditis elegans. Nat. Methods. 3, 839-844 (2006).
- Yang, X. W., Model, P., Heintz, N. Homologous recombination based modification in Escherichia coli and germline transmission in transgenic mice of a bacterial artificial chromosome. Nat Biotechnol. 15, 859-865 (1997).
- Court, D. L., Sawitzke, J. A., Thomason, L. C. Genetic engineering using homologous recombination. Annu.Rev.Genet. 36, 361-388 (2002).
- Westenberg, M., Bamps, S., Soedling, H., Hope, I. A., Dolphin, C. T. Escherichia coli MW005: lambda Red-mediated recombineering and copy-number induction of oriV-equipped constructs in a single host. BMC Biotechnol. 10, 27-27 (2010).
- Warming, S., Costantino, N., Court, D. L., Jenkins, N. A., Copeland, N. G. Simple and highly efficient BAC recombineering using galK selection. Nucleic Acids Res. 33, e36-e36 (2005).
- Penfold, R. J., Pemberton, J. M. An improved suicide vector for construction of chromosomal insertion mutations in bacteria. Gene. 118, 145-146 (1992).
- Praitis, V., Casey, E., Collar, D., Austin, J. Creation of low-copy integrated transgenic lines in Caenorhabditis elegans. Genetics. 157, 1217-1226 (2001).
- Puig, O. The tandem affinity purification (TAP) method: a general procedure of protein complex purification. Methods. 24, 218-229 (2001).
- Rigaut, G. A generic protein purification method for protein complex characterization and proteome exploration. Nat.Biotechnol. 17, 1030-1032 (1999).
- Achilleos, A., Wehman, A. M., Nance, J. PAR-3 mediates the initial clustering and apical localization of junction and polarity proteins during C. elegans intestinal epithelial cell polarization. Development. 137, 1833-1842 (2010).
- Maduro, M., Pilgrim, D. Identification and cloning of unc-119, a gene expressed in the Caenorhabditis elegans nervous system. Genetics. 141, 977-988 (1995).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。