需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
计算机辅助大型胰岛质量,粒度分布和结构的可视化和量化
摘要
介绍了新型计算机辅助大型采购及免疫组织化学染色的胰腺癌标本的分析方法:(1)虚拟切片捕捉整个节(2)大规模数据的质量分析;(3)重建二维虚拟切片(4)三维胰岛映射;(5)数学分析。
摘要
胰岛是一个独特的微型几种激素的分泌,如β细胞(胰岛素),α-细胞(胰高血糖素),和delta -细胞(生长抑素)中嵌入外分泌组织,包括1内分泌细胞组成的器官 - 整个胰腺的2%。身体和胰腺重量之间有一个密切相关。总β细胞的质量也相应增大,以弥补在体内对胰岛素的需求。逃脱此相称的扩张是胰岛的粒度分布。大型动物,如人类有着相似的小鼠胰岛与粒度分布,表明这种微型器官具有一定规模的限制功能。的大型动物胰腺无法产生比例较大的胰岛细胞的胰岛细胞数量增加,增幅较大的胰岛细胞在其整体的胰岛大小分布比例补偿。此外,胰岛内细胞成分和不同物种之间的架构,也表现出各种病理生理条件下的同一物种的一个惊人的可塑性。在本研究中,我们描述了生物图像数据分析的新方法,以便分析过程的自动化,允许大,异构的数据集合,在这种动态的生物过程和复杂的结构的研究分析。这些研究受到阻碍,由于偏见的抽样技术困难,并产生大规模的数据集,能够精确地捕捉胰岛生物学的生物过程的复杂性。在这里,我们显示的方法收集样本有限(或尽量减少样品的采集)和标准的实验设置内不偏不倚的“代表”的数据,并精确地分析复杂的三维结构的胰岛。计算机辅助自动化允许的收集和分析大规模数据集,并保证数据的公正解释。此外,胰岛的粒度分布和空间坐标(即X,Y,Z轴位置)的精确量化,不仅会导致一个准确的可视化胰岛的结构和组成,但也使我们能够确定在开发过程中和适应的模式改变条件通过数学建模。在这项研究中开发的方法以及许多其他系统和生物体的研究。
研究方案
1。创建虚拟切片的免疫组织化学染色图像
- 打开StereoInvestigator。在显微镜持有人持有样品放入干净的幻灯片和“收购”,然后点击“即时影像”(收购→现场图像),可视化立体声调查。确定每个通道的暴露水平;在我们具体的例子,通道2的DAPI,通道的GFP,RFP的通道,通道5 4 3 Cy5的通道6用于Cy7。使用“视频直方图”窗口,其中显示的荧光强度,以确定曝光量。适当的荧光强度达到强度尾巴视频直方图右端。在“相机设置”窗口中的暴露水平是可以改变的。视频直方图可以聚焦显微镜对焦轮。
- 在创建虚拟切片,轮廓点远离样品,这将标志着一个参考点在屏幕上点击,然后点击样本的轮廓周围的样本。当轮廓完成后,右击并选择“关闭的轮廓”连接起点和终点。
- 一旦轮廓是封闭的,捕捉一个虚拟切片样本(收购→获取虚拟切片)。当虚拟切片窗口选项打开时,选择“高速获得”以及通过取消选中旁边的框自动聚焦“手册。”保存该文件。
- 虚拟切片预聚焦,必须选择校准(点击右键→添加到站点列表对焦),手动对焦几个随机的部分,即,网站,在虚拟切片预览。当几个地点都集中,启动“虚拟切片(右击→启动虚拟片与预聚焦)。第一个样本的虚拟切片完成后,切换到下一个通道,并相应地调整曝光。每个通道重复步骤4和5。
2。计算机辅助的二维分析
量化胰岛
- 然后,该图像处理使用ImageJ宏名为“IHCVS,”首先准备加载的图像进行分析,然后进行量化的胰岛功能,如细胞成分(即每个区β-,α-和δ-细胞的人口,胰岛在面积的总和值)。栈是分割成单独的频道,在ImageJ的,每个通道显示一个单独的图像,基于免疫组织化学染色。每幅图像,然后自动阈值,后一个8位黑白图像,或口罩的阈值图像,制作。单独通道的图像,然后相加成一个复合面膜算术,ImageJ的内置的粒子复合图像进行分析。
- 投资回报率,然后确定,而不含颗粒比单一的β细胞小(<170微米;面积计算使用一个直径约15微米;原等人,2003年)。 “由此产生的投资回报是分析和量化使用ImageJ;量化可以包括周长(一个距离周围的区域),圆(那里的人数1.0描绘了一个完美的圆某种程度的圆度),FERET的直径(在一个区域内最长的距离)和中心每个分析区域的坐标,使胰岛分布可以使用这些参数的各种组合数学分析和绘制在三维图形,数据将被用于数学建模。一个单一的虚拟切片图像分析在30秒内。 ImageJ宏语言支持的循环语法嵌入到图像处理和分析脚本,可以分析多个图像。循环打开在一个给定的的目录中的所有文件,分析图像,和结果输出到一个新位置作为一个Excel文件。
- 汇总结果保存到Excel电子表格,存储数据,如面积,周长,圆形,FERET的直径,并为每个胰岛的胰岛中心,以及相应的数字图像标记。
计算分析和直方图安装
- Mathematica的脚本,运行后自动启动,使用过程中的电子表格收集的数据,金额达数以十万计的条目。运行的关键过程之一,是一个频率的分析,它构造用于研究粒度分布,也确定在数据的离群直方图。根据脚本,总胰岛面积测量和其他参数相比,特别是时间点或胰腺的年龄,然后相应地在图中显示。
3。免疫组织化学染色二维虚拟切片图像三维重建
虚拟切片的三维重建
- 脚本复制到该目录(即文件夹),(“im_jp2_2_tiff”)包含JP2图像。到控制台中键入“./im_jp2_2_tiff”,然后按Enter运行Linux的shell脚本。运行这个脚本会自动转换成TIFF文件,这是从虚拟片立体堆叠的建设和量化使用的格式的所有目录中的JP2图像。 (注:键入“CHMOD + X im_jp2_2_tiff”如果不运行脚本,将脚本转换成文本文件,以便它可以正常运行)
- 当所有拍摄的图像已经转换JP2 TIFF文件,他们准备使用ImageJ分析。
- 随着ImageJ,打开第一个样本(5张图像在这种情况下的DAPI,GFP,RFP,Cy5的,Cy7)的所有图像,并合并为一个图像(图像→颜色→合并通道)。每个样本重复这一过程。当所有的样本已合并为单一的图像,干净的图像,通过选择“多边形选择”工具不必要的地区,并填写(“编辑”→填写“)。 (调整图像的大小尺寸更小,如果有必要)。每个图像转换成RGB彩色图像(图像→类型→RGB颜色)。最后,创建一个堆栈的图像(图像→堆叠→图片到堆栈),之后,他们可以进一步“堆栈注册”插件一致,如有必要。最终,合并的渠道,然后结合图像创建3D的整个胰腺中的胰岛的蒙太奇。查看图像,点击“插件”,然后“3D查看器。”
4。胰岛映射
收集图像堆栈
- 位于整装胰岛细胞,在显微镜下。
- 打开Slidebook软件。访问“捕捉和集中控制”,按“CTRL + SHIFT + E”在“焦点控制”窗口,设置目标,20X或40X,胰岛的大小而定。适用于水,如果使用水浸泡物镜。
- 为了使用显微镜目镜,找到小岛,设置“斌”2X和“焦点控制”窗口中设置过滤器设置为User1。 “发射选择”应设置为100%的眼睛和中性密度为1。点击“绿色荧光蛋白EY”,然后“打开福陆公司”。
- 为了形象化Slidebook样品,返回的重点控制“窗口,切换的”过滤器设置“,”活“,并点击”绿色荧光蛋白DS。“中心的胰岛在摄像机1窗口,以及可能使用操纵杆。焦点胰岛细胞很容易就可以看出端倪中心附近的某处深度。
- 在“焦点控制”窗口中,“选择的”Z“选项卡。”点击设置在上左上角的窗口设置为参考点当前的深度。使用范围控制,滚动到的胰岛的功能,可以清楚地看出端倪的最高深度,然后点击“设置首页。”滚动到胰岛的底部,并点击“设置底部。”点击“GO”返回到参考点。
- 在捕捉窗口中,设置“斌因子”,“2X”,并设置“过滤器设置”,“活。”设置“捕捉型”,“3D”(只有3D)。在右侧窗口中,单击“使用顶部和底部的位置。”点击“返回捕获后参考点。” “步长”设定为“3”。
- 下方的“过滤器集箱,”选择的DAPI DSU,GFP,RFP的DSU DSU和Cy5的DSU。对于每一项,单击“查找最佳”下的“调整曝光”,然后点击“测试”,以确保所选的曝光是合适的。点击“开始”,开始捕获。
- 当捕捉完成后,作必要的调整水平,并为RFP的改变显示的颜色为白色。保存幻灯片,去查看→出口→TIFF系列。在年底破折号幻灯片的名称类型,并保存它。几十个人TIFF文件的创建,因此,它可能会有所帮助保持在一个单独的文件夹中的每个胰岛的形象栈。
映射图像堆栈
- 打开立体声调查。可视化的形象,开放的形象堆栈(文件→形象堆栈→图像堆栈打开)。在“图像缩放”菜单,“设置”图片“,以3.00微米之间的距离。要正确Slidebook 2X分级,选择“覆盖X和Y方向的”和“用户指定的X和Y来源”缩放“,然后输入X和Y 0.65
- 兴趣在胰岛中单击“宏”窗口,中心的形象。“标记至少一个单元格,使用适当的标记。 α细胞β细胞会出现绿色的,会出现红色,并三角洲细胞会出现白色的;使用标记(空心圆圈),以纪念β细胞,标记5(空心三角形)标记α细胞,并标记6(一个空心三角)三角洲细胞。应标明细胞在其细胞核内,会出现蓝色的,如果样品已经DAPI染色的中心。
- 至少一个单元格已被标记后,显示“正交视图”使用在顶部的菜单图标。检查“Z型过滤器”和“对称”。范围should设置为15.00。 0.0 Z水平开始,滚动使用鼠标滚轮的胰岛与相应的标记和标记每个单元。应标明每个单元只有一次在其深度的中心。后整理标记的Z -水平,“Z型过滤器”复选框可以取消。这将显示所有的Z -水平的标志。
- 当每一个细胞已被标记为“DAT”文件,保存文件,然后转到文件→导出跟踪。保存为“txt”文件的跟踪。点击“新的数据文件”来清除,然后再尝试新的胰岛映射的工作区。
5。代表性的成果:
编写的虚拟切片的免疫组织化学染色胰腺样本使研究所有的内分泌细胞(α,β,δ-细胞)在整个胰腺,双方共同为胰岛细胞(图1A),并在个别独立的通道(图1B)。随着计算机程序和脚本的协助下,一个大规模的数据质量分析,可以执行这些虚拟切片。具体来说,颗粒分析的复合口罩(图1C)含有胰岛面积,周长(周边地区的距离),圆(一个圆度的程度,其中1.0代表一个完美的圆)等参数作为统计表的输出, FERET每个胰岛的直径(在一个区域内的最长的距离)检测(图1D)。大型分析这些图像在生产总胰岛数量和规模分布直方图,以及α-,β-,和细胞三角洲地区的一个详细的比较结果。此外,每个虚拟切片是在约5微米的深度,和所有个人的二维虚拟切片进一步堆放创建一个三维重建整个胰腺样本。胰岛映射演示不仅捕获3D胰岛的另一个实例,但详细的电脑辅助分析。胰岛映射由不同的胰岛捕获(图2A)和随后的标记α-,β-和δ-细胞在不同的Z型飞机(图2B)在3D可视化的胰岛(图2C,D)。自动映射胰岛数学分析显示细胞的组成和体系结构,包括细胞与细胞间的距离(图2E)和细胞与细胞间的距离分布(图2F)的累积概率。
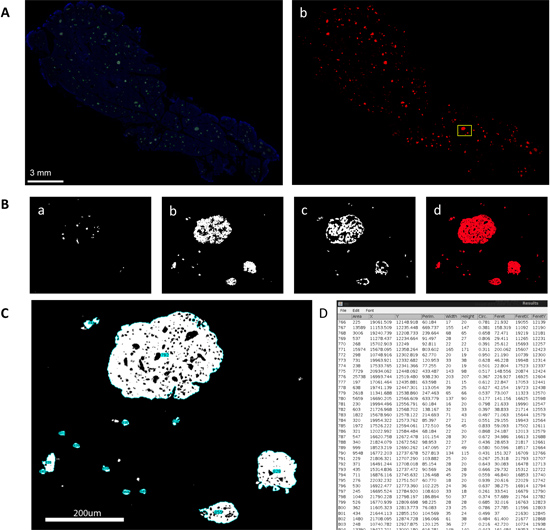
图1。大规模的采集和分析胰岛的分布,使用虚拟切片。 A.虚拟人胰腺部分切片的看法。 A.胰岛素免疫组化染色(绿),胰高血糖素(红色),生长抑素(白)的DAPI(蓝色)。 B.转换后自动阈值8位掩码。在每个通道的BB的意见是盒装的区域被放大。 A.三角洲细胞,B。 β细胞,C。 α-细胞和D。合并后的复合图像。 C.颗粒分析复合面膜后执行。请注意,每个胰岛的结构,包括小细胞群的编号为(蓝色亮点)。 D.统计表个别结构测量的各种参数,有对应的C所示的标签标识

图2虚拟切片的免疫组织化学分析。答:3D散点图图1显示的大小和形状各胰岛的分布参数,如面积,圆度和FERET的直径。 B.三维散点图图1显示细胞胰岛的组成和规模。 C.胰岛整个人类的截面分析的大小从图1中的分布符合对数正态分布的概率密度分布。 D.数学分析(β-细胞中的细胞成分比绿色,红色和蓝色的三角洲细胞在α-细胞)图1每个胰岛的有效直径斌。 E. A.胰岛随机抽样的免疫组织化学分析的粒度分布(左)。胰岛虚拟切片分析的粒度分布(右)。 B.对数正态分布随机抽样的免疫组织化学分析(红色)和虚拟切片(蓝色)的情节比较。
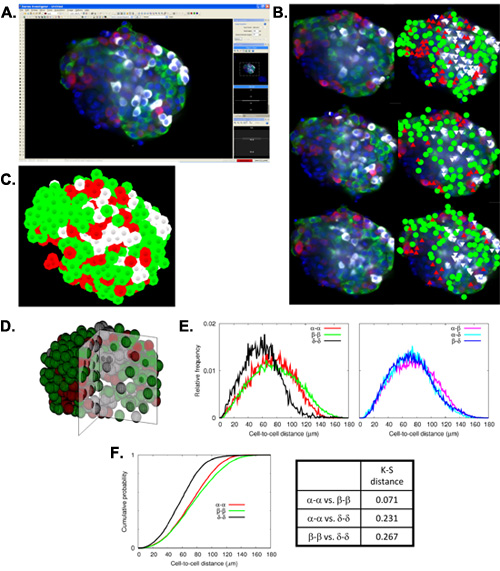
图3。胰岛细胞的组成和结构的映射和数学分析。答:屏幕捕获显示单一焦平面从三维图像上传到立体声研究者人类胰岛的重建堆栈(绿色,α-细胞β-细胞在红色,三角洲细胞在白色和蓝色的细胞核) 。乙:荧光图像(左)和对应数据(右)在三个不同的焦平面的显示间隔10微米。 :三维重建胰岛测绘数据有代表性的观点。 D:胰岛映射获得的坐标为基础的四分之一切胰岛的三维重建。电子邮件:细胞的组成和结构的数学分析。大号EFT。在一个单一的细胞群的细胞之间的细胞与细胞间的距离相对频率。右。相对频率之间的两个不同的细胞群的细胞与细胞间的距离。传真:柯尔莫哥洛夫 - 斯米尔诺夫(KS)的测试。左。累积概率细胞到细胞的距离分布,α- -α,β-β-,和三角洲,三角洲细胞。右。相应的三个累积概率KS距离。
讨论
计算机辅助大型可视化和量化这里介绍负担胰岛研究的四个重点:(1)大规模的胰腺标本的分析提供了一个全面的整体胰岛大小的分布和胰岛结构。 (2)细胞的组成和结构的三维重建和数学分析,进一步促进内的胰岛内分泌细胞的空间安排的考试。 (3)引人注目的胰岛可塑性可能会建议在不同物种间和各种病理生理条件下的同一物种进化收购代谢的变化的适应,而不是物种的差异。 (4)重排...
披露声明
致谢
支持这项研究是由美国公众卫生服务补助DK - 081527,DK - 072473和DK - 20595芝加哥糖尿病研究和培训中心(动物模型核心)的大学,Kovler家庭基金会的礼物。
材料
| Name | Company | Catalog Number | Comments | |
| Fluorescent microscope | Microscope | Olympus Corporation | IX-81 | |
| Stereo Investigator | Program | MBF Bioscience | ||
| MIP-GFP mice | Mice | Jackson Laboratory | ||
| Mathematica | Program | Wolfram | ||
| Image J | Program | National Institutes of Health | ||
| Slidebook | Program | Olympus |
参考文献
- Steiner, D. J., Kim, A., Miller, K., Hara, M. Pancreatic islet plasticity - Interspecies comparison of islet architecture and composition. ISLETS. 2, 135-145 (2010).
- Kim, A., Miller, K., Jo, J., Wojcik, P. l., Kilimnik, G., Hara, M. Islet architecture - a comparative study. ISLETS. 1, 129-136 (2009).
- Kilimnik, G., Kim, A., Jo, J., Miller, K., Hara, M. Quantification of pancreatic islet distribution in situ in mice. Am J Physiol Endocrinol Metab. 297, E1331-E1338 (2009).
- Hara, M., Dizon, R. F., Glick, B. S., Lee, C. S., Kaestner, K. H., Piston, D. W., Bindokas, V. P. Imaging pancreatic beta-cells in the intact pancreas. Am J Physiol Endocrinol Metab. 290, E1041-E1047 (2006).
- Hara, M., Wang, X., Kawamura, T., Bindokas, V. P., Dizon, R. F., Alcoser, S. Y., Magnuson, M. A., Bell, G. I. Transgenic mice with green fluorescent protein-labeled pancreatic beta -cells. Am J Physiol Endocrinol Metab. 284, E177-E183 (2003).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。