Method Article
条形码酵母图书馆竞争力的基因屏幕
* 这些作者具有相同的贡献
摘要
我们已经开发出全面的,无偏见的全基因组的屏幕,了解基因药物和基因与环境的相互作用。筛选这些突变集合的方法。
摘要
凭借在新一代测序技术的进步,我们几乎每天都获得新的基因组序列。这些进步的步伐正在加快,前途更大的深度和广度。鉴于这些非凡的进步,需要快速,并行的方法,以确定基因功能变得更加重要。集合的全基因组的缺失突变体, 酵母菌和大肠杆菌大肠杆菌曾担任基因功能的功能特性的工作母机,但这种方法是不可伸缩的,目前的基因缺失方法要求每个成千上万的基因组成基因组中被删除,并核实的。只有这项工作完成后,我们可以追求高通量的表型。在过去的十年,我们的实验室已提炼组合的竞争力,小型化,高通量的基因组范围内,可并行执行检测。这是可能的并行,因为DNA“标签”,或“条码”,到每个突变列入,条形码突变的代理服务和一个可以衡量条码丰评估突变体健身。在这项研究中,我们设法填补DNA序列和条形码突变集合之间的差距。而要做到这一点,我们引入一个结合转座子中断条码的方法,开辟了并行条码检测新测序,但较差的特点微生物。为了说明这种方法,我们提出一个新的白色念珠菌条形码中断收集和描述如何将两个芯片为基础的下一代测序为基础的平台,可以用来收集万- 100万在一次实验中基因-基因和基因药物的相互作用。
研究方案
1。背景资料
有几种方法产生突变体进行条码标签。目前的黄金标准是由财团实验室创建,并在2002年1完成的酵母基因敲除(YKO)集合。据介绍,由于原YKO已产生其他酵母集合;在不同的应变背景,使用过度表达的构造,并在其他微生物,如大肠杆菌大肠杆菌 2。同时,努力创建条形码的shRNA库进展迅速,事实上,许多这些哺乳动物集合的设计原则已经从酵母通过。为了演示如何条形码转座子可以快速,广泛适用于系统的突变体集合建立战略,我们专注于我们最近在人力,白色念珠菌的真菌病原体创建一个集合。条码屏幕S 中的成功是基于我们对白色念珠菌的工作这里使用的是酵母 ,并作为一个例子生物体。样品的协议,可以用来稍作修改屏幕上的任何可以在悬浮培养生长的生物。因为很少有生物体所需的高的转化率和高效率的有丝分裂重组,需要建立完善的的缺失突变体,因此我们开发了一个协议 ,它使用在体外转座子诱变mutagenize基因组DNA 库, 然后转化为白色念珠菌3这些条形码的基因片段4。灵感来自于成功的基因组中的原YKO收集和haploinsufficiency 9,全基因组的药物靶标和行动机制10,11 5-8基因网络的性质上的根本发现的作用,所有基因的必要性12,我们预计其他微生物扩大这种做法是非常富有成效的。
下面的协议承担所需的突变收集已创建(如YKO 或白色念珠菌扰乱收集),并作为单独归档株。对于应变建设的详细描述,请参阅1,13,14。
2。组合成一个池的单个突变体
- 允许一个星期的时间产生汇集细胞等份(可以无限期地储存于-80 ° C)。
- 完全解冻的利益株冰冻甘油股票,但不要让细胞保持解冻> 2小时。
- 消毒96针工具,浸在水中的脚工具,以消除任何剩余的细胞,其次是2骤降70%乙醇浴(如枪头框盖),火焰脚工具,1分钟冷却。小心火焰远离乙醇浴脚工具。乙醇浴水平应超过在水浴水平,以确保所有结转细胞被火烧和删除。更换水,每4-6 pinnings。
- 无菌的96孔针工具轻轻插入一个解冻的96孔板,漩涡和细胞转移到一个NUNC全方位装有YPD琼脂,包括适当的抗生素。拓展殖民地,直到他们达到的最大尺寸在30℃(2 - 3D)。为了保护板,我们发现它最有用的整合到一个单一〜384株的全方位托盘4个96孔板。
- 在殖民地长大后,请注意任何丢失或增长缓慢的菌株和列宾这些在〜2X其余株的细胞团。
- 工作中的微生物环境(火焰和无菌实验器皿,洪水5-10毫升媒体板,浸泡5分钟和悬浮殖民地与细胞吊具。倒入50毫升锥形离心管中的液体,加上细胞,并添加甘油15%或DMSO至7%(体积/体积)。
- 测量池的OD 600和调整(通过稀释或离心)到终浓度为50外径600 /毫升含15%甘油或7%DMSO的媒体。
- 在40μLPCR地带管和冻结卷分装在-80 ° C
3。实验池增长
此过程中概述图1。
- 在冰上解冻池等分(PCR管)。如果不使用机器人,跳到第5步。
- 立即稀释(gently!)为媒体与药物或选择条件的游泳池,一个总体积700μL,在48孔板接种0.0625 外径 600 。包括至少有一个适当的盘上的溶剂对照。对于延长实验,增长超过5代(即超过1),填写相邻井媒体或选择条件,但没有细胞。
- 用塑料板密封的密封,如果条件需要有氧增长(例如,非发酵碳源),使用21号针头刺破孔,在对每口井的侧膜密封。
- 生长在分光光度计,在30 ° C晃动实验determiNED晃动方案(如摇14分钟,在最高设置(或温控摇床),读井恢复晃动)。部分的细胞悬液,可收获机器人,并保存在用户定义的一代人的时间点,通常为5,10,15和20代增长机器人甲板上的冷板。 (http://med.stanford.edu/sgtc/technology/access.html,详情,请联系C. Nislow或G. Giaever)。
- 人工细胞的生长,在一个起点外径600 0.002接种在250 ml培养瓶中,50毫升的文化。摇匀,在250 rpm的细胞,直至达到最后的2.0 OD 600( 用于酿酒或念珠菌)〜10几代人的成长在30 ° C 。增长带来的额外代可稀释细胞在新鲜瓶中的OD600值在2.0至0.02。
- 收获至少600〜2 OD每个样品/安全锁离心管的时间点细胞单位。
注:务必收集细胞样本(即“T0时间点”)加入池1-2外径600直接从冰箱分装到1.5 ml管和加工,以评估任何新创建的池的初始应变表示下面。
4。基因组DNA提取,PCR和芯片杂交或测序
- 净化从1〜2的Zymo研究YeaStar套件细胞根据制造商的指示外径600(“第一议定书”,如果用酵母DNA),或另一种合适的方法具体的利益的有机体(标准酚/氯仿提取醇 沉作品基因组DNA多样的微生物)。如果使用的YeaStar试剂盒,300μL的0.1X TE洗脱的DNA,而不是在协议中指定的1X TE 60微升。基因组DNA,可以无限期地储存在-80 ° C
- 设置每个样品PCR反应,反应条件如下的一个uptags的downtags:33μLDDH 2 O,6μL10X PCR没有MgCl 2的缓冲,3μL50毫米MgCl 2的,1.2 μL 10毫米dNTPs,1.2μL50微米向上或向下引物组合,0.6μL,5 U /μLTaq DNA聚合酶,〜0.1微克15μL的基因组DNA。总成交量为60μL。 Thermocycle在下列条件下:94 ° C 3分钟,30个循环94℃30秒,55 ° C 30秒,72 °彗星30秒,然后72℃3分钟,并保持在4 ° C.检查凝胶产生的PCR产物;预计60 bp的两个PCR的产品是用于条码测序扩增产物)的杂交和130bp的扩增产物。 PCR产物,然后可以在-80℃无限期储存。
- Prewarm杂交炉温度至42 ° C和设立沸水浴和冰桶含有冰的水泥浆。
- 预湿的阵列,慢慢地与120μL1X杂交缓冲液灌装。
- 在杂交缓冲液中孵育42 ° C和10分钟20转。
- 准备90μL,每个样品的杂交组合,加上一个额外的缓冲区,如下:75μL2X杂交缓冲液,0.5μLB213控制寡核苷酸(0.2 FM /μL),12μL混合寡核苷酸(12.5分/μL),3μL 50X登哈特的解决方案)在0.5毫升锁顶管。
- 加入30μLuptag PCR和30μLdowntag PCR总体积为150μL到120μL的杂交组合。煮沸2分钟,至少2分钟,在冰水中设置。简要旋转管前使用。
- 从数组中删除的预杂交缓冲液,添加90μL杂交/ PCR混合物。为了防止蒸发,盖一个艰难的现场阵列垫圈。杂交16小时42℃,20 RPM。
- 刚准备每个样品600μL生物素标签的混合加一个额外的,如下:180μL20X SSPE的,12μL50X登哈特的,6μL1%吐温20(体积/体积),1μL1 mg / ml的链霉亲和素 - 藻红蛋白,401μL DDH 2澳所有的链霉亲和PE样品存放在黑暗中。到2毫升试管分装600μL。从芯片中删除强韧点。
- 慢慢地将阵列的杂交组合,通过吸液管,填补芯片与120μL洗A.总理Affymetrix公司流体站。
- 洗净的阵列使用一个Affymetrix公司流体站根据制造商的指示,使用以下修改“基因的Flex_Sv3_450”协议:1与洗净的额外步骤一(1周期,2混合)染色前,洗净乙温度42 ° C而不是对40℃,染色42 ° C而不是25 ° C。它也可以手动执行后的杂交洗,生物素染色,和后染色洗(见参考15第396页)。流体操作,运行的流体站“SHUTDOWN_450”协议。
- 洗净后,确保无气泡存在。如果需要添加90μL洗了吸管缓慢,直到气泡消失。如果有任何标志或SMudges阵列上的表面,用异丙醇的清洁玻璃窗和无绒布组织。应用垫片/隔新鲜强韧点,并放置在扫描仪的阵列。
- 在Affymetrix公司的GeneArray扫描仪扫描发射波长为560纳米。
5。阵列分析(参见图2为获得例如使用白色念珠菌中断收集)
- 离群掩蔽:由于Affymetrix公司TAG4阵列包含5个,每个条码的补充重复分散随机偏离的复制,可以屏蔽和丢弃任何条码探头。要做到这一点,为每个阵列功能,似乎是基于其信号相比,其他4个离群复制此功能,我们的软件首先检查的功能,环绕怀疑离群,生产与犯罪嫌疑人25特性矩阵在该中心的功能。如果相差超过10%来自其个人的修整复制平均每个(中间三个重复的平均值,去掉最高和最低的复制)> 13/25在这一地区进行探测,这个探头是从进一步的分析,然后丢弃。因为这样的离群点是最经常杂交后洗涤不一致的结果,我们扩大或“垫”含有怀疑探头的地区。垫等,由定义(包括5探针半径内的所有探测器的探头由(× 1 × 2)2 +(Y 1 - Y 2)2)半<6 × 1,× 2,Y 1 , Y 2 X和Y坐标的两个特点。最后,丢弃功能像素的标准差(包括在Affymetrix公司的阵列。CEL文件)/平均值功能像素的功能。删除离群值后,所有剩余的平均强度值复制。
- 删除标签:不可用,用低强度值标签质量差的结果,必须拆除。排斥截止为这些低强度探头,可以计算如下:
- 治疗控制对任何数组,计算出每个标签的日志2((I C - B克)/(I T - B G)), 其中i C是控制强度 ,I T是治疗强度,和b G是未分配的标记探针的平均强度。
- 配对应变uptag和downtag比率,每个标签对,两个样本中的两个标签的最小强度。依据这个最低强度的比例对。
- 使用50的滑动窗口大小的排名比对,计算uptag和downtag窗口内的比例对的相关性。也算在上一步中计算出的最低强度的平均。
- 25对滑动窗口,并重复前面的步骤,直到所有对已被突破。
- 绘制与所有的Windows uptag downtag相关的平均最低强度。
- 最后,选择强度阈值,我们一般使用强度值相关的第一个达到最高水平的80%。进一步的分析,国旗及删除低于此截止任何标记。
- 饱和度校正:由于每个芯片上的条形码的功能可以趋于饱和,对TAG4阵列的信号不是线性相关的标记浓度。要纠正这种饱和遵循参考16中所述的协议。
- 阵列正常化:各自为阵,分别uptags和downtags正常化。为了位数正常化,排名从每个uptags和强度递增的顺序downtags阵列获得的值。意味着每个uptags和downtags正常化,除以平均。所有阵列的平均正常化是第二步以下改造平均每个数组的原始数据。
- 计算灵敏度控制治疗比较分数:要使用日志2的敏感性指标比率:对于每一株,计算日志2((μC - B 克 )/(μ 吨 - B G)),其中μc是对照样本的平均强度,μ吨是处理样品的平均强度,b,g为平均强度的未分配的探针。以积极的日志2比例的菌株敏感的治疗,和那些有抗药性的负对数 2的比例。
6。条形码的酵母菌株的适用性评估,经测序
- 隔离的DNA微阵列删除池。
- 每次20 - MER uptag条码杂交Illumina的流通池所需的常用条码引物序列和序列组成的复合引物扩增(见Illumina的引物扩增图3和图表)。这些引物可用于无需额外的净化脱盐。 PCR是在100μL,Invitrogen公司白金PCR Supermix(货号11306-016)符合下列条件:95 ° C / 3分钟; 25周期94 ° C/30秒,55 ° C/30秒68 ° C/30秒; 68 ° C/10分钟。
- 净化Qiagen公司MinElute 96用友PCR纯化试剂盒(货号28051)的PCR产物(130bp)。
- PCR纯化,量化与Invitrogen公司QUANT - dsDNA的BR检测试剂盒(目录号Q32853)的DNA。不要依赖于260/280读数!
- 规范化的DNA浓度为10μg/ml和游泳池等体积归DNA。
- 12%的聚丙烯酰胺3-4小时,取决于使用电压TBE凝胶的独立汇集的DNA。
- 用溴化乙锭(SYBR GREEN以及工作)染色凝胶30分钟。
- 在长波紫外线灯箱的兴趣中找到的带(戴适当的面部防护),切出来,使用粉碎和提取的DNA,乙醇沉淀,浸泡法17。
- 确认适当大小的DNA(130bp)已被隔离,引物已被删除,使用安捷伦的生物分析仪的高灵敏度DNA试剂盒(目录号5067-4626)。
- 样品测序:
- Illumina公司GAIIx平台:
- 生成单读流通池群使用CBOT和单读集群生成工具包(猫号GD - 300 - 1001)。对于读1,向上和向下标签修改的测序引物都集中在100um的股票浓度,并添加到一个带管(每个120μLHT1 100um的测序引物0.6μL)。配方SR_Amp_Block_StripTubeHyb_v7.0是用于生成R1集群。
- 测序的基因组分析仪IIx的。以下18个测序周期,配对模块是用于去除合成的第一链和rehybridize的流通池,利用Illumina的R1(下面)。集群再生,并为5个周期序列捕获索引标签。
- Illumina公司GAIIx平台:
- 索引标签序列是用来斌序列分为实验箱。
- 在每个实验斌中,酵母条码序列相吻合,给每一个条码总数的计数。
- 计数位数正常化,因此每次实验有相同的计数分布。类比与条形码的微阵列健身实验,每一株健身缺陷率计算,并表示作为日志 2的比例(控制/处理) 。积极的健身缺损评分,在药物治疗期间的应变丰的下降,表明,野生型菌株中删除的基因版本,是需要抵抗的药物或抑制剂。
注:考虑到酒吧,以次作为替代阵列杂交。随着高通量测序的成本降低,标签丰度的读数,利用高通量测序是可行的,在许多情况下, 更多的成本18日生效。 通过这种方式,扩增PCR产物直 接测量作为“罪状”,而不是信号强度杂交数组 。 这消除了标记的交叉污染,饱和度,或非常高或非常低的信号强度所引起的问题中出现的假阴性和阳性。 此外,可以结合之前多次实验测序另外一个4-8基地DNA指数19。由于酵母的条形码是20基点,一个单一的,2步26-28基地捕捉复用指数和独特的条码读取,允许极端100 +复 。 在撰写本文的时候,酒吧,SEQ提供比条形码芯片的成本优势,此外,酒吧,SEQ本质上是灵活的读取/运行增加的数量,复用的水平,可以增加进一步降低成本。几个“中期能力”,所有主要的平台制造商定序,将进一步民主化酒吧SEQ,测序有可能成为选择的读数。
该协议也得到了验证Illumina公司HiSeq2000。
优秀示范的一个酒吧SEQ使用,以解决在酿酒酵母生长的控制生物的根本问题是在最近的一项研究等格雷沙姆20列出几个重要的实验设计和解释准则 。
7。汇集筛选数据的验证
从任何屏幕功能基因组学的结果,应验证孤立的文化在使用个别菌株。由于每个实验将在敏感菌株的数量方面不同,选择候选株的数量,以确认是有点乱。作为指导,日志2的比例或Z -得分排名最敏感的菌株和测试前的25-50%的考生(通常转化为2-3标准DEviations从池中的所有菌株)的平均值是成本和效益之间的良好平衡。个人确认,可在任何烧瓶中进行,但我们执行这些测试,使用100μL一个惊天分光光度计媒体开始接种600外径为0.06,每15分钟测量(见图 4)在96孔板增长5几代。
8。代表性的成果
一旦一个全基因组的屏幕完成后,阵列已规范化,每一株的行为相比,控制的治疗(例如,通过比较芯片强度或测序计数/应变)的数据是最容易在一个Excel文件中的基因操纵排名由log2比例控制/实验。在这种方式下,负面的log2比例更大,更敏感,特别是应变测试条件。这些Excel文件,可以绘制各种图形软件包。我们发现,最简单的情节Y轴的基因或在X轴的ORF名称的log2比率。在图2a所示的例子中,这样的情节克霉唑治疗(一个著名的抗真菌剂)所示。所有应变是显着敏感,治疗一个2的log2比例,以红色突出显示,和我们通常会在个人成长的每一个突变检测验证,在相同浓度的药物存在许多这样的菌株。在这个例子中,4株突出,NCP1,ERG2和2 ERG11独立等位基因,克霉唑已知的蛋白质目标。这4个基因中的每一个直接参与麦角固醇的生物合成,相当于胆固醇酵母。例如,NCP1编码的NADP -细胞色素P450还原酶,是麦角固醇的生物合成,并参与相关Erg11与协调监管。这个例子突出已知的药物靶标(Erg11)是确定在这种不偏不倚的屏幕,以及其他几个关键部件的目标的途径。最后,几个红色的突出的基因的代表可能在麦角固醇的生物合成或在不同的生物过程有关的基因。如上所述,在汇集的屏幕上的敏感检测到的每一株应验证其个体生长检测的灵敏度。在图2b所示的例子中,四株被证实为敏感克霉唑的基础上减少的增长,相对于野生型菌株,BWP17。这些个体的生长曲线突出等汇集的基因药物屏幕的一个重要特征,这是一个特定菌株的绝对排名,并不一定反映其确切的灵敏度水平。此外,图2b还显示,在这种情况下,不必为每一个基因的多个等位基因的价值 ,两个erg11受阻突变体略有不同的敏感性。这些中断的性质相关的敏感程度,可以提供额外的洞察到毒品的行动机制。
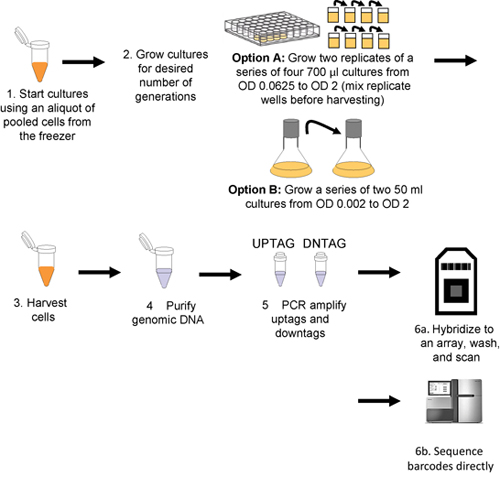
图1。汇集生长测定和条码检测的工作流程。文化接种汇集细胞解冻等份(步骤1),然后成长为几代人的所需数量(步骤2)无论是机器人(方案A)或手动(方案B)。细胞离心收集(步骤3)和基因组DNA中提取的干细胞(步骤4),然后从隔离,uptags和downtags独立扩增(步骤5),和一个数组(或直接测序步骤6b步骤6a)杂交。
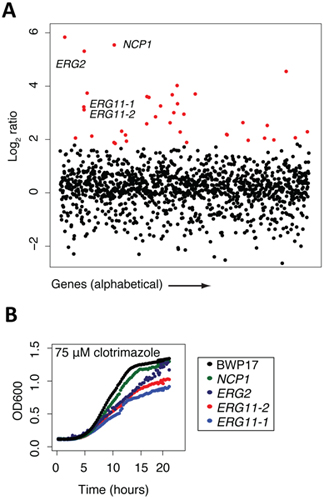
图2:在协议的某些点收集的样本数据。 (一)从筛选结果(13日改编)的样品数据。标签突变体库是增长20克霉唑和DMSO(对照组)的存在。日志2的比例(控制强度/治疗强度)计算和绘制的基因功能。高度敏感株(红色),包括克霉唑,ERG11p已知的目标。请注意,这个实验中经常揭示化合物的实际目标以外的其他敏感突变体。一般来说,这些突变体是那些行为与目标的综合,是那些一般应力/治疗反应的一部分,或未能确定的误报。 (二)确认数据的示例(改编13日) 。从汇集的增长分析的结果,可以验证,越来越多的个体文化的应变和野生型增长(黑色)相比较。
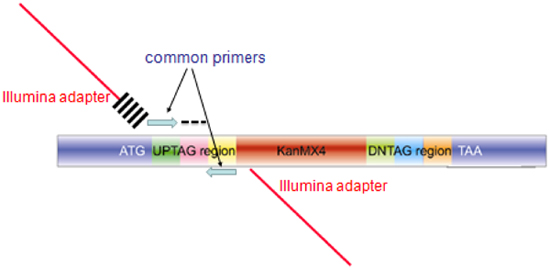
图3。汇集条码检测芯片杂交或条码测序扩增产物的结构。每个突变产生的扩增我n的集合包含集成的基因组(ATG和TAA标记的蓝色区域),独特的条形码(标记为AG和黑色破折号表示)的同源性。芯片杂交,蓝色的通用引物用于扩增一个60bp探针微阵列杂交。条码测序,在PCR反应中使用扩展的引物,包括编码Illumina的适配器(红色条),和6bases交叉的舱口指数)和蓝色的上游引物的共同引物序列,和相同的复合引物(减去6基期指数)为第二个引物。
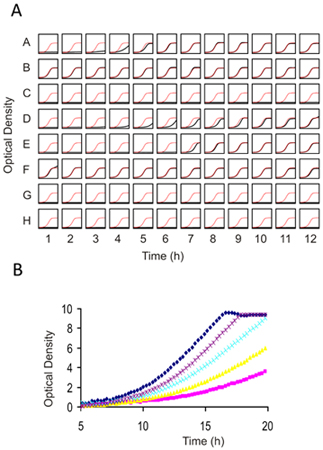
图4。个体生长实验1)预筛选化合物对野生型酵母确定一个适当剂量的全基因组筛查和2)从基因组宽屏幕确认结果。 (一)一个96孔平底板充满0.062 OD 100μL的细胞悬液。每口井可包含一脉相承(剂量测定)或不同的应变和药物组合(确认检测)。 2化合物(通常在DMSO溶解)添加液和细胞生长16-20 h不断摇动30 ° C。 DMSO终浓度不得超过2%。在这个例子中,以及在每个板块的的增长曲线绘制在黑对在红色的控制生长曲线图。 (二)高等教育几个prescreens覆盖另一个例子药物获得高分辨率图像。在此系列滴定,一个IC 10-15获得与紫色的剂量,将删除剖析(髋关节和啤酒花)的适当。由于在较高的光密度非线性,特餐(或任何类似的酶标仪)消耗臭氧层物质的使用与“传统”的1毫米路径长度比色皿获得的,必须进行校准。
讨论
在这里,我们提出了一个协议,适度的修改,可以很容易地适应广泛的现有馆藏的创建标签突变馆藏的条形码不同的微生物突变集合。我们强调,虽然我们已经报道了标签的转座子突变致病性酵母菌 C.一个协议白色念珠菌 ,一个非常类似的协议,可以适应各种各样的单细胞真菌。修改,该协议运作良好13中的细菌,和目前正在建设中一些额外的真菌和细菌的基因组的集合。目前,这个实验提供了唯一的综合性,全基因组基因的小分子的相互作用的公正屏幕。其中特别引人注目的检测功能是没有的基因或小分子的先验知识是必需的。尽管这些检测的范围和权力,其可转让给其他实验室已经有点阻碍,由最初的资本投资和信息工具,分析的结果。通过结合分析的强大工具的下一代序列读数,我们期望通过增加。
披露声明
没有利益冲突的声明。
致谢
我们感谢罗恩戴维斯,亚当Deutschbauer,整个臀部在多伦多大学的讨论和意见的跳实验室。 CN是由国家人类基因组研究所(批准号HG000205),RO1 HG003317,CIHR澳门币84305,加拿大癌症协会(#020380)的赠款支持。联办是由斯坦福大学的基因组培训计划(批准数的T32 HG00044从国家人类基因组研究所)和国立卫生研究院(批准号P01 GH000205)支持。 GG是在NHGRI RO1 HG003317和加拿大癌症协会,格兰特#020380,运输署和唐纳利测序中心的支持,部分是从加拿大创新到DRS基金会的赠款支持。布兰达安德鲁斯和杰克布拉特。 AMS是由多伦多大学打开奖学金支持。
材料
| Name | Company | Catalog Number | Comments |
| 抗生素 | 供应商和产品目录号 | ||
| 羧 | Sigma公司的一部分#C1613 | ||
| 卡那霉素 | Sigma公司的一部分#K1876 | ||
| 大观 | Sigma公司的一部分#S0692 | ||
| 氯霉素 | Sigma公司的一部分#C0378 | ||
| 清理和浓度试剂盒的DNA | |||
| QIAprep自旋小量制备试剂盒 | Qiagen公司,部分#27106 | ||
| HiSpeed质粒马克西套件 | Qiagen公司,部分#12663 | ||
| QIAquick PCR纯化试剂盒 | Qiagen公司,部分#28106 | ||
| QIAquick凝胶提取试剂盒 | Qiagen公司,部分#28704 | ||
| PCR和电泳试剂 | |||
| Taq DNA聚合酶(MG -免费)缓冲区 | New England Biolabs公司的一部分,#M0320L | ||
| 脱氧核苷酸的解决方案组合 | New England Biolabs公司的一部分,#N0447L | ||
| 25毫米氯化镁2 | 西格玛,部分#63036 | ||
| 琼脂糖,装载染料,核酸染色适用于凝胶电泳 | 各种 | ||
| 10X TAE缓冲液 | Sigma公司的一部分#T8280 | ||
| 1 KB PLUS DNA梯状 | Invitrogen公司的一部分#10787026 | ||
| YPD肉汤 | |||
| 10克酵母提取物 | Sigma公司的一部分#Y1625 | ||
| 细菌用胰蛋白胨20克 | BD公司的一部分,#211677 | ||
| 20克的葡萄糖 | Sigma公司的一部分#D9434 | ||
| 医学实验 | |||
| 多通道移液器(1000,200,和20μL) | 各种 | ||
| 一次性移液器水库 | 各种 | ||
| 15日和50 mL离心管 | 各种 | ||
| 96 - 384孔深井板 | 爱思进科学,一部分的P - 2ML - SQ - CS&P - 384240SQCS | ||
| 96孔和384孔PCR板和密封膜 | 各种 | ||
| 版辊密封多孔板 | Sigma公司的一部分#R1275 | ||
| 30 ° C和37 ° C摇晃增长的平板上的细菌和酵母和孵化器在管 | 各种 | ||
| 在体外转座子诱变 | |||
| EZ - Tn5的转座子 | 震中生物技术的一部分,#TNP92110 | ||
| 高通量转型 | |||
| Seqprep 96 HT质粒准备工具包 | 边缘生物系统公司的一部分,#84359 | ||
| 聚乙二醇,分子量3350 | 各种 | ||
| 醋酸锂 | Sigma公司的一部分#517992 | ||
| 6孔板,无菌 | 康宁,部分#3335 | ||
| 50毫克/毫升尿苷 | Sigma公司的一部分#U3750 | ||
| 100X的Tris - EDTA缓冲液 | Sigma公司的一部分#T9285 | ||
| 1X TE/0.1M LiOAc | 各种 | ||
| 鲑鱼睾丸DNA | Sigma公司的一部分#1626 | ||
| 增长的条形码集合 | |||
| 48孔板;如果增长板中的文化 | 格雷纳,部分#M9437 | ||
| 胶粘板密封 | ABgene,部分#AB - 0580 | ||
| 200毫升的培养瓶中 | 各种 | ||
| 能够分光光度计吸光度 | 各种 | ||
| 控温摇床,250毫升烧瓶或晃动分光光度计 | 各种 | ||
| 安全锁的离心管中,2毫升 | Eppendorf公司的一部分,0030 120.094 | ||
| 杂交设备 | |||
| 杂交炉640 | Affymetrix公司的一部分#800138 | ||
| 基因芯片流体站450 | Affymetrix公司,部分#00-0079 | ||
| GeneArray扫描仪3000 | Affymetrix公司,部分#00-0212 | ||
| 沸水浴浮动机架 | 各种 | ||
| 杂交耗材 | |||
| Genflex标签16K阵列V2 | Affymetrix,一部分#511331 | ||
| 登哈特的解决方案,集中50X | Sigma公司的一部分#D2532 | ||
| 链霉亲和,R -藻红蛋白共轭(萨佩) | Invitrogen公司的一部分#S866 | ||
| 安全锁的离心管,0.5毫升 | Eppendorf公司的一部分,0030 123.301 | ||
| 蝇头强硬点 | 多样化的生物技术,部分#LTTM - 1000 | ||
| 0.5 M EDTA | BioRad公司,部分#161-0729 | ||
| 10%吐温 | Sigma公司的一部分#T2700 | ||
| MES的自由酸水合物 | Sigma公司的一部分#M5287 | ||
| MES钠盐 | Sigma公司的一部分#M5057 | ||
| 5 M氯化钠 | 西格玛,部分#71386 | ||
| 20X SSPE | Sigma公司的一部分#S2015 | ||
| 分子生物学级水 | Sigma公司的一部分#W4502 | ||
| 杂交引物 | 各供应商(标准除盐) | ||
| Uptag | 5'GATGTCCACGAGGTCTCT 3' | ||
| Buptagkanmx4 | 5'生物素GTCGACCTGCAGCGTACG 3 | ||
| Dntag | 5'CGGTGTCGGTCTCGTAG 3' | ||
| Bdntagkanmx4 | 5'生物素GAAAACGAGCTCGAATTCATCG 3 | ||
| B213 | 5'生物素CTGAACGGTAGCATCTTGAC 3 | ||
| Uptagkanmx | 5'GTCGACCTGCAGCGTACG 3“ | ||
| Dntagkanmx | 5'GAAAACGAGCTCGAATTCATCG 3“ | ||
| Uptagcomp | 5'AGAGACCTCGTGGACATC 3“ | ||
| Dntagcomp | 5'CTACGAGACCGACACCG 3“ | ||
| Upkancomp | 5'CGTACGCTGCAGGTCGAC 3“ | ||
| Dnkancomp | 5'CGATGAATTCGAGCTCGTTTTC 3“ | ||
| 测序引物:Illumina公司平台 | 各供应商 | ||
| UpTag远期(100um的) | GGC GAC的阿拉木图5“民航局GCA GAA CGA GCT CTT CCG ATC T GAT GTC CAC GAG GTC的大老山隧道3” | ||
| UpTag反向(100um的) | GCG ACG的行政协调会5“学能测验的GAT ACC GAC中的ACT铁通台泥税务师CAC GAC GCT CTT CCG ATC T NNNNN GTC GAC CTG CAG总工会ACG的3' | ||
| DownTag远期(100um的) | GGC GAC的阿拉木图 5“ 民航局GCA GAA CGA GCT CTT CCG ATC T GAA AAC聚糖反恐委员会GAA TTC ATC地下3” | ||
| DownTag反向(100um的) | GCG ACG的行政协调会 5“ 学能测验的GAT ACC GAC中的ACT铁通台泥税务师CAC GAC GCT CTT CCG ATC T NNNNN CGG公司的TGT CGG大老山隧道总工会公司3` | ||
| 阅读1标签SEQ引物(100um的) | 5'GTC GAC CTG CAG总工会ACG的3' | ||
| 阅读1下标签SEQ引物(100um的) | CGG公司的TGT CGG大老山隧道CGT公司5'3' | ||
| 阅读指数测序引物(标准Illumina的R1底漆)(100um的) | 5“交流使铁通台泥税务师CAC GAC GCT CTT CCG ATC T 3” | ||
| 更多的测序试剂/设备 | |||
| Qiagen公司MinElute 96 UF PCR纯化试剂盒 | Qiagen公司,部分#29051 | ||
| 真空泵 | 任何供应商 | ||
| 马歇雷-纳格尔真空歧管 | 马歇雷纳格尔,部分#740 681 | ||
| Invitrogen公司QUANT它双链BR检测试剂盒 | Invitrogen公司的一部分#Q32853 | ||
| Invitrogen公司的量子位的检测管 | Invitrogen公司的一部分#Q32856 | ||
| 丙烯酰胺40%加1%N,N' -亚甲基双丙烯酰胺,37.5:1 | 生物RAD,部分#161-0148 | ||
| 三基地 | Sigma公司的一部分#T1503 - 1KG | ||
| 硼酸 | Sigma公司的一部分#B6768 - 500G | ||
| 0.5M EDTA,pH值8.0 | Teknova,部分#E0306 | ||
| 过硫酸铵 | Sigma公司的一部分#A3678 - 25G | ||
| TEMED | Bioshop,部分#TEM001.25 | ||
| 溴化乙锭溶液 | Bioshop,部分#ETB444.10 | ||
| 0.5M铵酯 | Teknova,部分#A2000 | ||
| 10MM镁醋酸水合物 | Sigma公司的一部分#M0631 - 100G | ||
| 为1mM EDTA,pH值8.0 | 0.5M EDTA,pH值8.0 | ||
| 乙醇 | 各种 | ||
| 醋酸钠,pH值5.2 | Teknova,部分#S0297 | ||
| 高速真空 | 各种 | ||
| 单读集群生成工具包 | Illumina公司的一部分,GD - 300 - 1001 | ||
| 36C测序试剂盒V4 | Illumina公司,部分FC - 104 - 4002 | ||
10X TBE缓冲液配方
| 应收 | 试剂 |
| 108克 | 三基地 |
| 55克 | 硼酸 |
| 40ML | 0.5M EDTA(pH值8.0) |
| DH 2 O至1L标志 | |
12%的聚丙烯酰胺凝胶配方
| 卷 | 试剂 |
| 5.8毫升 | 丙烯酰胺40%加1%N,N' -亚甲基双丙烯酰胺,37.5:1 |
| 12毫升 | DH 2 O |
| 2毫升 | 10X TBE |
| 140μL | 10%过硫酸铵 |
| 总成交量:20毫升 |
Uptag引物组合:
悬浮Uptag Buptagkanmx4在DDH 2 O的每100微米,然后以1:1的比例混合,终浓度为50微米。储存在-20 ° C。
Downtag引物组合:
悬浮Dntag Bdntagkanmx4 DDH 2 O在100微米,然后以1:1的比例混合,终浓度为50微米。储存在-20 ° C。
混合寡核苷酸:
重悬在DDH 2 O在100微米以下八个寡核苷酸(标准脱盐):
Uptag,Dntag,Uptagkanmx,Dntagkanmx,Uptagcomp,Dntagcomp,Upkancomp,Dnkancomp。
混合同等体积的八个寡核苷酸终浓度为12.5μM。
12X MES的股票:
10毫升,0.7克的MES游离酸水合物和1.93克MES钠盐溶于8毫升分子生物学级水。搅拌好后,检查pH值和调整,如果需要的话pH值6.5-6.7。加水到10毫升的总量。过滤消毒,并保存在4 ° C的光(例如,包装箔管)保护。替换,如果溶液变成明显的黄色或6个月后。
2X杂交缓冲液:
对于50毫升,混合12X MES的股票(从14年9月2日)8.3毫升,17.7毫升5 M氯化钠,4.0毫升的0.5 M EDTA,0.1毫升10%的吐温20(体积/体积),19.9毫升,过滤DDH 2 O。过滤消毒。
洗了:混合300毫升20X SSPE,1 mL 10%的吐温(体积/体积),699毫升DDH 2 O。过滤消毒。
洗净乙:混合150毫升20X SSPE,1 mL 10%的吐温(体积/体积),849毫升DDH 2 O过滤消毒。
条码测序引物
-标记引物序列的5'尾巴( 粗体 )Illumina的特定适配器纳入F和R引物序列。变量序列( 斜体字 )5 MER索引标签,用于在复用/索引读出。 3'尾巴(下划线)代表的共同引物侧翼uptag条码,需要放大酵母条码。
下调标记的引物序列5'尾巴是相同的5'UP标记的引物(Illumina的具体序列)的尾巴,然而,3'尾巴(下划线)所使用的共同引物扩增取代向下标签条形码。
参考文献
- Giaever, G. Functional profiling of the Saccharomyces cerevisiae genome. Nature. 418, 387-391 (2002).
- Baba, T. Construction of Escherichia coli K-12 in-frame, single-gene knockout mutants: the Keio collection. Molecular systems biology. 2, 2006.0008-2006.0008 (2006).
- Claus, H., Frosch, M., Vogel, U. Identification of a hotspot for transformation of Neisseria meningitidis by shuttle mutagenesis using signature-tagged transposons. Mol Gen Genet. 259, 363-371 (1998).
- Hava, D. L., Camilli, A. Large-scale identification of serotype 4 Streptococcus pneumoniae virulence factors. Molecular microbiology. 45, 1389-1406 (2002).
- Costanzo, M. The Genetic Landscape of a Cell. Science. 327, 425-431 (2010).
- Tong, A. H. Systematic genetic analysis with ordered arrays of yeast deletion mutants. Science. 294, 2364-2368 (2001).
- Pan, X. A robust toolkit for functional profiling of the yeast genome. Molecular cell. 16, 487-496 (2004).
- Schuldiner, M. Exploration of the Function and Organization of the Yeast Early Secretory Pathway through an Epistatic Miniarray Profile. Cell. 123, 507-519 (2005).
- Deutschbauer, A. M. Mechanisms of haploinsufficiency revealed by genome-wide profiling in yeast. Genetics. 169, 1915-1925 (2005).
- Giaever, G. Chemogenomic profiling: identifying the functional interactions of small molecules in yeast. Proceedings of the National Academy of Sciences of the United States of America. 101, 793-798 (2004).
- Lum, P. Y. Discovering modes of action for therapeutic compounds using a genome-wide screen of yeast heterozygotes. Cell. 116, 121-137 (2004).
- Hillenmeyer, M. E. The chemical genomic portrait of yeast: uncovering a phenotype for all genes. Science. 320, 362-365 (2008).
- Oh, J. A universal TagModule collection for parallel genetic analysis of microorganisms. Nucleic acids research. 38, e146-e146 (2010).
- Oh, J. Gene annotation and drug target discovery in Candida albicans with a tagged transposon mutant collection. PLoS pathogens. 6, (2010).
- Nislow, C., Giaever, G., Stark, I., Stansfields, M. J. R. Chapter 387. Yeast Gene Analysis. , 387-414 (2007).
- Pierce, S. E., Davis, R. W., Nislow, C., Giaever, G. Genome-wide analysis of barcoded Saccharomyces cerevisiae gene-deletion mutants in pooled cultures. Nature protocols. 2, 2958-2974 (2007).
- Sambrook, J., Russell, D. W. . Molecular cloning : a laboratory manual. , (2001).
- Smith, A. M. Quantitative phenotyping via deep barcode sequencing. Genome Res. , (2009).
- Hamady, M., Walker, J. J., Harris, J. K., Gold, N. J., Knight, R. Error-correcting barcoded primers for pyrosequencing hundreds of samples in multiplex. Nature. 5, 235-237 (2008).
- Gresham, D. System-Level Analysis of Genes and Functions Affecting Survival During Nutrient Starvation in Saccharomyces cerevisiae. Genetics. 187, 299-317 (2011).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。