Method Article
工作流的高含量,荧光标记的万能工具显微镜数据,支持开源软件的单个细胞定量
摘要
呈现是一个灵活的信息学的工作流程使得荧光标记的细胞的复用的基于图像的分析。工作流量化细胞核和细胞质的标记,并计算这些室之间的标记易位。提供了用于在96孔格式使用siRNA和可靠的方法为标记物的检测通过间接免疫荧光细胞的扰动过程。
摘要
在了解治理细胞贴壁哺乳动物组织培养模式的行为控制机制的进步越来越依赖于单细胞分析模式。方法,它们提供的复合数据反映从细胞群体的生物标记的平均值可能失去了反映所研究的生物系统的异质性亚群动力学。符合此,传统的方法正在取代,或与开发,以允许评估由高含量显微镜细胞测定的更复杂的形式的支持。这些测定法可能产生大量的荧光标记物,其由所附的专有软件包启用的图像,使得每个细胞的多参数测量。然而,相对高的资金成本,许多这些装置的过分专业化阻止包括方便许多研究者。
这里描述的是一种普遍适用的工作流程,从适用于图像使用大多数荧光显微镜的单个细胞的特定亚地区的多种荧光标记强度的量化。关键这个工作流程是免费提供的细胞Profiler软件1实施分辨单个细胞中这些图像,他们将段定义亚地区,提供荧光标记的强度值具体到这些地区。单个细胞的强度值,从图像数据中提取的该工作流程的主要目的,将说明与控制数据从一个屏幕的siRNA用于粘附人体细胞的G1检查点调节器的分析。然而,这里提出的工作流程可以适用于从细胞扰动的其它装置的数据的分析( 例如 ,化合物屏幕)和其它形式的基于荧光细胞标记,因此应为广泛的实验室是有用的。
引言
这里提出的工作描述了使用可免费获得的软件单元探查的执行的贴壁细胞,以确定个别的细胞和亚细胞限定区域的荧光显微镜图像算法引导击穿。这种方法被称为图像分割,使得成像单元的后续多参数分析通过定量定位于每个小区或亚细胞区(称为分割的对象)的荧光标记标记。此工作流构成用于使高含量的分析的基础,并意在作为该可进一步开发和修改,以适应多参数,在实验室中单个细胞的分析,而不访问专门的高含量的仪器或专有软件的工具。本稿件提供的文件包括一考定相关原始图像数据,算法设置和支持脚本生成描述的分析。所提供的算法设置˚F或细胞探查是为示例数据集和讨论部分细节的调整是什么可能是必要的,以便能够使用来自其他研究的图像数据进行了优化。
一旦量化数据已被使用Cell探查萃取,不同的实验室可以具有关于如何使用通过在原始数据的各个单元的值提供的信息不同的要求;这里示出的是由栅极被施加到原始数据的每个测定的一种方法。使用这些门中,数据被变换成响应二进制而言,允许联用的细胞发生反应由门限定的亚群不同处理的趋势可视化。栅极是基于为每个相关的测量获得适当的阴性和阳性对照的数据分布的观测置位。使用门的是如何管理原材料,基于细胞的测定的一个例子。这里还显示了使用核DNA强度我asurements在其原始形式中组合与门控的数据值的连续范围。其他的方法来管理的图像分析的数据,应考虑,这取决于研究的性质;统计替代使用门分配细胞亚群的报道策略2,系统的比较,总结了高含量的数据在大量的参数进行了报道3。
图像数据的高含量分析已经发现在细胞的药物-反应研究使用,反向遗传学和环境应激信号4 - 6。高内涵分析的优点源于以下事实荧光显微镜数据的算法分析可定量和空间参数可以同时跨越单个细胞7考虑。以这种方式,蜂窝结果对多个测定可以是测定-D交叉引用,差动行为efined细胞亚群可以在实验条件和测定法进行跟踪可以包括考虑形态变量。这里的策略和分析的工作流程所讨论的,作为其它的高含量的方法,是能够提供被交叉引用到单个细胞复用数据。产生的荧光显微镜图像和适用于数据从几十在低通量常规的基于荧光的显微镜产生的图像中的分析通过对成千上万的图像的高含量的方法西服研究使用自动化的高含量筛选平台制造。
工作流在这里示出与从其中分离测定法中任一核荧光标记物强度或荧光报告蛋白的核/细胞质易位,分别计测的例子的数据。工作流是灵活的,因为这些测定可单独考虑或联合取决于EACħ由不同的研究者给出的研究问题。该示例的数据产生作为RNA干扰(RNAi)实验( 图1)的一部分。小干扰RNA寡核苷酸(RNA干扰)被用来拦截特定蛋白质在HCT116人结肠直肠癌细胞,其导致对细胞周期蛋白依赖性激酶(CDK)的活性的两种荧光记者的变化。核视网膜细胞瘤蛋白在丝氨酸780(P-S780 RB1)的CDK6依赖性磷酸化是通过抗体染色评估。在相同的细胞中,CDK2活性(GFP-CDK2记者)的绿色荧光蛋白标记的报告是由它的核评定,其中在不存在CDK2活性的记者驻留在细胞核中并在CDK2激活梭进入细胞质的细胞质比率8。此外,每个小区的核DNA用DNA-嵌入染料染色,双苯甲亚胺,作为识别细胞和细胞核中定义的边界中的图像的装置,以及一个SA测量脱氧核糖核酸丰提供对细胞的细胞周期的位置信息( 图2)的。
CDK6和CDK2的活性是可检测从G1到细胞周期5的S期细胞中转和成功彼此9,10,正因为如此,在个体细胞中的两位记者之间的密切一致性的预期。此处所用的示范集分析,例如,siRNA的有效目标CDK6,视网膜母细胞瘤蛋白(RB1)和非靶向阴性对照( 表1)。 CDK6击倒应引出既减少在P-S780 RB1表位和细胞在细胞周期的G1期的累积。将RB1击倒用作试剂控制的磷酸S780抗体的特异性。从福尔马林荧光显微镜图像固定11,荧光染色的HCT116组织培养细胞可用于算法的图像分析。将得到的数字数据,然后用于交叉引用的记者和衡量不同击倒状态的影响。
通过这种类型的分析产生的数据的电位的大小可以是一个挑战正常分析工具。例如,所述个体单元中的数据可以大于一些电子表格软件将容纳。包括的是数据而进行简单,高重复性,监督处理,以帮助大型数据集分析Perl脚本。 Perl脚本被用于通过细胞探查,处理与特定的文件命名约定( 图3)的图像文件时生成的输出文件专门编写的,并允许每孔字段可变数量的分析中使用。它是将栅极个别细胞测定数据经常重要追踪细胞亚群5的趋势和这里示出的是使用的Perl脚本来标记根据一组预定的门对每个检测类型的每个单元。此外,还包括可选的Perl脚本这总结了各个孔(或条件)的数据的结果,提供:该组门中的细胞和原测定分数的平均值的百分比。查看数据的后者,更均匀的方式,是有效的,其中的反应影响所有或在井的大部分细胞。如上所讨论的,这种评估是比由单独的单元数据选通,其中响应局限于细胞群体内的一个子集,得到不太有用。
所描述的工作流的效用不被siRNA或描述的标志物分析限于扰动。研究已经用这种方法来测定使用的siRNA的组合,化学抑制剂和放射治疗组织培养实验和标记比其他CDK6和CDK2的活动5的评估反应。
在概念上,试验策略允许多种生物学有用的亚细胞区域中,以被自动注册在目前的荧光显微镜图像的单个细胞。因此,这种方法可以产生可通过技术,重点群体,而不是个别的细胞被错过的定量,多路复用的数据揭示的生物信息。稍作修改,所描述的方法和分析工作流程可以产生数量,单个单元格的数据对任何基于荧光检测输出和细胞生物反应,其中DNA含量的定量评估,核或细胞质荧光定量或这些标志物之间的穿梭两个隔室单独地或以多路复用方式是感兴趣。由于发布要求越来越朝着提交公开访问的原始数据,访问和熟悉的免费工具显微镜图像分析往往如这里所描述也将是直接有关的实验室寻求重新分析公布的数据。
研究方案
1.实验扰动和细胞标记为响应标记(反向转染的siRNA屏幕)
- 在无菌组织培养罩吸管70微升200nM的siRNA对1X siRNA的缓冲器在无菌,纯96孔板的孔中。稀释转染脂质成40体积的无血清DMEM培养基中,并分配105微升到每个孔中含有的siRNA。
注:稀释262.5微升脂质成10.5毫升无血清DMEM中产生一个主混合物适于整个96孔板的siRNA,提供2.6微升脂质的每孔。使用200nM的siRNA的起始浓度在该步骤将提供20纳米的工作浓度在步骤1.3中,但程序将工作工作浓度向下至5nm,与起始浓度相应的调整( 即 50纳米)。低工作浓度可能会降低的脱靶假阳性分数,尽管它们可以减轻目标响应的幅度,从而导致增加-圆盾的吨假阴性率。 - 混合板通过温和振动,在室温下十分钟。子划分所得175微升成三个50微升每个目标复制到治疗,96孔板用透明基底的不透明,组织培养。
- 反向转染通过在直接含有10%血清到50微升脂质的siRNA复合物150微升DMEM中分配,每孔8000细胞。使用HCT116人类结肠细胞稳定表达GFP标记标记报告CDK2活性5,8。没有进一步的混合是必要的。用无菌,粘合剂透气膜密封该板,以控制湿度和防止板"边缘效应",并将板放入培养箱,在37℃,5%CO 2的48小时。
- 吸出介质,使得媒体小的残留量保持在孔中。通过加入100微升4%缓冲的甲醛至每个孔中固定细胞,并培育在通风橱中在室温吨10分钟emperature。
- 由抽吸板拆下定影溶液。在这一点上要么停止实验通过洗涤该板3次用100μl磷酸盐缓冲盐水(PBS),然后储存密封,下在黑暗中加入100μl的PBS中,在4℃下进行长达一周,或继续进行透的细胞。
注意:我们尽快推荐处理板固定后,与一般喜欢存储完全处理的板。可以加入杀生物防腐剂如硫柳汞,叠氮化钠,或商业替代品,以防止micoroganismal增长。此外磷酸酶抑制剂的有利于维护磷酸表位,以及其它装置以保持蛋白质修饰状态可以是在相关检测上下文有用 - 从板除去PBS中,并通过加入100微升的透液透化细胞。孵育10分钟,在室温下不摇动。吸用多声道的通透的解决方案annel吸管。重复此步骤三次。
- 通过加入每孔100μl块溶液中30分钟,在室温下阻断细胞。由抽吸板取下块溶液,然后探测用50μl抗P-S780 RB1抗体的稀释500倍的2小时,在黑暗中在室温下将块溶液。
- 三次用100μl板洗涤溶液洗涤平板,离开上盘的溶液,每次5分钟。过夜,在黑暗中探测板在4℃下用50μl荧光标记的二级抗体稀释1000倍的块溶液补充有2微米的染色质特异性DNA染料双苯甲亚胺的。在4℃在黑暗中三次洗涤板作为前和储存密封,在100微升PBS中。图像中的两个星期之内的板。
2.成像和图像分割
- 使用共焦或转盘式荧光显微镜20倍的目标采取单独的16位的,在三个通道灰度TIFF图像对应的DNA染料,GFP和免疫荧光染色。捕获许多非重叠图像集,这里称为帧,将图像的大约1,000-2,000细胞每孔。
- 系统地命名图像文件,以便每个文件的名称是"实验名','井地址","帧号"和"信道标识符",以该顺序( 图3)的独特组合。示例数据集使用"蓝"(染色质DNA染色)或"绿色"(GFP)或"红"(免疫染色的荧光团),为信道标识符。阱地址,帧号和信道标识符被进一步上称为图像元数据。使用下划线符号以避免混淆以及与框架的元数据。
- 名称与指定的顺序,这些元数据元素的文件。这是必要的,以确保在随后的软件的步骤CORREctly组套图像进行分析。
- 下载并安装免费的细胞探查,活动的Perl社区版,R统计编程环境和RStudio。安装期间接受所有默认选项; PC用户安装Active Perl中应使有关路径,文件扩展名关联和脚本映射在提示的所有选项。活动Perl是可选的Mac用户,但他们会以其他方式需要在3.2步从终端命令行,而不是使用图标点击运行Perl脚本。
- 打开电池Profiler软件,点击"文件","导入管道",然后"来自文件",然后选择文件3_channels_pipeline.cppipe( 图S1A和S1B)。该文件中包含必要的软件来从所描述的文件名称约定解释图像文件的元数据的指令。细胞探查现在涉及的图像,从提取日核DNA和抗体强度ESE并使用GFP通道来计算的核与细胞质强度的比率为每个细胞检测到的( 图4和5)。
- 单击单元格Profiler窗口的左下角的按钮'查看输出设置"。在新屏幕的顶部是文本框标有"默认输入文件夹"和"默认输出文件夹"。一次一个,点击文件夹图标,以这些框的右边,并选择用于分析的图像文件的位置和目的地对所提取的数据,分别为( 图S1C)。
- 按"分析图像"按钮,在细胞探查的左下角开始进行图像分析。在屏幕的底部观察剩余时间的数据提取,则"停止分析"和"暂停"按钮。如果需要的话通过在任何时间,这是非常有用的选择的"暂停"按钮暂停分析当观看图像被分析(步骤2.8中所述)。
- 任选地,通过点击上的程序窗口( 图S1D)的极左面板眼图标打开窗户的任何的图像分析步骤。观察"IdentifyPrimaryObjects"窗口,那些"中等"和"第三Objects的检查,在细胞探查当前设置进行图像分割,适合(参见图1和讨论的意见修改这些设置)。
- 点击" 确定 "的,当分析完成后出现的消息框。转到位置"默认输出文件夹",所有的结果数据文件保存为逗号分隔值(.csv)文件( 图S2A)。
3.数据提取
- 找到新的文件'Nuclei.csv',它包含其中的从细胞探查输出。该文件包含单个单元格数据的荧光核抗体强度,核DNA的强度和GFP-CDK2记者比值( 图6A和S2A)。
注意:不同的实验室将要处理这种类型的数据,以满足自己的测定法的性质。建议用于当前数据是从每个处理条件,根据该抗体的数据,并使用所提供的Perl脚本'2_gate_classifier.pl'将GFP-CDK2记者值的单元格的门控。 - 所提供的Perl脚本文件'2_gate_classifier.pl"复制到同一文件夹中的"Nuclei.csv"数据文件( 图S2A)。双击该图标,Perl脚本,并出现提示时,键入跟着数据文件的全名由'的.csv"文件名名称中的细胞被门终于为抗体的门值的文件荧光和GFP-CDK2记者的数据。
注意:如何确定主要设置闸门和应用这些数据的分析,下面的代表数据部分和图6讨论(分析提供的数据使用"0.004"和"1.5",分别)。 Mac用户应当运行从输入终端的命令行Perl脚本:"perl的2_gate_classifier.pl"。 - 观察从与亚群的标签的原始细胞探查数据显示如何从每个孔各小区执行对两个栅极( 图6C)组合原始个体细胞测定值新创建的文件。
- 绘制数据使用单独的细胞亚群的标签通过打开RStudio软件各实验条件。点击"文件"和"打开文件",然后选择所提供的"analysis.r"文件。观察命令绘制图6B, 7和8中RStudio( 图S2B)的左上窗口。在左上窗口中,在线路5和6的双引号符号之间,键入包含选通数据的文件夹的计算机地址。包括驱动器号和文件本身,分别的名称( 如 "C:/分析文件夹/输出分析"和"nuclei_gated.csv")。
注意:如果RStudio用于第一次的给定计算机上,R图形包'ggplot2'将需要首先安装。这是一种新的安装RStudio中,之后,该步骤变得多余一次仅一步。要安装"ggplot2",单击叫RStudio的右下角的窗口上方"包"选项卡上,单击出现在这下面的"安装包"按钮。会出现一个新的窗口。键入"ggplot2"(省略报价)进入"包的步伐在这个新的窗口,最后单击"安装"按钮关闭该窗口,安装必要的ggplot2功能,并返回到主RStudio窗口从步骤3.6继续。 - 在RStudio左上方窗口高亮线1到17,然后点击"运行"按钮。这将进入实验数据,阈值和井位的细节到R( 图S2C)。 ř现在暂时持有的相关数据绘制。
- 突出显示下方17行的其余代码的各个块,并像以前一样单击"运行"按钮,创建相应的地块。观察图中RStudio的右下角的窗口并保存格式数通过点击"导出"按钮( 图S2D)。
- 同时关闭RStudio,单击"不保存"提示时。这可以防止在下次使用RStudio的,这将否则保存数据混乱从以前的会话。
结果
所产生的示例图像组使用反向转染的siRNA筛选方案已准备和使用Cell Profiler软件进行分析。所得数值的原始数据是这样的,每一个细胞都单独表示的,可追溯回其图像和良好的原点,测定几个荧光强度参数( 图6A)。对于确定的平均核荧光强度为P S780 RB1抗体和整合的DNA的强度为DNA染料定义核掩模每个单元被确定。 GFP平均强度值的每个细胞的细胞核和细胞质的区域也被记录下来,允许核的计算相对于GFP-CDK2记者细胞质荧光。下游的这些算法荧光强度测量使用了这些单个单元数据的以限定栅极两个测定中,核抗体染色和GFP-CDK2记者。细胞在th随后注释的测定结果,并使用这些标签,以使特定亚群ë依据待进一步的特征在于描述了第三个测量值(核DNA含量)。
直方图收集对每个试验中的原始荧光强度数据是评估细胞亚群的行为不同的条件下的一种有效方式。在图6B的直方图示出了从一式三份孔的每个RNAi敲低条件个别单元数据的人口分布。向左是用于核抗体强度和右边的数据被用于GFP-CDK2记者相应的数据。为P-S780 RB1抗体数据表明,细胞广泛存在于两个种群对本翻译后修饰和细胞群与RB1磷酸对S780的损失,可以区分作为细胞核强度的一个左手峰被富集当CDK6是的siRNA撞倒。同样的左手峰值当RB1本身是RNAi靶,反映了完全除去蛋白质,从而P-S780 RB1染色可见。与此相反,在相同的实验条件下对相同的细胞中,当通过将GFP-CDK2报告基因分析观察到的,显示出不同的动态中的各个单元中的数据。连续分布观察,只用单一的峰,但siRNA的扰乱细胞周期(siCDK6),使在该分布的右手肩部的延伸积累在G1期的结果( 即指示增强细胞表示存在增加在核/细胞质的GFP比,绘制在X轴)。
也示于图6B的直方图栅值(竖线)所选择的这两组实验数据的分布的基础上。用于P-S780 RB1抗体数据的规则是确定门位置为半高:在主的左肩最大宽度位置(右)峰值考虑阴性对照细胞的数据时(非靶向siRNA)。数据突出显示红色的细胞减少和缺乏P-S780 RB1,这是认同这个门。类似的门定位在所述比率值分布的相对肩部用于GFP-CDK2记者。将所得的高比率亚群的细胞,缺乏或功能降低CDK2活性,示于绿色。为了说明这两个实验的多路分析图6C显示了使用2_gate_classifier.pl perl脚本的原始数据( 图6A)转换成下面的批注文件既门价值的实现。这个新文件包括原始数据一起类标签的每个小区的新的列和用于区分它们两个闸值(0.004对抗体的数据和1.5的GFP-CDK2记者此时门分别使用) 。
经分类的各个细胞˚FROM中的两种测定现在有可能使用这些类的标签,以协助重复测定数据的注释的基础上,各敲低的条件。 图7示出散点图各个单元数据为P S780 RB1和GFP-的CDK2测定从例如数据对所有三个RNAi的条件设定。号码标注的散点图象限显示每个门控亚群的相对百分比,以整个为该击倒上下文和使用上述类标签R中产生。这些图表明,相对于转染的细胞与非靶向siRNA( 图7B),转染的细胞siCDK6揭示的净数据配给移都向下的Y轴(表示无RB1的磷酸化在丝氨酸780),并在正确的在X轴(表示低CDK2活性, 图7C)。这两个变化,预计这一目标击倒。与此相反,在从SIR数据B1转染的细胞( 图7A)显示抗体染色的符合表位的损失的损失,但影响不大在用于CDK2的记者的数据分布相比转染非靶向siRNA,暗示对GFP的没有很大的影响控制-CDK2记者源于RB1击倒。
为了进一步探索利用单个单元格数据,亚群分类和分析复用图8所示的散点图从综合DNA强度图7C一起配对直方图型材siCDK6数据。直方图的对涉及到的整个人口的相对的半部,分为两种抗体强度(散点图右)或GFP-CDK2记者比值(散点图以上)的基础上。核DNA强度为这些人群的定量展示了两个高峰的特点2N和4N DNA含量的左,右峰值,分别为。在我在图中所示的闸门的ntentions 6,7和8是这样的细胞识别为低用于P-S780 RB1(标记:P-S780-)或与来自GFP-CDK2记者高比率值(标记为:G1)会是在细胞周期的G1期。事实上,确定与任一这些测定的DNA图谱直方图亚群主要含有细胞,用2N DNA含量。在相反门人口DNA图谱(标记:P-S780 +或非G1)含有细胞的分布范围从2N至4N,符合这些细胞通过一系列的细胞周期的位置后G1期。
尽管这里的重点是产生和从荧光染色图像个别单元数据的分析,它也是有用的能够采取这些数据和良好的由阱基础总结每个测定以监测重复和性能之间的变异所有的井跨越数据整盘给定的实验中。 图9示出了从各siRNA处理归纳为平均值从一式三份孔的栅极内的百分比的细胞施加到A中的数据)在P-S780 RB1数据和B)中的GFP-CDK2记者数据。绘制在A和 B的值是由设有该手稿两个附加的Perl脚本生成; 'antibody_fluorescence_summary.pl'和'G1assay_summary.pl'上。这些脚本使用每孔用细胞探查(Nuclei.csv)创建的原始数据和报表数据为我)每个测量细胞总数以及,ii)本门内细胞的数量,iii)本门内%的细胞和IV)的算术指的是衡量,原始数据的井。如图此包括作为,着眼于个体治疗数据使用单独的单元数据的多路复用评估之前适于寻找跨越大套测定数据的选项图7和8。图表此处为两个实验,其适合看出对于在图6B中的直方图为P-S780 RB1和GFP-CDK2数据的非正常的数据分布"栅内ⅲ)百分细胞的显示图。这些脚本还计算"ⅳ)测得的,原始数据为该井的算术平均值",这将适合于同质种群的反应和前和实验后扰动正常数据分布的数据进行分析。
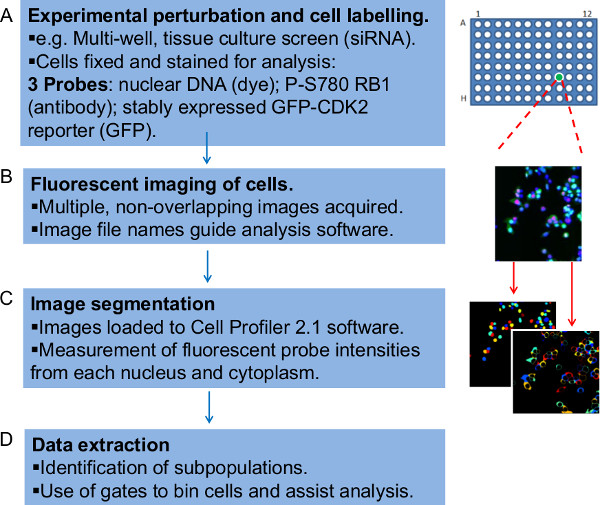
图1:在工作流来定量分析荧光标记的显微镜图像数据的步骤概述工作流在这里表示为4步骤(A)首先,有必要通过实验制备的细胞用于荧光成像。这里所描述的例子是,一个屏,其中siRNA治疗粘附人肿瘤细胞生长48小时,固定和染色在96孔组织培养板。不同的RNA干扰条件都存在一式三份,在板块内部不同孔。细胞染色用DNA染料,具体为RB1磷酸对CDK4和6选择性靶位丝氨酸-780(P-S780 RB1)的抗体,它们也稳定表达GFP-CDK2-记者,报告的G1细胞周期出口。统称这些荧光探针构成两个测定中的工作流中分别评估。(B)的生成,并命名为包括信息通过该图像分析软件可以组织数据并行显微镜图像为每个荧光探针(信道)。(C)中图像文件被加载到细胞Profiler软件,这在算法识别单个细胞和细胞核的相关对和细胞质屈服强度测量的三个荧光探针检测之前编辑只。(D)最后,Perl脚本用于组织生产的原料定量数据。此步骤适用大门的荧光强度数据,每一个细胞,有效分档细胞亚群成,可绘制,跟踪和盘问。 请点击此处查看该图的放大版本。
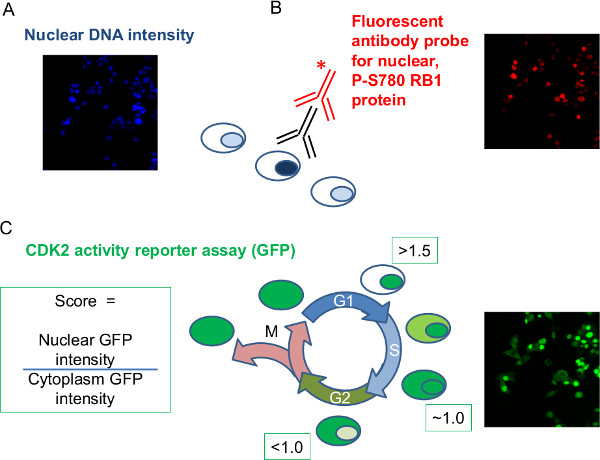
图2:实验数据可以通过图像分析从实施例的数据集进行成像和相应的强度测量每个细胞的采取得到的固定的,siRNA治疗,荧光标记的细胞。代表图像数据示出用于在图像分析记录的各参数(A)的核DNA的强度:核DNA染料的染色强度是用来收率每细胞核的DNA的量度(B)的磷酸化RB1核强度:免疫染色使用主(黑色)抗体和荧光标记的第二抗体(红色)使RB1磷酸化的强度测量在特定的用于P-S780 RB1 。S780每核(C)的 GFP-CDK2记者:使用稳定表达GFP标记报道蛋白,在与细胞周期的一组模式的细胞核和细胞质之间易位的细胞。成对的核和细胞质的GFP强度对每个小区的双重测量允许每细胞的比例,可以用来区分G1期从细胞周期的其余部分的计算。 3个siRNA目标将被用来说明数据;一个非靶向阴性对照siRNA; CDK6的siRNA作为阳性对照在扰动RB1磷酸化和细胞周期进展; RB1的siRNA建立抗体的特异性。K">点击此处查看该图的放大版本。

图3:组织图像文件之前的图像分析从组织培养板拍摄的图像会以允许图像分析软件对图像数据涉及回原始实验上下文系统命名。该信息被放置在文件名 的每个图像内:(A)由于各孔上的实验片可以对应于不同的RNAi目标或治疗,文件名 的井地址形式的一部分。(B)中的帧号是文件名 的一部分。由于每个孔成像收集多个不重叠的帧(C)的各帧的荧光探针分别成像;因此文件名也需要以反映通道的每个图像涉及到。 (D)的实施例的文件名 ,与井(G12),帧(2)与每个图像代表频道(蓝 ,红,绿)中的一个。虚线连接的文件名 元素为好,框架和通道的相关示意图,分别请点击此处查看该图的放大版本。

图4:使用细胞探查来测量核DNA和抗体染色随着提供管道文件中的设置(3_channels_pipeline.cppipe),核DNA和细胞探查图像分析软件测量荧光强度值抗体结合有关个别细胞(A)核被确定在DNA染色的"蓝色"通道图像。(B)< /强>将DNA染色的核的位置暂时在一个"核掩模'保持。该核掩模然后覆盖到(C)的蓝色和红色通道图像(DNA和抗体的荧光数据,分别地)和荧光值从图像片段,与掩模重叠记录针对每个识别出的小区。成功识别的单独的,相邻的核可以在视觉上评估了核掩模的外观。为了说明,示出盘旋在这个掩模图像,就是例子,其中所选择的设置,所述算法有误确定相邻细胞核作为单个核。调整算法设置,以尽量减少这些事件的讨论部分介绍。 请点击此处查看该图的放大版本。
/ftp_upload/51882/51882fig5highres.jpg"/>
图5:使用细胞探查来测量核和细胞质的GFP强度相对于细胞的细胞周期的位置的细胞核和细胞质之间的GFP标记CDK2记者易位。在同一时间,细胞探查计算每个细胞的DNA和抗体核强度( 图4),它也计算核到的GFP强度为每个小区的细胞质比率。(A)中的DNA染料的数据为每个图像被用于生成一个核掩模。(B)的细胞探查使用细胞核中与来自GFP-CDK2记者将GFP图像的种子各单元的位置相结合掩模,然后扩展到每个单元的外围,以估计每个单元的整体足迹。这成为一个新的,"细胞掩模'。(C)的细胞核掩模被从细胞掩模中减去,以产生一个甜甜圈状系列细胞质的轮廓,从而成为了"细胞质掩模'。(D)中的细胞核掩模和细胞质掩模被用来由细胞Profiler来测量双核和细胞质的GFP值。这些成对的值,然后利用细胞Profiler来计算比率,这是通知在细胞周期中的每个单元格的位置。 请点击此处查看该图的放大版本。
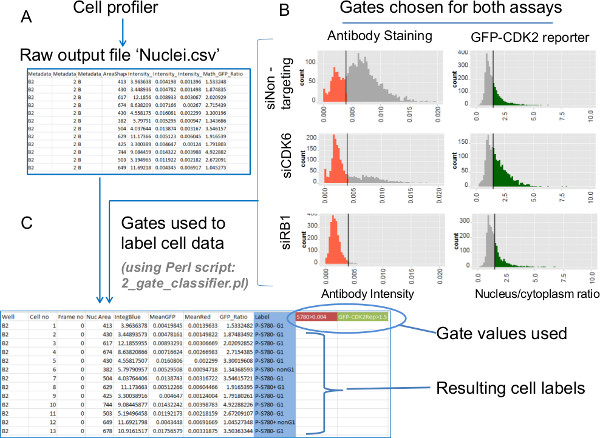
图6:数据提取-通过对测定值施加栅极加工原料个别单元数据从各个单元数据的抗体染色和GFP-CDK2报告分析生物趋势正在使用门控数据提取。原始数据直方图能识别合适的门值。然后这些强加一个Perl脚本。(一)分析与细胞探查所提供的设置图像文件的最终产品是逗号分隔值(.csv)文件。这些文件Çontain与每个不同的亚细胞片段的单个细胞的数据。文件'Nuclei.csv"包含有关使用核面具的所有选定的测量。这些测量包括核抗体强度,核DNA强度和GFP的比(核/细胞质)。核抗体强度(左)和GFP-CDK2记者比(右)从各个细胞的数据为每个siRNA敲除条件绘制的(B)的直方图。在显示的直方图的显示条为这些实验所需的停机位。上的直方图的彩色数据表示门控亚群。(C)的栅极为在乙所示的两种测定被施加到使用Perl脚本'2_gate_classifier.pl'的原始数据。脚本创建原始细胞探查器输出(Nuclei.csv)文件的修改后的副本,以协助后续的绘图。两个闸值被记录在新文件(在这里颜色突出显示)和一个新的"标签"列被添加。标签纸槽每个小区成基于对每个小区在两个门控测定值四种可能的亚组之一。这些标签用于后续的地块其特点各亚群,以及交叉引用的单元格探查产生的附加 参数的贡献计算。 请点击此处查看该图的放大版本。
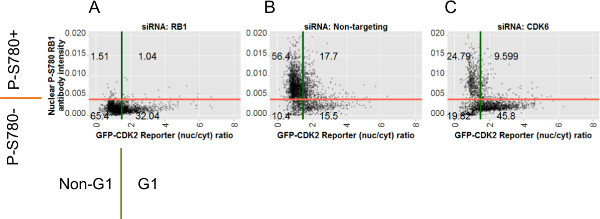
图7:散点图对于每种siRNA条件描绘原始数据的单个细胞和栅极位置散点图INDIVI的从对siRNA的条件的所有图像双节数据显示:(A)siRB1;(B)兴农靶向阴性对照;(C)siCDK6。暗算的Y轴是从抗P-S780 RB1核染色荧光值。作图在X轴是从GFP-CDK2记者计算出的相应的比率值。红色和绿色的条表示门,用于在P-S780 RB1栅极和GFP-CDK2记者门的位置,分别两个栅极分割的细胞分为四个亚群,并通过得到的象限的数字是从每个这些细胞的百分比数目。周围对于A轴的注释指示由2_gate_classifier.pl Perl脚本应用到每个单元中的四个可能的标签的元素。这些标签显示在相对 于它们各自的测定栅极和在R-脚本(analysis.r)被用来生成图的图6,图7和 8。 请点击此处查看该图的放大版本。
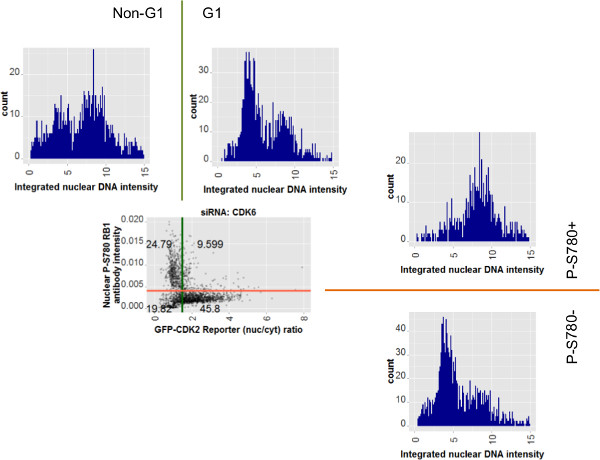
图8:由两个G1转运测定法定义的细胞亚群符合测定结果表明2N和4N DNA图谱为siCDK6单元中的数据的散点图,从图7C重复。周围的散点图的直方图与人口的子集综合核DNA力度。这些散点图上面涉及到GFP-CDK2记者分析。那些散点图的权利涉及到单独的核磷酸RB1抗体测量。着色栅极线延伸,以显示它们之间的关系的直方图。由该单元数据被选定为这些附加的地块栅极标签也示。细胞RB1损失磷酸丝氨酸780(P-S780-),或者那些具有高GFP-CDK2记者核到细胞质的比例(表明低CDK2活性)主要展示2N般的DNA图谱,而其对面的同行对每个各测定显示2N和4N,细胞的混合,后G1期人口特征的分布。 请点击此处查看该图的放大版本。
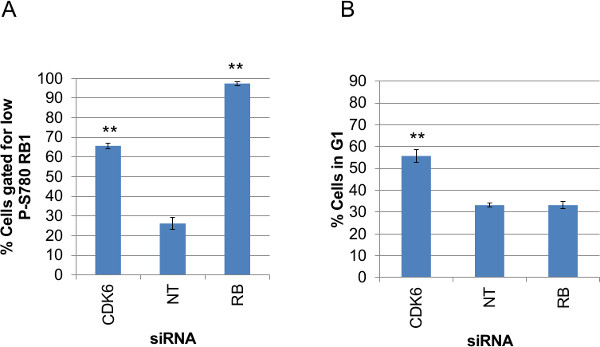
图9:摘要地块的门控(A)P-S780 RB1数据和三孔每个siRNA敲除条件(B)GFP-CDK2数据的每一个siRNA的情况汇总数据图门检测值。值是从使用的原始细胞探查器输出(Nuclei.csv)计算Perl脚本,"antibody_fluorescence_summary.pl"(A)或"G1assay_summary.pl"(B)。作图的值施加到每个测定的栅极内的百分比的细胞的手段。横道从一式三份孔中计算的标准误差。未成,同方差T检验P为每击倒条件相比,非靶向siRNA值显示在上面绘制数据,其中P <0.001(**)和P <0.05(*)。 请点击此处查看本一个更大的版本数字。
图S1。设置单元格Profiler软件进行图像分析。细胞探查的(A)截图之前的任何图像分析都已设置。细胞探查(B)截图已经被加载包含在"3_channels_pipeline.cppipe"算法细节后。高在左上角点亮标签表明该屏幕显示的参数进行分析的'LoadImages"阶段。点击下面这个列表中的其他部分将揭示的细节,在分析后续步骤。探查细胞与细节输入文件夹和输出文件夹进入(C)截图。(D)后,细胞探查的屏幕截图的"分析图像"按钮被点击,开始分析。叠加有三个新视窗表示通过从所分析的图像的软件中产生的算法制作面具。这些窗口通过点击"眼"的图标到打开位置来访问的旁边在分析相关步骤,在主细胞Profiler窗口的左上角。这些意见帮助用户验证产生的纸屑色口罩的设置是否同意随行,原来,灰度数据。
耳鼻喉科"> 图S2。使用Perl和RStudio的栅极单个细胞的数据并绘制得到的细胞亚群。(A)右图显示选择了从细胞探查分析接收输出的.csv文件(绿色图标)的文件夹。提供稿件(蓝色图标)的Perl脚本复制到该文件夹。突出的是"2_gate_classifier.pl"的Perl脚本,它一直双击鼠标产生对话框,在左侧面板中。显示的是加载"analysis.R'脚本后,立即就要门从"Nuclei.csv"文件中的单个单元格数据提示,输入相应的答案。(B)截图RStudio的,突出显示的是从A上传数据选通到命令绘图之前(在线路5和6的细节注释软件将需要根据其中选通数据位于所述计算进行调整用于分析R)。(RStudio权证),一旦数据被上传截图。(RStudio呈现出D)截图强调了代码须出示在右下窗口中显示的情节块。代码为每个小区由空行分隔和情节类型分组。| siRNA靶 | 板以及地址 |
| 非定向(NT) | E5,F5,G5 |
| 视网膜母细胞瘤(RB) | E7,F7,G7 |
| 细胞周期蛋白依赖性激酶6(CDK6) | B2,C2,D2的 |
表1:井地址和在实施例的数据集用对 应的siRNA的条件。
讨论
所描述的工作流构成的程序使用的siRNA,则后续的标记检测细胞的多孔扰动和最后使用的一系列软件支持的步骤,以便从所得的荧光显微镜图像提取的量化数据。该方法是集中交付核和细胞质强度值的单个细胞,其已在许多基于细胞的应用广泛的实际应用。这里使用的示例性数据是在一个siRNA的屏幕画面设定在其中两种荧光测定法G1期细胞周期中转进行测试和相关回核DNA含量的更直接的生物物理测量产生。
使用荧光DNA染色成像核DNA的是,在图像分割过程中不可缺少的步骤,因为它允许识别单个细胞,将所得'核掩模"作为出发点,以确定相应cytoplasmiC区。的GFP标记CDK2记者,其稳定表达在细胞中,给出了一个可变但比通过此隔室可被划定的细胞质背景信号始终更高。同样的分析管道应该适用于使用其它合适的荧光联记者和其响应于扰动蛋白质易位事件的分析。此外,利用细胞质特异的荧光染料取代的GFP-CDK2记者将允许替代使用此算法来测量细胞质的尺寸和单元中的图像的相对尺寸。
另一种设计考虑在这里所描述的图像分割策略是利用细胞探查的提供集成的强度值的DNA定量。强度积分值的核DNA染色数据允许在细胞核大小可能的变化,代表了紧密匹配见过的量化概况为碘化丙啶染色FACS数据。然而,积分强度可能无法提供适当的手段来评估蛋白质的功能,其中平均浓度,通过抗原的荧光强度平均值例举,更生物学相关比一细胞区室中的集成总量蛋白(和相关的荧光)的。因此平均强度值被用于在P-S780 RB1和GFP的数据。可以选择两种模式(平均或集成)的数据评估的细胞Profiler软件的"ExportToSpreadsheet"面板上发现的改变。
在3_channels_pipeline.cppipe文件中的分析设置为在实施例的数据集的图像进行了优化。新的图像设置与此协议分析将需要的文件名 采用命名约定上述( 图3)。另外,感光度值,以满足核DNA染色和阈值背景的亮度在新的图像组的强度可能需要被内细胞探查设置调整。给定的DNA染色保持用于建立各种图像分割掩码的关键作用,正确的灵敏度设置为这个信道的应用程序的关键是与Cell Profiler软件新的图像数据的成功分析。提供的电池事件探查器设置文件(3_channels_pipeline.cppipe)包含了适应分析新的数据最常有用的参数说明。这些说明是在在细胞探查主窗口在屏幕的顶部的文本框,并且包括指导改变灵敏度设置和调整要被分析的信道数。至于起诉的协议第2.8节,以测试设置新的图像数据,可能有必要观察期间的图像分析图像分割点击打开"眼"的图标为每个"标识...... Objects的协议的步骤( 图S1D )。具体地,图像数据的通过"IdentifyPrimaryOjects'可视化显示,如果核掩模被从DNA染色的图像正确识别。在为'IdentifyPrimaryOjects"模块单元Profiler软件页面阈值修正系数。反复试验调整,这个值将解决大多数核识别错误。值平衡的映衬下强度为每个图像的DNA的通道。阈值校正因子值铰链周围1,其中大于这是更严格的(好为清晰的图像)和小于1的宽(适合于图像用染色和背景之间更少对比度)。
个别单元数据的自Cell探查的原始输出可以以不同的方式,以适应其他研究的需要进行分析。这里示出的是使用Perl脚本来门适用于每两个小区中度量的参数,以便协助从数据中提取的生物趋势ð允许交叉引用额外的测量确定的亚群的。尽管它同样可以包括细胞探查的框架内浇口元素,这里用的替代路线提供了更大的灵活性和速度,特别是如果大的数据集需要被评估。在当前协议后图像采集阶段最慢的阶段是Cell Profiler软件的运行。细胞分析器这里用不同栅值运行而不强加门,以产生一个未选通的原始数据集可以与随后的Perl脚本更快地重新分析和,如果需要的话,重复。不是所有的研究将预先知道的合适的门值,因为这可能对任何给定的一组数据用试剂而变化,并有可能随着时间的推移。正是因此,建议生成柱状图,其描绘的小区探查的阳性对照和模型扰动细胞,以获得原始数据分布来确定合适的门VALUES感兴趣的参数。
Perl脚本编写接受细胞探查数据的严格定义的列结构,如果用户修改参数,输出的细胞事件探查器使用"ExportToSpreadsheet"设置的数量可能会停止工作。为了帮助实现票据包括Perl脚本文件中的设置修改。要看到在文本编辑器查看这些脚本,最好是程序员的文本编辑器设置为颜色代码的Perl元素( 例如 ,http://www.activestate.com/komodo-edit)。这些注释指明调整剧本,以适应变化中的数据格式。类似于Perl脚本,包含从绘制图像分析数据的数字说明中提供的R-代码文件(analysis.r),可以读取一个文本编辑器或RStudio软件,看看使用和适应附加说明。这些注释可以与正则表达式和Perl <细节进行补充SUP> 12和ggplot2 13包的R,这两者构成的基础数据是如何读,注释和作图,分别
使用荧光显微镜新的研究,以及保藏公开的出版物的原始数据是适合于分析的方法,例如那些在这里描述的。高含量数据的本质适合于递归分析依赖于任何特定观察者的研究兴趣不同的分析重点。虽然可以提出的数据的问题,是由原本所使用的探针的限制,图像数据通常可以有意义地重新分析超出该生成它们的研究范围。
披露声明
作者什么都没有透露
致谢
这项工作得到了补助CRUK 15043和CRUK 14251的支持。
我们感谢丹尼尔Wetterskog和嘉祺何为技术援助和手稿的批判性阅读。
材料
| Name | Company | Catalog Number | Comments |
| AllStars negative control siRNA | Qiagen | 1027280 | Negative control siRNA. |
| CDK6 siRNA | Dharmacon/ Custom synthesis | NA | Antisense sequence: 5' CUCUAGGCCAGUCUUCUUCUU |
| RB1 siRNA | Dharmacon/ Custom synthesis | NA | Antisense sequence: 5' GGUUCAACUACGCGUGUAATT |
| 5x siRNA buffer | Thermo Scientific | B-002000-UB-100 | To be diluted with nuclease-free, non-DEPC treated water. |
| Nulcease-free water (non-DEPC) | Applied Biosystems | AM9937 | For dilution of siRNA buffer. |
| Hiperfect | Qiagen | 301705 | Transfection lipid. |
| DMEM | Life Technologies | 41966052 | Pyruvate and high-glucose supplemented tissue culture media. |
| 96 well tissue culture plate | Falcon | 3072 | Plain, 96 well tissue culture plate for the parallel, compartmentalized mixing of transfection complexes. |
| Packard Viewplate | Perkin Elmer LAS | 6005182 | 96 well TC plate with optical base on which cells are transfected, grown, fixed and eventually imaged. Supplied with opaque, adhesive plate seals, which are used during storage of used plates. |
| Breathable membrane | Alpha Labs | LW2783 | Sterile, gas-permeable, adhesive membrane. |
| Neutral buffered formalin, 10% | Sigma-Aldrich | HT5012-1CS | 10% Formalin (4% formaldehyde) fixative used neat in protocol. |
| Triton X-100 | Sigma-Aldrich | X100PC | Non-ionic detergent |
| [header] | |||
| Tris | Sigma-Aldrich | T1503 | Trizma base (tris(hydroxymethyl)aminomethane) |
| Tween 20 | Sigma-Aldrich | P2287 | For plate wash solution. |
| Anti-P-S780 RB1 antibody | Abcam | ab32513 | Rabbit monoclonal |
| AlexaFluor647 Anti-rabbit | Invitrogen | A21245 | Highly cross-adsorbed, fluorescently labelled secondary antibody. |
| Hoechst 33342 (Bisbenzimide) | Sigma-Aldrich | B2261 | Fluorescent, chromatin-intercalating, DNA dye. |
| Permeabilization solution | NA | NA | 0.1% Triton X-100 in 50 mM Tris-buffered saline, pH 8.0. |
| Plate wash solution | NA | NA | Tris-buffered saline containing 0.1% Tween-20. |
| Block solution | NA | NA | 5% powdered milk in Tris-buffered saline and 0.1% Tween-20. For blocking plate prior to immuno-staining and dilution of antibodies. |
| Cell Profiler 2.1 | Broad Institute | http://www.cellprofiler.org/download.shtml | |
| Active Perl Community Edition | ActiveState | http://www.activestate.com/activeperl/downloads | |
| R programming environment | The R Foundation | http://www.r-project.org | |
| Rstudio | Rstudio | http://www.rstudio.com/ | |
参考文献
- Carpenter, A. E., et al. CellProfiler: image analysis software for identifying and quantifying cell phenotypes. Genome Biol. 7 (10), 100 (2006).
- Khan, A., Eldaly, H., Rajpoot, N. A gamma-gaussian mixture model for detection of mitotic cells in breast cancer histopathology images. J Pathol Inform. 4, 11 (2013).
- Selzer, P., Beibel, M., Gubler, H., Parker, C. N., Gabriel, D. Comparison of multivariate data analysis strategies for high-content screening. J Biomol Screen. 16 (3), 338-347 (2011).
- Lyman, S. K., et al. High content, high-throughput analysis of cell cycle perturbations induced by the HSP90 inhibitor XL888. PLoS One. 6 (3), e17692 (2011).
- Richardson, E., Stockwell, S. R., Li, H., Aherne, W., Cuomo, M. E., Mittnacht, S. Mechanism-based screen establishes signalling framework for DNA damage-associated G1 checkpoint response. PLoS One. 7 (2), e17692 (2012).
- Heynen-Genel, S., Pache, L., Chanda, S. K., Rosen, J. Functional genomic and high-content screening for target discovery and deconvolution. Expert Opin Drug Discov. 7 (10), 955-968 (2012).
- Krausz, E. High-content siRNA screening. Mol Biosyst. 3 (4), 232-240 (2007).
- Gu, J., Xia, X., et al. Cell Cycle-dependent Regulation of a Human DNA Helicase That Localizes in. DNA Damage Foci. Mol Biol Cell. 15 (7), 3320-3332 (2004).
- Mittnacht, S. Control of pRB phosphorylation. Curr Opin Genet Dev. 8 (1), 21-27 (1998).
- Mittnacht, S. The retinoblastoma protein--from bench to bedside. Eur J Cell Biol. 84 (2-3), 97-107 (2005).
- Nybo, K. GFP imaging in fixed cells. BioTechniques. 52 (6), 359-360 (2012).
- Schwartz, R. L., Foy, B. D., Phoenix, T. . Learning Perl - Making Easy Things Easy and Hard Things Possible. , 1-363 (2011).
- Wickham, H. . ggplot2: Elegant Graphics for Data Analysis. , 978-970 (2009).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。