需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
同心凝胶系统来研究基质微环境的三维细胞迁移的生物物理作用
摘要
机械性能和细胞外基质的组织强烈地影响细胞的三维迁移。 的体外方法来研究在生物物理变量的环境中,在人口和单个细胞水平的时空细胞迁移行为,进行说明。
摘要
细胞迁移的能力是在各种各样的细胞功能,在整个从胚胎发育寿命和伤口愈合肿瘤和肿瘤转移的关键。尽管强烈的研究努力,细胞迁移的基本生物化学和生物物理原理仍然没有完全了解,特别是在生理相关的三维(3D)的微环境。在这里,我们描述了在体外试验设计允许的三维细胞迁移行为定量检查。该方法利用了细胞的mechanosensing能力和倾向迁移到先前未被占据的细胞外基质(ECM)。我们使用高度侵袭性乳腺癌细胞的侵袭,MDA-MB-231,在胶原凝胶作为模型系统。细胞群的传播和单个细胞的迁移动力学数周的文化可以用活细胞成像进行监测和分析,以提取spatiotemporally分辨数据。此外中,该方法很容易适应于不同的细胞外基质,从而提供了一种简单而功能强大的方法来研究的生物物理因素对细胞迁移的微环境中的作用。
引言
细胞迁移起着各种生理反应,如胚胎发育,止血,和免疫反应,以及在病理过程,如血管疾病,炎症,癌症和1的关键作用。解剖底层细胞迁移的生物化学和生物物理因素,因此从根本上重要的是不仅要了解的细胞功能的基本原则,又能促进各种生物医学应用,例如在组织工程,抗转移和抗炎药物开发。由于体内观察是技术上的挑战,有很多的努力一直专注于体外再演细胞迁移。
体外方法来研究细胞迁移已经基本上被设计为检测在二维(2D)表面上,最显着的划伤或伤口愈合测定法2。这些试验提供了简单的实验装置,方便在线 -细胞成像,并提供有益的见解底层细胞迁移的各种生化机制。但是,这些测定法不占外基质(ECM)架构和重塑,这是在理解体内迁移关键方面。最近,人们已越来越多地认识到,3D培养模型,常常在基于胶原的基质3中,提供了更好的类似于体内情况的一个平台。事实上,细胞表现出的迁徙动力学是从那些在2D表面明显,特别是由于不同的维数的环境4。此外,基体的生物物理和机械性能灵敏影响细胞迁移5,包括在肿瘤细胞侵袭6的上下文中。
在这里,我们提出研究三维细胞迁移行为的ECM与生物物理特性,可以轻松地用不同的制备条件的方法。细胞是接种在"内凝胶"和被允许逃逸到并侵入最初无细胞"外凝胶"。该方法依赖于细胞的能力来识别的存在,并倾向成,无细胞迁移区域中的外凝胶,这是密切相关的细胞mechanosensing 7。在这项研究中,我们采用的胶原蛋白网络,通过高度侵袭性乳腺癌细胞,MD-MBA-231入侵的ECM。机械性能和两者的内部和外部的凝胶的微结构可以被调谐8和9,其特征实现生理学相关的条件。重建单元轨道和分析允许在两个群体水平和个体细胞水平的时空迁移行为的详细的定量检查。重要的是,同心凝胶系统的设置模仿面临迁移细胞,尤其是侵入癌细胞,从而提供了重要的见解的体内组织的拓扑对细胞迁移和转移的物理机制。
研究方案
1.细胞收获
- 得到的MD-MBA-231细胞从37℃,5%CO 2的培养箱中培养。分离,以0.5%胰蛋白酶-EDTA溶液从组织培养板上的细胞。使用1个毫升胰蛋白酶-EDTA溶液用于在T25培养瓶中的细胞进行培养。
- 沉淀细胞在15ml锥形管中,离心,在200×g下4分钟,吸出上清液,并悬浮细胞在5ml培养基。
- 计数细胞密度,ρ,使用血球。
注意:为了制备细胞接种内凝胶,将细胞悬液稍后会稀释10×达到最终细胞接种密度。因此,10×浓缩的细胞悬浮液是必需的。 - 计算达到10×细胞浓度所需介质的体积:
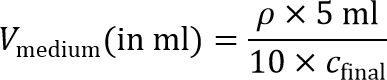
注:最后一个细胞接种密度,C 最终 ,大约215; 10 6个细胞/ ml的被推荐用于MD-MBA-231细胞,并用于在此协议。其他播种密度也可探索其他类型的细胞。 - 沉淀细胞再一次在一个15毫升的锥形试管离心,在200×g下4分钟,吸出上清液。
- 重悬的无血清细胞培养基中的细胞所需要的量(V 培养基在步骤1.3计算的)彻底以最小化细胞聚集。
注意:酚红是自体荧光,并且可以与荧光/反射成像干涉。使用不含酚红的培养基中可以考虑实现最佳的图像质量。
2.准备胶原蛋白解决方案
- 获取股票胶原溶液,10×PBS缓冲液,的Milli-Q H 2 O,0.1M NaOH和几个离心管。保留所有冰上以防止过早胶原蛋白的聚合,并保持无菌状态。
- 平衡无菌玻璃 -底菜在37℃培养箱预热吧。
注:在本协议中的所有卷进行了优化的12 mm孔玻璃底菜。如果其他菜类型时,相应地调整卷。 - 计算需要准备50微升2.4毫克/毫升的胶原溶液为基于所述胶原原料浓度的内凝胶(溶液Ⅰ)所需的体积。
注意:也可以使用其它的胶原蛋白的浓度为所述内凝胶。 - 在无菌的环境(通常是生物安全罩),慢慢加入5微升10×PBS缓冲液至所需的胶原原液量(在步骤2.3计算的)伴以轻轻旋转。注意避免气泡的形成。
- 调节混合物的7.4使用0.1M NaOH中使用校准的pH计的pH。作为一个粗略的指导,使用约5微升以使pH接近7.4(该量取决于该原料浓度和pH下)。
注意:请注意,参与这一卷步骤太小使用标准pH计。请使用以下招数之一:- 制备用于多个样品的胶原蛋白溶液。使用标准pH计调整在散装的pH值和整个样本分配胶原溶液。
- 可替代地,调整的胶原溶液的pH值在大的体积( 即 ,体积,允许使用标准的pH计)。注意,以使pH至最终pH所需的NaOH的量。缩小的容积,并使用氢氧化钠适量实验。确认使用石蕊试纸的pH值。
- 否则,使用微pH电极能够更准确地调节小批量的pH。
- 使溶液于45体积微升使用的H 2 O.执行上冰的所有步骤,以防止过早胶原蛋白的聚合。
3.形成同心凝胶文化
- 就拿预热的玻璃底菜(见步骤2.2)从日Ë孵化器。
- 添加5微升10×浓细胞悬液(步骤1.5准备)来解决一悬浮彻底。注意避免气泡的形成。该混合物现在有50微升的体积,并含有终胶原浓度(2.4毫克/毫升)和细胞密度(C 最终 = 2×10 6个细胞/ ml)。
- 慢慢加入20微升所述含细胞溶液Ⅰ至井的中心,以便它形成一圆顶状的液滴( 图1A)。注意避免泡沫形成在这一步。如果泡沫形成,但仔细赶快试试要么破裂,或用枪头吸出来。轻轻地放在培养皿放回培养箱让内部凝胶聚合45分钟。
- 准备解决O(用于外,无细胞胶原凝胶)的孵育步骤结束前约15分钟。
注意:外凝胶条件可以在胶原蛋白浓度和聚合pH值而言,以改变获得网络的不同的微观结构10。在这个协议中,着眼于一个1.5 - 聚合在pH 7.4 4.0毫克/毫升的胶原凝胶。- 基于该胶原原料浓度,计算需要准备200微升胶原溶液0在最终浓度所需的体积。
- 加入20微升10×PBS中缓慢缓冲到所需的胶原原液(在步骤3.4计算的)伴以轻轻旋转量。调节混合物的pH至用0.1M的NaOH与使用校准pH计的最终pH值。见注有关调节pH值至2.5步。
- 使溶液至200的最终体积使用微升的H 2 O.执行上冰的所有步骤,以防止过早胶原蛋白的聚合。
- 采取从孵化器的菜45分钟聚合内部凝胶后(见步骤3.3)。轻轻的内凝胶上添加180微升溶液O,以使溶液完全覆盖内凝胶和填充井( 图1B)。
- 小心执行此步骤不搅拌的溶液中,这可能会导致在外部凝胶不均匀纤维取向。注意避免接触内部凝胶用枪头,避免气泡或气泡的形成。如果泡沫形成,但仔细赶快试试要么破裂,或用枪头吸出来。轻轻将菜放回孵化器,让外胶聚合。
- 取从培养箱培养皿后聚合45分钟。凝胶应已相当固化,在这一点上,虽然它仍可以从底面脱落如果处理大致。
- 轻轻倾2毫升温热细胞培养基的菜( 图1C)。确保该凝胶在培养基中完全浸没。刷新介质每2 - 3天整个培养的持续时间。
4。活细胞成像
- 使用倒置共聚焦显微镜配备了长期活细胞成像能力进行成像。包括一个内置的孵育室的温度(37℃)和CO 2(5%)控制。接通显微镜和开始实验之前预热阶段至少1小时。
注意:使用物镜具有长工作距离,以优化细胞的观测和定位在3D凝胶。 - 孵育在含有5微升荧光细胞追踪染料30分钟介质的凝胶,以允许细胞在3D系统的准确定位。随后,通过洗涤用1×PBS中去除未结合的染料三次。之后,加入细胞培养基的培养皿中。
- 取菜从孵化器,并把它放在显微镜阶段( 图1D)。
注:活细胞成像能够在原则外凝胶的聚合反应后立即启动。然而,在这点上,小区in中的内部凝胶尚未普及。要检查已侵入外硅胶,活细胞成像可以开始24小时培养开始后(取决于细胞类型)的第一个细胞的迁移。作为粗略导,约12 - 需要14天培养的大多数细胞中的内凝胶进入外凝胶。 - 查看(两用粉饼的)的地区选择卷在内部凝胶周围外胶。后培养24小时,该细胞群体将已经扩散,越过内侧和外侧的凝胶之间的界面,并开始侵入外凝胶。
- 对于两用粉饼的,包括凝胶区域紧邻凝胶界面,中间区,和区域靠近外侧的凝胶7的郊区。排除的区域的距离小于50微米的底面和侧面,以及从该凝胶的顶部,以避免可能的边缘效应。每个两用粉饼一般测量647×647×100微米3(在X,y和z方向上,分 别地),以在z -stack5μm的间隔。
- 确保成像模式,信道/过滤器,曝光时间,和图像分辨率被正确地选择。对于胶原蛋白网络的无标记成像,同时采用激光共聚焦显微镜反射时间推移活细胞成像过程。
- 取样品的图像,画出了强度直方图,并调整增益和偏移来观察足够的信号,并避免饱和通过确保直方图在于零与最大强度之间。不整个实验的持续时间的任何更改变这些设置。
- 取10分钟,8小时(或更长,如果必要的话)的选择的两用粉饼的时间推移的图像,以一时间间隔,ΔT,。
5.细胞跟踪和数据分析
- 使用appropria 的 Z -stack图像进行定量图像后处理德的图像处理软件。
- 段的时间推移的图像来自动选择在(X,Y,Z,t)的三维细胞的位置。
- 对于每一帧,手动检查的定位精度和除去由于细胞碎片和细胞突起可能已被误认为细胞误报。除去从分析和分割重叠或连接成细胞的不同对象增殖性细胞。
- 生成由按时间顺序连接的每个单元的位置,在前面的步骤中获得的细胞的坐标(X,Y,Z,t)的三维时间推移单元轨道。
- 通过删除曲目大于阈轨道长度(通常为20分钟)较短消除随机和系统噪音。
- 如果必要,通过减去总净位移的轨道校正样品漂移。
- 计算单元的位移,尔斯/ ftp_upload / 52735 / 52735eq2.jpg"WIDTH ="80"/>,和细胞迁移的距离,
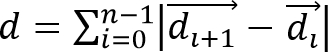 从所观察到的细胞的轨迹,其中,是表示一个小区的时间的三维位置的矢量和是时间点的总数。
从所观察到的细胞的轨迹,其中,是表示一个小区的时间的三维位置的矢量和是时间点的总数。 - 计算单元速度为S = D /(正•Δt),其中Δt是帧之间的时间间隔。计算使用P =量Δd/ D细胞迁移的方向性(或持久性)。持久性的这种简单措施意味着,对于P = 0的净位移是零和用于P = 1的轨迹是直线方向线。
结果
使用高度侵袭性乳腺癌细胞进行这里提出的同心凝胶测定中,MDA-MB-231,用2.4毫克/毫升的内胶原凝胶和= 2×10 6个细胞/ ml的细胞接种密度,作为一个例子。 如图2中 ,典型地经过培养几天,细胞违反内外凝胶接口,并开始侵入外凝胶。该细胞群主要传播径向向外。
外凝胶的聚合条件可被修改以研究对细胞迁移特性的矩阵的密度和机械性能的作用。 图3...
讨论
In this protocol we describe an in vitro assay to study the 3D migrational behavior of cells in matrix environments that topologically resemble ECMs encountered in vivo. There are three main strengths of this assay as compared to other currently available methods. First, this assay allows one to simultaneously examine the cell migration mechanisms at both population level and individual cell level. This opens up possibilities of studying collective cell migration13, which has to date been lar...
披露声明
作者什么都没有透露。
致谢
作者感谢W. Sun和K·詹森的批判性的讨论,并承认支持纳米生物力学实验室,在新加坡国立大学。 NAK承认支持由居里夫人IIF奖学金。
材料
| Name | Company | Catalog Number | Comments |
| Cell culture incubator | Fisher Scientific Pte Ltd | Model: 371, S/No 318854-6055 | |
| Confocal microscope | Nikon A1R | Inverted confocal laser scanning microscope equipped with incubator chamber | |
| Dulbecco's Modified Eagle's Medium (DMEM) | Life Technologies | 11965-092 | |
| Fetal Bovine Serum (FBS) | Life Technologies | 10082-147 | |
| Fluorescent CellTracker dye CMTMR | Life Technologies | C2927 | |
| Glass-bottom dish | IWAKI Cell Biology | 3931-035 | 35 mm diameter dish with 12 mm diameter glass-bottom well |
| Hemocytometer | iN CYTO | DHC-N01 (Neubauer Improved) | |
| Microprocessor pH meter | Hanna Instruments | pH 211 | |
| Nutragen Collagen | Advanced BioMatrix | #5010-D | Acid-solubilized bovine collagen type I (stock pH ~ 2) |
| Objective lens | Nikon | CFI Super Plan Fluor ELWD ADM 20XC, W.D. 8.2-6.9mm, NA 0.45. | |
| Penicillin-Streptomycin | Life Technologies | 15140-122 | |
| pH meter | Sartorius | S/No 29153352 | Basic pH Meter PB-11 |
| Trypsin-EDTA | Life Technologies | 15400-054 |
参考文献
- Horwitz, R., Webb, D. Cell migration. Curr Biol. 13 (19), R756-R759 (2003).
- Liang, C. C., Park, A. Y., Guan, J. L. In vitro scratch assay: a convenient and inexpensive method for analysis of cell migration in vitro. Nat. Protoc. 2 (2), 329-333 (2007).
- Provenzano, P. P., Eliceiri, K. W., Inman, D. R., Keely, P. J. Engineering three-dimensional collagen matrices to provide contact guidance during 3D cell migration. Curr. Prot. Cell Biol. 10, 10-17 (2010).
- Friedl, P., Sahai, E., Weiss, S., Yamada, K. M. New dimensions in cell migration. Nat. Rev. Mol. Cell Biol. 13 (11), 743-747 (2012).
- Grinnell, F., Petroll, W. M. Cell motility and mechanics in three-dimensional collagen matrices. Annu. Rev. Cell. Dev. Biol. 26, 335-361 (2010).
- Wirtz, D., Konstantopoulos, K., Searson, P. C. The physics of cancer: the role of physical interactions and mechanical forces in metastasis. Nat. Rev. Cancer. 11 (7), 512-522 (2011).
- Sun, W., Kurniawan, N. A., Kumar, A. P., Rajagopalan, R., Lim, C. T. Effects of migrating cell-induced matrix reorganization on 3D cancer cell migration. Cell. Mol. Bioeng. 7 (2), 205-217 (2014).
- Achilli, M., Mantovani, D. Tailoring mechanical properties of collagen-based scaffolds for vascular tissue engineering: the effects of pH, temperature and ionic strength on gelation. Polymers. 2 (4), 664-680 (2010).
- Kurniawan, N. A., Wong, L. H., Rajagopalan, R. Early stiffening and softening of collagen: interplay of deformation mechanisms in biopolymer networks. Biomacromolecules. 13 (3), 691-698 (2012).
- Sun, W., Lim, C. T., Kurniawan, N. A. Mechanistic adaptability of cancer cells strongly affects anti-migratory drug efficacy. J. R. Soc. Interface. 11 (99), 20140638 (2014).
- Guzman, A., Ziperstein, M. J., Kaufman, L. J. The effect of fibrillar matrix architecture on tumor cell invasion of physically challenging environments. Biomaterials. 35 (25), 6954-6963 (2014).
- Wolf, K., et al. Physical limits of cell migration: control by ECM space and nuclear deformation and tuning by proteolysis and traction force. J. Cell Biol. 201 (7), 1069-1084 (2013).
- Friedl, P., Gilmour, D. Collective cell migration in morphogenesis, regeneration and cancer. Nat. Rev. Mol. Cell Biol. 10 (7), 445-457 (2009).
- Vedula, S. R. K., Ravasio, A., Lim, C. T., Ladoux, B. Collective cell migration: a mechanistic perspective. Physiology. 28 (6), 370-379 (2013).
- Petrie, R. J., Doyle, A. D., Yamada, K. M. Random versus directionally persistent cell migration. Nat. Rev. Mol. Cell Biol. 10 (8), 538-549 (2009).
- Miron-Mendoza, M., Seemann, J., Grinnell, F. The differential regulation of cell motile activity through matrix stiffness and porosity in three dimensional collagen matrices. Biomaterials. 31 (25), 6425-6435 (2010).
- Mouw, J. K., et al. Tissue mechanics modulate microRNA-dependent PTEN expression to regulate malignant progression. Nat. Med. 20 (4), 360-367 (2014).
- Wolf, K., et al. Collagen-based cell migration models in vitro and in vivo. Semin. Cell Dev. Biol. 20 (8), 931-941 (2009).
- Wong, L. H., Kurniawan, N. A., Too, H. -. P., Rajagopalan, R. Spatially resolved microrheology of heterogeneous biopolymer hydrogels using covalently bound microspheres. Biomech. Model. Mechanobiol. 13 (4), 839-849 (2014).
- Gupton, S. L., Waterman-Storer, C. M. Spatiotemporal feedback between actomyosin and focal-adhesion systems optimizes rapid cell migration. Cell. 125 (7), 1361-1374 (2006).
- Provenzano, P. P., Inman, D. R., Eliceiri, K. W., Trier, S. M., Keely, P. J. Contact guidance mediated three-dimensional cell migration is regulated by Rho/ROCK-dependent matrix reorganization. Biophys. J. 95 (11), 5374-5384 (2008).
- Wolf, K., et al. Multi-step pericellular proteolysis controls the transition from individual to collective cancer cell invasion. Nat. Cell Biol. 9 (8), 893-904 (2007).
- Jansen, K. A., Bacabac, R. G., Piechocka, I. K., Koenderink, G. H. Cells actively stiffen fibrin networks by generating contractile stress. Biophys. J. 105 (10), 2240-2251 (2013).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。