需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
定义底物特异性的脂肪酶和磷脂酶考生
摘要
Many predicted (phospho)lipases are poorly characterized with regard to their substrate specificities and physiological functions. Here we provide a protocol to optimize enzyme activities, search for natural substrates, and propose physiological functions for these enzymes.
摘要
微生物产生的那些,以使可用于生物体外部基板分泌(磷)的脂酶广泛光谱。另外,其他(磷酸)脂肪酶可以在物理上与生产菌引起的内在脂质的营业额,并经常引起细胞膜的重塑有关。虽然势(磷)的脂肪酶可以用数目的算法时该基因/蛋白质序列可用来预测,经常没有获得的酶的活性,底物特异性,和潜在的生理功能的实验证据。这个手稿描述的测定条件与未知底物特异性,以及如何利用这些优化条件下在搜索用于相应(磷)的脂肪酶的天然底物准(磷)的脂肪酶的最优化。采用人工发色底物,如对硝基苯衍生物,可有助于检测小为在标准条件下的预测(磷)的脂肪酶的酶活性。具有遇到这样的次要的酶活性,酶测定法的不同参数可以在为了获得人造基底的更有效的水解而变化。已确定在其下的酶工作良好的条件之后,各种潜在天然底物应测定其降解,其可以采用不同的层析方法进行随后的处理。底物特异性为新酶的定义,通常提供假设这些酶,然后可通过实验测试的潜在生理作用。遵循这些指导原则,我们能够确定降解磷脂酰胆碱磷酸胆碱到甘油二酯和,在膜中生长时的磷限制条件细菌根瘤菌重塑的关键一步磷脂酶C(SMc00171)。两个预测patatin-就像有机体一样的磷脂酶(SMc00930和SMc01003),我们可以重新确定自己的底物特异性和澄清SMc01003是甘油二酯脂肪酶。
引言
基于甘油脂质,如三酰甘油和(甘油)磷脂构成了重要的可能是最知名的类脂质1。三酰基甘油(变量)的脂肪或油,其通常用作存储脂类,因此作为潜在的能量和碳的来源。标签可以通过脂肪酶,这是经常被生产菌分泌的消化外部变量并使它们可作为碳源退化。另外,脂肪酶已经被广泛地研究,多年来,由于其重要的生物技术应用2。
由于它们的两亲性质及其近圆筒状的形状,(甘油)磷脂展览成膜性,通常构成了双层膜3的主要类脂组分。在简单的微生物,如细菌大肠杆菌中,只有三个主要头组的变体,磷脂酰甘油(PG),心磷脂(CL),和phosphatidylethanolamine(PE)遇到,尽管人们应该知道,他们中的每一个都可以用在sn相当数量的不同脂肪酰基链的-1或SN -2位置引起大量不同分子种类4的被取代。其他细菌可能具有除了或代替其他磷脂。例如, 苜蓿中华根瘤菌 ,土壤细菌,其能够形成与豆科苜蓿( 紫花苜蓿 )一个固氮根瘤共生,包含除了PE的第二两性离子磷脂,磷脂酰胆碱(PC),5。另外,不含有脂质磷或甘油可能是细胞膜的两亲性并形成部分。例如,在磷限制生长条件下,在S根瘤菌 ,(甘油)磷脂主要由膜脂质不包含磷, 即,硫脂,鸟氨酸脂质和diacylglyceryl trimethylho取代moserine(DGTS)6。在细菌中,DGTS从二酰甘油(DAG)在一个两步通路7形成,但是对于DAG生成源不明确。脉冲追踪实验表明个人电脑可能是DGTS 8的前体,并使用在这个手稿,我们可以识别磷脂酶C(PLCP,SMc00171)中描述的方法,该方法是磷限制性的条件下形成并且可PC转换为DAG和磷酸8。
在另一项研究中,我们发现,酰基-CoA合成酶(FADD)S的缺陷型突变体进入生长9的固定相时的根瘤菌或大肠杆菌积累游离脂肪酸。尽管这些脂肪酸似乎是从膜脂质衍生自游离脂肪酸或解放出来的酶的确切来源没有已知的。再次,采用的策略在这个手稿概述,二patatin启动类10(磷)的脂肪酶(SMc00930和SMc01003),该对S中形成的游离脂肪酸贡献根瘤菌 11进行了预测。令人惊讶地,SMc01003使用的DAG作为底物将其转换为单酰甘油和最后甘油和游离脂肪酸11。因此,SMc01003是DAG脂肪酶(DGLA)。
虽然用于预测潜在(磷)存在一些算法脂酶12,13,它们的精确的功能和生理作用通常是不知道的。在这里,我们勾勒出一个协议,克隆和过度预测的或潜在的(磷)的脂肪酶。这份手稿解释酶检测如何开发和利用人工色底物的过度表达(磷)脂肪酶进行了优化。我们提供的例子,这些研究结果可能如何与优化的酶测定可遇到的实际(磷酸),酶底物,以及如何丰富我们的微生物生理学的理解。
研究方案
1.克隆和过度表达结构基因的脂肪酶预测
- 使用聚合酶链反应(PCR)14和特异性寡核苷酸( 表1)15,扩增感兴趣(smc01003,smc00930或smc00171)的基因,预测为脂肪酶编码或磷脂酶,从宿主生物的基因组DNA( 即, 苜蓿根瘤菌 )。
- 引入特定的限制性位点(有寡核苷酸的设计的序列)。消化与相应的限制性内切酶的扩增的DNA片段,并将其克隆到表达载体中,如PET系列16的质粒。
- 验证为克隆基因的正确DNA序列后,转化载体至表达菌株如大肠杆菌 BL21(DE3)pLysS中16。
- 准备表达宿主大肠杆菌的过夜预培养大肠杆菌 BL21(DE3)PLYSS,窝藏与克隆基因或空载体的各个pET载体,在含有20ml的Luria BERTANI肉汤(LB)17 100毫升培养瓶中加所需的抗生素。培养细胞在30℃(或在从该脂肪酶源自细菌的通常生长温度)。
- 使用过夜预培养物,接种在2L培养瓶中500毫升预热的LB培养基(加所需抗生素)在620nm(OD 620)= 0.05,以获得一个初始光密度。跟随在的OD 620 = 0.3培养物和生长,添加异丙基β-D-硫代半乳糖苷(IPTG)至100μM的最终浓度,并在搅动下孵育在30℃,为期4小时。
- 在温育期结束时,在5000 xg离心传送各培养到500ml的离心管并离心在4℃下30分钟。重悬细菌细胞沉淀在5ml悬浮缓冲液( 例如 ,SMc00930-和SMc01003表达细胞在50mM的Tris-HCl pH 8.0的和SMc00171表达细胞在50mM二乙醇胺 - 盐酸pH值9.8)。在-80°C,直到使用储存细胞悬液。
2.准备无细胞蛋白提取物和确定蛋白质浓度
- 除霜细菌细胞悬浮液并储存在冰上。传递细胞悬液通过冷压细胞三次,每2磅20000。离心5000 xg离心在4℃下除去完整细胞和细胞碎片30分钟。
- 离心后,从上清液用于后续分析准备的100和500微升等分试样,并将其存储在-80℃直至使用。
- 使用100微升等分试样中的一个由选择的方法或如所述18,以确定不同的无细胞提取物的蛋白质浓度。
3.使用人工基质用于优化酶的(磷)的脂肪酶活动
- 对于不同的酶活性的初始覆盖,使用通过水解产生有色产物人工基质,如对硝基苯酚( 第 -NP)。
- 对于人工对硝基苯酯底物已经过优化的酶测定(概述的磷脂酶C PLCP(SMc00171),以及对预测的patatin启动状磷脂SMc00930和SMc01003),在表2中记载的使用移液的方案。
- 当探索新的潜在的(磷)的脂肪酶,制备含有50mM Tris-盐酸,pH值8.5,100mM NaCl的,0.05%的Triton X-100,0.5mM的磷复合含有硝基苯基( 对硝基苯磷酸盐的第一标准酶测定,二- 对硝基苯基酯,磷酸对硝基苯癸酸盐,或对硝基苯基棕榈酸酯),和无细胞蛋白提取物(检查1,3,10,30,100,300,和1000微克)以1:1的总体积毫升在1毫升塑料CUVEttes。
注意:下面的连续测定对硝基苯酯的水解时使用的碱性pH值( 图1)。另外,使用单个时间点测定法的范围内的pH值,在温育期结束时加入NaOH以终止酶反应,并确保所有的p -NP存在于酚形式。 - 按照时间过程为在30℃下在5分钟内于405nm的增加吸光度,因为p -NP的形成,在分光光度计。通过确定每时间吸光度的增加的初始斜率量化p -NP的最初线性形成。
- 计算浓度(ΔC)的对p-NP使用朗伯-比尔定律(ΔA=εΔCd)1名的变化。
注:ΔA是吸光度确定的线性变化,ε是在各自的波长的摩尔消光系数(以M为单位-1 -1),d为光路(1厘米)的长度,和ΔC是浓度的(待确定的变化以M为单位)。- 考虑到分析体积为1毫升,计算形成在p -NP量。
注:金额=浓度X卷。 - 除以在它形成时形成在p -NP的量计算出的酶活性。通过蛋白质的这是负责生成该活动的量(以mg)除以酶活性确定特定的酶的活性。
- 考虑到分析体积为1毫升,计算形成在p -NP量。
- 通过比较蛋白质提取物挑起吸光度的变化,其中一个候选基因(smc00171,smc00930或smc01003)已表达了与港仅一个空向量提取物。
注意:为了继续执行下面的步骤,具体活动,造成其中的候选基因已被表达的蛋白提取物,应至少两次或间矿石比造成窝藏只有一个空的载体蛋白提取物的具体活动得到的数值。 - 进一步的实验中,选择这些条件,其中含有硝基苯- 对化合物的水解是最小的与无细胞提取物( 即,空载体)和的量,最明显形成p -NP的与p -nitrophenolate阴离子( 图1)可以在蛋白质提取物中使用的,其中一个候选基因已被表达进行观察。
- 确定在3.1的初始酶活性后,通过改变pH值,缓冲液类型优化用于各酶的测定条件,缓冲强度的NaCl浓度,洗涤剂如Triton X-100和不存在的或不同的二价阳离子的存在。
- 对于不同浓度的各变量,确定特定酶的活性(见3.1.4.2)(获得最高数目限定的条件最大酶活性的)。使用的每个变量所遇到的最佳条件的组合来定义一个优化的酶测定,其中每个变量是存在于它的最佳浓度。
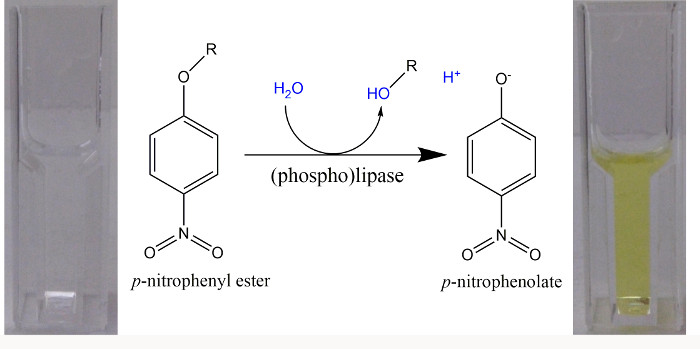
图1. 对 硝基苯酯在分光光度测定法(磷)的脂肪酶作为人工基质。当对硝基苯酯的水解,酸(R-OH)和对硝基苯酚( 对 -NP)形成。由于pK a为= 7.2酚类的 H +从对 -NP解离,在pH> 9.2的99%以上是在明亮的黄色p -nitrophenolate形式和18000 M -1 cm的摩尔消光系数- 1可以在波长使用为游离对 -nitrophenolate 22的量化405纳米。当使用具有pH为8.5缓冲,吸收在400 nm和14500 M -1厘米的摩尔消光系数确定-1采用23。 请点击此处查看该图的放大版本。
注:在已经确定的最佳条件为所关注的酶的活性,踏上寻求本脂肪酶的实/生理底物。原则上,利用2个,经常补充,方法来实现这个目标, 在体内方法或体外方法。
4.一脂肪酶的生理底物的体内鉴定
ove_content">注意: 在体内的方式,表达了在宿主生物体8,11感兴趣的脂肪,才能随着时间的推移脂肪酶的表达是否改变了host's血脂登记在另一个体内的方法,生成突变体缺乏兴趣8,11的基因并研究其脂质分布是否是由野生型版本6,8,11是不同的。为了得到生物体的脂质分布的定量评估,一个简单的方法包括放射性标记细胞化合物,提取脂质,通过色谱法将它们分离,并量化放射性标记的分离脂质。- 脂类的放射性标记。
- 在5毫升的所需培养基(复合培养基或定义基本培养基)的制备的兴趣( 大肠杆菌或S.根瘤菌 )有机体的过夜预培养,并在30℃下生长。
- 从预培养,接种20毫升的股份有限公司在100毫升培养瓶中箱新鲜培养基,得到初始OD 620 = 0.3的培养。
- 取培养物的等分试样(1ml)中在无菌条件下,并转移到14毫升无菌聚苯乙烯圆底管中。
- 添加[1- 14 C]乙酸盐(每毫摩尔60毫居里)的1 ml培养1微居里。
- 孵育在搅拌下在液体培养物在30℃,为期24小时。
- 在温育期结束时,转移培养到1.5ml微量离心管中并离心以12,000 xg离心在室温下5分钟。
- 重悬在100μl水中沉淀。在这一点上,存储所述细胞悬浮液在-20℃下或立即继续与极性脂质(第4.2节)的提取。
- 极性脂质的提取。
注:这里主要介绍的方法遵循布莱和代尔19日报道的过程。- 以100微升水细胞停赛的ension,添加375微升甲醇:氯仿溶液(2:1;体积/体积)。
- 涡旋30秒,并孵育在室温下5分钟。
- 离心5分钟,在室温下以12000 xg离心。
- 将上清转移至新的1.5 ml离心管中。
- 加氯仿的125微升和125微升水,旋涡30秒。
- 离心1分钟,在室温下12000 xg离心。
- 下部氯仿相转移到新的管中,并干燥,用氮气流。
- 溶解在100μl氯仿中干燥脂质:甲醇溶液(1:1;体积/体积)。
注意:在这一点上,5微升脂质溶液的等分试样可通过液体闪烁计数进行定量。 - 对于薄层色谱(TLC)分析,干下来其余95微升用氮气流和在20μl氯仿中再溶解干燥的脂质:甲醇溶液(1:1;体积/体积)。使用3微升等分对于TLC分析。
- 通过薄层色谱(TLC)极性脂质的分离。
注意:根据所述脂质类进行分析,固体和流动相的不同组合可能被用于分离。此处带电极性脂质和另一个,更适合中性极性脂质,采用高性能薄层色谱法(HPTLC)硅胶铝板作为固相的典型分离,进行了概述。- 由二维TLC(2D-TLC)带电极性脂质的分离。
- 在HPTLC硅胶铝板(10×10厘米),从板的边缘2厘米一角应用脂质样品的一个3微升等分试样。
- 准备与在第一维混合用于分离的流动相(140毫升氯仿60毫升甲醇,和10毫升水)。
- 大衣色谱纸TLC发展内部腔。
注意:这是为了确保该室的气体相将用于第一维流动相后迅速饱和(30分钟内)已经被添加到腔室和腔室已被封闭的玻璃板。 - 制备并混合流动相(130毫升氯仿,50ml甲醇,和20ml冰醋酸),用于在所述第二维分离,并转移到一第二薄层显影室内部涂覆有色谱纸,让腔饱和。
- 小心地将薄层硅胶铝板与干燥的脂质样品在封闭腔室中的第一维5转移到第一室和发展( 即 ,执行色谱法)该板60分钟。
- 从腔室中取出板,并让溶剂擦干在一个流罩30分钟。
- 90度相对于转动板到先前层析后,转移薄层硅胶铝板,在其上的脂质已经在一个维度被分离,给塞康ð腔和开发该板在第二维5 60分钟。
- 除去从腔室的薄片,让该溶剂干燥掉在一个流罩至少2小时。
- 中性极性脂质的分离。
- 适用于启动从板的边缘2cm的薄层硅胶铝板脂质样品3微升等分试样。如果多个样品在一维色谱分析,保持的不同示例应用点之间至少1.5厘米的距离。
- 制备并混合流动相(140毫升己烷,60毫升乙醚,和8ml乙酸),并转移到一个薄层显影室内部涂覆有色谱纸和覆盖有一个玻璃板,让腔饱和(30分钟)。
- 与干燥的脂质样品转移薄层硅胶铝板到腔和发展板在封闭室30分钟。
- 从室中取出板并让溶剂擦干在一个流罩2小时。
- 由二维TLC(2D-TLC)带电极性脂质的分离。
- 定量分离极性脂类的可视化。
- 一旦开发了薄层片材干燥,以光激励发光(PSL)屏幕孵育它在一个封闭的盒3天。
- 露出孵育屏幕到PSL扫描器并获取分离放射性标记的脂质的虚像。
- 使用PSL软件20进行量化。
- 可视化和个人极性脂质类的隔离。
- 孵育发达型TLC片在以1g碘晶体的存在下层析室10分钟。
注:分居脂质化合物会溶解碘和出现褐色斑点。 - 圈用铅笔斑点,把它们比作标准化合物( 即 1,2- dipalmitoyl- SN -甘油,二棕榈-L-α-磷酸的相对移动(R F)phatidylcholine,DL-α-甘油一棕榈酸酯,或棕榈酸),并确定它们可能属于哪脂质类。
- 在通风橱中,让碘在TLC板蒸发。
- 用刮铲的帮助下,刮含有从纸张目的产物的硅胶,并用100微升的水和375微升甲醇中的混合物中提取的硅胶化合物:氯仿溶液(2:1;体积/体积)。
- 根据布莱和代尔拔牙继续概述(4.2.2起)。
- 商店纯化脂质类在100μl的氯仿:甲醇溶液:;在-20℃直到使用(1体积/体积)。
- 孵育发达型TLC片在以1g碘晶体的存在下层析室10分钟。
5.脂肪酶的生理底物的体外鉴别
注意:在体外的方法,研究感兴趣的脂酶能否分离脂质或各个纯脂质的混合物转化成相应的hydrolysis产品在3.2定义为最佳的条件下进行。
- 使用酶分析吹打方案按表3为PC专用磷脂酶C SMc00171(见5.2),磷脂酶A(见5.3),以及DAG脂肪酶SMc01003(见5.4)的活动。
- PC专用磷脂酶C活性( 表3)的测定。
- 到1.5 ml离心管中,加每分钟5,000计数单位共14个 C标记的PC(CPM)和Triton X-100的解决方案。
- 混合和干燥在氮气流。
- 添加二乙醇胺-盐酸,pH值9.8的缓冲,以及NaCl和的MnCl 2溶液和二次蒸馏水,以获得99.5微升的最终体积。涡旋5秒。
- 加入0.5微升酶(5微克蛋白质)的( 即,无细胞提取物,其中过表达SMc00171存在),以引发反应。简单地混合。
- 孵育在30℃下4小时。
- 停经反应另外的甲醇250微升和125微升氯仿。
- 提取如先前所描述的脂质(见4.2)。
- 单独的脂质通过一维(1D)-tlc(参见4.3.2和4.4),并通过PSL成像分析它们。
- 磷脂酶A活性的测定( 表3)。
- 到1.5 ml离心管中,加总14 C标记的磷脂5000 CPM和Triton X-100的解决方案。
- 混合和干燥在氮气流。
- 为最终100μl的测定法中,添加的Tris-HCl,pH值8.5的缓冲,NaCl溶液和水。涡旋5秒。
- 添加5微升的酶(50微克蛋白质)( 即 ,无细胞提取物,其中过表达SMc00930或SMc01003存在)。
- 孵育在30℃下5小时。
- 停止通过加入甲醇250微升和125微升氯仿的反应。
- 提取如先前所描述的脂质(见4.2),独立他们通过使用1D型TLC130毫升氯仿,50ml甲醇,和20ml冰醋酸作为流动相,并且通过PSL成像分析它们。
- 甘油二酯(DAG)脂肪酶活性的测定。
- 14 C标记的DAG的制备。
- 放射性S.根瘤菌文化(见4.1),并提取极性脂质(见4.2)所描述的。独立S.利用在4.3.1中第二维分离所述的条件;(体积/体积20 130:50), 根瘤菌总脂质提取物通过1D-TLC氯仿:甲醇乙酸。
- 由碘染色可视化的PC和用铅笔标记磷脂酰胆碱(PC)的本地化。
- 按照4.5中所述隔离放射性标记的PC。
- 通过闪烁计数定量提取PC。
注:约32 CPM PC的预期。 - 治疗用0.1ü 产气荚膜梭菌磷脂酶C的PC(250,000 CPM)在50毫米的Tris-HCl,pH值7.2,0.5%的Triton X-100和10mM氯化钙2为在100μl的总体积2小时,停止加入甲醇250微升和125微升氯仿的反应。
- 因为他们通过1D-TLC(见4.3.2)以前和独立的描述中提取脂类。
- 从硅石板隔离的二酰基甘油和通过闪烁计数量化(如在4.2中描述)
- 二酰基甘油脂肪酶测定( 表3)。
- 到1.5 ml离心管中,加入14 C标记DAG 5000 CPM和Triton X-100的解决方案。
- 混合和干燥在氮气流。
- 为最终100μl的测定法中,添加的Tris-HCl(pH为9.0)缓冲液,NaCl溶液和二次蒸馏水。涡旋5秒。
- 发起通过添加5微升的酶(无细胞提取物的50微克蛋白质)的反应。
- 孵育在30℃下4小时。
- 停止通过加入250微升甲醇中的反应ð125微升如前所述氯仿和提取脂质(见4.2)。
- 分析由1D-TLC(见4.1.3.2)和随后的PSL成像中性极性脂质。
- 14 C标记的DAG的制备。
结果
PC专用磷脂酶C SMc00171的活性的双- 对 硝基苯磷酸酯
从大肠杆菌获得的无细胞提取物大肠杆菌 BL21(DE3)×pLysS中,其中有smc00171表达,进行了研究它们对水解二- 对硝基苯基磷酸酯的能力,使用分光光度酶促测定法中,测量形成p -NP。没有水解活性是?...
讨论
在过去的20年中,许多生物基因组研究已经被测序,虽然丰富的基因组序列数据已经产生,功能解释相对滞后,因此阻碍了我们的基因组功能的认识。在基因组中的基因功能的基础上相似已知功能或保守的基序的发生基因经常被分配。然而,给定基因的精确功能往往不是公知的。尤其,预计酶的结构基因不能由组学技术容易地探索由于这样的事实,大部分酶催化涉及两个基板和两个产品的复杂的?...
披露声明
作者什么都没有透露。
致谢
这项工作是由(在InvestigaciónCientíficaBASICA在Investigación连接国界德拉西恩西亚82614,153998,253549和178359以及118),并从提高妇女地位总署德从理事会全国国立科学城ŸTECNOLOGIA - 墨西哥(国家科学技术委员会和墨西哥)资助项目Asuntos去个人Académico-国立自治大学墨西哥(DGAPA,墨西哥国立自治大学; PAPIIT IN202616,IN203612)。
材料
| Name | Company | Catalog Number | Comments |
| Chloroform | JT Baker | 9180-03 | TLC analysis & Lipid extraction |
| Methanol | JT Baker | 9070-03 | TLC analysis & Lipid extraction |
| Acetic Acid | JT Baker | 9507-05 | TLC analysis & Lipid extraction |
| Hexanes | JT Baker | 9309-02 | TLC analysis & Lipid extraction |
| Diethylether | Sigma | 32203 | Enzymatic assays |
| bidistilled water | ANY | NA | Enzymatic assays |
| Tris Base | Sigma | T-1503 | Enzymatic assays |
| HCl | Baker | 9535-02 | Enzymatic assays |
| NaCl | Baker | 3624-01 | Enzymatic assays |
| Triton X-100 | Sigma | X-100 | Enzymatic assays |
| LB broth | ANY | NA | Bacterial growth, 10 g tryptone + 5 g yeast extract + 10 g NaCl per liter of bidistilled water |
| tryptone | Becton Dickinson and Company | 211705 | Bacterial growth |
| yeast extract | Becton Dickinson and Company | 212750 | Bacterial growth |
| TY broth | ANY | NA | Bacterial growth, 8 g tryptone + 3 g yeast extract + 66 mg CaCl2·2H2O per liter of bidistilled water |
| CaCl2·2H2O | Baker | 1332-01 | Enzymatic assays |
| isopropyl-β-D-thiogalactoside (IPTG) | Invitrogen | 15529-019 | Bacterial growth |
| Diethanolamine | Sigma | D-8885 | Enzymatic assays |
| MnCl2 | Sigma | 221279 | Enzymatic assays |
| Phospholipase A2 snake venom | Sigma | P0790 | Enzymatic assays |
| Phospholipase C Clostridium perfringens | Sigma | P7633 | Enzymatic assays |
| Bis-p-nitrophenyl phosphate | Sigma | 07422AH | Enzymatic assays |
| p-nitrophenyl stearate | Sigma | N3627 | Enzymatic assays |
| p-nitrophenyl dodecanoate | Sigma | 61716 | Enzymatic assays |
| p-nitrophenyl decanoate | Sigma | N0252 | Enzymatic assays |
| p-nitrophenyl palmitate | Sigma | N2752 | Enzymatic assays |
| p-nitrophenyl butyrate | Sigma | N9876 | Enzymatic assays |
| p-nitrophenyl octanoate | Sigma | 21742 | Enzymatic assays |
| Acetic Acid, sodium salt [1-14C] | Perkin Elmer | NEC084 | Bacterial growth |
| dimethylsulfoxide (DMSO) | JT Baker | 9224-01 | Enzymatic assays |
| Aluminium HPTLC silica gel 60 plates. Silica gel HPTLC plates size 20 x 20 cm, 25 sheets. | Merck | 105547 | TLC analysis & Lipid extraction |
| Spectrometer UV/VIS Lambda 35 | Perkin Elmer | NA | Enzymatic assays |
| Storm 820 Phosphorimager | Molecular Dynamics | NA | Photostimulable Luminescence scanner |
| Multipurpose Scintillation Counter | Beckman Coulter | NA | Radioactivity Quantification |
| French Pressure Cell | ThermoSpectronic | NA | Breakage of cells |
| chromatography paper 3MM Chr | Whatman | 3030917 | TLC analysis |
| Sinorhizobium meliloti 1021our | reference 11 | studied strain | |
| Escherichia coli BL21 (DE3) pLysS Competent cells | Novagen | 69451 | protein expression strain |
| pET9a vector | Novagen | 69431 | protein expression vector |
| pET17b vector | Novagen | 69663 | protein expression vector |
| sterile polystyrene round-bottom tube (14 ml) Falcon | Becton Dickinson | 352057 | radiolabeling of bacterial cultures |
| polypropylene microcentrifuge tubes (1.5 ml) | Eppendorf | 30125.15 | Enzymatic assays |
| 1,2-dipalmitoyl-sn-glycerol | Sigma | D9135 | lipid standard |
| L-α-phosphatidylcholine, dipalmitoyl | Sigma | P6267 | lipid standard |
| DL-α-monopalmitin | Sigma | M1640 | lipid standard |
| palmitic acid | Sigma | P0500 | lipid standard |
参考文献
- Nelson, D. L., Cox, M. M. . Lehninger, Principles of Biochemistry. , (2013).
- Jaeger, K. E., Eggert, T. Lipases for biotechnology. Curr. Opin. Biotechnol. 13 (4), 390-397 (2002).
- Dowhan, W., Bogdanov, M., Mileykovskaya, E. Functional roles of lipids in membranes. Biochemistry of Lipids, Lipoproteins and Membranes. , 1-37 (2008).
- Dowhan, W. Molecular basis for membrane phospholipid diversity: why are there so many lipids?. Annu. Rev. Biochem. 66, 199-232 (1997).
- De Rudder, K. E. E., Thomas-Oates, J. E., Geiger, O. Rhizobium meliloti mutants deficient in phospholipid N-methyltransferase still contain phosphatidylcholine. J. Bacteriol. 179, 6921-6928 (1997).
- Geiger, O., Röhrs, V., Weissenmayer, B., Finan, T. M., Thomas-Oates, J. E. The regulator gene phoB mediates phosphate stress-controlled synthesis of the membrane lipid diacylglyceryl-N,N,N-trimethylhomoserine in Rhizobium (Sinorhizobium) meliloti. Mol. Microbiol. 32 (1), 63-73 (1999).
- Klug, R. M., Benning, C. Two enzymes of diacylglyceryl-O-4'-(N,N,N,-trimethyl)homoserine biosynthesis are encoded by btaA and btaB in the purple bacterium Rhodobacter sphaeroides. Proc. Natl. Acad. Sci. USA. 98 (10), 5910-5915 (2001).
- Zavaleta-Pastor, M., et al. Sinorhizobium meliloti phospholipase C required for lipid remodeling duringphosphorus limitation. Proc. Natl. Acad. Sci. USA. 107 (1), 302-307 (2010).
- Pech-Canul, A., et al. FadD is required for utilization of endogenous fatty acids released from membrane lipids. J. Bacteriol. 193 (22), 6295-6304 (2011).
- Banerji, S., Flieger, A. Patatin-like proteins: a new family of lipolytic enzymes present in bacteria?. Microbiology. 150 (Pt 3), 522-525 (2004).
- Sahonero-Canavesi, D. X., et al. Fatty acid-releasing activities in Sinorhizobium meliloti include unusual diacylglycerol lipase. Environ. Microbiol. 17 (9), 3391-3406 (2015).
- Fischer, M., Pleiss, J. The Lipase Engineering Database: a navigation and analysis tool for protein families. Nucl. Acid. Res. 31 (1), 319-321 (2003).
- Sigrist, C. J. A., et al. New and continuing developments at PROSITE. Nucleic Acids Res. 41 (Database issue), D344-D347 (2013).
- Lorenz, T. C. Polymerase Chain Reaction: Basic Protocol Plus Troubleshooting and Optimization Strategies. J. Vis. Exp. (63), e3998 (2012).
- Untergasser, A., et al. Primer3 - new capabilities and interfaces. Nucl. Acids Res. 40 (15), e115 (2012).
- Studier, F. W., Rosenberg, A. H., Dunn, J. J., Dubendorff, J. W. Use of T7 RNA polymerase to direct expression of cloned genes. Methods Enzymol. 185, 60-89 (1990).
- Miller, J. H. . Experiments in Molecular Genetics. , (1972).
- Desjardins, P., Hansen, J. B., Allen, M. Microvolume Protein Concentration Determination using the NanoDrop 2000c Spectrophotometer. J. Vis. Exp. (33), e1610 (2009).
- Bligh, E. G., Dyer, W. J. A rapid method for total lipid extraction and purification. Can. J. Biochem. Physiol. 37 (8), 911-917 (1959).
- Molecular Dynamics. . Phosphorimager SI User´s Guide. , (1994).
- Dixon, M., Webb, E. . Enzymes: Third Edition. , (1979).
- Rudolph, A. E., et al. Expression, characterization, and mutagenesis of the Yersinia pestis murine toxin, a phospholipase D superfamily member. J. Biol. Chem. 274 (17), 11824-11831 (1999).
- Kato, S., Yoshimura, T., Hemmi, H., Moriyama, R. Biochemical analysis of a novel lipolytic enzyme YvdO from Bacillus subtilis. Biosci. Biotechnol. Biochem. 74 (4), 701-706 (2010).
- Peppelenbosch, M. P. Kinome profiling. Scientifica (Cairo). , (2012).
- Manafi, M., Kneifel, W., Bascomb, S. Fluorogenic and chromogenic substrates used in bacterial diagnostics. Microbiol. Rev. 55 (3), 335-348 (1991).
- Kuznetsova, E., et al. Enzyme genomics: Application of general enzymatic screens to discover new enzymes. FEMS Microbiol. Rev. 29 (2), 263-279 (2005).
- Scopes, R. K. . Protein Purification, Principles and Practice. , (2010).
- Geiger, O., Lopez-Lara, I. M., Sohlenkamp, C. Phosphatidylcholine biosynthesis and function in bacteria. Biochim. Biophys. Acta. 1831 (3), 503-513 (2013).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。