需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
电压钳荧光测定
摘要
本文介绍了使用荧光非天然氨基酸(fUAA)代替马来酰亚胺染料的常规电压钳子荧光测量(VCF)的增强,以探测离子通道中的结构重排。该程序包括非洲爪蟾卵母细胞DNA注射,RNA / fUAA共注射,以及同步电流和荧光测量。
摘要
电压钳式荧光法(VCF)一直是研究电生物膜蛋白的结构和功能的选择技术,其中荧光和电流的实时测量同时报告局部重排和全局功能。虽然高分辨率结构技术如低温电子显微镜或X射线晶体学提供了感兴趣的蛋白质的静态图像,但VCF提供了动态结构数据,使我们能够将结构重排(荧光)与动态功能数据(电生理学)联系起来。直到最近,用于蛋白质定点荧光标记的硫醇反应性化学物质限制了该方法的范围,因为所有可及的半胱氨酸,包括内源性半胱氨酸都将被标记。因此需要构建不含内源性半胱氨酸的蛋白质。标记也限于可从细胞外获得的位点侧。随着使用荧光非天然氨基酸 (fUAA),使用正交tRNA和tRNA合成酶对2特异性并入小荧光探针以响应终止密码子抑制而改变。 VCF改进仅需要DNA注射(tRNA /合成酶对)的两步注射程序,然后进行RNA / fUAA共注射。现在,标记细胞内和掩埋的位点是可能的,VCF的使用已经显着扩大。因此,VCF技术对于研究广泛的蛋白质变得有吸引力,更重要的是允许研究许多细胞溶质调节机制。
引言
具有各种化学和物理性质的200多种非天然氨基酸被遗传并入大肠杆菌 ,酵母和哺乳动物细胞中的蛋白质3 。非天然氨基酸通过正交工程化的tRNA /合成酶对而与特异性终止密码子相结合。改变蛋白质的遗传学方法为蛋白质结构和功能提供了宝贵的见解。在这里,我们提出使用电压钳夹荧光测量(VCF)与荧光UAA组合的方案。
在VCF中,同时观察位于荧光探针周围的功能数据和结构重排(〜5Å)可使我们以毫秒级分辨率获得动态信息1 。荧光探针在蛋白质定位运动时改变其淬灭状态。只有1-2埃的运动足以导致荧光的显着变化强度4 。鉴定目标蛋白质感兴趣位点后,通过点突变突变位点。经典地,残基已被突变为半胱氨酸,而现在,引入琥珀终止密码子(TAG)用于遗传fUAA并入。然后蛋白质被体外转录。
虽然其他表达系统( 例如哺乳动物细胞)可以使用5,6,7 , 非洲爪蟾卵母细胞因结构功能研究而较大,因为其尺寸较大,导致操作更容易,荧光强度更高(更多的荧光团),因此,信噪比。此外, 非洲爪蟾卵母细胞具有低内源性蛋白质的背景2,8,动物杆上的黑色色素沉着抵抗来自t的背景荧光他的细胞质手术切除非洲爪蟾卵母细胞,将编码fUAA特异性的正交tRNA / tRNA-合成酶对的DNA注射到卵母细胞核中。在孵育6-24小时后,将蛋白质RNA与fUAA共注射到卵母细胞的细胞溶质中,随后孵育2-3天。为了防止对fUAA(光漂白)的任何损坏,包括Anap在内的程序必须在红光下进行,以避免荧光团激发。
在切开的卵母细胞电压钳设置上研究卵母细胞,其安装在直立荧光显微镜上,电流和荧光变化同时记录9,10 。或者,可以使用双电极电压钳1或膜片钳构造11 。荧光被具有低RMS噪声的适当波长激发使用连接到具有高放大率的放大器的光电二极管记录的发射。
在电压钳荧光测定中使用荧光非天然氨基酸(fUAAs)有几个优点。一个是进入膜蛋白质的胞质侧;许多调节过程位于这里( 例如, Ca 2+ - 或核苷酸结合位点,电压门控离子通道的快速和闭态失活,开孔,模块耦合)。所有这些过程现在都可用于荧光标记。
另一个优点是探针的小尺寸导致蛋白质的较少干扰。到目前为止,fUAAs的两个正交tRNA / tRNA合成酶对已被设计为12,13 ,其中3-(6-乙酰基萘-2-基氨基)-2-氨基丙酸(Anap)是唯一已被用于非洲爪蟾卵母细胞的fUAA 如图2所示 ,"xref"> 8。 Anap是一种环境敏感的荧光团,分子量为272.3 g / mol,仅略高于色氨酸12 ( 图1A,1B )。由于其小的尺寸,与通过接头连接的常规荧光团(通常大于500g / mol)相比,荧光团可能引入较少的空间效应。此外,在Anap的情况下,荧光团比与半胱氨酸相连的位置更靠近蛋白质主链,因此,Anap正在探测更局部的重排。最后,在UAA-VCF中不再需要在常规VCF中去除内源性半胱氨酸以确保位点特异性标记,因此(i)使蛋白质(几乎)为其天然状态,(ii)允许VCF应用研究更广泛的蛋白质,其功能可能被半胱氨酸取代改变。

图1 :Anap和荧光光谱。 ( A )Anap的化学结构。 ( B )1nM Anap的归一化吸收光谱和发射光谱,证明Anap荧光对溶剂疏水性的敏感性。通过在350nm激发获得发射光谱。 请点击此处查看此图的较大版本。
使用荧光UAA的缺点是如果氨基酰化tRNA的量稀少,则异源蛋白质群体可能由终止密码子读取,翻译重新启动,C-末端截短的蛋白质或内源氨基酰化的串扰引起。在不存在fUAA和tRNA / tRNA合成酶对的情况下,应始终检查这种泄漏表达。我们处理了转交的问题国际重新启动以及如何规避N末端插入位置以前14 。然而,当fUAA,tRNA和tRNA合成酶以饱和量存在时,仅存在泄漏表达的可能性较低。
fUAA-VCF与常规VCF的关键程序差异在于注射和处理卵母细胞;注射编码tRNA和tRNA合成酶(pAnap)的DNA之后,引入Anap,其与蛋白质mRNA共注射,或者作为乙酰氧基甲基(AM)酯加入到孵育溶液中。
研究方案
青蛙操作按照加拿大指南进行,并经蒙特利尔大学道德委员会(CDEA,协议#15-042)的批准。
1. fUAA公司的mRNA准备
- 选择蛋白质感兴趣的位置,预期会发生构象变化。选择该区域中的氨基酸代替fUAA。
注意:位置的选择是基于预期的结构重新排列。如果存在高分辨率结构和预期运动的假设,则应该放置该化学物质的环境将会改变;这可能是介电常数(疏水性与亲水性环境)的变化,或者更可能是由另一种氨基酸猝灭。最好的猝灭剂是色氨酸。应该在一个状态(范德瓦尔半径的重叠)与猝灭剂接触,并将其与另一个。如果不存在高分辨率结构或模型,则必须扫描感兴趣的区域。在任一情况下,建议选择几个附近的位置以增加获得表达和荧光信号的概率。为了最小化蛋白质成熟和/或功能期间的空间效应,可以选择取代大和芳香族氨基酸(Phe,Trp,Tyr)。然而,作者经历了扫描fUAA插入的感兴趣区域,而不管取代的氨基酸如何,更有效率。 - 在选定的位点使用定点突变15插入琥珀终止密码子(TAG)。确保感兴趣的蛋白质不以琥珀终止密码子(TAG)结束。如果是这样,突变为不同的(赭色或蛋白石终止密码子)。扩增,分离和序列DNA。通过体外转录16获得蛋白质mRNA,并将mRNA存储在20℃或80℃。
- 手术从非洲爪蟾青蛙手术获得V或VI卵母细胞,如前所述17胶原酶脱胶。
- 根据批准的动物方案(本文为:3-氨基苯甲酸乙酯),用合适的麻醉剂麻醉青蛙。当他们不能轻蔑地对脚尖做出反应(失去撤回反射)时,则将其适当地麻醉以进行手术。
- 立即从麻醉溶液中取出青蛙,用清水彻底冲洗皮肤。这种冲洗将通过从皮肤表面去除未吸收的化学物质来防止动物进入更深层次的麻醉。
- 从一侧手术切除卵巢节点,并使用两个镊子仔细打开节点。孵育并搅拌含有1%(w / v)胶原酶的"标准卵母细胞溶液"(SOS)中的卵母细胞20-30分钟去流产用SOS溶液洗三次。
- 分别选择大型和健康的卵母细胞,并在注射前至少4小时将其添加到补充有抗生素(100U / mL青霉素,100μg/ mL链霉素,10mg / 100mL卡那霉素)和5%马血清的Barth溶液中至少4小时。
注意:经2-4次延迟的2 - 4次手术之后,通过与3-氨基苯甲酸乙酯长时间(> 1小时)孵育非洲爪蟾进行安乐死。
- 对于DNA的核注射,准备长而薄的注射针头以能够到达细胞核并避免损伤卵母细胞。用注射嘴填充油并将其安装在纳米注射器上。
- 在立体显微镜下安装纳米注射器,并使用镊子打破尖端的末端。排出油,直到没有气泡陷入尖端的末端。
- 将1μL0.1μg/#181;将L pAnap在含有NaOH(1%的1N NaOH)的无核酸酶的水上,在立体镜下在一片石蜡膜上,并用DNA填充注射尖端。
- 将40个卵母细胞转移到含有补充有抗生素的Barth溶液的网状包被的注射皿中。
注意:为了制作网眼注射皿,切出适当尺寸的800μm尼龙网填充聚苯乙烯培养皿。将氯仿加入中心,然后将网格放在顶部。将网格平直,直到塑胶套。 - 由于卵母细胞核位于动物(黑暗)极点,将注射针瞄准动物杆的中心并使其尖端到达动物半球的中心附近(或与RNA注射相比2-3倍的深度) )。将每个卵母细胞中的pAnap注入9.2 nL。薄的尖端和小的注射体积可能导致不规则的注射或阻塞的尖端。偶尔检查注射是否注入空气。
注意:DNA是否适当地注入细胞核是不确定的。因此,期望10-40%的卵母细胞不表达tRNA /合成酶对。参见讨论进一步阐述。 - 在2℃的Barth溶液中孵育卵母细胞,补充抗生素和5%马血清(HS)在18℃下孵育6-24小时,以使得能够稳定表达Anap特异性tRNA和tRNA合成酶。
注意:DNA孵育时间可以在注射RNA前几天持续,但不会增加表达。 - 准备用于RNA注射的纳米注射器(与步骤2.2相同,但注射头不需要像DNA注射那样薄)。此时只能在红灯下工作,以防止Anap的漂白。
- 将1μL1 mM Anap与1μL1-2μg/μLmRNA直接混合在一块石蜡膜上,并用混合溶液填充注射头。在植物的膜下方的Impale(亮)极,并在每个注射apAap注射的卵母细胞中注射46nL。
注意:所需的mRNA浓度取决于感兴趣的蛋白质。 - 孵育保存在盒子中的卵母细胞或包裹在铝箔中,在Barth溶液中补充抗生素和5%马血清,18℃下2-3天。每天与新鲜的Barth溶液进行交换,并去除死卵卵母细胞以避免污染。
- 如前所述安装切开的卵母细胞电压钳设备18 。
- 将电生理学记录系统安装在立式荧光显微镜上,将记录室安装在滑块上,允许在标准立体镜之间移动它以放置卵母细胞和显微镜进行荧光测量( 图2c )。
注意:切开的卵母细胞的几何形状不适合使用正常的tra在操作过程中记录光照明。因此,使用带有红色滤光片的"鹅颈式"卤素灯从顶部向侧面照亮。可以去除显微镜的冷凝器,使空间降低电生理室的阶段。 - 将光电二极管检测系统连接到荧光显微镜的C-mount出口端口( 图2a )。将光电流读数连接到数字信号处理器(DSP,模拟/数字 - 数字/模拟转换器)中的第二个输入通道。
- 使用100 W,12 V卤素灯作为荧光激发的光源。
注意:或者,可以使用Hg-燃烧器,但必须降低强度,以防止在记录期间太快的光漂白。只有当各个LED在激发范围内显示出显着的强度( 例如, Anap的〜350 nm)时才推荐使用LED照明。大多数白光LED不能进入紫外光谱。 - 插件在激励光源和显微镜之间设置电控快门,并将其控制(通常为TTL脉冲)连接到DSP的数字输出。在记录软件中记录TTL脉冲(参见制造商说明书),使快门在录制开始之前〜100 ms开始。这样,打开过程中的任何振动都不会影响记录。时间取决于快门的速度和振动。在记录结束前结束脉冲5ms, 如图4所示 。这样,也记录了总荧光的值。
- 在过滤器立方体塔架中插入适当的过滤器立方体(激发过滤器,分色镜和发射过滤器)。对于Anap,使用Ex:377/50 nm带通,二色409 nm长通和Em:470/40 nm带通。
- 按照前面所述的可视化开放式卵母细胞电压钳的准备步骤18 (琼脂桥制备,安装卵母细胞,皂角苷透化)。然而,在红灯下始终工作,以避免在记录之前漂白荧光团。放置卵母细胞时,请确保动物杆朝上。动物极膜下的色素沉着屏蔽来自细胞溶质的自发荧光,因此降低背景荧光。
- 将腔室滑动到显微镜并使用4X物镜进行对焦。
- 用电压感应V1电极(3 M KCl)冲击卵母细胞,切换到40X浸水(NA 0.8 - 0.9)的目标。专注于面向上的动物杆。
- 关掉红灯。通过转动过滤器立方体塔架和连接到光电二极管的光学出口端口,选择正确的过滤器立方体。打开halogen灯处于最高强度,并且快速打开快门2-5秒以读取源自卵母细胞的背景荧光强度。通过描述的设置,Anap的值应该在50-200 pA左右。
- 打开夹子,将浴室/护罩开关翻转到活动状态,并转动I台灯上的旋钮将膜电位(V1 - V2)调整到指令电位。
- 在录音软件中选择保持电位,步进协议,脉冲数量和长度等。记录电压依赖电流和Anap荧光强度。
- 要同时监测同一蛋白中的两个位置,将细胞外和可及的氨基酸突变为半胱氨酸,并除去其他半胱氨酸,以确保用硫醇化学特异性标记。
- 执行步骤2.1-2.5。
- 在VCF记录之前,将5μMTMR-马来酰亚胺的卵母细胞在标记溶液中孵育15分钟(或其他染料与Anap相比具有非重叠光谱)。
- 用标记溶液洗涤卵母细胞三次以除去多余的染料。
- 执行步骤4.1-4.6。
- 在过滤器立方体塔架中插入适当的TMR滤波器(激发滤光片,分色镜和发射滤光片)。转动过滤塔,切换到TMR过滤器立方体。
- 读取TMR的背景荧光,如步骤4.4中Anap所述。
注意:由于膜上的非特异性标记,硫醇化学的标记导致高背景荧光。因此,TMR背景荧光可能使放大器饱和(> 2,000 pA)。在这种情况下,不要降低光强度,而是通过向光电二极管添加偏移电流来简单地减去背景荧光。在市售系统中,使用检测器系统上的"采样保持"功能。注意实验室j中的背景荧光值(使用10X中性密度滤光片)ournal,因为这个值不会被记录(饱和)。 - 按照步骤4.6同时记录电压相关电流和TMR荧光强度。
2.卵母细胞的制备和注射
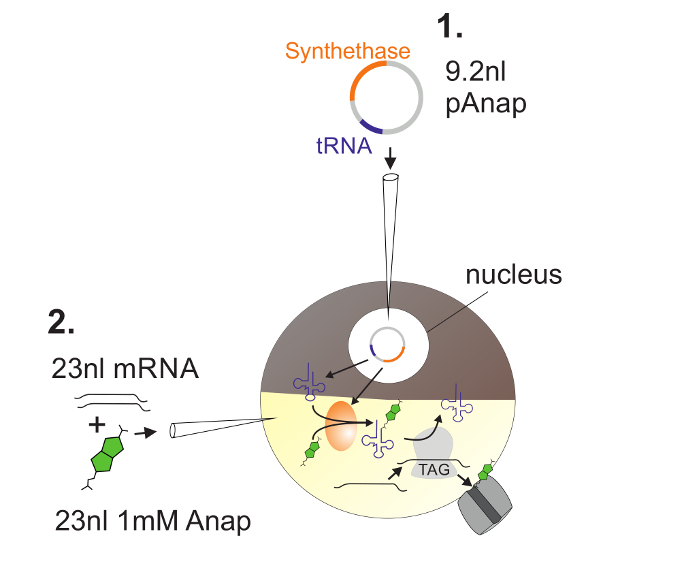
图 2 :DNA和RNA注射入 非洲爪蟾 卵母细胞的插图。
首先,将pAnap注射入非洲爪蟾卵母细胞核( 1 )。 6-24h后,将Anap和通道RNA共同植入植物极( 2 )。通过由pAnap编码的氨酰-tRNA合成酶,Anap将与携带琥珀终止反密码子的tRNA正交氨基酰化。这样,氨基酰化的Anap-tRNA被通道中插入的琥珀终止密码子的核糖体识别1 RNA,导致终止密码子的抑制和Anap的插入。 请点击此处查看此图的较大版本。
3. VCF设置

图3 / strong> :VCF设置。 ( A )VCF设置的侧视图,显示显微镜内的光路。过滤器立方体包含激发滤光片,分色镜和发射滤光片。 ( B )选择的卵母细胞室尺寸为上室半径(1)为3.4厘米,底室长度为2厘米为5.5厘米,底室宽度为3厘米,中间室宽度为4厘米为4厘米。 ( C )VCF设置的前视图。左侧的第一个眼睛是将卵母细胞安装在切开的电压钳夹室中并用于透化。然后,在右侧的第二次眼镜下,在显微镜下滑动腔室。这里,使用4X物镜将V1电极插入卵母细胞,并使用浸水式40X物镜记录荧光。 请点击此处查看此图的较大版本。
双色VCF
结果
图4显示了在存在pAnap和Anap的情况下,从表达快速灭活快速灭活的卵母细胞(IR),L382stop-W434F的卵母细胞获得的VCF记录的实例。 W434F突变阻断离子钾电流,这使得可以测量瞬态门控电荷位移(门电流)。门极电流(上限曲线)和Anap荧光强度变化(下限轨迹)同时记录去极化表明Anap成功纳入了位置L382。在这里,Anap位于在激活期间移动的四个S4跨膜螺旋中的每?...
讨论
与tRNA合成酶连续转录的tRNA 的体内氨基酰化使得可以获得荧光测量的高表达水平。为了有效的fUAA并入,pAnap正确注入细胞核是至关重要的。由于核的确切位置的不确定性,预计10-40%的DNA注射会失败,导致不表达(或泄漏表达)卵母细胞。因此,在没有Anap和pAnap的情况下检查表达是重要的,以识别泄漏通道情况下的当前表型和幅度。这样,通过适当的DNA注射(高表达,Anap dF,全长当前表型?...
披露声明
作者没有什么可以披露的。
致谢
pAnap是Peter Schultz博士(斯克里普斯研究所)的礼物。这项工作由加拿大卫生研究所MOP-102689和MOP-136894(加拿大RB)和加拿大创新基金会950-225005资助。
材料
| Name | Company | Catalog Number | Comments |
| Solutions | |||
| Barth's solution | |||
| NaCl | Sigma-Aldrich | S7653 | 90 mM |
| KCl | Fisher Scientific | BP366-500 | 3 mM |
| MgSO4 | Sigma-Aldrich | M-9397 | 0.82 mM |
| CaCl2 | Sigma-Aldrich | C-7902 | 0.41 mM |
| Ca(NO3)2 | Sigma-Aldrich | C-1396 | 0.33 mM |
| HEPES | Sigma-Aldrich | H4034 | 5 mM |
| NaOH hydrate | BDH | BDH7225-4 | pH 7.6 |
| Penicilin | Invitrogen | 15140122 | 100 U/mL |
| Streptomycin | Invitrogen | 15140122 | 100 µg/mL |
| Kanamycin | Invitrogen | 15160054 | 10 mg/100mL |
| Horse Serum (HS) | Invitrogen | 16050122 | 5% |
| SOS Standard Oocyte Solution | |||
| NaCl | Sigma-Aldrich | 746398 | 102 mM |
| KCl | Sigma-Aldrich | 746436 | 3 mM |
| MgCl2 | Sigma-Aldrich | M9272 | 1 mM |
| HEPES | Sigma-Aldrich | H4034 | 5 mM |
| External recording solution | |||
| N-methyl-D-glucamine (NMDG) | Alfa Aesar | L14282 | 115 mM |
| HEPES | Sigma-Aldrich | H4034 | 10 mM |
| Calcium hydroxide | Sigma-Aldrich | 239232 | 2 mM |
| MES hydrate | Sigma-Aldrich | 258105 | pH 7.2 |
| Internal recording solution | |||
| N-methyl-D-glucamine (NMDG) | Alfa Aesar | L14282 | 115 mM |
| HEPES | Sigma-Aldrich | H4034 | 10 mM |
| Ethylenediamine Tetraacetic Acid (EDTA) | Fisher Scientific | E478-500 | 2 mM |
| MES hydrate | Sigma-Aldrich | 258105 | pH 7.2 |
| Labeling solution | |||
| KOH | Fisher Scientific | P250-1 | 115 mM |
| HEPES | Sigma-Aldrich | H4034 | 10 mM |
| Calcium hydroxide | Sigma-Aldrich | 239232 | 2 mM |
| MES hydrate | Sigma-Aldrich | 258105 | pH 7.2 |
| TMR stock solution | |||
| Tetramethylrhodamine-5-maleimide (TMR) | Molcular Probes by Life Technologies | T6027 | 5 mM in DMSO |
| Anap stock solution | |||
| Anap | ABZENA (TCRS) | Custom synthesis TCRS-170 | 1 mM in nuclease-free water and 1% NaOH 1 N |
| Name | Company | Catalog Number | Comments |
| Material/Equipment | |||
| pAnap | Addgene | 48696 | |
| High Performance Oocyte Clamp | Dagan Corporation | CA-1B | |
| Gpatch Acquisition software | Department of Anesthesiology, University of California, Los Angeles | ||
| Analysis software | Department of Anesthesiology, University of California, Los Angeles | ||
| Recording Chamber | Custom machined | ||
| Photo diode detection system | Dagan Corporation | PhotoMax-200/PIN | |
| Electrical shutter driver | UNIBLITZ | VCM-D1 |
参考文献
- Mannuzzu, L. M., Moronne, M. M., Isacoff, E. Y. Direct physical measure of conformational rearrangement underlying potassium channel gating. Science. 271 (5246), 213-216 (1996).
- Kalstrup, T., Blunck, R. Dynamics of internal pore opening in K(V) channels probed by a fluorescent unnatural amino acid. Proc Natl Acad Sci U S A. 110 (20), 8272-8277 (2013).
- Xiao, H., Schultz, P. G. At the Interface of Chemical and Biological Synthesis: An Expanded Genetic Code. Cold Spring Harb Perspect Biol. 8 (9), (2016).
- Blunck, R. Chapter 9. Handbook of Ion Channels. , 113-133 (2015).
- Chatterjee, A., Guo, J., Lee, H. S., Schultz, P. G. A genetically encoded fluorescent probe in mammalian cells. J Am Chem Soc. 135 (34), 12540-12543 (2013).
- DeBerg, H. A., Brzovic, P. S., Flynn, G. E., Zagotta, W. N., Stoll, S. Structure and Energetics of Allosteric Regulation of HCN2 Ion Channels by Cyclic Nucleotides. J Biol Chem. 291 (1), 371-381 (2016).
- Shen, B., et al. Genetically encoding unnatural amino acids in neural stem cells and optically reporting voltage-sensitive domain changes in differentiated neurons. Stem Cells. 29 (8), 1231-1240 (2011).
- Aman, T. K., Gordon, S. E., Zagotta, W. N. Regulation of CNGA1 Channel Gating by Interactions with the Membrane. J Biol Chem. 291 (19), 9939-9947 (2016).
- Haddad, G. A., Blunck, R. Mode shift of the voltage sensors in Shaker K+ channels is caused by energetic coupling to the pore domain. J Gen Physiol. 137 (5), 455-472 (2011).
- Batulan, Z., Haddad, G. A., Blunck, R. An intersubunit interaction between S4-S5 linker and S6 is responsible for the slow off-gating component in Shaker K+ channels. J Biol Chem. 285 (18), 14005-14019 (2010).
- Kusch, J., et al. How subunits cooperate in cAMP-induced activation of homotetrameric HCN2 channels. Nat Chem Biol. 8 (2), 162-169 (2012).
- Lee, H. S., Guo, J., Lemke, E. A., Dimla, R. D., Schultz, P. G. Genetic incorporation of a small, environmentally sensitive, fluorescent probe into proteins in Saccharomyces cerevisiae. J Am Chem Soc. 131 (36), 12921-12923 (2009).
- Summerer, D., et al. A genetically encoded fluorescent amino acid. Proc Natl Acad Sci U S A. 103 (26), 9785-9789 (2006).
- Kalstrup, T., Blunck, R. Reinitiation at non-canonical start codons leads to leak expression when incorporating unnatural amino acids. Sci Rep. 5, 11866 (2015).
- Liu, H., Naismith, J. H. An efficient one-step site-directed deletion, insertion, single and multiple-site plasmid mutagenesis protocol. BMC Biotechnol. 8, 91 (2008).
- Beckert, B., Masquida, B. Synthesis of RNA by in vitro transcription. Methods Mol Biol. 703, 29-41 (2011).
- Goldin, A. L. Maintenance of Xenopus laevis and oocyte injection. Methods Enzymol. 207, 266-279 (1992).
- Rudokas, M. W., Varga, Z., Schubert, A. R., Asaro, A. B., Silva, J. R. The Xenopus oocyte cut-open vaseline gap voltage-clamp technique with fluorometry. J Vis Exp. (85), (2014).
- Zhao, J., Blunck, R. The isolated voltage sensing domain of the Shaker potassium channel forms a voltage-gated cation channel. Elife. 5, (2016).
- Posson, D. J., Ge, P., Miller, C., Bezanilla, F., Selvin, P. R. Small vertical movement of a K+ channel voltage sensor measured with luminescence energy transfer. Nature. 436 (7052), 848-851 (2005).
- Chanda, B., Asamoah, O. K., Blunck, R., Roux, B., Bezanilla, F. Gating charge displacement in voltage-gated ion channels involves limited transmembrane movement. Nature. 436 (7052), 852-856 (2005).
- Taraska, J. W., Puljung, M. C., Zagotta, W. N. Short-distance probes for protein backbone structure based on energy transfer between bimane and transition metal ions. Proc Natl Acad Sci U S A. 106 (38), 16227-16232 (2009).
- Baker, B. J., et al. Genetically encoded fluorescent sensors of membrane potential. Brain Cell Biol. 36 (1-4), 53-67 (2008).
- Miranda, P., et al. State-dependent FRET reports calcium- and voltage-dependent gating-ring motions in BK channels. Proc Natl Acad Sci U S A. , (2013).
- Sisido, M., Ninomiya, K., Ohtsuki, T., Hohsaka, T. Four-base codon/anticodon strategy and non-enzymatic aminoacylation for protein engineering with non-natural amino acids. Methods. 36 (3), 270-278 (2005).
- Hohsaka, T., Ashizuka, Y., Murakami, H., Sisido, M. Five-base codons for incorporation of nonnatural amino acids into proteins. Nucleic Acids Res. 29 (17), 3646-3651 (2001).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。