需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
利用彗星试验评价体外DNA 损伤
摘要
彗星试验是一种有效的方法来检测 dna 损伤, 包括单和双 dna 断裂。我们描述了碱性和中性彗星检测肿瘤细胞 DNA 损伤, 以评估化疗的治疗效果。
摘要
dna 损伤是每个细胞在其生命周期中常见的现象, 它被定义为基因组 dna 化学结构的改变。癌症治疗, 如无线电和化疗, 引入了大量额外的 DNA 损伤, 导致细胞周期阻滞和细胞凋亡, 以限制癌症的进展。在实验性癌症治疗过程中, DNA 损伤的定量评估是证明毒性剂有效性的关键步骤。在这项研究中, 我们的重点是单细胞电泳分析, 也称为彗星检测, 它可以量化单和双 DNA 断裂体外。彗星试验是一种高效、易于执行的 DNA 损伤定量方法, 具有较低的时间/预算要求和较高的重现性。在这里, 我们通过评价 olaparib/莫联合疗法对 U251 胶质瘤细胞的毒性作用, 强调了彗星试验在临床前研究中的效用。
引言
彗星试验是第一次开发的 Ostling 和艾里克·乔纳森在1984年通过证明 DNA 片段从细胞核迁移在一个中性条件下1。该技术后来由辛格的et al., 表明碱性条件极大地提高了检测的特异性和重现性的分析2。从那时起, 中性彗星检测主要用于检测双的 dna 断裂, 而碱性彗星检测对较小数量的 dna 损伤更敏感, 包括单和双链 dna 断裂、碱不稳定部位、dna dna 或dna 蛋白交联, 与不完全切除修复相关的 dna 单断裂3,4。这两种方法都允许对片段 dna 进行可视化, 并提供一种直接的方式来定量评估 dna 损伤。彗星试验被认为是一种敏感的方法,体外和体内遗传毒理学研究, 并适用于不同的研究领域, 如早期药物候选选择, 环境监测, 人类生物,以及 DNA 损伤和修复的基础研究5。
该法的原理是, 在电场下, 碎片 dna 从核体 (也称为 "彗星头") 迁移出来, 并在琼脂糖凝胶 (也称为 "彗星尾") 中形成 dna 染色。通过对这种单细胞电泳所形成的 "彗星" 进行分析, 可以量化 DNA 损伤的程度。尾力矩的计算可以进一步帮助比较不同实验组的 DNA 损伤。与传统的 DNA 损伤检测方法相比, 彗星试验具有直接、灵敏、价廉、相对简单等特点。
放疗和化疗是癌症治疗的共同策略, 在染色体上生成单链和双链 DNA 断裂6。最近的进展, DNA 修复抑制剂允许更有效的毒性效果的联合化疗, 因此, 可能会减少系统性副作用, 如贫血, 感染和骨髓抑制7,8. 在这项研究中, 我们展示了一个聚 (ADP-核糖) 聚合酶 (PARP) 抑制剂, olaparib (法律厅)9的研究。PARP 是一种丰富的核蛋白, 通过形成聚 (ADP-核糖) 聚合物10, 负责 DNA 基础切除修复。莫是一种口服的烷剂, 广泛应用于胶质瘤患者的治疗。通过使用彗星试验来量化 dna 损伤, 我们证明了 olaparib 与莫的结合, 深刻地增强了胶质瘤细胞的 dna 损伤, 这表明 olaparib/莫联合疗法是治疗胶质瘤的有效策略,与单独的莫11相比。
研究方案
1. 准备试剂
- 1x pbs
- 用900毫升 dH 2 O 稀释100毫升 10x pbs, 并使用 ph 计将 ph 值调整为7.4。储存在室温下.
- 溶解解决方案 (LS)
- 准备2.5 米氯化钠, 100 毫米二钠 EDTA, 10 毫米三碱, 200 毫米氢氧化钠在900毫升 dH 2 O; 它通常需要大约20分钟, 使混合物完全溶解。使用 ph 计将 ph 值调整到10。加入1% 的十二烷基酸和1% 的海卫 X-100, 并将最终的体积调整到1000毫升。冷却到4和 #176; C 至少在使用前30分钟.
- 碱性电泳溶液 (AES), ph 和 #62; 13
- 准备200毫米氢氧化钠和1毫米二钠 EDTA 在800毫升 dH 2 o 调整 ph 值并确保它是 ph 值和 #62; 13。将最后的音量调整到1000毫升。使用前要新鲜, 冷却到4和 #176; C 在使用前至少30分钟.
- 中性电泳 sollution (其他)
- 准备1000毫升中性电泳缓冲液, 将 100 mm 的三基色和 300 mm 的醋酸钠混合到1000毫升 dH 2 o. 将 pH 值调整为 9.0, 用冰醋酸。冷却到4和 #176; C 至少在使用前30分钟.
- DNA 沉淀解决方案 (DPS)
- 准备10毫升7.5 米醋酸铵库存。在50毫升的 DNA 沉淀溶液中, 用43.3 毫升95% 乙醇混合6.7 毫升7.5 米的醋酸铵。储存在室温下.
- 着色解决方案
- 添加1和 #181; 10,000x 和 #160; 绿色荧光核酸染色 ( 例如 , SYBR 绿色) 在30毫升三 edta 缓冲液 (10 毫米三盐酸, 1 毫米二钠盐, pH 7.4) 和存储在4和 #176; c. 保护光.
- 1% 低熔点琼脂糖
- 在微波炉中熔化1% 低熔点琼脂糖 (1 克在100毫升 dH
- 2 O)。每15-20 秒旋转一次琼脂糖, 以确保琼脂糖完全熔化。将琼脂糖放入37和 #176; C 水浴至少20分钟后使用.
- 预热吸管提示
- 将 P200 吸管尖端的窄端由 3 mm 切断, 并在37° C 和 #176 前加热; 移琼脂糖.
2。准备彗星幻灯片
- 幻灯片涂层
- 将1% 琼脂糖 (1 克在100毫升 dH 2 O) 放入微波炉中 2-3 分钟, 或直到琼脂糖完全融化。将玻璃显微镜滑入琼脂糖, 并使用无皮棉擦拭擦拭幻灯片的一侧.
- 在平坦的表面上放置幻灯片, 使其在50和 #176 时风干或发热; C 用于更快的干燥; 干燥后应形成透明的琼脂糖膜。在使用前将涂布的幻灯片放在37和 #176; C.
- 单细胞悬浮液的制备
- 培养和治疗胶质瘤细胞
- 培养 DMEM F-12 培养基中的 U251 镁细胞, 辅以 10% FBS、100 U/毫升青霉素、10和 #181; 37/毫升链霉素176;C 与 5% CO 2 .
- 用1毫升胰蛋白酶对3分钟的细胞进行消化, 用 DMEM 火腿 F-12 培养基和 FBS 中和胰蛋白酶。收集在15毫升管, 旋转在 300 x g 为4分钟, 吸气介质, 并暂停在 1x PBS 的 2 x 10 5 细胞/毫升的细胞.
注意: 细胞样本应在开始化验前立即准备好, 所有样品应在黑暗或暗淡的环境中处理, 以防止光的 DNA 损伤. - 将电池悬浮液与1% 熔融的低熔点琼脂糖 (37 和 #176; C) 在一个比例 1:10 (v/v), 轻轻地混合移上下, 并立即吸管30和 #181; 升到幻灯片上。使用吸管尖端的一侧来传播琼脂糖/细胞混合物, 以确保薄层的形成.
- 将幻灯片平放在4和 #176; C 在黑暗中为10分钟. 将凝胶时间提高到30分钟提高了样品在高湿度环境中的粘附性.
- 将幻灯片浸泡在4和 #176 中; C 是在黑暗中1小时到一夜.
- 培养和治疗胶质瘤细胞
3。单细胞电泳
- 进行碱性 (步骤 3.2) 或中性 (步骤 3.3) 彗星化验
- 为碱性彗星化验
- 轻轻地从 LS 中删除幻灯片, 耗尽多余的缓冲区, 并在 AES 中轻轻浸入1小时4#176 C 允许 DNA 的松弛把幻灯片放在黑暗中.
- 在电泳盘中添加 pre-chilled AES, 不要超过幻灯片的0.5 厘米 (这取决于电泳单位的大小), 将幻灯片放在里面并盖上盖子。将电源电压设置为1伏/厘米 (电极之间的长度), 并在4和 #176 上运行30分钟; C.
- 从幻灯片中排出多余的电泳溶液。在 dH 2 中轻轻地浸泡两次幻灯片, 在室温下为5分钟.
- 在室温下用70% 乙醇轻轻浸泡5分钟的幻灯片。继续执行步骤 4.
用于中性彗星试验的 -
- 轻轻地从 LS 中取出幻灯片, 排出多余的缓冲区, 然后在4和 #176 中轻轻地浸入其他30分钟; c. 在黑暗中保持幻灯片.
- 在电泳盘中添加 pre-chilled 中性电泳缓冲器, 不超过0.5 厘米以上的幻灯片 (这取决于电泳单位的大小), 将幻灯片放在里面并盖上盖子。将电源电压设置为1伏/厘米 (电极之间的长度), 并在4和 #176 上运行45分钟; C.
- 从幻灯片中排出多余的缓冲区。在室温下, 轻轻地浸泡幻灯片和 #160; 在 DPS 中为30分钟.
- 在室温下, 将70% 乙醇中的幻灯片轻轻地浸泡30分钟。继续执行步骤 4.
4。着色彗星幻灯片
- 37 和 #176 中的干燥幻灯片; C 为 10-15 分钟在黑暗中.
- 将 50-100 和 #181; 绿色荧光核酸染色溶液放在每个干燥的琼脂糖上, 在室温下为15分钟.
- 在 dH 中简要地冲洗幻灯片 2 O 和完全干燥在37和 #176; C 在黑暗中。进行图像采集和分析.
5。图像采集与分析
注意: DNA 断裂的可视化和量化是基于荧光显微镜和彗星检测软件 (参见 资料表 ) 12.
- 将幻灯片放在显微镜上, 并使用幻灯片夹。确保琼脂糖凝胶面向物镜。使用带有10x 物镜的荧光显微镜随机捕捉彩色彗星幻灯片中的图像。避开气泡周围的边缘和区域.
- 确保每颗彗星的尾部都是水平分布的。彗星头应该从左边和尾巴起源于右边.
- 将每张图片保存为具有明亮的 DNA 污点和深色背景的二进制 TIF 格式。使用和 #34 将图像加载到软件; 选择要分析和 #34 的文件; 按钮, 它位于工具栏的左侧。应该会出现一个图像视图窗口 ( 图 1 ).
- 在屏幕上绘制一个测量框, 并根据单元格的彗星调整其大小。单击 #34; 调整和 #34; 按钮根据图像设置头部、彗星和尾部的阈值, 然后单击并 #34; 启动测量和 #34; 按钮 ( 图 1 ).
- 使用框架选择一个单元格, 并通过在和 #34 上单击鼠标来激活测量; 测定彗星和 #34; 按钮; 强度图像显示在#34;p rofiles 和 #34; 具有所选测量参数的窗口。通过单击 "#34"; 存储结果和 #34; 按钮 ( 图 1 ), 可以保存结果.
注: 软件计算的参数包括彗星尾部的长度, 尾 DNA 的百分比, 尾矩 (TM) 和橄榄尾力矩 (OTM)。尾矩按如下公式计算:

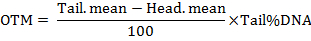
- 分析至少50单元格每次治疗.
结果
本协议描述了彗星试验执行和数据分析的 step-by 步骤工作流 (图 1)。从碱性和中性彗星的结果表明, 阿霉素治疗的 U251 细胞 (1 µM, 20 h) 的彗星尾巴较长, 并有较高的 dna 强度, 表明由于化疗的碎片 dna 大量积累 (图 2)。
对碱性或中性彗星的定量分析表明, 在阿霉素治疗后彗星尾形成明显?...
讨论
彗星试验是一个有效的工具, 以测量单和双 DNA 断裂的细胞水平。该法在遗传毒性和生物13的研究中被广泛应用为 "黄金标准", 从基底病变、DNA 通道、药物开发和碱敏部位不等。在本研究中, 我们分别显示了两个不同的 step-by 步骤的碱性和中性彗星分析的协议。结合单细胞电泳, 荧光显微镜和图像解释, 无论是碱性和中性的彗星检测提供定量的方法来评估的程度 DNA 损伤在体外
披露声明
作者没有什么可透露的。
致谢
这项研究得到了 NIH、癌症研究所和 CCR 的校内研究项目的支持。所有的作者都获得了来自 NIH, 癌症研究所和 CCR 的内部研究补助金。
材料
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| 10x PBS(Ca++, Mg++ free) | TEKnova | P0196 | |
| NaCl | Sigma | S5886 | |
| EDTA | TEKnova | E0308 | |
| Trizma base | Sigma | T1503 | |
| NaOH | Sigma | 72068 | |
| Sodium lauryl sarcosinate | Sigma | L7414 | |
| Triton X-100 | Sigma | 93443 | |
| Sodium acetate | Sigma | 32318 | |
| Glacial acetic acid | Sigma | 695092 | |
| Ammonium acetate | Sigma | A1542 | |
| SYBR Green | Invitrogen | S33102 | |
| Low melting point agarose | Invitrogen | 16520 | |
| Agarose | Invitrogen | 16500 | |
| 95% ethanol | WARNER-GRAHAM | #64-17-5 | |
| Trypsin | GIBICO | 25300-054 | |
| Name | Company | Catalog Number | Comments |
| Consumables | |||
| Glass tissue slides | ELECTRON MICROSCOPY SCIENCES | 63422-11 | |
| Kimwipes | KIMberly-Clark | ||
| 1.5 mL Microcentrifuge Tubes | DENVILLE | ||
| Pipette Tips | SHARP | ||
| Name | Company | Catalog Number | Comments |
| Equipments | |||
| Microwave | Avanti | ||
| Waterbath | PRECISION | ||
| Horizontal electrophoresis chamber | TREVIGEN | Cometassay ES II | |
| Power supply | Bio-Rad | ||
| Incubator | Quincy Lab | Model 12-140E | |
| Fluorescent microscope | Zeiss | LSM700 | |
| Micropipettor | Eppendorf |
参考文献
- Ostling, O., Johanson, K. J. Microelectrophoretic study of radiation-induced DNA damages in individual mammalian cells. Biochem Biophys Res Commun. 123 (1), 291-298 (1984).
- Singh, N. P., McCoy, M. T., Tice, R. R., Schneider, E. L. A simple technique for quantitation of low levels of DNA damage in individual cells. Exp Cell Res. 175 (1), 184-191 (1988).
- Tice, R. R., et al. Single cell gel/comet assay: guidelines for in vitro and in vivo genetic toxicology testing. Environ Mol Mutagen. 35 (3), 206-221 (2000).
- Shah, A. J., Lakkad, B. C., Rao, M. V. Genotoxicity in lead treated human lymphocytes evaluated by micronucleus and comet assays. Indian J Exp Biol. 54 (8), 502-508 (2016).
- Azqueta, A., Collins, A. R. The essential comet assay: a comprehensive guide to measuring DNA damage and repair. Arch Toxicol. 87 (6), 949-968 (2013).
- Goldstein, M., Kastan, M. B. The DNA damage response: implications for tumor responses to radiation and chemotherapy. Annu Rev Med. 66, 129-143 (2015).
- Gavande, N. S., et al. DNA repair targeted therapy: The past or future of cancer treatment?. Pharmacol Ther. 160, 65-83 (2016).
- Torgovnick, A., Schumacher, B. DNA repair mechanisms in cancer development and therapy. Front Genet. 6, 157 (2015).
- Weston, V. J., et al. The PARP inhibitor olaparib induces significant killing of ATM-deficient lymphoid tumor cells in vitro and in vivo. Blood. 116 (22), 4578-4587 (2010).
- Brown, J. S., O'Carrigan, B., Jackson, S. P., Yap, T. A. Targeting DNA Repair in Cancer: Beyond PARP Inhibitors. Cancer Discov. 7 (1), 20-37 (2017).
- Lu, Y., et al. Chemosensitivity of IDH1-Mutated Gliomas Due to an Impairment in PARP1-Mediated DNA Repair. Cancer Res. 77 (7), 1709-1718 (2017).
- Konca, K., et al. A cross-platform public domain PC image-analysis program for the comet assay. Mutat Res. 534 (1-2), 15-20 (2003).
- Valverde, M., Rojas, E. Environmental and occupational biomonitoring using the Comet assay. Mutat Res. 681 (1), 93-109 (2009).
- Collins, A. R. The comet assay for DNA damage and repair: principles, applications, and limitations. Mol Biotechnol. 26 (3), 249-261 (2004).
- Karbaschi, M., Cooke, M. S. Novel method for the high-throughput processing of slides for the comet assay. Sci Rep. 4, 7200 (2014).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。