需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
活蛙 (爪蟾) 胚胎单细胞代谢的微探针毛细管电泳质谱
摘要
我们描述的步骤, 使快速的原位取样的一个小部分, 高精度和最小的入侵使用 capillary-based 微取样, 以促进化学表征的代谢活动的快照活胚胎使用 custom-built 单细胞毛细管电泳和质谱仪平台。
摘要
单个细胞中小分子的量化为更好地理解胚胎发育的基本过程提供了新的潜能。为了能够直接在活胚胎中进行单细胞调查, 需要新的分析方法, 特别是那些敏感、有选择性、定量、健壮和可伸缩的不同细胞大小。在这里, 我们提出了一个协议, 使原位分析在自由发展的南非爪蛙 (爪蟾) 的单个细胞中的新陈代谢, 这是细胞和发育生物学的一个强有力的模型。这种方法使用毛细管微探针从胚胎中的单个识别细胞中抽出一个定义的部分, 使相邻的细胞保持完好, 以便随后进行分析。通过对高分辨率串联质谱仪进行微尺度毛细管电泳 (CE-ESI) 界面的耦合, 分析采集的细胞含量。这种方法可扩展到不同的细胞大小和兼容的复杂的三维结构发育的胚胎。作为一个例子, 我们证明了微探针单细胞 CE-ESI-ms 能够阐明的新陈代谢细胞异质性, 展开作为祖细胞在胚胎发育过程中产生的后代。除了细胞和发育生物学之外, 这里所描述的单细胞分析协议也可以用于其他细胞大小、细胞类型或动物模型。
引言
对胚胎发育的全面了解需要描述在发展中生物体的每个细胞中所展现的所有分子变化。当下一代测序与分子放大, 使深度测量单细胞转录1在开发的系统2,3, 相当少是已知的套件较小的分子在单个胚胎细胞中产生, 包括蛋白质, 特别是代谢物 (分子质量和 #60; ~ 1500 Da)。对内在和外在事件的快速和动态的反应, 代谢充当细胞的分子状态的一个强有力的描述符。因此, 单细胞代谢提高了在早期胚胎中追踪细胞异质性的空间和时间发展的潜能, 并为功能研究寻找新的分子。然而, 如果没有分子放大器可供这些分子, 检测代谢要求特殊灵敏度使用质谱 (MS), 这是技术的选择代谢物分析。
单细胞 MS 是一种技术的集合, 具有足够的灵敏度来测量单个细胞中的代谢物 (见评论4,5,6,7,8,9 ,10,11,12,13,14,15。细胞的重现性取样和代谢物的有效提取对单细胞中代谢物的成功检测至关重要。从爪胚胎中识别出的细胞的全细胞解剖, 使小分子和多肽的表征成为了16。其他方法采用 micropipettes, 以样本的个别活细胞, 其次是检测使用电喷雾电离 (ESI) MS。例如, 代谢物被测量在植物或哺乳动物细胞由单细胞录影 MS17, 压力探针18, 选拔探针19和流体力量显微镜20, 在其他技术之中21, 22、23、24。此外, 化学分离之前, 电离到单细胞 MS 工作流有效地简化了代谢, 从而减轻了潜在的干扰, 在离子生成前检测。重要的是, 分离还提供了复合特定的信息, 以协助分子鉴定。毛细管电泳 (CE) 已被用来检测代谢物在单一解剖25,26或 microsampled27神经元, 捕获小分子表型之间的差异。最近, 我们调整了 CE 到 ESI 串联 MS, 以使从爪蟾16,28的早期胚胎中解剖的单个细胞中的数百种代谢物的微量检测。这些研究揭示了胚胎细胞在发育早期的惊人代谢差异, 并导致发现代谢物与以前未知的发育影响16。
在这里, 我们提供了一个协议, 使检测的代谢物的单细胞直接在活脊椎动物胚胎使用微探针单细胞 CE-ESI-MS29,30。所选择的模型生物体是8至32细胞的x 蟾胚, 虽然这种方法也适用于后期的发展阶段和其他类型的模型生物体。该协议使用高分辨率成像系统指导下的多轴平移控制的锐化毛细管, 以在形态复杂的发育中的胚胎中, 在原位检测到 10 nL 部分的识别细胞。这种微探针可伸缩到较小的细胞, 并在几秒钟内操作, 这是足够快跟踪胚胎细胞谱系。在提取的极性或 apolar 小分子, 如代谢物和多肽, 从收集样本的〜4-5 µL 萃取液, 10 nL 的结果提取是分析在一个 custom-built CE 平台联用的一个 ESI 质谱仪。CE-ESI-MS 平台的构建和运行建立在其他地方所描述的协议之上。31,32轴 CE-ESI 接口是按照其他地方的描述构造的。31此平台在锥喷喷涂系统中保持, 以实现跟踪级别的灵敏度, 并具有在4-5 日志顺序动态范围内进行量化的能力 (相对28、29、30或绝对16)。CE-ESI-MS 平台提供了一个 60 amol 下限检测与8% 相对标准偏差 (RSD) 在定量超过 10 nM 到1µM 为小分子16, 这是足以表征内源代谢物在十。蟾单元格。Microprobed 细胞在胚胎发育的过程中继续分裂30, 允许对细胞代谢进行时空解析分析。事实上, 单细胞 CE-ESI-ms 可以用来发现细胞之间的新陈代谢差异占据了背腹16,29, 动物植物性的16, 和左右28发展轴以及细胞在x 蟾30中, 从一个共同的祖细胞中形成神经组织命中注定的背世系。除了在x 蟾胚30的不同发育阶段对单个胚胎细胞之间的代谢差异进行查询外, 我们预计这里描述的协议适用于广泛的生物分子和单细胞 microsampled 从胚胎发育的不同阶段, 以及其他类型的细胞和模型生物体。此外, 微探针可以用于 microsampling, 而不同的平台与微小的样品相兼容, 可以用于生物分子的分离和/或表征。
研究方案
所有与维护和处理爪蟾有关的协议都是由乔治·华盛顿大学机构动物保育和使用委员会批准的 (IACUC 号。A311)。
1. 取样器具、介质、溶剂和取样盘的制备
- 准备1x 斯坦伯格的溶液 (SS) 溶解在超纯水 (〜18.2 ω. cm 在25° c) 按以下顺序和在被表明的集中跟随一个标准协议33: 氯化钠 (58.2 毫米), 氯化钾 (0.67 毫米), 硝酸钙 (0.34 毫米), 硫酸镁 (0.83 毫米), 三盐酸盐 (4.19 毫米), 和三基 (0.66 毫米)。使 0.5x ss 由二倍, 和 0.1x ss 由十倍稀释, 1x ss 使用超纯水。
- 先制作2% 琼脂糖, 然后在120° c 的温度为20分钟, 准备好取样盘。还有液体时, 用溶液将 60 mm 培养皿的底部涂上。一旦琼脂糖凝胶冷却和固化, 火焰的六英寸的巴斯德吸管结束, 直到它形成一个球, 轻轻触摸的加热结束, 以压印5-10 口井, 〜1毫米深, 进入琼脂糖。
注: 这些井用于在取样过程中固定胚胎。 - 制备代谢物萃取溶剂。将溶剂的理化性质 (如: 如、极性和 pH) 调整到研究中感兴趣的分子类别。
注: 例如, 我们使用40% 甲醇和40% 乙腈在 LC-MS 级水中作为一种发现的方法, 主要针对极性代谢物, 和一些 apolar 代谢物和多肽28。 - 使头发循环使用干净的头发和巴斯德吸管的描述, 在其他地方的33 , 轻轻移动的胚胎在培养皿的最小摄动。
- 制造锥形尖端 micropipettes, 如图 1a所示。
- 首先, 将硼硅酸盐毛细管 (1000/500 µm 外/内直径) 置于燃烧棕色型毛细管拉拔器中, 其设置如下: 热 = 355;拉 = 65, 速度 = 80;时间 = 150。
- 接下来, 脱离的尖端的拉微使用一对罚款锋利钳获得一个毛细管尖端外径的〜20µm. 在显微镜下执行这一步骤, 以帮助精确和重现性。
注: 带小尖端的毛细血管在粘性细胞质的吸入过程中容易堵塞。虽然毛细血管具有较大的尖端, 当然有助于避免堵塞和吸入更多的细胞的内容, 大口径毛细血管可能会提出挑战, 在取样的较小的细胞, 并可能损害细胞为后续取样。仔细调整的压力和时间的渴望可能部分缓解这些挑战。我们发现 micropipettes 与〜20µm 外径理想的工作在这里提出。
2. Microsampling 单细胞和代谢物的提取
- 获得胚胎 (受精卵) 通过促性腺激素诱导的自然交配成人爪蟾或通过体外受精, 如在其他地方的协议33,34。
注意: 自然交配确保胚胎发育阶段交错, 而由体外受精获得的胚胎在供应方面更可靠。然而,体外受精需要牺牲成年雄性青蛙。 - 新制备2% 半胱氨酸 dejellying 溶液, 将4克半胱氨酸溶解到200毫升的超纯水中, 滴用 10 N 氢氧化钠溶液将溶液调节到 pH 值8。
- 取出在胚胎周围的果冻大衣, 因为他们开始切割成2细胞阶段如下: 让胚胎在 dejellying 溶液中休息2分钟, 然后轻轻地将它们旋转2分钟, 防止胚胎附着在收集表面菜.
- 轻轻地将碟中的内容倒入一个干净的烧杯中, 然后从烧杯中迅速醒酒 dejellying 溶液。立即用 0.1x SS 盖住鸡蛋, 冲洗剩下的 dejellying 溶液, 轻轻地旋转, 然后醒酒溶液。重复这一步骤四次彻底清洗的胚胎。
注意: 将胚胎的暴露范围限制在 dejellying 溶液中4分钟, 以确保存活。清除果冻大衣的全面协议在其他地方都有33。 - 在培养皿中将 dejellied 胚胎移植到 1x SS 中。为了减少板块内的拥挤, 每 100 mm 的100胚胎放置一盘33。
注: 含有胚胎的盘子可以在14-18 ° c 之间储存, 以减缓发育, 并在同一个父母交错发育阶段获得胚胎。关于生长和发育的温度依赖性的进一步指南发布在 Xenbase 和其他地方33,35,36,37。 - 排序裂解胚胎在2细胞阶段成一个单独的菜, 其中定型色素沉着自信地标志着背腹轴, 参照建立的细胞命运图38,39,40。
- 确定正确的裂解胚胎, 确保第一个沟沟, 其中 demarks 正中平面, 两黑暗 (腹) 和轻度 (背) 色素动物极点, 使两个一半是镜像图像41。
- 在多轴微 (手动或遥控) 上安装一个捏造的微。将微连接到微量。
- 用塑料转移吸管将5的8细胞胚胎吸入, 并将其转移到含有 0.5x SS 的取样盘中。
注: 在胚胎中根据色素沉着和位置识别细胞, 参照细胞命运图38,39,40。
注: 例如, 我们通过将-30 psi 的3脉冲应用到毛细管上, 例行地从细胞中吸出 10-15 nL 体积。这整个步骤持续〜5秒的愿望30。
- 紧密关闭小瓶, 以防止蒸发, 并将瓶子放回4° c 冰桶, 直到取样完成。将使用的微丢弃到锐器容器中, 以防止针刺危险。
注: 要确定吸气细胞的含量, 将吸入的油注入矿物油, 在那里获得球形的形状。这个球体的直径可以用显微镜来测量。计算吸气量: V = 4/3 π r3, 其中v是音量, r是球体的半径。
3. CE-电喷雾-质谱测量
- CE-ESI-质谱的标准和解决方案的制定
- 制备由1% 甲酸在 LC 级水中组成的背景电解质。
- 准备鞘液中含有50% 甲醇在 LC-MS 级水和0.1% 甲酸。
- 在鞘液中制备 50 nM 乙酰胆碱溶液, 每日评估 CE-电喷雾-质谱系统的性能。
- 准备150毫米氯化钠溶液作为质量校准标准为低m/z范围在正面离子方式。质量 (m/z) 的精度和 #60; 建议使用 10 ppm。按照质谱仪供应商的指示执行这一步骤。
注: 另一种方法是, 使用已知的m/z值的其他标准可以对质谱仪进行质量校准。
- CE-ESI 平台的构建
- 构建一个能快速垂直平移的 CE 注塑平台, 可进行一级 microvial 和样品装载。有关平台的构造和操作, 请参阅参考31中的详细信息。
- 按如下方式组装 CE-ESI 接口 (图 1c)。将电喷雾金属发射器 (130/260 µm 内/外直径和〜 35 mm 长度) 装入一个3端口的 T 型联合。通过电喷雾发射器将分离的 CE 毛细管 (40/105 µm 内/外直径和〜100厘米) 馈入, 使其能够凸出 ~ 40-100 µm 超出发射器尖端的距离。在显微镜下工作以帮助准确。
- 将鞘液毛细管 (75/360 µm 内/外径和 ~ 100 厘米长) 连接到剩余的端口以提供电喷雾溶液。使用适当的袖子和手指-拧紧连接, 以无泄漏操作的 CE-ESI 接口。有关此接口的程序集和 trouble-shooting 的详细信息, 请参阅以前的协议31,32 。
注: 毛细管尺寸影响信噪比 (S/N) 和间隔时间。例如, 窄孔和短毛细管使用更高的分离电压来促进快速分离42,43。此外, 根据研究中感兴趣的分子类型, 分离毛细管可以被涂敷以最小化/避免不需要的分子-毛细管壁相互作用44。
- 将鞘液毛细管 (75/360 µm 内/外径和 ~ 100 厘米长) 连接到剩余的端口以提供电喷雾溶液。使用适当的袖子和手指-拧紧连接, 以无泄漏操作的 CE-ESI 接口。有关此接口的程序集和 trouble-shooting 的详细信息, 请参阅以前的协议31,32 。
- 使用一个板持有人, 安装 CE-ESI 接口到一个三轴的翻译阶段和位置的电喷雾发射器尖端〜2毫米从质谱仪孔 (图 1c)。
- 为了清洁界面的成分, 通过电喷雾发射器在1µL/分钟和通过 CE 分离毛细管的方法, 通过提供喷雾液来冲洗。使用注射器泵以稳定的速度喂溶剂。
- 通过将注射器连接到毛细管入口端, 在每次测量前冲洗 CE 分离毛细管。使用足够大的注射器, 以尽量减少灌装和启动溶剂供应线。
注: 实验通常使用1毫升气密注射器提供电喷雾鞘溶剂和500µL 注射器冲洗分离毛细管。
- CE-电喷雾-MS 平台的验证及代谢产物的测定
注: 本步骤的目的是在分析单细胞提取物之前, 每天确认 CE-ESI-MS 仪器的分析灵敏度。
- 在冲洗分离毛细管5分钟后, 将其入口转移到位于不锈钢小瓶中的该溶液中。
- 将电喷雾发射器尖端〜2毫米从质谱仪孔和精细调整这个距离使用一个翻译阶段, 以产生在稳定的锥射流制度, 而监测喷雾使用显微镜 (参见参考文献31,45)。监测总离子电流 (TIC) 的稳定度为30-45 分钟, 以确保稳定运行。
- 应用〜 20 kV 的小瓶通过逐步扩大潜力超过〜十五年代, 通常产生〜7.5 µA 电流横跨分离毛细管使用1% 甲酸作为。在每次测量前, 确保系统稳定, 通过监测 TIC 剖面为〜5-10 分钟, 然后逐步降低分离 (CE) 的潜力, 以 0 V (地面)。
注意: 为了半自动这个过程, 我们使用一个自定义的软件来远程控制 CE 高压电源31。如果 ce-esi-ms 平台不稳定, 请首先在 esi 模式下测试 ce-esi-ms 平台, 然后在其他地方31中推荐的 ce-esi 操作模式下, 仔细评估不稳定源。简单地, 在仅 ESI 模式下测试平台, 关闭 CE 高电压, 并在锥射流喷涂系统中监视 TIC 30 分钟。如有必要, 请按以下方法处理错误: (i) 检查连接是否有泄漏;(ii) 用水、异丙醇、水和甲醇清洁电喷雾发射器;(三) de-gas 溶剂;(iv) 在再次测试前, 将发射器冲洗25分钟。如果 ce-esi 平台在仅 esi 模式下稳定, 但在 ce 分离过程中变得不稳定, 请检查系统的焦耳加热和/或电解: (i) 冲洗分离毛细管与25分钟, 重复实验;(ii) 使用较低的分离电位维持线性欧姆响应 (即, 线性 CE 电流与分离电压曲线);(iii) 检查 CE 毛细管的潜在损害, 如裂纹, 必要时更换毛细管。 - 分析〜 6 nL 的样品如下:
- 吸管1µL 的乙酰胆碱标准溶液进入注射瓶。
- 将分离的毛细管从一小瓶转移到注射瓶中。
- 在1s 中提起 CE 注塑阶段15厘米。
- 举行的阶段上升为六十年代水力注入〜 6 nL 样品入分离毛细管。
- 随后, 将舞台转换回起始水平 (与毛细管插座一致)。
- 轻轻地将毛细管进气道端移到其上。
- 紧接着, 加大了 CE 电压, 开始电泳分离。
- 启动 MS 数据获取。
注: 系统性能可采用任何化学标准进行表征。单细胞 CE-ESI-ms 为乙酰胆碱、蛋氨酸和组氨酸16提供了低 10 nM (60 amol) 检测的下限。注入的音量 (V 注), 插入到 nL 中的毛细管中, 取决于在注射过程中的高度差 (H, 厘米), 密度 (ρ, g cm-3) 和粘度 (η, 千克 m-1s-1), 长度 (L, m) 和 CE 的半径 (r, µm)毛细管和注入持续时间 (t 注, s).此关系由以下公式表示, 其中g (m s-2) 是重力加速度:
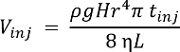
- 一旦检测到标准, 停止数据采集, 将分离电压逐步降低到0伏 (地面), 然后从孔口中检索到2厘米的发射器。在分析细胞提取物之前, 先将分离毛细管冲洗5分钟。
- 通过重复步骤3.3 来测量 10 nL 的单细胞提取物. 1-3. 3.5 使用九十年代向水力注入样品。
注意: 数据处理的目的是识别和量化单个细胞间的化合物。单细胞 CE-ESI-ms 协议生成的窄 electropherographic 峰值的典型基宽为几秒钟。通过执行数据分析 semi-manually, 可以使用以下步骤找到分子特征 (唯一的m/z值和唯一的迁移时间)。在图 2a中, 有代表性的分离显示出选定的代谢产物。
- 批量校准原始数据文件后数据获取。
注意: 我们使用来自甲酸钠簇的信号, 在从样品中分离出丰富的钠离子时产生, 它们在细胞中是原生存在的, 或者从胚胎培养基中提取。这一步骤的目的是通过确保高质量 (m/z) 的准确度, 优选和 #60; 5 mDa, 或 #60; 10 ppm, 在m/z 50-1, 000 之间, 以增强代谢产物的识别。在这里, post 数据获取校准使得有可能定期获得质量精度和 #60; 1 mDa, 或 #60; 2 ppm, 用于m/z 50-500。 - 使用处理脚本, 在检测到的质量范围内搜索分子特征。平均质量谱横跨每个峰值, 以确定准确质量, 并记下其相应的迁移时间。对于在m/z 50-500 范围内的质量代谢产物的鉴定, 使用 500 mDa 台阶窗口来监测具有 S/N 和 #62 的分子特征; 3。
- 手动或自动将峰值区域 under-the-curve 为每个分子特征。所产生的面积值被用作代谢物丰度的量度。
- 确定感兴趣的分子特征以高度信心如下 (参见图
- 首先, 将分子特征的精确质量与一个代谢物数据库 (例如, Metlin46和 HMDB47) 进行比较, 以 10 ppm 的精确度来获得假定的质量匹配列表。
- 接下来, 通过比较它们从细胞提取物中获得的串联质谱和代谢数据库中可用的数据, 或者用相应的化学标准测量的串联质谱来评估这些质量匹配。
- 最后, 通过将细胞提取物中记录的分子特征的迁移时间与相同的 CE-电喷雾-质谱仪分析的相关化学标准进行比较, 验证这些任务的有效性。
注意: 为了提高实验的吞吐量, 我们通常只识别在实验条件或细胞类型上有统计学差异的分子特征。有代表性的标识如图 2所示。为了识别具有高质量准确度的代谢产物, 我们建议每天对质谱仪进行外部校准, 在每次测量时使用内部标准进行 real-time 校准, 并/或外部质量校准每个测量建议使用文件数据获取 (例如, 用于甲酸钠簇)。
注意: 在我们的实验中, 使用了47的在线软件平台来执行后续数据分析的所有步骤, 包括以下步骤: i) 在每个 sample-set 的至少50% 中过滤分子特征 (例如, 单元格类型);二) 数据的正常化;统计 (例如, t测试) 和多元数据分析, 如主成分分析 (PCA) 和层次聚类分析 (HCA)。我们使用p和 #60; 0.05 (学生的t测试) 标记统计意义和折叠-变化≥1.5 注意生物学意义。
结果
我们最近使用微探针单细胞 CE-ESI-MS 来表征在自由发展的爪蟾胚29,30中单个识别细胞的代谢物。微探针使快速 (〜5秒/细胞),原位的愿望 10 nL 从一个单独的细胞, 同一细胞的多重愿望, 或几个不同的细胞在相同或后期发展阶段的活胚 (图 1b)。吸气细胞的含量是用合适的溶剂提取的, 然后用 custom-buil...
讨论
微探针 CE-电喷雾-MS 能够直接表征的代谢物在单细胞的活, 自由发展的胚胎。该方法的核心是两个技术子组件, 即原位毛细管 microsampling 和高敏 CE-电喷雾-MS. 与全细胞解剖相比, 毛细管 microsampling 具有快速操作的优点 (几秒钟与5分钟比较/细胞通过解剖), 与复杂的三维胚胎形态学的相容性, 以及在发育后期形成的更小的细胞的延展性。与解剖不同的是, 微探针取样会使胚胎中的其他细胞保持完好...
披露声明
作者没有什么可透露的。
致谢
这项工作得到了国家卫生研究院的资助 GM114854 (大法官) 和 CA211635 (大法官), 阿诺德和梅布尔贝克曼基金会贝克曼青年研究员补助金 (大法官), 杜邦青年教授奖 (大法官), 美国大众协会光谱研究奖 (对大法官) 和波斯菊俱乐部基础奖学金 (对 R.M.O. 和 E.P.P.)。本出版物所表达的意见和结论完全是作者的观点, 不一定代表资金来源的官方观点。
材料
| Name | Company | Catalog Number | Comments |
| Reagents for Embryo Culture Media | |||
| Potasium chloride | Fisher Scientific | BP 366-1 | |
| Magnesium sulfate | Fisher Scientific | M 65-3 | |
| Calcium nitrate | Sigma Aldrich | C1396 | |
| Cysteine | MP Biomedicals | 101444 | |
| Trizma hydrochloride | Sigma Aldrich | T3253 | |
| Trizma base | Sigma Aldrich | T1503 | |
| Sodium chloride | Fisher Scientific | 5641-212 | |
| Name | Company | Catalog Number | Comments |
| Metabolite Extraction Solvents | |||
| Acetonitrile (LC-MS-grade) | Fisher Scientific | A955 | |
| Methanol (LC-MS-grade) | Fisher Scientific | A456 | |
| Water (LC-MS-grade) | Fisher Scientific | W6 | |
| Name | Company | Catalog Number | Comments |
| Solvents and Standards for CE-ESI-MS | |||
| Formic acid (LC-MS-grade) | Fisher Scientific | A11710X1-AMP | |
| Methanol (LC-MS-grade) | Fisher Scientific | A456-4 | |
| Water (LC-MS-grade) | Fisher Scientific | W6 | |
| Sodium chloride | Fisher Scientific | 5641-212 | |
| Acetylcholine chloride | Acros Organics | 159170050 | |
| Name | Company | Catalog Number | Comments |
| Microprobe Fabrication Setup | |||
| Micropippette puller | Sutter Instrument Co. | P-1000 | |
| Borosilicate capillaries | Sutter Instrument Co. | B100-50-10 | |
| Fine sharp forceps: Dumont #5, Biologie/Dumoxel | Fine Science Tools (USA) Inc | 11252-30 | Corrosion resitant and autoclavable. |
| Name | Company | Catalog Number | Comments |
| Microprobe Sampling Setup | |||
| Micromanipulator | Eppendorf, Hauppauge, NY | TransferMan 4r | |
| Stereomicroscope | Nikon | SMZ18 | Should be vibrationally isolated. |
| Illuminator e.g. Goosenecks | Nikon | C-FLED2 | |
| Microinjector | Warner Instrument, Handem, CT | PLI-100A | |
| Transfer pipettes (Plastic, disposable) | Fisher Scientific | 13-711-7M | |
| Petri dish 60 mm and 80 mm | Fisher Scientific | S08184 | |
| Glass Pasteur Pipets ( Borosilicate, disposable) | Fisher Scientific | 13-678-20A | |
| Centrifuge | Thermo Scientific | Sorvall Legend X1R | |
| Name | Company | Catalog Number | Comments |
| CE-ESI-MS Setup | |||
| High voltage power supply | Spellman | CZE1000R | The HVPS may be controlled remotely using a low-voltage program generated by a personal computer. Caution: High voltage presents electrical shock hazard; all connective parts must be grounded or carefully shielded to prevent users from accidental exposure. |
| Syringe pumps (2) | Harvard Apparatus | 704506 | |
| Stereomicroscope | Amscope | SM-3BZZ | Stereomicroscope capable of 4.5× magnification, equipped with an illuminator to monitor the spraying mode of the CE-ESI interface. |
| XYZ translation stage | Thorlabs | PT3 | |
| XYZ translation stage | Custom-built | This platform is capable of loading nanoliter-amounts of sample into the separation capillary via hydrodynamic injection and supplying the BGE for CE. Both interfaces described in this work were able to inject 6–10 nL of sample within 1 min into a 1 m separation capillary | |
| Stainless steel sample vials | Custom-built | ||
| Stainless steel BGE vial | Custom-built | ||
| Fused silica capillary (40 µm/105 µm ID/OD; 100 cm) | Polymicro technologies | TSP040105 | |
| Fused silica capillary (75 µm/360 µm ID/OD; 100 cm) | Polymicro technologies | TSP075375 | |
| Stainless steel emitter with blunt tips (130/260 µm ID/OD) | Hamilton Co. | 21031A | For better performance, laser-cleave and fine-polish the emitter tip. |
| Syringes (gas-tight): 500 - 1000 µL | Hamilton Co. | 1750TTL | |
| Digital multimeter | Fluke | Fluke 117 | |
| High-resolution Mass Spectrometer | Bruker Daltonics | Maxis Impact HD | High-resolution tandem mass spectrometer equipped with an atmospheric-pressure interface configured for ESI |
| Tunning mixture for mass spectrometer calibration | Agilent technologies | ESI-L G1969-85000 | |
| Data Analysis ver. 4.3 software | Bruker Daltonics | ||
| Name | Company | Catalog Number | Comments |
| Ancillary Equipment | |||
| Vacuum concentrator capable of operation at 4–10°C | Labconco | 7310022 | |
| Analytical microbalance (XSE105DU) | Fisher Scientific | 01911005 | |
| Freezer (-20 °C) | Fisher Scientific | 97-926-1 | |
| Freezer (-80 °C) | Fisher Scientific | 88300ASP | |
| Refrigerated Incubator | Fisher Scientific | 11475126 | |
| Vortex-mixer | Benchmark | BS-VM-1000 |
参考文献
- Tang, F. C., Lao, K. Q., Surani, M. A. Development and applications of single-cell transcriptome analysis. Nat. Methods. 8 (4), S6-S11 (2011).
- Veselovska, L., et al. Deep sequencing and de novo assembly of the mouse oocyte transcriptome define the contribution of transcription to the DNA methylation landscape. Genome Biol. 16 (209), (2015).
- Tran, D. A., Bai, A. Y., Singh, P., Wu, X. W., Szabo, P. E. Characterization of the imprinting signature of mouse embryo fibroblasts by RNA deep sequencing. Nucleic Acids Res. 42 (3), 1772-1783 (2014).
- Wang, D. J., Bodovitz, S. Single cell analysis: the new frontier in 'omics'. Trends Biotechnol. 28 (6), 281-290 (2010).
- Svatos, A. Single-cell metabolomics comes of age: new developments in mass spectrometry profiling and imaging. Anal. Chem. 83 (13), 5037-5044 (2011).
- Rubakhin, S. S., Romanova, E. V., Nemes, P., Sweedler, J. V. Profiling metabolites and peptides in single cells. Nat. Methods. 8 (4), S20-S29 (2011).
- Bodenmiller, B., et al. Multiplexed mass cytometry profiling of cellular states perturbed by small-molecule regulators. Nat. Biotechnol. 30 (9), 858-889 (2012).
- Rubakhin, S. S., Lanni, E. J., Sweedler, J. V. Progress toward single cell metabolomics. Curr. Opin. Biotechnol. 24 (1), 95-104 (2013).
- Kleparnik, K., Foret, F. Recent advances in the development of single cell analysis: A review. Anal. Chim. Acta. 800, 12-21 (2013).
- Zenobi, R. Single-cell metabolomics: Analytical and biological perspectives. Science. 342 (6163), 1243259 (2013).
- Gholipour, Y., Erra-Balsells, R., Nonami, H. In situ pressure probe sampling and UV-MALDI MS for profiling metabolites in living single cells. Mass Spectrom (Tokyo). 1 (1), A0003 (2012).
- Comi, T. J., Do, T. D., Rubakhin, S. S., Sweedler, J. V. Categorizing cells on the basis of their chemical profiles: progress in single-cell mass spectrometry. J. Am. Chem. Soc. 139 (11), 3920-3929 (2017).
- Lombard-Banek, C., Portero, E. P., Onjiko, R. M., Nemes, P. New-generation mass spectrometry expands the toolbox of cell and developmental biology. Genesis. 55, e23012 (2017).
- Yang, Y. Y., et al. Single-cell analysis by ambient mass spectrometry. Trac-Trends Anal. Chem. 90, 14-26 (2017).
- Lanni, E. J., Rubakhin, S. S., Sweedler, J. V. Mass spectrometry imaging and profiling of single cells. J. Proteomics. 75 (16), 5036-5051 (2012).
- Onjiko, R. M., Moody, S. A., Nemes, P. Single-cell mass spectrometry reveals small molecules that affect cell fates in the 16-cell embryo. Proc. Natl. Acad. Sci. U. S. A. 112 (21), 6545-6550 (2015).
- Mizuno, H., Tsuyama, N., Harada, T., Masujima, T. Live single-cell video-mass spectrometry for cellular and subcellular molecular detection and cell classification. J. Mass Spectrom. 43 (12), 1692-1700 (2008).
- Nakashima, T., et al. Single-cell metabolite profiling of stalk and glandular cells of intact trichomes with internal electrode capillary pressure probe electrospray ionization mass spectrometry. Anal. Chem. 88 (6), 3049-3057 (2016).
- Pan, N., et al. The single-probe: A miniaturized multifunctional device for single cell mass spectrometry analysis. Anal. Chem. 86 (19), 9376-9380 (2014).
- Guillaume-Gentil, O., et al. Single-cell mass spectrometry of metabolites extracted from live cells by fluidic force microscopy. Anal. Chem. 89 (9), 5017-5023 (2017).
- Saha-Shah, A., Green, C. M., Abraham, D. H., Baker, L. A. Segmented flow sampling with push-pull theta pipettes. Analyst. 141 (6), 1958-1965 (2016).
- Hu, J., et al. Synchronized polarization induced electrospray: Comprehensively profiling biomolecules in single cells by combining both positive-ion and negative-ion mass spectra. Anal. Chem. 88 (14), 7245-7251 (2016).
- Zhang, L. W., Vertes, A. Energy charge, redox state, and metabolite turnover in single human hepatocytes revealed by capillary microsampling mass spectrometry. Anal. Chem. 87 (20), 10397-10405 (2015).
- Zhang, L. W., et al. In Situ metabolic analysis of single plant cells by capillary microsampling and electrospray ionization mass spectrometry with ion mobility separation. Analyst. 139 (20), 5079-5085 (2014).
- Lapainis, T., Rubakhin, S. S., Sweedler, J. V. Capillary electrophoresis with electrospray ionization mass spectrometric detection for single-cell metabolomics. Anal. Chem. 81 (14), 5858-5864 (2009).
- Nemes, P., Knolhoff, A. M., Rubakhin, S. S., Sweedler, J. V. Metabolic differentiation of neuronal phenotypes by single-cell capillary electrophoresis electrospray ionization mass spectrometry. Anal. Chem. 83 (17), 6810-6817 (2011).
- Aerts, J. T., et al. Patch clamp electrophysiology and capillary electrophoresis mass spectrometry metabolomics for single cell characterization. Anal. Chem. 86 (6), 3203-3208 (2014).
- Onjiko, R. M., Morris, S. E., Moody, S. A., Nemes, P. Single-cell mass spectrometry with multi-solvent extraction identifies metabolic differences between left and right blastomeres in the 8-cell frog (Xenopus) embryo. Analyst. 141 (12), 3648-3656 (2016).
- Onjiko, R. M., Plotnick, D. O., Moody, S. A., Nemes, P. Metabolic comparison of dorsal versus ventral cells directly in the live 8-cell frog embryo by microprobe single-cell CE-ESI-MS. Anal. Methods. , (2017).
- Onjiko, R. M., Portero, E. P., Moody, S. A., Nemes, P. In situ microprobe single-cell capillary electrophoresis mass spectrometry: Metabolic reorganization in single differentiating cells in the live vertebrate (Xenopus laevis) embryo. Anal. Chem. 89, 7069-7076 (2017).
- Nemes, P., Rubakhin, S. S., Aerts, J. T., Sweedler, J. V. Qualitative and quantitative metabolomic investigation of single neurons by capillary electrophoresis electrospray ionization mass spectrometry. Nat. Protoc. 8 (4), 783-799 (2013).
- Knolhoff, A. M., Nemes, P., Rubakhin, S. S., Sweedler, J. V., Wevers, R., Lutz, N., Sweedler, J. V. . Methodologies for Metabolomics. , 119-139 (2013).
- Sive, H. L., Grainger, R. M., Harland, R. M. . Early development of Xenopus laevis: a laboratory manual. , (2000).
- Moody, S. A. Cell lineage analysis in Xenopus embryos. Methods Mol Biol. 135, 331-347 (2000).
- Bowes, J. B., et al. Xenbase: a Xenopus biology and genomics resource. Nucleic Acids Res. 36, D761-D767 (2008).
- Karpinka, J. B., et al. Xenbase, the Xenopus model organism database; new virtualized system, data types and genomes. Nucleic Acids Res. 43 (D1), D756-D763 (2015).
- James-Zorn, C., et al. Xenbase: expansion and updates of the Xenopus model organism database. Nucleic Acids Res. 41 (D1), D865-D870 (2013).
- Moody, S. A. Fates of the blastomeres of the 16-cell stage Xenopus embryo. Dev. Biol. 119 (2), 560-578 (1987).
- Moody, S. A. Fates of the blastomeres of the 32-cell-stage Xenopus embryo. Dev. Biol. 122 (2), 300-319 (1987).
- Dale, L., Slack, J. M. W. Fate map for the 32-cell stage of Xenopus laevis. Development. 99 (4), 527-551 (1987).
- Klein, S. L. The first cleavage furrow demarcates the dorsal-ventral axis in Xenopus embryos. Dev. Biol. 120 (1), 299-304 (1987).
- Rollman, C. M., Moini, M. Ultrafast capillary electrophoresis/mass spectrometry of controlled substances with optical isomer separation in about a minute. Rapid Commun. Mass Spectrom. 30 (18), 2070-2076 (2016).
- Moini, M., Martinez, B. Ultrafast capillary electrophoresis/mass spectrometry with adjustable porous tip for a rapid analysis of protein digest in about a minute. Rapid Commun. Mass Spectrom. 28 (3), 305-310 (2014).
- Huhn, C., Ramautar, R., Wuhrer, M., Somsen, G. W. Relevance and use of capillary coatings in capillary electrophoresis-mass spectrometry. Anal. Bioanal. Chem. 396 (1), 297-314 (2010).
- Nemes, P., Marginean, I., Vertes, A. Spraying mode effect on droplet formation and ion chemistry in electrosprays. Anal. Chem. 79 (8), 3105-3116 (2007).
- Zhu, Z. J., et al. Liquid chromatography quadrupole time-of-flight mass spectrometry characterization of metabolites guided by the METLIN database. Nat. Protoc. 8 (3), 451-460 (2013).
- Wishart, D. S., et al. HMDB 3.0 The Human Metabolome Database in 2013. Nucleic Acids Res. 41 (D1), D801-D807 (2013).
- Liu, J. X., Aerts, J. T., Rubakhin, S. S., Zhang, X. X., Sweedler, J. V. Analysis of endogenous nucleotides by single cell capillary electrophoresis-mass spectrometry. Analyst. 139 (22), 5835-5842 (2014).
- Hubrecht, L., Nieuwkoop, P. D., Faber, J. . Normal table of Xenopus laevis (Daudin). A systematical and chronological survey of the development from the fertilized egg till the end of metamorphosis. , (1967).
- Grant, P. A., Herold, M. B., Moody, S. A. Blastomere explants to test for cell fate commitment during embryonic development. J. Vis. Exp. (71), (2013).
- Sellick, C. A., Hansen, R., Stephens, G. M., Goodacre, R., Dickson, A. J. Metabolite extraction from suspension-cultured mammalian cells for global metabolite profiling. Nat. Protoc. 6 (8), 1241-1249 (2011).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。