Method Article
单细胞基因组测序的超高通量微流控平台
摘要
单细胞测序揭示了生物系统中的基因型异质性, 但目前的技术缺乏对群落组成和功能的深入分析所必需的吞吐量。在这里, 我们描述了一个微流控流程的序列 > 5万单细胞基因组从不同的细胞群。
摘要
测序技术经历了从大批量到单细胞分辨率的范式转变, 以响应对生物系统中细胞异质性作用的逐渐理解。然而, 由于处理基因组测序的局限性, 大量人群的单细胞测序受到限制。本文描述了单细胞基因组测序 (SiC 序列) 的方法, 它利用液滴微流体分离、放大和条形码的单细胞基因组。微中的细胞封装允许分离纯化和 tagmentation DNA, 而微流控合并有效地将每个基因组与一个独特的单细胞寡核苷酸条码配对, 允许 > 5万单胞细胞被测序每次运行。测序数据由条形码 demultiplexed, 生成来自单个单元格的读取组。作为单细胞测序的高通量和低偏置方法, SiC 序列将使针对不同细胞群的更广泛的基因组研究得以实现。
引言
基因组作为细胞身份和功能的蓝图, 包含了有机体的编码潜能的整体。在基因组层面上对细胞生物学的理解可以解释异质细胞群中观察到的表型多样性。这种异质性在生物系统中很明显, 对人类健康和疾病有着广泛的影响。例如, 肿瘤细胞中基因复制数的变化与癌症的演变和扩散有关, 如1,2。在细菌感染中, 在一小部分基因组中存在的致病性岛屿可以水平转移, 并导致抗药性细菌的扩散3,4。在单细胞水平上研究基因组的一个主要挑战是可用的 DNA 数量少, 以及需要分析数以千计的细胞来取样基因型的全部多样性。由于这些原因, 实验吞吐量的限制阻碍了单细胞研究的有效性, 使结果偏向于最丰富的细胞。单单元隔离技术, 如流排序5,6, 光学镊子7, 在大容量凝胶8中嵌入, 微流体9能够处理数以百计的细胞进行测序;但是, 这仅代表大多数示例中的一小部分。单细胞基因组测序的方法具有极高的吞吐量, 可以更深入和更完整地分析细胞种群, 从而阐明基因型多样性在这些社区中的作用。
液滴微流体能够在数以百万计的 picoliter 反应器内对细胞和生物试剂进行高通量的操作。到目前为止, microdroplet 技术已经被用来研究不同组织的细胞之间的差异表达模式10,11,12, 深序列长分子13,14 、15, 并对单个单元格16进行染色质免疫沉淀排序 (芯片序列) 分析。事实上, microdroplets 能够进行高通量的、分割的操作, 使它们能够在单细胞基因组学中应用。然而, 这种技术的发展呈现出自己独特的技术挑战。细胞必须裂解, 纯化, 并以最小的偏差放大, 以均匀的样本细胞数量。另外, 与哺乳动物细胞中的 polyadenylated mRNA 记录不同, 基因组中没有可比的分子母题来促进目标核酸的捕获。由于这些原因, 单细胞基因组测序在 microdroplet 平台中难以实现。
在这项工作中, 我们提供了我们以前报告的单细胞微流控方法的详细协议, 能够在单个实验17中对成千上万个细胞的基因组进行排序。使用这种技术, 称为 SiC 序列, 细菌细胞被封装在微米尺度的水凝胶和单独裂解, tagmented, 并合并 microdroplet 含有一个独特的寡核苷酸条码, 这是拼接到细胞的基因组 DNA 通过单重叠扩展聚合酶链反应 (PCR)。水凝胶作为孤立的容器, 其中高分子量的基因组 DNA 被 sterically 包裹, 允许较小的分子, 如洗涤剂和裂解酶, 以获取和纯化 DNA 之前, 条形码18。此协议在几个小时内处理 > 5万个单元格, 导致条形码库准备好进行排序。按照顺序, 读取是根据他们的单单元条码序列 demultiplexed 的, 结果是由数以百万计的读取组成的数据集, 每个都带有一个蜂窝索引。
研究方案
1. 微流控器件制造
- 使用计算机辅助设计 (CAD) 软件准备微流控掩模设计 (如下所规定)。DWG;请参阅辅助文件)。在电路板胶片上有10µm 分辨率的供应商打印这些设计。
注: 对于多层微流控设备, 相应的掩码包含对齐标记。 -
对于每个设备, 按如下方式制造 SU-8 主模具 (图 1A)。
- 将约1毫升的 SU-8 3025 光刻胶倒入晶片中心, 制备3英寸直径的硅片。使用吸力, 在旋转涂布机卡盘上保护晶片。
- 有关每个设备的层厚度和旋转速度的列表, 请参见表 1 。为所有设备, 开始自旋涂层与三十年代在 500 rpm, 跟随三十年代在被表明的速度。
- 将 SU-8-silicon 晶片从自旋涂布机上取出, 并将其软烘烤到135°c 30 分钟. 允许晶片在烘烤后冷却到室温。
- 在准直190兆瓦, 365 nm 紫外线 LED 下, 用适当的微流控膜暴露 SU-8-silicon 晶片3分钟。
- 曝光后, 硬烘烤硅片上的热板设置为135°c 1 分钟。在这个烘烤步骤之后, 让晶片冷却到室温。
- 对于单层微流控设备, 跳到步骤1.2.7。对于多层微流控设备, 对光刻胶的第二层重复步骤 1.2.1 1.2.5 (图 1B)。
- 继第一次硬烘烤的单一层设备 (或第二个硬烘烤的多层设备), 通过浸泡到丙烯乙二醇单甲醚醋酸酯 (PGMEA) 浴30分钟, 开发该晶片。
- 晶片开发后, 使用含有 PGMEA 的喷瓶冲洗晶片。然后用含有异丙醇的喷瓶冲洗晶片, 然后将其放在135摄氏度的热板上, 使其干燥1分钟。
- 将晶片 (从今以后称为主) 放入一个培养皿中, 用于烷。
-
在步骤1.2 中编写的主控形状中, 继续使用由一个。
- 通过将硅基与固化剂结合在一起, 以质量11:1 的比例, 制备出其。用搅拌棒混合硅胶底座和固化剂。
- 将其放入脱气室并应用真空。允许在气泡不再可见 (通常为30分钟) 的情况下, 将其加在一起。
- 仔细地将脱气在母版上倒入, 到最后一层的5毫米厚. 再加一次, 以确保清除任何气泡。
- 脱气后, 在80°c 烘烤, 并在80分钟的主。
- 用剃刀刀片小心地从烤师傅身上切下固化后的板坯。确保所有的切割都在硅片的顶端。
注: 硅晶片上的任何切口都可能导致唇部防止均匀粘合。 - 使用0.75 毫米活检拳打进入口和插座。使用设备的功能端上的包装胶带去除任何灰尘和杂散光。
- 在等离子处理设备之前, 用异丙醇清洗50毫米 x 75 毫米的玻璃滑块, 并烘干。
- 对于等离子处理, 将该板和玻璃滑入等离子焊机中, 其特点是面朝上。使用1毫巴 O2等离子进行等离子处理1分钟. 将该设备与玻璃滑块结合, 将暴露的或正面的一面放在一起。
- 在等离子处理后, 将设备烘烤80摄氏度, 40 分钟。
- 最后, 将玻璃表面处理液注入其中一个入口, 使微流控通道疏水性。确保所有通道完全被溶液淹没, 并对每个 dropmaker 重复注入。烘烤处理后的设备在80°c 10 分钟, 以蒸发过剩的溶剂。
2. 琼脂糖微中细胞的封装
注意: 请参阅图 2A。
- 在1x 三 EDTA (TE) 缓冲器中制备1毫升的3% 瓦特/v 低熔点温度琼脂糖。将琼脂糖溶液保持在90摄氏度的热量块上, 直到注射器加载前立即。
-
准备细胞悬浮。
注: 该协议及其相关的微流控装置已被证实与细菌细胞, 无论是从冷冻库存或新鲜的准备工作。哺乳动物细胞, 根据细胞类型, 可能需要调整微流控通道尺寸, 以适应较大的细胞大小。- 并用重悬1毫升磷酸盐缓冲生理盐水 (PBS) 中的细胞。
- 计算 hemocytometer 中的单元格或按流排序。对于25µm 微, 目标细胞封装率为 1, 10, 在最终浓度为 2.4 x 107细胞/毫升时, 准备1毫升的细胞悬浮。
- 旋转细胞向下 3000 x g 3 分钟, 吸入上清和并用重悬的细胞颗粒在1毫升17% 伏/v 密度梯度介质 (参见材料表) 在 PBS。把它放在冰上直到注射器装上。
- 载入含氟油 (HFE) 的3毫升注射器, 含有2% 瓦特/w perfluoropolyether 聚乙二醇 (PFPE PEG) 表面活性剂, 适合它与27克针, 并将其放入注射器泵。
注: 适合所有注射器与27克针的微流控步骤在本议定书。为了减少意外刺伤的风险, 请将所有针头上的瓶盖保持在泵操作开始之前。 - 将细胞悬浮液和熔融琼脂糖装入1毫升注射器中, 同时配合27口径针头, 并将其放入注射器泵中。
- 保持琼脂糖注射器和泵温暖的小空间加热器, 以防止琼脂糖凝胶在注射器和进口油管。将空间加热器设置为高, 并将其放置, 使加热表面离注射器大约10厘米。确保在注射器上测量的温度约为80摄氏度。
注: 建议使用者在建议的距离内保持加热器, 以减少设备损坏的风险, 包括油管的熔化。 -
使用共流 dropmaking 设备生成25µm 微凝胶体滴。
注: 有关指示试剂入口和出口位置的设备示意图, 请参见图 3A 。- 使用聚乙烯 (PE) 油管将注射器针与微流控装置入口连接。在将管子插入设备之前, 先将泵从线上取出。
- 连接一条油管到出口, 并将自由端放在15毫升收集管。
- 使用以下 (推荐) 流速为 dropmaking: 800 µL/小时为 HFE 2% w/w PFPE PEG;200µL/小时的细胞悬浮在 PBS;和200µL/小时为3% 瓦特/v 琼脂糖。
- dropmaking 后, 将收集管放置在4°c 30 分钟, 以确保琼脂糖的完全凝胶。
3. 打破和洗涤琼脂糖微
- 使用装有20克针的3毫升注射器从收集管中取出较低的油层, 注意不要扰乱琼脂糖滴的顶层。
-
用 perfluorooctanol (PFO) 打破乳液。
- 添加1毫升的10% 伏/五 PFO 在 HFE 的琼脂糖滴。吸管这个解决方案上下1分钟, 彻底涂上乳液。
注意: 对于所有的微凝胶体洗涤步骤, 吸管的解决方案与1000µL 提示。微凝胶体悬浮液在吹打后应出现均匀且无团簇。 - 旋转圆锥管在 2000 x g 1 分钟收集琼脂糖微。PFO/HFE 上清液;微现在没有其表面活性剂层, 将会显得清晰。
- 添加1毫升的10% 伏/五 PFO 在 HFE 的琼脂糖滴。吸管这个解决方案上下1分钟, 彻底涂上乳液。
-
用正己烷的表面活性剂洗涤微。
注意: 己烷是一种挥发性有机溶剂, 在步骤3.3 中的洗涤应在通风罩中进行。- 加入2毫升的1% 伏/五山梨油酸酯非离子表面活性剂 (见材料表) 在己烷到琼脂糖微。吸管上下10x 混合, 确保微凝胶体颗粒完全破碎。
- 旋转管在 1000 x g 1 分钟收集微。吸入上清液去除表面活性剂/己烷溶液。
- 重复表面活性剂/己烷洗涤。
-
清洗水中的微, 去除任何残留的有机溶剂。
- 添加5毫升的春节缓冲 [0.1% 辛基醚非离子洗涤剂 (见材料表), 在 1x TE] 到锥形管。吸管上下10x 混合。
- 旋转圆锥管在 2000 x g 2 分钟收集微。吸入上清液以除去春节缓冲。
- 重复春节洗涤2x。
- 将5毫升 1x TE 缓冲器添加到锥形管中。吸管上下10x 混合。
- 旋转圆锥管在 2000 x g 2 分钟收集微。吸入上清, 取出 TE 缓冲器。
- 重复一下特勤洗。
- 通过将10µL 整除凝胶与1x 核酸染色 (见材料表) 进行染色, 以400X 放大率验证微下的细胞封装。
注: 微将在透明通道下清晰显示, 而细胞 DNA 将在 GFP 通道下荧光 (497/520 纳米激发/发射波长)。透明和荧光通道的叠加将显示微中的单元格, 如图 4A所示。
4. 通过裂解酶裂解琼脂糖中的细胞
- 用800µL 的 TE 缓冲器 (1x) 制备1毫升的裂解酶鸡尾酒, 2 µL 酵母裂解酶 (5 u/µL 库存, 10 u/毫升最终), 30 µL dithiothreitol (1 米股票, 30 毫米最后), 60 毫克溶菌酶 (冻干粉), 15 µL EDTA (0.5 米库存, 7.5 毫米决赛), 2 µL mutanolysin (100 u/µL 股票, 200 U/毫升最后), 2 µL 溶葡萄球菌酶 (10 u/µL 股票, 20 u/毫升最后) 和30µL 氯化钠 (1 M 股票, 30 毫米决赛)。
- 添加额外的 TE, 使体积为1毫升。用涡流混合解决方案。
- 将溶解酶溶液的整个1毫升加入不超过1毫升的洗涤微。混合吹打10x。在一个振动筛中孵育37摄氏度 > 2 小时的混合物 (最大为隔夜孵化)。
5. 基于洗涤剂的微凝胶体处理
-
通过溶解酶的溶解 (步骤 4), 洗涤微。
- 将微在 2000 x g 处旋转2分钟, 然后吸入上清。由于颗粒中分散的细胞碎片, 水滴会出现不透明的白色。
- 并用重悬微在5毫升10毫米的三盐酸缓冲器和吸管上下10x 混合。
- 将混合物向下旋转 2000 x 克, 2 分钟, 然后取出上清。
-
对微进行洗涤剂处理。
- 并用重悬的凝胶在一锂硫酸酯 (盖子) 裂解缓冲 (0.5% 瓦特/v 盖子在20毫米三 HCl) 和60µL 0.5 米 EDTA 到最后容量3毫升。
- 添加5µL 蛋白酶 K 酶 (800 U/毫升的股票)。吸管向上和向下的10x 混合, 然后孵化的混合物在42°c 在一个热块1小时溶解细胞膜和消化蛋白质。
-
经过洗涤剂处理后, 清洗微。
- 旋转圆锥管与微下来在 2000 x g 2 分钟. 取出上清。
- 用10毫升2% 伏/五吐温20的水冲洗微。吸管上下10x 混合。
- 旋转圆锥管在 2000 x g 2 分钟, 然后取出上清的愿望。
- 用10毫升100% 乙醇清洗微, 使残留酶失效。吸管上下10x 混合。
- 旋转圆锥管在 2000 x g 2 分钟, 然后取出上清的愿望。
- 用10毫升0.02% 伏/五吐温20的水冲洗微。吸管上下10x 混合。
- 旋转圆锥管在 2000 x g 2 分钟, 然后取出上清的愿望。
- 重复吐温20洗3x。在最后一次清洗前, 通过100µm 细胞过滤器将溶液移除, 以除去任何大团簇。
- 并用重悬微5毫升的10毫米三盐酸缓冲液, 以防止 DNA 降解。在 tagmentation (步骤 7) 之前, 微可存储在摄氏4摄氏度至1周。
6. 用数字 PCR 法生成条码滴
- 在低绑定管中的 1x TE 缓冲器中, 准备 500 pM 的条形底漆 (表 2) 的库存。在每次使用之前, 将底漆稀释为下午1点的工作库存, 并将其加热到70摄氏度, 在散热块上1分钟。
- 用75µL 的高保真热启动主组合制备 150-µL PCR 反应组合 (请参阅材料表) (2x), 42 µL PCR 级水, 3 µL DNA_BAR 底漆 (10 µM 库存, 0.2 µM 最后), µL 底漆 3 P7_BAR (10 µM 库存, 0.2 µM 决赛), 6 µL 条码稀释 (下午1点股票, 40 fM 决赛), 6 µL 吐温 20 (50% v/v 股票, 2% 决赛) 和15µL 的 PEG 6k (50% 瓦特/v 股票, 5% 决赛)。混合吹打上下10x。
- 将200µL 的 HFE 油放入注射器中, 用针将其 HFE。将 PE 油管的一部分连接到针上, 并将该线固定在手上。将 pe 油管的末端插入目标溶液中, 并小心地将 PCR 组合的所有150µL 到 PE 油管和注射器中。把注射器装进注射器泵里。
- 载入含氟油 (HFE) 的1毫升注射器, 含有 2% w/w perfluoropolyether 聚乙二醇 (PFPE PEG) 表面活性剂, 将其与针配合使用, 并将其放入注射器泵中。
-
使用共流 dropmaking 设备生成25µm 条码滴。
注: 有关指示试剂入口和出口位置的设备示意图, 请参见图 3A 。- 将 co 流 dropmaker 设备的单元格入口插入一小块铅焊料。
- 使用 PE 油管的熔融琼脂糖入口将装有 HFE 和 PCR 混合的注射器连接到微流控装置入口。在将管子插入设备之前, 先将泵从线上取出。
- 连接一条油管到出口, 并将自由端放在0.2 毫升 PCR 管。使用以下 (推荐) 流速为 dropmaking: 600 µL/小时为 HFE 2% w/w PFPE PEG 和200µL/小时为 PCR 组合。将滴入 PCR 管中, 每管大约有50µL 滴。
- dropmaking 后, 小心地从乳液中取出 HFE 油的下部, 使用凝胶加载吸管小贴士, 并用含有 5% w/w PFPE PEG 表面活性剂的 FC-40 含氟油代替。热循环以以下协议:98 °c 为3分钟, 40x (98 °c 为十年代, 62 °c 为二十年代, 72 °c 为二十年代), 72 °c 为5分钟, 然后举行在12°c。
注: 热循环液滴可以储存在4摄氏度, 可达1天。 -
验证条码放大和封装速率。
- 用2% 瓦特/w PFPE-PEG 表面活性剂, 在 HFE 上制备1x 核酸染色 (参见材料表);这种污渍在 HFE 中略有混溶, 将与水滴中的 DNA 结合。
- 将1µL 热循环条码乳液添加到10µL 的染色油中。在室温下孵化5分钟。
- 图像滴通过荧光显微镜 (GFP 通道, 497/520 nm 激发/发射波长) 在200X 放大和计数条码封装率。请注意, 信号将是离散的: 包含放大的条形码的水滴将荧光明亮, 而空的下落将显示黑暗 (图 4B)。
7. Tagmentation 基因组 DNA 在液滴中的提取
注意: 请参阅图 2B。
- 使用来自下一代测序库准备工具包的试剂准备500µL tagmentation 溶液 (请参见材料表)。使用7µL tagmentation 酶, 250 µL tagmentation 缓冲器, 243 µL 的 PCR 级水。混合涡流和旋转的混合物下来收集。将这个溶液装入一个 HFE 油支持的1毫升注射器中, 然后用针装入它。
-
准备回灌的微。
- 在 2000 x g 处旋转微凝胶体管2分钟, 并吸入上清。将200µL 的凝胶转移到 HFE 支持的注射器的顶端, 用凝胶加载吸管尖端, 用一小片胶带封住喷嘴。
- 使用 3 d 打印的离心机适配器 (请参阅补充文件 1和2), 在 3000 x g 处旋转微凝胶体注射器3分钟。
- 使用凝胶加载吸管尖端从注射器中取出液上清。把微凝胶体层推到注射器喷嘴的底座上。用针装注射器。
- 载入含氟油 (HFE) 的3毫升注射器, 含有 2% w/w perfluoropolyether 聚乙二醇 (PFPE PEG) 表面活性剂, 将其与针配合使用, 并将其放入注射器泵中。
- 将微重新封装成含有 tagmentation 试剂的水滴。
注: 有关指示试剂入口和出口位置的设备示意图, 请参见图 3B 。- 使用 PE 油管将含有 HFE、tagmentation 混合器和微的注射器连接到微流控装置入口。在将管子插入设备之前, 先将泵从线上取出。
- 连接一条油管到出口, 并将自由端放在一个空的1毫升注射器与活塞绘制到1毫升线。
- 使用以下 (推荐) 流速为 dropmaking: 2000 µL/小时为 HFE 2% w/w PFPE PEG, 200 µL/小时为微和500µL/小时为 tagmentation 组合。
- 用400X 放大倍数验证光镜下的微凝胶体封装速率。大约 80-90% 的水滴应该包含一个微凝胶体,如图4C 所示。
- 将含有 tagmentation 乳液的注射器装入针头, 并将其直立在热块或烤箱中1小时, 在55摄氏度处将基因组 DNA 碎片分解。
8. 单细胞条形码微流控双合并
注意: 请参阅图 2C。
- 为合并准备条码滴, 用 HFE 2% w/w PFPE PEG 代替 FC-40 油馏分。把这些水滴小心地转移到1毫升的注射器里, 用针把它装上, 放进注射器泵里。
- 将孵化和 tagmented 微凝胶体滴注射器装入注射器泵中。
- 准备500µL 的 PCR 组合。按照以下顺序添加试剂以防止沉淀形成: 140 µL PCR 级水, 10 µL P5_DNA 底漆 (10 µM 库存, 0.2 µM 最后), 10 µL P7_BAR 底漆 (10 µM 股票, 0.2 µM 最后), 50 µL 的 PEG 6k (50% 瓦特/v 库存, 5% 决赛), 50 µL 20 (50% v/v 股票, 5% 决赛), 250 µL Taq 大师组合 (2x) (参见材料表), 10 µL 等温聚合酶 (参见材料表) 和10µL 的中和缓冲器 (见材料表)。吹打上下10x 混合, 然后将混合物向下旋转以收集。将这个溶液装入一个 HFE 支持的1毫升注射器中, 将它与针头配合, 放入注射器泵中。
- 装载三3毫升含氟油 (HFE) 的注射器, 含有2% 瓦特/w perfluoropolyether 聚乙二醇 (PFPE PEG) 表面活性剂, 适合每个针头, 并将它们放入注射器泵中。
- 装载三1毫升注射器与2米氯化钠, 适合每个与针并且放在一边。
-
使用双合并装置将条码滴、tagmented 基因组液滴和 PCR 组合合并。
注意: 请参阅图 5中的设备示意图, 指示试剂和电极入口和插座的位置。- 将3氯化钠注射器连接到2电极入口和单护城河 inletusing PE 油管。对于电极, 手动对注射器施加压力, 直到电极完全充满盐溶液。填充电极后, 将手动压力应用到护城河注射器, 直到护城河填满。用一小块铅焊料堵住护城河的自由端。
- 将安装在泵上的 3 HFE 注射器连接到2个间隔油口和 dropmaking 油 inletusing 件 PE 油管。在将管子插入设备之前, 先将泵从线上取出。
- 连接 PCR 混合注射器, 微凝胶体滴注射器, 条形码滴注射器到他们各自的入口使用 PE 油管。在将所有液滴回注油管与抗静电枪连接到注射器针之前, 将其射出;抗静电处理降低了 PE 油管静电荷引起的液滴聚结风险。
- 将 PE 油管连接到出口, 并将游离端放在0.2 毫升的 PCR 管中。
- 使用鳄鱼夹子将电极注射器的针头连接到冷阴极荧光逆变器。将逆变器的直流电源设置为 2 v。
- 运行双合并设备的建议流量: 300 µL/小时的 tagmented 微凝胶体下降, 100 µL/小时的条码下降, 1500 µL/小时为 HFE 2% 瓦特/w PFPE PEG (dropmaking 油), 600 µL/小时的放大混合, 200 µL/小时 HFE 2% w/w PFPE-PEG (条形码间隔油), 和700µL/小时为 HFE 2% 瓦特/w PFPE-PEG (微凝胶体间隔油)。在每管中用大约50µL 的乳液将水滴收集到 PCR 管中。
- 在热循环之前, 小心地从乳液中取出较低层的 HFE 油, 使用凝胶加载吸管小贴士, 并用含有 5% w/w PFPE PEG 表面活性剂的 FC-40 氟化油代替。热循环以以下协议:65 °c 为5分钟, 95 °c 为2分钟, 30x (95 °c 为十五年代, 60 °c 为1分钟, 72 °c 为1分钟), 72 °c 为5分钟, 然后举行在12°c。
-
从热循环的水滴中恢复条形码 DNA。
- 将水滴放入离心管中, 用20µL 的 PFO 打破乳液。漩涡他们十年代混合。
- 旋转管在 1万 x g 1 分钟, 以分级混合物成水 (顶部) 和油 (底部) 阶段。用吸管小心地从管子上取下上层水层, 将其转移到新的离心管上。丢弃油相。
- 根据制造商的协议, 在自旋列中纯化条形码 PCR 产品, 并洗脱20µL 1x TE 缓冲器。
- 按照步骤9和10进行执行的排序和分析。
9. 图书馆的准备和排序
- 按照制造商的协议为碎片大小选择和量化, 准备单单元库进行排序。
- 将配对端库序列化为读1和读2的默认化学。使用自定义索引1底漆 I7_READ (表 2) 为 15 bp 索引读取, 对应于单单元条码。
10. 单细胞数据分析
注意: 定制 Python 脚本的质量控制和初步分析的 SiC 序列数据可以从 https://www.github.com/AbateLab/SiC-seq 下载。
- 运行脚本 "barcodeCleanup" 以执行条形码读取和导出单单元数据到 SQLite 数据库的质量控制。对于控件实验, 请将此脚本与 "-对齐" 标志集一起使用, 使读取与已知引用基因组对齐。
- 使用脚本 "纯度" 来分析条形码组 (用于控制实验) 的纯度, 并确认与图 6B一致的高纯度值。
结果
SiC 序列实验工作流包含3个用软光刻过程制作的微流控芯片 (图 1)。共流 dropmaker (图 3A) 生成25µm 的数字条码水滴, 用于标记基因组 DNA, 并使用唯一的单细胞标识符。条形码寡核苷酸由 15 bp 退化序列, 由 PCR 手柄两侧的放大 (表 2, 棒底漆)。条码被稀释到 femtomolar 浓度, 以实现单分子封装, 所有的水滴接收0或1条码片段 (s)。含有条形码的水滴放大, 产生了许多双绞型条码 amplicons 的拷贝。核酸染色用于验证成功的放大和量化条形码片段的封装速率 (图 4B)。微是由细菌细胞悬浮液和熔融琼脂糖凝胶在同等流速下产生的 (图 2A)。琼脂糖是准备在两次预期的最终浓度, 因为共流 dropmaking 过程有效稀释水溶液的系数为2。当琼脂糖冷却, 它固化成一个25µm 直径微凝胶体占据球形体积的水滴。
一系列的洗涤和裂解步骤净化微中的高分子量基因组 DNA (图 2B)。在破乳剂后, 在大体积内进行水洗, 稀释微量有机溶剂, 抑制下游酶处理。在显微镜下观察洗涤的微, 以验证细胞封装速率 (图 4A)。在微凝胶体悬浮液中加入含有广泛溶解活性的酶的鸡尾酒, 以消化细菌和真核微生物的细胞壁19。第二种蛋白酶 K 和洗涤剂的治疗会降低蛋白质和 solubilizes 细胞的碎片。
纯化 dna 的 Tagmentation 是在水滴中进行的, 以避免在微18之间扩散小 tagmented dna 片段导致潜在的交叉污染。液滴封装设备 (图 3B) 划分每个微凝胶体与一个缓冲和 tagmentation 酶, 它同时碎片双链 DNA, 同时也 "标记" 它与预先加载的寡核苷酸20。微被装入水滴作为接近包装的微粒, 达到封装率接近1微凝胶体为每下落与少量双峰21(图 4C)。
在微流控流的最后一步 (图 2C) 中, 一个设备执行双重合并操作, 组合1个条码降、1个含微凝胶体的降, 以及在受控的两步过程中的放大组合。首先, 含有 PCR 试剂的液滴被配对, 并与黄色 (图 5) 中显示的条形码下落合并。微流控通道中的盐水电极产生高电场梯度, 触发熔滴的合并。以类似的方式, 第一个合并的水滴与微凝胶体液滴配对, 并在以红色显示的区域中第二次合并。在单重叠扩展 (SOE) PCR 中采集液滴和热循环脱离芯片。条形码和 tagmented 基因组 DNA 重叠的互补端允许只有适当的条形码结构的融合和指数放大。
顺序数据首先通过读取质量进行筛选, 然后根据它们的 15 bp 单单元条码序列对读取进行分组分析。要认为条形码组是有效的, 应包含最少的读数数;这种阈值限制了对具有大量排序数据的单元格的分析, 并从数据集中删除了 PCR 变异的条形码 "孤儿"。在这个示例运行中, 最小值被设置为每组 7.5 kbps (每个50读取150个 bp)。条形码计数与组大小的直方图显示, 有效条码组的重要部分刚好高于阈值大小 (图 6A)。
在一个已知微生物群落组成的控制实验中, 用纯度和相对丰度指标来评价 SiC 序列的质量。在这里, 分析了由3革兰氏阴性菌、5革兰氏阳性菌和2株酵母组成的合成10细胞群落。给定条码组的纯度定义为组中最常见的基因组的读数映射数除以组中读取总数的个数。绝大多数条码组的纯度大于 0.95 (图 6B)。单元格类型的相对丰度是通过计算原始读取数和通过计数条形码组来计算的, 其中, 组分配了与其成员读取的一致性相对应的单元格类型 (图 6C)。大量的读取和条码组跟踪大致相同的比例, 表明细胞的人口正在取样, 使某些物种不包含在不成比例的小或大型条码组。从单个物种中绘制所有条码组的总覆盖范围, 表明整个基因组的覆盖率很高, 且很少或没有辍学区域 (图 7)。覆盖范围的均匀性可以通过规范化覆盖率值的频率分布进行验证, 大多数值围绕平均值 (图 7, 嵌入)。
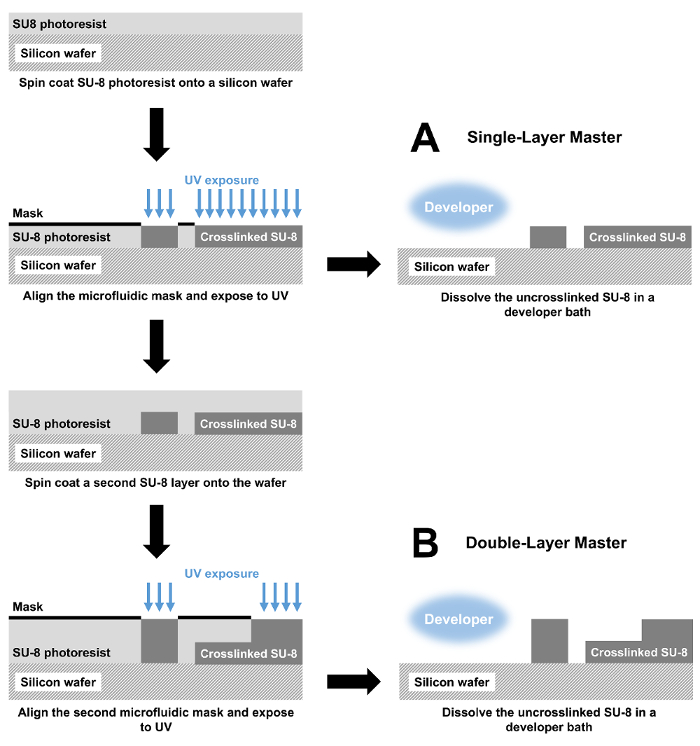
图 1: 通过光刻工艺制备微流控器件.(A) 具有单个特征高度的主模具由自旋涂覆一层 SU-8 光刻胶到硅片上制成。然后用光刻掩模和 UV 光进行图案化, 将暴露的 SU-8 交联。最后, uncrosslinked SU-8 溶解在显影液中。所产生的模具用于铸造与玻璃滑块粘合, 以产生完整的微流控装置。(B) 对于双层设备, 制造过程类似于从自旋涂层和曝光步骤开始。然后重复这些步骤以创建两层设备。请单击此处查看此图的较大版本.
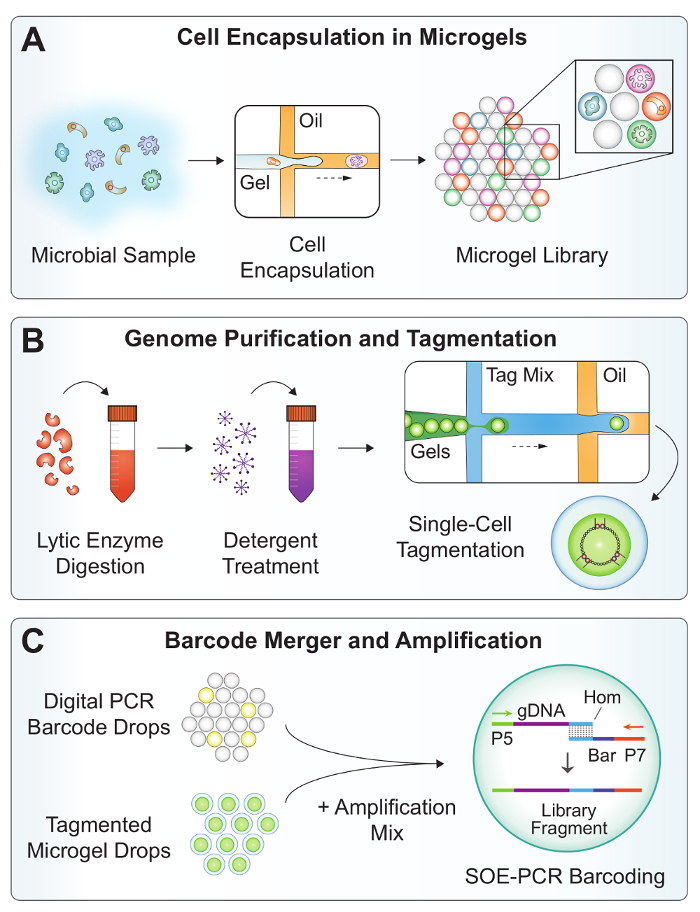
图 2:SiC 序列工作流概述.(A) 微生物悬浮液与 dropmaker 装置中的熔融琼脂糖共同流动, 以封装微中的单个细胞。(B) 微受到一系列清洗, 以净化细菌基因组 DNA。溶解酶消化革兰阳性菌和酵母细胞壁, 洗涤剂 solubilizes 细胞碎片。微被重新封装成水滴, tagmentation 以减少交叉污染。(C) 微流控合并结合了数字 PCR 条码、tagmented 微凝胶体基因组, 以及一个速率 > 1 赫的放大组合。非芯片的国企-PCR 拼接一个独特的单细胞条码到 tagmented 基因组, 并有选择地放大完全条形码结构。请单击此处查看此图的较大版本.
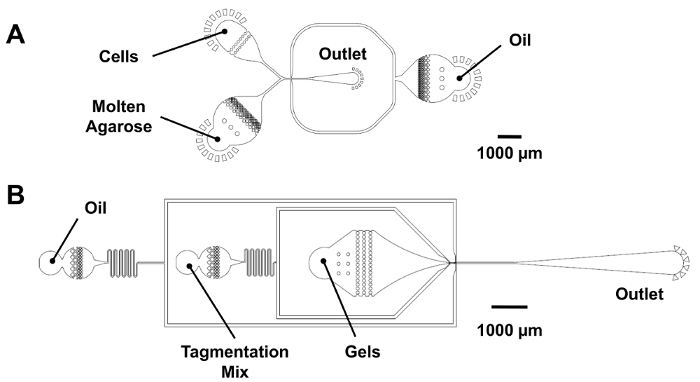
图 3:用于 dropmaking 和微凝胶体重新封装的微流控设备。(A) 此面板显示一个共流 dropmaker (25 µm 的特征高度)。细胞和熔融琼脂糖被引入到设备中, 在相同的流速, 以产生25µm 液滴在25µm x 25 µm 结。对于数字条码 dropmaking, 细胞入口堵塞, 并引入 PCR 组合进入琼脂糖入口。(B) 此面板显示微凝胶体重新封装设备 (25 µm 的特征高度)。微流入漏斗形入口, 以维持其紧密包装的订购, 并在25µm x 30 µm 路口重新封装之前接收 tagmentation 混合体。请单击此处查看此图的较大版本.

图 4: 水滴和微的显微照片.(A) 此面板显示在酶裂解之前洗涤了25µm 微。细菌被荧光染色, 以定量的封装速率。泊松负荷统计数据规定, 单元格的封装速率为 1, 在10滴或更少的情况下, 以最小化多封装事件的频率。(B) 此面板显示了一个荧光显微图像, 25 µm 数字条码水滴处理的核酸染色。含有放大条码片段的水滴产生强烈的荧光信号。(C) 此面板显示微重新封装在50µm 滴中。微的封闭包装使封装速率接近1凝胶每滴, 很少双峰。请单击此处查看此图的较大版本.
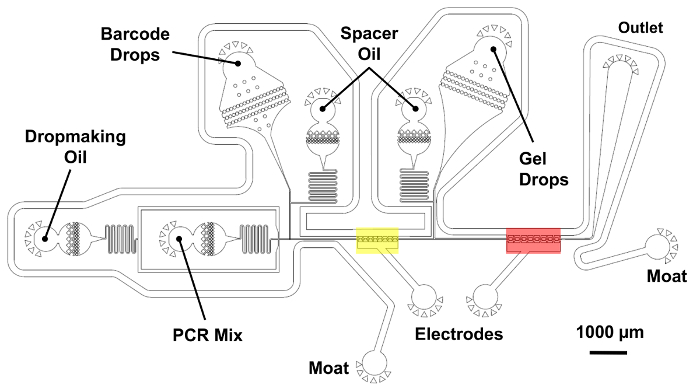
图 5: 单细胞基因组条形码的微流控双合并装置.两步合并操作对条形码水滴与 tagmented 基因组在高吞吐量。首先生成一滴 PCR 混合物, 并将其与在黄色中使用盐水电极显示的条形码水滴合并。接下来, 一个包含微凝胶体的水滴在红色显示的区域中第二次被引入并合并。油入口允许精确控制重新注入水滴之间的间距。条形码回注室及其间隔油放置在较短的25µm 层上, 以蓝色着色。所有其他设备功能属于较厚的层, 总高度为45µm。请单击此处查看此图的较大版本.
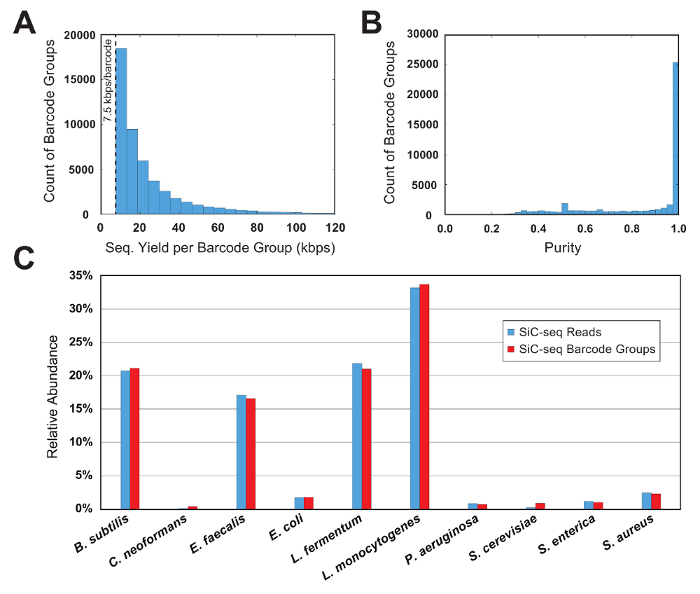
图 6:10 细胞合成微生物群落的条码组度量.(A) 此面板显示条码组大小的分布。当组大小增加时, 给定大小的组数呈指数递减。每个组的最小阈值为 7.5 kbps 限制对具有足够信息量的组进行分析, 并删除 PCR 突变序列 "孤儿"。(B) 此面板显示条码组纯度的分布。绝大多数 (> 90%) 的群体是非常高纯度 (> 95%)。(C) 此面板显示了在读取和条码组级别计算的10种的相对丰度。2计数方法产生相似的结果, 表明条码组的大小是一致的不同物种。请单击此处查看此图的较大版本.
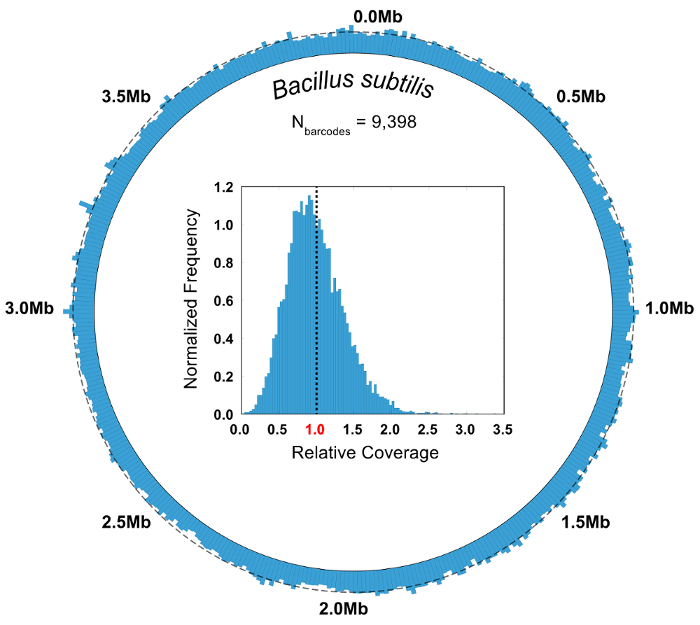
图 7: 聚合基因组覆盖率枯草芽孢杆菌条码组.将所有条码组的读数从映射到细菌枯草杆菌(N = 9398) 进行汇总并进行分析。圆形覆盖图说明了 SiC 序列读数的覆盖均匀性, 没有可观察到的辍学区域。绕圆周的虚线表示平均覆盖范围 (5.55x)。相对覆盖频率的嵌入直方图表明, 大部分的碱基覆盖在接近全基因组平均的深度, 由虚线表示。请单击此处查看此图的较大版本.
| 装置 | 第一层高度 (µm) | 第一层旋转速度 (rpm) | 第二层高度 (µm) | 第二层旋转速度 (rpm) |
| 共流 dropmaker | 25 | 4000 | N/A | N/A |
| 凝胶再 encapsulator | 25 | 4000 | N/A | N/A |
| 双重合并 | 25 | 4000 | 20 | 5000 |
表 1: 微流控设备制造参数.本表显示了在 SiC 序列工作流中使用的微流控器件的列表, 其所需的光刻胶自旋涂层的速度 (基于 SU-8 3025 的制造商规格)。
| 标签 | 序列 (5 ' > 3 ') | ||||
| 酒吧 | GCAGCTGGCGTAATAGCGAGTACAATCTGCTCTGATGCCGCATAGNNNNNNNNNNNNNNNTAAGCCAGCCCCGACACT | ||||
| DNA_BAR | CTGTCTCTTATACACATCTCCGAGCCCACGAGACGTGTCGGGGCTGGCTTA | ||||
| P7_BAR | CAAGCAGAAGACGGCATACGAGATCAGCTGGCGTAATAGCG | ||||
| P5_DNA | AATGATACGGCGACCACCGAGATCTACACTCGTCGGCAGCGTC | ||||
| I7_READ | GCCCACGAGACGTGTCGGGGCTGGCTTA | ||||
表 2: 底漆序列。
补充文件 1:请单击此处下载此文件.
补充文件 2:请单击此处下载此文件.
讨论
SiC 序列微流控流产生了数以千计的细菌细胞的单细胞基因组测序数据。将数字条形码拼接到微凝胶体封装的单元格的基因组上, 可以将数据的在硅片反褶积转化为来自同一单元格的条形码读取组。一个已知成分的微生物群落的控制实验是评价条形码组纯度的必要方法。大量低纯度组表明, 细胞封装率过高, 或有明显的水滴交叉污染发生在微流控处理步骤。根据泊松统计数据, 条形码和单元格应封装在1粒子的目标比, 每10滴, 以限制多个封装事件的速率小于5% 的所有非空滴。比这更高的封装速率会以指数的形式增加双峰的速率, 因此在 dropmaking 过程中对封装率的验证至关重要。用户在单个微凝胶体中应特别小心封装多个单元格, 因为共享同一条形码序列的不同单元格的读取不能 bioinformatically 分隔。如果1单元接收2不同的条形码, 条码组的纯度不受影响, 虽然丰度指标是在按条形码顺序计数时倾斜的。
由于合并条件不理想, 液滴交叉污染也可能出现。在成功的操作中, 微流控合并装置 (图 5) 可以 controllably 1 个条码液滴与1微凝胶体和一定量 PCR 试剂。非理想的流速会导致液滴配对不正确的比率: 例如, 1 条码可以与2微配对。协议中列出的所有流量都是估计的, 可能需要根据设备几何和雾滴大小的细微变化来调整。使用高速录音功能 (> 1万帧/秒) 的摄像头的用户应在微流控操作的开始和结束过程中验证正确的水滴合并。用户如果不使用高速相机, 就可以收集少量的合并输出, 并手动测量显微镜下的水滴大小。液滴尺寸应均匀: 过量的并入条形码或微凝胶体下降表明, 应相应减少回注率。
在处理微和 microdroplets 时, 应采取一些一般的预防措施, 以维护其完整性。微, 虽然机械健壮, 必须充分冷却之前, 打破和洗涤步骤, 以确保完整的凝胶。非球形微表明琼脂糖没有足够的时间凝固。清洗微时, 按所需的速度旋转悬浮液以避免产品损失。琼脂糖凝胶具有与水紧密匹配的折射率, 在管22中可能很难看到, 因此用户在吸入前应仔细识别凝胶液的边界。在实验室手套和油管上, 静态力23的累积使水中油滴容易发生聚结。因此, 我们建议在泵浦启动前, 用徒手装入滴注注射器, 并用防静电枪处理所有回灌线。通过缓慢旋转注射器中的乳液, 并手动吸出较大的水滴, 在顶部积聚, 由于其较大的浮力, 可以去除大的合并液滴。
SiC 序列是第一个证明单细胞基因组测序 > 5万细菌细胞的技术。该平台在现有方法的吞吐量方面具有显著优势, 并能够更深入地取样异构微生物群落。迄今为止, 单细胞基因组测序的微流控技术已使用 microchambers9和 microwells24进行细胞隔离和扩增, 但仅有数以上百的细胞的吞吐量。将单个单元格的流排序为 wellplates5,6不需要专门的微流控工具, 但具有类似的低吞吐量。鉴于来自环境的土壤和水样通常在物种级25、26的 > 1000 中具有 alpha 多样性, 因此, 由于它能够取样大量的生物体, 所以 SiC 序列是非常有利的。SiC 序列工作流适用于实验室文化、自然环境或活体宿主的细胞输入。细胞样品只需要在水悬浮和无大颗粒 (> 10 µm) 适合微流控封装。例如, 以前已将该方法应用于海水样本, 使用一系列洗涤和过滤步骤在封装17之前对单元格进行预处理。
SiC 序列协议从每个单元中生成相对稀疏的排序数据量, 可能不适用于所有应用程序。一些生物信息学算法, 如 "从头" 基因组组装或单核苷酸变体 (SNV) 调用需要更高的覆盖率深度才能有效地工作。相反, 可以通过分类 binning 方法27将条码组聚集在硅片中, 以便可以将算法应用于较大的读取集。碳化硅序列工作流的总体条形码效率相对较低, 在输入样本的可用性较低的情况下也可能带来挑战。SiC 序列依赖于泊松分布的条形码封装步骤, 因此大约10% 的细胞接收到一个分子条码, 并在最后的图书馆准备步骤中被放大。虽然这与其他基于 microdroplet 的条形码方案10类似, 但使用珍贵细胞样本的用户可能难以达到适当的库产量, 因此可能需要在最终的时间内增加 PCR 周期的数量。放大步骤。另一种潜在的解决方案为使用微流控技术的用户是在数字 PCR 步骤后排序正条码滴, 从而使整体条形码效率 > 85%28。
SiC 序列技术的一个潜在的未来方向是调整工作流, 用于哺乳动物细胞, 为新的临床单细胞研究铺平道路。例如, 对单癌细胞复制数变异的分析, 可以进一步了解异质性在肿瘤病理学中的作用2。或者, 将 SiC 序列与现有的方法相结合, 以探测和丰富感兴趣的 DNA 顺序29将使目标单细胞序列的亚群或罕见的细胞株进行排序。通过环境样本, 已知的代谢通路中的基因可以被定位, 并与相邻的基因进行分析, 以确定新的基因组岛。从人类宿主环境中, 低效的致病细菌样本可以被分离和序列在单细胞水平, 以更密切地检查他们的基因型的致病力的起源。
披露声明
与此工作流程有关的专利可能被授权给特派团生物, 其中 Adam r 减弱为股东。
致谢
这项工作得到了国家科学基金会通过职业奖 (赠款号 DBI-1253293) 的支持;国立卫生研究院 (NIH) (赠款编号 HG007233-01、R01-EB019453-01、1R21HG007233、DP2-AR068129-01、R01-HG008978);和国防高级研究项目机构生活铸造项目 (合同编号 HR0011-12-C-0065, N66001-12-C-4211, HR0011-12-C-0066)。
材料
| Name | Company | Catalog Number | Comments |
| 3" silicon wafers, P type, virgin test grade | University Wafers | 447 | |
| SU-8 3025 photoresist | Microchem | 17030192 | |
| Spin coater | Specialty Coating Systems | G3P-8 | |
| Photomasks | CadArt Servcies | (custom) | See Supplemental Files for mask designs |
| PGMEA developer | Sigma-Aldrich | 484431 | |
| Isopropanol | Sigma-Aldrich | 109827 | |
| Sylgard 184 silicone elastomer kit | Krayden | 4019862 | |
| Degassing chamber | Bel-Art | 42025 | |
| 0.75 mm biopsy punch | World Precision Instruments | 504529 | |
| Glass microscope slides (75 mm x 50 mm) | Corning | 294775X50 | |
| Aquapel (hydrophobic glass treatment) | Pittsburgh Glass Works | 47100 | |
| PE-2 polyethylene tubing | Scientific Commodities | B31695-PE/2 | |
| 1 mL syringes | BD | 309628 | |
| 27 gauge needles | BD | 305109 | |
| Syringe pump | New Era Pump Systems | NE-501 | |
| Novec HFE-7500 fluorinated oil (HFE) | 3M | 98-0212-2928-5 | |
| FC-40 fluorinated oil | Sigma-Aldrich | F9755 | |
| PEG-PFPE surfactant | Ran Biotechnologies | 008-FluoroSurfactant | |
| Space heater | Lasko | CD09250 | |
| Agarose, low gelling temperature | Sigma-Aldrich | a9414 | |
| TE (10X) | Rockland | mb-007 | |
| PBS 1X, pH 7.4 | E&K Scientific Products | EK-65083 | |
| OptiPrep (density gradient medium) | Sigma-Aldrich | d1556 | |
| 1H,1H,2H-Perfluoro-1-Octanol (PFO) | Sigma-Aldrich | 370533 | |
| Span 80 (sorbitane monooleate) | Sigma-Aldrich | s6760 | |
| Hexane | Sigma-Aldrich | 139386 | |
| Tween 20 (polysorbate 20) | Sigma-Aldrich | p2287 | |
| Lysozyme Type IV | MP Biomedicals | 195303 | |
| Mutanolysin | Sigma-Aldrich | M9901 | |
| Zymolyase (yeast lytic enzyme) | Zymo Research | e1004 | |
| Lysostaphin | Sigma-Aldrich | L7386 | |
| Sodium chloride | Sigma-Aldrich | S9888 | |
| EDTA | Sigma-Aldrich | E6758 | |
| Tris-HCl, pH 7.5, 1M | Invitrogen | 15567-027 | |
| Dithiothreitol (DTT) | Teknova | d9750 | |
| Lithium dodecyl sulfate | Sigma-Aldrich | L9781 | |
| Proteinase K | New England Biosciences | P8107S | |
| Ethanol, 200 Proof (100%) | Koptec | V1001 | |
| SYBR Green I (nucleic acid stain) | Invitrogen | S7563 | |
| PEG 6k | Sigma-Aldrich | 81260 | |
| Triton X-100 (octylphenol ethoxylate) | Sigma-Aldrich | t8787 | |
| Nextera DNA Library Prep Kit | Illumina | FC-121-1030 | |
| Phusion Hot Start Flex Master Mix (High-Fidelity Hot Start Master Mix) | New England Biosciences | m05365 | |
| Platinum Multiplex PCR Master Mix (Taq Master Mix) | Applied Biosystems | 4464263 | |
| Warmstart 2.0 Bst Polymerase (isothermal polymerase) | New England Biosciences | m0538m | |
| NT buffer from Nextera XT kit (neutralization buffer) | Illumina | FC-131-1024 | |
| Cold cathode fluorescent inverter | (custom) | (custom) | |
| DC power supply | Mastech | HY1503D | |
| Zerostat 3 anti-static gun | Milty | 5036694022153 | |
| 3D-printed centrifuge syringe holder | (custom) | (custom) | See Supplemental Files for 3D print file |
| Zymo DNA Clean & Concentrator-5 | Zymo Research | D4003 |
参考文献
- Navin, N., et al. Tumour evolution inferred by single-cell sequencing. Nature. 472 (7341), 90-94 (2011).
- Ni, X., et al. Reproducible copy number variation patterns among single circulating tumor cells of lung cancer patients. Proceedings of the National Academy of Sciences of the United States of America. 110 (52), 21083-21088 (2013).
- Schmidt, H., Hensel, M. Pathogenicity islands in bacterial pathogenesis. Clinical Microbiology Reviews. 17, 14-56 (2004).
- Martínez, J. L., Baquero, F. Interactions among strategies associated with bacterial infection: pathogenicity epidemicity, and antibiotic resistance. Clinical Microbiology Reviews. 15 (4), 647-679 (2002).
- Rinke, C., et al. Obtaining genomes from uncultivated environmental microorganisms using FACS-based single-cell genomics. Nature Protocols. 9 (5), 1038-1048 (2014).
- Rinke, C., et al. Insights into the phylogeny and coding potential of microbial dark matter. Nature. 499 (7459), 431-437 (2013).
- Zhang, H., Liu, K. K. Optical tweezers for single cells. Journal of the Royal Society Interface. 5 (24), 671-690 (2008).
- Xu, L., Brito, I. L., Alm, E. J., Blainey, P. C. Virtual microfluidics for digital quantification and single-cell sequencing. Nature Methods. 13 (9), 759-762 (2016).
- Gawad, C., Koh, W., Quake, S. R. Dissecting the clonal origins of childhood acute lymphoblastic leukemia by single-cell genomics. Proceedings of the National Academy of Sciences of the United States of America. 111 (50), 17947-17952 (2014).
- Macosko, E. Z., et al. Highly parallel genome-wide expression profiling of individual cells using nanoliter droplets. Cell. 161 (5), 1202-1214 (2015).
- Klein, A. M., et al. Droplet barcoding for single-cell transcriptomics applied to embryonic stem cells. Cell. 161 (5), 1187-1201 (2015).
- Rotem, A., et al. High-throughput single-cell labeling (Hi-SCL) for RNA-Seq using drop-based microfluidics. PLoS One. 10 (5), 1-14 (2015).
- Amini, S., et al. Haplotype-resolved whole-genome sequencing by contiguity-preserving transposition and combinatorial indexing. Nature Reviews Genetics. 46 (12), 1343-1349 (2014).
- Zheng, G. X. Y., et al. Haplotyping germline and cancer genomes with high-throughput linked-read sequencing. Nature Biotechnology. 34 (3), 303-311 (2016).
- Lan, F., Haliburton, J. R., Yuan, A., Abate, A. R. Droplet barcoding for massively parallel single-molecule deep sequencing. Nature Communications. 7, 11784 (2016).
- Rotem, A., et al. Single-cell ChIP-seq reveals cell subpopulations defined by chromatin state. Nature Biotechnology. 33 (11), 1165-1172 (2015).
- Lan, F., Demaree, B., Ahmed, N., Abate, A. R. Single-cell genome sequencing at ultra-high-throughput with microfluidic droplet barcoding. Nature Biotechnology. 35 (7), 640-646 (2017).
- Novak, R., et al. Single-cell multiplex gene detection and sequencing with microfluidically generated agarose emulsions. Angewandte Chemie Internation Edition. 50 (2), 390-395 (2011).
- Gill, C., Van De Wijgert, J. H. H. M., Blow, F., Darby, A. C. Evaluation of lysis methods for the extraction of bacterial DNA for analysis of the vaginal microbiota. PLoS One. 11 (9), 1-16 (2016).
- Picelli, S., et al. Tn5 transposase and tagmentation procedures for massively scaled sequencing projects. Genome Research. 24 (12), 2033-2040 (2014).
- Abate, A. R., Chen, C. H., Agresti, J. J., Weitz, D. A. Beating Poisson encapsulation statistics using close-packed ordering. Lab on a Chip. 9 (18), 2628 (2009).
- Jain, A., Yang, A. H. J., Erickson, D. Gel-based optical waveguides with live cell encapsulation and integrated microfluidics. Optic Letters. 37 (9), 1472 (2012).
- Karbaschi, M., Shahi, P., Abate, A. R. Rapid chemical-free breaking of microfluidic emulsions with a hand-held antistatic gun. Biomicrofluidics. 11 (4), 1-6 (2017).
- Gole, J., et al. Massively parallel polymerase cloning and genome sequencing of single cells using nanoliter microwells. Nature Biotechnology. 31 (12), 1126-1132 (2013).
- Chao, Y., et al. Metagenomic analysis reveals significant changes of microbial compositions and protective functions during drinking water treatment. Scientific Reports. 3 (1), 3550 (2013).
- Fierer, N., et al. Cross-biome metagenomic analyses of soil microbial communities and their functional attributes. Proceedings of the National Academy of Sciences of the United States of America. 109 (52), 21390-21395 (2012).
- Mande, S. S., Mohammed, M. H., Ghosh, T. S. Classification of metagenomic sequences: methods and challenges. Briefings in Bioinformatics. 13 (6), 669-681 (2012).
- Eastburn, D. J., et al. Microfluidic droplet enrichment for targeted sequencing. Nucleic Acids Research. 43 (13), e86 (2015).
- Clark, I. C., Abate, A. R. Finding a helix in a haystack: nucleic acid cytometry with droplet microfluidics. Lab on a Chip. 17 (12), 2032-2045 (2017).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。