需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
用相似有效的二元分类性能选择多个生物标记子集
摘要
现有算法为生物标志物检测数据集生成一个解决方案。该协议演示了多个类似的有效解决方案的存在, 并提供了一个用户友好的软件, 以帮助生物医学研究人员调查其数据集的建议的挑战。计算机科学家也可以在他们的生物标志物检测算法中提供这一特性。
摘要
生物标志物检测是高通量 "组学" 研究人员最重要的生物医学问题之一, 几乎所有现有的生物标志物检测算法都能通过对给定数据集的优化性能测量来生成一个生物标记子集。.然而, 最近的一项研究表明, 有多个生物标志物子集具有相似的有效甚至相同的分类性能。该协议提供了一种简单明了的方法, 用于检测具有二进制分类性能的生物标记子集, 优于用户定义的截止。该协议包括数据准备和加载、基线信息汇总、参数调整、生物标志物筛选、结果可视化和解释、生物标记基因注释、结果和可视化输出出版质量。所提出的生物标志物筛选策略是直观的, 并证明了开发生物标志物检测算法的一般规则。使用编程语言 Python 开发了用户友好的图形用户界面 (GUI), 使生物医学研究人员能够直接访问其结果。kSolutionVis 的源代码和手册可以从 http://www.healthinformaticslab.org/supp/resources.php 下载。
引言
二进制分类是生物医学领域中最常见、最具挑战性的数据挖掘问题之一, 用于构建对两组样本进行培训的分类模型, 其中最精确的判别功率为1,2,3,4,5,6,7. 然而, 生物医学领域产生的大数据具有固有的 "大 p 小 n" 范式, 其特征数通常远远大于样本6、8、9的数量。因此, 生物医学研究人员必须在使用分类算法之前减少特征维度, 以避免过度拟合问题8,9。诊断生物标志物被定义为从健康对照样本10,11分离某一疾病患者的检测特征的子集。患者通常被定义为阳性样本, 健康控制被定义为阴性样本12。
最近的研究表明, 生物医学数据集5具有相同或类似有效的分类性能的一个以上的解决方案。几乎所有的特征选择算法都是确定性算法, 只为同一数据集生成一个解决方案。遗传算法可以同时生成具有类似性能的多个解决方案, 但他们仍然尝试选择一个具有最佳健身功能的解决方案, 作为给定数据集13、14的输出。
特征选择算法可以大致分组为过滤器或包装12。筛选器算法根据要素相互独立的假设, 选择与二进制类标签的重要个人关联排名的顶级k要素15、16、17.尽管此假设对于几乎所有实际数据集都不适用, 但启发式筛选规则在许多情况下都很好地执行, 例如, mRMR (最小冗余和最大相关性) 算法、基于魏氏测试的功能筛选 (WRank)算法, 以及 ROC (接收机操作特性) 基于图的滤波 (ROCRank) 算法。mRMR 是一种有效的滤波算法, 因为它近似于一系列较小问题的组合估计问题, 与最大依赖特征选择算法相比, 其中每一个只涉及两个变量,因此使用更健壮的18、19的成对联合概率。但是, mRMR 可能低估某些功能的用处, 因为它不测量可以增加相关性的要素之间的交互, 因此错过了一些单独无用但仅在组合时有用的要素组合。WRank 算法计算一个非参数分数, 说明特征在两类样本之间的判别方式, 并以其对异常值20、21的鲁棒性而著称。此外, ROCRank 算法评估特定特征的 ROC 曲线 (AUC) 下的区域对于调查的二进制分类性能22、23的重要性。
另一方面, 包装器会评估给定要素子集的预定义分类程序的性能, 并通过启发式规则迭代生成, 并使用最佳性能测量24创建特征子集。包装通常优于分类性能中的过滤器, 但运行速度较慢25。例如, 正则随机林 (RRF)26、27算法使用贪婪规则, 方法是在每个随机林节点上评估训练数据子集上的要素, 其要素重要性分数由基尼索引评估.如果其信息增益不能提高所选要素的性能, 则选择新特征将受到惩罚。此外, 微阵列 (PAM)28、29算法的预测分析, 还有一个包装算法, 计算每个类标签的质心, 然后选择特征, 以缩小基因质心向整体类质心。PAM 具有强大的外围功能。
对于任何给定数据集, 可能需要具有顶级分类性能的多个解决方案。首先, 确定算法的优化目标是由一个数学公式定义的,例如最小误差率30, 这不一定是生物样本的理想选择。其次, 数据集可能具有多个、显著不同的解决方案, 具有类似的有效或甚至相同的性能。几乎所有现有的特征选择算法都将随机选择其中一个解决方案作为输出31。
本研究将介绍一种信息分析协议, 用于为任何给定的二进制分类数据集生成具有相似性能的多特征选择解。考虑到大多数生物医学研究人员不熟悉地学信息技术或计算机编码, 开发了一个用户友好的图形用户界面 (GUI), 以便快速分析生物医学二进制分类数据集。分析协议包括数据加载和汇总、参数调整、管道执行和结果解释。通过简单的单击, 研究人员能够生成生物标记子集和出版物质量可视化图。该协议已通过转录两个二进制分类数据集的急性淋巴细胞白血病 (ALL),即ALL1 和 ALL212进行了测试。ALL1 和 ALL2 的数据集是从 http://www.broadinstitute.org/cgi-bin/cancer/datasets.cgi 的广泛的研究所基因组数据分析中心下载的。ALL1 包含128个具有12625个特征的样本。这些样本中, 95 是 B 细胞全部, 33 是 T 细胞。ALL2 还包括100个具有12625个功能的样本。在这些样本中, 有65例患者复发和35例没有。ALL1 是一个简单的二进制分类数据集, 最小精度为四个过滤器和四个包装为 96.7%, 6 的8功能选择算法实现 100%12。虽然 ALL2 是一个比较困难的数据集, 但上面的8个特征选择算法达到了83.7% 精度12。通过封装算法检测到的56特性、基于相关性的特征选择 (CFS), 实现了最佳精度。
研究方案
注: 以下协议描述了主要模块的信息学分析程序和伪代码的详细信息。自动分析系统是使用 python 版本3.6.0 和 python 模块 (熊猫、abc、numpy、scipy、sklearn、sys、PyQt5、sys、mRMR、数学和 matplotlib) 开发的。本研究中使用的材料列在材料表中。
1. 准备数据矩阵和类标签
- 将数据矩阵文件作为制表符或逗号分隔的矩阵文件进行准备, 如图 1A所示。
注: 每一行都有要素的所有值, 第一项是要素名称。特征是基于微阵列的转录组数据集的 probeset id, 或者可能是另一个值 id, 如半胱氨酸残留及其在 methylomic 数据集中的甲基化值。每个列提供给定示例的要素值, 第一个项目是示例名称。一行由制表符 (图 1B) 或逗号 (图 1C) 分隔成列。制表符分隔的矩阵文件由文件扩展名. tsv 识别, 逗号分隔的矩阵文件具有扩展名. csv。此文件可能是通过将矩阵保存为. tsv 或. csv 格式的软件 (如 Microsoft Excel) 生成的。数据矩阵也可以通过计算机编码生成。 - 准备类标签文件作为制表符或逗号分隔的矩阵文件 (图 1D), 类似于数据矩阵文件。
注意: 第一列提供了示例名称, 每个示例的类标签都在标题为class的列中给出。在编码过程中考虑最大兼容性, 以便可以添加其他列。类标签文件可以格式化为. tsv 或. csv 文件。列类中的名称可以是任何术语, 并且可能有两个以上的示例类。用户可以选择任意两个类进行以下分析。
2. 加载数据矩阵和类标签
- 将数据矩阵和类标签加载到软件中。单击按钮加载数据矩阵以选择用户指定的数据矩阵文件。单击按钮加载类标签以选择相应的类标签文件。
注意: 两个文件加载后, kSolutionVis 将执行常规屏幕的兼容性两个文件。 - 从数据矩阵文件中汇总特征和样本。估计数据矩阵文件的大小。
- 从类标签文件中汇总示例和类。估计类标签文件的大小。
- 测试数据矩阵中的每个样本是否具有类标签。使用类标签汇总示例的编号。
3. 汇总和显示数据集的基线统计信息
- 单击 "汇总" 按钮, 无需任何指定的关键字输入, 软件将显示20索引要素和相应要素名称。
注意: 用户需要指定他们希望查找的功能名称, 以查看其基线统计信息和所有输入样本之间的相应值分布。 - 在文本框功能中提供关键字,例如"1000_at", 以查找要汇总的特定功能。单击 "汇总" 按钮以获取此给定要素的基线统计信息。
注意: 关键字可能出现在目标要素名称的任何位置, 方便用户搜索过程。 - 单击 "汇总" 以使用给定的关键字查找多个要素, 然后指定唯一功能 ID 以继续执行上述步骤总结一个特定要素。
4. 确定类标签和排名最高的要素数
- 在下拉框类中选择正数 ("P (33)") 和负数 ("N (95)") 类, 如图 2 (中间) 所示.
注意: 建议选择平衡的二进制分类数据集,即阳性和阴性样本数之间的差异极小。在两个下拉框中的每个类标签的名称之后, 还会在括号中给出样本数。 - 在下拉框Top_X (?)中, 选择10作为顶排要素 (参数pTopX) 的数量。功能子集的全面屏幕。
注: 软件会自动将所有要素按由每个要素的 t-检验计算的P值进行排序, 比较正负类。具有较小P值的特征在两类样本之间具有更好的辨别力。全面的筛选模块是计算密集型的。默认情况下, 参数pTopX为10。用户可以更改此参数的范围为10到 50, 直到他们找到满意的特征子集具有良好的分类性能。
5. 调整不同性能的系统参数
- 在所选分类器极限学习机 (榆木) 的下拉框中, 选择性能测量 (pMeasurement) 精度 (acc) bAcc (?) 。此参数的另一个选项是测量平衡精度 (bAcc)。
注意: 让 TP、FN、TN 和 FP 分别是真实阳性、假阴性、真底片和误报的数量。测量Acc是定义为 (tp + tn)/(tp + FN + TN + FP), 它最适用于平衡数据集6。但是, 对于Acc进行优化的分类器往往会将所有样本分配给负类, 如果负样本的数量远远大于正的样品。bAcc定义为 (sn + Sp)/2, 其中 sn = TP/(TP + FN) 和 Sp = tn/(tn + FP) 是正确预测的阳性和阴性样品的费率, 分别。因此, bAcc 对两个类的预测性能进行规范化, 并可能导致两个不平衡类的预测性能平衡。Acc是pMeasurement的默认选择。软件默认使用分类器榆木来计算分类性能。用户还可以从 SVM (支持向量机)、KNN (k 最近邻)、决策树或幼稚贝叶斯选择分类器。 - 在输入框pCutoff:中为指定的性能测量选择截止值 0.70 (参数pCutoff)。
注意: Acc和bAcc范围介于0和1之间, 用户可以指定一个值 pCutoff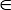 [0, 1] 作为截止显示匹配的解决方案。该软件进行了全面的功能子集筛选, pCutoff的适当选择将使3D 可视化更加直观和明确。pCutoff的默认值为0.70。
[0, 1] 作为截止显示匹配的解决方案。该软件进行了全面的功能子集筛选, pCutoff的适当选择将使3D 可视化更加直观和明确。pCutoff的默认值为0.70。
6. 运行管道并生成交互式可视化结果
- 单击 "分析" 以运行管道并生成可视化图, 如图 2 (底部) 所示。
注意: 左表提供了所有的要素子集及其pMeasurement , 由分类器榆木的10倍交叉验证策略计算, 如前5所述。使用当前参数设置为特征子集筛选过程生成两个3D 散点图和两行图解。 - 选择0.70 作为pMeasurement截止 (参数piCutoff、输入框值) 和10作为最佳要素子集 (参数piFSNum) 数量的默认值。
注意: 管道是使用参数pTopX、 pMeasurement和pCutoff执行的。检测到的要素子集可以使用截止piCutoff进一步筛选, 但是piCutoff不能小于pCutoff。因此, piCutoff初始化为pCutoff , 只有性能测量≥ piCutoff的要素子集才会被可视化。piCutoff的默认值为pCutoff。有时 kSolutionVis 检测到许多解决方案, 并且只有最佳piFSNum (默认值:10) 功能子集将被可视化。如果软件检测到的要素子集的数量小于piFSNum, 则所有要素子集都将可视化。 - 收集和解释软件检测到的功能, 如图 3所示。
注: 左框中的表显示检测到的要素子集及其性能测量值。前三列的名称为 "F1"、"F2" 和 "F3"。每个要素子集中的三要素都按其在一行中的排名顺序 (F1 < F2 < F3) 提供。最后一列提供了每个要素子集的性能测量 (acc或bAcc), 其列名 (acc或bAcc) 是pMeasurement的值。
7. 解释3D 散点图-使用3D 散点图可视化和解释具有相似有效二进制分类性能的特征子集
- 单击该按钮分析, 生成由软件检测到的最佳分类性能 (Acc或bAcc) 的前10个要素子集的3D 散点图, 如图 3 (中间框) 所示。按秩的升序对要素子集中的三要素进行排序, 并使用三要素的秩作为 F1/F2/F3 轴,即F1 < F2 < F3。
注: 点的颜色表示相应要素子集的二进制分类性能。数据集可能具有具有类似有效性能测量的多个要素子集。因此, 需要一个交互式和简化的散点图。 - 将值更改为0.70 在输入框pCutoff:并单击按钮分析以生成3D 散点图的要素子集与性能测量≥ piCutoff, 如图 3 (右框) 中所示。单击按钮3D 调整以打开一个新窗口, 手动调整3D 散点图的查看角度。
注: 每个要素子集的表示方式与上面的点相同。3D 散点图是以默认角度生成的。为便于3D 可视化和调谐, 可通过单击按钮3D 调整来打开一个单独的窗口。 - 单击该按钮减少以减少检测到的要素子集的冗余。
注意: 如果用户希望进一步选择功能三胞胎并最小化要素子集的冗余, 软件还会使用 mRMR 特征选择算法提供此功能。单击 "减少" 按钮后, kSolutionVis 将删除功能三胞胎中的这些冗余功能, 并重新生成表和上面提到的两个散点图。特征三胞胎的移除特征将替换为表中的关键字。F1/F2/F3 轴中无的值将被表示为piFSNum的值 (F1/F2/F3 的法线值的范围是 [1, top_x])。因此, 包含None值的点可能在3D 图中显示为 "异常点"。手动调谐的3D 图解可以在补充材料中的 "3D 点图的手动调谐" 中找到。
8. 查找基因注解及其与人类疾病的关联
注: 步骤8至10将说明如何从 DNA 和蛋白质的序列水平对基因进行注释。首先, 从32的数据库中检索上述步骤中每个生物标志物 ID 的基因符号, 然后将两个代表的 web 服务器分别用于从 DNA 和蛋白质的层次分析这个基因符号。服务器 GeneCard 提供了一个给定基因符号的全面功能注释, 而人类数据库中的在线孟德尔遗传 (OMIM) 提供了疾病-基因关联的最全面的组织。服务器 UniProtKB 是最全面的蛋白质数据库之一, 基于服务器组的预测系统 (GPS) 预测信号磷酸化是一个非常大的激酶列表。
- 将数据库 DAVID 的 web 链接复制并粘贴到 web 浏览器中, 然后打开该数据库的网页。单击图 4A中所示的链接基因 id 转换, 并输入数据集 ALL1 的第一个生物标记子集的特征 id 38319_at/38147_at/33238_at (图 4B)。单击链接基因列表, 然后单击提交列表, 如图 4B所示。检索感兴趣的注释, 然后单击显示基因列表(图 4C)。获取基因符号列表 (图 4D).
注: 此处检索的基因符号将用于后续步骤中的进一步功能注释。 - 将数据库基因卡的 web 链接复制并粘贴到 web 浏览器中, 然后打开该数据库的网页。在数据库查询输入框中搜索基因名称 CD3D, 并从基因卡33、34中查找该基因的注释, 如表 1和图 5A所示。
注: 基因卡是一个综合性的基因知识库, 提供命名、基因组学、蛋白质组学、亚细胞定位以及相关通路和其他功能模块。它还提供外部链接到各种其他生物医学数据库, 如 PDB/PDB_REDO35、Entrez 基因36、OMIM37和 UniProtKB38。如果要素名称不是标准基因符号, 请使用数据库 ENSEMBL 将其转换为39。CD3D 是基因 T 细胞受体 T3 三角洲链的名称。 - 将数据库 OMIM 的 web 链接复制并粘贴到 web 浏览器中, 然后打开该数据库的网页。搜索一个基因的名字 CD3D, 并找到这个基因的注释从数据库 OMIM37, 如表 1和图 5B所示。
注意: OMIM 现在作为人类基因与可遗传疾病的联系的最全面和权威性的来源之一。OMIM 是由维克多博士麦库西克, 以目录疾病相关的基因突变40。OMIM 现在涵盖了超过1.5万个人类基因和超过8500表型, 截至 12月1日st 2017。
9. 注释编码的蛋白质和翻译后修改
- 将数据库 UniProtKB 的 web 链接复制并粘贴到 web 浏览器中, 然后打开该数据库的网页。在 UniProtKB 的查询输入框中搜索基因名称 CD3D, 并从数据库38中查找此基因的注释, 如表 1和图 5C所示。
注: UniProtKB 收集蛋白质的丰富注解来源, 包括命名和功能信息。此数据库还提供指向其他广泛使用的数据库的外部链接, 包括 PDB/PDB_REDO35、OMIM37和 Pfam41。 - 将 web 服务器 GPS 的 web 链接复制并粘贴到 web 浏览器中, 然后打开此 web 服务器的网页。从 UniProtKB 数据库38中检索由生物标记基因 CD3D 编码的蛋白质序列, 并使用在线工具 GPS 预测蛋白质的后平移修饰 (PTM) 残留, 如表 1和图 5D所示。
注意: 生物系统是动态和复杂的, 现有数据库只收集已知信息。因此, 生物医学预测在线工具以及离线程序可能提供有用的证据来补充虚拟机制。GPS 已经开发和改进了超过12年7,42 , 可用于预测蛋白质的 PTM 残留在给定的肽序列43,44。工具也可用于各种研究课题, 包括预测蛋白质的亚细胞位置45和转录因子结合图案46等。
10. 注释蛋白质-蛋白质相互作用及其丰富的功能模块
- 将 web 服务器字符串的 web 链接复制并粘贴到 web 浏览器中, 然后打开此 web 服务器的网页。搜索 CD3D 和 P53 基因的列表, 并使用数据库字符串47查找其编排的属性。同样的过程可以使用另一个 web 服务器进行, 大卫32。
注意: 除了上述个别基因的注解外, 还有许多大型信息学工具可用于调查一组基因的性质。最近的一项研究表明, 个别不良标记基因可能构成一个大大改进的基因组5。因此, 对更复杂的生物标志物进行屏幕计算成本是值得的。数据库字符串可能会可视化已知或预测的交互连接, 而 David 服务器可能会在查询的基因47、32中检测具有重要表型关联的功能模块。还提供各种其他大型信息学分析工具。
11. 导出生成的生物标志物子集和可视化图解
- 将检测到的生物标记子集导出为. tsv 或. csv 文本文件以供进一步分析。单击按钮将表导出到所有检测到的生物标记子集的表下, 然后选择要另存为的文本格式。
- 将可视化图形导出为图像文件。单击每个图解下的 "保存" 按钮, 然后选择要另存为的图像格式。
注意: 该软件支持像素格式. png 和矢量格式. svg。像素图像很适合在计算机屏幕上显示, 而矢量图像可能会转换为期刊发布目的所需的任何分辨率。
结果
此工作流的目标 (图 6) 是检测多个具有类似效率的二进制分类数据集的生物标记子集。整个过程由两个示例数据集 ALL1 和 ALL2 从最近发布的生物标志物检测研究中提取12,48。用户可以按照补充材料中的说明安装 kSolutionVis。
数据集 ALL1 分析了 12 625 转录组功能 95 ...
讨论
本研究为用户指定的二进制分类数据集提供了易于遵循的多解决方案生物标志物检测和表征协议。该软件强调了用户友好性和灵活的导入/导出接口的各种文件格式, 使生物医学研究员可以很容易地使用软件 GUI 调查他们的数据集。本研究还强调了生成多个具有类似有效建模性能的解决方案的必要性, 以前许多现有的生物标记检测算法都忽略了这些方法。在未来, 新开发的生物标志物检测算法可能包?...
披露声明
我们没有与本报告有关的利益冲突。
致谢
这项工作得到了中国科学院战略优先研究计划 (XDB13040400) 和吉林大学启动补助金的支持。匿名审阅者和生物医学测试用户对提高 kSolutionVis 的可用性和功能的建设性意见表示赞赏。
材料
| Name | Company | Catalog Number | Comments |
| Hardware | |||
| laptop | Lenovo | X1 carbon | Any computer works. Recommended minimum configuration: 1GB extra hard disk space, 1 GB memory, 2.0MHz CPU |
| Name | Company | Catalog Number | Comments |
| Software | |||
| Python 3.0 | WingWare | Wing Personal | Any python programming and running environments support Python version 3.0 or above |
参考文献
- Heckerman, D., et al. Genetic variants associated with physical performance and anthropometry in old age: a genome-wide association study in the ilSIRENTE cohort. Scientific Reports. 7, 15879 (2017).
- Li, Z., et al. Genome-wide association analysis identifies 30 new susceptibility loci for schizophrenia. Nature Genetics. 49, 1576-1583 (2017).
- Winkler, T. W., et al. Quality control and conduct of genome-wide association meta-analyses. Nature Protocols. 9, 1192-1212 (2014).
- Harrison, R. N. S., et al. Development of multivariable models to predict change in Body Mass Index within a clinical trial population of psychotic individuals. Scientific Reports. 7, 14738 (2017).
- Liu, J., et al. Multiple similarly-well solutions exist for biomedical feature selection and classification problems. Scientific Reports. 7, 12830 (2017).
- Ye, Y., Zhang, R., Zheng, W., Liu, S., Zhou, F. RIFS: a randomly restarted incremental feature selection algorithm. Scientific Reports. 7, 13013 (2017).
- Zhou, F. F., Xue, Y., Chen, G. L., Yao, X. GPS: a novel group-based phosphorylation predicting and scoring method. Biochemical and Biophysical Research Communications. 325, 1443-1448 (2004).
- Sanchez, B. N., Wu, M., Song, P. X., Wang, W. Study design in high-dimensional classification analysis. Biostatistics. 17, 722-736 (2016).
- Shujie, M. A., Carroll, R. J., Liang, H., Xu, S. Estimation and Inference in Generalized Additive Coefficient Models for Nonlinear Interactions with High-Dimensional Covariates. Annals of Statistics. 43, 2102-2131 (2015).
- Li, J. H., et al. MiR-205 as a promising biomarker in the diagnosis and prognosis of lung cancer. Oncotarget. 8, 91938-91949 (2017).
- Lyskjaer, I., Rasmussen, M. H., Andersen, C. L. Putting a brake on stress signaling: miR-625-3p as a biomarker for choice of therapy in colorectal cancer. Epigenomics. 8, 1449-1452 (2016).
- Ge, R., et al. McTwo: a two-step feature selection algorithm based on maximal information coefficient. BMC Bioinformatics. 17, 142 (2016).
- Tumuluru, J. S., McCulloch, R. Application of Hybrid Genetic Algorithm Routine in Optimizing Food and Bioengineering Processes. Foods. 5, (2016).
- Gen, M., Cheng, R., Lin, L. . Network models and optimization: Multiobjective genetic algorithm approach. , (2008).
- Radovic, M., Ghalwash, M., Filipovic, N., Obradovic, Z. Minimum redundancy maximum relevance feature selection approach for temporal gene expression data. BMC Bioinformatics. 18, 9 (2017).
- Ciuculete, D. M., et al. A methylome-wide mQTL analysis reveals associations of methylation sites with GAD1 and HDAC3 SNPs and a general psychiatric risk score. Translational Psychiatry. 7, e1002 (2017).
- Lin, H., et al. Methylome-wide Association Study of Atrial Fibrillation in Framingham Heart Study. Scientific Reports. 7, 40377 (2017).
- Wang, S., Li, J., Yuan, F., Huang, T., Cai, Y. D. Computational method for distinguishing lysine acetylation, sumoylation, and ubiquitination using the random forest algorithm with a feature selection procedure. combinatorial chemistry & high throughput screening. , (2017).
- Zhang, Q., et al. Predicting Citrullination Sites in Protein Sequences Using mRMR Method and Random Forest Algorithm. combinatorial chemistry & high throughput screening. 20, 164-173 (2017).
- Cuena-Lombrana, A., Fois, M., Fenu, G., Cogoni, D., Bacchetta, G. The impact of climatic variations on the reproductive success of Gentiana lutea L. in a Mediterranean mountain area. International journal of biometeorology. , (2018).
- Coghe, G., et al. Fatigue, as measured using the Modified Fatigue Impact Scale, is a predictor of processing speed improvement induced by exercise in patients with multiple sclerosis: data from a randomized controlled trial. Journal of Neurology. , (2018).
- Hong, H., et al. Applying genetic algorithms to set the optimal combination of forest fire related variables and model forest fire susceptibility based on data mining models. The case of Dayu County, China. Science of the Total Environment. 630, 1044-1056 (2018).
- Borges, D. L., et al. Photoanthropometric face iridial proportions for age estimation: An investigation using features selected via a joint mutual information criterion. Forensic Science International. 284, 9-14 (2018).
- Kohavi, R., John, G. H. Wrappers for feature subset selection. Artificial intelligence. 97, 273-324 (1997).
- Yu, L., Liu, H. Efficient feature selection via analysis of relevance and redundancy. Journal of machine learning research. 5, 1205-1224 (2004).
- Wexler, R. B., Martirez, J. M. P., Rappe, A. M. Chemical Pressure-Driven Enhancement of the Hydrogen Evolving Activity of Ni2P from Nonmetal Surface Doping Interpreted via Machine Learning. Journal of American Chemical Society. , (2018).
- Wijaya, S. H., Batubara, I., Nishioka, T., Altaf-Ul-Amin, M., Kanaya, S. Metabolomic Studies of Indonesian Jamu Medicines: Prediction of Jamu Efficacy and Identification of Important Metabolites. Molecular Informatics. 36, (2017).
- Shangkuan, W. C., et al. Risk analysis of colorectal cancer incidence by gene expression analysis. PeerJ. 5, e3003 (2017).
- Chu, C. M., et al. Gene expression profiling of colorectal tumors and normal mucosa by microarrays meta-analysis using prediction analysis of microarray, artificial neural network, classification, and regression trees. Disease Markers. , 634123 (2014).
- Fleuret, F. Fast binary feature selection with conditional mutual information. Journal of Machine Learning Research. 5, 1531-1555 (2004).
- Pacheco, J., Alfaro, E., Casado, S., Gámez, M., García, N. A GRASP method for building classification trees. Expert Systems with Applications. 39, 3241-3248 (2012).
- Jiao, X., et al. DAVID-WS: a stateful web service to facilitate gene/protein list analysis. Bioinformatics. 28, 1805-1806 (2012).
- Rappaport, N., et al. Rational confederation of genes and diseases: NGS interpretation via GeneCards, MalaCards and VarElect. Biomedical Engineering OnLine. 16, 72 (2017).
- Rebhan, M., Chalifa-Caspi, V., Prilusky, J., Lancet, D. GeneCards: integrating information about genes, proteins and diseases. Trends in Genet. 13, 163 (1997).
- Joosten, R. P., Long, F., Murshudov, G. N., Perrakis, A. The PDB_REDO server for macromolecular structure model optimization. IUCrJ. 1, 213-220 (2014).
- Maglott, D., Ostell, J., Pruitt, K. D., Tatusova, T. Entrez Gene: gene-centered information at NCBI. Nucleic Acids Research. 39, D52-D57 (2011).
- Amberger, J. S., Bocchini, C. A., Schiettecatte, F., Scott, A. F., Hamosh, A. OMIM.org: Online Mendelian Inheritance in Man (OMIM(R)), an online catalog of human genes and genetic disorders. Nucleic Acids Research. 43, D789-D798 (2015).
- Boutet, E., et al. the Manually Annotated Section of the UniProt KnowledgeBase: How to Use the Entry View. Methods in Molecular Biology. 1374, 23-54 (2016).
- Zerbino, D. R., et al. Ensembl 2018. Nucleic Acids Res. , (2017).
- McKusick, V. A., Amberger, J. S. The morbid anatomy of the human genome: chromosomal location of mutations causing disease. Journal of Medical Genetics. 30, 1-26 (1993).
- Finn, R. D., et al. The Pfam protein families database: towards a more sustainable future. Nucleic Acids Research. 44, D279-D285 (2016).
- Xue, Y., et al. GPS: a comprehensive www server for phosphorylation sites prediction. Nucleic Acids Research. 33, W184-W187 (2005).
- Deng, W., et al. GPS-PAIL: prediction of lysine acetyltransferase-specific modification sites from protein sequences. Scientific Reports. 6, 39787 (2016).
- Zhao, Q., et al. GPS-SUMO: a tool for the prediction of sumoylation sites and SUMO-interaction motifs. Nucleic Acids Research. 42, W325-W330 (2014).
- Wan, S., Duan, Y., Zou, Q. HPSLPred: An Ensemble Multi-Label Classifier for Human Protein Subcellular Location Prediction with Imbalanced Source. Proteomics. 17, (2017).
- Zhang, H., Zhu, L., Huang, D. S. WSMD: weakly-supervised motif discovery in transcription factor ChIP-seq data. Scientific Reports. 7, 3217 (2017).
- Szklarczyk, D., et al. STRING v10: protein-protein interaction networks, integrated over the tree of life. Nucleic Acids Research. 43, D447-D452 (2015).
- Chiaretti, S., et al. Gene expression profile of adult T-cell acute lymphocytic leukemia identifies distinct subsets of patients with different response to therapy and survival. Blood. 103, 2771-2778 (2004).
- Rowley, J. D., et al. Mapping chromosome band 11q23 in human acute leukemia with biotinylated probes: identification of 11q23 translocation breakpoints with a yeast artificial chromosome. Proceedings of the National Academy of Sciences of the United States of America. 87, 9358-9362 (1990).
- Rabbitts, T. H., et al. The chromosomal location of T-cell receptor genes and a T cell rearranging gene: possible correlation with specific translocations in human T cell leukaemia. Embo Journal. 4, 1461-1465 (1985).
- Yin, L., et al. SH2D1A mutation analysis for diagnosis of XLP in typical and atypical patients. Human Genetics. 105, 501-505 (1999).
- Brandau, O., et al. Epstein-Barr virus-negative boys with non-Hodgkin lymphoma are mutated in the SH2D1A gene, as are patients with X-linked lymphoproliferative disease (XLP). Human Molecular Genetics. 8, 2407-2413 (1999).
- Burnett, R. C., Thirman, M. J., Rowley, J. D., Diaz, M. O. Molecular analysis of the T-cell acute lymphoblastic leukemia-associated t(1;7)(p34;q34) that fuses LCK and TCRB. Blood. 84, 1232-1236 (1994).
- Taylor, G. M., et al. Genetic susceptibility to childhood common acute lymphoblastic leukaemia is associated with polymorphic peptide-binding pocket profiles in HLA-DPB1*0201. Human Molecular Genetics. 11, 1585-1597 (2002).
- Wadia, P. P., et al. Antibodies specifically target AML antigen NuSAP1 after allogeneic bone marrow transplantation. Blood. 115, 2077-2087 (2010).
- Wilson, D. M., et al. 3rd et al. Hex1: a new human Rad2 nuclease family member with homology to yeast exonuclease 1. Nucleic Acids Research. 26, 3762-3768 (1998).
- O'Sullivan, R. J., et al. Rapid induction of alternative lengthening of telomeres by depletion of the histone chaperone ASF1. Nature Structural & Molecular Biology. 21, 167-174 (2014).
- Lee-Sherick, A. B., et al. Aberrant Mer receptor tyrosine kinase expression contributes to leukemogenesis in acute myeloid leukemia. Oncogene. 32, 5359-5368 (2013).
- Guyon, I., Elisseeff, A. An introduction to variable and feature selection. Journal of machine learning research. 3, 1157-1182 (2003).
- John, G. H., Kohavi, R., Pfleger, K. . Machine learning: proceedings of the eleventh international conference. , 121-129 (1994).
- Jain, A., Zongker, D. Feature selection: Evaluation, application, and small sample performance. IEEE transactions on pattern analysis and machine intelligence. 19, 153-158 (1997).
- Taylor, S. L., Kim, K. A jackknife and voting classifier approach to feature selection and classification. Cancer Informatics. 10, 133-147 (2011).
- Andresen, K., et al. Novel target genes and a valid biomarker panel identified for cholangiocarcinoma. Epigenetics. 7, 1249-1257 (2012).
- Guo, P., et al. Gene expression profile based classification models of psoriasis. Genomics. 103, 48-55 (2014).
- Xie, J., Wang, C. Using support vector machines with a novel hybrid feature selection method for diagnosis of erythemato-squamous diseases. Expert Systems with Applications. 38, 5809-5815 (2011).
- Zou, Q., Zeng, J., Cao, L., Ji, R. A novel features ranking metric with application to scalable visual and bioinformatics data classification. Neurocomputing. 173, 346-354 (2016).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。