Method Article
基于多重 CRISPR 增强器的细胞系干扰增强功能解剖
摘要
本协议描述了设计和执行多路化增强剂的步骤, 该方法与停用融合蛋白 SID4X-dCas9-KRAB (即增强器干扰) 相结合。该协议能够识别出调节基因表达的增强剂, 并促进调节共同靶基因的促促剂之间的关系的解剖。
摘要
多促进剂经常调节一个给定的基因, 但对于大多数基因, 它仍然不清楚哪些促进剂是必要的基因表达, 以及这些增强剂如何结合产生转录反应。随着数以百万计的促进剂被确定, 需要高通量的工具来确定增强功能的基因组范围。目前研究增强功能的方法包括使用核酸酶熟练的 Cas9 进行基因缺失, 但要研究多种促发剂的组合效应, 还是很难的, 因为多重连续克隆细胞系必须生成。在这里, 我们提出了一种基于 CRISPR 干涉的方法, 它允许在其内源位置同时对多个增强剂进行功能性审问。增强器-i 利用两个压制域融合到核酸酶缺乏 Cas9, SID 和 KRAB, 以实现增强失活通过组蛋白脱乙酰在靶位点。该协议利用导 rna 的瞬态转染, 使靶区的瞬态灭活, 尤其有效地阻断诱导转录反应的刺激在组织培养设置。增强剂-i 是高度特异的, 无论是在其基因组定位和它对全球基因表达的影响。从本协议获得的结果有助于了解增强剂是否有助于基因表达、贡献的大小, 以及其他附近促进剂对其贡献的影响。
引言
大规模的排序项目 (如编码1、路线图 Epigenomics2和幽灵3 ) 在人类基因组中对数以百计的细胞类型进行了数以百万计的假定增强。据估计, 每一个启动子与平均4.9 促进剂, 每个增强剂接触平均2.4 基因3, 表明基因表达往往是多种分布式调节相互作用的综合结果。还有一个重要的挑战是, 不仅要定义个体增强剂如何促进基因表达, 而且还要确定它们是如何结合来影响表达的。遗传方法通常用于确定模型有机体中的增强剂与果蝇4到小鼠5之间的关系。然而, 这些实验是耗时和低吞吐量的研究多促进剂在多个基因。
研究增强函数的一种方法是大规模的并行报道。这些化验可以同时筛选数以千计的 DNA 序列, 使其能够驱动表达的记者基因6。虽然这些化验结果表明, DNA 序列可以单独足以传达基因调控信息7, 他们来与在本地染色质上下文之外和与异源启动子进行的警告。此外, 在大量平行报道中分析的 DNA 序列的大小通常少于 200 basepairs, 可能排除相关的周围序列。重要的是, 由于记者的化验只是一次测量一个序列的活动, 他们没有考虑到在促进剂之间可能存在的复杂关系。因此, 虽然大量平行的记者化验可以是关于 dna 序列的内在活动的信息, 但他们不一定告诉我们的功能, 该 dna 序列的范围内的基因组。
最近开发的 CRISPR/Cas9 工具8促进了基因调控的研究, 因为它们允许在内源轨迹中删除增强剂。然而, 同时删除多个增强剂可能会导致基因组不稳定, 并且在单个细胞线中生成连续的强化剂删除是很耗时的。此外, 在修复后的删除站点上创建了新的基因组序列, 这一序列可能获得调节功能。Cas9 的另一种版本是专门用于调制基因表达的, 依赖于激活9、10或抑制11、12域到核酸酶不足形式 Cas9 的融合。(dCas9)。这些融合蛋白是理想的同时研究多个基因座, 因为它们不会物理改变 DNA 序列, 而是调整表观遗传学, 以询问一个调控区域。最广泛使用的压制融合是 KRAB, 招募 KAP1 联合阻遏复合体, 促进抑制相关组蛋白 H3 赖氨酸 9 trimethylation (H3K9me3)13的沉积。dCas9-KRAB, 也称为 CRISPR 干扰14, 已被用于目标和屏幕上的个体增强剂为他们的贡献基因表达15,16;但是, 它还没有针对多个区域同时进行优化。一个版本的复用 CRISPR 干扰促进剂, 马赛克序列17, 使用单细胞 RNA 序列作为读数, 但这种技术是昂贵的, 只适合研究高表达基因由于低灵敏度的单细胞RNA-
我们试图开发一种基于 CRISPR 干涉的方法来解剖组合增强函数在雌激素转录反应的背景下。大约一半的雌激素应答基因含有2或更多的增强剂, 受雌激素受体α (ER) 附近的18的约束, 这表明多重促愈剂可能参与雌激素的反应, 理解调控逻辑需要同时瞄准多个增强剂。在启动器中使用 CRISPR 干扰的初步研究表明, 并非所有的启动子都同样响应 KRAB 介导的镇压19, 我们推断, 增加一个独特的压制域, 以 dCas9 可以促进失活多样的促进剂。我们选择了 Mad1 (SID)20的 Sin3a 交互域, 因为它导致了组蛋白乙酰21的招募, 这将移除与转录活动相关的组织蛋白上的乙酰基团。重要的是, SID 域在将基因表达式融合到 dCas922和故事23时是有效的, Sin3a 在各种增强程序序列上下文24中被证明是一个强有力的压制性的共同因素。我们使用 SID4x-dCas9-KRAB (增强剂 i) 靶向10种不同的促进剂绑定的 er, 并确定 er 结合点 (ERBS), 是必要的雌激素转录应答的4基因18。我们还瞄准了促进剂的组合, 以确定在雌激素转录应答的生产中合作的地点。我们发现, 多达50个站点可能同时具有可检测的基因表达变化的目标。利用芯片序列和 RNA 序列, 我们证明了增强剂 i 是一种高度特异的技术, 同时研究多促能器。
在本协议中, 我们描述了执行增强器 i 的步骤, 这是一种灵活的技术, 能够同时在组织培养环境中同时进行多种促进剂的功能研究。增强器-i 与基因删除高度相关, 但提供了依赖于组蛋白乙酰 (HDACs) 的瞬态失活。通过通过瞬变转染提供导 rna, 而不是通过病毒载体稳定集成, 该协议避免了 H3K9me3 的沉积和潜在扩散。本协议详细介绍了通过吉布森组装进行 RNA 的设计和克隆, 利用 lipofection 对导 rna 进行转染, 并通过 qPCR 对由此产生的基因表达变化进行分析。我们还包括评估增强剂的特异性的方法-i 靶向基因组和转录的水平。该技术是在人类癌细胞系中应用 ER 结合促进剂研究基因调控的同时, 适用于任何哺乳动物增强剂的解剖。
研究方案
1. 细胞系稳定表达 SID4X-dCas9-KRAB 的生成
注: 在这里提出的转染条件和药物浓度已优化为石川细胞, 子宫内膜癌细胞系, 生长在 RPMI 1640 媒体补充10% 血清和1% 青霉素/链霉素 (完全 RPMI)。其他细胞系可能需要不同的转染条件和药物浓度。用户还可以在野生型细胞中进行瞬态转染实验, 而不是生成稳定的细胞系, 用质粒表达 SID4X-dCas9-KRAB 和引导 RNA 表示粒;然而, 瞬态 transfections 的结果可能难以重现, 因为 SID4X-dCas9-KRAB 的水平可能因转染而异。
- 在 30-50% 汇合 (大约30万石川细胞) 在3毫升完全 RPMI 的6井板材至少2个井的板材石川细胞。
- 从细胞中吸取介质。用 1x PBS (pH 7.4) 清洗细胞一次。吸入 PBS 和添加胰蛋白酶 (4 毫升的10厘米菜或5毫升的 T-75 瓶)。
- 在37摄氏度孵化细胞5分钟, 每2分钟检查分离细胞, 轻轻摇动血管。
- 一旦细胞分离, 吸管 trypsinized 细胞向上和向下几次, 和吸管轻轻地沿着血管的一侧释放任何附着的细胞。
- 将细胞转移到15毫升锥形管, 在 250 x g 处旋转细胞向下5分钟。
- 在 5-10 毫升的培养基中吸入胰蛋白酶并并用重悬细胞。如有必要, 使用 P1000 吸管分离细胞团簇。
- 计数单元格, 并确定所需的体积为30万细胞每井在总容积3毫升。将单元格添加到2个6井板的独立井中。用完整的 RPMI 填充每个井到3毫升。
- 在电镀后的前15分钟内, 每隔5分钟轻轻摇动盘子, 以确保电池均匀分布在板材上。使用显微镜, 以确保细胞从井中间分散。
- 在24小时内电镀, 执行以下 transfections 使用适当的转染试剂为细胞线感兴趣。对于石川细胞, 请按照下面所述的步骤操作。
注: 本协议假定采用阳离子脂质体为基础的转染试剂。电穿孔为细胞类型提供了一种替代方法, 对这些试剂非常敏感, 或具有 lipofection 的低转染效率。在尝试增强剂 i 试验之前, 必须对所感兴趣的细胞线进行转染条件的优化。- 在1.7 毫升离心管中, 稀释2.5 微克 SID4X-dCas9-KRAB 质粒和 800 ng 质粒, 将荧光蛋白转化为无血清介质, 使管中的最终体积为155µL, 质粒的最终浓度为0.020 µg/µL。
- 在另一管中, 稀释3.3 µg 的质粒, 不含有新霉素电阻盒, 如 pCMV-GFP, 在无血清的介质中, 使管中的最终体积为155µL, 质粒的最终浓度为0.020 µg/µL。
- 用离心涡旋每管, 并向下旋转。
- 将转染试剂 (材料表) 的9.9 µL 添加到每个管中。涡流简单地混合在低速。用离心旋转管子。
- 室温下将试管孵化至少5分钟, 但不超过20分钟。
- 在生物安全柜, 添加150µL 的准备 DNA: 试剂混合滴到一个井在6井板。重复的另一管制备的 DNA: 试剂组合。轻轻地旋转盘子, 把盘子放回孵化器。
- 2天后转染, 改变媒体和补充与 G418 到最后集中的 600 ng/ul。这种浓度可能需要优化的细胞类型。
- 改变完整的 RPMI 媒体, 并补充与 G418 每隔一天 2-4 周, 直到控制转染的细胞死亡, 以及含 SID4X-dCas9-KRAB 的水井变得汇合。细胞恢复所需的确切时间将取决于细胞的加倍时间。
- 当细胞变得汇合时, 通过一个 T-25 或 T-75 容器完全 RPMI 与较低剂量 G418 (300 ng/ul 为石川细胞)。在这段经文中, 分别将2整除数10万细胞 (大约1/10 个6井板的 th) 成2个分别为 RNA 和 DNA 隔离的1.7 毫升离心管.旋转这些管子 (5 分钟, 250 x g), 去除胰蛋白酶由吹打, 并且冷冻管子在-20 °c 为将来使用。
- 使用商用试剂盒分离基因组 DNA, 使用 "pAC95_PCR" 或 "SID4X_PCR" 引物 (表 1) 进行 PCR, 以验证细胞系内是否存在融合蛋白。将基因组 dna 从亲本线提取为阴性对照, SID4x-dCas9-KRAB 质粒 dna 作为阳性对照。使用高保真聚合酶大师组合与 50-100 ng 基因组 DNA 和以下循环条件:98 °c 为三十年代, 25 个周期 (98 °c 为十年代, 58 °c 为三十年代, 72 °c 为2分钟), 72 °c 为5分钟, 保持在摄氏4摄氏度。
- 为了验证融合蛋白在 RNA 水平上的表达, 请使用商业上可用的试剂盒从细胞线提取的 rna 进行 qPCR。使用 "dCas9_qPCR" 底漆 (表 1) 和本协议步骤6.3 中提供的一步 qPCR 协议。
- 为了验证融合蛋白水平的表达, 在细胞系上对裂解物进行一个西方印迹。使用反旗或抗 HA 抗体检测融合蛋白。
2. 指南 RNA 设计
注: 本协议设计用于 U6 指南 RNA 克隆载体, 由教会实验室创建, 可在 Addgene (Addgene 41824) 上使用。要创建包含与41824相同克隆策略的嘌呤霉素电阻的此向量的版本, 我们将多个克隆站点从该向量移动到 pGL3-U6-sgRNA-PGK-puromycin 向量 (Addgene 51133) 中。无论是 Addgene 41824 或我们的版本与嘌呤霉素 (Addgene 106404) 都符合以下概述的克隆战略。
- 获得 600-900 basepairs 的 DNA 序列为每个调节区域的利益。使用转录因子绑定站点和/或染色质可访问性, 以指导有关在何处定义感兴趣区域 (图 2A)。
注意: 虽然图 2A中的示例具有上游和下游增强功能, 但也可以针对位于内含子内的管理元素。 - 使用 FASTA 格式将在单个文本文件中获取的所有序列放在一起。
- 确定至少一个负面控制区域, 在实验条件下不应发生变化, 如基因的启动子没有在感兴趣的细胞线中表达。获取此区域的 DNA 序列, 并将其添加到 FASTA 格式的文本文件中。
注意: 我们使用引导 rna 瞄准IL1RN启动子25作为对我们目标的所有区域的负控制。用户还可以选择不包含转录因子绑定站点的感兴趣区域附近的基因间序列作为负控制。然而, 如果多个定位点同时被瞄准, 一个负控制区域简化实验设计和结果解释。如果靶向增强器是内含子的, 那么将内含子区域定位在同一个轨迹上, 而不包含一个假设的调控元素作为额外的负控制可能是有用的, 因为 dCas9 融合可能会干扰转录。 - 确定积极的控制区域, 例如作为感兴趣的监管区域的假定目标的发起人, 或在感兴趣的细胞线上高度转录的基因的发起人。获取这些区域的 DNA 序列, 并将其添加到 FASTA 格式的文本文件中。
- 在生成的 DNA 序列上使用诸如 e 脆26 (http://www.e-crisp.org/E-CRISP/) 的程序, 以找到低目标的导 rna (理想情况下为 0-3)。导 rna 由一个 protospacer 相邻的母题 (PAM) 上游的20核苷酸组成, 它从化脓中获取 dCas9 的形式 "NGG"。
- 在电子脆化网站上, 使用下拉菜单选择感兴趣的有机体。基因组组件出现在物种名称的右侧。
- 选择输入为 FASTA 序列单选按钮。从上面复制 FASTA 序列并将它们粘贴到对话框中。确保每个序列都包含 FASTA 标头。
注: 最多可同时查询多达50个序列。 - 在下拉菜单中选择中单选按钮和单个设计。
- 单击按钮开始 sgRNA 搜索。将会打开一个新的浏览器选项卡, 结果将显示出来。通过单击按钮将所有查询序列的 Excel 格式化表格报表下载到一起, 下载候选序列。
- 使用 Excel 或文本编辑程序打开表格式报表。
- 使用 UCSC 基因组浏览器 BLAT 候选全长 gRNA 序列 (23 basepairs) 的基因组。
- 在浏览器中, 导航到 UCSC 基因组浏览器网站 (http://genome.ucsc.edu)。在我们的工具部分下, 找到单词BLAT并单击它。将会打开 BLAT 搜索工具。
- 使用位于BLAT 搜索基因组文本下的下拉菜单选择感兴趣的有机体和基因组组件。
- 从电子脆片生成的表格式报告中复制指南 RNA 序列, 并将其粘贴到对话框中。确保每个序列都有一个唯一的 FASTA 头, 然后单击对话框底部的提交。
注: 最多可同时检查25个序列。在 " BLAT 搜索结果" 页上, 将显示每个指南 RNA 序列的对齐方式, 每行表示一个对准。理想的情况是, 每个指南 rna 应该有一个对齐, 表明该导 rna 的唯一性。 - 如果可能, 避免与基因组中多个位置对齐的参考线。
- 要检查指南 RNA 在感兴趣区域内的定位和分布, 请单击操作部分下的浏览器链接, 查看查询的指南 rna 之一。基因组浏览器将出现, 并将集中在选定的指南 RNA。使用页面顶部的缩小按钮可视化在感兴趣区域内由电子脆识别的其他指南 rna 的分布情况。
- 选择4最好的非重叠指南 RNA 序列分布在整个感兴趣的区域 (图 2B)。如果感兴趣的区域超过 600 bp, 考虑增加 1-2 其他指南。避免引导 rna 与 homopolymeric 伸展和极端 GC 的内容, 因为这些功能可能会阻碍指南核糖核酸克隆过程, 并减少引导 rna 靶向效率。
- 选择参考线后, 为每个所需的指南创建一个包含完整指南 RNA 序列 (23 核苷酸) 的文件, 然后从 3 ' 端移除 5 ' 核苷酸以及 PAM (NGG)。这一步骤有助于寡核苷酸排序。
- 将以下序列添加到寡核苷酸序列的 5 ' 末端: GTGGAAAGGACGAAACACCG。
- 将以下序列添加到寡核苷酸序列的 3 ' 末端: GTTTTAGAGCTAGAAATAGC。
注: 最后的序列应该是59核苷酸长, 看起来像这样: GTGGAAAGGACGAAACACCG 目标 (19 nt)-GTTTTAGAGCTAGAAATAGC。 - 确保每个管理元素的目标与增强剂-我有至少4独特的寡核苷酸为它设计。订购这些序列连同表 1中列出的 "U6_internal" 底漆。
3. 指南 RNA 克隆
注: 指南 RNA 克隆通过吉布森组装已经证明是高效的, 在我们的手中, 产生数以百计的殖民地每板块, 很少, 如果任何殖民地只存在于向量控制。这种效率对于在汇集克隆过程中保持复杂性至关重要。吉布森组装克隆的另一个优点是, 用户不必担心在其试图插入 U6 克隆载体的指南 RNA 中存在限制酶切割站点。尽管如此, 如果需要, 该协议可以根据传统的限制酶克隆进行修改。
- 重建指南 RNA 寡核苷酸在最后集中100微米在超纯水 (RNase, 无 DNase)。每个感兴趣的区域应该至少有4个单独的指南 RNA 寡核苷酸。
- 对于每个感兴趣的管理区域, 创建一个与感兴趣区域对应的所有寡核苷酸的池。在离心管中, 结合每个区域的每个个体重组导 RNA 寡核苷酸的 5 ul。混合池涡流, 然后删除 1 ul 和稀释这整除1:200 在超纯水。
注意: 如果需要, 这些针对单个管理区域的池可以进一步组合以生成针对多个区域的复杂池。最多可在单个池中同时锁定多达50个管理区域 (图 3C)。 - 使用 U6 引物进行短 PCR, 在吉布森组装之前将同源区域附加到寡核苷酸。大约40个基地将被添加到每个寡聚, 产生一个 ~ 100 bp 产品, 包含足够的同源性的 U6 向量两端。
- 对于每个指南 RNA 池, 建立了一个 20 ul PCR 与高保真聚合酶主混合和以下组件: 1 ul 稀释寡糖池从步骤 3.2, 1 ul 的 U6 前底漆 (10 微米), 1 ul U6 反向底漆 (10 微米), 水高达 20 ul。
- 孵育在热循环仪以以下情况:98 °c 为三十年代, 10 个周期 (98 °c 为十年代, 55 °c 为三十年代, 72 °c 为2分钟), 72 °c 为5分钟, 并且举行在4°c。
- 运行 5 ul 的反应在 1-2% 琼脂糖凝胶与低分子量阶梯。最终产品应该是 ~ 100 basepairs (图 2C)。
- 用基于柱的 DNA 纯化试剂盒清理延伸反应, 并在试剂盒中提供的 20 ul 洗脱缓冲器中洗脱。
注: 由于产品短, 避免使用珠基清洁, 这是设计, 以排除小碎片小于 100 bp。 - 使用荧光计或分光光度计量化纯化的 DNA (预期产量为 10-20 ng/ul)。指南 RNA 插入物可以存储在-20 °c, 或可以立即使用在吉布森装配与线性化的 U6 向量。
- 为了准备 U6 克隆载体为吉布森组装, 建立了限制酶文摘。如果要执行许多吉布森装配反应, 建立多个文摘, 以确保切割载体的充分屈服。
- 使用20单位的 AflII 酶和1微克的质粒在 20 ul 反应与适当的限制酶缓冲。孵育37°c 为1-2 小时。
- 在 20 ul 的洗脱缓冲器中, 用珠子或以柱为基础的 DNA 纯化和洗脱的试剂盒清理文摘。使用荧光计或分光光度计量化纯化的 DNA。样品可以冻结在-20 摄氏度以后使用。
- 在准备好的向量上执行吉布森组件并插入。
- 在冰上设置吉布森装配反应。使用 50 ng 的向量和 7 ng 的插入在 20 ul 反应。稀释在超纯水中插入 1:10, 以方便吹打。建立一个仅向量吉布森装配反应, 使用 50 ng 的载体和取代插入与水。
- 孵化的吉布森组装反应为15分钟在50摄氏度, 其次是保持在4摄氏度。
- 把组装的产品转移到冰上。在冰上用超纯水稀释组装后的产品1:4。例如, 将吉布森装配产品的 5 ul 添加到 15 ul 的超纯水中。
- 转换稀释的吉布森组装产品。
- 解冻高效的能力细胞在冰上, 使 25 ul 整除数每一个转变。如果需要针对多个站点的复杂池, 请将足够的单元格解冻到不同的管道中, 以执行同一复杂 gRNA 池的多个独立转换。
- 对于每个稀释的产品, 添加 1 ul 的这种稀释到一个1.7 毫升离心管含有 25 ul 的主管细胞。混合通过简单地弹管。将管子在冰上孵化30分钟。
- 热休克细胞为三十年代在42°c, 然后立刻转移到冰为2分钟。
- 添加 300 ul SOC 介质 (2% 胰蛋白胨, 0.5% 酵母萃取物, 10 毫米氯化钠, 2.5 毫米氯化钾, 10 毫米氯化镁 2, 10 毫米 MgSO 4 和20毫米葡萄糖), 并让细胞恢复 1 h 在37°c 与震动 (300 rpm).在此期间, 加热琼脂板与氨苄西林/羧苄青霉素到37°c 在一个孵化器。每个变换使用一个盘子。
- 板 50 ul 的细胞和放置在一个37摄氏度孵化器一夜之间的板块。对于针对单个站点的池, 直接放置在含有氨苄西林/羧苄青霉素 (1 毫克/毫升) 的 3-5 毫升磅肉汤(材料表) 中, 并在一夜之间以 250 rpm 在37°c 为 minipreps 进行孵化。
- 收获细胞并分离 DNA。
- 对于针对多个站点的大型引导 RNA 库, 请使用一个刮刀将各个板块的所有菌落收集成一个 maxiprep (150 毫升液体培养)。这可以通过将5毫升的磅与适当的抗生素倒入50毫升猎鹰管中, 并将菌落刮入管子来促进。对于针对单个站点的库, 将板块刮成一个 miniprep (3-5 毫升液体培养)。
- 孵化这些文化与适当的抗生素为 3-5 小时在37°c 与震动在250转每分钟。
- 使用试剂盒进行 DNA 提取, 结果无内毒素眼影。
- 使用荧光计或分光光度计量化 DNA。质粒可以立即用于转染或储存在-20 °c, 以供将来使用。
- 为确定针对单个站点的小池的 U6 向量中的导 RNA 序列是否存在, 请使用表 1中列出的 "U6_PCR_R" 底漆在已准备好的 miniprep 上进行的桑格排序。由于聚集的导 rna, 19 basepair gRNA 目标序列将产生混合基地, 但 U6 促进者和指南 RNA 脚手架围绕这个序列应该是完整的。
4. 转染增强剂 i
注: 为了成功地阻断雌激素的反应, 使用增强剂 i 在石川细胞, 有必要在 5-7 天前剥夺雌激素的细胞转染, 保持在酚红色自由 RPMI 与10% 木炭剥离血清和1%青霉素/链霉素。如果试图阻止雌激素的反应, 细胞在转染过程中和后应培养。我们建议使用苯酚红游离胰蛋白酶在完全酚红游离 RPMI 细胞的通路。
- 在转染前的前一天, 将细胞 (野生型或稳定表达 SID4X-dCas9-KRAB) 在24井板上, 在 30-50% 融合 (为石川细胞每井6万个细胞)。板足够的细胞, 这样 transfections 可以重复执行, 包括用控制引导 rna 转染的水井。 确保细胞均匀分布在井中, 通过轻轻摇动板后, 细胞电镀和步骤1.1.7。
注: 本协议假定采用阳离子脂质体为基础的转染试剂。电穿孔为细胞类型提供了一种对这些试剂非常敏感的替代方法。在尝试增强剂 i 试验之前, 必须对所感兴趣的细胞线进行转染条件的优化。 - 第二天, 根据选择的转染试剂的指示, 准备 transfections。对于石川细胞, 对24井板的每一个井, 使用总质粒的 550 ng。将质粒稀释为0.020 微克/ul 在无血清介质中的最终浓度 (1.1 微克的 DNA 在转染2井总容积的 52 ul 中)。使用 3 ul 的转染试剂, 每1微克的 DNA, 漩涡和孵化, 如步骤1.2 所述。将最终混合物的 25 ul 添加到每个井中。
注: 为了目标组合的网站, 使用相同重量的质粒为每个单独的站点, 然后用控制质粒填充剩余的重量 (空指南 rna 克隆载体或指南 rna 瞄准消极控制区域例如IL1RN启动子)。对于瞬态 transfections, 使用 3:2 Cas9 融合的比值: 引导 RNA 质粒。可以添加含有荧光记者的质粒来监测转染效率。 - 在 36 h 后转染, 改变介质使用酚红色游离 RPMI 与10% 木炭剥离的血清和1% 青霉素/链霉素 (为石川细胞) 和供应嘌呤霉素 (最终浓度: 1 微克/毫升) 和新霉素 (最终浓度: 300 ng/毫升)。如果细胞对转染试剂敏感, 则可以更早地改变培养基, 但不应在24小时后再转染抗生素。
注: 在收割细胞前添加抗生素至少要等待24小时。表达式的变化由于增强剂-我可以发现早在48小时后转染和5天后转染。如果与已被剥夺雌激素的石川细胞合作, 在抗生素治疗后的一天执行 8-小时 10 nM 17β雌二醇 (E2) 诱导, 然后立即收获细胞。
5. 细胞收获和 RNA 提取
- 用1% β巯基乙醇 (BME) 制备裂解缓冲液。确保有足够的裂解-BME 混合物 (300 ul 为每个井被收获)。
- 使用吸尘器吸入介质。
- 用同样数量的 1x pbs (500 ul) 和吸吸来洗涤细胞一次, 以尽可能多地去除 pbs。
- 使用多通道吸管在每个井中添加 300 ul 的裂解 BME 溶液。把溶解液的 8-10 倍的吸管, 并转移到一个深井板或1.7 毫升离心管在冰上。RNA 可以立刻被提取, 或者裂解物可以被冷冻在-80 °c 为将来处理。
- 要从裂解物中提取 RNA, 请使用包括 DNase 治疗在内的商业上可用的工具包。洗脱最小推荐体积的超纯水 (无 RNase, 无 DNase) 或洗脱缓冲和量化 RNA。对于少量的样品, 使用荧光计或分光光度计。对于大量的样品, 使用荧光探针检测 RNA 和测量在一个板块阅读器。样品可在-80 摄氏度之前或之后冻结。
6. 使用一步法 qPCR 和 RNA 序列量化基因表达变化
- 获得 qPCR 引物的兴趣基因和至少一个管家基因表达附近的目标基因水平, 并没有改变, 在实验条件。理想情况下, 这些引物将跨越一个外显子-显子的连接, 以避免扩增基因组 DNA。
- 测试这些引物在 RNA 获得从细胞线感兴趣。使用熔体曲线分析验证单个产品的生产。如果没有生产单个产品, 则测试附加的底漆对。
- 对于每个增强 i 和控制指南 RNA 处理的样本, 确定有多少基因必须在该样本中测定。这组基因应该包括管家基因, 如CTCF或GAPDH。
- 建立 qPCR 反应。
- 将所有样品稀释到相同浓度的水中, 这样50的总 RNA 很容易 pipetted, 每个反应都有足够的稀释 rna。例如, 稀释 rna 到 16.6 ng/ul 和使用 3 ul 的 rna 在每个反应。在设置主混合时, 将 RNA 放在冰上。
- 为每个基因准备单独的主混合, 用商用的一步 qPCR 套件来测量。使用 20 ul 反应量与每个底漆 1 ul (10 微米库存解决方案)。在冰上设置这些反应。
- 在适合于热循环仪的反应板中, 添加 RNA 样本, 后跟主混合。用印版封口机密封, 用涡流或吹打轻轻搅拌。简要地离心板 (140 x g 六十年代) 确保液体在井的底部。
- 孵育板材在热循环仪如下 (或作为套件指示):48 °c 为30分钟, 95 °c 为10分钟, 40 个周期 (95 °c 为十五年代, 60 °c 为1分钟)。
- 获取每个样本中测量的每个基因的 Ct 值。采用比较 Ct 的方法确定基因表达的变化。
- 从每个样本感兴趣的每个基因的 ct 中减去管家基因的 ct, 生成规范化的 ct 值。
- 对于对照治疗样品, 取平均每种基因的规范化 Ct 值。对数基 2-规模褶皱镇压然后可以计算通过减去规范化的增强剂-i 治疗样本 Ct 为每个基因从相同的价值, 对照治疗样本的对应基因。
- 为了确定基因表达的全球变化后促进剂 i 治疗, 准备样品的 RNA 排序使用一个商业上可用的套件兼容用户的测序技术。使用500的 RNA 用于启动材料, 并为至少2个生物复制准备库。
7. 用芯片序列验证 SID4X-dCas9-KRAB 的特定基因组靶向性
注: SID4X-dCas9-KRAB 融合蛋白同时含有标志位位标记和 HA 表位标记, 但用抗旗抗体获得最佳的芯片序列结果。如果需要, 用户可以执行额外的芯片序列实验的转录因子可能受增强剂 i, 或 H3K27ac, 一个标记的增强活性, 减少的增强剂-i。但是, 每个芯片序列实验都需要 10 x 106单元格, 因此应相应地进行规划。
- 染细胞与增强剂 i. 池。
- 板 10 x 106细胞在 15 cm 组织培养皿。每道菜代表1个芯片序列实验的1因素的兴趣。
- 第二天, 染细胞使用每盘20微克的总 DNA。对于稳定表达 SID4X-dCas9-KRAB 的细胞系中的 transfections, DNA 应该是引导 rna 的质粒池, 目标是所有感兴趣的部位, 也可以选择一种表达荧光蛋白的质粒。对于瞬态 transfections, 使用 3:2 dCas9 融合蛋白的比值: 引导 RNA 池。对于其他 TFs 或组蛋白修饰的芯片序列, 执行至少一个附加控制指南 RNA 转染另一盘。
- 将嘌呤霉素 (1 微克/毫升) 和新霉素 (300 ng/毫升) 的菜肴用 24-48 h 后转染处理。在收获染色质之前至少要等24小时。
- 从盘子里收获染色质。
注: 研究增强 i 对石川细胞 ER 基因组结合的影响, 对转染对照导 rna 和增强剂的菜肴进行1小时10纳米 E2 治疗, 收获前。为了研究增强剂 i 对 H3K27ac 的影响, 在收割前用对照导 rna 和增强剂转染的盘子上执行 8 h 10 nM E2 诱导。- 对每道菜应用 500 ul 37% 甲醛 (最后浓度为 1%)。简单地旋转盘子。让盘子在室温下坐10分钟。
- 加入1毫升的2.5 米甘氨酸 (最后浓度为125毫米)。简单地旋转盘子。
- 用甲醛和甘氨酸把培养基倒掉。添加一个相同体积 (20 毫升) 的冷 1x PBS。
- 把 PBS 倒掉吸入吸尘器, 以消除尽可能多的 PBS。把盘子放在冰上。
- 添加 3-5 毫升的冷 1x PBS 或法纳姆裂解缓冲 (5mM 管 pH 值 8.0, 85 毫米氯化钾, 0.5% NP-40) 与1x 蛋白酶抑制剂 (刚使用前添加) 每个板块。用刮刀刮盘子, 将溶液转移到冰上的15毫升锥形管上。
- 颗粒染色质通过在离心机中旋转向下管5分钟, 在4摄氏度, 1000 x g. 丢弃上清, 并将小球储存在-80 °c 以供将来使用, 或使用反旗抗体或抗体来进行选择的芯片序列协议转录因子或组蛋白修饰的兴趣 (H3K27ac, H3K9me3)。
结果
图 1显示了协议中描述的工作流示意图。为了确定在雌激素调控基因MMP17附近的 ER 增强增强剂的贡献, 它有3个结合点在附近由芯片序列 (图 2A) 定义, 指南 rna 为每个区域被设计了。为了设计导 rna, 选择了一个 600-900 bp 围绕每个 ER 结合点的序列的窗口, 并将其放入向导 RNA 设计程序中。结果引导 RNA 序列与0-2 被预言的目标站点被排列了对人类基因组使用 BLAT。为确定目标 (图 2B), 选择了四个跨越了芯片序列和 DNaseI 超敏的区域的非重叠导 rna。添加了附加序列 (表 1), 以方便下游克隆, 并订购了所产生的59核苷酸片段。到达后, 引导 rna 被稀释和聚集的站点, 并进行了短期 PCR 添加同源区域之前, 吉布森组装。图 2C在使用 "U6_internal" 引物 (表 1) 的短 PCR 后显示预期的指南 rna 产品, 它将在 59 basepair 指南 RNA 片段的每一端添加 20 basepairs 序列, 从而产生 100 basepair 序列。在吉布森组装后, 这些导 RNA 池被转化为细菌, 并在第二天制备质粒 minipreps。图 2D显示了增强器解剖实验的结果, 在该试验中, 在MMP17附近的多个增强剂是单独使用的, 并且是用促进剂 i 进行组合的。以增强器为目标的站点-我用黑色六边形表示。引导 RNA 质粒靶向表明的地点被转染成雌激素剥夺的石川细胞系稳定表达 SID4X-dCas9-KRAB。两天后, 媒体被改变, 嘌呤霉素被添加, 以丰富的转染细胞。第二天, 这些细胞是在8小时10纳米雌二醇治疗后收获的。RNA 被分离, 并且一步 qPCR 被执行了。在此示例中, 站点1和2对于MMP17的完全雌激素响应是必需的, 而站点3在这些条件下不作出贡献 (图 2D, 第二条车道)。当只有2或3的站点处于活动状态 (vi 和 vii) 时, 雌激素的反应类似于没有活动的站点 (viii), 这表明这些站点不能独立贡献。站点1可以自行贡献一些表达式 (v), 但当站点1和2处于活动状态 (iv) 时, 就会看到最大的活动。
为了同时操作近4种不同基因的10促进剂 (图 3A), 生成了包含42个增强器指南和16个启动器参考线的导 rna 复合池。指南 rna 寡核苷酸在最初的指南 rna 引伸 PCR (步骤 3.3) 之前汇集了, 并且结果 PCR 产品被纯化了并且结合了空嘌呤霉素 U6 克隆载体用吉布森汇编。在吉布森装配之后, 进行了多次独立变换并进行了电镀。板块被刮成磅, 允许在 maxiprep 之前长出 2-4 小时。图 3B显示, 当这些指南 RNA 池被转染成雌激素剥夺的石川细胞系稳定表达 SID4X-dCas9-KRAB 和治疗如上所述 (图 2D) 时, qPCR 基因表达的代表性减少。增强剂的减少-我是类似于那些通过靶向启动者的假定目标基因。图 3C显示了在使用增强剂 i 的情况下, 导 rna 稀释对降低雌激素反应的影响。1:50 稀释的指南 RNA 池靶向增强剂附近的G0S2仍然产生显著的减少基因表达, 表明增强剂-我可以被用于目标多达50个网站一次。但是, 停用可以被稀释, 表明数以百计的站点不能同时被瞄准, 除非使用更灵敏的检测方法。

图1。使用增强器的多路增强剂解剖协议示意图指南 rna (红色和蓝色) 是设计使用电子脆和选择使用 UCSC 基因组浏览器。四引导 rna 被选择横跨感兴趣的区域 (转录因子结合站点由芯片序列定义)。指南 RNA 寡核苷酸已汇集的利益区 (红和蓝) 进行 PCR, 添加同源区域 (橙色) 之前, 吉布森组装和转换。结果质粒池通过 lipofection 转染成细胞系, 与 SID4X-dCas9-KRAB 质粒结合, 稳定地表达 SID4X-dCas9-KRAB 或与野生型细胞。引导 RNA 质粒池可以单独转染, 一次瞄准一个站点, 或者组合在一起, 同时瞄准多个站点。转染细胞用抗生素治疗, 以丰富含有导 rna 的细胞。在72小时后转染, 细胞被收获。核酸可以提取 qPCR, RNA 序列, 或芯片-后向请单击此处查看此图的更大版本.
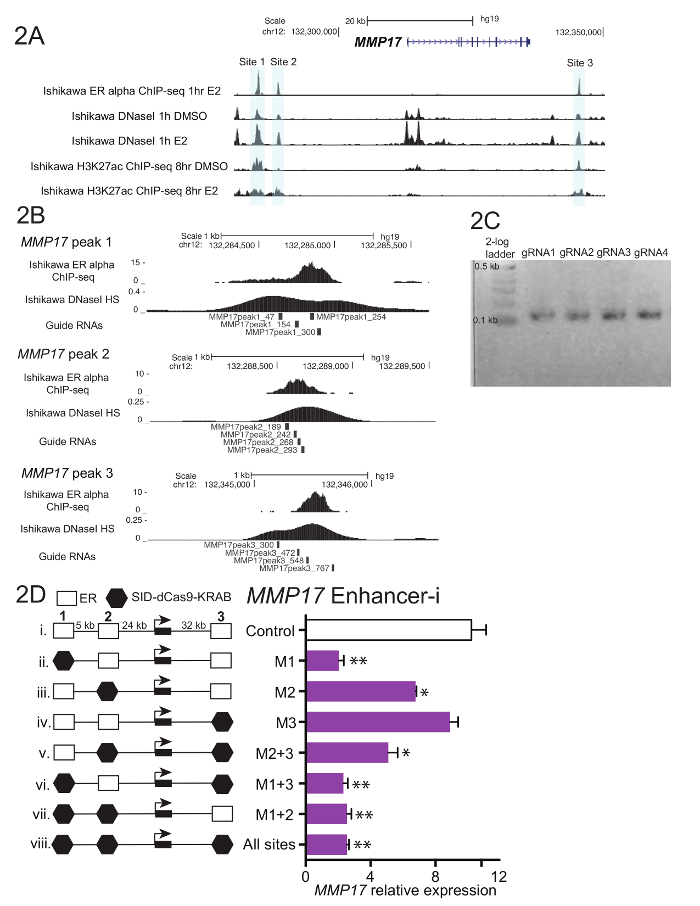
图2。指南 RNA 设计和增强器解剖为MMP17.(A)基因组浏览器在 MMP17 附近瞄准的 ER α增强增强剂 (灰色) 的截图.此数字已从卡尔顿 ( et .) 中进行了修改。18. (B)指南 RNA 设计为3个绑定站点18。由芯片序列定义的 ER 的绑定站点是目标, 4 导 rna 在这个区域内平铺。DNaseI 灵敏度信号, 它跨越绑定站点, 也可以用来定义目标序列的指导 RNA 设计。从 10 nM 雌二醇处理的石川细胞中提取了两种芯片序列和 DNaseI 的数据, 1 h. (C)代表指南 RNA 顺序, 为吉布森装配准备好, 经过短 PCR 添加同源区域。(D) MMP17的相对表达式, 通过 qPCR 对特定区域的靶向增强 i 和 8-h10 nM 雌二醇治疗后测量。表达式相对于CTCF和MMP17在未用雌二醇处理的单元格中的表达式级别。控制指南 rna 瞄准 IL1RN 的启动子.所有误差线都代表 SEM, 双星号指示p < 0.01 和单星号指示p < 0.05 在配对 t 测试。此数字已从卡尔顿 ( et al) 中进行了修改。18.请单击此处查看此图的更大版本.
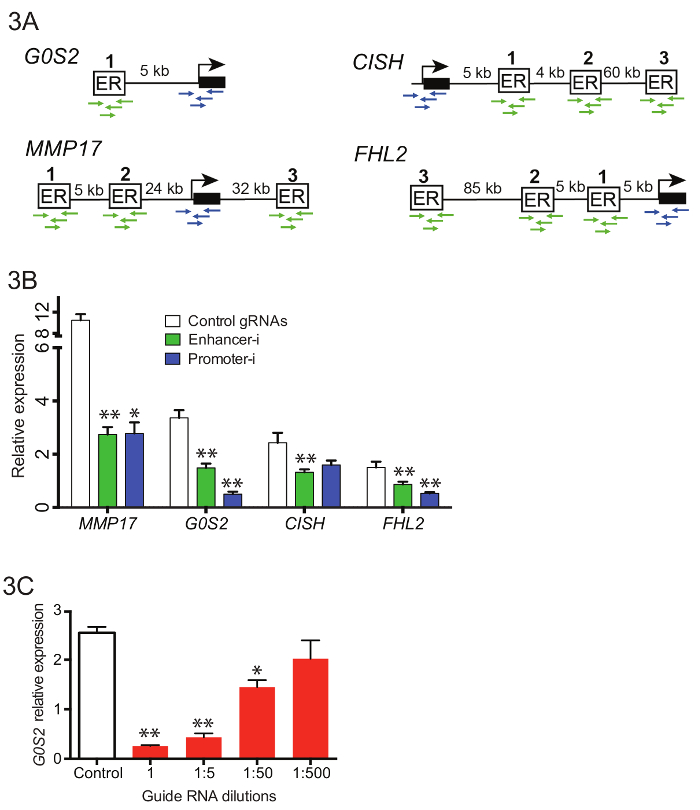
图3。同时用联合增强剂 i. 在不同基因附近的多个促进剂的靶向性.(A) "绑定站点" 和 "启动器" 的示意图, 其目标是汇集增强器-i。(B)在 E2 治疗后, 用增强型质粒池 (绿色)、启动子 i. 质粒池 (蓝色) 或控制 gRNAs (白色)18转染的石川细胞对表达的影响 qPCR。用增强剂 i 观察所有基因的显著减少。此数字是从卡尔顿 ( et al) 中修改的。18. (C)在 E2 治疗后, 以不同数量的导 rna 为靶向G0S2转染的石川细胞对G0S2表达水平的影响。即使有少量的导 RNA (1:50 稀释), 也可以看到显著的减少, 这表明多达50个站点可能同时被瞄准。所有误差线都代表 SEM, 双星号指示p < 0.01 和单星号指示p < 0.05 在配对 t 测试。请单击此处查看此图的较大版本.
| 名字 | 序列 |
| U6_internal_F | TTTCTTGGCTTTATATATCTTGTGGAAAGGACGAAACACCG |
| U6_internal_R | GACTAGCCTTATTTTAACTTGCTATTTCTAGCTCTAAAAC |
| U6_PCR_F | CCAATTCAGTCGACTGGATCCGGTA |
| U6_PCR_R | AAAAAAAGCACCGACTCGGTGCCA |
| gRNA_qPCR_F | GCTAGAAATAGCAAGTTAAAATAAGGCTAGTCCG |
| gRNA_qPCR_R | AAAAAGCACCGACTCGGTGCC |
| dCas9_qPCR_F | GTGACCGAGGGAATGAGAAA |
| dCas9_qPCR_R | AGCTGCTTCACGGTCACTTT |
| pAC95_PCR_F | AGAAGAGAAAGGTGGAGGCC |
| pAC95_PCR_R | CGTCACCGCATGTTAGAAGG |
| SID4X_PCR_F | CAATAGAAACTGGGCTTGTCG |
| SID4X_PCR_R | TCGTGCTTCTTATCCTCTTCC |
表1。引物用于引导 RNA 的延伸和排序, qPCR 和检测的融合蛋白。
讨论
本协议描述了一种简单而灵活的方法来解剖增强函数在内源基因组轨迹没有物理改变 DNA 序列。虽然在概念上与以前发布的 CRISPR 干扰协议使用 dCas9-KRAB27相似, 但增强器-i 在3主要方式上与这些协议不同。首先, 增强器-i 利用 MAD120的 SIN3A 交互域来实现增强停用。利用 HDAC 抑制剂可以抢救增强剂失活, 提示失活的主要机制是 HDAC 依赖性的。不像 CRISPR 干扰 dCas9-KRAB, 增强剂-i 不导致沉积 H3K9me3。这很可能是由于增强剂-i 依赖于瞬态引入导 rna, 而细胞在转染后3天被收割。在 CRISPR 干扰中, H3K9me3 的增加在7天后转导12中观察到。最后, 增强 i 协议提供了一个针对多个站点同时同时监控目标效率的策略。在马赛克序列17中, dCas9-KRAB 用于同时针对多个增强剂, 但这种技术依靠单细胞 RNA 测序来识别表达变化, 许多基因 (如雌激素应答基因) 由于低单细胞 RNA-下增强因子的灵敏度-i 提供了一个可靠的方法来研究促进剂单独和组合为任何基因。
增强剂的最关键的步骤-i 是转染, 这应该是优化的细胞线的兴趣。该协议依赖于嘌呤霉素处理, 以丰富的转染细胞, 但有可能的联合转染引导 rna 与荧光蛋白和分类的荧光细胞使用流式细胞术可能被证明是一个更好的浓缩方法的一些细胞类型。我们建议通过 qPCR 来监测导 rna 和 SID4x-dCas9-KRAB 的表达水平, 以诊断和确认转染。如果指南 rna 水平低 (周期阈值 > 30), 用户也可以考虑其他指南 rna 生产策略, 如体外转录28。也有可能, 尽管高 gRNA 水平, 引导 rna 靶向 SID4x-dCas9-KRAB 蛋白是低效的, 在这种情况下选择不同的指南 rna 序列可能是必要的。通过对增强 i 治疗细胞的染色质对融合蛋白进行芯片序列处理, 可以对靶向的效率进行监测。如果在感兴趣的区域有 SID4x-dCas9-KRAB 的高信号, 并且没有发现其假设的目标基因的表达变化, 那么该区域在研究的条件下可能不会对该基因的表达做出贡献。
增强器的一个潜在限制是, 如果太多的站点同时被瞄准, 则非目标效果可能会累积。然而, CRISPR 干扰策略对于击倒的目标效果要比 rna29少, 特别是当使用多克隆细胞线表示 dCas9-KRAB 时。当我们同时瞄准10个地点时, 我们已经看到了 SID4X-dCas9-KRAB 的基因组结合, 我们没有确定基因表达的变化, 因为这些绑定事件。由于某些促进剂可能会接触到多个促进剂和/或其他促进剂, 有可能许多基因可能会改变表达后, 针对单一的增强剂, 虽然不清楚这种形式的基因调控是常见的。为了确认所观察到的表达式更改是由于针对特定的增强器, 而不是目标效果, 用户可以使用两组不同的非重叠导向 rna 来针对同一区域进行增强。此外, 利用核酸酶能力的 Cas9 对该区域的遗传缺失可以进一步证实其对基因表达的影响。
由于增强剂-i 功能通过组蛋白脱乙酰, 它是可能的, 其失活能力仅限于有明显水平的组蛋白乙酰化。有各种各样的替代性压制融合, 可能更有效地针对特定的增强剂。DNA 甲基融合到 dCas9 可以用来减少基因表达时, 针对远端促进剂30, 但这种镇压往往不是短暂的。另一个压制融合使用 GATA1 (FOG1) 域的朋友, 它导致组蛋白 H3 赖氨酸 27 trimethylation 和压抑基因表达的水平类似 dCas9-KRAB 跨各种细胞系和促进者31。有趣的是, 添加更多的 FOG1 到 dCas9 减少了启动器的压制潜力, 这表明 SID 域的一个副本可能提供更多的增强失活比目前使用的4拷贝在增强剂 i。在上述 dCas9 融合的不同组合中, 某些位点可能受益于双目标。例如, 可以通过同时转导 dCas9-DNMT3a 和 dCas9-KRAB32来实现稳定的长期压制。这些压制性融合大多只针对一个单一的轨迹, 在同一时间, 它仍然不清楚哪些是最有效地操作多个促进剂同时。
增强剂 i, 虽然一个适当的方法来研究促进者的组合, 少数基因, 仍然有点有限的吞吐量, 如果用户希望研究假定促增剂为数以百计的基因。这种技术的未来应用将结合成像技术, 同时在多个样本中量化多个基因。重要的是, 这些技术与从裂解液中直接检测 rna 分子是相容的, 消除了耗时的 rna 分离的需要。这些适应将有助于审讯更大的增强剂。
披露声明
作者没有什么可透露的。
致谢
这项工作得到了 nih/NHGRI R00 HG006922 和 nih/NHGRI R01 HG008974 和猎人癌症研究所的支持。J.B.C. 在遗传学 T32GM007464 的 NIH 培训计划中得到了支持。
材料
| Name | Company | Catalog Number | Comments |
| ZR 96-well Quick-RNA Kit | Zymo Research | R1053 | |
| Power SYBR Green RNA-to-CT 1-Step | Applied Biosystems | 4389986 | |
| AflII restriction enzyme | NEB | R0520S | |
| Phusion High-Fidelity PCR Master Mix with HF Buffer | NEB | M0531L | |
| NEBuilder HiFi DNA Assembly Master Mix | NEB | E2621L | |
| FuGENE HD | Promega | E2312 | |
| DNA Clean & Concentrator Kit | Zymo Research | D4013 | |
| Buffer RLT Plus | Qiagen | 1053393 | |
| b-estradiol | Sigma-Aldrich | E2758 | |
| Human: Ishikawa cells | ECACC | 99040201 | |
| H3K27ac rabbit polyclonal | Active Motif | 39133 | |
| H3K9me3 rabbit polyclonal | Abcam | ab8898 | |
| FLAG mouse monoclonal | Sigma-Aldrich | F1804 | |
| ER alpha rabbit polyclonal | Santa Cruz | sc-544 | |
| pGL3-U6-PGK-Puro plasmid | Addgene | 51133 | Shen et al., 2014 |
| gRNA_cloningVector plasmid | Addgene | 41824 | Mali et al., 2013 |
| AflII U6 puromycin plasmid | Addgene | 106404 | Carleton et al., 2017 |
| SID4X-dCas9-KRAB plasmid | Addgene | 106399 | Carleton et al., 2017 |
| 2-Mercaptoethanol | Sigma-Aldrich | M6250-10ML | |
| UltraPure DNase/RNase-Free Distilled Water | ThermoFisher Scientific | 10977-023 | |
| Opti-MEM I Reduced Serum Medium | Gibco | 31985070 | |
| KAPA Stranded mRNA-Seq Kit, with KAPA mRNA Capture Beads | Kapa Biosytems | KK8420 | |
| Pierce Protease and Phosphatase Inhibitor Mini Tablets | ThermoFisher Scientific | A32959 | |
| Formaldehyde solution | Sigma-Aldrich | 252549-25ML | |
| Geneticin Selective Antibiotic (G418 Sulfate) (50 mg/mL) | ThermoFisher Scientific | 10131035 | |
| LB Broth | ThermoFisher Scientific | 10855001 | |
| Quick-DNA Miniprep Kit | Zymo Research | D3020 | |
| Quick-Load Purple 2-Log DNA Ladder | NEB | N0050S |
参考文献
- Consortium, E. P. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Roadmap Epigenomics, C., et al. Integrative analysis of 111 reference human epigenomes. Nature. 518 (7539), 317-330 (2015).
- Andersson, R., et al. An atlas of active enhancers across human cell types and tissues. Nature. 507 (7493), 455-461 (2014).
- Perry, M. W., Boettiger, A. N., Bothma, J. P., Levine, M. Shadow enhancers foster robustness of Drosophila gastrulation. Curr Biol. 20 (17), 1562-1567 (2010).
- Lam, D. D., et al. Partially redundant enhancers cooperatively maintain Mammalian pomc expression above a critical functional threshold. PLoS Genet. 11 (2), e1004935 (2015).
- Patwardhan, R. P., et al. Massively parallel functional dissection of mammalian enhancers in vivo. Nat Biotechnol. 30 (3), 265-270 (2012).
- Savic, D., et al. Promoter-distal RNA polymerase II binding discriminates active from inactive CCAAT/ enhancer-binding protein beta binding sites. Genome Res. 25 (12), 1791-1800 (2015).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Cheng, A. W., et al. Multiplexed activation of endogenous genes by CRISPR-on, an RNA-guided transcriptional activator system. Cell Res. 23 (10), 1163-1171 (2013).
- Hilton, I. B., et al. Epigenome editing by a CRISPR-Cas9-based acetyltransferase activates genes from promoters and enhancers. Nat Biotechnol. , (2015).
- Kearns, N. A., et al. Functional annotation of native enhancers with a Cas9-histone demethylase fusion. Nat Methods. 12 (5), 401-403 (2015).
- Thakore, P. I., et al. Highly specific epigenome editing by CRISPR-Cas9 repressors for silencing of distal regulatory elements. Nat Methods. 12 (12), 1143-1149 (2015).
- Groner, A. C., et al. KRAB-zinc finger proteins and KAP1 can mediate long-range transcriptional repression through heterochromatin spreading. PLoS Genet. 6 (3), e1000869 (2010).
- Qi, L. S., et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression. Cell. 152 (5), 1173-1183 (2013).
- Fulco, C. P., et al. Systematic mapping of functional enhancer-promoter connections with CRISPR interference. Science. 354 (6313), 769-773 (2016).
- Joo, J. Y., Schaukowitch, K., Farbiak, L., Kilaru, G., Kim, T. K. Stimulus-specific combinatorial functionality of neuronal c-fos enhancers. Nat Neurosci. 19 (1), 75-83 (2016).
- Xie, S., Duan, J., Li, B., Zhou, P., Hon, G. C. Multiplexed Engineering and Analysis of Combinatorial Enhancer Activity in Single Cells. Mol Cell. 66 (2), 285-299 (2017).
- Carleton, J. B., Berrett, K. C., Gertz, J. Multiplex Enhancer Interference Reveals Collaborative Control of Gene Regulation by Estrogen Receptor alpha-Bound Enhancers. Cell Syst. 5 (4), 333-344 (2017).
- Gilbert, L. A., et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell. 154 (2), 442-451 (2013).
- Ayer, D. E., Laherty, C. D., Lawrence, Q. A., Armstrong, A. P., Eisenman, R. N. Mad proteins contain a dominant transcription repression domain. Mol Cell Biol. 16 (10), 5772-5781 (1996).
- Alland, L., et al. Role for N-CoR and histone deacetylase in Sin3-mediated transcriptional repression. Nature. 387 (6628), 49-55 (1997).
- Konermann, S., et al. Optical control of mammalian endogenous transcription and epigenetic states. Nature. 500 (7463), 472-476 (2013).
- Rennoll, S. A., Scott, S. A., Yochum, G. S. Targeted repression of AXIN2 and MYC gene expression using designer TALEs. Biochem Biophys Res Commun. 446 (4), 1120-1125 (2014).
- Stampfel, G., et al. Transcriptional regulators form diverse groups with context-dependent regulatory functions. Nature. 528 (7580), 147-151 (2015).
- Perez-Pinera, P., et al. RNA-guided gene activation by CRISPR-Cas9-based transcription factors. Nat Methods. 10 (10), 973-976 (2013).
- Heigwer, F., Kerr, G., Boutros, M. E-CRISP: fast CRISPR target site identification. Nat Methods. 11 (2), 122-123 (2014).
- Parsi, K. M., Hennessy, E., Kearns, N., Maehr, R. Using an Inducible CRISPR-dCas9-KRAB Effector System to Dissect Transcriptional Regulation in Human Embryonic Stem Cells. Methods Mol Biol. , 221-233 (2017).
- Romanienko, P. J., et al. A Vector with a Single Promoter for In Vitro Transcription and Mammalian Cell Expression of CRISPR gRNAs. PLoS One. 11 (2), e0148362 (2016).
- Smith, I., et al. Evaluation of RNAi and CRISPR technologies by large-scale gene expression profiling in the Connectivity Map. PLoS Biol. 15 (11), e2003213 (2017).
- Liu, X. S., et al. Editing DNA Methylation in the Mammalian Genome. Cell. 167 (1), 233-247 (2016).
- O'Geen, H., et al. dCas9-based epigenome editing suggests acquisition of histone methylation is not sufficient for target gene repression. Nucleic Acids Res. 45 (17), 9901-9916 (2017).
- Amabile, A., et al. Inheritable Silencing of Endogenous Genes by Hit-and-Run Targeted Epigenetic Editing. Cell. 167 (1), 219-232 (2016).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。