需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
通过大刀DNA完整性检测评估结肠直肠癌风险和患病率
Erratum Notice
摘要
提出的诊断FL-DNA试剂盒是一种节省时间和用户友好的方法,用于确定存在结肠直肠癌病变的可靠概率。
摘要
如今,凳子DNA可以通过几种方法进行分离和分析。通过qPCR测定可以检测粪便中DNA的长片段,它提供了出现前肿瘤或肿瘤性结肠直肠病变的可靠概率。这种方法,称为荧光长DNA(FL-DNA),是一个快速,非侵入性的程序,是一个改善的主要预防系统。该方法基于对基因组DNA特定靶点进行定量扩增,对粪便DNA完整性进行评估。特别是,对超过200bp的DNA片段的评估可以检测出具有极高特异性的结肠直肠病变患者。然而,该系统和所有目前可用的粪便DNA测试提出了一些需要解决的一般问题(例如,应进行测试的频率和在每个时间点为每个个体收集的粪便样本的最佳数量)。然而,FL-DNA的主要优点是有可能将其与目前用于CRC筛查程序的测试(称为免疫化学基粪便隐匿血测试(iFOBT)结合使用。事实上,两个测试都可以在同一样本上进行,从而降低成本,并更好地预测结肠直肠病变的最终存在。
引言
结肠直肠癌(CRC)源于一个多步骤的过程,其中健康的上皮慢慢发展成腺瘤或息肉,随着时间的推移,它逐渐发展成恶性癌。,2尽管CRC的发病率很高,但在过去十年中,死亡人数呈下降趋势。事实上,筛查计划采用的早期诊断工具已导致早期发现和去除肿瘤前腺瘤或息肉4。然而,由于不同的技术限制,这些方法都不是最佳的。事实上,为了提高灵敏度和特异性,许多粪便DNA测试都是单独提出的,或与当前常规诊断测试55、66结合提出。
通常,健康的粘血症会排入粪便流中凋亡的结肠细胞,而患病的粘卵菌会去角质非凋亡结肠细胞。长度为200bp或以上的片段是非凋亡DNA的特征。这种DNA被称为长DNA(L-DNA),已成为CRC早期诊断的可利用生物标志物。L-DNA可以从粪便标本中分离出来,并使用体外诊断FL-DNA试剂盒77、8、9、10、11、128,9,10,11,12进行qPCR量化。
该测试包括两个检测FL-DNA片段的检测,范围从138bp到339bp。每个测定允许放大FL-DNA(FAM)以及尖峰DNA(HEX)。为了确保所有片段的最佳放大,测试分为两个检测(称为"A"和"B")。A检测检测APC基因(NM_001127511)的外子14和TP53基因(NM_001276760)的外子7片段。B检测检测出APC基因(NM_001127511)的外子14片段和TP53基因(NM_001276760)的外子5和8的两个区域。测定不区分检测到的区域。尖峰DNA对应于Oncorhynchus keta鲑鱼DNA,并能够验证手术是否正确,并检查是否存在抑制剂,从而可能产生假阴性结果。使用标准曲线方法通过绝对定量评估FL-DNA浓度,并表示为ng/反应。
FL-DNA方法是一种非侵入性和廉价的粪便DNA测试,结合免疫化学为基础的粪便隐匿血测试(iFOBT),目前用于CRC筛查程序,并允许更好地预测CRC和/或高风险腺瘤病变12。
研究方案
2013年至2015年,在梅尔多拉(FC,意大利FC)的科罗马诺洛科学组织(IRST)招募了患者。注册患者进入 IRSTB002 协议,经 IRST 道德委员会 - IRCCS AVR (25/10/2012,第 1 条) 批准。所有方法均按照相关准则和条例执行。所有患者均获得书面知情同意。
1. 从凳子中提取DNA
- 使用工具包准备凳子样本(参见材料表)。根据制造商的说明,通过执行萃取来选择和处理粪便材料。直接放大纯化DNA或在-20°C储存,以便随后分析。
2. 准备阳性对照、标准、尖峰DNA和临床样本
- 编制标准和样品
- 为了准备正控制、标准、尖峰DNA和所有临床样本,请离心正控制、标准和尖峰DNA的分值,然后通过添加正确数量的提供水重新悬浮每个试剂(见下文)。然后,小心地旋涡正控制,标准和尖刺DNA,然后离心机10s。要完全重新悬浮干试剂,在使用前将液体试剂储存在室温 (RT) 下 30 分钟。
- 正对照是干格式的人类DNA。用 750 μL 的水重新悬浮每个等位值。
- 尖刺DNA是鲑鱼(Oncorhynchus keta)DNA,它被用作外源内控,以验证从粪便中提取的DNA样本中可能存在抑制剂。用 100 μL 的水重新悬浮每个等位。
- 要准备标准曲线,请从库存解决方案开始生成四个 1:5 稀释。标准点必须为 10 ng/反应、2 ng/反应、0.4 ng/反应和 0.08 ng/反应。
- 为了准备正控制、标准、尖峰DNA和所有临床样本,请离心正控制、标准和尖峰DNA的分值,然后通过添加正确数量的提供水重新悬浮每个试剂(见下文)。然后,小心地旋涡正控制,标准和尖刺DNA,然后离心机10s。要完全重新悬浮干试剂,在使用前将液体试剂储存在室温 (RT) 下 30 分钟。
- 制备1倍尖峰DNA
- 使用前直接准备尖峰DNA控制。
- 通过将 5 μL 的 FL-DNa 尖峰与 20 μL 无菌水混合,准备 1 倍尖峰DNA控制。将根据要分析的样本数量加上正对照制备 1 倍的 DNA 控制样本数量。
- 样品的制备
- 将 75 μL 的样品(临床样品或阳性对照)与 25 μL 的 1 倍尖峰 DNA 混合,总体积为 100 μL。
3. 使用qPCR易PGX放大和测定FL-DNA值
注:含有针对人类DNA和内部控制的特定引物和探针的完整扩增混合物以8个井带提供,用于FL-DNA混合A和FL-DNA混合B。 标准、阳性和阴性对照,样品必须用两种冻干混合物进行放大。临床样本只能用两种冻干混合物重复进行扩增。
- 有关 qPCR 仪器和操作软件,请参阅材料表。
- 打开操作软件并设置板和热型材:
- 设置板,如表 1所示。
- 将第 1 列中的所有八个位置的井类型设置为标准。
- 将 A2 和 B2 井的井类型设置为NTC。
- 将 C2 和 D2(正控件)的井类型设置为未知。
- 将所有其他位置的井类型设置为"未知"。
- 选择所有 96 个位置,然后添加染料FAM和HEX。单击同步板。
- 根据表 2设置热配置文件。
- 设置板,如表 1所示。
- 将所需的条数离心 10 s,将内件带到管的底部。
- 轻轻地从条带上取下密封件,同时注意保留内容物,并添加到相应的条带:负控制:20 μL水;样品:20μL的DNA;标准曲线:标准 1、2、3 或 4 的 20 μL;正控制:正控制20μL。
- 使用 8 条平光盖和涡旋小心关闭所有条带几秒钟。
- 将条带离心 10 s 并加载到仪器中。然后,开始运行。
- 打开操作软件并设置板和热型材:
4. 数据分析
注:数据分析可以根据软件自动或手动执行(参见材料表)。
- 在运行结束时,选择"FAM:FL-DNA-A"和"HEX:IC"的列 A、C、E、G,选择 B、D、F、H 列,用于"FAM:FL-DNA-B"和"HEX:IC"。FAM
- 为标准数量起始量设置以下内容:A1 和 B1 井的 10 ng/反应,C1 和 D1 的 2 ng/反应,E1 和 F1 的 0.4 ng/反应,G1 和 H1 的 0.08 ng/反应。
- 将 FAM(FL-DNA A 和 FL-DNA B)和 HEX (IC) 通道的阈值荧光值设置为 150。
- 在"结果表"框中,单击"列选项" |选择全部|好的,使用各自的 Cq (+R) 和 +R 最后值在两个通道中获取结果。
注:这些值由实时 PCR 仪器软件提供。*R 最后对应于与最后一个放大周期相归一的荧光值。 - 在"结果表"框中,右键单击表以打开上下文菜单,然后单击"发送到 Excel"以导出原始数据。
- 检查标准的值以验证标准曲线的适用性。
- 对于每个 FL-DNA 组合,请检查 R2 [R2 ([R)] 列] 和效率 ["效率 (%)"列* 值。如果它们处于可接受的范围内,可以根据制造商的说明进行相应的分析(表 3)。
- 如果 FAM 通道的结果不在预期范围内,请省略标准曲线的一个点,然后重新分析运行。
- 使用以下公式确定负控件和正控件的值,将"无 Cq"值视为零:
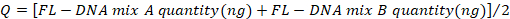
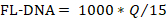
- 将获取的值与表 4中报告的值进行比较。
- 如果反应控制位于预期值范围内,请继续分析样本。
注:验证获得的 Cq 值是否来自实际放大反应(sigmoidal 荧光曲线),而不是从伪影(线性荧光曲线)生成。 - 要分析样品是否适合每种FL-DNA混合,请比较 HEX 通道的 Cq 值。如果值为 +16,请继续分析样本。如果该值为 <16,或者没有 Cq,则很可能是由于 FL-DNA 尖峰的点胶错误。因此,无法分析样本。
- 使用以下公式计算正控件的"HEX"通道中的 Cq 值的平均值:

- 使用以下公式计算示例复制的"HEX"通道中 Cq 值的平均值:
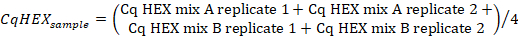
- 根据以下公式计算 _CqHEX 值:
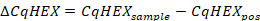
- 将样本的 _CqHEX 值与表 5中报告的值进行比较。
- 对于每个混合(混合 A 和混合 B),将 FAM 通道的 Cq 值与表 6中报告的 Cq 值进行比较。
- 要确定每个合适样品的 FL-DNA 值,请使用以下公式,将"无 Cq"值视为零:
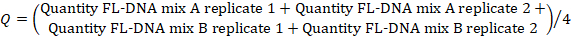
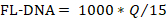
注:结肠直肠癌的风险和患病率是iFOBT和FL-DNA评估的函数,根据Rengucci等人12获得的Fagan诺莫图结果(表7)。
结果
该协议的工作流如图1所示。工作流根据这些步骤结果提供两个控制步骤和不同的操作。首先,如果样本显示不适合的控件,则必须重复放大。其次,如果放大被抑制,样品必须从开始再加工或归类为无价值。
图 2显示了正负样本产生的荧光曲线。(A) 所示是合适的正样本的示例。HEX 通道上的采样信号在可接受的范围内。正信号高于 FAM ...
讨论
先前的研究已经表明,由手动和半自动方法提取的粪便的DNA完整性分析,可以代表早期发现结肠直肠病变的替代方法,87、8、9、10、11、12。,9,10,11,12多年来,为检测结肠直肠癌开发了分子、非侵入性筛查测试,但由于方法耗时,与其他筛查测试相比?...
披露声明
莫拉·孟吉是Diatech制药遗传学公司的全职员工。
致谢
作者没有承认。
材料
| Name | Company | Catalog Number | Comments |
| 1.5 mL and 2 mL polypropylene twist-lock tubes (DNase-, RNase-, DNA-, PCR inhibitor-free) | Consumables required for DNA extraction and Real Time PCR | ||
| Absolute Ethanol (quality of analytical degree) | Reagent required for DNA extraction | ||
| Benchtop centrifuge | Maximum speed of 20000 x g. Instrument required for DNA extraction | ||
| EasyPGX analysis software version 2.0.0 | Diatech Pharmacogenetics | RT800-SW | Analysis software |
| EasyPGX centrifuge/vortex 8-well strips | Diatech Pharmacogenetics | RT803 | Instrument recommended for the Real Time PCR assay |
| EasyPGX qPCR instrument 96 | Diatech Pharmacogenetics | RT800-96 | Instrument recommended for the Real Time PCR assay |
| EasyPGX ready FL-DNA | Diatech Pharmacogenetics | RT029 | Kit required for the Real Time PCR assay |
| Micropipettes (volumes from 1 to 1.000 µL) | Consumables required for DNA extraction and Real Time PCR | ||
| Powder-free disposable gloves | Consumables required for DNA extraction and Real Time PCR | ||
| QIAamp Fast DNA Stool | Qiagen | 51604 | Kit recommended for the DNA extraction and purification from stool |
| Sterile filter tips DNase-, RNase-free (volumes from 1 to 1.000 µL) | Consumables required for DNA extraction and Real Time PCR | ||
| Thermal block e.g. EasyPGX dry block | Diatech Pharmacogenetics | RT801 | Instrument required for DNA extraction |
| Vortex e.g. EasyPGX centrifuge/vortex 1.5 ml | Diatech Pharmacogenetics | RT802 | Instrument required for DNA extraction |
参考文献
- Fearon, E. R. Molecular Genetics of Colorectal Cancer. Annual Review of Pathology. 6, 479-507 (2011).
- Sears, C. L., Garrett, W. S. Microbes, Microbiota, and Colon Cancer. Cell Host and Microbe. 15, 317-328 (2014).
- Levin, B., et al. Screening and Surveillance for the Early Detection of Colorectal Cancer and Adenomatous Polyps, 2008: A Joint Guideline From the American Cancer Society, the US Multi-Society Task Force on Colorectal Cancer, and the American College of Radiology. Gastroenterology. 134, 1570-1595 (2008).
- Bosch, L. J., et al. Molecular tests for colorectal cancer screening. Clinical Colorectal Cancer. 10, 8-23 (2011).
- Ahlquist, D. A. Molecular detection of colorectal neoplasia. Gastroenterology. 138, 2127-2139 (2010).
- Calistri, D., et al. Fecal multiple molecular tests to detect colorectal cancer in stool. Clinical Gastroenterology and Hepatology. 1, 377-383 (2003).
- Calistri, D., et al. Detection of colorectal cancer by a quantitative fluorescence determination of DNA amplification in stool. Neoplasia. 6, 536-540 (2004).
- Calistri, D., et al. Quantitative fluorescence determination of long-fragment DNA in stool as a marker for the early detection of colorectal cancer. Cellular Oncology. 31, 11-17 (2009).
- Calistri, D., et al. Fecal DNA for noninvasive diagnosis of colorectal cancer in immunochemical fecal occult blood test-positive individuals. Cancer Epidemiology Biomarkers and Prevention. 19, 2647-2654 (2010).
- De Maio, G., et al. Circulating and stool nucleic acid analysis for colorectal cancer diagnosis. World Journal of Gastroenterology. 20, 957-967 (2014).
- Rengucci, C., et al. Improved stool DNA integrity method for early colorectal cancer diagnosis. Cancer Epidemiology Biomarkers and Prevention. 23, 2553-2560 (2014).
Erratum
Formal Correction: Erratum: Evaluation of Colorectal Cancer Risk and Prevalence by Stool DNA Integrity Detection
Posted by JoVE Editors on 9/28/2020. Citeable Link.
An erratum was issued for: Evaluation of Colorectal Cancer Risk and Prevalence by Stool DNA Integrity Detection. An affiliation was updated.
The first affiliation was updated from:
Istituto Scientifico Romagnolo per lo Studio e la Cura dei Tumori (IRST)
to:
Istituto Scientifico Romagnolo per lo Studio e la Cura dei Tumori (IRST) IRCCS, Meldola, Italy
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。