需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
使用声学纳米分配技术实现高通量DNA质粒多路复用和转染
摘要
该协议使用声滴喷射技术描述了384孔板中哺乳动物细胞的高通量质粒转染。耗时、易出错的 DNA 点胶和复用,以及转染试剂点胶,由软件驱动,由纳米点胶设备执行。然后,这些细胞被播种在这些预填充的井中。
摘要
细胞转染是许多生物学研究不可或缺的,需要控制许多参数,以实现准确和成功。通常在低吞吐量下执行,而且既耗时又容易出错,在多路复用多个质粒时更是如此。我们开发了一种简单、快速、准确的方法,利用声滴喷射 (ADE) 技术在 384 孔板布局中执行细胞转染。本研究中使用的纳米分配器装置基于该技术,允许从源井板高速向目标井板精确输送纳米体积。它可以根据预先设计的电子表格分配和复用DNA和转染试剂。在这里,我们提出了一个基于ADE的高通量质粒转染的最佳方案,这使得在共转染实验中达到高达90%的效率和近100%的共转染成为可能。我们扩展了初始工作,提出了一个基于用户友好的基于电子表格的宏,能够管理多达四个质粒/孔从包含多达1,536个不同的质粒的库,以及一个基于平板电脑的移液指南应用程序。宏设计了源板的必要模板,并为纳米分配器和基于平板电脑的应用生成即用型文件。四步转染协议涉及 i) 使用经典液体处理机的稀释剂,ii) 质粒分布和多路复用,iii) 纳米分配器的转染试剂点,以及 iv) 预填充孔上的细胞电镀。所述基于软件的ADE质粒多路复用和转染控制,即使是该领域的非专家也能快速、安全地执行可靠的细胞转染。此方法能够快速识别给定单元类型的最佳设置,并可转用到更大规模和手动的方法。该协议简化了非池化筛选策略中的人类ORFeome蛋白(基因组中开放阅读框架[ORF])表达或基于CRISPR-Cas9的基因功能验证等应用。
引言
这里介绍的方法详细介绍了如何在384孔板中使用声基液体纳米分配器在高通量的哺乳动物细胞中执行DNA质粒多路复用和转染,即使是对于该领域的非专家也是如此。最近公布的方法1允许在一个实验中执行多达384个独立的质粒DNA复用和转染条件,在不到1小时。转染细胞群中的共转染。此协议使转染更容易,因为大多数繁琐、耗时且容易出错的步骤现在都是软件驱动的(有关概述,请参阅图1)。已作出进一步努力,开发专用工具,以提高易用性,同时避免整个过程中的人为错误,并促进成功的转染,即使是外地的非专家。所述协议包括一个"用户友好"的宏电子表格,我们开发了该电子表格,以便管理 384 个独立的转染条件,每个孔中最多具有四个质粒的多路复用可能性。宏自动生成源板的模板,以从起始库存解决方案加载预期的 DNA 质粒体积,并在已输入的实验设计后启动纳米分配器软件所需的文件。由于在384孔源盘中手动分配DNA是繁琐且容易出错的,我们还开发了一个基于平板电脑的专用应用程序,在根据模板分配DNA溶液的同时指导用户。
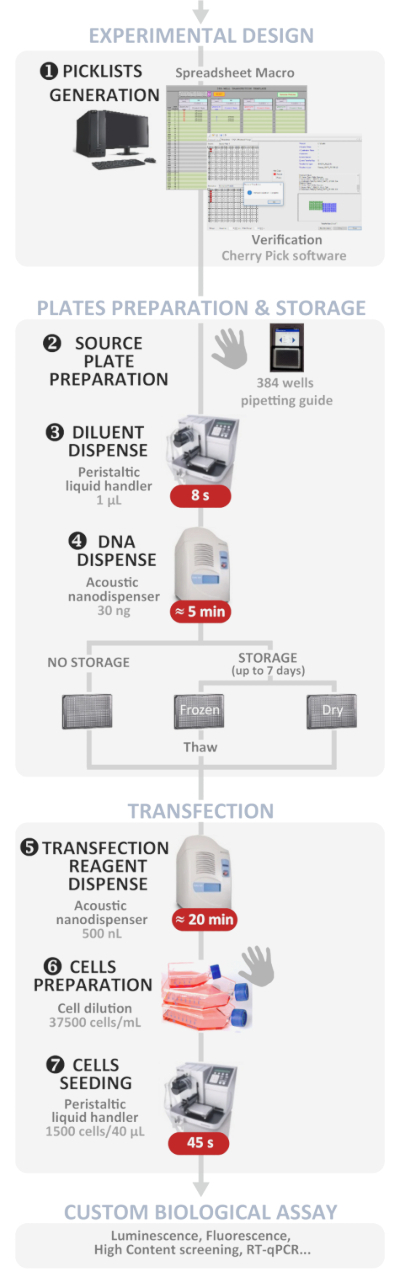
图1:实验工作流。最佳自动化高通量反向转染协议(从实验设计到定制生物测定)的原理表示。手动步骤由手符号指示,每个步骤的大致时间写在红色框中。请点击此处查看此图的较大版本。
许多基于细胞的实验从质粒DNA转染开始,即使许多专用试剂已经和仍在开发中,以提高转染效率和/或简化程序,仍有许多工作要做2,3,4.DNA质粒细胞转染涉及几个步骤,以达到高效率,如初始复杂接受,内皮逃逸,和细胞质传输到细胞核5,6。除了钙沉淀或物理技术,如电穿孔或显微注射使用专用装置7,现代化学方法已侧重于加强DNA细胞交付,同时降低细胞细胞毒性8, 9.使用脂质或阳离子聚合物形成脂质体状复合物,以及最近的非脂体聚合物化学系统使转染更容易和更有效率10。尽管有这些发展,细胞转染仍然需要具体技能,以准确执行,因为大多数物理或化学转染协议要求科学家手动准备每个DNA转染反应条件,因此影响吞吐量。为了规避这个问题,利用化学转染试剂11、12、13开发了反向转染方案,使用户能够更快地测试或组合几个质粒。在这些协议中,在将细胞播种到复合物上之前,与转染试剂形成核酸复合物。然而,这些反向协议仍然受到DNA溶液的手动处理和每个独立条件的组合的限制。虽然以96孔板格式进行它们是可行的,但DNA的制备和分配将单调乏味,并且可能会有错误。当需要不同数量的DNA质粒并相互多路复用时,细胞转染就更难实现,而且更加耗时,人为错误也变得相当不可避免。尽管很少多路复用DNA转染条件,但以反向转染方法扩展到384孔板格式,由于以下原因,成为一个不可能的挑战。i) 要管理的DNA量、转染试剂或反应混合物体积低于每口井的1μL。ii) 384个独立条件下质粒的多用性变得极其复杂。384口井的输送也非常耗时,而且容易出错。事实上,在预期井中分配正确的解决方案是很难管理的,因为已经分配的低容量不允许在空井和已满井之间进行目视监控。v) 最后,由于执行必要点胶步骤所需的时间,在加入细胞之前,蒸发干燥混合物的风险很高。总之,建立高通量DNA质粒转染测定的限制因素似乎是检测的小型化,这意味着小批量多路复用和管理,不能再手动处理,但也很难在由传统的围静态液体处理机的可靠方式。
为了证明自动化(如测定和获得高通量)的难度,到目前为止,只发布了几次自动转染的尝试:使用商业液体处理装置和磷酸钙沉淀的96 孔板格式最近,一个利波莱克斯试剂,和一个微流体芯片,使280个独立的转染15,但需要这方面的专业技能。另一种方法,去磷,允许液体悬浮,并导致流体操纵和混合,用于执行DNA转染在24至96孔板格式16。虽然可行,但这种方法的通量极低,因为细胞与DNA转染混合物的混合需要在播种前每一点孵育60s。这意味着整个 96 孔板的持续时间至少为 96 分钟。此外,由于这项工作是使用目前市场上没有的内部设计和制造设备完成的,因此该协议还远远不能让广大生物学家的观众满意。相反,在过去几年中,纳米体积分配器设备出现了一种易于使用的基于软件的声学点胶技术。利用聚焦声能,这些器件允许严格控制小液体体积从2.5 nL到500 nL从源板喷射到目的地17。这种技术称为声滴喷射 (ADE),具有许多优点:它是全自动的、非接触的、无提示的、准确、精确且高度可重复的,并且具有高通量18。首次致力于提供二甲基硫酸盐(DMSO)解决方案,设置已增强,以分配水缓冲液19。因此,声学纳米分配器似乎适用于反向细胞转染协议,并可以规避上述大多数手动限制。由于以前没有使用该技术描述过质粒转染尝试,我们最近评估了基于声学的点胶系统是否适合执行反向细胞转染。
利用纳米分给器的吞吐量和易用性,我们优化了HeLa细胞的反向转染方案,通过交叉测试几个参数,这些参数可以影响384孔单板的DNA转染,即总DNA量和源DNA起始浓度、稀释体积、转染试剂和扩散细胞数量。开发的协议规避了上述细胞转染的手动限制,与其他自动转染尝试具有若干优点。首先,它被小型化,从而允许通过节省DNA质粒制剂和转染试剂实现具有成本效益的转染试剂。其次,它比手动协议(即使是初学者)具有更高的吞吐量和可重现性,因为整个 384 孔板的转染可在 1 小时内实现。最后,它是软件驱动的,允许控制分配的DNA数量和多个质粒的多路复用。事实上,多亏了纳米分配器软件(材料表),用户可以制定一个研究计划来控制从定义的源井板到目的地的孔板的分配量。
此处提出的协议主要面向那些能够使用纳米分配器并希望在高通量下设置转染实验的用户,但也适用于那些希望通过以下形式快速优化特定细胞类型的转染参数的人。将该协议应用于在高吞吐量下交叉测试多个参数。事实上,我们已经表明,通过这种纳米尺度协议识别的优化参数可以转用于更大规模和手动转染实验。最后,由于本协议中使用的转染试剂允许根据制造商进行DNA或siRNA转染,该协议也对那些旨在执行基因过度表达或敲除的阵列方法的人感兴趣。预填充DNA的目的地板可在转染检测使用前7天保存,而不会失去功效,这是此类应用下协议的另一个优点。
研究方案
1. 提前准备
- 准备蠕动液体处理程序
注:对于协议的稀释剂和单元分配步骤,必须准备专用程序,同时考虑到使用板的点胶头高度和步骤意图。- 对于 1 μL 稀释剂点胶步骤,安装 1 μL 盒式磁带,并准备步骤 1.1.1.1 和 1.1.1.2 中所述的设置程序。
- 将流速参数调整为"高",以获得最佳吞吐量,因为此步骤中预计不会造成生物材料损坏。将分配高度调整到 9.6 mm(根据所使用的细胞培养板,补充图1),以便在分配期间使 1 μL 跌落接触井底。
注: 此步骤对于避免在分配头上保留液滴,直到达到足够的体积才能下降至关重要。 - 将板的透明高度调整到 14.4 mm,以便在每行分配后,分配头在板上自由位移。目视控制蠕动液体处理程序头高度的正确设置:在点胶时确保点胶头上不留任何掉落液滴,并验证头部是否足够高,以便在分配每行后能够位移头部。
注意:避免跌落保留是一个关键参数,因为它会影响豁免体积的准确性。
- 将流速参数调整为"高",以获得最佳吞吐量,因为此步骤中预计不会造成生物材料损坏。将分配高度调整到 9.6 mm(根据所使用的细胞培养板,补充图1),以便在分配期间使 1 μL 跌落接触井底。
- 要分配 40 μL 电池悬架,请安装 10 μL 盒式磁带,并准备步骤 1.1.2.1-1.1.2.2 中所述的设置程序。
- 将流速参数调整为"低",以以低速分配细胞,以避免剪切应力和对井底的高冲击对细胞造成潜在损害。将分配高度调整到 11.43 mm(根据所使用的细胞培养板,补充图1),高度足以降低在点胶过程中对井底的细胞影响,但足够低,以避免液滴滞留在分配头。将板的透明高度调整到 16 mm,以便在分配每行后使分配头自由位移在板上。
- 目视控制蠕动液体处理程序头高度的正确设置:在点胶时确保点胶头上不留任何掉落液滴,并验证头部是否足够高,以便在分配每行后能够位移头部。
注: 避免丢弃保留是一个关键参数,因为它将导致分配不可靠的细胞编号。
- 对于 1 μL 稀释剂点胶步骤,安装 1 μL 盒式磁带,并准备步骤 1.1.1.1 和 1.1.1.2 中所述的设置程序。
- DNA质粒制剂(经典小制备提取方案)
- 在LB介质中生长转化的DH5+细菌菌株,辅以125μg/mL阿霉素选择抗生素(材料表),在37°C下在轨道摇床(材料表)上温和搅拌(200rpm)。
- 收获2mL的培养物,在6000 x g下通过离心5分钟粒细胞,并丢弃上清液。
- 用含有RNase A的250 μL再悬浮缓冲液重新悬浮细胞颗粒(材料表)。根据制造商的说明,加入250 μL的解液缓冲液,并在室温下孵育5分钟。
- 加入300μL的中和缓冲液(材料表)和涡流,停止分解反应。在11,000 x g下将管子离心5分钟。
- 在2mL收集管中放置一个新的质粒小柱(材料表),在11,000 x g下离心1分钟,将上清液分化在柱中。
- 丢弃流过,并将微型柱放回收集管中。
- 根据制造商的说明,用500μL的可选洗涤缓冲液(材料表)和离心机在11,000 x g下清洗质粒小柱1分钟。
- 丢弃流过,并将质粒小柱放回收集管中。
- 根据制造商的说明,在11,000 x g下加入700μL的洗涤缓冲液(材料表)和离心机1分钟。
- 在 11,000 x g下,将流通和离心的质粒小柱及其收集管再丢弃 2 分钟,以干燥硅膜。
- 将干燥的质粒小柱放入新的1.5 mL管中,在60°C下加入30μL的蒸馏水,在室温下孵育2分钟,然后在11,000 x g下将其离心1分钟。
- 丢弃质粒小柱,保持含有纯化DNA质粒的脱氧核糖核酸酯。
- 使用微体积分光光度计(材料表)测量洗脱DNA的DNA浓度。
- 打开分光光度计并选择DNA 测量设置。
- 将分光光度计的取样臂和移液器 1 μL 水提升到测量底座上,以执行空白校准。
- 放下采样臂,开始空白测量,然后等待完成。
- 抬起取样臂,从上部和下部底座上擦拭样品。
- 将DNA溶液的移液器1μL移液到下部底座上进行测量。
- 降低取样臂,开始DNA浓度测量,并等待完成。
- 抬起取样臂,从上部和下部底座上擦拭样品。
- 对于进一步的 DNA 浓度测量,重复步骤 1.2.13.5-1.2.13.7。
- 测量完成后,将DNA溶液储存在4°C,直到使用。
2. 实验设计和生成选择列表,以驱动基于 ADE 的点胶
注:开发了一个专用的"用户友好"电子表格宏,以384孔板格式管理DNA数量,并混合多达4个质粒。基于输入的实验设计,该宏生成必要的文件,以驱动基于ADE的DNA转染协议由纳米分配器。为了生成这些文件,必须在模板表中填写多个字段,如图2所示。
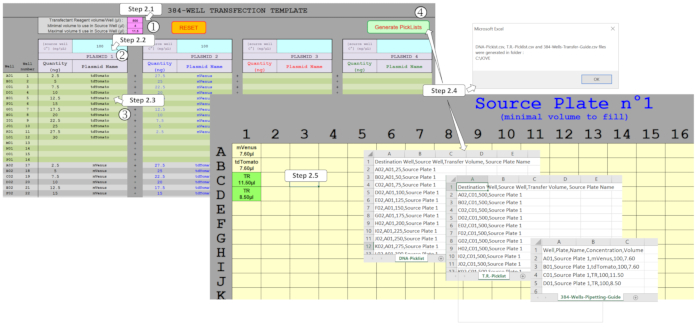
图 2:生成选择列表,以使用电子表格宏驱动 ADE 豁免。必须填充几个参数,即(1) 转染试剂 (TR) 和源板中使用的最小/最大体积, (2) 源板中要分配的初始质粒浓度, 和 (3)全板设计,包括384孔中预期的质粒量和复用。4)生成选择列表激活允许验证不同的字段,一旦正确填写,DNA 和 TR 分配的选择列表和必要的源板模板将自动生成。请点击此处查看此图的较大版本。
- 在粉红色字段中输入纳米分配器协议参数。将转染试剂 (TR) 混合物值设置为 500 nL。将源板孔中的最小体积值设置为 4 μL.将源板孔的最大体积设置为 11.25 μL。
注: 此处使用的纳米分配器在一次 ADE 运行中最多只能传输 500 nL。这些粉红色字段预先填充了建议的值,但可以根据用户需要进行修改。 - 输入 100 纳克/μL DNA 起始浓度与基础 DNA 对应的蓝色字段。
注: 此值是以前定义的最佳浓度,但可以根据不同的用户需求进行修改。 - 在灰色/绿色字段中输入所需的 DNA 量。输入 384 口井的量和质粒名称,确保在几口油井中使用相同的质粒时拼写相同。
- 生成源板设计、选择列表文件和移液引导文件。单击"生成选择列表"以允许宏从相应工作表上收集的数据生成 DNA-选择列表.csv、T.R.-Picklist.csv 和 384-Wells-移位指南.csv 文件。如果请求,请更正充满橙色的单元格值,因为它表示纳米分配器无法处理的错误或体积。
- 从源板工作表打印模板。标有在井中填充的质粒名称和最小体积。同样,转染试剂混合物卷,接下来必须填补在以下井指示为TR,并突出显示在绿色。
3. 使用384孔移液导应用进行DNA源板制备
- 使用蒸馏水将储存的DNA质粒从步骤1.2.14稀释至100纳克/μL。
- 将 384 孔网格校准到板尺寸:打开平板电脑上的 384 孔移液导引应用(图 3)。将源板放在下屏幕的网格上,并在左上角校准菜单中,单击+或- (或使用红色光标)以增强或减小网格和井的大小,以便将绿色孔调整到板的四个角孔.

图 3:使用384井移液导引应用。(1) 384 孔网格的校准到板尺寸;(2) 使用双面胶带将通用 3D 打印板适配器安装到平板电脑上;(3) 将板放在适配器上;(4) 将网格移位以将其居中到安装的板上。(5) 校准步骤的锁定。(6) 打开 384 口井移液指南.csv 文件。(7) 给定文件清单,申请将指明预期源板名称、试剂(DNA或转染试剂)、浓度和分配到目标井中的体积,该图孔将逐一照亮。(8) 左右箭头按钮允许用户按照移液指南,根据电子表格宏源板模板轻松分配试剂。请点击此处查看此图的较大版本。
- 使用双面胶带将 3D 打印板适配器安装到屏幕上,以避免点胶时源板移动。如果需要,使用旋转箭头和向上/向下/右/左按钮移动校准的网格,以将屏幕上的网格调整到板位置。正确校准和定位网格和井尺寸后,勾选"锁定"校准框。
- 单击"文件"并打开384 口移液指南.csv文件。按照屏幕说明手动分配指示质粒的指定体积,以与预期板的正确目标相对应的白色突出显示的浓度指示的浓度。在 DNA 分配过程中使用-或+箭头返回或进一步。到达第一个转染试剂溶液加载时停止点胶。
- 完成 DNA 分配后,从适配器中取出源板。如果必须填充多个源板,则在适配器上放置新的源板,并按照点胶说明进行操作。DNA分配完成后,将充满DNA的源板(在1,500 x g处2分钟)离心,以确保适当的液体调平,并去除导致基于ADE的转移不准确的气泡。
4. 基于蠕动液体处理机的1μL稀释剂在目标板中的分配
注:在生物安全柜中执行步骤 4.1-4.5。
- 用喷雾消毒剂(材料表)喷洒1μL盒头,并允许此溶液进入尖端支架。吸收吸收纸上的残留消毒剂。将 1 μL 盒卡安装在蠕动液体处理程序设备上。打开设备并确保盒式类型设置正确 (1 μL) 以及板格式 (384 孔)。
- 消毒管的整个流明:将管机构(将八根管子放在一起)插入无菌容器中,并填充 5 mL 的 70% 酒精。使用蠕动液体处理机的充注功能,首先冲洗管道中的酒精,然后通过5 mL蒸馏水和5 mL的无血清介质冲洗(Dulbecco的改性Eagle的介质[DMEM]辅以100 U/mL青霉素-链霉素;材料表),连续填入同一容器。通过目视检查所有液位,确保没有堵塞任何尖端。
- 用无血清培养基填充新的无菌容器,填充10 mL的预温无血清介质,并潜水管组织者。按蠕动液体处理程序的质键约 10 s。再次,通过目视检查所有液体流,确保没有尖端堵塞。
- 向板中填充 1 μL 稀释剂。将无菌 384 孔培养板(目的地)放在蠕动液体处理程序板托架上,并取下其盖子。
- 运行预校准程序,在 384 孔板的每个孔中分配 1 μL。点胶时间约为 8 s。然后更换 384 孔板的盖子。
注: 或者,此步骤可以在安全柜中使用多通道微移液器手动处理。
5. 执行测量以控制手动分配的数量
注:有关详情,请参见图4。
- 运行 nano 分配器程序,转到诊断选项卡,勾选源板出箱,将源板加载到板架上,然后勾选输入板。当出现提示时,选择384LDV_AQ_B2将纳米点胶设置为水缓冲点胶模式,然后按"确定"。
- 在"杂项"菜单中选择"调查",然后单击"启动"。选择预填充的井进行分析,然后单击"转到"按钮。验证测得的体积与预期体积匹配,并确保没有井装的体积超过 12 μL,因为这将避免传输。
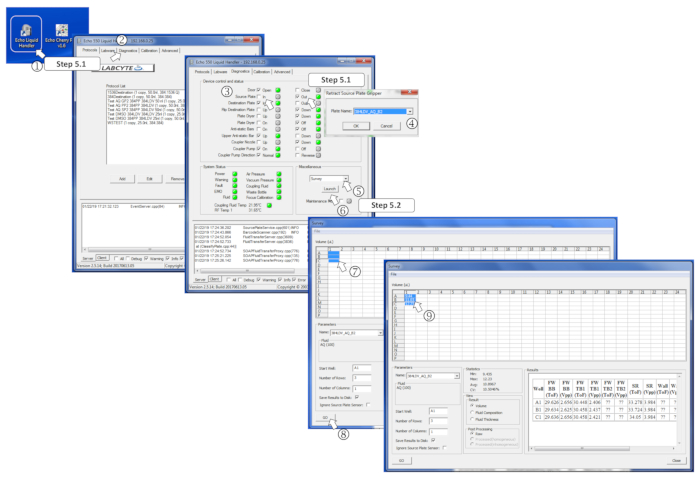
图 4:定义测量软件参数。(1) 启动纳米分配器程序。(2) 打开诊断选项卡 . (3)插入源板,勾选源板,然后,在。(4) 在提示时在菜单中定义源板类型。(5) 在"杂项"框中,在下拉菜单中选择"调查"。(6) 点击"启动"启动调查计划。(7) 选择要测量的预填充井。(8) 单击 Go 开始分析。(9) 测量完成后,测量的体积将写在相应的选定井中。请点击此处查看此图的较大版本。
6. ADE驱动的DNA分配到目的地板
- 运行选择列表软件,将 384 孔源和目标板类型分别设置为 384_LDV 和 Greiner 384PS_781096(图 5)。通过选择384LDV_AQ_B2,将器件设置为水缓冲点胶模式, 然后取消勾选"优化传输吞吐量"。

图 5:基于选择名单的豁免的性能。(1) 启动纳米分配器软件。在"协议"选项卡中,选择 (2) 样本板格式, (3) 目标板类型和 (4) 取消标记"优化传输吞吐量"。(5)选择"选择列表"选项卡 (6)单击"导入"并选择正确的 *.csv 文件(DNA-选择列表或 T.R.-选择列表)。(7) 选中后,单击"导入"。(8)单击"播放"并保存协议。(9) 通过单击"模拟"或 (10)单击"运行"启动已编程的豁免来执行分配模拟。请点击此处查看此图的较大版本。
- 选择"选择列表"选项卡,点击导入,选择DNA选择列表.csv文件。单击"播放"并保存协议。单击"模拟"以执行编程分配模拟,以确保选择列表与预期的实验设计匹配。完成后,单击"关闭"。
- 单击"播放",然后运行启动点胶程序:当系统询问时,将请求的源板(DNA 溶液手动填充)和目标板(稀释剂填充)插入纳米点胶器中。
注:整个 384 孔板的点胶时间约为 5-20 分钟,具体取决于所选体积和实验设计中的分配总数。 - 或者,暂停这里的协议,因为稀释剂和DNA填充的板可以处理干或冷冻存储长达7天。对于干燥存储,让盘子在室温下在工作台上干燥,然后以同样的方式存放。解冻和离心机(在1,500 x g下2分钟)冷冻储存的板,然后再用于转染步骤(第7节)。
7. ADE驱动的转染试剂分配
- 在生物安全柜中,在无血清介质中暂时稀释脂聚聚物转染试剂至1x最终浓度。Vortex,并立即根据宏设计的预定义源板(s)分配此转染试剂混合物,并使用步骤 3.4 中所述的预校准 384 孔移液导应用。
注:一旦将源板装上试剂,请勿将其离心,因为离心后不会注意到转染。 - 运行 nano 分给器程序以执行第 5 节所述的"测量",以便控制源板的所有手动填充TR孔的体积,以避免因体积超过 12 μL 而导致的点胶错误。
- 单击"重置"以清除选择列表软件中 DNA 选择列表的样本列表,并验证设备参数是否仍设置为水缓冲器以及所使用的源板类型和目标板类型,如步骤 6.1 所示。
- 单击"导入"并选择 TR-选择列表.csv 文件。单击"播放"并保存协议(如果出现提示),并且(这是可选的,但强烈建议)执行编程转染试剂混合物的模拟,通过单击模拟按钮。完成后,单击"关闭"。
- 单击"播放", 然后运行按钮启动点胶程序:根据请求,将源板(TR 混合填充)和目标板(稀释剂和 DNA 填充)放入纳米点胶器中。
注:在分配 500 nL TR 混合物时,整个 384 孔板的点胶时间小于 20 分钟。 - 在将TR添加到DNA后,在室温下孵育15-30分钟,如制造商的协议所示。
8. 基于蠕动液体处理程序的细胞分配
- 准备蠕动液体处理程序,用于分配细胞。使用 Aniospray Surf 29 消毒剂喷洒 10 μL 盒式头并吸收纸张上的残留物,对其进行消毒。将盒式卡盒安装在蠕动液体处理程序设备上,将盒式类型设置更改为 10 μL,并确保板格式设置为 384 孔。
- 按照步骤 4.2 中所述对 10 μL 盒式管进行消毒。在无菌容器中潜水管,用5 mL的70%酒精冲洗管,然后用5 mL的蒸馏水,最后,用5 mL的无血清介质,连续填充在同一容器中,直到每个管都是空的。
- 准备细胞悬浮液以点胶。从交集的HeLa细胞B10培养皿中,用1倍磷酸盐缓冲盐水(PBS)溶液清洗细胞1x,然后在37°C下用胰蛋白酶/EDTA分离细胞5分钟。
- 通过在培养盘中加入10 mL的完整培养基(DMEM辅以10%胎儿牛血清和100 U/mL青霉素-链霉素;见材料表),验证显微镜下细胞分离,并停止胰蛋白酶/EDTA作用。在50 mL管中采集细胞,使用马拉塞兹细胞或自动细胞计数器在显微镜下对细胞进行计数。
- 在完整培养基(即1,500个细胞/40μL)中,以37,500个细胞/mL的浓度制备至少25 mL的HeLa细胞悬浮液,以制作一个完整的384孔板,以确保管注和40μL/孔分配。
- 要分配细胞,用制备的细胞悬浮液填充新的无菌容器,并搅拌它,以避免沉淀导致分配细胞密度不准确。将管式组织者插入此溶液中,然后按Prime按钮,直到电池悬架开始从点胶头冲洗。通过目视检查所有管的液体流量,确保没有堵塞任何尖端,并确保每个管都装有电池悬浮液。
- 将 DNA 和 TR 填充的 384 孔目的地板加载到蠕动液体处理程序板托架上,并取下其盖子。运行预校准程序,在完整的 384 孔板(即 1,500 个电池/孔)上分配 40 μL 的电池悬浮液。点胶时间约为 8 s,更换 384 孔板的盖子。
注:或者,可以使用多通道微移液器手动分配 40 μL 电池悬架。
9. 定制生物测定(细胞转染效率监测)
注:按照实验设置和实验意图,使用所需的发光、荧光、高含量筛选和逆转录定量聚合酶链反应(RT-qPCR)的方法。在协议的这一节中,细胞转染效率是通过自动荧光显微镜和图像分析来评估的。
- 在水饱和环境中用5%的CO2在37°C下孵育板,直到蛋白质表达正常。
注:这里,使用TdTomato-和mVenus表达的质粒,为HeLa细胞使用48小时的孵育时间来监测转染效率。 - 通过反转板去除培养基48 h转染后,使用蠕动液体处理程序(10μL盒),加入30μL/孔10%的正口,并在室温下孵育15分钟。
- 通过反转板来去除形式;然后,在室温下孵育细胞15分钟,在1xPBS溶液中稀释0.1纳克/mL Hoechst。
- 将80μL的1x PBS调整至pH= 8,将细胞清洗3倍15分钟,以恢复形式素溶液孵育步骤6.9 pH损失的高荧光信号。
- 使用自动荧光显微镜,以10倍物镜和适当的发射滤镜集(4,6-二酰胺-2-phenyininole[DAPI]、dsRed和荧光素)按顺序获取两个或三个荧光通道(Hoechst、tdTomato和mVenus)的图像等子亚酸[FITC],分别)。
- 为了评估转染效率,使用图像分析软件,使用基于核染色的脚本分析来确定转染效率。
结果
f 为了确定ADE技术是否可用于自动反向转染协议,我们使用红色荧光tdTomato表达质粒,通过荧光显微镜监测细胞转染效率。首先,为了确定最佳转染参数,对不同的稀释量和总DNA进行了交叉测试。稀释体积用于允许DNA液滴,一旦分配,蔓延到整个井,以规避在初步实验中观察到的不均匀转染(即,仅在井的中心)。如图6A所示,使用脂聚聚物试剂20转...
讨论
为给定细胞系建立和优化精确的高通量转染方法需要科学家遵循本节中描述的一些关键参数。我们强烈鼓励从整个协议中的建议值开始,因为这些针对 HeLa 细胞优化的设置也证明对 HEK 细胞有效。然而,由于最佳参数可能取决于细胞系和转染试剂,因此可以通过改变细胞数量、稀释体积、总DNA量和转染试剂的性质、浓度,甚至体积来定义最佳条件。在HeLa细胞1优化该协议期间。
披露声明
作者没有什么可透露的。
致谢
作者披露了对本文的研究、作者和/或出版物的下列财政支持:因塞姆、里尔大学、里尔巴斯德研究所、北方理事会以及PRIM-HCV1和2(Recherche)(联邦国际医药协会、国家重建局(ANR-10-EQPX-04-01)、Feder(12001407(D-AL)和欧洲共同体(ERC-STG INTRACELLTB n=260901)。作者要感谢S.Mouru博士、B.Villemagne博士、R.Ferru-Clément博士和H.Groult博士对手稿的批判性回顾和更正。
材料
| Name | Company | Catalog Number | Comments |
| 384LDV Microplate | Labcyte | LP-0200 | |
| 384-well Microplate μClear Black | Greiner | 781906 | |
| Ampicilin | Sigma | A9393-5G | Selection antibiotic for bacteria transformed with ampicilin expressing vector |
| Android Tablet | Samsung | Galaxy Note 8 | used to guide the user while the source plate manual dispense |
| Aniospray Surf 29 | Anios | 2421073 | disinfectant to clean the MicroFlo head |
| Columbus software | Perkin Elmer | image analysis software | |
| Dulbecco's Modified Eagle Medium (DMEM), high glucose, GlutaMAX Supplement, pyruvate | Thermo Fisher Scientific | 10566032 | cell culture medium |
| Echo Cherry Pick 1.5.3 software | Labcyte | Software enabling ADE-based dispenses by the Echo550 device from a *.csv file; nanodispenser software | |
| Echo550 | Labcyte | ADE-based dispenser | |
| Fetal Bovine Serum | Thermo Fisher Scientific | 16000044 | to add in cell culture medium |
| Formalin solution, neutral buffered, 10% | Sigma-Aldrich | HT501128-4L | to fix cell |
| HeLa cells | ATCC | HeLa (ATCC® CCL-2™) | |
| Hoechst 33342, Trihydrochloride, Trihydrate | Thermo Fisher Scientific | H3570 | 10 mg/mL Solution in Water |
| INCell Analyzer 6000 | GE Healthcare | 29043323 | automated laser-based confocal imaging platform |
| LB medium | Thermoischer Scientific LB Broth Base (Lennox L Broth Base)®, powder | 12780052 | culture medium for bacteria growth |
| Lysis Buffer (A2) | Macherey-Nagel | 740912.1 | Buffer from the NucleoSpin Plasmid kit used to prepare plasmid from bacterial culture |
| MicroFlo 10µL cassette | Biotek Instruments Inc | 7170013 | to use with the Microflo Dispenser |
| MicroFlo 1μL cassette | Biotek Instruments Inc | 7170012 | to use with the Microflo Dispenser |
| MicroFlo Dispenser | Biotek Instruments Inc | 7171000 | peristaltic pump-based liquid handler device |
| Microvolume spectrophotometer | Denovix | DS-11 Spectrophotometer | Measure the DNA concentration of samples |
| mVenus plasmid | mVenus cDNA was cloned by enzymatic restriction digestion and ligation in Age1/BsrG1 sites of the tdTomato-N1 plasmid | Vector type: Mammalian Expression, Fusion Protein: mVenus | |
| Neutralization Buffer (A3) | Macherey-Nagel | 740913.1 | Buffer from the NucleoSpin Plasmid kit used to prepare plasmid from bacterial culture |
| NucleoSpin Plasmid kit | Macherey-Nagel | 740588.50 | used to prepare plasmid from bacterial culture |
| Optimal-Modified Eagle Medium (Opti-MEM) Medium | Thermo Fisher Scientific | 31985070 | |
| optional Wash bufferWash Buffer (A4) | Macherey-Nagel | 740914.1 | Buffer from the NucleoSpin Plasmid kit used to prepare plasmid from bacterial culture |
| orbital shaker | incubated large capacity shaker | 444-7084 | Used to grow bacteria under gentle agitation and 37°C |
| Penicillin-Streptomycin | Thermo Fisher Scientific | 15140122 | 10,000 U/mL |
| Phosphate-Buffered Saline | Thermo Fisher Scientific | 10010001 | |
| Plasmid mini-columns | Macherey-Nagel | 740499.250 | Silica membrane mini-column to prepare plasmid from bacterial culture |
| Resuspension Buffer (A1) | Macherey-Nagel | 740911.1 | Buffer from the NucleoSpin Plasmid kit used to prepare plasmid from bacterial culture |
| RNAse A | Macherey-Nagel | 740505 | Enzyme from the NucleoSpin Plasmid kit used to prepare plasmid from bacterial culture |
| tdTomato-N1 plasmid | Addgene | Plasmid #54642 | Vector type: Mammalian Expression, Fusion Protein: tdTomato |
| TransIT-X2 Dynamic Delivery System | Mirus Bio | MIR 6000 | |
| Wash Buffer (AW) | Macherey-Nagel | 740916.1 | Buffer from the NucleoSpin Plasmid kit used to prepare plasmid from bacterial culture |
| 3D printer | Creality | CR10S | used to print the plate adapter |
| Blender Software | https://www.blender.org/ Free software under GNU General Public License (GPL). | version 2.79b | used to design the plate adapter |
参考文献
- Colin, B., Deprez, B., Couturier, C. High-Throughput DNA Plasmid Transfection Using Acoustic Droplet Ejection Technology. SLAS Discovery: Advancing Life Sciences R & D. , 2472555218803064 (2018).
- Mirus Bio. . Optimising Transfection Performance. , (2019).
- Thermo Fisher Scientific. . Factors Influencing Transfection Efficiency | Thermo Fisher Scientific - FR. , (2019).
- Boussif, O., et al. A versatile vector for gene and oligonucleotide transfer into cells in culture and in vivo: polyethylenimine. Proceedings of the National Academy of Sciences of the United States of America. 92 (16), 7297-7301 (1995).
- Figueroa, E., et al. A mechanistic investigation exploring the differential transfection efficiencies between the easy-to-transfect SK-BR3 and difficult-to-transfect CT26 cell lines. Journal of Nanobiotechnology. 15 (1), 36 (2017).
- Kirchenbuechler, I., Kirchenbuechler, D., Elbaum, M. Correlation between cationic lipid-based transfection and cell division. Experimental Cell Research. 345 (1), 1-5 (2016).
- Zhang, Z., Qiu, S., Zhang, X., Chen, W. Optimized DNA electroporation for primary human T cell engineering. BMC Biotechnology. 18 (1), 4 (2018).
- Cao, D., et al. Transfection activity and the mechanism of pDNA-complexes based on the hybrid of low-generation PAMAM and branched PEI-1.8k. Molecular bioSystems. 9 (12), 3175-3186 (2013).
- Bos, A. B., et al. Development of a semi-automated high throughput transient transfection system. Journal of Biotechnology. 180, 10-16 (2014).
- Colosimo, A., et al. Transfer and expression of foreign genes in mammalian cells. BioTechniques. 29 (2), 314-318 (2000).
- Villa-Diaz, L. G., Garcia-Perez, J. L., Krebsbach, P. H. Enhanced transfection efficiency of human embryonic stem cells by the incorporation of DNA liposomes in extracellular matrix. Stem Cells and Development. 19 (12), 1949-1957 (2010).
- Sabatini, D. M. . Reverse transfection method. , WO2001020015A1 (2001).
- Raymond, C., et al. A simplified polyethylenimine-mediated transfection process for large-scale and high-throughput applications. Methods (San Diego, CA). 55 (1), 44-51 (2011).
- Junquera, E., Aicart, E. Recent progress in gene therapy to deliver nucleic acids with multivalent cationic vectors. Advances in Colloid and Interface Science. 233, 161-175 (2016).
- Woodruff, K., Maerkl, S. J. A High-Throughput Microfluidic Platform for Mammalian Cell Transfection and Culturing. Scientific Reports. 6, 23937 (2016).
- Vasileiou, T., Foresti, D., Bayram, A., Poulikakos, D., Ferrari, A. Toward Contactless Biology: Acoustophoretic DNA Transfection. Scientific Reports. 6, 20023 (2016).
- Hadimioglu, B., Stearns, R., Ellson, R. Moving Liquids with Sound: The Physics of Acoustic Droplet Ejection for Robust Laboratory Automation in Life Sciences. Journal of Laboratory Automation. 21 (1), 4-18 (2016).
- Grant, R. J., et al. Achieving accurate compound concentration in cell-based screening: validation of acoustic droplet ejection technology. Journal of Biomolecular Screening. 14 (5), 452-459 (2009).
- Sackmann, E. K., et al. Technologies That Enable Accurate and Precise Nano- to Milliliter-Scale Liquid Dispensing of Aqueous Reagents Using Acoustic Droplet Ejection. Journal of Laboratory Automation. 21 (1), 166-177 (2016).
- Day, R. N., Davidson, M. W. The fluorescent protein palette: tools for cellular imaging. Chemical Society Reviews. 38 (10), 2887-2921 (2009).
- Zielinski, D., Gordon, A., Zaks, B. L., Erlich, Y. iPipet: sample handling using a tablet. Nature Methods. 11 (8), 784-785 (2014).
- Brunner, S., et al. Cell cycle dependence of gene transfer by lipoplex, polyplex and recombinant adenovirus. Gene Therapy. 7 (5), 401-407 (2000).
- Nii, T., et al. Single-Cell-State Culture of Human Pluripotent Stem Cells Increases Transfection Efficiency. BioResearch Open Access. 5 (1), 127-136 (2016).
- Noonan, D. J., Henry, K., Twaroski, M. L. A High-Throughput Mammalian Cell-Based Transient Transfection Assay. Signal Transduction Protocols. 284, 051-066 (2004).
- . . Transfection | TransIT Transfection Reagents | Mirus Bio. , (2015).
- American Type Culture Collection. . General protocol for transfection of stem cells, primary cells, and continuous cell lines with ATCC TransfeX Transfection Reagent. , (2017).
- American Type Culture Collection. . Transfection Reagents for Nucleic Acid Transfer into ATCC Cells. , (2017).
- de Los Milagros Bassani Molinas, M., Beer, C., Hesse, F., Wirth, M., Wagner, R. Optimizing the transient transfection process of HEK-293 suspension cells for protein production by nucleotide ratio monitoring. Cytotechnology. 66 (3), 493-514 (2014).
- Promega. . FuGENE® 6 Transfection Reagent. , (2019).
- Olden, B. R., Cheng, Y., Yu, J. L., Pun, S. H. Cationic polymers for non-viral gene delivery to human T cells. Journal of Controlled Release: Official Journal of the Controlled Release Society. 282, 140-147 (2018).
- Park, E., Cho, H. B., Takimoto, K. Effective gene delivery into adipose-derived stem cells: transfection of cells in suspension with the use of a nuclear localization signal peptide-conjugated polyethylenimine. Cytotherapy. 17 (5), 536-542 (2015).
- Wood, R. W., Loomis, A. L. The physical and biological effects of high-frequency sound-waves of great intensity. The London, Edinburgh, and Dublin Philosophical Magazine and Journal of Science. 4 (22), 417-436 (1927).
- Mamat, U., et al. Eliminating Endotoxin at the Source - A Novel Competent Cell Line with Modified Lipopolysaccharide for Low-Endotoxin Plasmid Production. , (2014).
- Ivanova, N. V., Kuzmina, M. L. Protocols for dry DNA storage and shipment at room temperature. Molecular Ecology Resources. 13 (5), 890-898 (2013).
- Lesnick, J., Lejeune-Dodge, A., Ruppert, N., Jarman, C. . High-Precision Cell Dispensing with the Labcyte Echo® Liquid Handler. , (2017).
- Yang, X., et al. A public genome-scale lentiviral expression library of human ORFs. Nature Methods. 8 (8), 659-661 (2011).
- Peng, J., Zhou, Y., Zhu, S., Wei, W. High-throughput screens in mammalian cells using the CRISPR-Cas9 system. The FEBS journal. 282 (11), 2089-2096 (2015).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。