需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
用于评估肿瘤细胞中的微RNA水平、功能和相关靶基因的体外协议
摘要
该协议使用基于探针的实时聚合酶链反应(PCR)、磺胺胺B(SRB)测定、3'未翻译区域(3'UTR)克隆和荧光素酶测定来验证感兴趣的miRNA的目标基因,并了解miRNA的功能。
摘要
微RNA (miRNA) 是小型调节RNA,可识别调节包括癌症在内的多种疾病中的许多细胞内信号通路。这些小型调节RNA主要与其目标信使RNA(mRNA)的3'未翻译区域(3'UTR)相互作用,最终导致mRNA解码过程的抑制和目标mRNA降解的增强。基于表达水平和细胞内功能,miRNA能够作为致癌和肿瘤抑制性mRNA的调节因子。在数百甚至数千个计算预测目标中识别 miRNA 的真正目标基因是识别感兴趣的 miRNA 的作用和基本分子机制的关键步骤。各种miRNA目标预测程序可用于搜索可能的miRNA-mRNA相互作用。然而,最具挑战性的问题是如何验证感兴趣的miRNA的直接靶基因。该协议描述了如何识别与miRNA功能相关的miRNA靶点的关键方法的可重复策略。该协议提供了一个实用的指南,用于使用基于探针的实时聚合酶链反应(PCR)、磺胺B(SRB)测定,在miRNA模拟转染后发现miRNA水平、功能和相关目标mRNA。,剂量反应曲线生成,和荧光素酶测定以及基因的3'UTR克隆,这是正确理解单个miRNA的作用所必需的。
引言
微RNA(miRNA)是一种小型调节RNA,主要通过对真实靶基因1中的3'未翻译区域(3'UTR)做出反应来调节信使RNA(mRNA)的转换和降解过程。miRNA的表达可以通过转录和转录后机制调节。这种调节机制的不平衡导致包括癌症在内的众多疾病中不受控制和独特的miRNA表达水平。单个 miRNA 可以与不同的 mRNA 进行多种交互。相应地,单个 mRNA 可以由各种 miRNA 控制。因此,细胞内信号网络受到明显表达的miRNA的复杂影响,通过这种影响,生理疾病和疾病可以启动和恶化2,3,45,6.虽然在各种癌症中观察到了miRNA的改变表达,但调节癌细胞与miRNA一起进行的分子机制在很大程度上仍不为人所知。
累积的证据表明,miRNA的致癌或肿瘤抑制作用取决于癌症的类型。例如,通过瞄准叉头盒o3(FOXO3),miR-155促进结肠直肠癌7、8的细胞增殖、转移和化学抗药性。相比之下,胶质瘤细胞入侵的限制性是由miR-107通过神经原位位同源蛋白2(NOTCH2)表达9的调节引起的。评估miRNA-目标相互作用与miRNA功能是一个不可或缺的部分,更好地了解miRNA如何调节各种生物过程在健康和疾病状态10。此外,miRNA真正靶点的发现,可以进一步为基于miRNA的抗癌药物治疗提供微调的策略。然而,miRNA领域的主要挑战是确定miRNA的直接目标。在这里,详细的方法作为可重复的实验方法,用于miRNA靶基因的测定。miRNA目标鉴定的成功实验设计涉及各种步骤和注意事项(图1)。比较肿瘤细胞和正常细胞中的成熟miRNA水平是选择感兴趣的miRNA的常见程序之一(图1A)。对选定的miRNA进行功能研究,以检测miRNA对细胞增殖的影响,对于缩小感兴趣的miRNA最佳潜在候选靶点列表非常重要(图1B)。基于miRNA的实验验证功能,需要用miRNA靶向预测程序对公司文献和数据库进行系统审查,以搜索与基因功能最相关的信息(图1C)。通过实施荧光素酶测定以及基因3'UTR的克隆、实时PCR和西方印迹(图1D),可以识别感兴趣的miRNA的真实目标基因。当前协议的目标是提供关键实验、基于探针的实时聚合酶链反应 (PCR)、磺胺 B (SRB) 分析后 miRNA 模拟转染、剂量-反应曲线生成以及荧光酶测定以及基因3'UTR的克隆。目前的协议对于更好地了解单个 miRNA 的功能以及 miRNA 在癌症治疗中的含义非常有用。
Access restricted. Please log in or start a trial to view this content.
研究方案
1. 成熟微RNA (miRNA) 表达分析
-
成熟 miRNA 补充 DNA (cDNA) 合成
- 加入总RNA的254纳克和4.5μL的脱氧核糖核酸I(DNase I)混合物,然后将超纯水加入PCR带管,以补制成高达18μL(图2A)。根据反应总数,使用足够量的DNase I混合物,制备从多个细胞系纯化的每个总RNA样品的反应。
注:DNase I混合物由DNase I(1.8 μL)、核糖酸酶抑制剂(0.3 μL)和25 mM MgCl 2(2.4 μL)组成。为了重新获取总RNA,应用了一种基于柱的提取方法,而不是使用基于苯酚氯仿的提取方法。据报道,使用基于苯酚氯仿的提取方法11、12时,某些miRNA的提取产量可能因细胞数量而异。 - 在热循环器中孵育管子。在37°C下运行管子10分钟,在90°C下进行5分钟孵育,热灭活DNase I。孵育后立即将管子放在冰上。
- 将7.1 μL的DNaseI处理总RNA到2组新管中,然后加入1.5μL的反义引基,用于甘油醛-3-磷酸脱氢酶(GAPDH)基因(图2B)。
注:在此步骤中,cDNA合成的总RNA量为100 ng。GAPDH反义引物的库存浓度为10μM。添加GAPDH反义引物是使用基因特异性引物方法生成GAPDH cDNA的。 - 使用热循环器孵育管。在80°C下开始5分钟,随后在60°C下反应5分钟。孵育后立即将管子放在冰上。
- 在每个反应中加入3.4 μL的逆转录(RT)酶混合物(图2B)。RT酶混合物由100mM脱氧核苷酸三磷酸盐(0.15μL)、10xRT缓冲液(1.5μL)、核糖酸酶抑制剂(0.75μL)和逆转录酶(1μL)组成。根据反应总数准备足够的混合物。
- 在每个反应中为特定的miRNA添加3μL的5倍RT引漆(图2B)。
注:每种反应的总体积为15μL。 - 使用热循环器运行管。在16°C下开始30分钟,随后在42°C下反应30分钟,最后在85°C下持续5分钟,在4°C下保持任何剩余时间(图2B)。单链cDNA在此步骤中为同一管中的特定miRNA和GAPDH基因生成。
- 加入总RNA的254纳克和4.5μL的脱氧核糖核酸I(DNase I)混合物,然后将超纯水加入PCR带管,以补制成高达18μL(图2A)。根据反应总数,使用足够量的DNase I混合物,制备从多个细胞系纯化的每个总RNA样品的反应。
-
实时聚合酶链反应(PCR)和数据分析
- 以1:49的比例用超纯水稀释每个cDNA。
- 为特定的miRNA和GAPDH制备反应混合物(表1)。为了检测特定的miRNA和GAPDH,为每个cDNA样品设置三联体反应。
- 执行实时 PCR 和数据分析 (图 2C)。使用比较 CT方法13、14分析数据。
2. 微RNA(miRNA)模拟转染
注:miRNA-107是从步骤1中选择的。由于miRNA-107在肿瘤细胞中与正常细胞相比具有低调节性,因此可以推测miRNA-107是一种肿瘤抑制miRNA。在肿瘤细胞中与正常细胞(如miRNA-301)相比,在肿瘤细胞中向上调节的miRNA,对miRNA-301的反义寡核苷酸可应用于步骤2、3和4。
- 使用计数室装置对细胞进行计数,并在 96 孔板中为细胞板板。细胞密度为每口孔的细胞密度为2 x 103个细胞/100 μL。不要使用含有青霉素-链霉素(P/S)的细胞培养基,因为P/S会降低转染效率。
-
准备一组转染混合物,在第二天以miRNA控制模拟和miRNA-107模拟的几种最终浓度转染细胞(图3)。
- 从miRNA控制模拟或miRNA-107模拟的库存(25μM浓度),稀释和添加相应量的控制模拟或miRNA-107模拟在减少血清培养中,以及转染试剂使用微离心管(图3A)。使用微移液器轻轻混合含有混合物的寡果。寡聚物的总量(miRNA模拟控制 + miRNA-107 模拟)在每个井中应相同。空白孔包括100μL的细胞培养基和含有无细胞转染试剂的降血清培养基。
- 在细胞培养罩中孵育10分钟后,再次轻轻混合含有混合物的寡果,然后将50μL的混合物加入每个孔中。将转染细胞保存在细胞培养箱中。在6-12小时孵育后,用含有胎儿牛血清(FBS)和P/S的新鲜细胞培养基剂替换含有培养基的转染试剂。进一步孵育细胞72小时。miRNA模拟的总治疗持续时间为96小时。
3. 硫二胺 B (SRB) 测定
-
细胞固定
- 取出板中每个孔中的细胞培养基,并迅速将100μL的10%三氯乙酸(TCA)填充到每个孔中。小心地从每个孔中吸出细胞培养基,以避免任何细胞损伤和脱离底部。
注:在 50 mL 蒸馏水中加入 20 g TCA 粉末,制备 40% TCA。从 40% TCA 起,通过蒸馏水以 1:3 稀释比稀释 40% TCA,使 TCA 达到 10%。 - 将含有 10% TCA 的盘子放在冰箱 (4 °C) 中 1 小时。
- 将盘子浸入水箱中,然后晾干,洗几次盘子。通过敲击板,清除井内多余的水,直到井中没有剩余水。在进行下一步之前,将盘子放在实验室工作台上晾干。
- 取出板中每个孔中的细胞培养基,并迅速将100μL的10%三氯乙酸(TCA)填充到每个孔中。小心地从每个孔中吸出细胞培养基,以避免任何细胞损伤和脱离底部。
-
细胞染色
- 移液器 50 μL 0.4% SRB 溶液进入每个井中,包括空白井。轻轻摇动板,直到 0.4% SRB 溶液始终覆盖井底。
注:将 0.4 g SRB 粉末加入 100 mL 的 1% 醋酸中,制备和使用 0.4% SRB 溶液。仔细摇动溶液以混合。将 0.4% SRB 溶液的瓶子包裹在铝箔等轻质保护材料中。将 0.4% SRB 溶液存放在冰箱中。 - 孵育40分钟至60分钟后,用1%醋酸冲洗板洗板。清洗板,直到未绑定的染料被完全冲走 (图 3B)。
- 在进行下一步之前,将盘子放在实验室工作台上晾干。
注:在进行步骤 3.3 之前,板应完全干燥。
- 移液器 50 μL 0.4% SRB 溶液进入每个井中,包括空白井。轻轻摇动板,直到 0.4% SRB 溶液始终覆盖井底。
-
吸收测量
- 将Tris基溶液(10 mM)移液器100μL插入相应的井中,包括空白井。将板放在摇床上 10 分钟,测量 492 nm 的吸光度。
4. 生成剂量-响应曲线
- 分析电子表格中的 SRB 分析数据。从每个组的吸收值中减去空白吸收率,并计算每个组的吸收值的平均值 (AVE) 和标准差 (STD)。
- 计算平均吸收率百分比 (AVE%)以及标准差(STD%)的偏差使用 SRB 测定的吸收值的每个组。
注:miRNA对照模拟治疗组的AVE%为100%。使用以下公式计算 STD%: STD% = (每个组的 STD / 对照模拟处理组的 AVE 吸收率) x 100。 - 通过垂直对齐这些数据,将原始数据(包括治疗浓度、AVE% 和 STD%) 导入软件。由于未定义日志 0,因此将 X 轴的第一个浓度设置为接近 0 的值(例如,0.01)。
- 单击"创建图形"选项卡并选择"简单散点误差栏"。选择工作表列作为符号值,然后单击"下一步"。在数据格式面板中,选择XY对,然后单击"下一步"。在"选择数据"面板中选择相应的数据列。单击"完成"按钮以创建绘图。
注:X 轴表示浓度,Y 轴指示每个浓度的平均吸收率 (AVE)百分比,误差条指出每个浓度的标准偏差百分比 (STD%)。 - 双击 X 轴可修改轴的比例类型和缩放。将比例类型从线性更改为日志。分别将开始和结束范围编号修改为 0.01 和 200。
-
右键单击任何散点图,选择"曲线拟合",然后转到用户定义的子类别。选择剂量响应曲线,单击"下一步"按钮,然后单击"完成"按钮。剂量-响应曲线现在与报告选项卡一起生成 (图 4A)。
- 要在软件中输入公式 1 以生成剂量-响应曲线,请单击"分析"选项卡并选择"回归向导"。转到公式类别中的用户定义,然后单击"新建"按钮。将公式 1、变量、初始参数和约束插入相应的空白框(图 4B,C)。单击"添加为"按钮并将方程的名称设置为剂量-响应曲线。公式名称现在生成在公式类别中的用户定义的子类别中。f表示公式 1 中细胞活力(细胞存活百分比)的百分比。
公式 1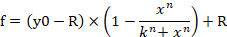
- 转到报表选项卡,然后检查 n、k 和 R 值。
注:y0表示miRNA对照模拟组100%细胞存活,n表示希尔型系数(图的斜率),k表示miRNA-107模拟的浓度,产生50%的miRNA-107模拟的最大效果(一半最大抑制浓度,IC50),R 表示残留未受影响的分数(电阻分数)15。用于生成剂量-响应曲线的方程将 y0 到 R 值(如果有)的范围从 100% 识别为 100%(图 4A)。因此,有必要获取基于 y0 到零值范围计算的调整后 k (IC50)值 (图 4A)。调整的k(IC50)以及其他ICx值(例如,IC10到IC90)可以使用公式2获得,该公式从公式1派生。公式 2 从公式 1 的推导在补充图 1中指示。
公式 2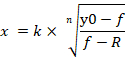
- 双击应用公式 2 的单元格上的鼠标左键。使用公式 2 和生成的剂量响应曲线中的参数,它可用于计算 ICx 的调整后值,范围从 IC10到 IC90(图 4D)。
- 输入等号后跟公式,单元格中带有括号开头。输入公式时,通过将美元符号添加到相应的列和行,将 n、k 和 R 的值固定为绝对单元格引用,以便在自动将公式填充到行时不会更改这些固定值(图 4D)。或者,可以使用公式 2 手动计算调整后的值。
注:IC90值未确定,因为 R 值大于 10。此外,如果 R 值高于 20,则 IC80的值也不确定 (图 4D)。
5. 对感兴趣的微RNA直接靶基因的验证
注:在进行SRB测定等功能实验后,miRNA-107被确认为肿瘤抑制miRNA,miRNA-107直接针对肿瘤基因是高度可行的。使用 miRNA 目标预测程序(如 TargetScan (http://www.targetscan.org/vert_71/)检查所有预测目标基因的列表,然后根据包括 PubMed 和基因卡。
- 克隆 3' 未翻译区域 (UTR) 的底漆设计
- 将基因名称放入GeneCards(https://www.genecards.org/)中,然后单击基因符号。 通过单击基因的Ensembl ID来评估 Ensembl 基因组浏览器,然后单击成绩单表中的转录ID。之后,单击左侧基于脚本的显示列表中存在Exons。
- 复制 3' UTR 的核苷酸序列并将其粘贴到引物设计程序中。再次从该程序复制序列并将其粘贴到字处理器中。检查miRNA结合序列的存在,以及是否存在用于克隆的限制酶位点。
注:如果 3' UTR 中没有限制酶识别位点,则为克隆选择的限制酶可用于下一步。 - 在引体设计程序中,接受 3' UTR 序列,并开始设计具有以下条件的正向和反向引力。长度:20-30核苷酸,Tm:45-58 °C,GC%:40-60%。两个引基的 Tm 值之间的差值应小于 5 °C。本研究中使用的引种序列在补充图2中提供。在设计引体中添加限制性酶识别序列以及4个随机核苷酸。
- 梯度PCR
- 制备25μL的PCR反应混合物,包括每一退火温度设计的引体(表2)。根据反应总数准备足够的混合物。通过移液混合溶液,并将25μL的反应混合物加入每个管中。使管离心几秒钟。
- 执行从变性步骤到扩展步骤的 35-40 PCR 周期。将 PCR 循环设置为以下步骤:98 °C 1 分钟(1 个循环,聚合酶激活步骤),95 °C 10 s(变性步骤),45 °C-68 °C 30 s(退火步骤),68 °C(延长步长,每 1000 bp 10 s 1min),68 °C 3 分钟(终止步骤),最后冷却到4°C。
- 运行 PCR 产品,并在 1% 的糖凝胶上使用 DNA 阶梯检查带。找到最佳退火温度 (图 5A)。在下一步,使用最佳退火温度再次放大基因的 3' UTR。
- 双重消化
- 使反应混合物包括两种限制性酶,XhoI(或AsiSI)和NotI,在一个管(表3)。使用水浴(37 °C)孵育混合物3-4小时。
- 在1%的甘蔗凝胶上运行双重消化产物,然后在紫外线下切割带。在荧光酶载体的情况下,在凝胶上运行之前,用10U的碱性磷酸酶反应双消化载体,再持续1小时,以防止在结扎步骤中重新循环。
- 从切除的带中纯化双消化PCR产物和荧光素酶载体。
- PCR 产物与荧光素酶载体的结扎
- 使20μL的结扎反应混合物,包括DNA结合酶(表4)。
注:PCR 产物(插入)与荧光素酶载体的摩尔比可以是 3:1。1:1 或 2:1。 - 将管短暂离心10-15s,使用热循环器在16°C孵育过夜。
注:或者,该管可在4°C下孵育2-3天进行结扎。在此步骤中,PCR 插入物将被克隆到位于雷尼拉报告基因下游的区域(图 5B)。将miRNA结合到基因的克隆3'UTR中,可以降低肾上体的活性。萤火虫荧光素酶用于雷尼拉表达水平的规范化。
- 使20μL的结扎反应混合物,包括DNA结合酶(表4)。
- 转化和菌落 PCR
- 将结扎混合物(3-5 μL)加入含有合格细胞的管中。轻轻敲击管子,将其放在冰上(20 分钟)。
注:在加入结扎混合物之前,在冰上解冻合格的细胞。 - 快速、轻轻地将管转移到热块。热休克后(42 °C,30 s-1 分钟),将管子放在冰上 20 分钟。
- 在卢里亚-贝尔塔尼 (LB) 琼脂板上传播合格的细胞。在孵化器(37°C)中一夜之间生长出合格的细胞。
注:琼脂素(50-100 μg/mL)包含在琼脂板中。 - 挑选单个菌群,在含有超纯水的8条管中重新悬浮大肠杆菌。重复此步骤,从随机选择的4-8菌落中重新悬浮大肠杆菌(图5C)。
- 将25μL的大肠杆菌悬浮液转移到另一组8条管中。现在,有2组大肠杆菌悬浮液管。
注:一个管用于殖民地PCR,另一个用于接种。用于接种的大肠杆菌悬浮液可暂时储存在4°C(图5C)。 - 使用大肠杆菌悬浮液执行菌落 PCR。此步骤旨在确定菌落是否包含插入。选择最佳菌落接种和分离含有3'UTR基因的荧光酶载体(图5C)。
注:对所选基因的每个 3' UTR 重复步骤 5.1-5.5。遵循表2所示的PCR反应状况,用大肠杆菌悬浮液替换基因组DNA。
- 将结扎混合物(3-5 μL)加入含有合格细胞的管中。轻轻敲击管子,将其放在冰上(20 分钟)。
- 路西法酶测定
- 准备 24 孔板。在每个孔使用1-2 x 104个细胞在500μL细胞培养基。不要使用含有P/S的细胞培养基进行转染,因为使用P/S会降低转染效率。
- 通过对照模拟或使用转染试剂模拟特定的miRNA(图5D)将荧光素酶载体的50 ng转染到细胞中。如果筛选特定 miRNA 在多个浓度下模拟的效果,请保持每个井中的寡果总量不变(参见步骤 2)。
- 第二天使用磷酸盐缓冲盐水(PBS)洗净井内侧两次。
- 在测量荧光素酶活性之前,将200 μL的赖沙试剂涂入井中,并充分进行细胞赖沙。
注:将盘子放在摇盘上至少 15 分钟。 - 将5-10μL的细胞赖沙液转移到新管中,加入100μL试剂I。
注:阅读10-15s的萤火虫荧光素酶活性。 - 在同一管中加入100μL试剂II,然后通过移液混合两次。使用灯具仪读取 10-15 s 的雷尼拉荧光素酶活性。对每个示例重复步骤 5.6.5 和 5.6.6。
- 计算雷尼拉与萤火虫的比率 (图 5E)。
注:萤火虫的活动代表荧光素酶结构到细胞的转染效率。
Access restricted. Please log in or start a trial to view this content.
结果
成功和准确地确认miRNA水平对于解释基于miRNA在疾病发展和进展中预期作用的miRNA分类的数据非常重要。使用基于探针的定量PCR在三个胰腺细胞系中测量了miRNA-107和miRNA-301的水平。在相同反应中合成特定miRNA和参考基因的cDNA可以提高数据的可重复性。PANC-1和CAPAN-1是人类胰腺导管腺癌细胞系,而HPNE是一种不朽的胰腺导管细胞系,通过携带人类端粒酶逆转录酶(hTERT)基因的逆转录病毒表达载体进行转导。?...
Access restricted. Please log in or start a trial to view this content.
讨论
确定具有目标miRNA功能的真正miRNA靶点的策略对于理解miRNA的多重作用是必不可少的。miRNA靶基因的识别可以作为解释由miRNA在细胞中调节的细胞信号事件的指南。揭示miRNA的功能重要的靶基因可以为开发基于miRNA的癌症治疗提供基本知识。
微阵列、小RNA库测序、深度测序、原位PCR逆转录酶和北方印迹等多种方法,利用从细胞系和组织中分离出的总RNA,可用于探索m...
Access restricted. Please log in or start a trial to view this content.
披露声明
作者没有什么可透露的。
致谢
这项研究得到了韩国国家科学研究基金会(NRF)基础科学研究计划的支持,该基金会由韩国教育部资助(2017R1D1A3B03035662);和Hallym大学研究基金,2017年(HRF-201703-003)。
Access restricted. Please log in or start a trial to view this content.
材料
| Name | Company | Catalog Number | Comments |
| 15 mL conical tube | SPL Life Sciences | 50015 | |
| 24-well plate | Thermo Scientific | 142475 | |
| 50 mL conical tube | SPL Life Sciences | 50050 | |
| 6-well plate | Falcon | 353046 | |
| 6x DNA loading dye | Real Biotech Corporation | RD006 | 1 mL |
| 8-cap strip | Applied Biosystems | N8010535 | For cDNA synthesis |
| 8-tube strip | Applied Biosystems | N8010580 | For cDNA synthesis |
| 96-well plate | Falcon | 353072 | |
| Acetic acid | Sigma | A6283-1L | 1 L |
| Agarose A | Bio Basic | D0012 | 500 g |
| Alkaline phosphatase | New England Biolabs | M0290S | 10,000 U/mL |
| Ampicillin | Bio basic Canada Inc | AB0028 | 25 g |
| AriaMx 96 tube strips | Agilent Technologies | 401493 | For real time PCR |
| AriaMx real-time PCR system | Agilent Technologies | G8830A | qPCR amplification, detection, and data analysis |
| AsiSI | New England Biolabs | R0630 | 10,000 units/mL |
| CAPAN-1 cells | ATCC | HTB-79 | |
| Cell culture hood | Labtech | Model: LCB-1203B-A2 | |
| Counting chambers with V-slash | Paul Marienfeld | 650010 | Cells counter |
| CutSmart buffer | New England Biolabs | B7204S | 10X concentration |
| DMEM | Gibco | 11965-092 | 500 mL |
| DNA gel extraction kit | Bionics | DN30200 | 200 prep |
| DNA ladder | NIPPON Genetics EUROPE | MWD1 | 1 Kb ladder |
| DNase I | Invitrogen | 18068015 | 100 units |
| Dual-luciferase reporter assay system | Promega | E1910 | 100 assays |
| Fetal bovine serum | Gibco | 26140-079 | 500 mL |
| HIT competent cells | Real Biotech Corporation(RBC) | RH617 | Competent cells |
| HPNE cells | ATCC | CRL-4023 | |
| LB agar broth | Bio Basic | SD7003 | 250 g |
| Lipofectamine 2000 | Invitrogen | 11668-027 | 0.75 mL |
| Lipofectamine RNAiMax | Invitrogen | 13778-075 | 0.75 mL |
| Luminometer | Promega | Model: E5311 | |
| Microcentrifuge tube | Eppendorf | 22431021 | |
| Microplate reader | TECAN | Infinite F50 | |
| miRNA control mimic | Ambion | 4464058 | 5 nmole |
| miRNA-107 mimic | Ambion | 4464066 | 5 nmole |
| miRNeasy Mini Kit | Qiagen | 217004 | 50 prep |
| Mupid-2plus (electrophoresis system) | TaKaRa | Model: AD110 | |
| NotI | New England Biolabs | R3189 | 20,000 units/mL |
| Oligo explorer program | GeneLink | For primer design | |
| Optical tube strip caps (8x Strip) | Agilent Technologies | 401425 | For real time PCR |
| Opti-MEM | Gibco | 31985-070 | 500 Ml |
| PANC-1 cells | ATCC | CRL-1469 | |
| Penicillin/streptomycin | Gibco | 15140-122 | 100 mL |
| Phosphate buffer saline | Gibco | 14040117 | 1000 mL |
| Plasmid DNA miniprep S& V kit | Bionics | DN10200 | 200 prep |
| PrimeSTAR GXL DNA polymerase | TaKaRa | R050A | 250 units |
| Shaker | TECAN | Shaking platform | |
| Shaking incubator | Labtech | Model: LSI-3016A | |
| Sigmaplot 14 software | Systat Software Inc | For dose-response curve generation | |
| Sulforhodamine B powder | Sigma | S1402-5G | 5 g |
| SYBR green master mix | Smobio | TQ12001805401-3 | Binding fluorescent dye for dsDNA |
| T4 DNA ligase | TaKaRa | 2011A | 25,000 U |
| TaqMan master mix | Applied Biosystems | 4324018 | 200 reactions, no AmpErase UNG |
| TaqMan microRNA assay (hsa-miR-107) | Applied Biosystems | 4427975 | Assay ID: 000443 (50RT, 150 PCR rxns) |
| TaqMan microRNA assay (hsa-miR-301) | Applied Biosystems | 4427975 | Assay ID: 000528 (50RT, 150 PCR rxns) |
| TaqMan miR RT kit | Applied Biosystems | 4366597 | 1,000 reactions |
| Thermo CO2 incubator (BB15) | ThermoFisher Scientific | 37 °C and 5% CO2 incubation | |
| Trichloroacetic acid | Sigma | 91228-100G | 100 g |
| Trizma base | Sigma | T4661-100G | 100 g |
| Ultrapure water | Invitrogen | 10977-015 | 500 mL |
| Veriti 96 well thermal cycler | Applied Biosystems | For amplification of DNA (or cDNA) | |
| XhoI | New England Biolabs | R0146 | 20,000 units/mL |
参考文献
- He, L., Hannon, G. J. MicroRNAs: small RNAs with a big role in gene regulation. Nature Reviews Genetics. 5 (7), 522-531 (2004).
- Park, J. K., Doseff, A. I., Schmittgen, T. D. MicroRNAs Targeting Caspase-3 and -7 in PANC-1 Cells. International Journal of Molecular Sciences. 19 (4), (2018).
- Park, J. K., et al. MicroRNAs-103/107 coordinately regulate macropinocytosis and autophagy. Journal of Cell Biology. 215 (5), 667-685 (2016).
- Henry, J. C., et al. miR-199a-3p targets CD44 and reduces proliferation of CD44 positive hepatocellular carcinoma cell lines. Biochemical and Biophysical Research Communications. 403 (1), 120-125 (2010).
- Hoefert, J. E., Bjerke, G. A., Wang, D., Yi, R. The microRNA-200 family coordinately regulates cell adhesion and proliferation in hair morphogenesis. Journal of Cell Biology. 217 (6), 2185-2204 (2018).
- Anfossi, S., Fu, X., Nagvekar, R., Calin, G. A. MicroRNAs, Regulatory Messengers Inside and Outside Cancer Cells. Advances in Experimental Medicine and Biology. 1056, 87-108 (2018).
- Khoshinani, H. M., et al. Involvement of miR-155/FOXO3a and miR-222/PTEN in acquired radioresistance of colorectal cancer cell line. Japanese Journal of Radiology. 35 (11), 664-672 (2017).
- Gao, Y., et al. MicroRNA-155 increases colon cancer chemoresistance to cisplatin by targeting forkhead box O3. Oncology Letters. 15 (4), 4781-4788 (2018).
- Catanzaro, G., et al. Loss of miR-107, miR-181c and miR-29a-3p Promote Activation of Notch2 Signaling in Pediatric High-Grade Gliomas (pHGGs). International Journal of Molecular Sciences. 18 (12), (2017).
- Akbari Moqadam, F., Pieters, R., den Boer, M. L. The hunting of targets: challenge in miRNA research. Leukemia. 27 (1), 16-23 (2013).
- Brown, R. A. M., et al. Total RNA extraction from tissues for microRNA and target gene expression analysis: not all kits are created equal. BMC Biotechnology. 18 (1), (2018).
- Kim, Y. K., Yeo, J., Kim, B., Ha, M., Kim, V. N. Short structured RNAs with low GC content are selectively lost during extraction from a small number of cells. Molecular Cell. 46 (6), 893-895 (2012).
- Schmittgen, T. D., Livak, K. J. Analyzing real-time PCR data by the comparative C(T) method. Nature Protocols. 3 (6), 1101-1108 (2008).
- Livak, K. J., Schmittgen, T. D. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) Method. Methods. 25 (4), 402-408 (2001).
- Park, J. K., Seo, J. S., Lee, S. K., Chan, K. K., Kuh, H. J. Combinatorial Antitumor Activity of Oxaliplatin with Epigenetic Modifying Agents, 5-Aza-CdR and FK228, in Human Gastric Cancer Cells. Biomolecules & Therapeutics. 26 (6), 591-598 (2018).
- Xia, X., et al. Downregulation of miR-301a-3p sensitizes pancreatic cancer cells to gemcitabine treatment via PTEN. American Journal of Translational Research. 9 (4), 1886-1895 (2017).
- Lee, K. H., et al. Epigenetic silencing of MicroRNA miR-107 regulates cyclin-dependent kinase 6 expression in pancreatic cancer. Pancreatology. 9 (3), 293-301 (2009).
- van Tonder, A., Joubert, A. M., Cromarty, A. D. Limitations of the 3-(4,5-dimethylthiazol-2-yl)-2,5-diphenyl-2H-tetrazolium bromide (MTT) assay when compared to three commonly used cell enumeration assays. BMC Research Notes. 8, 47(2015).
- Wang, P., Henning, S. M., Heber, D. Limitations of MTT and MTS-based assays for measurement of antiproliferative activity of green tea polyphenols. PloS One. 5 (4), e10202(2010).
- Wu, L., Belasco, J. G. Let me count the ways: mechanisms of gene regulation by miRNAs and siRNAs. Molecular Cell. 29 (1), 1-7 (2008).
- Jin, Y., Chen, Z., Liu, X., Zhou, X. Evaluating the microRNA targeting sites by luciferase reporter gene assay. Methods in Molecular Biology. , 117-127 (2013).
- Ma, Z., et al. Gamma-synuclein binds to AKT and promotes cancer cell survival and proliferation. Tumour Biology. 37 (11), 14999-15005 (2016).
- Pan, Z. Z., Bruening, W., Giasson, B. I., Lee, V. M., Godwin, A. K. Gamma-synuclein promotes cancer cell survival and inhibits stress- and chemotherapy drug-induced apoptosis by modulating MAPK pathways. Journal of Biological Chemistry. 277 (38), 35050-35060 (2002).
- Martinez-Sanchez, A., Murphy, C. L. MicroRNA Target Identification-Experimental Approaches. Biology (Basel). 2 (1), 189-205 (2013).
- Lee, E. J., et al. Expression profiling identifies microRNA signature in pancreatic cancer. International Journal of Cancer. 120 (5), 1046-1054 (2007).
- Nuovo, G. J., et al. A methodology for the combined in situ analyses of the precursor and mature forms of microRNAs and correlation with their putative targets. Nature Protocols. 4 (1), 107-115 (2009).
- Schmittgen, T. D., et al. Real-time PCR quantification of precursor and mature microRNA. Methods. 44 (1), 31-38 (2008).
- Diederichs, S., Haber, D. A. Dual role for argonautes in microRNA processing and posttranscriptional regulation of microRNA expression. Cell. 131 (6), 1097-1108 (2007).
- Orellana, E. A., Kasinski, A. L. Sulforhodamine B (SRB) Assay in Cell Culture to Investigate Cell Proliferation. Bio Protocol. 6 (21), (2016).
- Lawrie, C. H. MicroRNAs in hematological malignancies. Blood Reviews. 27 (3), 143-154 (2013).
- Quah, B. J., Warren, H. S., Parish, C. R. Monitoring lymphocyte proliferation in vitro and in vivo with the intracellular fluorescent dye carboxyfluorescein diacetate succinimidyl ester. Nature Protocols. 2 (9), 2049-2056 (2007).
- Xing, Z., Li, D., Yang, L., Xi, Y., Su, X. MicroRNAs and anticancer drugs. Acta Biochimica et Biophysica Sinica. 46 (3), 233-239 (2014).
- Moeng, S., et al. MicroRNA-107 Targets IKBKG and Sensitizes A549 Cells to Parthenolide. Anticancer Research. 38 (11), 6309-6316 (2018).
- Chou, T. C. Drug combination studies and their synergy quantification using the Chou-Talalay method. Cancer Research. 70 (2), 440-446 (2010).
- Flamand, M. N., Gan, H. H., Mayya, V. K., Gunsalus, K. C., Duchaine, T. F. A non-canonical site reveals the cooperative mechanisms of microRNA-mediated silencing. Nucleic Acids Research. 45 (12), 7212-7225 (2017).
Access restricted. Please log in or start a trial to view this content.
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。