需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
生物信息学管道,用于准确、高效地分析植物中的微RNA转录
* 这些作者具有相同的贡献
摘要
生物信息学管道,即miRDeep-P2(简称miRDP2),具有更新的植物miRNA标准和经过全面检查的算法,可以准确有效地分析植物中的微RNA转录,特别是对于具有复杂和大型基因组的物种。
摘要
微RNA (miRNA) 是 20- 24 核苷酸 (nt) 内源性小RNA (sRNA), 广泛存在于植物和动物中,在转录后水平上调节基因表达起着有效作用。过去十年中,用新一代测序 (NGS) 方法测序 sRNA 库被广泛用于识别和分析 miRNA 转录体,从而迅速增加了 miRNA 发现。然而,由于测序sRNA库的深度增加以及植物基因组的大小和复杂性,植物miRNA注释中出现了两个主要挑战。首先,许多其他类型的sRNA,特别是来自sRNA库的短干扰RNA(siRNA),被许多计算工具错误地批号为miRNA。其次,在具有庞大和复杂基因组的植物物种中分析miRNA转录体,这成为一个极其耗时的过程。为了克服这些挑战,我们最近通过采用新的过滤策略、彻底检查评分算法和合并新更新的植物 miRNA,将 miRDeep-P(miRNA 转录组分析的常用工具)升级到 miRDeep-P2(简称 miRDP2)注释条件。我们在基因组复杂性增加的五种代表性植物(包括阿拉伯拟南芥、水稻、番茄、玉米和小麦)中针对测序sRNA种群测试了miRDP2。结果表明,miRDP2处理这些任务的效率非常高。此外,miRDP2 在灵敏度和准确性方面优于其他预测工具。综合起来,我们的研究结果证明miRDP2是分析植物miRNA转录酶的快速和准确的工具,因此是帮助社区更好地在植物中对miRNA进行分文的有用工具。
引言
过去二十年来生物学中最令人兴奋的发现之一是sRNA物种在调节基因组1的不同功能中的增殖作用。特别是,miRNA在真核生物中构成20-24ntsRNA的重要类别,主要在转录后水平上作为重要的基因调控器,在整个生命周期发育阶段以及刺激和应激反应2、3中发挥作用。在植物中,miRNA产生于称为pri-miRNA的主要转录本,通常由RNA聚合酶II转录为单个转录单元4、5。由进化保存的细胞机械(动物中的Drosha RNase III,植物中的DICER样)处理,pri-miRNA被切除到直接的miRNA前体,前miRNA,其中包含形成分子内茎环结构的序列6,7。然后,将预miRNA加工成双链中间体,即miRNA双工,由功能链、成熟miRNA和功能性较低的伙伴miRNA®2、8组成。在加载到RNA诱导沉默复合物(RISC)后,成熟的miRNA可以基于序列互补性识别其mRNA靶点,导致负调节功能2,8。miRNA要么破坏目标成绩单,要么阻止目标翻译,但前者在植物8,9中占主导地位。
自从偶然发现线虫10、11号线虫中第一个miRNA以来,许多研究都致力于miRNA鉴定及其功能分析,特别是在NGS方法的提供之后。NGS方法的广泛应用极大地促进了计算工具的利用,这些工具旨在捕捉miRNA的独特特性,如前体的干环结构及其在成熟miRNA和miRNA®上序列读取的优先积累。因此,研究人员在识别不同物种的miRNA方面取得了显著成功。基于先前描述的概率模型12,我们开发了miRDeep-P13,这是从NGS数据中发现植物miRNA的第一个计算工具。miRDeep-P是专门旨在征服解码植物miRNA的挑战,具有更多的可变前体长度和大型的副体家族13,14,15。发布后,这个程序已被下载数千次,并用于在40多个植物物种16中对miRNA转录子进行批过。在基于NGS的工具(如miRDeep-P)的推动下,公共miRNA存储库miRBase17中注册的miRNA数量急剧增加,目前托管的miRNA项目超过38,000个(版本22.1),而2008年仅为500个miRNA项目(版本2.0)。
然而,植物miRNA注释带来了两个新的挑战。首先,高误报率严重影响了植物miRNA注释16、19的质量,原因如下:1)由于缺乏严格的miRNA注释标准,NGS sRNA库中的内源性短干扰RNA(siRNA)被错误地注释为miRNA;2) 对于没有先验miRNA信息的物种,根据NGS数据预测的假阳性很难消除。以miRBase为例,Taylor等人20号在公共存储库21(第21版)中发现了三分之一的植物miRNA条目,缺乏令人信服的支持证据,甚至四分之三的植物miRNA家族也值得怀疑。其次,它成为一个极其耗时的过程,预测植物miRNA与大和复杂的基因组16。为了克服这些挑战,我们更新了 miRDeep-P,增加了新的过滤策略,彻底修改了评分算法,集成了植物 miRNA 注释的新标准,并发布了新版本 miRDP2。此外,我们使用 NGS sRNA 数据集测试了 miRDP2,其基因组大小逐渐增大:阿拉伯拉多普西、大米、番茄、玉米和小麦。与其他五种广泛使用的工具及其旧版本相比,miRDP2 分析了这些 sRNA 数据,并更快地分析 miRNA 转录,提高了准确性和灵敏度。
miRDP2 封装的内容
miRDP2 包由六个文档化的 Perl 脚本组成,这些脚本应由准备好的 bash 脚本按顺序运行。在六个脚本中,三个(convert_bowtie_to_blastpl、filter_alignments.pl和excise_candidate.pl)是从miRDeep-P继承的。 其他脚本是从原始版本修改的。六个脚本的函数如下所述:
preprocess_reads.pl筛选输入读取,包括太长或太短的读取(<19 nt 或 >25 nt),读取与 Rfam ncRNA 序列相关,以及读取的 RPM(读取百万)小于 5。然后,该脚本检索与已知 miRNA 成熟序列相关的读取。输入文件是 FASTA/FASTQ 格式的原始读取和 bowtie2 输出的读取映射到 miRNA 和 ncRNA 序列。
计算 RPM 的公式如下:
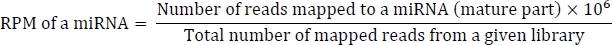
convert_bowtie_to_blast.pl将蝴蝶结格式更改为 BLAST 解析格式。BLAST 解析格式是从标准 NCBI BLAST 输出格式派生的自定义表格分隔格式。
filter_alignments.pl过滤深度测序读取到基因组的对齐方式。它过滤部分对齐以及多对线读取(用户指定的频率截止)。基本输入是 BLAST 解析格式的文件。
excise_candidate.pl使用对齐的读取作为指南,从参考序列中剪切出潜在的前体序列。基本输入是 BLAST 解析格式的文件和 FASTA 文件。输出是 FASTA 格式的所有潜在前体序列。
mod-miRDP.pl需要两个输入文件,签名文件和结构文件,通过改变评分系统与植物特定参数从核心miRDeep-P算法修改。输入文件是点括号前体结构文件和读取分发签名文件。
mod-rm_redundant_meet_plant.pl需要三个输入文件:mod-miRDP.pl生成的chromosome_length、前体和original_prediction。它生成两个输出文件,非冗余预测文件和预测文件筛选新更新的工厂 miRNA 标准。有关输出文件格式的详细信息,请见第 1.4 节。
研究方案
1. 安装和测试
- 下载所需的依赖项:鲍蒂222和RNAfold23。建议使用已编译的包。
- 下载Bowtie2,一个读取映射工具,从它的家庭网站(http://bowtie-bio.sourceforge.net/bowtie2/index.shtml)。
- 下载RNAfold,维也纳包的工具,用于预测RNA二次结构,从http://www.tbi.univie.ac.at/~ivo/RNA/。
- 在安装 miRDP2 之前,请确保正确安装这两个依赖项,并自定义 bash 环境文件(例如 .bashrc)以为这两个依赖项设置正确的 PATH。
注:其他映射工具,如鲍蒂24也适合miRDP2;鲍蒂或鲍蒂2可在1.1.3版后使用。
- 要下载 miRDP2 包,请转到https://sourceforge.net/projects/mirdp2/files/latest_version/并获取 tarball 文件。
- 在安装 miRDP2 之前,请确保 Perl 位于 PATH 中。要安装 miRDP2,请将下载的 tarball 文件的所有内容提取到一个文件夹中(命令行,如 1.4.2 中),然后将文件夹路径设置为 PATH。
注: 建议使用至少 8 GB RAM 和 100 GB 存储的计算机或计算节点运行 miRDP2。 - 测试 MiRDP2 管道。
- 要测试 miRDP2 是否已正确安装,请使用测试数据和https://sourceforge.net/projects/mirdp2/files/TestData/中找到的预期输出。测试数据包含一个格式化的GSM测序文件和一个阿拉伯拉多普西斯塔利亚纳基因组文件。
- 将所有下载的文件移动到当前工作目录:
mv miRDP2-v_.tar.gz TestData.tar.gz ncRNA_rfam.tar.gz
cd - 提取压缩的焦油球文件:
tar _xvzf miRDP2-v_.tar.gz
tar @xvzf 测试数据.tar.gz
塔尔·xvzf ncRNA_rfam.tar.gz - 建立阿拉伯基因组参考索引:
bowtie2-build -f ./测试数据/TAIR10_genome.fa ./测试数据/TAIR10_genome - 构建 ncRNA 参考索引:
bowtie2-build -f ./ncRNA_rfam.fa ./1.1.3/脚本/索引/rfam_index - 运行 miRDP2 管道:
bash ./1.1.3/miRDP2-v1.1.3_pipeline.bash @g /testData/TAIR10_genome.fa -i ./ 测试数据/TAIR 10_genome +f /测试数据/GSM2094927.fa _o .
注: 使用的 Linux 命令采用粗体和斜体字体,命令行选项以斜体显示。*指示 miRDP2 的版本(当前版本为 1.1.3)。bowtie2-build 命令大约需要 10 分钟,miRDP2 管道应在几分钟内完成
- 检查测试输出。
- 请注意,名为"GSM2094927-15-0-10"的文件夹在中自动生成,其中包含所有中间文件和结果。
- 检查标签分隔输出文件 GSM2094927-15-0-10_filter_P_prediction,预测 miRNA 的最终输出,包含指示染色体 ID、链方向、代表性读取 ID、前体 ID、成熟 miRNA 位置、前体)的列位置、成熟序列和前体序列。请注意从此文件派生的附加床文件,以便进一步分析。
- 检查文件"progress_log",其中提供有关已完成的步骤的信息,以及包含程序输出和警告的文件"script_log"和"script_err"。
注意:目前,我们已经在两个Linux平台上测试了miRDP2,包括集群服务器上的CentOS版本6.5和PC Windows系统上的Cygwin 2.6.0,miRDP2应该在支持Perl的类似系统上工作。
2. 识别新颖的 miRNA
- 在运行管道之前,请确保将输入读取预处理为正确的格式。
注: miRDP2 的新版本 1.1.3 可以接受原始 FASTQ 格式文件作为输入,尽管格式化读取的过程与以前的版本一样执行。- 首先,从深度排序读取的 5' 和 3' 端卸下适配器(如果存在)。
- 其次,将深度排序读取解析为 FASTA 格式。
- 第三,删除冗余,以便使用单个且唯一的 FASTA 条目表示具有相同序列的读取。
- 最后,确保所有 FASTA 标识符都是唯一的。每个序列标识符必须以"_x"和整数结尾,指示在深度排序数据集中检索到的精确序列的副本数。确保唯一 FASTA 标识符的一种方法是在 ID 中包含一个正在运行的数字。有关参考,请参阅测试数据(https://sourceforge.net/projects/mirdp2/files/TestData/) 中的文件 GSM2094927.fa。
- 有关正确格式化的读取示例,请参阅以下内容:
>read0_x29909
TTGGGAAGGGGCTCTA
>read1_x36974
TTCCACCTCTCTTTG
>read2_x32635
TTCCACCTCTCTTTTT
- 生成参考索引。
- 对于基因组参考,为了节省时间,从iGenomes网站(https://support.illumina.com/sequencing/sequencing_software/igenome.html)下载Bowtie2索引文件,如果感兴趣的物种的基因组序列已经编制索引。否则,用户索引引用序列并保留索引文件一段时间,直到项目完成,因为基因组序列可能需要重新编制索引。有关如何索引基因组参考的详细信息包含在 bowtie2 手册 (http://bowtie-bio.sourceforge.net/bowtie2/manual.shtml) 中。
- 还需要另一个非miRNA ncRNA索引来过滤其他非编码RNA片段中的噪声序列。该文件是来自Rfam的主要ncRNA序列的集合,包括rRNA、tRNA、snRNA和snoRNA。要构建此索引,请参阅第 1.4 部分,因为索引应正确放置和命名,即 /脚本/索引/rfam_index。
- 运行 miRDP2。
- 要使用 miRDP2 从深度测序数据中检测新的 miRNA,请使用包中的 bash 脚本启动分析管道(可以在步骤 1.4 中找到示例):
//////////_pipeline-o >-f
其中 * 指示管道 bash 脚本的版本。有三个参数可以修改:1) 读取可以映射到的不同位置的数量,2) 运行 bowtie2 的不匹配数,3) RPM 阈值(每百万读取)。分别使用 +L、-M 和 +R 选项修改这些选项。第 3.1 节对此作了详细说明。
- 要使用 miRDP2 从深度测序数据中检测新的 miRNA,请使用包中的 bash 脚本启动分析管道(可以在步骤 1.4 中找到示例):
- 检查 miRDP2 输出。
- 请注意,输出文件夹将在 下自动生成,并命名为"-15-0-10";最后 3 个数字分别指示参数 1、2 和 3 的值(本例中为默认值)。文件 _filter_P_prediction包含满足新更新的植物 miRNA 注释标准的最终预测 miRNA 的信息。有关输出文件格式的详细信息,请放在第 1.4 部分中。
3. 使用 miRDP2 进行修改和警告
- 可修改的参数
- 使用"-L"选项设置读取可映射到多少位置的限制(参数 1)。读取映射到太多站点可能与重复序列相关联,并且不太可能与 miRNA 相关联。默认设置为 15。对于特定物种,如果有具有许多成员的miRNA家族,第一个参数可以手动增加以适应基因组景观。
- 使用"-M"选项设置弓形(参数 2)允许的不匹配。默认设置为 0。
- 使用"-R"选项设置可能与成熟 miRNA 相对应的读取阈值(参数 3)。为了减少时间消耗和误报,请按 RPM 筛选读取。只有超过一定 RPM 阈值的读取才能表示 miRNA 的成熟序列,而不是背景噪声,并将保留以进行进一步分析。默认设置为 10 RPM。
- 请注意,更改这些参数可能会影响性能和时间消耗。通常,增加参数 1 和参数 2 以及减少参数 3 将生成不太严格的结果和更长的运行时间,反之亦然。
- 冗余和 miRNA*
- 请注意,miRDP2 的输出 miRNA 可能与已知的 miRNA 不同。我们发现,这主要是由于两个原因之一:成熟miRNA的异质性或miRNA和miRNA®的相对丰度。我们发现,这不会影响前体的最佳长度选择和已知miRNA基因的轮廓。
结果
本文描述的miRNA注释管道miRDP2适用于5个基因组长度逐渐增加的植物物种的10个公共sRNA-seq库,包括阿拉伯拟南芥、Oryza sativa(大米)、索兰姆碱化(番茄)、Zea Mays(玉米)和三联体(小麦)(图1A)。总体而言,对于每个物种,来自不同组织的2个代表性sRNA库(折叠成唯一的读取,协议部分的细节)及其索引基因组序列作为两个?...
讨论
随着NGS的出现,从越来越多的sRNA测序数据中发现了大量的miRNA位点,这些物种的数量不断增加,不同的物种29,30。在集中式社区数据库miRBase21中,沉积的miRNA项目在过去十年中增加了近100倍。然而,与动物中的miRNA相比,植物miRNA具有许多独特的特征,使得识别/注释更加复杂13,14。
披露声明
作者没有什么可透露的。
致谢
这项工作得到了北京农林科学院(KJCX201917、KJCX20180425和KJCX20180204)对XY和中国国家自然科学基金(31621001)的LL支持。
材料
| Name | Company | Catalog Number | Comments |
| Computer/computing node | N/A | N/A | Perl is required; at least 8 GB RAM and 100 GB storage are recommended |
参考文献
- Ghildiyal, M., Zamore, P. D. Small silencing RNAs: an expanding universe. Nature Reviews Genetics. 10 (2), 94-108 (2009).
- Bartel, D. P. MicroRNAs: target recognition and regulatory functions. Cell. 136 (2), 215-233 (2009).
- Moran, Y., Agron, M., Praher, D., Technau, U. The evolutionary origin of plant and animal microRNAs. Nature Ecology Evolution. 1 (3), 27 (2017).
- Xie, Z., et al. Expression of Arabidopsis MIRNA genes. Plant Physiology. 138 (4), 2145-2154 (2005).
- Zhao, X., Zhang, H., Li, L. Identification and analysis of the proximal promoters of microRNA genes in Arabidopsis. Genomics. 101 (3), 187-194 (2013).
- Bologna, N. G., Mateos, J. L., Bresso, E. G., Palatnik, J. F. A loop-to-base processing mechanism underlies the biogenesis of plant microRNAs miR319 and miR159. EMBO JOURNAL. 28 (23), 3646-3656 (2009).
- Rogers, K., Chen, X. Biogenesis, turnover, and mode of action of plant microRNAs. Plant Cell. 25 (7), 2383-2399 (2013).
- Voinnet, O. Origin, biogenesis, and activity of plant microRNAs. Cell. 136 (4), 669-687 (2009).
- Iwakawa, H. O., Tomari, Y. The Functions of MicroRNAs: mRNA Decay and Translational Repression. Trends in Cell Biology. 25 (11), 651-665 (2015).
- Lee, R. C., Feinbaum, R. L., Ambros, V. The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14. Cell. 75 (5), 843-854 (1993).
- Wightman, B., Ha, I., Ruvkun, G. Posttranscriptional regulation of the heterochronic gene lin-14 by lin-4 mediates temporal pattern formation in C. elegans. Cell. 75 (5), 855-862 (1993).
- Friedlander, M. R., et al. Discovering microRNAs from deep sequencing data using miRDeep. Nature Biotechnology. 26 (4), 407-415 (2008).
- Yang, X., Li, L. miRDeep-P: a computational tool for analyzing the microRNA transcriptome in plants. Bioinformatics. 27 (18), 2614-2615 (2011).
- Meyers, B. C., et al. Criteria for annotation of plant MicroRNAs. Plant Cell. 20 (12), 3186-3190 (2008).
- Yang, X., Zhang, H., Li, L. Global analysis of gene-level microRNA expression in Arabidopsis using deep sequencing data. Genomics. 98 (1), 40-46 (2011).
- Kuang, Z., Wang, Y., Li, L., Yang, X. miRDeep-P2: accurate and fast analysis of the microRNA transcriptome in plants. Bioinformatics. , (2018).
- Kozomara, A., Birgaoanu, M., Griffiths-Jones, S. miRBase: from microRNA sequences to function. Nucleic Acids Research. 47 (1), 155-162 (2019).
- Griffiths-Jones, S., Saini, H. K., van Dongen, S., Enright, A. J. miRBase: tools for microRNA genomics. Nucleic Acids Research. 36, 154-158 (2008).
- Axtell, M. J., Meyers, B. C. Revisiting Criteria for Plant MicroRNA Annotation in the Era of Big Data. Plant Cell. 30 (2), 272-284 (2018).
- Taylor, R. S., Tarver, J. E., Hiscock, S. J., Donoghue, P. C. Evolutionary history of plant microRNAs. Trends in Plant Science. 19 (3), 175-182 (2014).
- Kozomara, A., Griffiths-Jones, S. miRBase: annotating high confidence microRNAs using deep sequencing data. Nucleic Acids Research. 42, 68-73 (2014).
- Langmead, B., Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nature Methods. 9 (4), 357-359 (2012).
- Lorenz, R., et al. ViennaRNA Package 2.0. Algorithms for Molecular Biology. 6, 26 (2011).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biology. 10 (3), 25 (2009).
- An, J., Lai, J., Sajjanhar, A., Lehman, M. L., Nelson, C. C. miRPlant: an integrated tool for identification of plant miRNA from RNA sequencing data. BMC Bioinformatics. 15, 275 (2014).
- Lei, J., Sun, Y. miR-PREFeR: an accurate, fast and easy-to-use plant miRNA prediction tool using small RNA-Seq data. Bioinformatics. 30 (19), 2837-2839 (2014).
- Evers, M., Huttner, M., Dueck, A., Meister, G., Engelmann, J. C. miRA: adaptable novel miRNA identification in plants using small RNA sequencing data. BMC Bioinformatics. 16, 370 (2015).
- Mathelier, A., Carbone, A. MIReNA: finding microRNAs with high accuracy and no learning at genome scale and from deep sequencing data. Bioinformatics. 26 (18), 2226-2234 (2010).
- Zhu, Q. H., et al. A diverse set of microRNAs and microRNA-like small RNAs in developing rice grains. Genome Research. 18 (9), 1456-1465 (2008).
- Fahlgren, N., et al. MicroRNA gene evolution in Arabidopsis lyrata and Arabidopsis thaliana. Plant Cell. 22 (4), 1074-1089 (2010).
- Fromm, B., et al. A Uniform System for the Annotation of Vertebrate microRNA Genes and the Evolution of the Human microRNAome. Annual Review of Genetics. 49, 213-242 (2015).
- Blevins, T., et al. Identification of Pol IV and RDR2-dependent precursors of 24 nt siRNAs guiding de novo DNA methylation in Arabidopsis. Elife. 4, 09591 (2015).
- Zhai, J., et al. A One Precursor One siRNA Model for Pol IV-Dependent siRNA Biogenesis. Cell. 163 (2), 445-455 (2015).
- Werner, S., Wollmann, H., Schneeberger, K., Weigel, D. Structure determinants for accurate processing of miR172a in Arabidopsis thaliana. Current Biology. 20 (1), 42-48 (2010).
- Mateos, J. L., Bologna, N. G., Chorostecki, U., Palatnik, J. F. Identification of microRNA processing determinants by random mutagenesis of Arabidopsis MIR172a precursor. Current Biology. 20 (1), 49-54 (2010).
- Vitsios, D. M., et al. Mirnovo: genome-free prediction of microRNAs from small RNA sequencing data and single-cells using decision forests. Nucleic Acids Research. 45 (21), 177 (2017).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。