Method Article
调整降解以实现特定和高效的蛋白质消耗
摘要
在这里,我们提出一个协议,使用β-est AID系统,有效和具体地耗尽酵母糖精中感兴趣的蛋白质。
摘要
植物辅助素结合受体 TIR1 可识别含有特定辅助诱导脱粒 (AID) 在辅助素存在时的蛋白质,针对它们进行降解。该系统在许多非植物真核生物中被利用,因此,一种带有AID图案的目标蛋白在添加辅助物后降解。TIR1表达式的级别至关重要;即使缺乏辅助蛋白,过度的表达也会导致AID标记的蛋白质降解,而低表达会导致缓慢耗竭。建立了β-雌二醇诱导AID系统,在β-雌二醇诱导启动子的控制下表达TIR1。在添加辅助剂之前,通过改变使用β-雌二醇的孵育时间,TIR1的含量是可调的。该协议描述了如何使用AID系统快速耗尽目标蛋白质。适当的β-雌二醇孵育时间取决于靶蛋白的丰度。因此,有效耗竭取决于最佳时序,这也最大限度地减少了与辅助剂无关的损耗。
引言
条件突变,如温度敏感突变体,是研究基本蛋白质的有力工具,允许在允许条件下细胞生长,但在不容许的条件下导致功能丧失。然而,细胞代谢可能严重干扰生长条件的变化,需要诱导缺陷,也可能产生偏离目标的影响。已经开发出几种方法,其中感兴趣的蛋白质有条件地隔离1,或者通过添加小分子控制其表达2,3。该协议使用辅助蛋白和辅助诱导脱粒 (AID) 系统来有效耗尽目标蛋白质。
AID系统起源于植物,其中一种辅助蛋白(在本议定书中使用indole-3-醋酸(IAA),刺激Aux/IAA蛋白与TIR1的相互作用,TIR1是SCF U3泛性联苯复合物4的成员。SCF复杂相互作用导致Aux/IAA家族蛋白多聚体化,导致其降解为蛋白酶体5,6。
该系统以前被改造用于酵母糖精7,8通过表达来自酵母细胞的 Oriza sativa (osTIR) 的 TIR1 蛋白质, 在那里它能够与内源酵母相互作用SCF 复合体。感兴趣的蛋白质被标记从Aux/IAA蛋白IAA17的图案,以瞄准它的降解。IAA17 的功能截断后来被开发,如 AID®8、9、10,包含来自阿拉伯拟南芥IAA17的 43 个氨基酸辅助素敏感主题,以及一个表位标记,以启用检测。
该系统最初适应在萌芽酵母7,8表达的osTIR1蛋白质从酵母GAL启动子。表达需要转向生长介质,以角质素作为唯一的碳源,不幸的是,这导致了二次转移,细胞代谢发生了广泛的变化11。另一方面,据报道,TIR1的构成表达在缺乏辅助蛋白/IAA12的情况下,如果表达水平高,则可能导致目标蛋白降解,而低TIR1表达会导致低效耗竭。开发了一种名为β-est AID的改进AID系统,其中osTIR受一个可调节的诱导启动子控制,该启动子可调谐到靶蛋白,对细胞代谢的影响最小。为此,构建了一个人工转录因子(ATF),其中VP16病毒转录活化剂与雌激素受体和四个Zn手指DNA结合域(DBD)融合。当β-雌二醇(雌激素)存在时,ATF可以进入细胞核,并通过与启动子(Z4EVpr)13、12结合来诱导osTIR转录。
在加入β-雌二醇12后,osTIR表达通常可检测到约20分钟。然而,osTIR表达的最佳持续时间,以实现有效消耗带辅助蛋白的标记蛋白,同时在添加辅助蛋白之前避免耗竭,需要为每个目标蛋白进行经验测定。这种预孵育的大致时间可以从糖霉菌基因组数据库中的丰度值(SGD https://www.yeastgenome.org/)中估计。如图1所示,丰富的蛋白质Dcp1(2880至4189分子/细胞)需要40分钟的β-雌二醇预孵育,没有观察到与辅助素无关的消耗。较不丰富的蛋白质Prp2(172至211分子/细胞)在预孵育后仅20分钟就严重耗尽。建议在此初始估计时间之前或之后测试两个额外的预孵育时间(20 分钟是建议的最短时间)。最佳预孵育时间是靶蛋白在添加辅助蛋白之前没有耗尽的时间,一旦添加辅助蛋白,消耗是可以接受的,或者蛋白质水平接近可能达到的最低水平。因此,从图1b,对于预孵化30分钟的Prp22,在添加辅助后10分钟的水平没有下降多少。与40分钟的预孵育和15分钟的IAA相比,在几乎没有额外消耗的情况下,用超过10分钟的辅助剂孵育或超过30分钟的预孵育没有好处,特别是因为有证据表明非辅助性依赖在40分钟内耗尽。对于预孵育40分钟的Dcp1(在添加辅助剂之前蛋白质水平约为100%的最后一点),使用辅助蛋白消耗15至20分钟是可以接受的。建议尽量缩短耗竭时间,以减少对细胞代谢的二次影响14。
本文演示如何使用β-est AID系统,通过优化osTIR表达β-雌醇孵育的时间,在添加辅助蛋白之前,在IAA添加时实现快速目标蛋白质消耗而不消耗。
研究方案
注:有关图形摘要,请参阅图2。
1. 应变准备
-
使用ura3-应变,引入β-est AID系统(即编码β-雌二醇反应转录因子(ATF)和osTIR的基因),并标记目标蛋白的AID®(参见图3和表1,了解该过程的摘要)。
- 将15 pZTRK(G418 电阻标记)或 pZTRL(LEU2 标记)质粒(可从酵母遗传资源中心获得)转换为ura3-酵母菌株,或使用质粒作为模板生产用于基因组的 PCR 产品集成。
- PCR 使用来自 pZTRK 或 pZTRL 的质粒中的高保真聚合酶放大 ATF(在质粒图图上标记的 Z4EVATF)和 osTIR。使用引物与50至60碱基3'扩展与同源基因组区域,通过同源重组16直接集成。有关两个组分的基因组集成,请参阅表 1了解引体和条件。
注:菌株pZ4EV-NTR1的成分已经集成到基因组中(可从日本酵母遗传资源中心获得)。 - 确保使用 Longtine 过程17标记目标蛋白 ID+(参见图 3b和表 1)。
- 在没有β-雌二醇和IAA存在的情况下对菌株执行生长分析,以确定AID®标签是否影响生长,并预测在步骤1.5中使用的生长速度。
2. 消耗的一般程序
- 计算收集所有样本所需的区域性;例如,OD600的 0.8 的培养值为 10 mL,足以用于单个样本的蛋白质、RNA 和 DNA 提取,因此对于 6 个样本,至少需要 60 mL 培养。
- 从隔夜培养,在 OD600 0.1 到 0.2 处建立足够的新培养,并在 30°C 下保持增长。建议使用丰富的培养基,如YPDA,但可以使用其他生长条件:
酵母提取物 12 % 蛋白胨 20克 葡萄糖 20克 亚胺硫酸盐 40毫克 H2O 到 1 升
注:高压灭菌或过滤器灭菌;过滤器灭菌是首选的肽/糖复合物,通过高压灭菌沉淀物在样品收集中使用的甲醇。 - 准备接收样品。
- 将甲醇的预期样品量的30%至50%放入管中。例如,如果要采集 10 mL 样品,将 5 mL 甲醇放入 15 mL 猎鹰管中,并紧闭管。关闭后,将管子贴上标签,放在干冰上或-80°C冷却。
警告:将甲醇在烟机罩中分配。 - 标记 1.5 mL 管,用于长期储存样品,并放入冰中冷却。
- 在冰上冷却足够的 H2O(每个样品至少 1 mL)。
- 将甲醇的预期样品量的30%至50%放入管中。例如,如果要采集 10 mL 样品,将 5 mL 甲醇放入 15 mL 猎鹰管中,并紧闭管。关闭后,将管子贴上标签,放在干冰上或-80°C冷却。
- 预测文化的发展。收集样本的目标OD约为0.7至0.8,但孵育前步骤(用β-雌二醇诱导osTIR的孵育)需要更早开始,以便培养在样品时达到大约正确的OD收集。
注:建议在实验中使用的条件中执行增长曲线,以便可以估计此起始 OD。 - 一旦达到预孵育开始的目标OD,将样品(通常为10 mL)放入含有冷甲醇的预制备管中。短暂反转以混合并放回干冰中。
注:如果方便的话,样品可以在大约5分钟后移动到水冰中。 - 立即加入β-雌二醇,1μL/mL培养(最终浓度为10μM);在移液器中预测了β-雌二醇,以便减少收集样品和添加β-雌二醇之间的时间。通过剧烈旋转快速混合。
- 继续像以前那样种植培养(步骤2.2),用β-雌二醇孵育(这是"预孵化"步骤),以达到最佳时间(用于确定最佳预孵育时间,见图1)。
- 准备添加 IAA(辅助剂)。占用步骤 2.10 所需的 IAA 量(即每 mL 培养值 0.5 μL 的 IAA)。这使得步骤 2.20 更快。
- 作为步骤 2.5 收集示例。
- 立即将 IAA 0.5 μL/mL 的培养值添加到步骤 2.8 中准备的 750 μM 最终浓度中。通过剧烈旋转快速混合。
- 根据您的实验设计,按照步骤 2.5 收集样品。要么是单个样品,在预期蛋白质将可靠地耗尽时,要么在一个耗尽的时间过程中有多个样品。例如,5 分钟间隔便于计时,并提供一系列蛋白质水平。如图1所示,优化策略将给出适当时间的指示。
- 处理样品。
- 将样品放在冰上,如果尚未完成。确保没有样品冻结;如果有,轻轻温暖在手,不断反转,使温度不上升局部。
注:这是最好的做手,因为样品的温度可以评估,它应该总是感到寒冷。放在冰上。这不是暂停点 - 一旦所有样本都是流动的,继续执行下一步。 - 收集所有样品且不再冷冻后,以 3,500 x g旋转 2 分钟(如果可能为 4°C)。
- 倒掉甲醇/中混料,放回冰上;如果所有液体都已去除,请不要担心。
- 将细胞颗粒悬浮在 1 mL 的冰冷 H2O(从步骤 2.3.3)中,并转移到冰上标记的 1.5 mL 管(步骤 2.3.2 中准备)。
- 以 >15,000 x g的时间短暂旋转(例如,总时间 10 s),以重新颗粒细胞,放回冰上并去除液体。
- 通过吸入拆下 H2O。细胞颗粒可储存在-20°C或-80°C进行长期储存。
- 将样品放在冰上,如果尚未完成。确保没有样品冻结;如果有,轻轻温暖在手,不断反转,使温度不上升局部。
- 检查蛋白质已经耗尽的水平由西方斑点分析18。
注:对于大多数目的,可以从单个细胞颗粒中提取足够的蛋白质19和/或核酸,尽管稀有的RNA物种可能需要更多的样本量。
结果
消耗的代表性示例如图1所示。图中提出的三个实验是Pp2、Prp22和Dcp1蛋白质消耗的优化实验。低丰度,乘以体Prp2和Prp22蛋白在40分钟预孵化后,用β-雌二醇先于20%后耗尽至20%以下,随后与辅助蛋白一起耗尽15分钟。较长的预孵育时间会导致更快的耗竭,但在添加辅助剂之前也表现出不良的蛋白质消耗。相比之下,较丰富的Dcp1在相同处理下仅耗尽至约30%,但60分钟的预孵化后,在相同辅助剂处理下,以添加辅助剂之前耗尽为代价,导致消耗至13%。使用β-雌二醇进行50分钟的预孵育和15分钟的辅助蛋白可能在较短的时间点内取得类似的结果,因此可能更为理想。
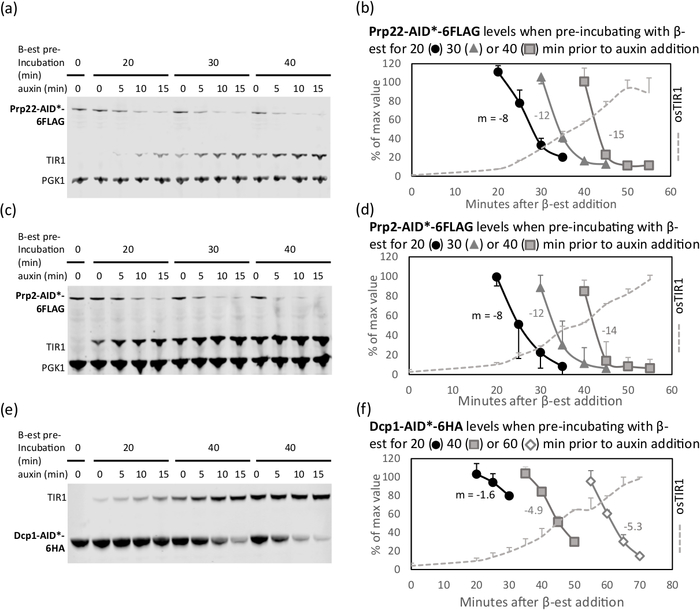
图1:通过调节β-雌二醇预孵育的持续时间,可以调整消耗率。西方斑点18靶蛋白:(a和b)Prp22-AID_-6FLAG,(c和d)Prp2-AID_-6FLAG,以及(e和f)Dcp1-AID_-6HA,来自预孵化的β-雌二醇(β-est)培养物,为20、30、40或60分钟前,辅助添加12。每条巷子里都装有等量的总蛋白质。Pgk1 被检测为可视加载控件,但面板e除外,其中 Pgk1 和 Dcp1 共同迁移。面板a、c和e中蛋白带的定量分别显示在b、d和f面板中。 作为损耗率的度量,计算每个曲线的线性截面(从初始值的 100% 到 30%)的斜率 (m)。最佳预孵育时间是蛋白质水平仍然接近非诱导水平的时间(100%)和随后的消耗速度很快。对于Dcp1( f), 60分钟的预孵育时间太长,因为蛋白质已经开始降解,在缺乏辅助蛋白,而20分钟太短,因为蛋白质不会明显耗尽在这段时间内。在预孵育40分钟后,15分钟使用辅助蛋白,因为蛋白质大约70%耗尽,虽然20分钟会导致进一步耗尽,但也可能导致二次效应。误差条表示两个生物复制的标准偏差。对于每个实验,将显示一个代表性的斑点。此图派生自以前的出版物9。请点击此处查看此图的较大版本。
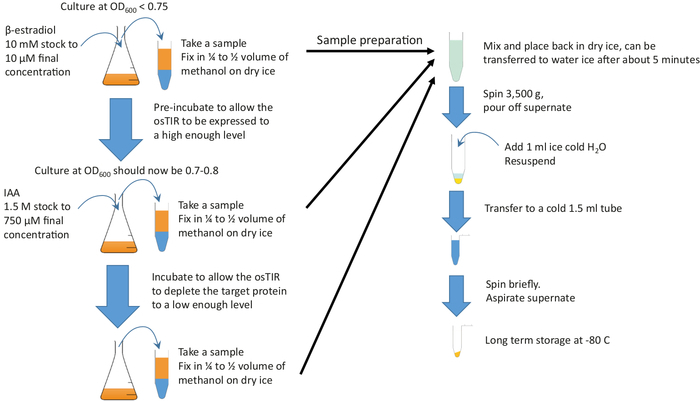
图 2:图形摘要。在富培养基和所需温度下生长的足够培养基中加入β-雌二醇,以便开始预孵育。在加入IAA(辅助剂)开始耗尽之前,在所需的预孵育时间内继续增长。孵化前和耗尽时间取决于要耗尽的蛋白质,但预孵育时间通常在20-60分钟之间,消耗时间一般在10至20分钟之间。g 耗竭。这些样品在颗粒和储存前被快速固定在冷甲醇中。请点击此处查看此图的较大版本。
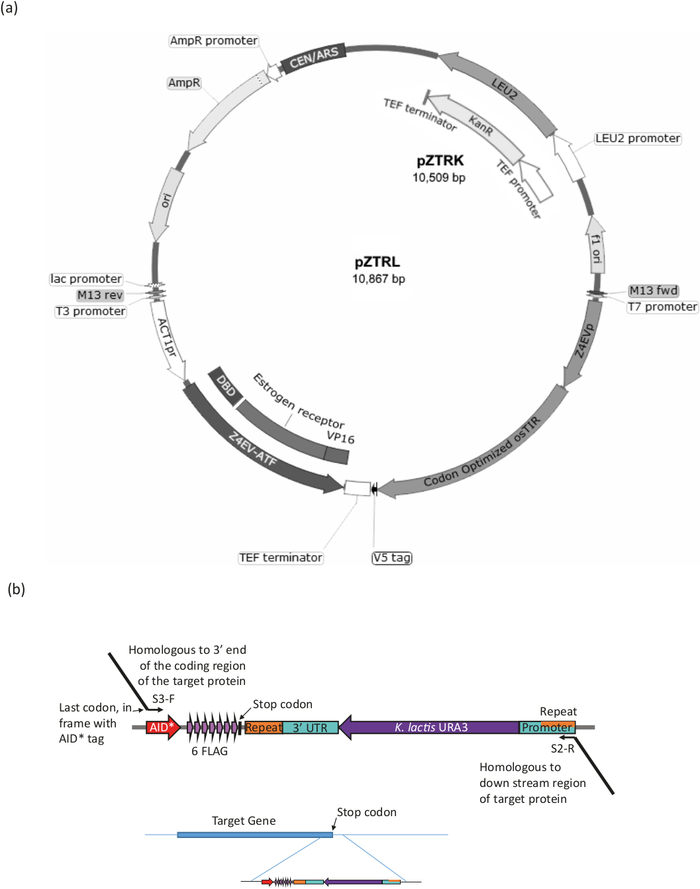
图 3:B-est 系统的应变生成。(a) 要使用 AID® 系统(pZTRL(LEU2) 或 pZTRK(卡纳霉素 (G418) 抗性)生成酵母菌株,应将质粒引入菌株,或者,ATF 和 osTIR 可插入到基因组中。PCR从3'端引物生成的片段的同源重组(参见图3b和表1)。(b) 靶蛋白的C端标记是通过PCR扩增的质粒pURA3-AID_-6FLAG(pURA3_AID_-6HA仅在标记中不同,可以以完全相同的方式处理),使用长汀引基锡S3-F和S2-R 与 3' 扩展同源到目标蛋白的 3' 端。前引基扩展应包括最后一个氨基酸科顿在框架与AID®标签的开头,并且不得包括停止科顿。反向引信扩展应为编码区域下游的区域。一旦插入基因组,失去URA3标记的细胞(通过在标记两端发现的相同区域之间的同源重组)可以通过5-FOA的生长来选择,即反选择URA3细胞。请点击此处查看此图的较大版本。
| a. 引子序列 | |||||||
| 目标 | 位置 | 底漆 | 名字 | 序列 | Tm (°C) | ||
| pZTRL | 516 | F | pZTRL_F | <-同源区->GCGACAGCATCACCACACTTCG | 61.23 | ||
| 7897 | R | pZTR_R | CGCCGCCTCTCTGCAGA<-同源地区(RC)-> | 61.30 | |||
| pZTRK | 9154 | F | pZTRK_F | <-同源地区->ACGTGAGCCATATATATATTGCGCCCCC | 59.40 | ||
| 5897 | R | pZTR_R | CGCCGCCTCTCTGCAGA<-同源地区(RC)-> | 61.30 | |||
| pURA3-AID_-6FLAG 或 pURA3_AID_6HA | F | S3-F | <-同源区->CGTACGCAGCGAC | 59.21 | |||
| R | S2-R | 同源地区(RC)-> | 52.76 | ||||
| pZRTL/K 用于放大 +-est AID 系统 | |||||||
| pURA3-AID®-6FLAG/6HA 放大AID®和表位标记目标蛋白(龙汀程序) | |||||||
| <-同源区 -> | 与要插入系统的侧翼区域的区域同源。该区域越长,修改成功的可能性就越大;建议使用 50 - 100 个底座。 | ||||||
| <-同源区 (RC) - > | 区域与要插入系统的侧翼区域同源,请记住使用反向补码。如上所述,这个区域越长越好。 | ||||||
| Tm (°C) | 使用 %GC 方法的 Tm 50 mM NaCl | ||||||
| b. PCR 混合 | |||||||
| 组件 | 体积 (μL) | ||||||
| 模板 | <10 | ||||||
| NEB Phusion HF 缓冲器 (5x)* | 100 | ||||||
| 正向底漆 100 μM | 2.5 | ||||||
| 反向底漆 100 μM | 2.5 | ||||||
| dNTP 每个 10 mM | 10 | ||||||
| H2O | 到 500 | ||||||
| • 也可以使用 NEB Phusion GC 缓冲器 (5x),但不可取 | |||||||
| 制作这种混合物,分成10个管,每个50μl混合,并执行PCR作为表1 c。 | |||||||
| 通过在胶凝剂上运行来检查 PCR 的工作 | |||||||
| 将所有成功反应组合成一管和乙醇沉淀物 | |||||||
| 使用 PCR 生产的所有材料对酵母进行转化 | |||||||
| c. PCR 条件 | |||||||
| 步 | 温度 (°C) | 时间 | |||||
| 初始变性 | 98 | 30 s | |||||
| 25-35 周期 | 变性 | 98 | 10 s | ||||
| 退火 | 45~60 | 20 s | |||||
| 扩展 | 72 | 30 s/kb | |||||
| 最终扩展 | 72 | 10分钟 | |||||
| 保持 | 8 | ||||||
| 对于龙汀底漆集(S3-F 和 S2-R),在 45°C 时退火,pZTRL/K 底漆的发火温度为 60°C | |||||||
| 对于龙汀 PCR 延长 3 分钟,对于 pZTRL/K 延长 3 分钟 | |||||||
表1:底漆序列、PCR混合物和PCR条件。
讨论
优化良好的方案可以产生目标蛋白的快速和高效消耗。确定使用β-雌二醇的近似预孵育时间很重要,因为这增加了耗竭的可重复性,但可以容忍预孵育时间的微小变化。另一方面,由于蛋白质水平下降非常迅速,在添加辅助剂后必须注意时间。
这种方法的一个优点是,通过不同的预孵育时间与β-雌二醇和IAA孵育时间的组合,可以实现调谐的消耗。例如,如果需要,通过减少预孵育时间,目标蛋白可以更缓慢地耗尽。
与 OsTIR 构成的系统系统相反,β-est AID 系统具有某些优势。例如,如果靶蛋白对生存至关重要,则 osTIR 的调节表达可以避免目标蛋白过早耗尽。此外,osTIR的表达可以调整,以适应目标蛋白的丰度和降解的易感性,并且消耗可以是快的,也可以是缓慢的。与拉帕霉素不同,两种小分子效应器β-雌二醇和辅助素,在这里使用的条件下不会干扰酵母代谢,不像在锚离系统1中使用的雷帕霉素。
应该注意的是,标记一些蛋白质会破坏它们的功能,这是任何目标耗尽系统的问题。在这种情况下,当 C 端标记不起作用时,N 终端标记可能工作。此外,并非所有蛋白质都会被有效耗尽;例如,目标蛋白上的 AID 标记可能无法访问 osTIR 蛋白。因此,在AID标记后,在优化β-雌二醇预孵化和辅助素处理的时间之前,应测试每个靶蛋白对生长标记的任何影响,并确定消耗是否有效。
此 AID® 系统非常简单,可与任何不涉及进一步生长的后续实验程序兼容,例如蛋白质、DNA 或 RNA 分析或显微镜。此外,该系统与硫标签结合,以纯化新生的RNA20时效果良好。
该系统提供了一种快速、具体和可重复的方法,在不影响酵母细胞代谢的情况下消耗蛋白质。
披露声明
作者没有什么可透露的。
致谢
感谢简·里德发起这一计划,芭芭拉·特劳为发展,瓦希德阿斯兰扎德为"乌拉循环"建设和苏珊娜德卢卡斯为许多有益的讨论。这项工作得到了墨西哥国家科学委员会(CONACYT)和爱丁堡大学生物科学学院的奖学金,以及威康大学获得IEM博士学位[105256]的资助,以及威康资助[104648]给JD Beggs.韦康细胞生物学中心的工作得到了威尔康核心基金的支持[092076]。
材料
| Name | Company | Catalog Number | Comments |
| Adenine sulphate | Formedium | DOC0230 | |
| Agar | Formedium | AGA03 | |
| Β-estradiol | Sigma Aldrich | E2758-1G | 10 mM solution in ethanol. Store at -20 °C |
| DMSO | Alfa Aesar | 42780 | DMSO should be solid at 4 °C |
| Glucose | Fisher Scientific | G/0500/60 | |
| IAA 1H-Indole-3-acetic acid | Across Orgainics | 122150100 | Auxin analogue. 1.5 M in DMSO. The solution will be a russet colour and darken as time goes on; a deep red solution should be discarded and a new one made. Store at -20 °C. |
| Methanol | Fisher Scientific | M/4000/PC17 | CAUTION Toxic and flammable |
| Phusion High-Fidelity DNA Polymerase | NEB | M0530 | |
| Peptone | Formedium | PEP03 | |
| SCSM single drop-out –ura | Formedium | DSCS101 | |
| Yeast Extract | Formedium | YEA03 | |
| Yeast nitrogen base without amino acids with amonium sulphate | Formedium | CYN0410 |
参考文献
- Haruki, H., Nishikawa, J., Laemmli, U. K. The Anchor-Away Technique: Rapid, Conditional Establishment of Yeast Mutant Phenotypes. Molecular Cell. 31, 925-932 (2008).
- Bellí, G., Garí, E., Piedrafita, L., Aldea, M., Herrero, E. An activator/repressor dual system allows tight tetracycline-regulated gene expression in budding yeast. Nucleic Acids Research. 26, 942-947 (1998).
- Alexander, R. D., et al. RiboSys, a high-resolution, quantitative approach to measure the in vivo kinetics of pre-mRNA splicing and 3′-end processing in Saccharomyces cerevisiae. RNA. 16, 2570-2580 (2010).
- Deshaies, R. J., Joazeiro, C. A. P. RING Domain E3 Ubiquitin Ligases. Annual Review of Biochemistry. 78, 399-434 (2009).
- Tan, X., et al. Mechanism of auxin perception by the TIR1 ubiquitin ligase. Nature. 446, 640-645 (2007).
- Teale, W. D., Paponov, I. A., Palme, K. Auxin in action: signalling, transport and the control of plant growth and development. Nature Reviews Molecular Cell Biology. 7, 847-859 (2006).
- Nishimura, K., Fukagawa, T., Takisawa, H., Kakimoto, T., Kanemaki, M. An auxin-based degron system for the rapid depletion of proteins in nonplant cells. Nature Methods. 6, 917-922 (2009).
- Morawska, M., Ulrich, H. D. An expanded tool kit for the auxin-inducible degron system in budding yeast. Yeast. 30, 341-351 (2013).
- Kubota, T., Nishimura, K., Kanemaki, M. T., Donaldson, A. D. The Elg1 Replication Factor C-like Complex Functions in PCNA Unloading during DNA Replication. Molecular Cell. 50, 273-280 (2013).
- Brosh, R., et al. A dual molecular analogue tuner for dissecting protein function in mammalian cells. Nature Communications. 7, 11742 (2016).
- DeRisi, J. L., Iyer, V. R., Brown, P. O. Exploring the metabolic and genetic control of gene expression on a genomic scale. Science. 278, 680-686 (1997).
- Mendoza-Ochoa, G. I., et al. A fast and tuneable auxin-inducible degron for depletion of target proteins in budding. Yeast. , (2018).
- McIsaac, R. S., et al. Synthetic gene expression perturbation systems with rapid, tunable, single-gene specificity in yeast. Nucleic Acids Res. 41, e57 (2013).
- Prusty, R., Grisafi, P., Fink, G. R. The plant hormone indoleacetic acid induces invasive growth in Saccharomyces cerevisiae. PNAS. 101, 4153-4157 (2004).
- Geitz, D., St Jean, A., Woods, R. A., Schiest, R. H. Improved method for high efficiency transformation of intact yeast cells. Nucleic Acids Research. 20, 1425 (1992).
- Widlund, P. O., Davis, T. N. A high-efficiency method to replace essential genes with mutant alleles in yeast. Yeast. 22, 769-774 (2005).
- Longtine, M. S., et al. Additional modules for versatile and economical PCR-based gene deletion and modification in Saccharomyces cerevisiae. Yeast. 14, 953-961 (1998).
- Eaton, S. L., et al. A Guide to Modern Quantitative Fluorescent Western Blotting with Troubleshooting Strategies. Journal of Visualized Experiments. , e52099 (2014).
- Volland, C., Urban-Grimal, D., Géraud, G., Haguenauer-Tsapis, R. Endocytosis and degradation of the yeast uracil permease under adverse conditions. Journal of Biological Chemistry. 269, 9833-9841 (1994).
- Barrass, J. D., et al. Transcriptome-wide RNA processing kinetics revealed using extremely short 4tU labeling. Genome Biology. 16, 282 (2015).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。