需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
小鼠肝细胞核球状估计的高通量原位方法
摘要
我们提出了一种稳健、经济、灵活的方法,用于测量固定/冷冻组织样本中肝细胞数和核球菌的变化,而不需要流动细胞测量。我们的方法提供了肝细胞学的强大样本范围特征,非常适合跟踪肝损伤和疾病的进展。
摘要
当肝脏受伤时,肝细胞数量减少,而细胞大小、核大小和脂肪增加。非乳腺细胞的膨胀,如胆囊细胞、肌瘤细胞、祖细胞和炎症细胞也表明慢性肝损伤、组织重塑和疾病进展。在此协议中,我们描述了一种简单的高通量方法,用于计算与损伤、慢性疾病和癌症相关的肝脏细胞组成的变化。我们展示了如何利用从二维(2D)组织部分提取的信息来量化和校准样品内的肝细胞核球,并使用户能够在原位定位肝脏内的特定多西子。我们的方法需要获得固定/冷冻肝材料、基本免疫细胞化学试剂和任何标准的高含量成像平台。它是标准流细胞学技术的有力替代品,需要破坏新收集的组织、空间信息丢失和潜在的分解偏差。
引言
哺乳动物肝脏中的肝细胞可以经历停滞的细胞因子细胞生成双核细胞,DNA内复制产生多倍体核,含量高达16N的DNA含量。产后发育、衰老和应对各种细胞压力时,细胞和核障碍总体增加1。多倍化过程是动态的和可逆的2,尽管它的精确生物功能仍不清楚。增加的普洛伊与增殖能力降低4、遗传多样性2、适应慢性损伤5和癌症保护6有关。肝细胞性改变发生由于昼夜节律改变7,并律下8。最值得注意的是,肝脏的脂肪轮廓因损伤和疾病9而改变,令人信服的证据表明,特定的蛋白核变化,如增加+8N核或2N肝细胞的丧失,为跟踪非酒精性脂肪肝疾病(NAFLD)进展3,10,10或病毒感染的差分影响11提供了有用的签名。3
一般来说,肝损伤和再生与肝细胞细胞大小和核面积增加有关,同时肝细胞总数减少,特别是那些具有2NDNA含量10,11,11的肝细胞。肝脏中的帕伦奇马尔损伤也经常伴有非泛状细胞(NPCs)的扩增,包括基质肌纤维细胞、炎症细胞和二能肝祖细胞。高通量方法,提供肝细胞数和核球的定量细胞学概况,同时也考虑到NPC的变化,因此具有相当大的潜力,作为研究和临床工具,以跟踪肝脏在损伤和疾病期间的反应。最近对肝细胞癌人体样本中的普西迪光谱进行实地分析也表明,核球在肿瘤内急剧增加,在更具攻击性的肿瘤亚型中特别扩增,减少分化和TP53 13的损耗。因此,在核策略定量评估方面的方法学进步很可能有助于今后对肝癌的预后分析。
本协议介绍了一种灵活的高通量方法,用于对小鼠肝组织部分进行比较分析,它提供了肝细胞数的详细细胞分析、NPC响应和用于估计核高倍的内部校准方法(图1)。肝细胞在核尺寸和核测量表之前,通过肝细胞核因子4α(HNF4+)免疫标签与NPC区别开来。"最小DNA含量"通过将平均Hoechst 33342强度(DNA密度的代理)与插值的三维(3D)核体积集成,估计为所有圆形核掩膜"最低DNA含量"。然后,使用NPC校准肝细胞最小DNA含量,以生成核球状轮廓。
使用高含量成像进行图像采集、核分割和图像分析,使包含数万个细胞的大面积二维(2D)肝部分进行筛选。提供一个定制程序,用于自动处理高含量图像分析数据,以生成所有圆形肝细胞核的样本范围的ploidy轮廓。这是使用免费下载软件,计算基于立体图像分析(SIA)10,11,14,15的核策略。10,11,14,15SIA方法以前曾被流细胞测定证实为一种准确(尽管费力)的方法,用于估计肝脏14中的肝细胞核糖核,假设循环核形态和核大小与DNA含量之间的单调关系。在本议定书中,这两个核参数都是通过核测量和Hoechst 33342标签的评估来衡量的。计算每个核掩膜的"最小DNA含量"后,使用NPC校准肝细胞核糖核糖,这些NPC具有已知的2-4NDNA含量,因此作为有用的内部控制。
与传统的流细胞测量方法16相比,所述方法使肝细胞核球菌能够就地评估,并且不需要获得新的组织或分解方法,这些方法可能会偏袒结果,难以标准化。与所有基于 SIA 的方法一样,由于赤道平面外较大核的切片,2D 采样对核策略子类 >2N 的代表性不足。全组织蛋白体剖面还描述了所有圆形肝细胞核掩码的最低DNA含量,并且不直接区分单核肝细胞和具有同一双核核核的两个离散("不接触")核。然而,这项议定书的简单性使得可以相当的余地加以调整,以考虑到核间隔或细胞周界分析等其他参数,这将有助于识别双核细胞,从而更详细地评估细胞结构。
Access restricted. Please log in or start a trial to view this content.
研究方案
所有动物实验以前都得到CIPF道德委员会的批准。老鼠被安置在费利佩中心(西班牙瓦伦西亚)的无病原体设施中,注册为实验动物饲养者、用户和供应中心(注册号。根据现行适用的欧洲和西班牙动物福利条例(RD 53/2013),ES 46 250 0001 002)。
1. 组织收获和样品制备
注:此协议描述了如何在不事先固定或冷冻保存的情况下冻结组织。对于以前固定/冷冻保存的样本,请转到第 2 节并省略步骤 3.1。所有分析均使用12-16周的成年雌性C57BL/6小鼠进行。
- 通过芬太尼/五巴比妥内腹注射来牺牲动物,然后进行宫颈脱位。当小鼠朝方腹腔一侧时,打开腹腔,用钳子抓住皮肤,用手术剪刀从下腹部底部到胸骨底部进行垂直切口,从而打开腹腔并露出肝脏。
- 使用细钳小心地取出胆囊,解剖肝脏,并在充满磷酸盐缓冲盐水 (PBS) 的 10 厘米培养皿中冲洗选定的肝球。
注:建议比较每种动物的同一肝叶,在这种情况下,使用了中位叶。 - 在室温 (RT) 下,使用最佳切割温度 (OCT) 介质填充标记的低温。避免 OCT 气泡。如果它们出现,请使用针或移液器尖端将其推到模具边缘。
- 将肝卵泡嵌入一个填充的OCT低温中,并立即将其放在干冰上,以确保快速冻结。在-80°C下储存冷冻,直到冷冻。
2. 冷冻切除
- 在干冰上运输低温,以避免组织退化。在低温解剖之前,冷冻素内平衡设置为-20°C20分钟。
- 通过在塑料低温器底部施加压力来喷射样品。将液体 OCT 涂抹到 RT 的加热样品盘上,在低温塔中放置,并附加 OCT 嵌入式肝脏样本。施加温和的压力,等待 3 分钟,让 OCT 冻结,确保样品粘在磁盘上。
注意:避免尽可能用手指处理样品,以避免组织退化。 - 将样品锁定到冷冻器的臂中并调整方向,使样品边缘与低温铲刀平行。切成样品,直到达到组织。
- 在 6 μm 厚度下对样品进行切片。将标有聚酰胺涂层的幻灯片放在样品上 5 s,让样品粘在幻灯片上。将幻灯片放在 RT 上 3-5 分钟,然后,为了获得最佳效果,请直接转到第 3 节。
注:为了处理多个新鲜冷冻样品,通过暂时将幻灯片存放在干冰上的滑箱中,直到处理所有样品,可以重现结果。使用此方法时,允许所有幻灯片在继续第 3 节之前对 RT 进行平衡。可以使用形式固定石蜡嵌入式(FFPE)样品,但通过这种方法增加了背景自荧光。从FFPE样品中继续,通过在聚酰胺处理的滑轨上捕捉40°C水浴的部分,在4μm.安装处进行切片。在 60°C 下加热 1 小时,然后在含有二甲苯 (x2)、乙醇 100%(x2)、96% (x2)、70% (x1) 和 dH2O (x1) 的 Coplin 罐子中通过串行 RT 哈希(5 分钟)进行脱麻。要暴露抗原,在 90°C 下将幻灯片放入西酸缓冲液中 20 分钟,然后再在 RT 的 PBS 中回火幻灯片。
3. 荧光免疫标签
- 在RT将幻灯片转移到装有PBS的Coplin罐中,用温和的搅拌(重复3倍)将幻灯片转移到PBS填充的Coplin罐中,然后洗涤3分钟,在PBS中涂抹1 mL4%甲醛(PFA),将组织部分固定在烟雾罩中。
注:从现在起到免疫染色过程结束,避免样品干燥。 - 使用疏水笔干燥每个组织部分周围的区域并环绕。在 RT 中使用 0.5% 的非离子表面活性剂(即 Triton X-100)在 RT 中渗透 15 分钟。然后用温和的搅拌(重复2倍)在PBS填充Coplin罐子中洗涤3分钟。
- 块使用过滤溶液1%牛血清白蛋白(BSA),5%马血清,0.2%非离子表面活性剂在PBS(在RT至少1小时)。
- 在深色潮湿的染色室中,在4°C的夜间阻塞缓冲液中稀释原发HNF4®抗体(有关抗体和特定稀释物的材料表)。
- 将幻灯片放入充满 PBS 的 Coplin 罐子中,并使用温和的搅拌(重复 4x)洗涤 3 分钟。
- 孵化与Alexa-488结合的二次抗体和Hoechst稀释在过滤1%BSA和0.2%非离子表面活性剂在PBS在RT在黑暗的湿润染色室2小时(见材料表抗体和特定的稀释)。
- 将幻灯片放入充满 PBS 的 Coplin 罐子中,并使用温和的搅拌(重复 4x)洗涤 3 分钟。使用温和的搅拌(重复2倍),在 ddH 2 O 中洗涤 3 分钟。
- 将两滴荧光安装介质放在盖玻片(24 x 60 mm)上,并在滑道上放置滑轨,通过施加柔和的压力消除气泡,从而安装滑道。对于长期存储,用透明指甲油在边缘密封盖玻片,并储存在 4 °C 的黑暗中。
- 在继续之前,使用传统的荧光显微镜检查幻灯片,以确保良好的固定和免疫标记。
注: 有关预期结果,请参阅图 2A,B。
4. 荧光图像采集
注:对于此步骤,需要一个支持自动荧光图像采集的高含量成像平台(材料表)。
- 打开成像系统并打开新的采集协议。
- 选择 10x 目标,记下视野区域(本例中为 0.6 mm2)。
- 设置参数,使用适当的激励和发射滤清器(根据步骤 3.6)获取荧光图像。对于 Hoechst 和 Alexa-488,选择分别具有 390/18 和 438/24 nm 发射和 432.5/48 和 475/24 nm 发射的"DAPI"和"GFP"通道。
- 聚焦样品并确保信号强度不饱和。确保所有图像捕获的时间与所有图像的曝光时间相同,或使用在曝光时间内校正荧光强度的系统。
- 扫描样品并获取足够的图像,以获得组织部分的完整覆盖(根据样本大小,大约 20-50 个视场)。
- 查看图像数据库,手动消除 (i) 聚焦不良的场,(ii) 每个组织部分边界处的场(以避免偏置细胞密度计算),以及 (iii) 包含组织部分折叠/物理损坏区域(如果存在)。"
5. 自动荧光图像分析
注:此步骤需要适当的图像分析软件(材料表),能够:(1) 自动识别 405 nm(核分段)图像中的 Hoechst 标记核;(2) 评估平均 Hoechst 核强度和近值测量值;(3) 阈值分析以确定 488 nm (HNF4]) 下核荧光的 +/- 状态。需要一些基本的操作员培训/专业知识来目视评估和调整程序中的分段和阈值参数,以确保核和 HNF4+/- 状态得到最佳门控(图 2)。
- 在图像分析软件中,从步骤 4.5 中打开包含 Hoechst (405 nm) 和 HNF4+ (488 nm) 图像的采集文件,并创建新的分析协议。
- 定义用于核分割(霍赫斯特,405 nm)和肝细胞/NPC阈值分析(HNF4+,488 nm)的波长。
- 调整软件的核分割参数(如"最小核区域"和核检测"灵敏度"),以确保核以最佳方式分离。
注:肝细胞的良好分段应优先于NPC。肝细胞核是特征圆的(四分位数范围:40~64 μm2)。NPC核,如正弦内皮核核,在形状上是扁平的/椭圆形的或不规则的,通常比肝细胞小,包装更紧密(四分位数范围:30~43 μm2)。对于小鼠肝脏,使用了最小核面积±23 μm2和检测"灵敏度"65%(有关预期结果,参见图2C,D)。灵敏度根据像素团的强度确定如何识别为单个核,在进行自动图像分析之前,应对用户设置的每个样本进行经验测试。 - 在 488 nm 处修改阈值强度,以确保肝细胞 (HNF4+) 和非甲形细胞 (HNF4+-) 的最佳浇注。
注: 有关预期结果,请参阅图 2C,D。阈值强度值是相对的,取决于染色效率和采集设置,如激光强度。因此,用户应将其标准化。使用已知的HNF4+-细胞,如内皮细胞和圆体NPC作为内部负对照和双核肝细胞核作为染色的积极参考。在对整个数据集应用分析参数之前,使用少量图像测试分析参数,以确保良好的核分割和强度阈值分离。 - 选择要量化的下列核参数:(1)基于Hoechst染色(μm2)的核区域,(2)平均核高强度(RU),(3)核伸长系数(原子核短轴与原子短轴的平均比, 其中中心对称[非长)物体的值为 1,(4) Nuc 1/(形式因子),均值核"圆度"指数按周界 2/(4= x 面积)计算)。值范围从 1 到无穷大,其中 1 是一个完美的圆,(5) HNF4+ 状态(正-1 或负 -0),以及 (6) 基于"重心"(cg)的核 x/y 坐标,这是一种以亚像素精度从灰度图像中定位对象中心的方法。
- 运行所有示例数据集的分析,并将数值数据从步骤 5.5 导出到电子表格软件。
6. 数据分析
注:数据分析步骤可以使用任何标准电子表格软件执行。
- 计算肝细胞和非肝细胞细胞数。
- 通过将视场数乘以视场区域(步骤4.2),计算每个样本分析的肝脏部分总面积。

- 使用为每个肝脏部分生成的电子表格文件,仅选择 HNF4+ 核来筛选数据。计算分析的HNF4+核的总数,并将其除以分析的总面积,以获得每个样品的平均肝细胞密度(图2F)。

- 通过筛选 HNF4+-单元格的电子表格(图 2E),对非计算单元执行相同的计算。
- 通过将视场数乘以视场区域(步骤4.2),计算每个样本分析的肝脏部分总面积。
- 计算肝细胞核大小分布。
- 使用电子表格软件,筛选数据仅选择HNF4+核。
- 直方图中核区域的绘图值(图2G)。将箱宽度设置为 5 μm2。
注:根据步骤 6.1.1,可以校正区域(核/mm2)的频率值。
- 执行肝细胞核球分析。
注:步骤 5.6 中的电子表格数据用于为每个样本生成核策略配置文件。此过程已自动化,可以使用自定义书面软件执行,该软件可免费下载,支持信息和演示数据集,https://github.com/lukeynoon(参见补充文件).为希望调整方法的用户提供源代码。下面概述了该算法的说明以及安装和使用说明。程序使用电子表格数据自动将肝细胞核分成两组;(1)具有"简单"圆核和(2)代表具有±2c倍性的双核细胞的"复杂"非圆核。下一步计算所有"简单"核的最低核DNA含量(核区域和DNA密度的函数)。然后,后续步骤使用HNF4+-核作为已知的2-4N内部控制自动校准HNF4++肝细胞核策略。- 下载并安装软件。
- 从:下载打包的应用程序:https://github.com/lukeynoon
- 启动 MATLAB。导航到工具行程的 APP 选项卡,单击"安装应用程序"并打开称为"Ploidy_Application.mlapp 安装"的下载应用程序。将显示一条消息以确认成功安装。
注: 应用程序现已准备就绪,并将保留在工具跳闸的 APP 选项卡中。
- 格式化输入数据。
注:在自动核策略分析之前,所有包含高含量成像数据的电子表格文件(步骤 5.6)都应按照以下说明进行存储和格式化。- 在每个导出的数据文件中 (.XLS 97-2004 工作簿)从步骤 5.6 中,包括一个称为"细胞度量值"的工作表,其中包含列中列的 ploidy 分析所需的所有数据(图 3A)。确保包含列标题名称的电子表格布局与图 3A保持不变,因为分析方法通过搜索这些名称来查找正确的列数据(请参阅补充文件中的演示数据集以供参考)。例如,如果高含量图像分析软件不生成"光通量"列(图 3A),则在同一位置手动插入"光通量"列,即列 K 并将其填充为零。
- 对于每个实验条件(例如"受伤-d14"),提供一个控制数据集,用于计算2-4N核复核校准的内部控制(步骤6.3.4.3)。在这里,从未经处理的成年垃圾伴侣中选择肝脏样本("控制-d0";图3B+D。
- 对于生物复制(根据条件),将每个电子表格存储在其自己的文件夹中(如图3B所示)。以增量方式命名文件夹前缀,例如"示例 1、示例 2、示例 3...样值N",根据其中包含的文件名。因此,每个数据集文件夹(例如"Control-d0")都应包含一系列子文件夹("示例 1"、"Sample2"等),每个文件夹都包含具有相同相应名称的电子表格文件。
- 运行应用程序。
- 在 MATLAB 中,通过单击工具跳闸的 MY APPS 选项卡中的图标(图3C),启动"Ploidy_Application"。将显示Ploidy_Application图形用户界面 (GUI) (图 3C)。
- 单击"路径以控制数据"按钮以导航到控件数据复制所在的文件夹(例如,"控制-d0")。然后,此数据路径将显示在接口中(例如/用户/桌面/控制 d0)。
- 接下来,在"文件夹前缀"中键入要为输出文件指定的名称(例如,"示例")。
注: 此前缀可以更改为任何文本,前提是文件夹和文件名保持增量命名。 - 单击"路径到其他数据"按钮并导航到比较数据复制所在的文件夹(例如,"受伤-d14")。然后,此数据路径将显示在接口中(例如/用户/桌面/受伤-d14)。
- 单击"运行"!分析完成后,状态栏将改为"分析完成!.."。
注:应用程序将报告,每个样本,"简单"核分层到+2n,2n+4n,4n_8n和8n+的绝对计数和总的百分比(图3D)。这些文件将自动保存在每个示例文件夹中,如:"Count_2n.txt"、"Count_2n_to_4n.txt"、"Count_4n_to_8n.txt"、"Count_8n_and_higher.txt"、"Percentage_2n.txt"、"Percentage_2nto4n.txt"、"Percentage_4nto8n.txt"、"Percentage_8n_and_higher.txt"。Count_2n.txtCount_2n_to_4n.txt" "Count_4n_to_8n.txt" "Count_8n_and_higher.txt" "Percentage_2n.txt" "Percentage_2nto4n.txt" "Percentage_4nto8n.txt" "Percentage_8n_and_higher.txt"Ploidy_Application将自动为每个样本保存一个列表,即"Ploidy_All_Hepatocytes.txt"和"Ploidy_NonHepatocytes.txt"中"简单"肝细胞和非肝细胞核的所有单独ploidy估计值。对于控制数据集,该方法还保存了为 ploidy 分层计算的最小 DNA 含量阈值(请参阅步骤 6.3.4.3.7)中的名为"Normalised_Thresholds_Control"。最后,应用程序将为控件和称为"摘要"的选定比较条件数据生成一个文件夹。此文件夹包含两个子文件夹,"Ploidy"和"分层",其中包含提供的所有样本的平均值(图 3D)。
- 方法的说明。
注:以下部分详细介绍了核普洛伊迪分析软件使用的方法。如果用户选择不使用该应用程序,可以使用电子表格软件手动计算核策略配置文件,然后执行这些步骤。- 根据核测量,将核分离成"简单"或"复杂"。
- 计算所有核的"循环指数",定义为核"伸长因子"除以"Nuc 1/(形式因子)",其中值 1.0 表示一个完美的圆。
注:"核伸长"和"Nuc 1/(形式因子)"是评估互补、非重叠形态测量标准的物体"循环性"的两个离散度量。前者测量对象的长轴和短轴,而后者将对象的周长与其区域的长度进行比较。为了加强本议定书中使用的核循环的定义,这两项测量结果已合并为一个单一的"循环指数"。以前使用所述方法估计核废料的方法只使用核伸长17。虽然使用这种方法获得了可接受的结果,但作者指出,复合的"循环指数"改善了单核和双核半核细胞中人工选择的核的鉴别(未显示的数据)。 - 将循环指数 = 0.8 的核分类为"复杂",将 > 0.8 分类为"简单"。
- 计算所有核的"循环指数",定义为核"伸长因子"除以"Nuc 1/(形式因子)",其中值 1.0 表示一个完美的圆。
- 估计所有"简单"核的"最小"DNA含量(m)。
- 使用公式计算核半径 (r):

- 使用球体公式的体积计算核体积 (v):
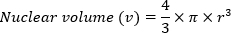
- 使用公式为最小DNA含量 (m) 生成相对值:

- 使用公式计算核半径 (r):
- 使用 NPC (HNF4+-) 核作为内部 2-4N 控制校准数据集。
注:NPC 具有 2-4N DNA 含量,具体取决于细胞周期状态。因此,NPC"最小"DNA含量(NPCm)的平均值随伤害而增加(图4A)。通过建立表示 4c 阈值的 NPCm的上限(图 4B),校准误差最小化。- 在电子表格中,仅选择带有值为"m"值的 NPC 核,该值位于模式的 1 个标准差 (SD) 内(这可过滤出可能的分段误差中的噪声)。
- 在此过滤范围内,检查核区域及其相应的均值 Hoechst 强度(图4C)。
- 以最大核霍赫斯特强度估计此过滤范围内最小的核区域(即曲线在筛选数据集中改变方向的点,如图4C中的红色圆圈所示)。此值表示 2N-4N 过渡状态 (t),其 4c 核的采样占主导地位于 2c 核之上,从而产生平均 Hoechst 强度的最大值。
注: 此值由软件自动确定;但是,电子表格用户可以手动选择此点作为过渡大小。 - 通过按照步骤 6.3.4.2 计算此过渡大小 (tm) 表示的最小 DNA 含量。
- 要估计 NPCm数据集的 4N 肩,在 tm的值中添加 1 SD 。所得数字(图4B)描述了用于核糖体分层(S4c)的NPC最小DNA含量的上限。
- 对所有"控制"样本重复步骤 6.3.4.3.1_6.3.4.3.5。
注:例如,在图 3中,未受伤的控制肝("控制 d0")用作控制条件。 - 计算"控制"样本的平均4c分层阈值(S4c),并用它来推断2c(S2c)和8c(S8c)边界,以最小的DNA含量(m)。分层阈值由软件自动生成和存储(步骤 6.3.3.3)。
注:根据研究设计,可以为每个情况或特定条件(例如健康控制肝脏)计算平均分层阈值。但是,核 Ploidy 分析软件要求将一组 2 个文件中的一个指定为"控制",以便计算相对 ploidy 值。 - 使用步骤 6.3.4.3.7 中生成的 S2c值计算所有原子核的 ploidy 值,如下:

- 根据以下标准,将"简单"肝细胞 (HNF4+) 核放入 2c/4c/8c/>8c 支架中:"2c" HNF4+ = p = 2;"4c" HNF4+ = 2 < p = 4;"8c" HNF4+ = 4 < p = 8;">8c" HNF4+ = 8 < p.
- 为了重建ploidy子群的空间模式,根据获取这些子组的相应字段,将每个样本电子表格中的核数据分开。然后使用关联的核 x/y 坐标(从步骤 5.5 开始)以 2D 为单位绘制 ploidy 子组(图 5C)。
- 根据核测量,将核分离成"简单"或"复杂"。
- 下载并安装软件。
Access restricted. Please log in or start a trial to view this content.
结果
该方法已用于测量胆静损伤对成年小鼠肝脏的影响,通过喂养动物0-21天与肝毒性饮食含有0.1%3,5-二氧碳基-1,4-二氢胶碱(DDC)17。慢性DDC喂养导致肝细胞损伤增加,NPC的皮球和外皮扩张。用户应注意,在核策略中可能存在小鼠应变和年龄依赖性差异,所有分析均使用12-16周的成年雌性C57BL/6小鼠进行。
在HNF4+?...
Access restricted. Please log in or start a trial to view this content.
讨论
介绍了一种高含量、高通量的方法,用于分析毛毛肝中肝细胞核细胞核球的组织重塑和估计。熟悉该过程后,用户可以在 3-5 天内处理、成像和分析多个样本,生成大型可测试数据集,从而提供肝脏健康的详细签名。鉴于样品制备方法的简单性,以及分析的大量细胞和组织区域(平均14 mm2/样本),结果非常可靠,具有很高的可重复性。图像捕获和分析的自动化还可以消除这些重要步骤中?...
Access restricted. Please log in or start a trial to view this content.
披露声明
作者没有什么可透露的。
致谢
这项工作由西班牙MINECO政府赠款BFU2014-58686-P(LAN)和SAF-2017-84708-R(DJB)资助。LAN得到全国MINECO Ramón和Cajal奖学金RYC-2012-11700和计划GenT奖(瓦伦西亚大学,CDEI-05/20-C)和FMN的支持,由瓦伦西亚将军ACIF/2016/020的区域ValI+D学生。RP要感谢EwaK.Paluch教授的资助。我们感谢艾丽西亚·马丁内斯-罗梅罗博士(CIPF细胞测量服务)对IN细胞分析仪平台的帮助。
Access restricted. Please log in or start a trial to view this content.
材料
| Name | Company | Catalog Number | Comments |
| 3,5-diethoxycarboxynl-1,4-dihydrocollidine diet (DDC) | TestDiet | 1810704 | Modified LabDiet mouse diet 5015 with 0.1% DDC |
| Alexa Fluor 488 donkey anti-goat IgG (H+L) | Invitrogen | A11055 | Dilution 1:500 |
| Bovine Serum Albumin | Sigma-Aldrich | A7906 | |
| Cryostat Leica CM1850 UV | Leica biosystems | CM1850 UV | Tissue sectioning |
| Fluorescent Mounting medium | Dako | S3023 | |
| GraphPad Prism | GraphPad Software | Prism 8 | Statistical software for graphing data |
| Hoechst 33342 | Sigma-Aldrich | B2261 | Final concentration 5 µg/mL |
| IN Cell Analyzer 1000 | GE Healthcare Bio-Sciences Corp | High-Content Cellular Imaging and Analysis System | |
| MATLAB | MathWorks | R2019a | Data analytics software for automated analysis of nuclear ploidy |
| Microscope coverslides | VWR International | 630-2864 | Size of 24 x 60 mm |
| Microsoft Office Excel | Microsoft | Speadsheet software | |
| OCT Tissue Tek | Pascual y Furió | 4583 | |
| Paraformaldehyde | Panreac AppliChem | 141451.121 | |
| Pen for immunostaining | Sigma-Aldrich | Z377821-1EA | 5mm tip width |
| Polysine Microscope Slides | VWR International | 631-0107 | |
| Rabbit polyclonal Anti-HNF4α | Thermo Fisher Scientific | PA5-79380 | Dilution 1:250 (alternative) |
| Rabit polyclonal Anti-HNF4α | Santa Cruz Biotechnology | sc-6556 | Dilution 1:200 (antibody used in the study) |
| Tween 20 | Sigma-Aldrich | P5927 |
参考文献
- Gentric, G., Desdouets, C. Polyploidization in liver tissue. American Journal of Pathology. 184 (2), 322-331 (2014).
- Duncan, A. W., et al. The ploidy conveyor of mature hepatocytes as a source of genetic variation. Nature. 467 (7316), 707-710 (2010).
- Gentric, G., Desdouets, C. Liver polyploidy: Dr Jekyll or Mr Hide? Oncotarget. 6 (11), 8430-8431 (2015).
- Wilkinson, P. D., et al. The Polyploid State Restricts Hepatocyte Proliferation and Liver Regeneration in Mice. Hepatology. 69 (3), 1242-1258 (2019).
- Wilkinson, P. D., et al. Polyploid Hepatocytes Facilitate Adaptation and Regeneration to Chronic Liver Injury. The American Journal of Pathology. 189 (6), 1241-1255 (2019).
- Zhang, S., et al. The Polyploid State Plays a Tumor-Suppressive Role in the Liver. Developmental Cell. 44 (4), 447-459 (2018).
- Chao, H. W., et al. Circadian clock regulates hepatic polyploidy by modulating Mkp1-Erk1/2 signaling pathway. Nature Communications. 8 (1), 2238(2017).
- Celton-Morizur, S., Merlen, G., Couton, D., Margall-Ducos, G., Desdouets, C. The insulin/Akt pathway controls a specific cell division program that leads to generation of binucleated tetraploid liver cells in rodents. Journal of Clinical Investigation. 119 (7), 1880-1887 (2009).
- Wang, M. J., Chen, F., Lau, J. T. Y., Hu, Y. P. Hepatocyte polyploidization and its association with pathophysiological processes. Cell Death & Disease. 8 (5), e2805(2017).
- Gentric, G., et al. Oxidative stress promotes pathologic polyploidization in nonalcoholic fatty liver disease. Journal of Clinical Investigation. 125 (3), 981-992 (2015).
- Toyoda, H. Changes to hepatocyte ploidy and binuclearity profiles during human chronic viral hepatitis. Gut. 54 (2), 297-302 (2005).
- Miyaoka, Y., et al. Hypertrophy and Unconventional Cell Division of Hepatocytes Underlie Liver Regeneration. Current Biology. 22 (13), 1166-1175 (2012).
- Bou-Nader, M., et al. Polyploidy spectrum: a new marker in HCC classification. Gut. , (2019).
- Danielsen, H., Lindmo, T., Reith, A. A method for determining ploidy distributions in liver tissue by stereological analysis of nuclear size calibrated by flow cytometric DNA analysis. Cytometry. 7 (5), 475-480 (1986).
- Guidotti, J. E., et al. Liver Cell Polyploidization: A Pivotal Role for Binuclear Hepatocytes. Journal of Biological Chemistry. 278 (21), 19095-19101 (2003).
- Severin, E., Meier, E. M., Willers, R. Flow cytometric analysis of mouse hepatocyte ploidy - I. Preparative and mathematical protocol. Cell and Tissue Research. 238 (3), 643-647 (1984).
- Manzano-Núñez, F., et al. Insulin resistance disrupts epithelial repair and niche-progenitor Fgf signaling during chronic liver injury. PLoS Biology. 17 (1), e2006972(2019).
- Morales-Navarrete, H., et al. A versatile pipeline for the multi-scale digital reconstruction and quantitative analysis of 3D tissue architecture. eLife. 4, e11214(2015).
- Baratta, J. L., et al. Cellular organization of normal mouse liver: A histological, quantitative immunocytochemical, and fine structural analysis. Histochemistry and Cell Biology. 131 (6), 713-726 (2009).
- Pandit, S. K., et al. E2F8 is essential for polyploidization in mammalian cells. Nature Cell Biology. 14 (11), 1181-1191 (2012).
- Vinogradov, A. E., Anatskaya, O. V., Kudryavtsev, B. N. Relationship of hepatocyte ploidy levels with body size and growth rate in mammals. Genome. 44 (3), 350-360 (2001).
- Tanami, S., et al. Dynamic zonation of liver polyploidy. Cell and Tissue Research. 368 (2), 405-410 (2017).
- Kudryavtsev, B. N., Kudryavtseva, M. V., Sakuta, G. A., Stein, G. I. Human hepatocyte polyploidization kinetics in the course of life cycle. Virchows Archiv B Cell Pathology Including Molecular Pathology. 64 (1), 387-393 (1993).
- Gentric, G., Celton-Morizur, S., Desdouets, C. Polyploidy and liver proliferation. Clinics and Research in Hepatology and Gastroenterology. 36 (1), 29-34 (2012).
- Uhlén, M., et al. Tissue-based map of the human proteome. Science. 347 (6220), 1260419(2015).
Access restricted. Please log in or start a trial to view this content.
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。