需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
应用活细胞成像和低温电子断层扫描解决军团菌肺瘤点/Icm分泌系统的时空特征
摘要
细菌细胞成像是一种新兴的系统生物学方法,侧重于定义静态和动态过程,这些过程决定了大型大分子机器的功能。在这里,定量活细胞成像和低温电子断层扫描的集成用于研究军团菌肺瘤IV型分泌系统架构和功能。
摘要
军团菌的点/Icm分泌系统是一种复杂的IV型分泌系统(T4SS)纳米机器,在细菌极处进行定位,并调解蛋白质和DNA基质向靶细胞的输送,这一过程通常需要细胞到细胞直接接触。我们最近通过低温电子断层扫描(cryo-ET)解决了Dot/Icm仪器的结构,并表明它形成了一个细胞包络跨度通道,连接到细胞质复合体。应用两种补充方法,保持标本的原生结构,荧光显微镜在活细胞和低温-ET中,允许蛋白质的原位可视化和同化每个机器部件相对于其他点/Icm子单位的生产时间。为了研究极性定位的要求,并描述与T4SS机器生物成因相关的动态特征,我们将编码超级文件夹绿色荧光蛋白的基因融合到Dot/Icm ATPase基因的原生位置。以下方法集成了活细胞和低温-ET的定量荧光显微镜,以量化这些蛋白质在完整细菌细胞中的极性定位、动力学和结构。应用这些方法研究军团菌肺炎T4SS有助于描述Dot/Icm系统的功能,并可用于研究利用T4SS或其他类型的细菌分泌复合物的各种细菌病原体。
引言
军团菌肺杆菌(L.肺炎杆菌),军团病的病因,栖息在淡水水库,细菌通过感染和复制水生自由游泳原生动物传播。L. 肺尘埃球菌在吸入饮用水源中的气溶胶细菌时,导致人类疾病爆发。在受感染的细胞中,宿主通路的颠覆使L.肺炎杆菌能够延缓其所在的真空细胞的内皮成熟,并促进支持细菌复制的细胞舱的生物生成。这个过程是由一个专门的细菌类型IVB分泌系统(T4BSS)称为Dot/Icm及其的300多个"效应器"蛋白质的品种,在感染期间被转移到宿主细胞群素,以促进细胞功能1,2,3,4,5的操纵。缺乏功能点/Icm仪器的突变体不能将效应器输送到宿主细胞索中,在细胞内复制方面存在缺陷,在疾病6、7的动物模型中具有avirule。
许多细菌物种已经开发出了感染过程所需的极其复杂和动态的多组分机。其他T4BSS,如Dot/Icm系统也是必不可少的细胞内复制细菌病原体,如科西拉布鲁内蒂和里基西拉格丽利。虽然T4BSS与原型型IVA系统具有进化关系,后者调解DNA转移,并能提供有限的有效蛋白,但Dot/Icm系统拥有几乎两倍的机器部件,并提供各种各样的效应器。据推测,这种组件数量的扩大使Dot/Icm设备能够轻松容纳和集成新的效应器8,9。
我们最近使用低温电子断层扫描(cryo-ET)就地解决了Dot/Icm仪器的结构,并表明它形成了一个细胞包络跨度通道,连接到细胞质复合体。进一步分析表明,通过与细胞体ATPase DotO的相互作用,圆细胞状ATPase DotB与L.肺尘埃球细胞极的点/Icm系统有关。我们发现,DotB在大多数细菌细胞中显示一个循环球状运动,这表明这种ATPase存在于动态的细胞群中,但也与极性点/Icm复合物有关联。此外,DotO 形成与内膜复合体相关的 DotO 二分器的 hexamic 组件,DotB hexamer 连接到此细胞质复合物的基座。DotB-DotO 能量复合体的组装创建了一个细胞质通道,通过 T4SS(图 1)10引导基材的易位。
尽管最近取得了这些进展,但关于Dot/Icm系统如何发挥作用以及每种蛋白质如何组装形成一个活性装置8知之甚少。发现Dot/Icm T4SS的调节回路对于理解宿主-病原体相互作用的分子机制至关重要。因此,我们讨论如何使用活细胞显微镜和低温-ET来检测和表征使用超级文件夹GFP(sfGFP)标记的基本L.肺活血杆菌/Icm系统组件。使用定量荧光显微镜,DotB 的极定位将在野生类型背景中定义,或者当 IV 型系统被删除时。延时显微镜将用于量化点/Icm细胞学的定位和动力学差异。
与其他体外系统相比,实时成像和低温ET等两种互补方法的组合应用提供了优势。这两种方法均以完整的细胞执行,并保护T4BSS的自然环境,从而最大限度地减少样品制备过程中原生结构的干扰。由于蛋白质的过度表达可能会损害分泌器的分泌器的分泌器,sfGFP融合通过异位交换返回到军团菌染色体,以便每个融合在单拷贝中编码,并且表达由内源促进者驱动。色谱编码聚变的可视化能够量化在规定的时间点表达的蛋白质的确切水平。Cryo-ET 在确定分泌系统结构方面也有许多优点。最显著的优点是低温-ET样品由冷冻完整细胞组成,在细菌细胞结构中保留原生复合物。因此,低温-ET可能比生化纯化方法更可取,后者提取膜复合物,并可能从核心设备中去除外周蛋白或修改整体结构。此外,用笨重的蛋白质(如 sfGFP)标记感兴趣的蛋白质,可添加低温-ET 可检测的质量,并有助于将 Dot/Icm 仪器的不同子复合物映射到低温-ET 获得的结构上。
这种方法是一个强大的工具,用于发现在细菌细胞膜中组装的多分子复合物的结构信息。解释使用这些技术阐明的结构将有助于现场了解 T4BSS 组件的工作原理、为什么函数需要这么多组件、组件如何在更复杂范围内交互以及这些组件的功能子组件执行。
Access restricted. Please log in or start a trial to view this content.
研究方案
注:所有涉及肺尘埃球菌生长、操作和成像的程序都应按照当地准则在生物安全2级实验室进行。
1. 使用全位交换和双选择策略将sfGFP插入L.肺炎嗜血杆菌染色体(图2,图3)
- 克隆成基因置换载体pSR47S11以下序列:1000bp上游感兴趣的站点,然后是sfGFP序列,然后是1000bp下游感兴趣的站点(图2)。sfGFP 序列应放置在 N 端或 C 端的框架中,其链接器包含四到八个氨基酸。将生成的病媒转换为大肠杆菌DH5_pir。后来,单菌菌提取物(CYE)agar12的单菌体L.肺炎杆菌(受体)含有100μg/mL链霉素,在37°C下生长5天(图3)。
- CYE-agar-链霉素上的条纹L.肺炎杆菌,在37°C(重斑)10下生长2天。带 25 μg/mL 氯霉素的 LB Agar 上用 pRK600 助种质粒(帮助器)13转化了大肠杆菌 DH5+。在含有50微克/mL卡那霉素的LBAgar上,将大肠杆菌DH5_pir(供体)划破。
- 进行三亲交配:在未选择的情况下,将三个菌株的斑块覆盖在CYE加盘上,在37°C下孵育4~8小时,从而孵育一个帮手、捐赠者的蚁群和受体。当负对照在相同时间段内孵育帮助者-受体菌株组合和捐赠者-受体菌株组合时。
- 在含有100微克/mL链球菌霉素的CYE加上反应500μL中重新悬浮交配反应,并在37°C下生长5天。在含有100微克/mL链霉素的CYEAgar上产生的克隆的条纹四,在37°C下生长5天。
- 在CYE Agar上克隆16个,含有5%蔗糖和100微克/mL链霉素,在37°C下生长5天。然后,这些克隆的条纹32在CYE Agar上含有100μg/mL链球菌霉素,在含有100微克/mL链球菌霉素和10μg/mL假霉素的CYE加上,在37°C下生长5天。
2. 将sfGFP集成到L.肺炎染色体中的克隆人分离
- 对CYE-agar-链霉素板上的卡那霉素敏感的条纹克隆,确认sfGFP插入具有菌落PCR的染色体中。使用与 sfGFP 基因和感兴趣的染色体区域互补的底漆来放大插入结。
- 将 10 μM 底漆溶液中的每个溶液和一个聚位混合 0.5 μL,最终体积为 12.5 μL,在 95 °C 下变性 10 分钟。在冰上冷却 10 分钟,加入 12.5 μL 的 2x PCR 主混合溶液,并执行 PCR 分析。
- 在37°C下,在CYE-agar-链霉素板上生长2天的孤立菌落的重斑块。通过抗GFP抗体的免疫膨胀,检查sfGFP融合的表达水平和稳定性。
3. L. 肺杆菌的活细胞成像与荧光标记点/Icm成分
- 准备琼脂糖垫
- 在水中生产约 30 mL 的 1% 低熔脂糖溶液。微波炉在玻璃瓶中约90s,偶尔旋转,直到琼脂完全溶解。
- 将两张 22 x 22 x 0.15 mm3玻璃幻灯片放在 25 x 75 x 1.1 mm3玻璃幻灯片的边缘,一张放在另一个玻璃幻灯片的顶部。将两个 22 x 22 x 0.15 mm3玻璃幻灯片堆叠在另一个边缘。
- 约1 mL的熔融琼脂放入两个上部玻璃幻灯片之间的中心滑移,然后将另一张 25 x 75 x 1.1 mm3滑道放在熔融阿加罗斯的顶部。尽量避免形成气泡。在 4 °C 下冷却幻灯片 15 分钟。
- 使用手术刀或剃刀刀片,轻轻地将垫子切成小方块,±5 x 5 mm2。在 25 x 75 x 1.1 mm3玻璃滑轨上固定双面胶粘剂 17 x 28 x 0.25 mm3框架,并在滑轨上放置多个垫。
- 图像采集
注:以下步骤用于由 SlideBook 6.0 控制并配备固态照明器、CCD 单色摄像机和 100 倍客观透镜(1.4 个数值光圈)的显微镜。如果需要,请使用具有适当硬件和软件配置的替代显微镜设备,这些配置可根据协议设置进行自定义。- 将一块重片的L.肺炎杆菌溶解在1 mL的ddH2O,涡流和移液2+3μL的稀释到垫上。将 50 x 24 x 0.15 mm3盖玻片轻轻放在粘合框架上。
- 在捕获窗口中将 ND 调整为 180。将分箱调整到2⁄2,并使用488 nm通道在500~1000 ms之间暴露样品。
4. 极性定位和点/Icm成分动力学的定量
注: 以下步骤设计用于使用 2 x 2 分箱获得/像素为 0.129 μm 的图像。
- sfGFP融合蛋白极性量化 (图 5)
- 调整图像对比度,使细菌清晰可见。使用区域工具放置一个 0.25 x 1.3 μm2矩形,从极点开始并延伸到细胞质。矩形必须精确地保持在细菌边界内。
- 标记至少 200 种细菌,并使用"区域"遮罩按钮向感兴趣的区域创建蒙版。在蒙版统计和蒙版范围下,选择对象。然后,在"特征和强度"下,标记平均强度和方差。
- 导出数据并计算每个细菌的极性分数,作为方差与平均强度之间的比率。
- 对于高通量应用,使用相位目标和适当的冷凝器设置来获取具有相位和 488 nm 通道的图像。确保选择细菌完全分离的视场。
- 调整图像的相通道对比度,使细菌清晰可见。打开双通道图像,启动"创建分段蒙版"窗口并将通道更改为相位。
- 使用"定义对象"按钮调整适当的阈值并删除小对象。在"优化蒙版"下,选择"删除边缘对象",然后分离彼此相邻的细菌蒙版。
- 计算每个单元的 488 通道中信号的极性分数,如前所述步骤 4.1.2_4.1.3 中所述。
- sfGFP融合蛋白动力学的定量(图6)
注:按照第 3 节中的说明准备图像采集示例。动态定义为强度随时间的变化,以下步骤设计用于短成像周期(即几分钟)。如果需要更长的成像周期,添加到垫上适当的补充剂。- 在"图像捕获"窗口中,在间隔框中标记"延时",在间隔框中输入间隔时间,并在"时间点"框中输入2。获得连续两张L.肺炎嗜血杆菌的图像,表达感兴趣的荧光蛋白。
- 调整图像对比度,直到单元格清晰可见。请按照图 6A和下面的说明放置三个不同的蒙版。使用区域工具将 0.25 x 0.25 μm 正方形放置在至少 400 个单元格的中间。
- 使用"区域"遮罩按钮创建感兴趣方块的蒙版(蒙版 1)。创建新的空掩码(掩码 2),并使用像素工具或多边形工具标记至少 25 个随机单元格的整个单元格区域,用于计算荧光漂白。创建新的空掩码(蒙版 3),并使用大型画笔工具标记单元格之间的区域,这些区域将用于背景减法。
- 在蒙版统计和蒙版范围下,选择蒙版 1的对象和蒙版 2。然后,在"特征和强度"下,选择"平均强度"并导出两个蒙版的数据。对于蒙版 3,导出整个蒙版的平均强度。
- 使用以下公式计算掩码 1 中每个对象的荧光强度变化:

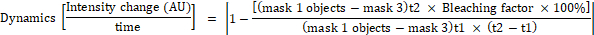
其中掩码1是放置在细胞中心的0.25 μm × 0.25 μm平方,掩码2覆盖整个细胞,掩码3是细胞之间的背景,t1是第一时间点的平均强度,t2是第二时间点的平均强度。
5. 使用冷冻-ET 检测 sfGFP 质量密度
- 样品制备、数据收集和重建
- 在37°C下,在CYE-agar-链霉素板上生长48小时,在CYE-agar-链霉素板上生长一个重片L.肺炎杆菌,表达荧光标记的点/Icm蛋白。重新挂起 ddH2O 到 OD600 ±0.7 的单元格。将5 μL胶体金颗粒(BSA 跟踪器,10 nm)添加到细胞悬浮液的20μL。
- 移液器5 μL的细胞混合物到新鲜发光释放的孔碳网格(R 2/1在Cu 200网格上),让站在1分钟,用滤纸和冻结在液体乙醚使用重力驱动的柱塞仪器,如前所述的14,15。
- 使用配备场发射枪、能量过滤器、Volta 相位板和直接检测装置的 300 kV 透射电子显微镜对冷冻水合试样进行成像。以 26,000 倍和 42,000 倍的放大倍率收集单轴倾斜系列,从而在试样级别分别达到 5.4 ×/像素或 3.4 ×/像素的像素大小。
- 使用地形包 SerialEM 以 ±0 μm 散焦收集图像堆栈,倾斜角度范围介于 -60° 和 +60° 之间,增量为 3°,累积剂量为 +60 e-/+2,16。使用 MotionCor217将每个堆栈中的剂量分馏电影图像对齐。使用 TOMOAUTO14组装漂移校正的堆栈。
- 通过 IMOD 标记相关对齐18对齐漂移校正堆栈。使用SIRT方法19重建图,用于分割和直接图像分析和WBP方法20。
- sfGFP 融合样品的子光度图分析
- 使用地形包I3(0.9.9.3)进行子光度分析14、21、22。
注: 对齐过程是迭代的,每次迭代由生成引用和分类蒙版的三个部分组成,子光度图对齐和分类,类平均值相互对齐。 - 使用 4 x 4 x 4 装箱的子色调图进行初始对齐。合并属于类平均值的粒子,并显示与 sfGFP 对应的电子密度。使用 sfGFP 聚变对粒子进行排序后,使用 2 x 2 x 2 装箱的亚圆图对感兴趣区域(如 Dot/Icm 细胞质 ATPase 复合物)进行聚焦对齐,以获得高分辨率结构。
- 使用地形包I3(0.9.9.3)进行子光度分析14、21、22。
Access restricted. Please log in or start a trial to view this content.
结果
在两个步骤中采用双选择进行同源重组,用于构造 sfGFP 的定义插入。在第一步中,进行了三亲交配,其中从大肠杆菌帮助器菌株MT616的pRK600偶联质质质体(IncP质粒)被调动到捐赠者大肠杆菌菌株与自杀载体pSR47S包含sfGFP基因的两个同源区域,转移奥里特的起源和杆菌子选择基因囊肿。接下来,从pRK600的合并系统协助将pSR47S衍生物动员到L.肺炎杆菌(Lp01,
Access restricted. Please log in or start a trial to view this content.
讨论
阐明细菌分泌系统的功能是全面了解宿主-病原体相互作用的关键。分泌系统是复杂的机器,可以将效应剂蛋白质注入宿主细胞,在某些情况下,它促进建立支持细菌复制的亚细胞利基。上述方法为研究呼吸系统病原体军团菌肺杆菌的Dot/Icm分泌系统提供了重要的新工具,为有效器易位机制提供了线索,对其致病性至关重要。这种方法可以通过利用活细菌细胞成像和结构研究来解剖其分子水?...
Access restricted. Please log in or start a trial to view this content.
披露声明
作者没有什么可透露的。
致谢
华盛顿特区和C.R.R.得到NIH的支持(R37AI041699和R21AI130671)。D.P.、B.H.和J.L得到国家卫生研究院(R01AI087946和R01GM107629)的支持。
Access restricted. Please log in or start a trial to view this content.
材料
| Name | Company | Catalog Number | Comments |
| 10 nm colloidal gold particles | Aurion | 25486 | |
| 100x Plan Apo objective (1.4 NA) | Nikon | ||
| ACES | Sigma-Aldrich | A9758 | |
| Activated charcoal | Sigma-Aldrich | C5510 | |
| Agaroze GPG/LMP, low melt | American bioanalytical | AB00981 | |
| Bacto dehydrated agar | BD | 214010 | |
| CoolSNAP EZ 20 MHz digital monochrome camera | Photometrics | ||
| Gene Frame, 1.7x2.8 cm, 125 µL | Fisher Scientific | AB-0578 | |
| Holey Carbon grid R 2/1 Cu 200 mesh | Quantifoil | Q225-CR1 | |
| Iron(III) nitrate nonahydrate | Sigma-Aldrich | 216828 | |
| K2 Summit camera for cryo-EM | GATAN | ||
| L-Cysteine | Sigma-Aldrich | C7352 | |
| Microscope cover slides 22x22 mm | Fisher Scientific | 12-542B | |
| Microscope cover slides 24x50 mm | Fisher Scientific | 12-545K | |
| Microscope slides 25x75x1 mm | Globe Scientific | 1380 | |
| SlideBook 6.0 | Intelligent Imaging Innovations | ||
| Spectra X light engine | Lumencor | ||
| Taq 2X Master Mix | New England BioLabs | M0270 | |
| Titan Krios | Thermo Fisher Scientific | ||
| Yeast Extract | BD | 212750 |
参考文献
- Franco, I. S., Shuman, H. A., Charpentier, X. The perplexing functions and surprising origins of Legionella pneumophila type IV secretion effectors. Cellular Microbiology. 11, 1435-1443 (2009).
- Burstein, D., et al. Genome-scale identification of Legionella pneumophila effectors using a machine learning approach. PLOS Pathogens. 5, 1000508(2009).
- Ninio, S., Roy, C. R. Effector proteins translocated by Legionella pneumophila: strength in numbers. Trends in Microbiology. 15, 372-380 (2007).
- Vogel, J. P., Andrews, H. L., Wong, S. K., Isberg, R. R. Conjugative transfer by the virulence system of Legionella pneumophila. Science. 279, 873-876 (1998).
- Isberg, R. R., O'Connor, T. J., Heidtman, M. The Legionella pneumophila replication vacuole: making a cosy niche inside host cells. Nature Reviews Microbiology. 7, 13-24 (2009).
- Roy, C. R., Berger, K. H., Isberg, R. R. Legionella pneumophila DotA protein is required for early phagosome trafficking decisions that occur within min of bacterial uptake. Molecular Microbiology. 28, 663-674 (1998).
- Archer, K. A., Roy, C. R. MyD88-Dependent Responses Involving Toll-Like Receptor 2 Are Important for Protection and Clearance of Legionella pneumophila in a Mouse Model of Legionnaires' Disease. Infection and Immunity. 74, 3325-3333 (2006).
- Nagai, H., Kubori, T. Type IVB Secretion Systems of Legionella and Other Gram-Negative Bacteria. Frontiers in Microbiology. 2, 136(2011).
- Kubori, T., Nagai, H. The Type IVB secretion system: an enigmatic chimera. Current Opinion in Microbiology. 29, 22-29 (2016).
- Chetrit, D., Hu, B., Christie, P. J., Roy, C. R., Liu, J. A unique cytoplasmic ATPase complex defines the Legionella pneumophila type IV secretion channel. Nature Microbiology. 3, 678-686 (2018).
- Merriam, J. J., Mathur, R., Maxfield-Boumil, R., Isberg, R. R. Analysis of the Legionella pneumophila fliI gene: intracellular growth of a defined mutant defective for flagellum biosynthesis. Infection and Immunity. 65, 2497-2501 (1997).
- Feeley, J. C., et al. Charcoal-yeast extract agar: primary isolation medium for Legionella pneumophila. Journal of Clinical Microbiology. 10, 437-441 (1979).
- Andrews, H. L., Vogel, J. P., Isberg, R. R. Identification of linked Legionella pneumophila genes essential for intracellular growth and evasion of the endocytic pathway. Infection and Immunity. 66, 950-958 (1998).
- Morado, D. R., Hu, B., Liu, J. Using Tomoauto: A Protocol for High-throughput Automated Cryo-electron Tomography. Journal of Visualized Experiments. (107), e53608(2016).
- Hu, B., Lara-Tejero, M., Kong, Q., Galan, J. E., Liu, J. In Situ Molecular Architecture of the Salmonella Type III Secretion Machine. Cell. 168, 1065-1074 (2017).
- Mastronarde, D. N. Automated electron microscope tomography using robust prediction of specimen movements. Journal of Structural Biology. 152, 36-51 (2005).
- Zheng, S. Q., et al. MotionCor2: anisotropic correction of beam-induced motion for improved cryo-electron microscopy. Nature Methods. 14, 331-332 (2017).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD. Journal of Structural Biology. 116, 71-76 (1996).
- Gilbert, P. Iterative methods for the three-dimensional reconstruction of an object from projections. Journal of Theoretical Biology. 36, 105-117 (1972).
- Radermacher, M. Weighted Back-projection Methods. Electron Tomography. , 245-273 (2007).
- Winkler, H., et al. Tomographic subvolume alignment and subvolume classification applied to myosin V and SIV envelope spikes. Journal of Structural Biology. 165, 64-77 (2009).
- Winkler, H., Taylor, K. A. Accurate marker-free alignment with simultaneous geometry determination and reconstruction of tilt series in electron tomography. Ultramicroscopy. 106, 240-254 (2006).
- Prevost, M. S., Waksman, G. X-ray crystal structures of the type IVb secretion system DotB ATPases. Protein Science. 27, 1464-1475 (2018).
- Miklos, G. L., Rubin, G. M. The role of the genome project in determining gene function: insights from model organisms. Cell. 86, 521-529 (1996).
- Reyrat, J. M., Pelicic, V., Gicquel, B., Rappuoli, R. Counterselectable markers: untapped tools for bacterial genetics and pathogenesis. Infection and Immunity. 66, 4011-4017 (1998).
- Yu, J. Single-Molecule Studies in Live Cells. Annual Review of Physical Chemistry. 67, 565-585 (2016).
- Stewart, P. L. Cryo-electron microscopy and cryo-electron tomography of nanoparticles. Wiley Interdisciplinary Reviews: Nanomedicine and Nanobiotechnology. 9, (2017).
Access restricted. Please log in or start a trial to view this content.
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。