Method Article
用于大规模表观遗传学研究的半自动ChIP-Seq程序
Erratum Notice
摘要
本文描述了使用ChIP液体处理平台对与特定组蛋白修饰(H3K27ac)相关的DNA区域的半自动,微量尺度的ChIP-Seq方案,然后使用标记进行文库制备。该程序包括出于质量和数量目的的对照评估,并且可以用于其他组蛋白修饰或转录因子。
摘要
染色质免疫沉淀后进行测序(ChIP-Seq)是一种强大且广泛使用的方法,用于分析与特定组蛋白修饰(如H3K27ac)相关的染色质DNA,以帮助鉴定顺式调节DNA元件。完成 ChIP-Seq 的手动过程是劳动密集型的,技术上具有挑战性,并且通常需要大细胞数(> 100,000 个细胞)。这里描述的方法有助于克服这些挑战。详细介绍了完整的半自动,微量规模的H3K27ac ChIP-Seq程序,包括细胞固定,染色质剪切,免疫沉淀和测序文库制备,用于细胞数输入小于100,000个细胞的48个样品。半自动平台减少了技术可变性,提高了信噪比,并大大减少了劳动力。因此,该系统可以通过减少反应量来降低成本,限制昂贵的试剂数量,如酶、磁珠、抗体和所需的手动操作时间。ChIP-Seq方法的这些改进非常适合以高度可重复的方式对细胞数量有限的临床样品进行大规模表观遗传学研究。
引言
ChIP-Seq测定法的广泛使用来确定与特定组蛋白修饰相关的DNA片段,部分原因是它能够识别顺式调节DNA元件,包括活性增强剂,启动子,消音剂,异染色质和其他1,2,3,4。识别整个基因组中的非编码调节区域已经显示出有价值的见解,可以更好地了解健康和疾病中的基因调控4。该实验室先前的工作已经使用ChIP-Seq来证明顺式调节元件可以在不同的细胞类型中发挥重要作用5。转录因子(TF)ChIP测定已被用于显示疾病相关风险的单核苷酸多态性6。
将ChIP-Seq用于人类临床样品具有挑战性,主要是由于细胞数量或所需组织样品的限制。因此,该领域一直在共同努力改进和微观尺度这些技术,因此,出现了几种分析方法,例如CUT&TAG5,7,8,9,10,11,12。该测定利用转座酶来标记和分离由特异性抗体9结合的基因组区域。该技术已经能够将细胞数量减少到1,000s,在某些情况下减少到单细胞,但是,由于使用活细胞的要求,该技术在转化研究和临床设置中的使用已经显示出局限性9,12。活细胞要求使得临床样品在逻辑上难以处理,如果不同时处理样品,可能会引入批处理效应。其他人已经优化了甲醛固定细胞的微尺度技术,包括ChIPmentation11的开发,它在这里以高通量的方式进行调整。使用固定细胞允许存储样品,直到收集和随后处理所有样品,以最大限度地减少批量效应。
在这里,描述了一种半自动微量级的ChIP-Seq测定,它减少了实验动手时间来分析组蛋白修饰10。半自动方法允许高通量 ChIP-Seq 检测,允许在短短 5 天内完全处理多达 48 个样品并准备好进行测序,使用 ChIP 液体处理程序,每个样品只需 10,000 个细胞。处理程序以自主方式完成免疫沉淀(IP)和随后的洗涤,这有助于减少样品之间的变异性。半自动方法将48个样品的手动操作时间降低了15小时以上,并降低了技术变异性,使得能够以可重复和快速的方式对原代或培养细胞进行大规模表观遗传学研究。该协议解释了高质量ChIP-Seq从头到尾的过程。如果特定机器不可用,该协议仍将是手动设置和故障排除 ChIP-Seq 实验的有用资源。
该测定是用三种不同的原代人免疫细胞类型和一种培养的细胞系(HUT78 – ATCC:TIB-161)进行的。为清楚起见,该方案分为七个部分:细胞固定,通过超声处理剪切染色质,自动染色质免疫沉淀,通过DNA片段标记制备文库,文库扩增,文库纯化,然后DNA定量。有关缓冲液配方,请参阅 补充表1。
研究方案
拉霍亚过敏和免疫学研究所(LJI;IRB 协议编号SGE-121-0714)批准了这项研究。招募了健康志愿者,并在书面知情同意后提供白细胞分离术样本。
1. 细胞固定
- 将细胞悬浮液浓度降至1至2 x 106 个细胞/ mL,在15 mL管(<10 mL悬浮液)或50 mL管(10-30 mL细胞)中完整的细胞培养基。如果<1×106 个细胞,请在1.5 mL管中使用0.5mL培养基。
- 轻轻涡旋细胞悬浮液,滴加10x细胞固定缓冲液(1:10;vol:vol),并在室温(RT)下低速旋转10分钟。
- 通过轻轻涡旋停止反应,并以1:20(vol:vol)的比例加入2.5M甘氨酸。将管倒置几次,并在冰上孵育至少5分钟。
- 在4°C或冰上进行其余步骤。在4°C下以 800×g 旋转管5分钟并丢弃上清液。
- 用5 mL冰冷的1x PBS轻轻重悬沉淀,并在冰上孵育2分钟。
- 用1 mL冰冷的1x PBS重复1.4和1.5,并将样品转移到预冷的1.5 mL管中(标记为长期储存)。如果适用,建议制备等分试样。
- 在4°C下以1,200×g旋转管,并在不干扰细胞沉淀的情况下除去尽可能多的上清液。 在液氮中快速冷冻颗粒。储存在-80°C。
注意:处理液氮时采取适当的保护措施。
2. 染色质剪切
注意:该方案针对在0.65mL低结合管中具有0.3至3×106 个细胞的沉淀物剪切进行了优化。
- 从-80°C取出带有冷冻细胞沉淀的样品管并储存在干冰上,以避免在加入裂解缓冲液之前沉淀的任何解冻。此步骤至关重要。
- 向沉淀中加入70μL新鲜的RT完全裂解缓冲液,并在室温下保持1分钟。
- 在不引入任何气泡的情况下重悬沉沉淀1分钟,然后在室温下孵育细胞悬浮液1分钟,然后将样品放在冰上。
- 将重悬的沉淀转移到0.65mL低结合管中并保持在冰上。
注意:为了获得可重复的超声处理,在对样品进行超声处理之前,仅用空白管运行3-6个周期来预热超声仪。 - 将样品放入超声仪的管架中,并用装满70μL水的平衡管填充任何间隙。在开始超声处理之前,将样品留在水浴中约1分钟。
- 执行 x 个周期 (取决于细胞类型)的超声处理,每个周期 16 s ON / 32 s OFF。
注意:此步骤需要验证实验来确定有效超声处理的最佳循环数。 - 每3个循环后,从超声仪中取出样品,轻轻涡旋并脉冲旋转管子,然后将其放回支架中。确保管外侧没有小液滴,因为这会导致气泡形成。
- 完成必要的循环后,在4°C下以>14,000×g旋转样品15分钟。 将上清液转移到新的,预冷却的,低结合的0.65mL低结合管中并保持在冰上。
- 对部分超声处理样品进行脱杂交联,以检查超声处理效率。
- 将1-7μL上清液(相当于约250ng剪切染色质)转移到0.2mL PCR管中,并在室温下用短期裂解缓冲液使体积达到10μL。
- 加入1μL RNase A,在37°C下以800rpm孵育30分钟,然后加入1μL蛋白酶K。
- 在55°C下孵育2小时,以1,000转/分振荡。
- 使用荧光定量测定法10 除去2μL脱交联样品进行DNA定量(不建议使用分光光度计,因为肥皂和降解的蛋白质会在结果中产生偏倚)。
- 将剩余的样品在1.2%琼脂糖凝胶上以70 V.用核酸染料(1:20,000)染色1小时,并使用UV透射仪读取凝胶。
- 制备染色质储备等分试样进行储存(用完全裂解缓冲液将样品稀释至20μL中的25ng / μL)。将所有剪切的染色质储存在-80°C。
3. 用于组蛋白修饰的自动 ChIP-Seq
注意:此协议设计为在 ChIP 液体处理程序上运行。虽然系统可以使用定制的缓冲液,但所有缓冲液都随 ChIP 套件一起提供。本节中使用的带有8个管的ChIP条是特定于ChIP液体处理器的。
- 将16个样品等分试样与500ng剪切的染色质从-80°C转移到20μL中,并将它们放在冰上以缓慢解冻染色质。一旦完全解冻,短暂涡旋和脉冲旋转。
- 染色质的制备
- 将100μL补充有1x蛋白酶抑制剂和20mM丁酸钠(完全tC1缓冲液)的tC1缓冲液移入两个ChIP 8管条中。
- 将每个染色质样品的20μL转移到含有100μL完整tC1缓冲液的ChIP 8管条的适当管中。通过将80μL完整的tC1缓冲液加入染色质管中来洗涤染色质管,然后将染色质管转移回ChIP 8管条的相应管中,最终体积为200μL。
- 抗体的制备
- 计算抗体的体积,使得每个管中含有0.5μg抗体。
抗体体积 = (样本数 x 每个反应的抗体) / 抗体浓度 - 将计算出的抗体量加入500μLtBW1缓冲液中。快速涡旋和脉冲旋转。
- 将70μLtBW1移液到两个ChIP 8管条中,并将30μL抗体+ tBW1加入每个管中。这将使每个试管的总体积达到100μL。
- 计算抗体的体积,使得每个管中含有0.5μg抗体。
- 磁珠的制备
- 彻底涡旋蛋白质A珠溶液。对于0.5μg抗体,将5μL微珠移液到一组新的ChIP 8管条中并脉冲旋转。
- 用标记的空 ChIP 8 管条填充 ChIP 液体处理装置的最后一行。
- 遵循 ChIP-16-IPure-200D 程序规范,将所有条带放置在 ChIP 液体处理器机器中。将缓冲液添加到正确的位置,但使用tW4而不是tE1缓冲液。
注意:组织这一天,以便 ChIP 液体处理程序将在一夜之间执行 ChIP。该程序将运行约16小时,用于16个样品。这标志着第1天的结束。
4. 文库适配器转座酶集成,用于文库制备
- 将热混混器预设为 37 °C 和 500 rpm。冷却磁铁,在冰上放置0.2 mL管条。
- 对于16个样品,在冰上制备440μL标签缓冲液。将 53 μL 移液到单个新的 8 管条中,并保持在冰上。
- 在新的0.2 mL 8管试纸中,加入220μL冷tC1缓冲液并保持在冰上。8个条形管可以保持这个体积,并且仍然可以封盖。
- 从ChIP液体处理机(第12排)中取出"IP样品"试纸管,并在脉冲旋转之前盖上管子。使用磁铁捕获8管条2分钟的珠子,并小心地除去上清液。
- 将25μL标记缓冲液转移到具有多通道的磁珠中,从磁体中取出,然后轻轻混合,直到磁珠均匀(移液器设置为20μL时上下约5次)。
- 盖上管子,放入预热的热混合器中,孵育3分钟。增加时间会降低文库制备的效率。
- 将试管转移到冷却的金属架上,并向每个样品中加入100μL冷却的tC1缓冲液。将多通道移液器设置为80μL并混合样品,直到珠子均匀,停止标记反应。
- 将样品放回ChIP液体处理机中,然后继续 Washing_for_IP reacts_16_Ipure的洗涤程序。确保用tC1缓冲液洗涤两次,用tW4洗涤两次。洗脱应按照程序布局的标记完成,并带有缓冲液tE1。
- DNA的脱杂交联
- 除去ChIP液体处理程序最后一行中的ChIP 8管条,并向每个样品中加入2μL RNase A。
- 盖上试管,脉冲旋转,用多通道移液器轻轻混合珠子,直到混合物均匀,然后重新盖上试管。
- 将样品在热混合器中在37°C和900rpm下孵育30分钟。
- 从热混合器中取出样品,加入2μL蛋白酶K.加入后遵循与4.9.2相同的步骤。
- 将样品在热混合器中孵育4小时,在55°C和1,250rpm下,然后在65°C下以1,000rpm过夜。
注意:这是第 2 天的结束。
5. 标记DNA片段纯化
- 用适当的样品编号标记16个1.5 mL试管,并从DNA纯化试剂盒中加入400μL DNA结合缓冲液。
- 从热混合器中取出8管条,然后脉冲旋转条带,以确保保留任何蒸发的产品。将条带放在8条磁铁上以捕获珠子。
- 将100μL脱杂环联的DNA转移到每个1.5mL管中。将100μLDNA结合缓冲液加入8管条中以洗涤磁珠,然后转移到适当的1.5mL管中。
- 涡旋约10秒,脉冲旋转1.5 mL管。
- 在柱中加入含有DNA结合缓冲液和ChIP样品的600μL。
- 在10,000 x g下旋转样品20 s,然后用流出物重新加载色谱柱。在相同的条件下再次旋转并丢弃流出。
- 用200μL洗涤缓冲液洗涤柱两次(与前一步相同的离心),并丢弃流出物。
- 通过在12,000×g下离心2分钟来干燥色谱柱。
- 将色谱柱转移到新的1.5 mL收集管中,并将9μL温TE缓冲液(预热至55°C)直接加入柱基质中。让色谱柱孵育1分钟,然后在10,000×g下离心1分钟。
- 将9μL洗脱液转移到一组适当的新8管条中。
- 像以前一样用8μLTE缓冲液再次完成洗脱。将洗脱液转移到适当的8管条中(每个样品的最终体积17μL)并保持在冰上。
6. 纯化样品的扩增和尺寸选择
- 以下步骤使用qPCR确定最佳扩增所需的循环次数(CtD – Ct测定)
- 通过将CtD混合缓冲液的内容物乘以样品数量来为所有样品准备CtD混合物。
- 将3.6μLCtD混合物分配到qPCR板中,并加入1.4μL标记的DNA样品(约占总体积的10%)。执行以下 qPCR:98 °C 持续 3 分钟,72 °C 持续 5 分钟,98 °C 持续 30 秒,在 98 °C 下循环 26 次循环 10 秒,63 °C 持续 30 秒,72 °C 循环 30 秒。
- 通过将 AMP 混合缓冲液的内容量乘以样本数量,为所有样品准备 Amp 混音。将14μL标记的DNA分配到PCR板的单独孔中,然后向每个样品中加入2.5μL两个测序指数引物(25μM)(最终反应体积为50μL)。
- 通过多通道移液器混合样品,并以适当的循环次数执行CtD中使用的扩增程序。
注意:这是一个很好的停止点,因为扩增的样品可以在-20°C下储存几周。但是,纯化可以在同一天完成。对于48个样品,步骤3 - 6.5与另外两个单独的批次一起完成,然后在一个批次中扩增,如下所述。 - 如下所述对标记的DNA进行扩增后、大小选择和定量。这通常可以用48个样品完成(可以根据需要用更少的样品完成)。
- 将90μL顺磁珠(1:1.8比例)加入每个孔中,混合,并使其在室温下孵育2分钟。
- 使用板磁铁捕获珠子并丢弃上清液。用200μL新鲜80%乙醇洗涤珠子3次,而不会破坏珠子沉淀。
- 最终洗涤后,用20μL吸头去除任何多余的乙醇,并将珠子干燥10分钟或直到珠丸中出现裂缝。
- 当板仍在磁铁上时,向每个孔中加入40μL预热的水。密封板,彻底涡旋,并短暂脉冲旋转板。
- 通过将板放回磁体上并将40μL洗脱转移到新的"样品"板中来捕获磁珠。样品现在被纯化,接下来的步骤浓缩了200-1,000 bp的片段。
- 可选的QC步骤:从样品中取出4μL并转移到QC板中。将4μL水倒回样品中。这决定了大片段的百分比。
- 向样品中加入22μL顺磁珠(1:0.55比例),小心混合,并在室温下孵育2分钟。
- 置于磁铁上捕获磁珠5分钟,并将上清液转移到"样品"板的柱7-12中。从磁体中取出板并加入30μL磁珠(最终比例为1:1.3)。仔细混合,让它在室温下静置2分钟。
- 捕获珠子5分钟,然后丢弃上清液。
- 如前所述,用200μL新鲜80%乙醇洗涤所有微珠3次(步骤6.6.2 – 6.6.3)。
- 一旦沉淀干燥,用8μL预热的TE缓冲液将DNA洗脱到每个孔中,同时仍在磁铁上。
- 从磁铁上取下板,密封并彻底涡旋。让板在室温下孵育2分钟,脉冲旋转,然后将板放回磁铁上2分钟。将上清液转移到新板(板2)。上清液中。
- 为了获得最大的回收率,用额外的8μL预热的TE缓冲液重复洗脱。将样品放入适当的孔中,使每个样品具有16μL的最终文库。
注意:在此步骤结束时,应该有两个板(如果没有完成QC板,则一个板)。QC板将具有预先选择的片段,第二板应具有48孔的最终文库(总共16μL)。
7. 使用荧光测定法对最终文库和 QC 样品进行定量
- 使用荧光定量测定或类似方法完成DNA定量。
- 如果QC定量完成,则确定<1,000 bp的样品损失百分比。损失不应超过约20% - 如果损失更多,则应用的珠子比率可能存在问题。
- 确定每个样品的片段大小,优选使用毛细管电泳机。要计算摩尔浓度,请使用以下等式:[文库浓度(ng / μL)* 106]/[660 * 中位数片段大小(bp)])。
注:文库已准备好合并(等摩尔量)并按照标准的下一代测序程序进行测序。
结果
作为概念验证,ChIP-Seq为六名人类供体完成了三组免疫细胞类型:朴素CD4 T细胞(CD4),经典单核细胞(MO)和自然杀伤细胞(NK),如前所述通过FACS分选富集13。带下划线的过程由 图 1所示的九个不同的过程组成。
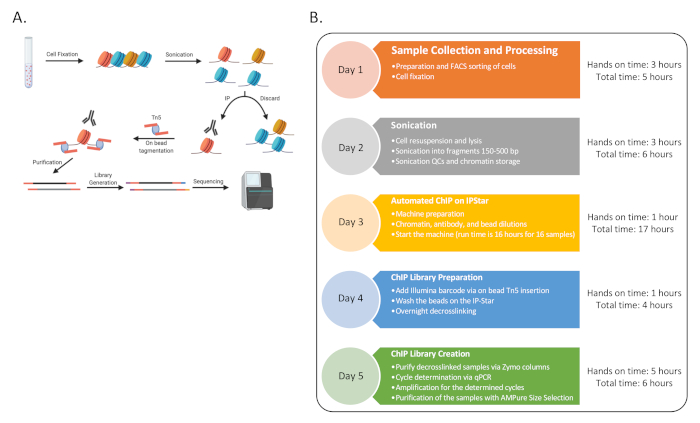
图1:程序的一般流程图(A)整个过程的卡通(在BioRender中生成)。(B) 协议所有主要步骤的流程图,以及与每天相关的估计动手和总时间。测序可能在第5天结束时或之后进行多轮。时间线也可以在整个星期内错开,其中连续的第3-4天可以在一周内多次完成,以生成48个ChIP样本。请点击此处查看此图的放大版本。
通过流式细胞术13分离细胞后,离心分离的细胞,固定细胞并如上所述储存。一旦收集了所有样品,就如上所述,将样品裂解并准备进行12次分批的色度剪切。对于每个样品,达到最佳超声处理的循环次数为10。定量测量以及剪切染色质片段大小的测量表明,我们的方法在三组免疫细胞上具有很高的重现性(图2A)。将不同的人免疫细胞分批进行超声处理,并且产量非常一致,>70%的样品在100-500 bp之间,持续14个周期(16 s ON,每个周期32 s OFF)。此时,超声处理后具有大碎片的样品(<100-500 bp之间的70%样品)被认为是失败的。这些样品可以超声处理1-2个额外的循环,或者被丢弃并随后用来自另一个沉淀的细胞替换。我们的方法显示,没有一个样品需要更多的超声处理或被消除,这表明该过程的绝对稳健性。
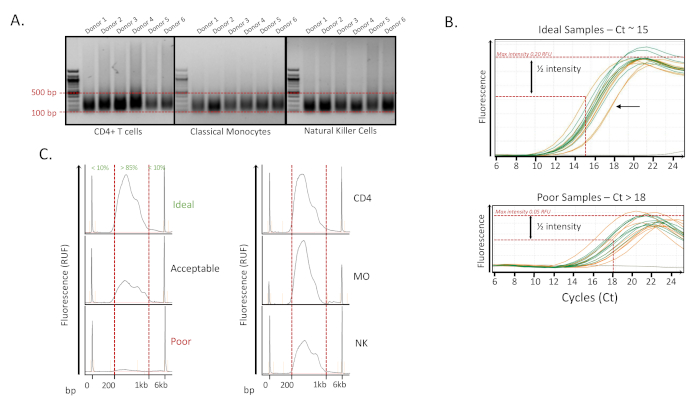
图2:预测序QC实例(A)1.2%琼脂糖凝胶显示出超声处理的重现性。6种供体的超声处理样品,分为三种细胞类型:朴素CD4 T细胞(CD4),经典单核细胞(MO)和自然杀伤细胞(NK)。将样品超声处理14个周期(每个周期16 s ON,32 s OFF)。对于每个样品,将约200ng脱杂环联染色质上样加载在1%琼脂糖凝胶上。如果超过70%的片段在100-500 bp以内,则样品被认为是好的(B)顶部 - 分析qPCR扩增曲线,以确定扩增的最佳循环数(Ct,其中最大强度为1/2)。理想样品的 Ct 约为 15,扩增最多可比测量的 Ct 多 2 个周期。箭头是 Ct 大于 18 的坏示例。底部 - 显示了一组 Ct 大于 18 的不良样本示例。这些样品也显示出较低的荧光强度。(C)左 - 片段分析仪电泳迹线显示了扩增和尺寸选择后最终标记库的分布情况。片段库超过85%的样品位于200-1,000 bp以内,被认为是良好的样品。荧光峰强度的测量也被认为是一个重要的QC参数,事实上,如果信号较低,样品不太可能很好地测序。右 - 显示了CD4,MO和NK中阳性样本的示例。请点击此处查看此图的放大版本。
定量后,将样品在具有H3K27ac抗体的ChIP液体处理程序上运行,然后用Tn5转座酶标记。为了通过qPCR确定适当的扩增循环次数,使用了10%的标记样品。为了确定样品扩增的循环次数,我们找到样品强度为循环测定平均最大值一半的循环(图2B)。Ct值超过18的样品在测序后表现不佳,因此其Ct值表明ChIP样品失败。这些样品在扩增后通常也产生较低量的DNA。Ct值等于或小于15的样品(100,000个细胞输入)是理想的,15至18之间的样品是可以接受的,但测序后不太一致。对于输入细胞少于100,000个的样品,Ct值通常在15到18之间,但不需要超过18个周期来产生足够的产物进行测序。
在DNA标记扩增后,纯化文库并选择大小,以获得理想的大小分布,范围从200到1,000 bp,用于NextGen测序。每个文库的大小分布评估已经完成,因为当超过85%的DNA片段在200至1,000 bp之间时,获得了最佳测序数据(图2C)。值得注意的是,由于加载相同数量的DNA(通过荧光定量测量),因此注意到荧光强度较低的样品通常测序较差(图2C)。
测序后,应用基于ENCODE ChIP-Seq指南的标准质控品5,14,15。
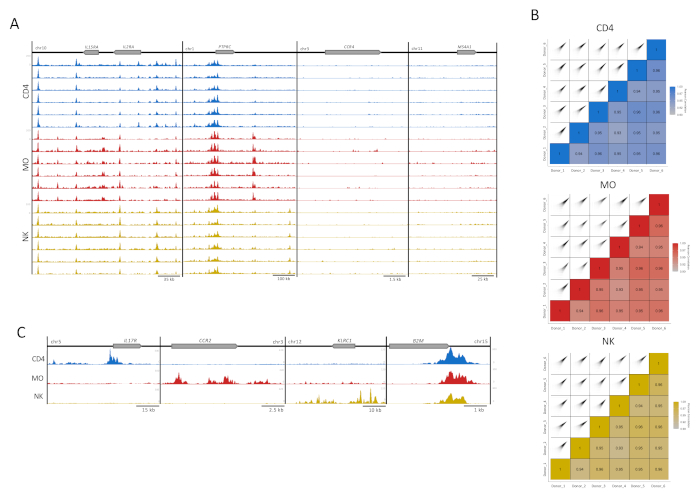
图3:免疫细胞样品的再现性。(A) H3K27ac 跟踪(UCSC 基因组浏览器,最大强度,平滑功能为 4,均具有相同比例的 Y 轴)中每种细胞类型(CD4、MO 和 NK)中的 6 个供体(每个重复 100,000 个细胞)。图中显示了四个示例性位点,两个具有(IL2RA位点和PTPRC),两个没有富集H3K27ac(CCR4和MS4A1)。(B)Pearson对供体之间的相关性和相应的相关图使用300 bp扩展和500 bp窗口在MEDIPS包内对每种细胞类型重复16个。(C)每种细胞类型的合并供体文件显示细胞类型特异性区域(CD4的IL17R,MO的CCR2和NK的KLRC1)中的H3K27ac轨迹(UCSC基因组浏览器最大强度,平滑功能为4)和所有细胞类型中存在的家养基因B2M。请点击此处查看此图的放大版本。
对于视觉质量控制,制备了H3K27ac富集轨迹,以显示在UCSC基因组浏览器中。对于四个基因位点,每个样品的单独轨迹显示出高映射质量和信噪比,反映了我们测定的高一致性和稳健性(图3A)。左侧的两个位点在这些细胞类型中具有表达良好的基因,而右侧两个位点中的基因未表达并用作背景对照13(图3A)。此外,MEDIPS分析包被用作测序后变量来评估技术重复之间的相关指数(图3B)5,16,17,建立500 bp bins16的读取富集水平的相关程度。对于大多数成对比较,Pearson相关性指数显示超过90%的相关性,表明生物重复之间的高度一致性(图3B)。将具有可接受相关性的重复项合并在一起,以提高信噪比。虽然细胞类型特异性位点在适当的细胞中显示出高富集性,但家养基因(B2M)显示出非常一致的组蛋白修饰(图3C)。对于分析,合并来自重复的轨迹将增加富集,增强特定信号,包括重要的细胞类型特异性增强子,并降低人类样品固有的个体间变异性5。
虽然这项研究使用了100,000个细胞,但在人类培养的T细胞系(HUT78)中,只有10,000个细胞具有很高的可重复性。从少于100,000个细胞的样品中进行的ChIP-Seq数据集之间的相关性分析显示出高再现性和相关性,低至10,000个细胞(图4A)。
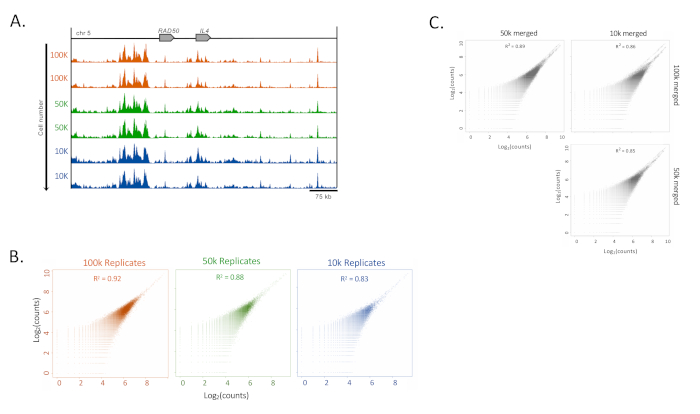
图4:低输入样品的再现性(A)H3K27ac ChIP-Seq在HUT-78细胞(T细胞淋巴瘤细胞系)中100,000至10,000的细胞的一致性示例。轨迹(UCSC基因组浏览器,最大强度,平滑功能4,所有具有相同比例的Y轴)显示IL4位点。(B) PEARSON 相关性,在 MEDIPS 包16中使用 300 bp 扩展和 500 bp 窗口进行重复。 (C) 使用与 (B)16中相同的 MEDIPS 参数,对不同细胞数组(100,000、50,000 和 10,000 个细胞)之间的 Pearson 相关性。请点击此处查看此图的放大版本。
Pearson相关性分析显示高相关指数(83%至92%),表明低细胞数样品的信号维持。然而,随着细胞数量的减少以及相关系数的下降,背景增加了(图4B)。为了保持低背景信号,合并了技术重复项,并测试了组间的相关性(图4C)。
| 10X细胞固定缓冲液 | |
| 复合 | 最终浓度 |
| 甲醛溶液 | 11% |
| 氯化钠 | 100 平方米 |
| 乙二胺四乙酸烷值,pH 值 8.0 | 1 毫米 |
| 乙二醇, pH值 8.0 | 0.5 毫米 |
| 肝素,pH 值 7.5 | 50 毫米 |
| 完全裂解缓冲液 | |
| 复合 | 最终浓度 |
| 三氢盐酸盐,pH 值 8.0 | 50 毫米 |
| 乙二胺四乙酸烷值,pH 值 8.0 | 10 毫米 |
| 安全数据表 | 0.25% |
| 丁酸钠 | 20 毫米 |
| 蛋白酶抑制剂鸡尾酒 | 1 倍 |
| 短期裂解缓冲液 | |
| 复合 | 最终浓度 |
| 三氢盐酸盐,pH 值 8.0 | 50 毫米 |
| 乙二胺四乙酸烷值,pH 值 8.0 | 10 毫米 |
| 安全数据表 | 0.25% |
| 标签组合 | |
| 复合 | 最终浓度 |
| 三氢盐酸盐,pH 值 8.0 | 10 毫米 |
| 氯化镁 | 5 毫米 |
| N,N-二甲基甲酰胺 | 10% |
| Illumina tagmentation enzyme | 1:24 卷:卷 |
| CtD 混合 | |
| 复合 | 每个样品(μL) |
| 下一个Era 指数引物 A (25 μM) | 0.275 |
| 下一个Era 指数底漆 B (25 μM) | 0.275 |
| 2 个 KAPA 高保真热启动预混合 | 2.75 |
| 1:1000 SYBR 绿色染料 | 0.11 |
| ROX钝化染料 | 0.11 |
| 水 | 填充至 4 μL |
| 放大器组合 | |
| 复合 | 每个样品(μL) |
| 2 个 KAPA 高保真热启动预混合 | 27.5 |
| 水 | 填充至 31 μL |
补充表1:缓冲液配方。
补充表2:Spearman和Pearson样本对6个供体和每种细胞类型的相关性。请点击此处下载此表格。
讨论
这里描述的方法扩展了ChIPmentation程序11,该程序通过自动化和微缩放方案,在DNA纯化之前实现标记文库制备方案。自ChIP-Seq开始以来,所需的细胞数急剧减少,从组蛋白的约2000万个细胞减少到数百个甚至单细胞1,7,10,12,18,19,20,21。这些新开发的方法通过提高灵敏度并允许测试罕见的临床细胞群,可以更深入地了解顺式调节机制如何在细胞中起作用5,6,12,17。例如,最新和流行的程序之一,称为CUT&TAG,作为强大而灵敏的ChIP-Seq替代方案9。它产生出色的信噪比,因为Tn5酶与蛋白质A共价结合,并识别具有高特异性的ChIP抗体的Fc链9。Tn5-酶的背景活性降低,因为酶在与靶抗体9结合之前不起作用。然而,这种方法在临床环境中的实施是有限的,因为它需要非固定的活细胞。此外,从低渗细胞核中去除DNA片段可能会对染色质产生负面影响,因为它在测定过程中被去除。使用新鲜细胞和活细胞的必要要求是罕见临床样本和大量样本的问题来源,因为大型队列可能需要数年才能收集5。另一种类型的方法,drop-ChIP,在处理ChIP19之前,优雅地使用微流体装置来生成基于液滴的标签。然而,它使用高度专业化的微流体装置,虽然可以完成单细胞ChIP-Seq,但它也仅限于使用活细胞7,8,9,18,19。依赖于ChIP-Seq的较新方法,如PLAC-Seq或HiChIP,试图理解ChIP-Seq峰22,23之间的3维(3D)相互作用。这些3D方法令人兴奋,因为它们正在识别整个基因组中的顺式调节或TF介导的相互作用,并更好地理解感兴趣的细胞类型,健康组织和疾病背景下基因表达的调节。
要使方案成功,需要考虑几个关键步骤,例如超声染色质的质量和抗体的质量。剪切效率至关重要,如果染色质未被很好地超声处理,则测定效率会急剧下降24。由于所需的细胞数量,超声处理是ChIP-Seq的一个具有挑战性的方面。在协议中使用的超声仪上,效率在300,000个细胞下急剧下降。这是ChIP-Seq中一个具有挑战性的方面,因为在该水平下进行超声处理通常需要酶分碎,这不太公正。因此,超声处理是真正的微尺度ChIP-Seq的主要限制因素。其他超声处理平台和市售试剂盒被测试用于超声处理染色质,但这里使用的超声仪具有最强大和可重复的结果。超声仪的另一个优点是不必购买专门的管子来运行超声处理,这降低了处理大量样品时的成本。为了获得最佳的超声处理,首先,如上所述对超声处理器进行预热非常重要。其次,要裂解沉淀,建议在裂解时使移液器尖端接触管的底部,以分解具有更多物理约束的细胞。第三,在超声处理之前形成的任何气泡都会阻碍样品均匀超声处理的能力。如果在裂解过程中形成任何气泡,用移液器将其除去非常重要。如果不去除大量样品,这可能具有挑战性,但是如果将尖端轻轻地压在气泡上,则可以缓慢地将其抽出而不会损失太多样品。最后,在确定循环次数时,完成一个时间过程,其中每三个循环,取出样品,纯化并在琼脂糖凝胶上运行。避免样品超声/超声处理,因为这会降低ChIP效率。如果样品未超声处理,则大片段会对 ChIP-Seq 质量产生负面影响 24。另一方面,如果样品过度超声处理,则存在目标表位在此过程中丢失的风险。
ChIP-Seq的另一个重要部分是抗体的质量。在进行任何大规模研究之前,有必要优化将要使用的抗体。目标是获得基因组已知区域的显着高信噪比,另一个是可重复性。如果抗体拉动大量背景信号,则可能建议使用更大的输入或尝试不同的批次/供应商。这将增加开始大规模实验之前的时间,但这是必不可少的一步。为了测试信噪比,建议将qPCR用于已知是抗体靶标的区域和已知不存在的另一个区域。已经注意到组蛋白修饰比TF更强大,更容易优化。
上述方案为以半自主、微尺度方式进行高通量组蛋白修饰 ChIP-Seq 提供了一种稳健的方法。该方法限制了手动操作时间,并提高了手动ChIP-Seq的再现性。先前在实验室中完成的研究使用手动ChIP进行技术重复,并获得0.505的Spearman相关性平均值,然而,使用半自动系统,不同供体之间的Spearman相关性与NK细胞平均值为0.66(补充表2)。这也减少了约40%的动手时间。这里描述的方法已经针对组蛋白修饰进行了优化(此处显示了H3K27ac,但该协议不需要对其他方法进行任何修改),并且只需要对TF ChIP-Seq进行微小的修改即可实现。尽管抗体的质量很高,但主要的修改将是超声处理时间,并且可能是IP期间使用的缓冲液。通常,对于TF ChIP测定,该方法可能更好地用于稍长的染色质片段(范围约为350-800 bp),因为TF:DNA复合物可能不太能够通过严格的超声处理来维持6。缓冲液可能还需要更改为自定义混合物或其他行业可用的试剂盒,因为TF的行为可能与组蛋白修饰不同。
尽管自动ChIP液体处理机仅针对10,000个细胞进行了测试,但在较低的染色质浓度下,可重复性明显降低。因此,不建议将方案用于少于10,000个细胞,其中100,000个细胞是最佳条件。该协议也是使用工业ChIP缓冲区完成的,这是一项额外的费用,但提供了更高质量的数据。可以根据超声处理条件修改方案(只要剪切的染色质保持在相同的范围内),可以为免疫沉淀定制缓冲液(IP;可能需要优化),或者可以使用ChIP液体处理程序。该协议的一个限制是使用ChIP液体处理器,这可能是一项昂贵的投资,并且一次只能运行16个样品。ChIP液体处理器仅限于小规模反应,不建议使用大于100万的细胞数。但是,无需它,即可通过手动完成 IP 和清洗步骤来完成协议。如果IP和洗涤是手工完成的,则完成测定的时间将增加,再现性可能会降低,但运行高质量的ChIP-Seq实验,本指南仍然有用。值得注意的是,其他液体处理程序可以适应运行半自动ChIP反应。
总而言之,该系统的主要优点是高通量性,因为IP和洗涤步骤是自主完成的。因此,可以完成连续几轮的ChIP实验,允许在5天内完全处理多达48个样品并准备好进行测序,与手动ChIP-Seq实验相比,动手时间有限。另一个好处是增加了可重复性,因为ChIP-Seq可能很难获得高度可重复的结果。其他方法要么需要活细胞,复杂的微量移液系统,要么需要手工完成的工作。该系统必须针对低输入样品(<10,000个细胞)进行优化,最终允许单细胞ChIP反应。该系统还能够适应较新的ChIP方法,例如PLAC-Seq和HiChIP 22,23。
披露声明
作者没有什么可透露的。
致谢
我们感谢Vijayanand实验室成员的技术支持和建设性讨论,并感谢Diagenode的Sharron Squazzo博士和Geoffrey Berguet先生在超声器和ChIP液体处理机和协议方面的技术援助。这项工作得到了NIH资助的AI108564,R01HL114093,S10RR027366(BD FACSAria II)和S10OD016262(Illumina HiSeq 2500)。
材料
| Name | Company | Catalog Number | Comments |
| 200 µl tube strips (8 tubes/strip) + cap strips | Diagenode | C30020002 | Strip tubes for use on the IP Star; ChIP 8-tube strip |
| AMPure XP for PCR Purification | Beckman Coulter | A63880 | SPRI beads |
| Axygen 0.6 mL MaxyClear Snaplock Microcentrifuge Tube | Corning | MCT-060-C | 0.65 mL low binding tube |
| Bioruptor Pico Sonicator | Diagenode | B01060010 | Sonicator used in the lab but others can be used |
| ChIP DNA Clean & Concentrator (Capped Columns) | Zymo Research | D5205 | DNA clean-up kit |
| Dynabeads Protein A for Immunoprecipitation | ThermoFisher | 10001D | |
| EDTA (0.5 M), pH 8.0, RNase-free | ThermoFisher | AM9260G | |
| EGTA pH 8.0 | Millipore Sigma | E3889-25G | |
| Eppendorf ThermoMixer C | Eppendorf | 2231000667 | |
| Formaldehyde solution | Millipore Sigma | 252549-1L | |
| Glycine | Millipore Sigma | 50046-250G | |
| H3K27ac polyclonal antibody - Premium | Diagenode | C15410196 | |
| HEPES (1 M) pH 7.5 | ThermoFisher | 15630080 | |
| IDT for Illumina Nextera DNA Unique Dual Indexes | Illumina | 20027213 | |
| Illumina Tagment DNA Enzyme and Buffer Small Kit | Illumina | 20034197 | |
| IP-Star Compact Automated System | Diagenode | B03000002 | Automated system for ChIP-Seq studies; ChIP liquid handler |
| KAPA HiFi HotStart ReadyMix | Roche | KK2601 | PCR mix |
| Medium reagent container for SX-8G IP-Star Compact | Diagenode | C30020003 | |
| MgCl2 (magnesium chloride) (25 mM) | ThermoFisher | R0971 | |
| N,N-Dimethylformamide | Millipore Sigma | D4551-250ML | CAUTION - low flash point |
| NaCl (5 M), RNase-free | ThermoFisher | AM9760G | |
| PBS (10X), pH 7.4 | ThermoFisher | 70011044 | |
| PCR Flex-free 8-tube stripes, attached individual optical caps | USA Scientific | 1402-4700 | 8 strip tubes, 0.2 mL 8-tube strip |
| Proteinase Inhibitor Cocktail | Millipore Sigma | P8340 | |
| Proteinase K Solution (20 mg/mL), RNA grade | ThermoFisher | 25530049 | |
| PureLink RNase A (20 mg/mL) | ThermoFisher | 12091021 | |
| Quant-iT PicoGreen dsDNA Reagent | ThermoFisher | P7581 | Used in the flourescence quantification |
| QuantStudio 6 Flex Real-Time PCR System | ThermoFisher | 4485699 | qPCR |
| ROX Reference Dye | ThermoFisher | 12223012 | |
| Sodium butyrate | Millipore Sigma | 303410-100G | |
| SYBR Gold Nucleic Acid Gel Stain (10,000X Concentrate in DMSO) | ThermoFisher | S11494 | nucleic acid dye |
| SYBR Green I Nucleic Acid Gel Stain - 10,000X concentrate in DMSO | ThermoFisher | S7563 | |
| TE Buffer | ThermoFisher | 12090015 | |
| Tips (bulk) | Diagenode | C30040020 | Tips for the IP Star |
| True MicroChIP Kit | Diagenode | C01010130 | Contains all the buffers for the IP; ChIP kit |
| UltraPure 1M Tris-HCI, pH 8.0 | ThermoFisher | 15568025 | |
| UltraPure SDS Solution, 10% | ThermoFisher | 24730020 |
参考文献
- Barski, A., et al. High-resolution profiling of histone methylations in the human genome. Cell. 129 (4), 823-837 (2007).
- Johnson, D., Mortazavi, A., Myers, R., Wold, B. Genome-Wide Mapping of in Vivo Protein-DNA Interactions. Science. 316, 1497-1502 (2007).
- Mikkelsen, T. S., et al. Genome-wide maps of chromatin state in pluripotent and lineage-committed cells. Nature. 448 (7153), 553-560 (2007).
- Furey, T. S. ChIP-seq and beyond: new and improved methodologies to detect and characterize protein-DNA interactions. Nature Reviews Genetics. 13 (12), 840-852 (2012).
- Seumois, G., et al. Epigenomic analysis of primary human T cells reveals enhancers associated with TH2 memory cell differentiation and asthma susceptibility. Nature Immunology. 15 (8), 777-788 (2014).
- Schmiedel, B. J., et al. 17q21 asthma-risk variants switch CTCF binding and regulate IL-2 production by T cells. Nature Communication. 7, 13426 (2016).
- Ai, S., et al. Profiling chromatin states using single-cell itChIP-seq. Nature Cell Biology. 21 (9), 1164-1172 (2019).
- Brind'Amour, J., et al. An ultra-low-input native ChIP-seq protocol for genome-wide profiling of rare cell populations. Nature Communication. 6, 6033 (2015).
- Kaya-Okur, H. S., et al. CUT&Tag for efficient epigenomic profiling of small samples and single cells. Nature Communication. 10 (1), 1930 (1930).
- Youhanna Jankeel, D., Cayford, J., Schmiedel, B. J., Vijayanand, P., Seumois, G. An Integrated and Semiautomated Microscaled Approach to Profile Cis-Regulatory Elements by Histone Modification ChIP-Seq for Large-Scale Epigenetic Studies. Methods Molecular Biology. 1799, 303-326 (2018).
- Schmidl, C., Rendeiro, A. F., Sheffield, N. C., Bock, C. ChIPmentation: fast, robust, low-input ChIP-seq for histones and transcription factors. Nature Methods. 12 (10), 963-965 (2015).
- Skene, P. J., Henikoff, S. An efficient targeted nuclease strategy for high-resolution mapping of DNA binding sites. Elife. 6, 21856 (2017).
- Schmiedel, B. J., et al. Impact of Genetic Polymorphisms on Human Immune Cell Gene Expression. Cell. 175 (6), 1701-1715 (2018).
- Landt, S. G., et al. ChIP-seq guidelines and practices of the ENCODE and modENCODE consortia. Genome Research. 22 (9), 1813-1831 (2012).
- Chen, L., et al. Genetic Drivers of Epigenetic and Transcriptional Variation in Human Immune Cells. Cell. 167 (5), 1398-1414 (2016).
- Lienhard, M., Grimm, C., Morkel, M., Herwig, R., Chavez, L. MEDIPS: genome-wide differential coverage analysis of sequencing data derived from DNA enrichment experiments. Bioinformatics. 30 (2), 284-286 (2014).
- Engel, I., et al. Innate-like functions of natural killer T cell subsets result from highly divergent gene programs. Nature Immunology. 17 (6), 728-739 (2016).
- Grosselin, K., et al. High-throughput single-cell ChIP-seq identifies heterogeneity of chromatin states in breast cancer. Nature Genetics. 51 (6), 1060-1066 (2019).
- Rotem, A., et al. Single-cell ChIP-seq reveals cell subpopulations defined by chromatin state. Nature Biotechnology. 33 (11), 1165-1172 (2015).
- Wang, Q., et al. CoBATCH for High-Throughput Single-Cell Epigenomic Profiling. Molecular Cell. 76 (1), 206-216 (2019).
- Consortium, E. P. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Fang, R., et al. Mapping of long-range chromatin interactions by proximity ligation-assisted ChIP-seq. Cell Research. 26 (12), 1345-1348 (2016).
- Mumbach, M. R., et al. HiChIP: efficient and sensitive analysis of protein-directed genome architecture. Nature Methods. 13 (11), 919-922 (2016).
- Diagenode. The Ultimate Guide for Chromatin Shearing Optimization with Bioruptor Standard and Plus. Diagenode. , (2012).
Erratum
Formal Correction: Erratum: A Semiautomated ChIP-Seq Procedure for Large-scale Epigenetic Studies
Posted by JoVE Editors on 9/14/2020. Citeable Link.
An erratum was issued for: A Semiautomated ChIP-Seq Procedure for Large-scale Epigenetic Studies. An author's name was updated.
The name was corrected from:
Pandurangan Vijayanad
to:
Pandurangan Vijayanand
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。