需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
细胞扩散过程中细胞边缘动力学的定量分析
摘要
在此协议中,我们介绍了基于活细胞显微镜的细胞扩散检测的实验程序。我们为荧光标记细胞的无偏见分割和细胞扩散过程中的跛脚足动力学定量分析提供了开源计算工具。
摘要
细胞扩散是一个动态过程,其中悬浮在介质中的细胞附着在基材上,并把自己从圆形压扁成薄而分散的形状。在细胞基底附着物之后,细胞形成一块从细胞体中散发出的薄薄的跛脚状。在lamellipodia中,球状作用素(G-actin)单体聚合成密集的丝状活性蛋白(F-actin)网状物,推动对等离子膜,从而提供细胞扩散所需的机械力。值得注意的是,控制跛脚细胞聚合的分子分子对许多其他细胞过程至关重要,如细胞迁移和内分泌。
由于扩散细胞形成横跨整个细胞外围并持续向外扩张的连续跛脚状体,细胞扩散检测已成为评估跛脚突起动力学的有效工具。虽然已经制定了细胞传播检测的若干技术实施,但目前缺乏对工作流程的详细描述,其中包括分步协议和数据分析的计算工具。在这里,我们描述了细胞扩散检测的实验过程,并提出了一个开源工具,用于定量和公正地分析细胞边缘动力学在扩散过程中。当与药理学操作和/或基因沉默技术相结合时,此协议可适应调节跛脚突出的分子参与者的大规模屏幕。
引言
拉梅利波德突起是生长细胞前部形成的突出细胞骨结构。在lamilipodia,在Arp2/3复合物的帮助下,行为物的聚合和福明斯创造了一个快速生长的分支行为网格,推动对等离子膜1,2。网格工程产生的推动力在物理上推动细胞向前推进1,3,4,5。Arp2/3复杂或信号通路的耗竭,对于跛脚突出至关重要,通常会损害细胞迁移6,7。虽然跛脚足缺细胞的迁移也已报告8,9,跛脚足在细胞迁移中的重要性是显而易见的,因为这种突起结构的耗竭扰动细胞通过复杂的生物微环境移动的能力6,10。
理解迁移细胞中跛脚足的调控的一个主要障碍是跛脚突出动力学的自然变异性、大小和形状11、12、13、14。此外,最近的研究表明,跛脚皮迪亚表现出复杂的突出行为,包括波动,周期性,加速突出14,15。与迁移细胞6、16的高度可变的跛脚足症相比,在细胞扩散过程中形成的跛脚足足12更为均匀。由于扩散和迁移细胞的突起活动是由相同的大分子组件驱动的,包括分支作用网络、收缩性活体蛋白束和基于内格林的细胞基质粘附17、18,因此扩散细胞被广泛用作研究跛脚体动力学调控的模型。
细胞扩散是一个动态的机械化学过程,悬浮中的细胞首先通过基于内格林的附着力17、19、20粘附粘附,然后通过延长基于作用的突起21、22、23进行扩散。在扩散阶段,从细胞体中喷出的跛脚足部突出异位和持久,很少或根本没有收回或停滞12。最常用的细胞扩散协议是端点检测,其中扩散细胞在电镀19,24后在不同时间固定。这些检测虽然快速而简单,但检测跛脚皮迪亚动态特征的变化的诊断能力有限。为了确定控制跛脚足动力学的分子机制,Sheetz小组率先对活扩散细胞进行了定量分析,并发现了细胞边缘突起11、12、22的许多基本特性。这些研究表明,活细胞传播检测是细胞生物学实验室工具箱中一种强大而有力的技术。尽管如此,细胞生物学界目前还无法为活细胞传播检测提供详细的协议和开源计算工具。为此,我们的协议概述了成像活传播细胞的程序,并提供了一个自动化的图像分析工具。为了验证这种方法,我们使用Arp2/3抑制作为实验治疗,并表明抑制Arp2/3复合体的功能不会阻止细胞扩散,但会导致细胞突起速度显著降低,以及细胞边缘突起的稳定性,从而产生锯齿状细胞边缘。这些数据表明,活细胞成像和自动图像分析的结合是分析细胞边缘动力学和识别调节跛脚皮迪亚的分子组件的有用工具。
研究方案
1. 细胞播种
注:描述的细胞扩散协议使用小鼠胚胎成纤维细胞(MEF)执行,表达PH-Akt-GFP(PIP3/PI(3,4)P 2的荧光标记)。该细胞系通过CRISPR介质基因编辑,通过基因学整合PH-Akt-GFP(添加基因#21218)的表达结构而生成。然而,其他在基因组中短暂表达或集成的荧光标记也可以用于此测定。为了实现最佳图像分割,我们建议使用细胞质均匀分布的荧光标记,例如细胞溶胶 GFP。
- 培养一个10厘米的细胞盘到90%的汇流。
- 一旦细胞达到适当的共体,将22毫米×22毫米盖片(#1.5;0.17毫米厚度)放入35毫米细胞培养盘中。涂上 400μL 的纤维素,在 PBS 中稀释为最终浓度为 2.5μg/mL。
注:检测所需的盖片数量由实验条件和技术复制品的数量决定。 - 将带有纤维素涂层盖唇的 35 mm 盘放入 37 °C、5% CO2 孵化器中 1 小时。
- 用盖子从孵化器中取出盘子。吸气纤维素,用PBS轻轻在盖片周围管道两到三次,用PBS清洗盖片。
- 从10厘米的细胞盘中吸气细胞培养介质,用PBS清洗。
- 将 650 μL 的 0.05% trypsin-EDTA 添加到 90% 的细胞汇合盘中,使菜向倾斜以均匀地分配酶。将带试剂的菜放入孵化器中1分钟。
- 用细胞从孵化器中取出盘子。将 10 mL 的细胞培养介质添加到 15 mL 离心机管中。快速在菜中再添加 10 mL 的介质,以淬灭试穿素。
- 将试用细胞的 1 mL 放入 15 mL 离心机管中,以稀释细胞。将管子的内容上下吹,以确保细胞在介质中的均匀分布。对于聚合倾向较高的细胞类型,建议通过细胞过滤器过滤细胞(100μm网格大小),以尽量减少细胞团块的发生。
- 从管子,移液器500 - 1000μL的稀释细胞到35毫米的菜包含盖唇。
- 轻轻摇动盘子,均匀地展开细胞。确保盖片处于 ±10% 的共存度(±50,000 个细胞/mL),并根据需要调整稀释细胞的体积。
注:细胞处于如此低的共体中的目的是确保每个视域中有1-2个极化细胞,用于在整个细胞扩散获取中集中目标。 - 将 10 厘米盘中剩余细胞的 1/5 传递到每个治疗条件下的一个 6 厘米的盘中。将经过的菜肴和带盖子的 35 mm 盘放入孵化器过夜。
注意:这些细胞将被分析为传播动力学。
2. 药物孵化和细胞恢复
- 在两个 15 mL 离心机管中各加入 5 mL 细胞培养介质,在两个 50 mL 离心管中各加入 20 mL 无苯酚红色 DMEM。
注:管对的数量(15 mL + 50 mL)应与实验条件的数量相对应。 - 为了测试Arp2/3对细胞扩散的重要性,移液器要么将Arp2/3、CK-666的药理抑制剂,或控制治疗(如DMSO)放入每对离心管中,达到所需的浓度。
- 从孵化器中取出经过的 6 厘米菜肴(见第 1.11 步),然后吸气介质。用温暖的 PBS 洗碗。
- 将CK-666或DMSO补充的15mL离心管的内容添加到每个通道中。给每道菜贴上正确的药物治疗标签,并将菜肴放入孵化器中一小时。
- 从孵化器中取出盘子,吸气媒体。用温暖的 PBS 清洗洗碗,以便彻底去除所有剩余的酚红色介质。
- 将 230 μL 的 0.05% trypsin-EDTA 添加到每个 6 厘米的菜中,并孵育细胞 1 分钟。
注意:如果适用,可以用非催化细胞粘附阻滞剂代替肌氨基。 - 从孵化器中取出盘子。每次治疗,将5mL的药物补充酚红色免费DMEM添加到指定为"管B"的15mL离心机管中。在相关菜中添加相同介质的额外 5 mL 以淬灭试剂。将菜内物品转移到指定为"管A"的15mL离心机管中。
- 将1mL的细胞从A管转移到B管中,每次治疗重复。
- 将A和B管放入孵化器45分钟,使细胞从尝试中恢复。稍微松开离心管的盖子,然后放入孵化器,允许二氧化碳 渗透。
注意:不同细胞类型的恢复时间可能有所不同。虽然在我们的实验中,45分钟长的恢复对细胞生存能力的影响微乎其微,但有些细胞类型在暂停中维持太久时可能会经历异常。因此,我们建议从经验上确定最佳恢复时间。最佳恢复时间使细胞快速同步扩散,样本中无死细胞或凋亡细胞。
3. 磁室制备
- 确保可容纳 22 mm x 22 mm 方形盖唇的 1 井香酰细胞磁室的所有部分在使用前均已清洁。
- 将带盖唇的 35 mm 盘从孵化器中取出(参见步骤 1.11)。吸气细胞培养介质,用温暖的 PBS 清洗盖子。
- 使用一对钳子从 35 mm 盘中取出盖片,然后轻轻地将盖片放在磁室的底部板上。
- 将硅胶垫片放在盖片顶部。
注意:放置不当的硅胶垫片是磁室漏水的最常见原因。确保垫片位于底部板的凹痕中,并且不会超过凹痕。 - 将主体连接到底部板上。
注意:做这部分非常缓慢。一个好的提示是用一只手按住底部板,同时将主体放在顶部。这确保了主体的磁铁不会将底部板向上抬起,从而可能取代并裂开盖片。 - 在磁室中加入 1 mL 的药物补充酚红色免费 DMEM。采取无绒组织,并仔细擦在主体和底部板之间的外壳,以检查任何泄漏。
注意:如果有泄漏,请快速吸气媒体,然后从步骤 3.4 重新开始。 - 将透明盖降低到主体上以包围磁室。
- 先用水喷洒实验室组织,擦拭磁室底部(盖片,而不是金属部分)。之后,用少量的70%乙醇喷洒第二个实验室组织并擦拭,小心不要裂开盖子。
4. 图像采集
- 将舞台顶部孵化器和目标加热器预热至 37 °C,并将舞台顶部孵化器中的 CO2 水平设置为 5%。
注意:如果阶段顶部孵化器未连接到 CO2 供应,应向细胞培养介质补充 25 mM HEPES 以保持恒定的 pH 7.4。 - 将足够的浸入油应用于预热的 60 倍、1.4 N.A. 油目标。
注:我们在此协议中使用 60X、1.4 N.A. 油浸入目标,因为它的视野相当大,光收集效率卓越。如果需要更大的视场,只要图像的信号噪声比大于 2.5,就可以使用较低的放大目标(例如20 倍)。 - 将完成的磁室和管 B(步骤 2.9) 带到共焦显微镜中。将磁室放在舞台顶部孵化器上。
注意:将磁室轻轻地放在舞台上,以避免在浸入式油中产生气泡。 - 使用 GFP 通道将焦点对准荧光电池。确保细胞边缘清晰且定义良好。
- 从管 B 中取出磁室和移液器 500 μL 的透明盖进入磁室。将透明盖重新放在磁室顶部。
- 为了识别适合细胞扩散分析的细胞,搜索尚未附着在盖唇上但不再滚动的细胞的"光环"。处于盖唇附件早期阶段的细胞也是很好的候选者,但图像采集必须迅速才能捕获传播。
- 为绿色通道配置延时图像采集,包括四个视图字段,以 6 秒的间隔进行映像。
注:由于不同细胞类型间跛脚足突起速度的变异性高,应经验确定最佳帧速率。我们实验中使用的6秒的成像间隔是分析许多间皮细胞和上皮细胞的良好起点。然而,传播非常快的细胞(例如免疫细胞)可能需要更高的帧速率(更短的成像间隔)。细胞传播电影的最佳帧速率可确保后续帧之间突出的单元格边缘的 2-5 像素位移。考虑到用于识别细胞扩散高原的曲线拟合的准确性,最佳帧速率还应确保细胞扩散快速膨胀阶段细胞边缘位移的测量值为50-100。视场数量应根据曝光时间、采集点之间的距离和舞台移动速度进行调整。建议用户确定可以以所需的帧速率获得的视场的最大数量。 - 在确定合适的视场后,保存显微镜阶段的 X 和 Y 坐标。继续确定封面上彼此相对接近的另外三个视图字段。将显微镜阶段的坐标保存到每个所需的视场。
注意:强烈建议优化视场之间的舞台运动路径,以尽量减少任何不必要的样本移动。这种优化可以手动或自动执行。过多的样本运动会减慢采集速度,并可能导致细胞在下降时从视线中滚出。 - 以 6 秒帧速率获取图像 15 分钟,并保存文件。如果需要进行更多收购,则从步骤 4.6 开始重复。
5. 细胞扩散过程中细胞面积、圆形和突起动力学分析
- 为数据处理和分析准备图像
注:软件需要以.tiff格式显示图像,并需要像素大小作为输入参数。这两项要求都可以使用收购软件或斐济(在此协议中)实现。如果满足了这些要求,则继续执行步骤 5.2。- 安装斐济应用程序的最新版本(https://imagej.net/Fiji/Downloads)。
- 使用斐济打开延时图像。
- 通过选择 图像>属性复制图像的像素大小。将像素大小以 μm 复制并粘贴到记事本/Word 中。
- 为了分析细胞分布区域和圆形,将延时图像保存为蒂夫图像堆栈。自定义构建分析软件不支持专有文件格式。通过选择 文件>保存为>蒂夫保存单个单元格蒂夫图像堆栈。
- 安装Python IDE(间谍)和必要的包(皮辛普尔吉 和 蒂夫档案)用于数据处理和分析。
注:初始设置仅需要安装 Python 和包。- 延时电影将在 Spyder IDE 中使用自定义构建的 Python 脚本进行分析。要下载 Spyder IDE,请下载 Anaconda 分销商 (https://www.anaconda.com/products/individual),其中包括 Spyder IDE 和此分析所需的大多数库和包。
- 安装蟒蛇,并通过阿纳康达导航器发射间谍。
- 在 IPython 控制台选项卡(位于 Spyder 的右下角)中,复制并粘贴以下命令: 点安装 PySimpleGUI 并按下输入键。运行此命令将安装启动图形用户界面 (GUI) 所需的包。
- 在同一控制台中,复制并粘贴以下命令: 点安装 tifffile 并按 下输入 键。运行此命令将安装保存图像作为 tiff 文件所需的包。
- 从补充文件下载所有 Python 脚本或 GitHub 中更新最多的脚本:https://github.com/ernestiu/Cell-spreading-analysis.git
- 在细胞扩散过程中量化细胞面积和细胞形状因子
- 通过选择 Spyder 顶部面板中的打开文件选项或使用快捷方式 Ctrl + O打开主分析脚本"cell_spreading_GUI.py"。
- 通过在顶部面板中选择"运行文件"或使用快捷方式 F5打开单元格分布分析GUI。
- 单击"细胞传播区域"选项卡(图 3A)。
- 选择使用浏览按钮分析的蒂夫图像。
注:所选文件必须是蒂夫文件。 - 指定保存数据输出(例如,单元格掩码、值)的目的地目录。
- 指定数据输出设置:
- 保存口罩:保存在分割过程中生成的手机口罩。
- 导出数据:将包含所有分析数据的优秀电子表格(.xlsx)导出到目的地文件夹。
所有扩展单元的单元格区域、圆形和纵横比将作为 Excel 电子表格保存在目标文件夹中。细胞区域的计算方式如下: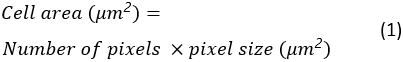
细胞循环度是衡量细胞与完美圆细胞的接近程度的指标。其计算方式如下: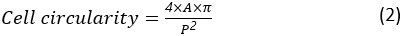
其中A和P分别是细胞区域和细胞周长。细胞的纵横比表示细胞的拉长程度。扩散单元应具有接近 1 的纵横比。纵横比计算如下: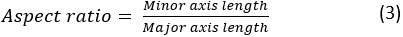
- 保存轮廓:将单元格边界轮廓叠加在目的地文件夹中。
- 指定分割设置
- 显示细分:在分析过程中显示 Spyder 控制台中的分割结果。
- 最小的细胞区域(μm2):输入细胞区域的最低值,包括连接开始阶段的细胞区域值。面积小于此阈值的对象将不被视为分布单元。此数字将影响分割过程。
- 指定图像参数。
- 采集间隔:在几秒钟内输入图像采集频率。
- 像素大小(μm):输入准备分析图像时记录的像素大小。
- 图像位深度:输入相机/探测器的位深度。
- 单击 "运行"。如果出现错误,将会在 Spyder 的控制台中显示错误消息。否则,图像分析过程将显示在控制台中。
注:显示在控制台/绘图部分(取决于 Spyder 设置)中的第一个图像显示视图领域中识别的所有单元格。放置在细胞周围的绿色框表示适合分割和分析的分布细胞。灰色框表示不适合分析的单元格。已识别的传播单元总数也将显示在控制台选项卡中。该软件绘制了细胞区域(蓝色)和细胞循环(红色)作为时间的函数。这些图表允许用户评估细胞分割的准确性。成功的分割为细胞区域产生单调增加曲线。要获得细胞扩散区域的代表性曲线,应手动从图形中删除滞后阶段。滞后阶段包括细胞开始扩散前的细胞区域测量。滞后阶段由快速波动表示,如细胞区域图(图 3C 右图)所示。
6. 使用基图对细胞扩散过程中的细胞边缘动力学进行量化
- 在运行分析之前,作物分布细胞的原始电影,以创建单个扩散细胞的时间系列。
- 使用斐济工具栏中的 矩形 工具手动选择封装 单个单元格的感兴趣区域 (ROI)。(为确保投资回报率完全封装扩散单元,请使用滚动功能在任何时候检查投资回报率。
- 右键单击投资回报率并选择 重复。
- 检查 重复堆栈 并单击 "确定"。
- 通过选择 Spyder 工具栏中的 "打开文件" 按钮或使用快捷方式 Ctrl + O打开主分析脚本"cell_spreading_GUI.py"。如果GUI已经打开,直接转到步骤6.3。
- 通过在顶部面板中选择 Run 文件 或使用快捷方式 F5 (图 3B) 打开单元格分布分析 GUI。
- 单击 "基莫克生成器"和"分析 "选项卡。
- 使用浏览按钮选择蒂夫图像进行分析。
注:自有文件格式, 例如 ,nd2、lif、禅宗,不支持脚本。 - 指定目标文件夹以保存输出数据(单元格掩码和值)。
- 指定输出设置。
- 导出数据:将优异的电子表格(.xlsx)导出到包含基莫格的相对单元边缘位置和缩回事件的目的地文件夹。
- 指定图像参数:
- 采集间隔:在几秒钟内输入图像采集频率。
- 像素大小(μm):输入在准备步骤 5.1.3 中分析图像时录制的像素大小。
- 最小的细胞区域(μm2):输入细胞区域的最低值,包括连接开始阶段的细胞区域值。面积小于此阈值的对象将不被视为分布单元。此数字将影响分割过程。
- 图像位深度:输入相机/探测器的位深度。
- 单击 "运行"。如果出现错误,将会在 Spyder 的控制台中显示错误消息。否则,将在控制台中显示突起动力学定量的摘要。将有4对缩还频率和突出速度测量,这是从从细胞的顶部,底部,左和右部分产生的4个kymgraph提取。收回频率计算如下:
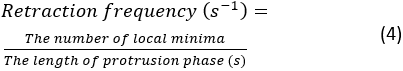
注:这个数字表明,在传播过程中,跛脚的缩回频率。平均突起速度通过突出开始和基图上的高原点之间的斜率来测量。分段后,控制台中将显示一个摘要基莫图。要保存摘要图,请右键单击该图并保存图像。
结果
上述协议描述了扩散细胞活细胞成像的实验程序,以及用于细胞扩散动力学定量分析的计算工具。计算工具可以采用低通量或高通量格式,以识别调节细胞前沿作用聚合机械的分子玩家。
实验程序的示意图表示在图1中描述。细胞扩散检测是在不朽的小鼠胚胎成纤维细胞上进行的,稳定地表达了标有eGFP25的Akt蛋白激酶的丛斯特林同源(P...
讨论
描述的细胞扩散检测允许连续跟踪形态变化(如细胞大小和形状)和细胞边缘运动(即突起速度和缩回频率),这是大多数细胞扩散协议19,24中缺少的功能。虽然常用的端点细胞扩散检测允许确定细胞扩散速度,但这些测定无法解决细胞边缘运动的时间动力学。缺乏时间信息限制了检测和量化跛脚突起-缩回周期变化的能力。
披露声明
作者没有什么可透露的。
致谢
这项工作得到了康诺特基金新调查员奖、加拿大创新基金会、NSERC发现赠款计划(授予RGPIN-2015-05114和RGPIN-2020-05881)、曼彻斯特大学和多伦多大学联合研究基金以及多伦多大学XSeed项目的支持。
材料
| Name | Company | Catalog Number | Comments |
| 0.05% Trypsin (0.05%), 0.53 mM EDTA | Wisent Bioproducts | 325-042-CL | |
| 10.0 cm Petri Dish, Polystyrene, TC Treated, Vented | Starstedt | 83.3902 | |
| 15 mL High Clarity PP Centrifuge Tube, Conical Bottom, with Dome Seal Screw Cap, Sterile | Falcon | 352097 | |
| 1-Well Chamlide CMS for 22 mm x 22 mm Coverslip | Quorum Technologies | CM-S22-1 | |
| 35 mm TC-treated Easy-Grip Style Cell Culture Dish | Falcon | 353001 | |
| 50 mL Centrifuge Tube, Transparent, Plug Seal | Nest | 602002 | |
| 6.0 cm Cell Culture Dishes Treated for Increased Cell Attachment, Sterile | VWR | 10861-658 | |
| Arp2/3 Complex Inhibitor I, CK-666 | Millipore Sigma | 182515 | |
| Camera, Prime 95B-25MM | Photometrics | ||
| Dimethyl Sulfoxide, Sterile | BioShop | DMS666 | |
| DMEM, 1x, 4.5 g/L Glucose, with L-Glutamine, Sodium Pyruvate and Phenol Red | Wisent Bioproducts | 319-005 CL | |
| DMEM/F-12, HEPES, No Phenol Red | Gibco | 11039021 | |
| D-PBS, 1X | Wisent Bioproducts | 311-425 CL | |
| Fetal Bovine Serum | Wisent Bioproducts | 080-110 | |
| Fiji Software | ImageJ | ||
| HEPES (1 M) | Gibco | 15630080 | |
| Human Plasma Fibronectin Purified Protein 1 mg | Millipore Sigma | FC010 | |
| Immersion Oil | Cargille | 16241 | |
| L-Glutamine Solution (200 mM) | Wisent Bioproducts | 609-065-EL | |
| MEM Non-Essential Amino Acids Solution (100X) | Gibco | 11140050 | |
| Micro Cover Glasses, Square, No. 11/2 22 x 22 mm | VWR | CA48366-227-1 | |
| Microscope Body, Eclipse Ti2-E | Nikon | ||
| Objective, CFI Plan Apo Lambda 60X Oil | Nikon | MRD01605 | |
| Penicillin-Streptomycin | Sigma | P4333 | |
| Spinning Disk, Crest Light V2 | CrestOptics | ||
| Spyder | Anaconda | ||
| Stage top incubator | Tokai Hit | ||
| Statistics Software, Prism | GraphPad | ||
| Tweezers, Style 2 | Electron Microscopy Sciences | 78326-42 |
参考文献
- Mullins, R. D., Heuser, J. A., Pollard, T. D. The interaction of Arp2/3 complex with actin: Nucleation, high affinity pointed end capping, and formation of branching networks of filaments. Proceedings of the National Academy of Sciences. 95 (11), 6181-6186 (1998).
- Yang, C., Czech, L., Gerboth, S., Kojima, S., Scita, G., Svitkina, T. Novel Roles of Formin mDia2 in Lamellipodia and Filopodia Formation in Motile Cells. PLoS Biology. 5 (11), 317 (2007).
- Mogilner, A., Oster, G. Cell motility driven by actin polymerization. Biophysical Journal. 71 (6), 3030-3045 (1996).
- Mogilner, A., Oster, G. Force Generation by Actin Polymerization II: The Elastic Ratchet and Tethered Filaments. Biophysical Journal. 84 (3), 1591-1605 (2003).
- Pollard, T. D., Borisy, G. G. Cellular Motility Driven by Assembly and Disassembly of Actin Filaments. Cell. 112 (4), 453-465 (2003).
- Wu, C., et al. Arp2/3 is critical for lamellipodia and response to extracellular matrix cues but is dispensable for chemotaxis. Cell. 148 (5), 973-987 (2012).
- Steffen, A., et al. Rac function is crucial for cell migration but is not required for spreading and focal adhesion formation. Journal of cell science. 126, 4572-4588 (2013).
- Gupton, S. L., et al. Cell migration without a lamellipodium. The Journal of Cell Biology. 168 (4), 619-631 (2005).
- Dimchev, V., et al. Induced Arp2/3 Complex Depletion Increases FMNL2/3 Formin Expression and Filopodia Formation. Frontiers in Cell and Developmental Biology. 9, 634708 (2021).
- Leithner, A., et al. Diversified actin protrusions promote environmental exploration but are dispensable for locomotion of leukocytes. Nature cell biology. 18 (11), 1253-1259 (2016).
- Giannone, G., Dubin-Thaler, B. J., Döbereiner, H. -. G., Kieffer, N., Bresnick, A. R., Sheetz, M. P. Periodic Lamellipodial Contractions Correlate with Rearward Actin Waves. Cell. 116 (3), 431-443 (2004).
- Dubin-Thaler, B. J., et al. Quantification of Cell Edge Velocities and Traction Forces Reveals Distinct Motility Modules during Cell Spreading. PLoS ONE. 3 (11), 3735 (2008).
- Suraneni, P., Rubinstein, B., Unruh, J. R., Durnin, M., Hanein, D., Li, R. The Arp2/3 complex is required for lamellipodia extension and directional fibroblast cell migration. The Journal of cell biology. 197 (2), 239-251 (2012).
- Wang, C., et al. Deconvolution of subcellular protrusion heterogeneity and the underlying actin regulator dynamics from live cell imaging. Nature Communications. 9 (1), 1688 (2018).
- Dimchev, G., et al. Lamellipodin tunes cell migration by stabilizing protrusions and promoting adhesion formation. Journal of cell science. 133 (7), 239020 (2020).
- Burnette, D. T., et al. A role for actin arcs in the leading-edge advance of migrating cells. Nature cell biology. 13 (4), 371-381 (2011).
- Yamada, K. M., Kennedy, D. W. Dualistic nature of adhesive protein function: fibronectin and its biologically active peptide fragments can autoinhibit fibronectin function. The Journal of Cell Biology. 99 (1), 29-36 (1984).
- Cai, Y., et al. Nonmuscle Myosin IIA-Dependent Force Inhibits Cell Spreading and Drives F-Actin Flow. Biophysical Journal. 91 (10), 3907-3920 (2006).
- Humphries, M. J. Cell adhesion assays. Molecular Biotechnology. 18 (1), 57-61 (2001).
- Cavalcanti-Adam, E. A., Volberg, T., Micoulet, A., Kessler, H., Geiger, B., Spatz, J. P. Cell Spreading and Focal Adhesion Dynamics Are Regulated by Spacing of Integrin Ligands. Biophysical Journal. 92 (8), 2964-2974 (2007).
- Dubin-Thaler, B. J., Giannone, G., Döbereiner, H. -. G., Sheetz, M. P. Nanometer Analysis of Cell Spreading on Matrix-Coated Surfaces Reveals Two Distinct Cell States and STEPs. Biophysical Journal. 86 (3), 1794-1806 (2004).
- Gauthier, N. C., Fardin, M. A., Roca-Cusachs, P., Sheetz, M. P. Temporary increase in plasma membrane tension coordinates the activation of exocytosis and contraction during cell spreading. Proceedings of the National Academy of Sciences. 108 (35), 14467-14472 (2011).
- Wolfenson, H., Iskratsch, T., Sheetz, M. P. Early Events in Cell Spreading as a Model for Quantitative Analysis of Biomechanical Events. Biophysical Journal. 107 (11), 2508-2514 (2014).
- Guan, J. -. L., Berrier, A. L., LaFlamme, S. E. Cell Migration, Developmental Methods and Protocols. Methods in molecular biology. 294, 55-68 (2004).
- Raucher, D., et al. Phosphatidylinositol 4,5-Bisphosphate Functions as a Second Messenger that Regulates Cytoskeleton-Plasma Membrane Adhesion. Cell. 100 (2), 221-228 (2000).
- Machacek, M., Danuser, G. Morphodynamic Profiling of Protrusion Phenotypes. Biophysical Journal. 90 (4), 1439-1452 (2006).
- Zack, G. W., Rogers, W. E., Latt, S. A. Automatic measurement of sister chromatid exchange frequency. The journal of histochemistry and cytochemistry official journal of the Histochemistry Society. 25 (7), 741-753 (1977).
- Bardsley, W. G., Aplin, J. D. Kinetic analysis of cell spreading. I. Theory and modelling of curves. Journal of cell science. 61, 365-373 (1983).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。