Method Article
探索病毒感染时的m6A和m5C表继转录组:HIV示例
摘要
RNA修饰在病毒感染中的作用才刚刚开始被探索,并可能突出新的病毒 - 宿主相互作用机制。在这项工作中,我们提供了一个管道来研究病毒感染背景下的m6A和m5C RNA修饰。
摘要
在过去几年中,RNA修饰在生物过程中的作用一直是越来越多的研究的重点,现在被称为表转录组学。其中,N6-甲基腺苷(m6A)和5-甲基胞嘧啶(m5C)RNA修饰已经在mRNA分子上被描述,并且可能在调节细胞过程中起作用。因此,表观转录组学是除转录组学分析之外还必须考虑的新调节层,因为它也可以通过暴露于任何化学或生物制剂(包括病毒感染)来改变或调节。
在这里,我们提出了一个工作流程,该工作流程允许在感染或未感染人类免疫缺陷病毒(HIV)的细胞中同时分析m6A和m5C标记的联合细胞和病毒表观转录组学景观。在从HIV感染和未感染的细胞中分离和片段化mRNA后,我们使用两种不同的程序:MeRIP-Seq,一种基于RNA免疫沉淀的技术,以富集含有m6A标记的RNA片段和BS-Seq,一种基于亚硫酸氢盐转化的技术,以单核苷酸分辨率鉴定m5C标记。在甲基化特异性捕获后,为高通量测序制备RNA文库。我们还开发了一个专用的生物信息学管道,以独立于其基础表达谱来鉴定差异甲基化(DM)转录本。
总体而言,该方法允许同时探索多个表观转录标记,并在病毒感染或任何其他细胞扰动时提供DM转录本的图谱。这种方法为识别新的参与者和细胞反应的新机制提供了新的机会,例如促进或限制病毒复制的细胞因子。
引言
众所周知,RNA分子可以被修饰,迄今为止已经描述了150多种转录后修饰1。它们包括将化学基团(主要是甲基)添加到RNA分子的嘧啶和嘌呤环的几乎任何位置2。这种转录后修饰已被证明在转移RNA(tRNA)和核糖体RNA(rRNA)中具有高度富集性,并且最近在mRNA分子上也有描述。
新技术的兴起,如下一代测序(NGS),以及识别明确化学修饰的特异性抗体的产生,首次允许在转录组范围内研究特定化学修饰的位置和频率。这些进步使人们更好地理解了RNA修饰,并绘制了mRNA分子上的几种修饰3,4。
虽然表观遗传学研究了DNA和组蛋白修饰在转录组调控中的作用,但表转录组学以类似的方式关注RNA修饰及其作用。对表观转录组修饰的研究为突出可能调谐各种细胞过程(即RNA剪接,导出,稳定性和翻译)的新调节机制提供了新的机会5。因此,最近的研究发现细胞和病毒RNA中病毒感染的许多表观转录组修饰也就不足为奇了6。迄今为止研究的病毒包括DNA和RNA病毒;其中,艾滋病毒可以被视为一个开创性的例子。总而言之,在病毒感染的背景下发现RNA甲基化可能允许研究尚未描述的病毒表达或复制机制,从而提供新的工具和靶标来控制它们7。
在HIV表观转录组学领域,病毒转录本的修饰已被广泛研究,并表明这种修饰的存在有利于病毒复制8,9,10,11,12,13。迄今为止,可以使用各种技术在转录组范围内检测表观转录组标记。最常用的m6A鉴定技术依赖于免疫沉淀技术,如MeRIP-Seq和miCLIP。虽然MeRIP-Seq依靠RNA片段化来捕获含有甲基化残基的片段,但miCLIP基于RNA-antibody UV交联时产生α-m6A抗体特异性特征突变,从而实现更精确的映射。
m5C修饰的检测可以通过类似于m6A检测(m5C RIP)的基于抗体的技术来实现,也可以通过亚硫酸氢盐转化或AZA-IP或miCLIP来实现。Aza-IP和m5C miCLIP都使用特定的甲基转移酶作为诱饵,在通过RNA甲基化时靶向RNA。在Aza-IP中,靶细胞暴露于5-氮杂胞苷,导致胞苷类似物5-氮杂胞苷位点随机引入新生RNA中。在miCLIP中,NSun2甲基转移酶经过基因改造,具有C271A突变14,15。
在这项工作中,我们专注于以HIV为模型,对受感染细胞中m6A和m5C修饰的双重表征。经过方法学优化,我们开发了一种结合甲基化RNA免疫沉淀(MeRIP)和RNA亚硫酸氢盐转化(BS)的工作流程,允许在细胞和病毒环境中同时在转录组范围内探索m6A和m5C表观转录组组标记。该工作流程可以在细胞RNA提取物以及从病毒颗粒中分离的RNA上实施。
甲基化RNA免疫沉淀(MeRIP)16 方法允许在转录组范围内研究m6A,并且迄今为止已有一系列m6A特异性抗体17。该方法包括使用m6A特异性抗体选择性捕获含有m6A的RNA片段。该技术的两个主要缺点是(i)分辨率有限,高度依赖于RNA片段的大小,因此提供了包含甲基化残基的近似位置和区域,以及(ii)执行分析所需的大量材料。在下面的优化方案中,我们将片段大小标准化为约150 nt,并将起始物质的量从10μg聚A选择的RNA(目前建议的起始材料量)减少到仅1μg的聚A选择RNA。我们还通过与m6A肽的竞争方法进行洗脱,而不是使用基于苯酚的技术或蛋白酶K的更常规和更不具体的洗脱方法,最大限度地提高了与特异性抗体结合的m6A RNA片段的回收效率。然而,这种基于RIP的测定的主要局限性仍然是次优分辨率,不允许鉴定精确修饰的A核苷酸。
目前可以使用两种不同的方法对m5C标记进行分析:基于RIP的方法,具有m5C特异性抗体和RNA亚硫酸氢盐转化。由于RIP在甲基化残基鉴定方面仅提供有限的分辨率,因此我们使用亚硫酸氢盐转化,可以提供单核苷酸分辨率。RNA暴露于亚硫酸氢盐(BS)会导致胞嘧啶脱氨,从而将胞嘧啶残基转化为尿嘧啶。因此,在RNA亚硫酸氢盐转化反应过程中,每个非甲基化的胞嘧啶被脱氨并转化为尿嘧啶,而在5位的甲基的存在具有保护作用,防止BS诱导的脱氨并保留胞嘧啶残基。基于BS的方法允许以单碱基分辨率检测m5C修饰的核苷酸,并评估每个转录本的甲基化频率,从而深入了解m5C修饰动力学18。然而,该技术的主要局限性依赖于甲基化残基的假阳性率。事实上,BS转化对具有可接触C残基的单链RNA有效。然而,紧密RNA二级结构的存在可能会掩盖N5C位置并阻碍BS转化,导致非甲基化C残基不转化为U残基,从而出现假阳性。为了规避这个问题并最大限度地减少假阳性率,我们应用了3轮变性和亚硫酸氢盐转化循环19。我们还在样品中引入了2个对照,以估计亚硫酸氢盐转化效率:我们增加了ERCC测序对照(非甲基化标准化和市售序列)20 以及聚A-耗尽RNA,以评估亚硫酸氢盐转化率,并通过RT-PCR验证是否存在已知且保守的甲基化位点C4447, 另一方面,在28S核糖体RNA上21。
在病毒学领域,将这两种表观转录组学研究方法与下一代测序和准确的生物信息学分析相结合,可以深入研究m6A和m5C动力学(即,病毒感染时可能发生的RNA修饰时间变化,并且可以发现一系列新的治疗相关靶点供临床使用)。
研究方案
1. 细胞制备
注意:根据细胞类型及其RNA含量,细胞的起始数量可能会有所不同。
- 有足够的细胞来获得200-500μg的总RNA或5-7μg的聚A选择RNA。例如,50 x 106 SupT1细胞在用苯酚基试剂提取时应产生约500μg的总RNA,因此对于测试的每个单独条件都是必需的。
- 根据实验设计准备所需数量的细胞,从而根据测试的条件数量(感染,时间点,治疗)。如果实验的目的是在感染后24小时获得未感染的细胞和HIV感染的细胞,则总共需要100×106 个细胞,一半用于非感染条件,一半用于感染条件。
2. 核糖核酸提取
- 从细胞:用苯酚 - 氯仿提取RNA
- 对于每种情况,通过离心收集细胞(例如,50×106)并丢弃上清液。
- 向每个50×106 细胞沉淀中加入5mL苯酚基试剂,并通过上下移液几次进行混合。
- 在室温下孵育5分钟,以允许完全裂解。裂解细胞可以储存在-80°C或直接处理。
注意:如果需要,细胞也可以在1.5 mL管中以每管10 x 10 6 个细胞的等分试样中分离,并在1 mL苯酚基试剂中裂解,以便更方便储存。 - 加入1 mL氯仿,倒置混合。
- 在室温下孵育3分钟。
- 在2,000×g和4°C下离心15分钟。
- 移出水相(上相)并转移到新管中。通过将管子倾斜45°并小心地移出溶液来完成水相的转移。
注:水相的量可能因样品而异,但应接近添加到样品中的氯仿量 (即 1 mL)。 不要转移任何相间或有机层! 使用锁相管或相位器管可以促进这一过程。 - 向水相中加入0.5mL的100%分子级异丙醇。
- 在-80°C下孵育1小时,使RNA沉淀。
- 在12,000×g和4°C下离心10分钟以沉淀沉淀RNA。
- 弃去上清液并将RNA沉淀重悬于1mL的75%分子生物学级乙醇中。短暂地涡旋。
- 在7,500×g和4°C下离心5分钟,弃去上清液。
- 风干沉淀15分钟。
- 将沉淀重悬于20μL无RNase水中并转移到新管中。
- 用额外的20μL水洗涤空管以最大限度地提高RNA回收率,并与前20μL体积一起池化。
- 使用分光光度计定量总RNA,并使用片段分析仪评估RNA质量。
- 来自病毒颗粒:使用基于柱的病毒RNA提取试剂盒提取RNA
注意:使用苯酚基试剂从病毒颗粒中提取RNA会导致低质量的病毒RNA和低质量的文库。因此,应支持基于色谱柱的 RNA 提取。使用载体RNA进行RNA洗脱和回收的RNA提取试剂盒不适合该程序,应避免使用。由于HIV RNA是多腺苷化的,直接提取RNA而不进一步分离mRNA就足以进入MeRIP-Seq和BS-Seq管道。通常,来自普遍感染细胞的1-2 mL病毒上清液应提供足够的RNA来执行整个工作流程。- 通过将150μLβ-巯基乙醇加入30mL裂解缓冲液来制备缓冲液。通过加入96 mL 100%乙醇来重建病毒洗涤缓冲液。
- 收集含病毒的上清液并离心以沉淀细胞碎片,以最大限度地减少细胞RNA污染。
- 将 1 mL 病毒上清液转移到 15 mL 管中。
- 将3 mL病毒RNA缓冲液加入1 mL病毒样品中,并通过涡旋混合。
- 将700μL样品转移到柱中,插入收集管中。
- 在室温下以13,000×g离心2分钟。
- 丢弃流式流出。
- 重复前面的3个步骤,直到整个样品被处理,因此所有RNA都被捕获在二氧化硅基质柱上。
- 向色谱柱中加入500μL病毒洗涤缓冲液。
- 在室温下以10,000×g离心1分钟。丢弃流式流出。
- 向色谱柱中加入200μL病毒洗涤缓冲液。
- 在室温下以10,000×g离心1分钟。丢弃流式流出。
- 将色谱柱放入空收集管中。
- 在室温下以10,000×g离心1分钟,以进一步丢弃任何剩余的洗涤缓冲液污染物。
- 小心地将色谱柱转移到1.5 mL管中。
- 将20μL不含DNA酶/RNase的水直接加入柱基质的中心,并在室温下以10,000×g离心30秒。
- 将另外10μL不含DNA酶/RNase的水直接加入柱基质的中心,然后再次离心30秒。
- 使用分光光度计定量总RNA,并使用片段分析仪评估RNA质量。
注意:如果检索到的RNA质量很高,RNA完整性/质量编号>9,则可以使用任何方法进行RNA提取。总RNA可以储存在-80°C直至进一步加工。
3. 通过聚A选择寡核苷酸(dT)25分离mRNA
注意:由于细胞提取物中存在高度甲基化的核糖体RNA,因此强烈建议通过rRNA消耗或优先通过poly-A阳性选择分离poly-A RNA。该步骤是可选的,应仅对细胞RNA样品执行,以获得更高分辨率的测序结果。如果分析非多聚腺苷化病毒RNA的甲基化,则倾向于rRNA耗尽而不是poly-A选择,或者最终对总RNA进行分析。
-
用于聚A捕获的磁珠制备
- 通过涡旋>30秒重悬Oligo(dT)25 磁珠浆料瓶。
- 将 200 μL 磁珠转移到 1.5 mL 管中。根据要处理的RNA样品的总量准备带有磁珠的试管数量。
注意:一管含有200μL Dynabead储备溶液对应于1mg磁珠,可以容纳75μg总RNA的样品。 - 将试管放在磁铁上1分钟,然后丢弃上清液。从磁铁上取下管子。
- 加入1 mL结合缓冲液(20 mM Tris-HCl,pH 7.5,1.0 M LiCl,2 mM EDTA),并通过涡旋重悬。将试管放在磁铁上1分钟,然后丢弃上清液。从磁铁上取下管子。重复。
- 将洗涤的磁珠重悬于100μL结合缓冲液中。
-
总核糖核酸制备
- 用不含RNase的水以0.75μg/μL的终浓度稀释总RNA,相当于75μg/100μL。
注意:如果RNA浓度较低,请按如下所述进行操作,而不修改体积。 - 通过每管分配100μLRNA样品,将总RNA等分到多个试管中。
- 向每个RNA样品中加入100μL结合缓冲液。
- 将总RNA加热至65°C2分钟以破坏二级结构。
- 立即放在冰上,直到准备好继续下一步。
注意:孵育时间可能因待处理样品的数量而异,但不应超过1小时,以避免任何RNA降解。
- 用不含RNase的水以0.75μg/μL的终浓度稀释总RNA,相当于75μg/100μL。
-
多边形 A 选择
- 向每个RNA管(从步骤3.2开始),加入100μL洗涤的磁珠(从步骤3.1开始)。
- 通过上下移液彻底混合,并在室温下结合在旋转轮上15分钟。
- 打开所有管子,将它们放在磁铁上1分钟,然后小心地除去所有上清液。
- 在新试管中回收上清液并留待第二轮RNA捕获(步骤3.3.14),以改善poly-A最终回收率。
- 从磁体中取出试管,加入200μL洗涤缓冲液(10mM Tris-HCl,pH 7.5,0.15 M LiCl,1mM EDTA)。通过小心移液4至5次混合。
- 将管放在磁铁上1分钟,然后丢弃上清液。
- 重复洗涤步骤一次(重复步骤3.3.5和3.3.6)。
- 加入20μL冰冷的10mM Tris-HCl,从珠中洗脱聚-A RNA。
- 在80°C孵育2分钟。
- 将试管放在磁铁上,并迅速将含有聚-A RNA的上清液转移到新的无RNase管中。将管子放在冰上。
- 重复洗脱步骤(步骤3.3.8至3.3.10)以提高产量。
- 用200μL洗涤缓冲液洗涤相同的珠子一次。通过小心移液4至5次混合。
- 放在磁铁上1分钟,丢弃洗涤缓冲液。
- 将步骤3.3.4中的流通装置添加到磁珠中,并重复从结合到洗脱的过程(步骤3.3.2至3.3.10)。暂时将RNA洗脱物保存在单独的试管中。
注意:可选地,再次将相当于步骤3.3.4的上清液保存在新管中,因为它可以用作对照。在程序结束时,通过乙醇沉淀或选择的基于色谱柱的方法(即RNA清洁和浓缩器)纯化和浓缩RNA。该样品对应于聚A耗尽的RNA样品,可用作亚硫酸氢盐转化的对照(步骤8.2.2)。 - 用分光光度计定量洗脱的RNA,并保持2μL等分试样,以使用片段分析仪进一步评估RNA质量。
注意:聚-A RNA可以储存在-80°C,直到需要。
4. 核糖核酸工作流程
- 将细胞多A RNA(mRNA)和病毒RNA样品分成2个等分试样,专用于各自的表观转录组学分析管道:
(i)5μg细胞mRNA或1μg病毒RNA用于MeRIP-Seq和输入对照(转到步骤5至7和步骤9)。
(ii)1μg细胞mRNA或500ng病毒RNA用于BS-Seq(转到步骤8和9)。
5. 核糖核酸片段化
注意:RNA片段化使用RNA片段化试剂进行,适用于MeRIP-Seq和对照RNA样品。这是一个非常重要的步骤,需要仔细优化才能获得介于 100-200 nt 之间的片段。
- 将mRNA的总体积分成0.2 mL PCR管和18μL mRNA /管。
注意:快速工作。不要一次使用超过8个样品以获得可重复的结果。扩大卷规模并不能保证可重复和均匀的碎片。 - 在70°C下加热热循环仪。
- 在每个PCR管的边缘加入2μL片段化试剂。
- 关闭试管并向下旋转(以便试剂同时与8个管的RNA接触)。
- 将样品在预热的热循环器中在70°C下孵育15分钟。
- 孵育结束后,在每个管中快速加入2μL停止溶液。
- 旋转下来,坐在冰上,直到准备好继续下一步。
注意:孵育时间可能因待处理样品的数量而异,但不应超过1小时,以避免任何RNA降解。 - 对所有样品重复该过程(如果等分试样超过8个)。
- 将试管混合在一起,并使用RNA清洁和浓缩器试剂盒(步骤6)或任何定制的基于柱的试剂盒进行RNA纯化,以去除缓冲液并回收水中干净的片段RNA。
6. 核糖核酸纯化
注意:此步骤可以通过乙醇沉淀或使用任何一种基于色谱柱的RNA纯化和浓缩方法(即RNA清洁和浓缩器)进行。
- 将纯化的RNA洗脱或重悬于总体积为50-75μL的DNA酶/不含RNA酶的水中。
注意:如果使用基于色谱柱的方法,强烈建议进行两轮洗脱,以确保最大回收率。 - 使用分光光度计定量纯化的片段化mRNA,并使用片段分析仪评估RNA质量。
- 保留100 ng片段化mRNA作为文库制备和测序的输入对照(转到步骤9)。剩余的片段化mRNA(最小2.5μg)可用于MeRIP(转到步骤7.2)。
7. 美里普
注意:使用特定的抗m6A抗体(测试条件)或使用抗IgG抗体(阴性对照),每个免疫沉淀(IP)至少需要2.5μg片段mRNA。
- 用于免疫沉淀的磁珠制备
- 对于每个样品,通过用3.2 mL无核酸酶水稀释800μLmRNA IP缓冲液5x(50mM Tris-HCl pH 7.4,750 mM NaCl,0.5%Igepal CA-630和无核酸酶水),在新的锥形管中制备4mL 1x IP缓冲液。
注意:至少需要2次反应(一次测试和一次IgG对照)。 - 将管子放在冰上。
- 标记适当数量的1.5 mL微量离心管,以获得所需IP反应的数量:
n个管(测试)用于抗m6A抗体。
n管(阴性对照)用于正常小鼠IgG。 - 通过反转和涡旋重悬磁珠(例如,Magna ChIP蛋白A / G)。不应看到任何珠团。
- 对于计划的每次反应, 将25μL磁珠转移到微量离心管中。
- 相对于所用磁珠的原始体积(即, 每25 μL磁珠250 μL 1x IP缓冲液),添加10倍以上的1x IP缓冲液(从步骤7.1.1开始)。
- 通过轻轻地上下移液几次来混合珠子,以完全重悬。
- 将管子放在磁选机上1分钟。
- 取出并丢弃上清液,确保不吸出任何磁珠。从磁铁上取下管子。
- 重复洗涤步骤(步骤7.1.6至7.1.9)。
- 将磁珠重悬于每 25 μL 原体积磁珠的 100 μL 1x IP 缓冲液中。
- 每25μL原始体积的磁珠加入5μL抗体(1μg/ μL)。
n管(测试)与抗m6A抗体(克隆17-3-4-1)[1μg/μL]。
n管(阴性对照)与正常小鼠IgG(1μg/ μL)。 - 在室温下在旋转轮上孵育30分钟,以使抗体与磁珠偶联。
- 将管子放在磁选机上1分钟。丢弃上清液。从磁体中取出试管,将抗体 - 微球混合物重悬于100μL1x IP缓冲液中。
- 对于每个样品,通过用3.2 mL无核酸酶水稀释800μLmRNA IP缓冲液5x(50mM Tris-HCl pH 7.4,750 mM NaCl,0.5%Igepal CA-630和无核酸酶水),在新的锥形管中制备4mL 1x IP缓冲液。
- 核糖核酸免疫沉淀
- 为每个2.5μgmRNA样品制备500μLRI反应混合物,如下所示:2.5μg在100μL片段RNA中(从步骤6.12开始);295μL无核酸酶水;5 μL 40 U / μL RNase抑制剂;和 100 μL 5x IP 缓冲液。
- 向每个抗体 - 微球混合物中加入500μLRI反应混合物(步骤7.1.14〜100μL)。通过轻轻移液几次以完全重悬珠子来混合。放在冰上。
- 将所有RIP管在旋转轮上孵育2小时,温度为4°C。
- 短暂地离心MeRIP反应,以从盖子和管侧旋转液滴。将管子放在磁选机上1分钟。
- 将上清液转移到新的离心管中,小心不要干扰磁珠 。
注意:可以保留流通作为控制,以验证 RIP 效率(请转到步骤 7.3.9)。 - 从磁铁上取下管子。通过加入500μL冷1x IP缓冲液洗涤珠子。通过轻轻移液几次来混合珠子,以完全重悬珠子。
- 将管子放在磁选机上1分钟,然后丢弃上清液。
- 重复洗涤程序(步骤7.2.6-7.2.7)两次,共3次洗涤。
- 将试管放在冰上,然后立即进行洗脱。
- 洗脱
- 通过将10mg N6-甲基腺苷,5'-单磷酸钠(m6A)溶解在1.3mL无核酸酶水中来制备20mM m6A溶液。准备150μL等分试样并储存在-20°C。
- 对于每个样品(测试和对照):通过混合以下组分制备225μL洗脱缓冲液:45μL5x IP缓冲液,75μL20mM m6A,3.5μL40U / μL RNase抑制剂和101.5μL无核酸酶水。
- 向微珠(步骤7.2.9)中加入100μL洗脱缓冲液(从步骤7.2.9开始)。通过轻轻移液几次以完全重悬珠子进行混合。
- 将所有管孵育1小时,并在4°C下在摇臂上连续摇晃。
- 短暂离心RIP反应,从盖子和管侧旋转液滴。将管子放在磁选机上1分钟。
- 将含有洗脱RNA片段的上清液转移到新的1.5mL微量离心管中。注意不要吸出珠子,因为这会增加背景噪音。
- 重复洗脱步骤(7.3.3至7.3.6),再次加入100μL洗脱缓冲液,在4°C下孵育1小时,并在磁分离后收集洗脱液。
- 将来自同一样品的所有洗脱液混合(总洗脱体积应为200μL)。
- 通过乙醇沉淀或通过选择的基于色谱柱的方法(即RNA清洁和浓缩器)纯化洗脱的RNA和流过(可选,从步骤7.2.5开始)。
- 使用高灵敏度检测试剂盒,使用片段分析仪评估流过和洗脱样品的RNA数量和质量。如果RNA的质量令人满意,请继续进行文库制备和高通量测序(步骤9)。
注意:在MeRIP上检索的RNA量非常低,并且迫切需要高灵敏度检测试剂盒以确保定量。如果没有可用的生物分析仪,则可以盲目地进行文库制备。
8. RNA亚硫酸氢盐转化
- 控制和试剂制备
- ERCC混合峰值对照:按照制造商的说明添加ERCC混合物,建议将0.5μL未稀释的ERCC混合物加入500ng mRNA中。这种控制可以帮助评估亚硫酸氢盐转化的效率。
- 以1/1000的比例(即500 pg的聚A耗尽RNA用于500 ng的mRNA)刺突Poly-A耗尽RNA(从步骤3.3.14开始)。该样品富含核糖体RNA,因此应含有28S rRNA,这是亚硫酸氢盐转化的阳性对照。
注意:总RNA也可以用作阳性对照,而不是多A耗尽的RNA。 - 使用RNA甲基化试剂盒(例如,Zymo EZ)进行亚硫酸氢盐转化。
- RNA洗涤缓冲液:使用前将48 mL 100%乙醇(或52 mL 95%乙醇)加入12 mL RNA洗涤缓冲液浓缩液中。
- 亚硫酸氢盐 转化率
注:亚硫酸氢盐转化是使用市售的RNA亚硫酸氢盐转化试剂盒按照制造商的程序进行的,如下所述。- 在0.2 mL PCR管中,加入1000ng的mRNA(或300至1000ng之间)。添加峰值对照:1μLERCC混合物(步骤8.1.1)和1000pg聚A耗尽RNA(步骤8.1.2)。用不含DNA酶/RNase的水完成高达20μL的体积。
- 向每个20μLRNA样品中加入130μLRNA转化试剂。
- 通过上下移液来混合样品。
- 短暂旋转,以确保管的盖子或侧面没有液滴。
- 将PCR管置于热循环仪中并执行以下步骤:在70°C下变性5分钟;在54°C下转换45分钟;重复变性和转化步骤共3个周期;然后无限期地保持在4°C。
注:三个变性和亚硫酸氢盐转化循环可确保样品的亚硫酸氢盐转化完成。样品可以储存在-80°C或直接处理。 - 继续进行柱内脱硫。将色谱柱放入空的收集管中,并向色谱柱中加入250μLRNA结合缓冲液。
- 将样品(步骤8.2.5中的约150μL)上样到含有RNA结合缓冲液的柱中,并通过上下移液进行混合。
- 向柱中的样品-RNA结合缓冲液混合物中加入400μL95-100%乙醇。合上盖子,通过倒置柱子几次立即混合。
- 全速离心(≥ 10,000 x g)30 秒。丢弃流式流出。
- 向色谱柱中加入200μLRNA洗涤缓冲液,全速离心30秒。
- 向色谱柱中加入200μLRNA脱硫缓冲液,并在室温下孵育30分钟。孵育后,全速离心30秒。丢弃流式流出。
- 向色谱柱中加入400μLRNA洗涤缓冲液,全速离心30秒。用额外的400μLRNA洗涤缓冲液重复洗涤步骤。丢弃流式流出。
- 在清空的收集管中全速离心柱2分钟。将色谱柱转移到无RNase的管中。
- 将≥10μL不含DNA酶/RNase的水直接加入柱基质中,并在室温下孵育1分钟。全速离心30秒。
注意:我们通常在20μL的体积中洗脱。洗脱的RNA可以立即使用或在-20°C下储存长达3个月。长期储存,请保持在-80°C。 - 取出2.5 μL用于片段分析仪对RNA质量和数量的评估,然后进行文库制备和高通量测序(步骤9)。
- 取4μL转化RNA用于亚硫酸氢盐转化控制效率(步骤8.3)。
- 通过 RT-PCR 控制亚硫酸氢盐转化率
注意:此步骤可确保在进行测序之前亚硫酸氢盐转化成功。来自 智人的 28S核糖体RNA将被用作RNA甲基化分析的阳性对照,因为位置4447(GenBank加入#NR_003287)的C残基被描述为100%甲基化。
引物序列:
H 28SF 底漆: 5'-GGGGTTTTAYGATTTTTTTTTGTTGG-3'
H 28SR 底漆: 5'-CCAACTCACRTTCCCTATTAATAATAAAC-3'- 使用高容量cDNA逆转录试剂盒制备逆转录(RT)反应混合物。在冰上解冻试剂盒组件,并在冰上制备RT预混液,如下所示:
4 μL亚硫酸氢盐转化RNA(来自步骤8.2.14):
2 μL 10xRT缓冲液
0.8 μL 25x dNTP 混合物 [100 mM]
2 μL 10x 室温随机引物
1 μL 多文逆转录酶
1 μL RNase 抑制剂
9.2 μL 无核酸酶 H2O
注意:每次RT反应应包含20μL最终体积在0.2mL PCR管中。 - 将管放入热循环仪中,采用以下RT程序:25°C10分钟;37 °C 120分钟;85 °C持续5分钟;然后在4°C下无限期地。
- 准备PCR反应,用PCR校对酶特异性扩增28S rRNA。在冰上解冻试剂盒组件,轻轻涡旋并短暂离心。在冰上或冰冷的金属板架上制备PCR预混液,如下所示:
0.6 μL 10 μM H 28SF 底漆
0.6 μL 10 μM H 28SF 底漆
6.5 μL 模板 cDNA
22.5 μL DNA聚合酶预混液
注意:每个PCR反应应包含20μL最终体积在0.2mL PCR管中。 - 将试管放入热循环仪中,采用以下PCR程序:在95°C下初始变性5分钟;45个变性(95°C持续15 s),退火(57°C持续30 s)和伸长率(72°C持续15 s)循环,在72°C下最终伸长10分钟,然后在4°C无限期保持。
- 在2%琼脂糖凝胶上运行10μL反应。预期波段大小为 130 - 200 bp。
- 使用高容量cDNA逆转录试剂盒制备逆转录(RT)反应混合物。在冰上解冻试剂盒组件,并在冰上制备RT预混液,如下所示:
- PCR产物的测序
- 使用所选的基于色谱柱的方法纯化PCR产物,以除去酶和dNTP残基,并在至少20μL无DNase/RNase水中洗脱扩增的DNA。
- 用分光光度计量化纯化的DNA。
- 测序反应
- 使用40 ng PCR产物/测序反应。
- 使用H 28SF和H 28SR引物在两个方向上进行序列。
- 将序列与已知的未转化序列(28S核糖体N5(RNA28SN5))对齐。检查位置 C4447 是否存在 C 残留物,以及其他地方是否存在 T 残留物而不是 C 残留物。
9. 文库制备和高通量测序
- 使用mRNA试剂盒(例如,Illumina TruSeq Stranded)准备用于测序的文库,在Elute-Prime-Fragment步骤处启动实验方案并遵循制造商说明。
- 然而,对于输入的RNA-Seq和MeRIP-Seq样品,将样品在80°C下孵育2分钟,仅质化,但不会进一步片段化它们。
- 使用Illumina平台进行测序。测序反应可以根据偏好和实验设计进行,无论是单端还是成对端,长度至少为100 nt。
第10章 生物信息学分析
- m6A 数据处理
- 运行 FASTQC24 以评估 m6A 中的读取质量,并从排序中输入 FASTQ 文件。
- 运行 Atropos25 以从读取中修剪低质量的端部和适配器序列。在运行 Atropos 中设置以下参数。
- 删除以下适配器序列:AGATCGGAAGAG、CTCTTCCGATCT、AACACTCTTTCCCT、AGATCGGAAGAGCG、AGGGAAAGAGTGTT、CGCTCTTCCGATCT。
- 使用以下 Phred 质量截止值:5,用于修剪制造商指定的低质量端(https://support.illumina.com/downloads/illumina-adapter-sequences-document-1000000002694.html)。
- 修剪后使用以下最小读取长度:25 个碱基对。
- 将 GRh38 人类基因组和 HIV [集成线性 pNL4-3Env-GFP] 参考合并为 FASTA 格式。
- 使用 HISAT226 为合并的引用编制索引。
- 对修剪的读取运行 HISAT2 以与索引引用对齐。使用默认的 HISAT 参数。
- 使用SAMtools27对对齐的读取进行排序和索引。
- 运行 SAMtools stat 和 Qualimap 228, 对序库进行对准后质量检查。
- (可选)使用 multiQC29 收集和汇总上一步中的质量度量值。
- HIV基因组在5'LTR和3'LTR中具有同源的634 bp序列:使用SAMtools重新对齐从5'LTR读取到相应的3'LTR区域的多映射。
- 为了识别m6A峰值,请运行峰值调用软件MACS230 (v 2.1.2)。仔细选择MACS2运行参数,以确保RNA-Seq数据的正确功能,因为峰调用会受到基因表达水平的影响,而短外显子可能会被误称为峰。因此,必须从m6A信号中减去输入信号,而无需MACS2对基于DNA的数据进行常规平滑处理。将以下参数应用于 MACS2 中的"callpeak"子命令:
-keep-dup auto(控制 MACS2 对重复读取的行为,"auto"允许 MACS 根据二项分布(使用 1e-5 作为 p 值截止值)计算完全相同位置的最大读取次数)
-g 2.7e9 (人类基因组的大小,以 bp 为单位)
-q 0.01(调用显著峰值的最小 FDR 截止值)
-nomodel(绕过为ChIP-Seq实验量身定制的移位模型的构建)
-slocal 0
-llocal 0(将此参数和前一个参数设置为 0 允许 MACS2 直接减去输入读取 m6A 读取的内容,而无需平滑)
-extsize 100(片段的平均长度,单位为 bp)
-乙 - 运行 MACS2 的差分峰值调用子命令"bdgdiff"以比较受感染样本与未感染样本。'bdgdiff' 将上一步中由 'callpeak' 生成的 bedGraph 文件作为输入。对于每个时间点,使用"bdgdiff"运行感染样本与未感染样本的比较,从m6A信号中减去相应的输入信号并提供附加参数:-g 60 -l 120。
- m5C 数据处理
- 运行 Cutadapt31 以从原始读取中修剪适配器序列,并使用以下参数:
适配器"AGATCGGAAGAGCACACGTCTGAAC"
-最小长度 = 25。 - 使用seqkit32反向补充修剪后的读数,因为测序协议从反向链产生读数。
- 运行 FastQC 以检查读取质量。
- 以FASTA格式合并GRh38人类基因组和HIV [集成线性pNL4-3Env-GFP]参考。
- 将合并的引用与来自 meRanTK 包33 的应用程序 meRanGh 编制索引。
- 使用以下参数与 meRanGh 对齐:
-UN 允许将未映射的读取写入输出文件
-MM 允许将多映射读取写入输出文件
-bg 用于在床上的输出图形
-mbgc 10 按覆盖范围筛选报告的区域(至少读取 10 次覆盖率) - HIV基因组在5'LTR和3'LTR中具有同源的634 bp序列:使用SAMtools将多图分析从5'LTR重新排列到相应的3'LTR区域。
- 通过 meRanTK 提供的 meRanCall 工具运行甲基化调用,并使用以下参数:
-rl = 126,读取长度
-ei = 0.1,甲基化速率p值计算的误差区间
-cr = 0.99,预期转换 - 运行 MeRanTK 的实用工具 estimateSizeFactors.pl,用于估计每个样本的大小因子。大小因子将在下一步中用作参数。
- 运行 MeRanCompare,用于在 12、24 和 36 小时内对未感染与受感染进行差异甲基化分析。应用以下参数:显著性值 .01 作为报告的最小阈值和上一步中的大小因子。
- 运行 Cutadapt31 以从原始读取中修剪适配器序列,并使用以下参数:
结果
该工作流程已被证明可用于研究m6A和m5C甲基化在HIV感染中的作用。为此,我们使用CD4 + T细胞系模型(SupT1),我们要么感染HIV,要么未经治疗。我们开始了每个条件5000万个细胞的工作流程,并获得了平均500μg的总RNA,RNA质量数为10(图1A-B)。在poly-A选择后,我们为每个条件检索了10至12μg的mRNA(约占总RNA的2%)(图1B)。在这一点上,我们使用5μg聚A选择RNA用于MeRIP-Seq管道,1μg用于BS-Seq管道。由于HIV RNA是多腺苷基化的,因此不需要进一步的作用,可以直接应用MeRIP-Seq和BS-Seq程序。
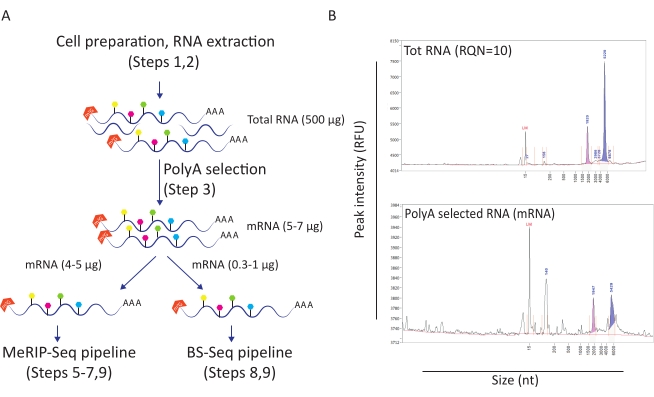
图1:用于下游应用的RNA制备。A) 描述同时用于 MeRIP-Seq 和 BS-Seq 管道的 RNA 制备和分布的工作流程。每个填充的六边形形状代表一种RNA修饰类型,例如m6A(绿色)或m5C(粉红色)。指示了进行实验所需的RNA物质量。 B)代表性结果,描绘了总RNA提取(上图)和poly-A选择(下图)的预期RNA分布曲线(大小和数量)。将样品装有标准灵敏度试剂盒的片段分析仪上,以便在进入特定的MeRIP-Seq和BS-Seq程序之前评估RNA质量。RQN: RNA质量数;nt:核苷酸。请点击此处查看此图的放大版本。
MeRIP-Seq管道是一种基于RNA免疫沉淀的技术,可以研究沿RNA分子的m6A修饰。为此,首先将RNA片段化,然后与偶联到磁珠上的m6A特异性抗体一起孵育以进行免疫沉淀和捕获。然后对富含MerIP的RNA片段和未触及的(输入)部分进行测序和比较,以鉴定m6A修饰的RNA区域,从而鉴定m6A甲基化的转录本(图2A)。该技术的分辨率取决于RNA片段化的效率。事实上,较短的片段允许更精确地定位m6A残基。在这里,将细胞多A选择的RNA和病毒RNA在20μL最终体积中与RNA片段化缓冲液在15分钟内进行基于离子的片段化,以获得100-150 nt的RNA片段。从5μgmRNA开始,我们回收了4.5μg片段RNA,相当于90%的回收率(图2B)。我们使用100 ng片段化、纯化的RNA作为输入对照,直接进行文库制备和测序。剩余的RNA(~4.4μg)根据MeRIP-Seq管道进行处理,该管道首先孵育片段RNA,其微球与抗m6A特异性抗体或抗IgG抗体结合作为对照。2.5μg片段RNA的m6A特异性RIP(MeRIP)允许检索约15ng经过文库制备和测序的m6A富集物质(图2B)。正如预期的那样,具有抗IgG控制的RIP没有产生足够的RNA以进行进一步分析(图2B)。
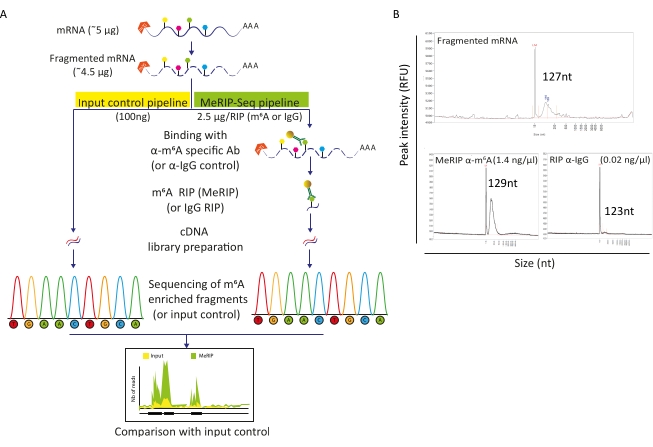
图 2:MeRIP-Seq 管道。A) MeRIP-Seq 工作流程和输入控制的示意图。在poly-A选择后,将样品片段化成120-150 nt块,并在测序前直接进行测序(100ng,输入对照),或用于RNA免疫沉淀(2.5μg,RIP),抗m6A特异性抗体或抗IgG抗体作为阴性对照。B)代表性结果显示片段化(上图)和RIP(下图,MeRIP:左,IgG对照:右)时的预期RNA分布曲线(大小和数量)。将样品加载到片段分析仪上以评估RNA质量和浓度,然后进一步处理以进行文库制备和测序。使用RNA标准灵敏度试剂盒进行片段化RNA分析,而免疫沉淀RNA使用高灵敏度试剂盒。请点击此处查看此图的放大版本。
BS-Seq管道允许在核苷酸分辨率下探索m5C RNA修饰,并导致m5C甲基化转录本的鉴定。在亚硫酸氢盐转化时,非甲基化的胞嘧啶转化为尿嘧啶,而甲基化的胞嘧啶保持不变(图3A)。由于亚硫酸氢盐转化过程的苛刻条件 (即 高温和低pH值),转化的mRNA高度降解(图3B),但这不会干扰文库制备和测序。亚硫酸氢盐的转化仅对单链RNA有效,因此可能受到二级双链RNA结构的阻碍。为了评估C-U转换的效率,我们引入了两个控件。作为阳性对照,我们利用了先前描述的在28S rRNA23的位置C4447中存在高度甲基化的胞嘧啶。在RT-PCR扩增和对甲基化位点周围的200 bp片段进行测序后,我们可以观察到所有胞嘧啶都成功转化为尿嘧啶,从而在DNA序列中作为胸腺嘧啶出现,除了位于4447位置的胞嘧啶保持不变。作为亚硫酸氢盐转化率的控制,我们使用市售的合成ERCC RNA序列。该混合物由已知的,非甲基化和多腺苷化的RNA序列组成,具有各种二级结构和长度。在文库制备和测序时,我们专注于这些ERCC序列来计算转化率,这可以通过计算所有ERCC序列和每个样品中总C残基中转化的C的数量来执行。我们获得了99.5%的转化率,证实了亚硫酸氢盐转化反应的效率和成功(图3D)。
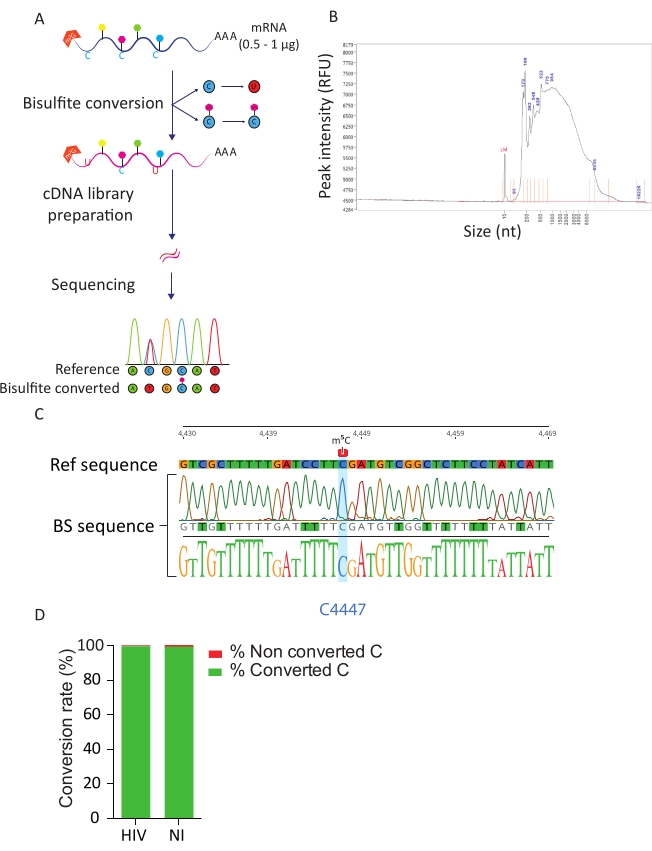
图 3:BS-Seq 管道。A) BS-Seq工作流程的示意图。在选择聚A时,样品暴露于亚硫酸氢盐中,导致非甲基化C残基的C到U转化(由于脱氨)。相反,甲基化C残基(m5C)不受亚硫酸氢盐处理的影响,保持不变。 B)亚硫酸氢盐转化RNA分布曲线(大小和数量)的代表性结果,在用标准灵敏度试剂盒的片段分析仪上分析。 C)电泳图显示28S rRNA中100%甲基化C周围区域4447处的RT-PCR扩增子的代表性测序结果(以蓝色突出显示)。相反,由于亚硫酸氢盐转化成功,参考序列的C残基被鉴定为扩增子序列中的T残基。 D)通过分析HIV感染和非感染细胞中的ERCC尖峰序列来评估C-U转化率。平均转化率为99.5%。 请点击此处查看此图的放大版本。
进一步处理富含M6A的样品,亚硫酸氢盐转化样品和输入对照,以进行文库制备,测序和生物信息学分析(图4)。根据实验设计和所解决的生物学问题,可以应用多种生物信息学分析。作为原理证明,我们展示了一种潜在应用(即差异甲基化分析)的代表性结果, 该分析侧重于鉴定HIV感染时诱导的差异甲基化转录本。简而言之,我们研究了转录本在非感染细胞和HIV感染细胞中的m6A或m5C甲基化水平,独立于它们的基因表达水平,以进一步了解RNA甲基化在病毒生命周期中的作用。在基因表达归一化后,我们鉴定ZNF469转录本根据感染状态被差异化为m6A甲基化,实际上该转录本在未感染细胞中未甲基化,而它在HIV感染时显示出几个甲基化峰(图5A)。对m5C的类似差异甲基化分析显示,PHLPP1转录本含有几种甲基化残基,这些残基在HIV条件下往往更频繁地甲基化(图5B)。在这种情况下,两项分析都表明HIV感染会影响细胞表转录组。
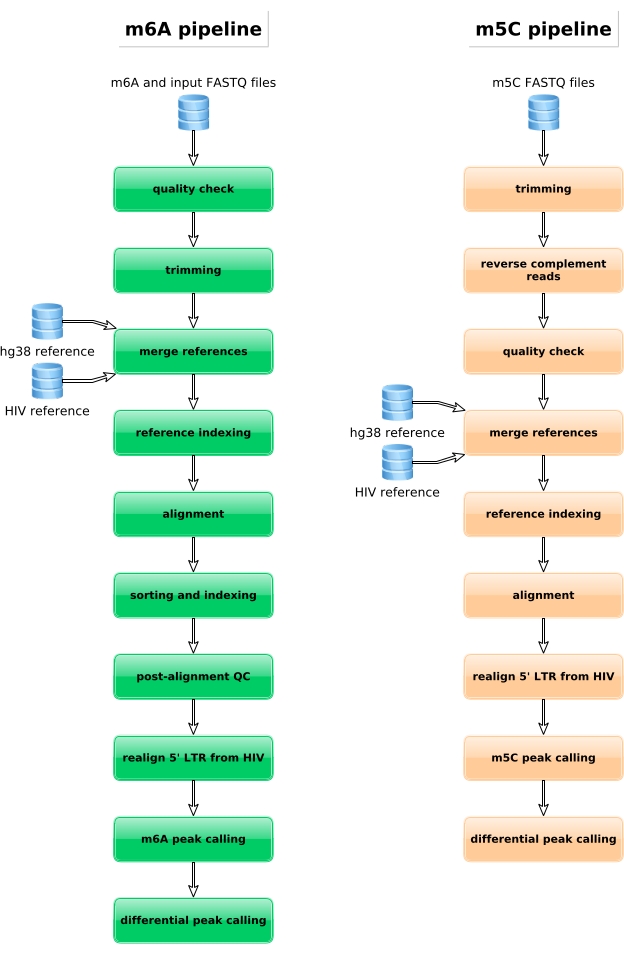
图 4:用于分析 m6A 和 m5C 数据的生物信息学工作流程的示意图。 请单击此处查看此图的大图。
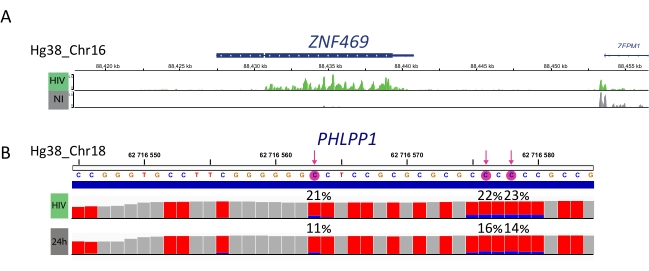
图5:感染时差异甲基化转录本的示例。A)代表性结果显示HIV感染(绿色)和非感染(灰色)细胞中ZNF459转录本的m6A甲基化。峰值强度(在输入表达式减法时)显示在 y 轴上,并沿 x 轴显示在染色体中的位置。差异甲基化分析显示,ZFN469转录本在HIV感染时被高甲基化。 B)m5C甲基化基因在HIV感染(上泳道)和非感染(下泳道)细胞中的代表性结果。每个条形的高度表示每个核苷酸的读取次数,并允许进行覆盖率评估。每个C残基用红色表示,甲基化C的比例用蓝色表示。在每个C残基上方报告精确的甲基化速率(%)。箭头突出显示了具有统计学意义的差异甲基化C.使用IGV查看器可视化样品。 请点击此处查看此图的放大版本。
讨论
RNA修饰在病毒感染中的作用在很大程度上仍然是未知的。更好地了解表观转录组修饰在病毒感染中的作用可能有助于寻找新的抗病毒治疗靶点。
在这项工作中,我们提供了一个完整的工作流程,可以研究受感染细胞的m6A和m5C表观转录组。根据生物学问题,我们建议使用聚A选择的RNA作为起始材料。虽然是可选的,但由于管道可以与总RNA一起使用,但重要的是要记住,rRNA和小RNA都是高度修饰的,并且含有大量甲基化残基。这可能导致有意义的测序数据的质量和数量下降。
但是,如果研究的重点是非聚腺苷酸化RNA,则应调整RNA提取步骤,以避免丢弃小RNA(在基于柱的RNA提取的情况下),并优先使用核糖体消耗技术而不是poly-A选择进入管道。
为了确保高质量的RNA,正确的片段化以及合适的m6A富集和BS转化RNA质量用于文库制备,我们强烈建议使用片段分析仪或生物分析仪。但是,此设备并非始终可用。作为替代方案,RNA的质量,mRNA和片段RNA的大小也可以通过琼脂糖凝胶上的可视化来评估。或者,可以在不事先评估RNA数量的情况下进行文库制备。
我们使用基于抗体的MeRIP-Seq16技术来探索m6A表观转录组学景观。该技术基于RNA免疫沉淀,是成功的;但是,有些步骤需要仔细优化,并且可能很关键。虽然m6A甲基化被描述为主要发生在共识序列RRA * CH中,但该基序在mRNA分子上非常频繁,并且不允许精确鉴定甲基化位点。因此,实现可重复且一致的RNA片段化,产生小RNA片段,以提高基于RIP的分辨率至关重要。在该协议中,我们建议使用优化的程序,在我们的实验环境中提供可重复和一致的结果;但是,此碎片步骤可能需要根据特定的示例特征进行进一步优化。
最近描述了一种允许m6A直接测序的新技术。它基于使用特定的逆转录酶变体,这些变体表现出独特的RT特征,作为对遇到m6A RNA修饰的反应24。经过仔细优化,该技术可以规避MeRIP-Seq面临的主要限制(减少初始材料的数量并允许更高的分辨率)。为了探索m5C修饰,我们决定使用亚硫酸氢盐转化技术,以便在核苷酸分辨率下检测修饰的C残基。为了降低由于RNA二级结构的存在而导致的假阳性率,我们进行了3个变性/亚硫酸氢盐转化循环,并通过使用ERCC尖峰对照进一步控制亚硫酸氢盐转化率性能。与该技术相关的局限性之一是亚硫酸氢盐转化非常苛刻,三个变性/亚硫酸氢盐转化循环可能会降解某些RNA,从而降低分辨率。但是,在我们的设置中,我们选择采用可能略低的分辨率,以提高数据集的质量。
由于这些优化和对照,我们能够提供可靠和健全的工作流程,可以利用该工作流程来研究在病毒感染,宿主 - 病原体相互作用或任何暴露于特定治疗的背景下的表观转录组学景观及其改变。
披露声明
作者没有什么可透露的。
致谢
这项工作得到了瑞士国家科学基金会的支持(赠款31003A_166412和314730_188877)。
材料
| Name | Company | Catalog Number | Comments |
| AccuPrime Pfx SuperMix | Invitrogen | 12344-040 | |
| anti-m6A antibody _Clone 17-3-4-1 | Millipore | MABE1006 | |
| Chloroform | Merck | 67-66-3 | |
| ERCC | Invitrogen | 4456740 | |
| EZ RNA Methylation Kit | Zymo Research | EZR5001 | |
| Fragment analyzer RNA Kit - HS RNA Kit | Agilent | DNF-472-0500 | |
| Fragment analyzer RNA Kit - RNA Kit | Agilent | DNF-471-0500 | |
| High-Capacity cDNA Reverse Transcription Kit | Applied Biosystem | 4368814 | |
| Illumina TruSeq Stranded mRNA | Illumina | 20020594 | |
| Magnetic Beads A/G Blend | Merck | 16-663 | |
| N6-Methyladenosine, 5′-monophosphate sodium salt (m6A) | Sigma Aldrich | M2780-10MG | |
| Normal Mouse IgG | Merk | 12371 | |
| Oligo(dT)25 | Life Technologies | 61005, | |
| PCRapace | Stratec | 1020220300 | |
| Quick RNA Viral Kit | Zymo Research | 1034 | |
| RNA Clean & Concentrator | Zymo Research | R1015 | |
| RNA Fragmentation Reagent | Ambion | AM8740 | |
| RNase Inhibitor | Ambion | AM2684 | |
| Trizol | TRIzol Reagent | 15596026 |
参考文献
- Machnicka, M. A., et al. MODOMICS: a database of RNA modification pathways--2013 update. Nucleic Acids Research. 41, 262-267 (2013).
- Zaccara, S., Ries, R. J., Jaffrey, S. R. Reading, writing and erasing mRNA methylation. Nature Reviews Molecular Cell Biology. 20 (10), 608-624 (2019).
- Davalos, V., Blanco, S., Esteller, M. SnapShot: Messenger RNA Modifications. Cell. 174 (2), 498 (2018).
- Saletore, Y., et al. The birth of the Epitranscriptome: deciphering the function of RNA modifications. Genome Biology. 13 (10), 175 (2012).
- Zhao, B. S., Roundtree, I. A., He, C. Post-transcriptional gene regulation by mRNA modifications. Nature Reviews Molecular Cell Biology. 18 (1), 31-42 (2017).
- Netzband, R., Pager, C. T. Epitranscriptomic marks: Emerging modulators of RNA virus gene expression. Wiley Interdisciplinary Reviews: RNA. 11 (3), 1576 (2020).
- Pereira-Montecinos, C., Valiente-Echeverria, F., Soto-Rifo, R. Epitranscriptomic regulation of viral replication. Biochimica et Biophysica Acta. 1860 (4), 460-471 (2017).
- Lichinchi, G., et al. Dynamics of the human and viral m(6)A RNA methylomes during HIV-1 infection of T cells. Nature Microbiology. 1, 16011 (2016).
- Courtney, D. G., et al. Epitranscriptomic Addition of m(5)C to HIV-1 Transcripts Regulates Viral Gene Expression. Cell Host & Microbe. 26 (2), 217-227 (2019).
- Kennedy, E. M., et al. Posttranscriptional m(6)A Editing of HIV-1 mRNAs Enhances Viral Gene Expression. Cell Host & Microbe. 19 (5), 675-685 (2016).
- Tirumuru, N., Wu, L. HIV-1 envelope proteins up-regulate N (6)-methyladenosine levels of cellular RNA independently of viral replication. Journal of Biological Chemistry. 294 (9), 3249-3260 (2019).
- Tirumuru, N., et al. N(6)-methyladenosine of HIV-1 RNA regulates viral infection and HIV-1 Gag protein expression. Elife. 5, (2016).
- Cristinelli, S., Angelino, P., Janowczyk, A., Delorenzi, M., Ciuffi, A. HIV Modifies the m6A and m5C Epitranscriptomic Landscape of the Host Cell. Frontiers in Virology. 1 (11), (2021).
- Khoddami, V., Cairns, B. R. Transcriptome-wide target profiling of RNA cytosine methyltransferases using the mechanism-based enrichment procedure Aza-IP. Nature Protocols. 9 (2), 337-361 (2014).
- Hussain, S., Aleksic, J., Blanco, S., Dietmann, S., Frye, M. Characterizing 5-methylcytosine in the mammalian epitranscriptome. Genome Biology. 14 (11), 215 (2013).
- Dominissini, D., Moshitch-Moshkovitz, S., Salmon-Divon, M., Amariglio, N., Rechavi, G. Transcriptome-wide mapping of N6-methyladenosine by m6A-seq based on immunocapturing and massively parallel sequencing. Nature Protocols. 8 (1), 176-189 (2013).
- Dominissini, D., et al. Topology of the human and mouse m6A RNA methylomes revealed by m6A-seq. Nature. 485 (7397), 201-206 (2012).
- Shobbir Hussain, J. A., Blanco, S., Dietmann, S., Frye, M. Characterizing 5-methylcytosine in the mammalian epitranscriptome. Genome Biology. 14 (215), (2013).
- Amort, T., et al. Distinct 5-methylcytosine profiles in poly(A) RNA from mouse embryonic stem cells and brain. Genome Biology. 18 (1), 1 (2017).
- Endrullat, C., Glökler, J., Franke, P., Frohme, M. Standardization and quality management in next-generation sequencing. Applied & Translational Genomics. 10, 2-9 (2016).
- Schaefer, M., Pollex, T., Hanna, K., Lyko, F. RNA cytosine methylation analysis by bisulfite sequencing. Nucleic Acids Research. 37 (2), 12 (2009).
- Cristinelli, S., Angelino, P., Janowczyk, A., Delorenzi, M., Ciuffi, A. HIV Modifies the m6A and m5C Epitranscriptomic Landscape of the Host Cell. biorxiv. 1 (11), (2021).
- Squires, J. E., et al. Widespread occurrence of 5-methylcytosine in human coding and noncoding RNA. Nucleic Acids Research. 40 (11), 5023-5033 (2012).
- Aschenbrenner, J., et al. Engineering of a DNA Polymerase for Direct m(6) A Sequencing. Angewandte Chemie (International ed. in English). 57 (2), 417-421 (2018).
- Didion, J. P., Martin, M., Collins, F. S. Atropos: specific, sensitive, and speedy trimming of sequencing reads. PeerJ. 5, 3720 (2017).
- Kim, D., Langmead, B., Salzberg, S. L. HISAT: a fast spliced aligner with low memory requirements. Nature Methods. 12 (4), 357-360 (2015).
- Li, H., et al. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 25 (16), 2078-2079 (2009).
- Okonechnikov, K., Conesa, A., García-Alcalde, F. Qualimap 2: advanced multi-sample quality control for high-throughput sequencing data. Bioinformatics. 32 (2), 292-294 (2016).
- Ewels, P., Magnusson, M., Lundin, S., Käller, M. MultiQC: summarize analysis results for multiple tools and samples in a single report. Bioinformatics. 32 (19), 3047-3048 (2016).
- Zhang, Y., et al. Model-based Analysis of ChIP-Seq (MACS). Genome Biology. 9 (9), 137 (2008).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet Journal. 17 (1), (2011).
- Shen, W., Le, S., Li, Y., Hu, F. SeqKit: A Cross-Platform and Ultrafast Toolkit for FASTA/Q File Manipulation. PLOS ONE. 11 (10), 0163962 (2016).
- Rieder, D., Amort, T., Kugler, E., Lusser, A., Trajanoski, Z. meRanTK: methylated RNA analysis ToolKit. Bioinformatics. 32 (5), 782-785 (2015).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。