需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
通过工程、条形码和筛选腺相关病毒 (AAV) 衣壳变体分离下一代基因治疗载体
摘要
AAV肽显示文库生成,随后通过条形码对具有新颖特性的候选物进行验证,以创建下一代AAV。
摘要
源自腺相关病毒(AAV)的基因递送载体是治疗遗传疾病的最有前途的工具之一,令人鼓舞的临床数据和几种AAV基因疗法的批准证明了这一点。AAV载体成功的两个主要原因是(i)先前分离出具有不同特性的各种天然存在的病毒血清型,以及(ii)随后建立了强大的分子工程技术,并在高通量中再利用。最近实施的策略进一步提高了这些技术的潜力,用于在DNA和RNA水平上对选定的AAV衣壳进行条形码编码,允许它们在单个动物的所有主要器官和细胞类型中进行全面和平行的 体内 分层。在这里,我们提出了一个包含这组互补途径的基本管道,使用 AAV 肽显示来代表可用衣壳工程技术的各种库。因此,我们首先描述了生成AAV肽显示库的关键步骤,用于 体内 选择具有所需特性的候选药物,然后演示如何对最有趣的衣壳变体进行条形码编码以进行二次 体内 筛选。接下来,我们举例说明了创建用于下一代测序(NGS)文库的方法,包括条形码扩增和衔接连接,最后概述了NGS数据分析中最关键的步骤。由于这里报告的协议是通用和适应性强的,研究人员可以很容易地利用它们来丰富他们最喜欢的疾病模型中的最佳AAV衣壳变体和基因治疗应用。
引言
基因转移疗法是在细胞中引入遗传物质以修复、替换或改变细胞遗传物质,以预防、治疗、治愈或改善疾病。体内和离体基因转移依赖于不同的递送系统,非病毒和病毒。病毒已经自然进化,可以有效地转导其靶细胞,并可用作递送载体。在基因治疗中使用的不同类型的病毒载体中,腺相关病毒已被越来越多地使用,因为它们缺乏致病性、安全性、低免疫原性,最重要的是它们能够维持长期、非整合的表达1,2,3。AAV基因治疗在过去十年中取得了相当大的成就;三种疗法已被欧洲药品管理局和美国食品和药物管理局批准用于人类3,4。一些临床试验也正在进行中,以治疗各种疾病,如血友病、肌肉、心脏和神经系统疾病,详见其他专题3。尽管取得了数十年的进步,但基因治疗领域近年来经历了一系列挫折4,最重要的是临床试验中的死亡5由于剂量限制性毒性而被搁置,特别是对于巨大的组织,如肌肉,或难以触及的组织,如大脑6。
目前用于临床试验的AAV载体属于天然血清型,但少数例外1。AAV工程为开发具有卓越器官或细胞特异性和效率的载体提供了机会。在过去的二十年中,已经成功应用了几种方法,例如肽显示,环交换,衣壳DNA洗牌,容易出错的PCR和靶向设计,以生成具有不同性质的单个AAV变体或其文库7。然后对它们进行多轮定向进化,以选择其中具有所需特性的变体,如其他地方1,3所述。在所有衣壳进化策略中,肽显示AAV文库使用最广泛,这是由于一些独特的性质:它们相对容易生成,并且可以实现高多样性和高通量测序,从而允许跟踪它们的进化。
第一个成功的肽插入AAV文库是在大约20年前描述的。在第一个中,Perabo等人8构建 了一个修饰的AAV2衣壳文库,其中随机生成的寡核苷酸池入质粒中,位置对应于VP1衣壳蛋白的氨基酸587,在从衣壳突出的三倍轴上。使用腺病毒混合感染,AAV文库通过多轮选择进化,最终的重新靶向变体被证明能够转导亲本AAV28难治的细胞系。此后不久,Müller等人9引入了 用于文库生产的两步系统,这是对该协议的重大改进。最初,质粒文库与腺病毒辅助质粒一起用于产生含有嵌合衣壳的AAV文库。该AAV穿梭文库用于感染低感染多重性(MOI)的细胞,目的是为每个细胞引入一个病毒基因组。与腺病毒的混合感染可确保产生具有匹配基因组和衣壳9的AAV。大约十年后,Dalkara10 使用 体内 定向进化创造了7m8变体。该变体具有10个氨基酸插入(LALGETTRPA),其中三个充当接头,并在玻璃体内注射后有效地靶向视网膜外层10。这种工程衣壳是一个非凡的成功故事,因为它是迄今为止为数不多的进入临床的工程衣壳之一11。
随着二代测序(NGS)技术的引入,该领域经历了第二次推动。Adachi et al.12 在 2014 年和 Marsic et al.13 在 2015 年的两篇出版物展示了 NGS 以高精度跟踪条形码 AAV 衣壳文库分布的能力。几年后,条形码区域的NGS适应肽插入区域以跟随衣壳的演变。Körbelin等人14进行了NGS引导的筛选,以确定基于肺靶向AAV2的衣壳。NGS分析有助于计算三个评分:选择轮之间的富集评分,确定组织特异性的一般特异性评分,最后是综合评分14。Gradinaru实验室15在同一年发表了基于Cre重组的AAV靶向进化(CREATE)系统,该系统有助于细胞类型特异性选择。在这个系统中,衣壳文库携带一个Cre可逆开关,因为polyA信号两侧有两个loxP位点。然后将AAV文库注射到Cre小鼠中,其中polyA信号仅在Cre+细胞中倒置,为反向PCR引物与衣壳基因内的正向引物结合提供模板。这种高度特异性的PCR救援能够鉴定AAV-PHP。可以穿过血脑屏障的B变体15。该系统进一步发展为M-CREATE(Multiplexed-CREATE),其中NGS和合成文库生成被集成到管道16中。
来自Maguire实验室17的基于RNA的改进版本iTransduce允许在DNA水平上选择功能转导细胞并表达其基因组的衣壳。肽显示文库的病毒基因组包括由普遍存在的启动子控制的Cre基因和在p41启动子控制下的衣壳基因。将该文库注射到tdTomato上游具有loxP-STOP-loxP盒的小鼠中。用AAV变体转导的细胞,这些变异体表达病毒基因组,因此Cre表达tdTomato,并与细胞标记结合,可以进行分类和选择17。类似地,Nonnenmacher等人18 和Tabebordbar等人19 将衣壳基因库置于组织特异性启动子的控制之下。注射不同动物模型后,使用病毒RNA分离衣壳变体。
另一种方法是使用条形码来标记衣壳库。Björklund实验室20 使用这种方法对肽插入衣壳文库进行条形码编码,并开发了条形码理性AAV载体进化(BRAVE)。在一个质粒中,将Rep2Cap盒克隆在倒置末端重复序列(ITR)侧向表达黄色荧光蛋白(YFP)的条形码标记转基因旁边。使用 帽 末端和条形码开头之间的loxP位点, 体外 Cre重组产生足够小的NGS片段,从而允许肽插入与唯一条形码(查找表,LUT)相关联。使用质粒文库进行AAV生产,并在 体内 应用后再次使用NGS20筛选mRNA中表达的条形码。当衣壳文库包含整个衣壳基因的变体(即洗牌文库)时,需要使用长读长测序。一些小组使用条形码来标记这些不同的库,这使得NGS具有更高的读取深度。Kay 实验室21 用 帽 polyA 信号下游的条形码标记了高度多样化的衣壳洗牌文库。第一步,生成条形码质粒文库,并将洗牌衣壳基因文库克隆到其中。然后,MiSeq(短读长,高读深)和PacBio(长读长,低读深)NGS以及Sanger测序的组合用于生成他们的LUT21。2019年,Ogden和来自ChurchLab 22 的同事使用在每个位置都有单点突变、插入和缺失的文库描绘了AAV2衣壳对多种功能的适应性,最终实现了机器引导设计。为了生成文库,合成衣壳基因的较小片段,用条形码标记,进行下一代测序,然后克隆到完整的衣壳基因中。NGS数据用于生成LUT。然后仅使用条形码和短读取序列对库进行筛选,这反过来又允许更高的读取深度22。
条形码文库主要用于筛选已知、天然和工程变体的池,经过几轮衣壳文库的选择或独立于衣壳进化研究。这种文库的优点是有机会筛选多个衣壳,同时减少动物数量并最大限度地减少动物之间的变异。将这项技术引入AAV领域的第一批研究是在大约十年前发表的。Nakai实验室12用一对12 核苷酸条形码标记了191个双丙氨酸突变体,覆盖AAV9的VP1上的氨基酸356至736。使用NGS,在 体内筛选 文库的半乳糖结合和其他性质12。Marsic及其同事在1年后使用双条带分析描绘了AAV变体的生物分布13。最近一项针对非人灵长类动物的研究比较了使用不同递送途径的29个衣壳在中枢神经系统中的生物分布23。我们的实验室最近发布了183种变体的条形码AAV库屏幕,其中包括天然和工程AAV。DNA和RNA水平上的这些筛选导致在小鼠中鉴定出高度肌致性AAV变体24以及在小鼠大脑中显示出高细胞类型特异性的其他变体25。
在这里,我们描述了这项工作中使用的方法,并对其进行了扩展,以包括AAV肽显示文库的筛选。这包括生成AAV2肽显示文库,用于定量的数字液滴PCR(dd-PCR)方法,最后是用于分析AAV变体的NGS管道,部分基于Weinmann及其同事的工作24。最后,提供了条形码AAV文库的生成和同一出版物中使用的NGS管道的描述。
研究方案
1. AAV2随机7-mer肽显示文库制备
注意:为了制备AAV2随机肽显示文库,将简并寡核苷酸合成为单链DNA,将其转换为双链DNA,酶解,连接受体质粒并电穿孔。
- 简并寡核苷酸的设计
- 对简并寡核苷酸进行排序,避免密码子偏差。在寡核苷酸5'CAGTCGGCCAG AG W GGC (X01)7 GCCCAGGCGGCTGACGAG 3'中,X01对应于20个密码子,每个密码子编码20个氨基酸中的一个。W可以是A或T,产生密码子AGA或AGT,其编码氨基酸精氨酸(R)或丝氨酸(S)。
- 订购扩增引物:5' CTCGTCAGCCGCCTGG 3'(详见 图1 )。这会产生以下蛋白质插入片段:R/S GX 7。理论多样性计算如下:1 x 2 x 207 = 2.56 x 109 个唯一变体。
注意:应该注意的是,这种多样性可能会受到转换效率的限制。
- 第二链合成
- 用TE缓冲液将两个寡核苷酸(简并寡核苷酸和扩增引物)重悬至100μM终浓度。
- 对于PCR反应,用1 μL每个引物、10 μL缓冲液、1.5 μLDMSO、0.5 μLdNTP (10 mM)、0.5 μL高保真热启动聚合酶II和35.5 μL无核酸酶水进行50 μL反应。
- 将反应转移到热循环仪中,并在98°C下运行预孵育步骤10秒,然后在98°C下运行10秒,在59°C下运行30秒,在72°C下运行10秒的三个循环,然后在72°C下运行5分钟和最后的冷却步骤。
- 使用核苷酸去除试剂盒纯化反应,并在 100 μL 无核酸酶水中洗脱。
- 通过在生物分析仪上分析来确认第二链合成的效率(见 图2)。根据制造商的说明,从DNA 1000试剂盒中将1 μL反应加载到微流控芯片中,从而分析双链插入片段的大小和纯度。该试剂盒经过优化,可测量 25-1,000 bps 的双链 DNA 片段的大小和浓度。
- 用TE缓冲液将两个寡核苷酸(简并寡核苷酸和扩增引物)重悬至100μM终浓度。
- 插入片段和质粒载体的消化
- 在最终的 100 μL 反应体积中,用 10 μL 10x 缓冲液和 5 μL BglI 酶消解 85 μL 纯化的插入片段(详见 图 1 )。在37°C孵育过夜。使用核苷酸去除试剂盒纯化,在 50 μL 无核酸酶水中洗脱,并使用分光光度计中的"寡核苷酸 DNA"类型进行定量。
- 在最终的 200 μL 反应体积中,用 20 μL 10x 缓冲液和 10 μL SfiI 酶消化 10 μg 具有复制能力的 AAV 质粒 (pRep2Cap2_PIS)26 (ITR 侧翼病毒基因组)(详见 图 1 )。在50°C孵育过夜。使用凝胶提取试剂盒在1%琼脂糖凝胶上纯化载体,然后使用DNA纯化试剂盒进行额外的纯化步骤。量化分光光度计中的浓度。
- 插入片段与载体的连接
- 在 20 μL 连接反应中,将 955 ng 质粒载体与 45 ng 插入片段与 2 μL 缓冲液和 2 μL 连接酶连接。在16°C孵育过夜,然后在70°C下孵育10分钟以热灭活连接酶。
- 转化、复杂性计算和质粒文库制备
- 按照制造商的说明使用DNA纯化试剂盒纯化反应。在约80%的无核酸酶起始体积水中洗脱反应,并储存在冰上以进行后续转化。
- 转化电感受态细胞:在冰上解冻一瓶电感受态细胞10分钟。然后将 1-2 μL 纯化的连接反应加入 30 μL(一瓶)电感受态细胞中,并通过轻轻敲击进行混合。接下来,小心地将细胞/DNA混合物移液到预冷的1 mm间隙电穿孔比色皿中,不要引入气泡。
- 使用以下设置电穿孔:1800 V、600 Ω 和 10 μF。在电穿孔脉冲的 10 秒内,将 970 μL 预热的恢复培养基(随电感受态细胞一起提供)加入比色皿中,并通过移液混合。最后,将细胞转移到微量离心管中,并在37°C以250rpm孵育1小时。为了达到所需的多样性,进行10-100次反应,孵育后,将所有反应集中在一个管中。
- 通过在 PBS 中稀释 10 μL 混合转化 10、100 或 1,000 倍并铺展 100 μL 在含有适当抗生素(75 mg/mL 氨苄西林)的营养琼脂平板上来计算多样性。将琼脂平板在37°C孵育过夜,然后计数琼脂平板上的菌落。
- 理论多样性计算如下:
理论最大多样性 = 10 x 稀释因子 x 菌落数 x 电穿孔反应数。
注意:要确认文库质量,请通过Sanger测序对至少20个菌落进行测序。大多数克隆都应包含插入片段,并且所有克隆都应该是唯一的。 - 将含有适当抗生素的LB培养基接种400-1,000mL与其余的混合转化物,并在37°C,180rpm下孵育过夜。
- 质粒文库的制备
- 从过夜培养中,制备甘油储备液(在无核酸酶水中混合等体积的细菌培养物和50%甘油溶液,并在-80°C下冷冻),并使用质粒最大试剂盒纯化质粒文库。
- AAV病毒文库的制作
- 如前所述制备病毒文库27。使用转染试剂(如聚乙烯亚胺(PEI))将质粒文库(pRep2Cap2_PI,肽插入片段)与辅助性腺质粒转染到HEK293T细胞中。
- 3天后收集细胞,并对其进行三个冻融循环。使用氯化铯梯度超速离心纯化病毒裂解物,然后缓冲液交换至PBS,最后浓缩病毒颗粒。
- 使用 dd-PCR 进行 AAV 载体滴定
- 在 198 μL 无核酸酶水中连续稀释 2 μL AAV 载体原液,得到 1:106 的最终稀释度。每次使用 200 μL 移液器充分混合。添加一个无模板对照 (NTC) 作为阴性对照。
注意:可以测定其他较低或较高的稀释度(1:105-1:107)。 - 准备 20x 引物-探针混合物。向 1.5 mL 离心管中加入 3.6 μL 的 100 μM 引物(正向和反向、Rep2 和 ITR)、100 μM dd-PCR 探针(Rep2 和 ITR)各 1 μL 和 3.6 μL 无核酸酶的水。
注意:AAV文库是使用FAM标记探针检测的转基因靶向引物探针组(Rep2)和用HEX标记探针检测的ITR靶向引物探针组测量的。 - 通过加入 5.5 μL 样品、1.1 μL 20x 引物-探针混合物、11 μL 用于探针的 dd-PCR 超级混合物(无 dUTP)和 4.4 μL 无核酸酶水,制备 22 μL PCR 反应。这分别产生引物和探针的浓度为900 nM和250 nM。
- 使用液滴发生器产生液滴,将反应转移到96孔板中,将板放入热循环仪中,并在94°C下运行变性步骤10分钟,然后在94°C下运行40个30秒的循环,在58°C下运行1分钟。 接下来,在98°C下热灭活聚合酶10分钟,并加入最后的冷却步骤。在液滴读取器中读取反应并继续分析28.
- 使用分析软件打开保存的dd-PCR板文件。使用一维振幅选项卡中的阈值工具(荧光振幅与事件编号)以NTC为指导,分离每个通道的负液滴和正液滴,并将数据导出到csv文件。
- 要计算载体浓度,首先使用以下公式计算校正因子CF:
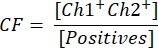
CF确定转基因阳性液滴[阳性]与转基因和ITR [Ch1+ Ch2+]阳性的比例,以确保检测功能性载体颗粒。最终矢量浓度c现在可以使用以下公式计算:
DF是稀释因子(1:105-1:107 如前所述)。每 20 μL/孔反应的拷贝数对应于 5 μL 稀释样品。因子 1,000 将比例校正为 VG/mL(病毒基因组/mL)。示例性滴定结果如 表1 和 图3所示。
- 在 198 μL 无核酸酶水中连续稀释 2 μL AAV 载体原液,得到 1:106 的最终稀释度。每次使用 200 μL 移液器充分混合。添加一个无模板对照 (NTC) 作为阴性对照。
- NGS对AAV病毒库的分析
- 通过使用校对聚合酶试剂盒(2x;见 图4)建立20 μL PCR反应,扩增96核苷酸肽插入片段。向反应中加入 1 μL 含有 1 x 108 vg、100 μM 引物(NGS_forward 和 NGS_reverse 各 0.5 μL 和 10 μL 酶混合物的 AAV 储备液。用无核酸酶水将最终体积调节至 20 μL。
- 将反应转移到热循环仪中,并在98°C下运行变性步骤3分钟,然后在98°C下运行30-35个循环,每次10秒,在59°C下运行10秒,在72°C下运行20秒,然后在72°C下运行5分钟和最后的冷却步骤。
- 使用PCR纯化试剂盒纯化样品。在分光光度计中定量浓度,并运行3%琼脂糖凝胶以验证纯度和片段大小。
- 根据制造商关于制备NGS文库的说明,使用低复杂度样品试剂盒的文库系统处理PCR片段。用30ng的PCR片段进行末端修复反应,然后进行衔接连接和PCR扩增10个循环。使用PCR纯化试剂盒纯化反应。
- 根据制造商的说明,使用DNA试剂盒在生物分析仪上处理最终产品以验证大小和纯度。
- 使用荧光计定量扩增子并汇集它们。在荧光计上再次量化最终的合并NGS文库(根据制造商的说明),并在生物分析仪上验证质量。
- 使用 75 周期高输出套件,以单端 (SE) 模式对 NGS 文库进行测序,读取长度为 84,索引 1 为 8。
注意:本文中示例的测序是在海德堡EMBL的GeneCore设施(http://www.genecore.embl.de/)进行的。 - 使用 Python 3 和 biopython 分析 NGS 测序数据。这些文件可以在 https://github.com/grimmlabs/AAV_GrimmLab_JoVE2022(或者 https://doi.org/10.5281/zenodo.7032215)找到。NGS分析由两个步骤组成。
- 在第一步中,在序列文件中搜索满足某些条件的序列(插入位点两侧存在识别序列)(参见 图4,步骤1.9.8.5)。这是使用脚本 (Script#1) 和提供所需信息的配置文件完成的。一旦确定了正确的序列,程序就会提取序列并将其存储在输出文件中,输出文件是与排序文件同名的 txt 文件。
- 第二步是分析输出文件。文库中的序列从九个氨基酸插入片段中的六个核苷酸(AGWggc,W = A / T)中的任何一个开始。基于该起始序列,肽被翻译。这将生成包含肽变体 (PV) 的输出文件。
- 准备两个文件夹:脚本和数据。到"数据"文件夹,复制排序生成的 gzip 压缩文件。到脚本文件夹,复制以下文件,Python 文件:脚本#1_DetectionExtraction_JoVE_Py3.py;Python 文件: 脚本#2_PV_extraction_and_ranking_Py3.py;配置文件:Barcode_Script_JoVE.conf;和查找表 (LUT) 文件:Zuordnung.txt。
- 在运行脚本之前,请在脚本文件夹中编辑以下文件。打开"Zuordnung.txt"文件,并添加两个制表符分隔的列,即 gzip 文件的名称(第 1 列)和所需的最终名称(第 2 列;制表符分隔的值)。
注意:示例 txt 文件位于 GitHub 文件夹"PV_analysis_script"中。GitHub 文件夹中提供的文件是为分析上述库中的三个示例数据而准备的:xaa.txt.gz、xab.txt.gz 和 xac.txt.gz。还提供了输出文件。 - 更改配置文件"Barcode_Script_JoVE.conf"中的以下变量:
my_dir = "~/数据/"
filename_sample_file = "~/Script/Zuordnung.txt"
特定于序列的变量:BCV_size = 27,BCV左侧 = TCCAGGGCCAG,BCV 右侧 = GCCCAGG,BCVloc = 30,BCV保证金 = 8,BCVleft_revcomp = GCCGCCTGGGC,BCVright_revcomp = CTGGCCC,BCVloc_revcomp = 41(有关详细信息,请参见 图 4 )。 - 使用以下命令调用变体序列检测和提取:
>python3 ~/Script#1_DetectionExtraction_JoVE_Py3.py ~/Barcode_Script_JoVE.conf
注意:输出是带有提取的DNA序列及其读取次数的txt文件。此文件的标头包含统计数据(即读取总数和提取的读取)。这些数据将传输到下一个文件。这些 txt 数据是 Script#2 的输入文件,其中对 DNA 序列进行翻译、排序和分析。 - 使用以下命令执行 PV 提取和分析:
>python3 ~/Script#2_PV_extraction_and_ranking_Py3.py ~/Barcode_Script_JoVE.conf - 分析脚本#2的文本输出文件。Script#2 的输出文件使用"Zuordnung.txt"中 LUT 的第二列命名,扩展名基于分析类型。
注意:确保三个输出文件在第一行包含统计数据("# 有效 PV 读取"、"# 无效 PV 读取"和"# 唯一 PV 读取")、第一列包含输入 txt 文件中每个 DNA 序列的索引(脚本 #1 的输出)和以下列:(1) "...analyzed_all.csv":"样品:"(DNA序列),"#"(读取次数),"Frw或Rev"(正向或反向读取)和"PV"(翻译的肽序列)。无效序列在最后两列中有"NA"和"无效"。(2) "...analyzed_validSeq.csv":与上一个文件相同,筛选出有效序列。(3) "...analyzed_PV.csv":"PV"(翻译肽序列)、"#"(读取次数)和"计数"(先前文件中的 FRW 和 rev 计数合并,计数给定 1 或 2)。 - 根据用户的需求,使用可用的软件可视化输出文件。
2. AAV2随机7聚体肽显示文库选择
- 在定量和质量控制(第1节)之后使用AAV库在选择的模型中进行定向进化,以迭代选择具有所需特性的候选物(见图5)16,18,21。
注意:然后,这些候选库用于生成条形码库,如下文第 3 节所述。
3. 条形码AAV衣壳文库制备与分析
注意:在肽显示屏中鉴定一组潜在特异性和有效的AAV衣壳后,验证鉴定的肽序列的功能,并将其与一组常用或描述良好的参考AAV衣壳变体进行比较。为此,将衣壳序列插入到没有 ITR 的 Rep/Cap 帮助程序结构中。
- 制作条形码AAV库
- 使用三质粒系统对每个衣壳变体进行重组AAV生产,如前所述24。
注意:为了区分不同的衣壳变体,ITR侧翼报告转基因质粒含有长度为15个核苷酸的独特条形码。条形码位于增强黄色荧光蛋白(EYFP)和polyA信号之间的3' UTR(非翻译区域)(见 图6A)。EYFP表达由强大的泛在巨细胞病毒(CMV)启动子驱动,可提供足够水平的RNA转录本。 - 设计长度为15个核苷酸的条形码,均聚物小于3个核苷酸,GC含量为<65%29,汉明距离大于4个核苷酸24。
- 与带有独特条形码的转基因质粒分别生产每个衣壳。这样,每个衣壳变体都标有不同的条形码,以实现其特定的跟踪(见 图 6B)。
- 使用三质粒系统对每个衣壳变体进行重组AAV生产,如前所述24。
- 使用 dd-PCR 进行 AAV 载体滴定
- 按照前面1.8节所述,用YFP引物对替换Rep2引物对,进行AAV滴定。
- 量化单个AAV产品,并汇集每个产品的等量,以生成最终的条形码库。
- 再次定量最终文库以检查最终浓度和质量(见 图7)。
- 条形码AAV文库 在体内 应用
- 将条形码的AAV库系统地应用于所选的模型系统(例如在小鼠24中系统地应用)。
- 根据实验收集ON和OFF目标组织(即肝,肺,心脏,横膈膜,平滑肌,十二指肠,胰腺,结肠,二头肌,卵巢,胃,内耳,肾脏,腹主动脉,胸主动脉,脑,棕色和白色脂肪以及脾脏)或细胞类型。将它们在-80°C下冷冻,提取DNA / RNA,并应用NGS定量分析,如下一节所述。
- 脱氧核糖核酸/核糖核酸提取
- 使用 DNA/RNA 迷你试剂盒从感兴趣的组织中提取 DNA 和 RNA。
- 将一小块感兴趣的组织(1mm3,约5mg)放入2mL反应管中。
- 向组织中加入 350 μL 与β-巯基乙醇 (1%) 和 5 mm 钢珠混合的裂解缓冲液(在通风橱下处理含有 β-巯基乙醇的样品)。
- 在组织裂解器中以40 Hz均匀化组织45秒。
- 加入 10 μL 蛋白酶 K (10 mg/mL),并在 55 °C 下孵育 15 分钟,同时以 400 rpm 振荡。
- 在室温下以20,000× g 离心3分钟,收集上清液,然后继续使用DNA / RNA试剂盒的制造商方案。
- 将洗涤步骤分成两步,每步加入 350 μL 洗涤缓冲液。在这些洗涤步骤之间,用不含RNase的DNase I消化色谱柱上的残留DNA.将根据制造商的说明制备的80μLDNase I溶液加入色谱柱上,并在室温下孵育15分钟。
- 用无核酸酶水从色谱柱中洗脱RNA/DNA。将分离的RNA储存在-80°C,将gDNA储存在-20°C。
- cDNA合成
- 在逆转录反应之前,将RNA样品进行另一轮15-30分钟的DNase I处理(用于从RNA样品中完全去除污染DNA)。将 1 μL DNase I 溶液、4 μL 缓冲液(随试剂盒提供)和无核酸酶水加入到 40 μL 至 212 ng RNA 的最终体积中。在室温下孵育30分钟,并在70°C下加热灭活10分钟。
- 根据制造商的说明使用试剂盒使用 150 ng RNA 合成 cDNA。包括不含逆转录酶的对照,以确保样品中没有污染病毒DNA。cDNA储存在-20°C。
注意:用于最佳逆转录的起始RNA量可能因组织类型和相应组织中的预期转导效率而异。
- AAV病毒库(in-vivo) 由 NGS
- 为了以低成本实现高测序深度,如前所述,通过Illumina测序 进行 NGS(第1.9节)。扩增条形码序列,然后将测序接头连接到扩增子。
- 由于扩增子两侧的测序接头的读取长度较短,因此在设计时,请检查扩增子是否足够小,以确保NGS读取中存在条形码序列。对于病毒基因组和病毒转录本中的条形码测序,PCR扩增子设计为113 bp长(见 图8)。
- 用引物BC-seq向前和BC-seq反向扩增条形码区域。准备以下 PCR 反应:0.5 μL 高保真 DNA 聚合酶、10 μL 5x 缓冲液、每 100 μM 引物 0.25 μL (BC-seq fw/BC-seq rv)和 1 μl 10 mM dNTP。使用 25 ng 的 cDNA 或 DNA/反应作为模板,并用无核酸酶水将最终体积调节至 50 μL。
- 在干净的PCR罩下制备PCR预混液以避免污染。使用以下循环条件:在98°C下30秒,然后在98°C下循环40次10秒,在72°C下循环20秒,最后在72°C下5分钟步长。
- 包括PCR对照,以确认PCR预混液中不存在污染DNA。对于cDNA样品,包括没有逆转录酶的对照。最后,包括一个带有 AAV 输入库的示例。此信息将用于生成分析中使用的Normalization_Variant.txt文件。
- 在PCR纯化之前,通过凝胶电泳验证每个样品的PCR片段的大小。后者通过使用市售磁珠或基于柱的DNA纯化系统来实现(参见 材料表)。
- 根据制造商的说明,使用文库系统制备低复杂度样品的NGS文库,如前面的第1.9节所述。
- 通过dsDNA HS试剂盒 测定 DNA浓度,并如前所述分析文库的质量(第1.9.6节),然后进行合并。在荧光计上量化合并文库,并在生物分析仪上评估质量。
- 按照第 1.9.7 节中的讨论执行 NGS 测序。
- 通过qPCR量化转基因(病毒基因组)和管家基因的拷贝数,以评估DNA上组织或器官之间混合文库的分布。
- 按如下方式设置 30 μL qPCR 反应,以确定 EYFP(转基因)和 GAPDH(甘油醛 3-磷酸脱氢酶,管家基因)的拷贝数:
- 准备 EYFP 的 60 倍引物/探针混合物(1.5 μM YFP_fw、1.5 μM YFP_rv和 0.6 μM YFP_probe;参见材料表)。使用GAPDH引物/探针混合物(见 材料表)确定管家基因的拷贝数。在冰上设置反应。
- 准备PCR预混液(15 μL,参见 材料表),为所有样品和标准品添加60倍引物/探针混合物(0.5 μL)(要计算标准品的拷贝数,请使用以下链接:http://cels.uri.edu/gsc/cndna.html)。在冰上设置反应。
- 将 15.5 μL 预混液转移到 96 孔板中,并向相应的孔中加入 14.5 μL 样品(总 DNA 浓度为 75 ng)或标准品。用箔密封96孔板,涡旋并短暂旋转。
- 将 10 μL 的每个样品一式两份转移到 384 孔板中。用箔密封板,并在4°C下以800× g 旋转5分钟。
- 使用50°C的初始温度在热循环仪中孵育反应混合物2分钟,然后在95°C下进行10分钟的初始活化步骤。 在95°C下进行40个变性循环15秒,在60°C下退火/延伸1分钟24。
- 要获得二倍体基因组(dg)的数量,请使用GAPDH拷贝数并除以2。然后,取EYFP拷贝数的值除以dg的数量,得到每个二倍体基因组(vg/dg)的载体基因组。使用此值生成用于生物信息学分析的Normalization_Organ.txt文件。
- 像Weinmann等人一样对NGS测序数据进行分析。24,使用 Python3 (https://github.com/grimmlabs/AAV_GrimmLab_JoVE2022) 中的自定义代码。该工作流程包括检测由侧翼序列引导的条形码序列、它们的长度和位置 (Script#1_BarcodeDetection.py),以及分析一组组织的条形码富集和分布 (Script#2_BarcodeAnalysis.py)。
- 检测条形码并将其分配给 AAV 变体。将测序数据作为存档的 fastq 文件放在一个目录中(例如,"Data_to_analyze")。输入文库的测序数据文件包含在此目录中,仅用于计算输入库中的衣壳比例。
- 在执行脚本之前,请创建两个制表符分隔的文本文件:衣壳变体文件(参见示例文件"变体.txt"),其中包含分配给 AAV 衣壳变体名称的条形码序列,以及带有可能污染的条形码序列的污染文件(参见"污染.txt"),这些文件来自可能的污染(实验室中提供的其他条形码,导致污染)。
- 最后,编辑配置文件"Barcode_Script.conf"以包含以下信息:包含排序数据的文件夹路径(例如,"Data_to_analyze"),条形码侧翼区域的序列,它们的位置以及条形码检测的窗口大小(类似于1.9.8.5,见 图8)。
- 使用以下命令调用条形码检测,并提供 Script#1_BarcodeDetection.py 和配置文件的路径:
>python3 ~/Script#1_BarcodeDetection.py ~/Barcode_Script.conf
注意:Script#1_BarcodeDetection.py 执行的输出是文本文件,其中包含每个衣壳变体的读取计数以及从原始数据中恢复的读取总数。 - 通过执行 Script#2_BarcodeAnalysis.py 和以下 txt 文件来评估条形码 AAV 衣壳在组织或器官中的分布:
- 在"Zuordnung.txt"文件中,将从条形码检测运行中获得的每个 txt 文件的名称分配给组织/器官名称:第一列中的 txt 文件名称和制表符分隔分配中的相应组织/器官名称。
注意:有关示例,请检查"示例"文件夹 (https://github.com/grimmlabs/AAV_GrimmLab_JoVE2022)。值得注意的是,组织/器官名称可以包括定义cDNA或gDNA测量和生物重复数(M1,M2等)的字符。 - 创建一个"器官.txt"文本文件,其中包含ON和OFF目标器官的名称列表,这些名称对应于作业"Zuordnung.txt"文件中给出的名称(请参阅"示例"文件夹:https://github.com/grimmlabs/AAV_GrimmLab_JoVE2022)。
- 创建"Normalization_Organ.txt"和"Normalization_Variant.txt"制表符分隔的文本文件,其中包含所有衣壳变体和所有器官/组织的标准化值。在"Normalization_Organ.txt"文件的第一列中,写下为每个器官给出的名称(如在分配文件"Zuordnung.txt中),在第二列中写下相应组织的归一化值,在第3.6.11节中生成。
- 用衣壳名称列表填充"Normalization_Variant.txt"文件的第一列,用池库中每个衣壳的读取计数的规范化值填充第二列(可以根据第一个脚本生成的输入库的 txt 输出文件计算归一化)。
- 通过指定上述所有其他文件的完整路径来编辑配置文件。执行脚本 #2_BarcodeAnalysis.py 作为:
>python3 /Script#2_BarcodeAnalysis.py ~/Barcode_Script.conf
注意:条形码分析脚本输出多个文件:基于前面描述的多个归一化步骤,具有不同组织内衣壳分布的相对浓度(RC)值的文本文件,以及将文本文件数据合并为合并矩阵数据的电子表格文件。后者可用于聚类分析和可视化。 - 可视化数据并对基质数据进行聚类分析,以区分衣壳特性并根据跨组织的 RC 谱评估它们的相似性。使用其他脚本PCA_heatmap_plot。放置在存储库中的 R:
>rscript --香草 ~/PCA.R ~/相对浓度.xls
注意:该脚本将相对浓度.xls文件作为输入,并生成分层聚类热图和主成分分析 (PCA) 两个图。 - 若要修改绘图(热图轴、PCA 主成分)或 png 参数(颜色、大小、标记),请打开 R 脚本并按照注释部分中提供的说明进行操作。
- 在"Zuordnung.txt"文件中,将从条形码检测运行中获得的每个 txt 文件的名称分配给组织/器官名称:第一列中的 txt 文件名称和制表符分隔分配中的相应组织/器官名称。
结果
生成 AAV2 肽显示库。 作为选择工程AAV的第一步,描述了质粒文库的生成。肽插入片段是使用简并引物产生的。将密码子的组合从64个减少到20个具有消除终止密码子和促进NGS分析的优点,方法是减少DNA上的文库多样性,而不是蛋白质水平上的文库多样性。寡核苷酸插入片段以单链DNA形式购买(图1),通过PCR反应将其转化为双链DNA。该反应的质量在生物分析仪中?...
讨论
在该协议中,概述了肽显示AAV衣壳工程和条形码AAV文库筛选以及文库组成和衣壳性能的生物信息学分析所需的步骤。该协议侧重于促进这些类型文库的生物信息学分析的步骤,因为大多数病毒学实验室在编程技能方面落后,以匹配他们在分子生物学技术方面的熟练程度。正如引言中所述,这两种类型的库在文献中都有广泛的描述,并且可以相对容易地复制。
作为第一步,概?...
披露声明
D.G.是AaviGen GmbH.的联合创始人,D.G.和K.R.是与产生免疫逃避AAV衣壳变体相关的未决专利申请的发明人。其余作者没有什么可透露的。
致谢
D.G.非常感谢德国研究基金会(DFG)通过DFG合作研究中心SFB1129(Projektnummer 240245660)和TRR179(Projektnummer 272983813)以及德国感染研究中心(DZIF,BMBF;TTU-艾滋病毒 04.819)。
材料
| Name | Company | Catalog Number | Comments |
| Amplification primer | ELLA Biotech (Munich, Germany) | - | Second-strand synthesis of oligonucleotide insert |
| Agilent DNA 1000 Reagents | Agilent Technologies (Santa Clara, CA, USA) | 5067-1504 | DNA fragment validation |
| Agilent 2100 Bioanalyzer System | Agilent Technologies (Santa Clara, CA, USA) | G2938C | DNA fragment validation |
| AllPrep DNA/RNA Mini Kit | Qiagen (Venlo, Netherlands) | 80204 | DNA/RNA extraction |
| Agilent DNA 1000 Reagents | Agilent Technologies (Santa Clara, CA, USA) | 5067-1504 | NGS Library preparation |
| Agilent 2100 Bioanalyzer System | Agilent Technologies (Santa Clara, CA, USA) | G2938C | NGS Library preparation |
| BC-seq fw: | IDT (San Joce, CA, CA, USA) | ATCACTCTCGGCATGGACGAGC | NGS Library preparation |
| BC-seq rv: | IDT (San Joce, CA, CA, USA) | GGCTGGCAACTAGAAGGCACA | NGS Library preparation |
| β-Mercaptoethanol | Millipore Sigma (Burlington, MA, USA) | 44-420-3250ML | DNA/RNA extraction |
| BglI | New England Biolabs (Ipswich, MA, USA) | R0143 | Digestion of double-stranded insert |
| C1000 Touch Thermal Cycler | Bio-Rad (Hercules, CA, USA) | 1851196 | dd-PCR cycler |
| dNTPS | New England Biolabs (Ipswich, MA, USA) | N0447S | NGS Library preparation |
| ddPCR Supermix for probes (no dUTP) | Bio-Rad (Hercules, CA, USA) | 1863024 | dd-PCR supermix |
| Droplet Generation Oil for Probes | Bio-Rad (Hercules, CA, USA) | 1863005 | dd-PCR droplet generation oil |
| DG8 Cartridges for QX100 / QX200 Droplet Generator | Bio-Rad (Hercules, CA, USA) | 1864008 | dd-PCR droplet generation cartridge |
| DG8 Cartridge Holder | Bio-Rad (Hercules, CA, USA) | 1863051 | dd-PCR cartridge holder |
| Droplet Generator DG8 Gasket | Bio-Rad (Hercules, CA, USA) | 1863009 | dd-PCR cover for cartridge |
| ddPCR Plates 96-Well, Semi-Skirted | Bio-Rad (Hercules, CA, USA) | 12001925 | dd-PCR 96-well plate |
| E.cloni 10G SUPREME Electrocompetent Cells | Lucigen (Middleton, WI, USA) | 60081-1 | Electrocompetent cells |
| Electroporation cuvettes, 1mm | Biozym Scientific (Oldendorf, Germany) | 748050 | Electroporation |
| GAPDH primer/probe mix | Thermo Fischer Scientific (Waltham, MA, USA) | Mm00186825_cn | Taqman qPCR primer |
| Genepulser Xcell | Bio-Rad (Hercules, CA, USA) | 1652660 | Electroporation |
| High-Capacity cDNA Reverse Transcription Kit | Applied Biosystems (Waltham, MA, USA) | 4368814 | cDNA reverse transcription |
| ITR_fw | IDT (San Joce, CA, USA) | GGAACCCCTAGTGATGGAGTT (https://signagen.com/blog/2019/10/25/qpcr-primer-and-probe-sequences-for-raav-titration/) | dd-PCR primer |
| ITR_rv | IDT (San Joce, CA, USA) | CGGCCTCAGTGAGCGA (https://signagen.com/blog/2019/10/25/qpcr-primer-and-probe-sequences-for-raav-titration/) | dd-PCR primer |
| ITR_probe | IDT (San Joce, CA, USA) | HEX-CACTCCCTCTCTGCGCGCTCG-BHQ1 (https://signagen.com/blog/2019/10/25/qpcr-primer-and-probe-sequences-for-raav-titration/) | dd-PCR probe |
| Illumina NextSeq 500 system | Illumina Inc (San Diego, CA, USA) | SY-415-1001 | NGS Library sequencing |
| KAPA HiFi HotStart ReadyMix (2X)* | Roche AG (Basel, Switzerland) | KK2600 07958919001 | NGS sample prepration |
| MagnaBot 96 Magnetic Separation Device | Promega GmbH (Madison, WI, USA) | V8151 | Sample prepration for NGS library |
| NanoDrop 2000 spectrophotometer | Thermo Fischer Scientific (Waltham, MA, USA) | ND-2000 | Digestion of double-stranded insert |
| NGS_frw | Sigma-Aldrich (Burlinght, MA, USA) | GTT CTG TAT CTA CCA ACC TC | NGS primer |
| NGS_rev | Sigma-Aldrich (Burlinght, MA, USA) | CGC CTT GTG TGT TGA CAT C | NGS primer |
| NextSeq 500/550 High Output Kit (75 cycles) | Illumina Inc (San Diego, CA, USA) | FC-404-2005 | NGS Library sequencing |
| Ovation Library System for Low Complexity Samples Kit | NuGEN Technologies, Inc. (San Carlos, CA, USA) | 9092-256 | NGS Library preparation |
| PX1 Plate Sealer | Bio-Rad (Hercules, CA, USA) | 1814000 | dd-PCR plate sealer |
| Pierceable Foil Heat Seal | Bio-Rad (Hercules, CA, USA) | 1814040 | dd-PCR sealing foil |
| Phusion High-Fidelity DNA-Polymerase | Thermo Fischer Scientific (Waltham, MA, USA) | F530S | Second-strand synthesis of oligonucleotide insert |
| PEI MAX - Transfection Grade Linear Polyethylenimine Hydrochloride (MW 40,000) | Polysciences, Inc. (Warrington, PA, USA) | 24765-1G | AAV library preparation |
| ProNex Size-Selective Purification System | Promega GmbH (Madison, WI, USA) | NG2002 | Sample prepration for NGS library |
| Phusion Hot Start II Polymerase | Thermo Fischer Scientific (Waltham, MA, USA) | F549L | NGS Library preparation |
| Proteinase K | Roche AG (Basel, Switzerland) | 5963117103 | DNA/RNA extraction |
| pRep2Cap2_PIS | ITR-Rep2Cap2-ITR vector. Peptide insertion site within the Cap2 ORF, manufactured/prepared in the lab | ||
| QX200 Droplet Generator | Bio-Rad (Hercules, CA, USA) | 1864002 | dd-PCR droplet generator |
| QX200 Droplet Reader | Bio-Rad (Hercules, CA, USA) | 1864003 | dd-PCR droplet analysis |
| QIAquick Nucleotide Removal Kit | Qiagen (Venlo, Netherlands) | 28306 | Second-strand synthesis of oligonucleotide insert purification |
| QIAquick Gel Extraction Kit | Qiagen (Venlo, Netherlands) | 28704 | Plasmid vector purification |
| QIAGEN Plasmid Maxi Kit | Qiagen (Venlo, Netherlands) | 12162 | Plasmid library DNA preparation |
| Qiaquick PCR Purification kit | Qiagen (Venlo, Netherlands) | 28104 | Sample prepration for NGS library |
| Qubit fluorometer | Invitrogen (Waltham, MA, USA) | Q32857 | NGS Library preparation |
| Qubit dsDNA HS | Thermo Fischer Scientific (Waltham, MA, USA) | Q32851 | NGS Library preparation |
| QuantiFast PCR Master Mix | Qiagen (Venlo, Netherlands) | 1044234 | Taqman qPCR |
| rep_fw | IDT (San Joce, CA, USA) | AAGTCCTCGGCCCAGATAGAC | dd-PCR primer |
| rep_rv | IDT (San Joce, CA, USA) | CAATCACGGCGCACATGT | dd-PCR primer |
| rep_probe | IDT (San Joce, CA, USA) | FAM-TGATCGTCACCTCCAACA-BHQ1 | dd-PCR probe |
| RNase-free DNase | Qiagen (Venlo, Netherlands) | 79254 | DNA/RNA extraction |
| SfiI | New England Biolabs (Ipswich, MA, USA) | R0123 | Digestion of vector |
| 5 mm, steel Beads | Qiagen (Venlo, Netherlands) | 69989 | DNA/RNA extraction |
| TRIMER-oligonucleotides | ELLA Biotech (Munich, Germany) | - | Degenerate oligonucleotide |
| T4 Ligase | New England Biolabs (Ipswich, MA, USA) | M0202L | Plasmid library ligation |
| TissueLyserLT | Qiagen (Venlo, Netherlands) | 85600 | DNA/RNA extraction |
| YFP_fw | IDT (San Joce, CA, USA) | GAGCGCACCATCTTCTTCAAG | dd-PCR primer |
| YFP_rv | IDT (San Joce, CA, USA) | TGTCGCCCTCGAACTTCAC | dd-PCR primer |
| YFP_probe | IDT (San Joce, CA, USA) | FAM-ACGACGGCAACTACA-BHQ1 | dd-PCR probe |
| Zymo DNA Clean & Concentrator-5 (Capped) | Zymo research (Irvine, CA, USA) | D4013 | Vector and Ligation purification |
参考文献
- Wang, D., Tai, P. W. L., Gao, G. Adeno-associated virus vector as a platform for gene therapy delivery. Nature Reviews Drug Discovery. 18 (5), 358-378 (2019).
- Muhuri, M., Levy, D. I., Schulz, M., McCarty, D., Gao, G. Durability of transgene expression after rAAV gene therapy. Molecular Therapy. 30 (4), 1364-1380 (2022).
- Li, C., Samulski, R. J. Engineering adeno-associated virus vectors for gene therapy. Nature Reviews Genetics. 21 (4), 255-272 (2020).
- Kuzmin, D. A., et al. The clinical landscape for AAV gene therapies. Nature Reviews Drug Discovery. 20 (3), 173-174 (2021).
- Mullard, A. Gene therapy community grapples with toxicity issues, as pipeline matures. Nature Reviews Drug Discovery. 20 (11), 804-805 (2021).
- Nature Biotechnology. Gene therapy at the crossroads. Nature Biotechnology. 40 (5), 621 (2022).
- Becker, P., et al. Fantastic AAV Gene Therapy Vectors and How to Find Them-Random Diversification, Rational Design and Machine Learning. Pathogens. 11 (7), 756 (2022).
- Perabo, L., et al. In vitro selection of viral vectors with modified tropism: the adeno-associated virus display. Molecular Therapy. 8 (1), 151-157 (2003).
- Muller, O. J., et al. Random peptide libraries displayed on adeno-associated virus to select for targeted gene therapy vectors. Nature Biotechnology. 21 (9), 1040-1046 (2003).
- Dalkara, D., et al. In vivo-directed evolution of a new adeno-associated virus for therapeutic outer retinal gene delivery from the vitreous. Science Translational Medicine. 5 (189), (2013).
- Sahel, J. A., et al. Partial recovery of visual function in a blind patient after optogenetic therapy. Nature Medicine. 27 (7), 1223-1229 (2021).
- Adachi, K., Enoki, T., Kawano, Y., Veraz, M., Nakai, H. Drawing a high-resolution functional map of adeno-associated virus capsid by massively parallel sequencing. Nature Communications. 5, 3075 (2014).
- Marsic, D., Mendez-Gomez, H. R., Zolotukhin, S. High-accuracy biodistribution analysis of adeno-associated virus variants by double barcode sequencing. Molecular Therapy-Methods & Clinical Development. 2, 15041 (2015).
- Korbelin, J., et al. Pulmonary targeting of adeno-associated viral vectors by next-generation sequencing-guided screening of random capsid displayed peptide libraries. Molecular Therapy. 24 (6), 1050-1061 (2016).
- Deverman, B. E., et al. Cre-dependent selection yields AAV variants for widespread gene transfer to the adult brain. Nature Biotechnology. 34 (2), 204-209 (2016).
- Ravindra Kumar, S., et al. Multiplexed Cre-dependent selection yields systemic AAVs for targeting distinct brain cell types. Nature Methods. 17 (5), 541-550 (2020).
- Hanlon, K. S., et al. Selection of an efficient AAV vector for robust CNS transgene expression. Molecular Therapy-Methods & Clinical Development. 15, 320-332 (2019).
- Nonnenmacher, M., et al. Rapid evolution of blood-brain-barrier-penetrating AAV capsids by RNA-driven biopanning. Molecular Therapy-Methods & Clinical Development. 20, 366-378 (2021).
- Tabebordbar, M., et al. Directed evolution of a family of AAV capsid variants enabling potent muscle-directed gene delivery across species. Cell. 184 (19), 4919-4938 (2021).
- Davidsson, M., et al. A systematic capsid evolution approach performed in vivo for the design of AAV vectors with tailored properties and tropism. Proceedings of the National Academy of Sciences. 116 (52), 27053-27062 (2019).
- Pekrun, K., et al. Using a barcoded AAV capsid library to select for clinically relevant gene therapy vectors. Journal of Clinical Investigation Insight. 4 (22), (2019).
- Ogden, P. J., Kelsic, E. D., Sinai, S., Church, G. M. Comprehensive AAV capsid fitness landscape reveals a viral gene and enables machine-guided design. Science. 366 (6469), 1139-1143 (2019).
- Kondratov, O., et al. A comprehensive study of a 29-capsid AAV library in a non-human primate central nervous system. Molecular Therapy. 29 (9), 2806-2820 (2021).
- Weinmann, J., et al. Identification of a myotropic AAV by massively parallel in vivo evaluation of barcoded capsid variants. Nature Communications. 11 (1), 5432 (2020).
- Kremer, L. P. M., et al. High throughput screening of novel AAV capsids identifies variants for transduction of adult NSCs within the subventricular zone. Molecular Therapy-Methods & Clinical Development. 23, 33-50 (2021).
- Borner, K., et al. Pre-arrayed pan-AAV peptide display libraries for rapid single-round screening. Molecular Therapy. 28 (4), 1016-1032 (2020).
- Kienle, E., et al. Engineering and evolution of synthetic adeno-associated virus (AAV) gene therapy vectors via DNA family shuffling. Journal of Visualized Experiments. (62), e3819 (2012).
- Furuta-Hanawa, B., Yamaguchi, T., Uchida, E. Two-dimensional droplet digital PCR as a tool for titration and integrity evaluation of recombinant adeno-associated viral vectors. Human Gene Therapy Methods. 30 (4), 127-136 (2019).
- Lyons, E., Sheridan, P., Tremmel, G., Miyano, S., Sugano, S. Large-scale DNA barcode library generation for biomolecule identification in high-throughput screens. Scientific Reports. 7 (1), 13899 (2017).
- Korbelin, J., et al. Optimization of design and production strategies for novel adeno-associated viral display peptide libraries. Gene Therapy. 24 (8), 470-481 (2017).
- Korbelin, J., Trepel, M. How to successfully screen random adeno-associated virus display peptide libraries in vivo. Human Gene Therapy Methods. 28 (3), 109-123 (2017).
- Herrmann, A. K., et al. A robust and all-inclusive pipeline for shuffling of adeno-associated viruses. American Chemical Society Synthetic Biology. 8 (1), 194-206 (2019).
- Choudhury, S. R., et al. In vivo selection yields AAV-B1 Capsid for central nervous system and muscle gene therapy. Molecular Therapy. 24 (7), 1247-1257 (2016).
- Buschmann, T., Bystrykh, L. V. Levenshtein error-correcting barcodes for multiplexed DNA sequencing. BMC Bioinformatics. 14, 272 (2013).
- Buschmann, T. DNABarcodes: an R package for the systematic construction of DNA sample tags. Bioinformatics. 33 (6), 920-922 (2017).
- Li, B., et al. A comprehensive mouse transcriptomic BodyMap across 17 tissues by RNA-seq. Scientific Reports. 7 (1), 4200 (2017).
- Clarner, P., et al. Development of a one-step RT-ddPCR method to determine the expression and potency of AAV vectors. Molecular Therapy-Methods & Clinical Development. 23, 68-77 (2021).
- Zolotukhin, S., Vandenberghe, L. H. AAV capsid design: A Goldilocks challenge. Trends in Molecular Medicine. 28 (3), 183-193 (2022).
- Brown, D., et al. deep parallel characterization of AAV tropism and AAV-mediated transcriptional changes via single-cell RNA sequencing. Frontiers in Immunology. 12, 730825 (2021).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。