需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
人类干细胞来源的中脑多巴胺能神经元的表型分析
摘要
该方案描述了人中脑多巴胺能神经元的细胞培养,然后进行免疫染色和从获得的微观高内涵图像生成神经元表型图谱,从而可以识别由于遗传或化学调节引起的表型变异。
摘要
帕金森病 (PD) 与一系列导致中脑多巴胺能 (mDA) 神经元丢失的细胞生物学过程有关。许多目前的 体外 PD细胞模型缺乏复杂性,没有考虑多种表型。人诱导多能干细胞 (iPSC) 衍生的 mDA 神经元的表型分析可以通过同时测量 PD 相关细胞类型中的一系列神经元表型来解决这些缺点。在这里,我们描述了一种从市售的人类mDA神经元中获取和分析表型谱的协议。神经元特异性荧光染色面板用于可视化细胞核、α-突触核蛋白、酪氨酸羟化酶 (TH) 和微管相关蛋白 2 (MAP2) 相关表型。所描述的表型分析方案具有可扩展性,因为它使用 384 孔板、自动液体处理和高通量显微镜。使用健康供体 mDA 神经元和携带富含亮氨酸的重复激酶 2 (LRRK2) 基因中 PD 连接的 G2019S 突变的 mDA 神经元来举例说明该协议的效用。两种细胞系均用LRRK2激酶抑制剂PFE-360处理,并测量表型变化。此外,我们还演示了如何使用聚类或机器学习驱动的监督分类方法分析多维表型图谱。所描述的协议将特别引起从事神经元疾病建模或研究人类神经元中化合物效应的研究人员的兴趣。
引言
帕金森病 (PD) 中的多种细胞生物过程受到干扰。例如,线粒体功能障碍、氧化应激、蛋白质降解缺陷、囊泡运输和内溶酶体功能的破坏与中脑多巴胺能 (mDA) 神经元丢失有关,常见于 PD1。因此,帕金森病似乎涉及多种疾病机制,这些机制可以相互作用并相互恶化。研究这种机制相互作用的一种有用方法是创建中脑多巴胺能 (mDA) 神经元的综合表型指纹或图谱。
表型分析是一种方法,涉及根据一组可测量的特征创建样本的图谱,其次,它涉及根据该图谱对样本进行预测 2,3。分析的目标是捕获各种特征,其中一些特征以前可能与疾病或治疗无关3.因此,分析可以揭示意想不到的生物过程。表型分析通常依赖于荧光染色的细胞,并且已经开发了标准化检测方法,例如细胞绘画,以创建表型图谱 4。例如,最近,表型分析已被应用于小分子的表征或仅基于患者来源的成纤维细胞 5,6 的 PD 亚型的准确预测。尽管取得了这些进展,但表型分析很少应用于有丝分裂后分化细胞,例如表达PD相关突变(如LRRK2 G2019S)的人诱导多能干细胞(iPSC)衍生的mDA神经元。iPSC衍生模型的重大挑战包括分化批次或基因型之间存在细微或可变的病理特征,以及孤立的PD表型不能捕捉到疾病的全部复杂性。此外,虽然 iPSC 神经元模型具有生理相关性,但由于担心技术复杂性,它们很少用于 PD 药物发现过程 7,8。
我们之前开发了一种强大的方法来测量人类 mDA 神经元中多种与 PD 相关的病理生理表型表型,这些神经元对遗传和化合物诱导的表型变化都很敏感9。本文详细描述了该方法的进一步优化版本,以从mDA神经元创建表型图谱(图1)。与先前描述的表型分析方法相比,该协议具有几个优点,例如使用高质量的mDA神经元和技术可重复性。该协议首次以高度可扩展的方式在化学扰动后实现生理相关的有丝分裂后mDA神经元的表型分析。完全分化和冷冻保存的 mDA 神经元是市售的,可显著降低批次间的分化变异性。其次,通过使用明确定义的实验设计(即培养持续时间或避免边缘孔)、自动液体处理和自动显微镜,可以进一步减少技术变异性。此外,本文还概述了使用无监督聚类或监督分类方法进行表型谱分析的初始步骤,指出了如何分析表型谱数据。该协议将用于对遗传或化学扰动诱导的mDA神经元的表型变化感兴趣的研究人员,特别是当需要高度可扩展的研究设置时,例如,在筛选活动期间或当要研究较少数量的化合物的影响时,例如,确定毒性作用。总之,预计人类神经元表型分析的应用是研究复杂疾病相关表型和表征候选药物细胞效应的宝贵技术。
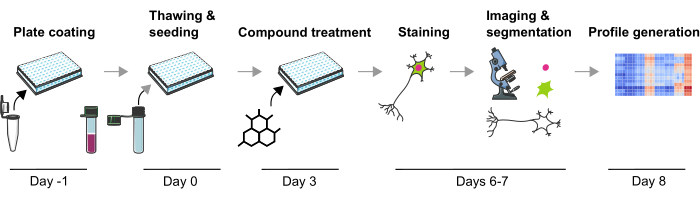
图 1:从人类 iPSC 衍生的 mDA 神经元生成基于图像的表型图谱的实验方案示意图。 请点击这里查看此图的较大版本.
研究方案
1. 准备用于神经元接种的培养基和培养板(第 1 天)
- 为了在第 1 天准备用于神经元接种的板,请在使用前将层粘连蛋白加热至室温 (RT)。通过将层粘连蛋白储备溶液(0.1mg / mL)稀释在冷PBS + / +(含Ca2+ 和Mg2 +)中1/10来制备层粘连蛋白溶液。
注:所有试剂均列在 材料表中。溶液和缓冲液的组成见 表1-4。 - 然后,向聚-D-赖氨酸(PDL)预涂384孔板的每个孔中加入25μL层粘连蛋白溶液,并在4°C下孵育过夜。 包被的板可以在4°C下储存长达一周。
注意:该协议可以在此处暂停长达一周。使用塑料薄膜密封板。 - 准备完整的维持培养基并在4°C下储存长达一个月(表1)。
2. 神经元解冻(第 0 天)
- 为了在第0天解冻神经元,将水浴预热至37°C,并将完全维持培养基平衡至RT,避光。
- 将装有商业获得的冷冻神经元(见 材料表)的小瓶从液氮罐中取出,并将其放在干冰上。然后将小瓶放入水浴中2分钟。液体完全解冻后,用70%乙醇对小瓶进行消毒。
- 用P1000移液管(约370μL)吸出解冻的神经元,并将它们转移到50mL离心管中,无需上下移液。接下来,用 630 μL 完全维持培养基冲洗小瓶,在 50 mL 试管中逐滴(45° 角)分配。分配时缓慢搅拌。
注意:至关重要的是要逐滴缓慢分配培养基,以避免细胞渗透破裂。45°角和轻微搅拌可最大限度地减少移液过程中的高局部渗透压。 - 同样,用 P1000 移液器在 50 mL 离心管中加入 1 mL 完全维持培养基。然后,缓慢加入 2 mL 完全维持培养基。分配时小心搅拌。
- 对细胞进行计数。制备含有 10 μL 台盼蓝和 10 μL 细胞悬浮液的微管,并加入计数室载玻片 (10 μL)。使用自动细胞计数仪(参见 材料表)或手动进行计数。
- 计数后,在室温下以400× g 离心含有神经元的50mL管5分钟,并除去上清液。使用 P1000 移液器和 1 mL 完全维持培养基小心地重悬沉淀。然后添加必要的体积以达到所需浓度(300,000 个细胞/mL,另请参阅下一步)。
3. 在准备好的平板上接种神经元(第 0 天)
- 为了在第0天将神经元接种到准备好的平板上,将包被的平板从冰箱中取出,将它们放在细胞培养罩下,让它们平衡至室温约30分钟。
- 在接种前,用自动液体处理器或 16 通道移液器吸出 15 μL 包衣溶液。每孔留出约 10 μL,以防止损坏涂层。
- 接下来,用 16 通道移液管每孔分配 50 μL 含有 300,000 个细胞/mL(在步骤 2.6 中制备)的细胞溶液,每孔产生 15,000 个接种神经元,每孔最终体积为 60 μL。
- 在 384 孔板中,避免使用第 1、2、23、24 列和 A、B、O 和 P 行,以尽量减少可能影响表型谱的边缘效应。用 80 μL PBS 填充未使用的空孔。将板在37°C和5%CO2下孵育。
4. 介质变化或复方处理(第 3 天)
- 根据孔数,在室温下预热适当体积的完全维持培养基。
- 如果需要更改介质,请继续执行步骤 4.4。如果需要复合处理,请验证化合物原液浓度和使用的溶剂(水、DMSO、甲醇等)。
- 制备1.5倍浓缩化合物溶液,以测试所有所需浓度。使用完全维持培养基制备化合物稀释液。
注意:作为中性对照,使用与被测化合物浓度相同的相应溶剂。如果使用不同浓度的多种或未知化合物,建议进行单独的剂量反应实验,以测量溶剂对表型特征的影响。 - 如果使用自动移液系统,则将 60 μL 的 1.5x 化合物溶液加入 384 孔储存板中。或者,使用 16 通道移液器。如果需要更换培养基,请添加 60 μL 完全维持培养基。
- 使用自动移液系统,从含神经元的板中每孔吸出并弃去 40 μL 培养基,以保持 20 μL/孔。然后将 40 μL/孔的 1.5x 化合物溶液从 384 孔储存板添加到每个孔中以获得所需的最终浓度。
注意:关键是不要进行完全的培养基更换,而是始终将残留培养基留在孔中,以防止损坏神经元地毯或涂层。 - 如果神经元培养超过本协议中描述的6天,则每2-3天更换一次培养基。
5. 神经元固定和染色(第 6 至 7 天)
- 为了在第 6 天和第 7 天固定和染色神经元,请使用自动液体处理器进行所有分配和洗涤步骤。或者,使用 16 通道移液器。
- 通过在1x PBS中稀释Triton X-100来制备10%Triton X-100溶液。涡旋直到溶液均匀。储存在4°C。
- 为了固定神经元,分配 20 μL/孔的 16% PFA,最终浓度为 4%。将板在室温下孵育30分钟,并用1x PBS洗涤3次。最后一次洗涤后留下 20 μL/孔的 PBS。
注意:PFA 被认为是一种已知会引起口腔、皮肤和呼吸道毒性的有害物质。它还对眼睛构成威胁,并可能导致基因突变和癌症。正确处理PFA除了确保适当的通风外,还需要使用合适的个人防护设备,例如眼睛和面部防护装置。防止PFA释放到环境中是很重要的。 - 对于透化和封闭,准备2x封闭溶液(表2)。
- 加入 20 μL/孔的 2x 封闭溶液(1x 终浓度),在室温下孵育 1 小时,并用 PBS 洗涤一次。洗涤后保留 20 μL/孔的 PBS。
- 对于一抗(参见 材料表)染色,制备2x一抗染色缓冲液(表3)。
- 加入20μL/孔的2x原染色缓冲液(1x终浓度),并在4°C下孵育过夜。 第二天早上,用PBS清洗3次。洗涤后留下 20 μL/孔的 PBS。
- 对于二抗(参见 材料表)染色,制备2x二抗染色缓冲液(表4)。
- 加入 20 μL/孔的 2x 二染色缓冲液(1 x 终浓度),在室温下避光孵育 2 小时,并用 PBS 洗涤 3 次。最后一次洗涤后留下 100 μL PBS/孔。
- 在板上添加铝密封,以尽量减少蒸发。或者,用塑料薄膜和铝箔盖住板。继续进行图像采集,或将板储存在4°C。
注意:该协议可以在此处暂停长达一周。如果观察到背景荧光,请考虑增加阻断时间。如果染色不足,请尝试增加一抗浓度或孵育时间。
- 在板上添加铝密封,以尽量减少蒸发。或者,用塑料薄膜和铝箔盖住板。继续进行图像采集,或将板储存在4°C。
6. 荧光染色神经元成像(第 7 天)
- 在第 7 天获取铺板、培养和染色神经元的图像。理想情况下,使用自动共聚焦荧光显微镜(参见 材料表)。或者,手动获取图像。
- 分别使用 405 nm、488 nm、561 nm 和 647 nm 激光获取 Hoechst、TH、α-突触核蛋白和 MAP2 通道(图 2)。
注:要生成足够数量的详细数据用于表型分析,请使用 40 倍物镜并使用由 3 个相隔 2 μm 的 Z 切片组成的 Z 堆栈采集 16 个田/孔。如果神经元地毯中存在与移液相关的损伤,请尽量避免对这些区域进行成像。 - 根据显微镜和相机的不同,分别调整四个荧光通道中每个通道的曝光时间和激发强度,以获得荧光强度的最佳动态范围。
注意:成像软件通常提供直方图来确定理想的曝光时间。如果直方图在低信号范围内向左偏移太多,则曝光时间太短或激发强度太低。如果右侧最大信号电平处出现急断,则信号值饱和。在这种情况下,请降低激发强度或缩短曝光时间。 - 以无损和开放的格式存储图像,例如 .tif。
7. 图像处理(第 8 天)
- 创建定量表型图谱需要图像分割和表型特征提取。使用PhenoLink软件进行图像分割和特征提取(材料表)。PhenoLink的安装说明可以在GitHub存储库(https://github.com/Ksilink/PhenoLink)中找到。
注意:图像分割用于识别和分离图像中的不同对象或区域,而表型特征提取用于分析和提取这些区域的相关信息。CellProfiler 10、ImageJ/FIJI11、Napari 12 或 Knime Analytics Platform13 等多种替代软件解决方案可用于从多通道荧光图像中提取定量信息。 - 对经过照明校正的原始图像执行图像分割。根据经验确定每个板的相应荧光通道强度阈值,使背景信号最小,并且所需的分段信号对应于原始图像中的信号。在同一天处理和染色的板通常需要相当的通道强度阈值才能进行分割。
- 定义细胞核大小和强度,将活细胞与死细胞分开。使用 40 倍图像时,请保留所有其他默认参数并运行软件。每孔将计算 126 个定量图像特征(补充表 1)。
- 使用得到的表格定量数据来构建表型图谱,并比较来自不同细胞系或处理条件的表型图谱。每行对应一个生物学条件(井),每列对应一个确定的表型特征。
注意:我们提供了一个示例输出文件以及数据分析管道来说明其用法(请参阅 材料表)。此外,图 3 显示了表型 图 谱的组成。
8. 表型图谱生成和可视化(第 8 天)
- 如果您的计算机上没有安装 Python 和 Jupyter,请安装 Anaconda 发行版并打开 Jupyter 软件。下载提供的 Jupyter 笔记本和所有其他提供的文件,并将它们保存在同一目录中(请参阅 材料表)。使用 Jupyter 软件打开 Jupyter 笔记本文件。
注意:Anaconda 是一个免费的开源平台,用于编程语言(如 Python)。该平台附带了 Python 解释器 Jupyter,它可以执行提供的 Jupyter 笔记本来创建和分析表型图谱 (https://github.com/Ksilink/Notebooks/tree/main/Neuro/DopaNeuronProfiling)。 - 使用 Jupyter 软件逐个单元执行 Jupyter 笔记本。提供的示例数据 .fth 和要求 .txt 文件需要与 Jupyter 笔记本位于同一目录中。每个 Jupyter 笔记本单元都带有注释,以解释其功能。
注意:从数据加载和缩放开始,以正确的顺序使用 Jupyter 笔记本,并逐个单元格地进行到最后,这一点至关重要。所有数据和图形输出都将存储在 Jupyter 笔记本源目录中新创建的文件夹中。 图 4 描述了工作流和工作流输出。
结果
mDA神经元中的表型分析是量化细胞生物学的多个方面及其在实验调节过程中的变化的有效方法。为了举例说明这种方法,本研究使用了冷冻保存的 LRRK2 G2019S 和健康的供体 mDA 神经元。这些神经元已经分化了大约 37 天,是有丝分裂后和表达神经元标志物(TUBB3 和 MAP2)和多巴胺能神经元标志物,包括酪氨酸羟化酶 (TH) 与 FOXA2 联合使用,而神经胶质标志物胶质纤维酸性蛋白 (GFAP) 缺失
讨论
表型分析是一种通过应用荧光染色、显微镜和图像分析来测量细胞中大量表型的技术3。可以跨细胞系或其他实验条件获得和比较表型图谱,以了解细胞生物学中的复杂变化,这些变化在使用单次读数时可能会被忽视。在这里,我们描述了表型分析在人类 iPSC 衍生的 mDA 神经元中的应用,这是一种经常用于模拟 PD 细胞生物学的细胞类型17,18,19。
披露声明
所有作者均受雇于Ksilink。
致谢
作者要感谢Ksilink的所有同事,感谢他们为所提出的方案设计提供的宝贵帮助和讨论。
材料
| Name | Company | Catalog Number | Comments |
| Anti- chicken – Alexa 647 | Jackson ImmunoRearch | 703-605-155 | Immunofluorescence |
| Anaconda | https://www.anaconda.com/download | ||
| Anti-Map2 | Novus | NB300-213 | Immunofluorescence |
| Anti-mouse - Alexa 488 | Thermo Fisher | A11001 | Immunofluorescence |
| Anti-rabbit - Alexa 555 | Thermo Fisher | A21429 | Immunofluorescence |
| Anti-Tyrosine Hydroxylase | Merck | T2928 | Immunofluorescence |
| Anti-α-synuclein | Abcam | 138501 | Immunofluorescence |
| Bravo Automated Liquid Handling Platform with 384ST head | Agilent | If no liquid handler is available, the use of an electronic multichannel pipette is recommended. | |
| Confocal microscope | Yokogawa | CV7000 | The use of an automated confocal fluorescence microscope is recommended to ensure image quality consistency. |
| Countess Automated cell counter | Invitrogen | Cell counting before seeding. Can also be done using a manual counting chamber. | |
| DPBS +/+ | Gibco | 14040-133 | Buffer for washing |
| EL406 Washer Dispenser | BioTek (Agilent) | If no liquid handler is available, the use of an electronic multichannel pipette is recommended. | |
| Formaldehyde Solution (PFA 16 %) | Euromedex | EM-15710-S | Fixation before staining |
| Hoechst 33342 | Invitrogen | H3570 | Nuclear staining |
| iCell Base Medium 1 | Fujifilm | M1010 | Base medium for neurons |
| iCell DPN, Donor#01279, Phenotype AHN, lot#106339, 1M | Fujifilm | C1087 | Apparently healthy donor |
| iCell DPN, Donor#11299, Phenotype LRRK2 G2019S, phenotype PD lot#106139 | Fujifilm | C1149 | Donor carrying LRRK2 G2019S mutation |
| iCell Nervous System Supplement | Fujifilm | M1031 | Supplement for base medium |
| iCell Neural Supplement B | Fujifilm | M1029 | Supplement for base medium |
| Jupyter Python Notebook | In-house development | https://github.com/Ksilink/Notebooks/tree/main/Neuro/DopaNeuronProfiling | Notebook to perform phenotypic profile visualization and classification from raw data. |
| Laminin | Biolamina | LN521 | Plate coating |
| PFE-360 | MedChemExpress | HY-120085 | LRRK2 kinase inhibitor |
| PhenoLink | In-house development | https://github.com/Ksilink/PhenoLink | Software for image analysis |
| PhenoPlate 384w, PDL coated | Perkin Elmer | 6057500 | Pre-coated plate for cell culture and imaging. This plate allows imaging of all wells using all objectives of the Yokogawa CV7000 microscope. |
| Storage plates Abgene 120 µL | Thermo Scientific | AB-0781 | Necessary for compound dispensing using the Vprep pipetting system. If not available, the use of an electronic multichannel pipette is recommended. |
| Triton | Sigma | T9284 | Permeabilization before lysis |
| Trypan Blue | Sigma | T8154-20ML | Determination of living cells |
| Vprep Pipetting System | Agilent | Medium change and compound dispensing. Alternatively, an electronic multichannel pipette can be used. |
参考文献
- Panicker, N., Ge, P., Dawson, V. L., Dawson, T. M. The cell biology of Parkinson's disease. The Journal of Cell Biology. 220 (4), 202012095 (2021).
- Caicedo, J. C., et al. Data-analysis strategies for image-based cell profiling. Nature Methods. 14 (9), 849-863 (2017).
- Chandrasekaran, S. N., Ceulemans, H., Boyd, J. D., Carpenter, A. E. Image-based profiling for drug discovery: due for a machine-learning upgrade. Nature Reviews Drug Discovery. 20 (2), 145-159 (2021).
- Bray, M. -. A., et al. Cell Painting, a high-content image-based assay for morphological profiling using multiplexed fluorescent dyes. Nature Protocols. 11 (9), 1757-1774 (2016).
- Schiff, L., et al. Integrating deep learning and unbiased automated high-content screening to identify complex disease signatures in human fibroblasts. Nature Communications. 13 (1), 1590 (2022).
- Ziegler, S., Sievers, S., Waldmann, H. Morphological profiling of small molecules. Cell Chemical Biology. 28 (3), 300-319 (2021).
- Cobb, M. M., Ravisankar, A., Skibinski, G., Finkbeiner, S. iPS cells in the study of PD molecular pathogenesis. Cell and Tissue Research. 373 (1), 61-77 (2018).
- Elitt, M. S., Barbar, L., Tesar, P. J. Drug screening for human genetic diseases using iPSC models. Human Molecular Genetics. 27 (R2), 89-98 (2018).
- Vuidel, A., et al. High-content phenotyping of Parkinson's disease patient stem cell-derived midbrain dopaminergic neurons using machine learning classification. Stem Cell Reports. 17 (10), 2349-2364 (2022).
- Stirling, D. R., Swain-Bowden, M. J., Lucas, A. M., Carpenter, A. E., Cimini, B. A., Goodman, A. CellProfiler 4: improvements in speed, utility and usability. BMC Bioinformatics. 22 (1), 433 (2021).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Sofroniew, N., et al. . napari: a multi-dimensional image viewer for Python. , (2022).
- Berthold, M. R., et al. KNIME: The Konstanz Information Miner. Data Analysis, Machine Learning and Applications. , 319-326 (2008).
- Fathi, A., et al. Diverging Parkinson's Disease Pathology between patient-derived GBAN370S, LRRK2G2019S and engineered SNCAA53T iPSC-derived Dopaminergic Neurons. bioRxiv. , (2023).
- Wang, Y., Huang, H., Rudin, C., Shaposhnik, Y. Understanding How Dimension Reduction Tools Work: An Empirical Approach to Deciphering t-SNE, UMAP, TriMap, and PaCMAP for Data Visualization. Journal of Machine Learning Research. 22 (201), 1-73 (2021).
- Ke, G., et al. LightGBM: A highly efficient gradient boosting decision tree. Advances in Neural Information Processing Systems. 30, (2017).
- Avazzadeh, S., Baena, J. M., Keighron, C., Feller-Sanchez, Y., Quinlan, L. R. Modelling Parkinson's Disease: iPSCs towards Better Understanding of Human Pathology. Brain Sciences. 11 (3), 373 (2021).
- Sánchez-Danés, A., et al. Disease-specific phenotypes in dopamine neurons from human iPS-based models of genetic and sporadic Parkinson's disease. EMBO Molecular Medicine. 4 (5), 380-395 (2012).
- Oosterveen, T., et al. Pluripotent stem cell derived dopaminergic subpopulations model the selective neuron degeneration in Parkinson's disease. Stem Cell Reports. 16 (11), 2718-2735 (2021).
- Hughes, R. E., et al. Multiparametric high-content cell painting identifies copper ionophores as selective modulators of esophageal cancer phenotypes. ACS Chemical Biology. 17 (7), 1876-1889 (2022).
- Akbarzadeh, M., et al. Morphological profiling by means of the Cell Painting assay enables identification of tubulin-targeting compounds. Cell Chemical Biology. 29 (6), 1053-1064 (2022).
- Schiff, L., et al. Integrating deep learning and unbiased automated high-content screening to identify complex disease signatures in human fibroblasts. Nature Communications. 13 (1), 1590 (2022).
- Way, G. P., et al. Morphology and gene expression profiling provide complementary information for mapping cell state. Cell Systems. 13 (11), 911-923 (2022).
- Feng, Y., Mitchison, T. J., Bender, A., Young, D. W., Tallarico, J. A. Multi-parameter phenotypic profiling: using cellular effects to characterize small-molecule compounds. Nature Reviews Drug Discovery. 8 (7), 567-578 (2009).
- Antonov, S. A., Novosadova, E. V. Current state-of-the-art and unresolved problems in using human induced pluripotent stem cell-derived dopamine neurons for parkinson's disease drug development. International Journal of Molecular Sciences. 22 (7), 3381 (2021).
- Miller, J. D., et al. Human iPSC-based modeling of late-onset disease via progerin-induced aging. Cell Stem Cell. 13 (6), 691-705 (2013).
- Bezard, E., Gross, C. E., Brotchie, J. M. Presymptomatic compensation in Parkinson's disease is not dopamine-mediated. Trends in Neurosciences. 26 (4), 215-221 (2003).
- Wu, Y., Le, W., Jankovic, J. Preclinical Biomarkers of parkinson disease. Archives of Neurology. 68 (1), 22-30 (2011).
- Verstraelen, P., et al. Systematic quantification of synapses in primary neuronal culture. iScience. 23 (9), 101542 (2020).
- Liu-Yesucevitz, L., et al. ALS-Linked mutations enlarge TDP-43-enriched neuronal RNA granules in the dendritic arbor. The Journal of Neuroscience. 34 (12), 4167-4174 (2014).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。