需要订阅 JoVE 才能查看此. 登录或开始免费试用。
在计算机中合成用活性羰基化合物修饰的氨基酸,以使用分子动力学模拟评估结构效应
摘要
在这里,我们描述了一种用活性羰基物种修饰的氨基酸残基的优化和参数化方案,适用于蛋白质系统。实验步骤包括结构设计和优化、电荷分配、参数构建和蛋白质系统的制备。
摘要
脂质过氧化衍生的反应性醛对蛋白质进行羰基化,导致蛋白质交联、寡聚化和聚集,导致细胞内损伤、细胞功能受损,并最终导致细胞死亡。它已在衰老和几种与年龄相关的慢性病中被描述。然而,与蛋白质靶标功能丧失相关的结构变化的基础仍不清楚。因此,描述了用脂肪酸氧化衍生的活性羰基物种羰基化的氨基酸的计算机构建新参数的途径。构建了具有 4-羟基-2-壬烯醛 (HNE)、4-羟基-2-己烯醛 (HHE) 的 Cys、His 和 Lys 的 Michael 加合物,以及 4-氧代-2-壬烯醛 (ONE) 的呋喃环形式,而丙二醛 (MDA) 直接连接到每个残基上。该协议描述了构造、几何优化、电荷分配、缺失键、角度、二面角参数及其对每个改性残基结构的验证的详细信息。因此,通过对不同蛋白质系统(如硫氧还蛋白、牛血清白蛋白和膜 Zu-5-锚蛋白结构域)进行分子动力学模拟,采用均方根偏差 (RMSD)、均方根波动 (RMSF)、结构二次预测 (DSSP) 和溶剂可及表面积分析 (SASA),测量了与这些脂质衍生物羰基化诱导的结构效应, 等等。
引言
在不断追求了解具有氧化修饰的蛋白质的分子行为的过程中,计算化学已成为广泛科学研究领域的基本支柱。这依赖于使用能够解释电子系统中物理现象的理论模型,使用数学方程来描述分子的原子行为。在这种环境中,蛋白质的计算模拟是分析分子系统原子行为的关键工具。基于对结构行为、能量计算和构象状态1 的评估,这些方法成为预测生物分子系统行为的战略盟友。
这些模拟专门用于研究结构变化和评估蛋白质系统中生物功能的丧失或获得。然而,当应用于包含由序列中的共价翻译后修饰形成的修饰残基的蛋白质系统时,计算方法显示出明显的局限性。这是因为许多可用方法缺乏参数适应力场的资源,而这些力场与最常见的蛋白质分子动力学模拟程序包兼容 2,3,4,5,6。因此,计算软件兼容的力场自适应参数的标准化对于促进拓扑和原子坐标与控制系统势能方程7 的精确耦合至关重要。
为了应对这些挑战,已经使用 从头到尾 的方法开发了一种适用于新的修饰氨....
研究方案
1. 新型改性氨基酸的设计与优化
注意:此阶段涉及绘制修饰残基的结构并优化其能量。
- 设计修改后的结构并优化其结构。
- 使用计算化学软件包绘制与脂质过氧化衍生的反应性醛结合的氨基酸分子,即使用 HNE、HHE、MDA 和 ONE。一旦修饰,在氨基酸的羧基末端绘制出甲胺基的形状。在氨基端,画一个乙酰基来模拟修饰氨基酸的肽键,如图 1 所示。
- 单击 Clean 图标 进行结构清理。对于结构优化,请点击 Calculate > Gaussian calculation setup... ,或 Ctrl+G,然后点击 General ,取消选中 Write connectivity。单击 Job Type > Optimization,如图 2 所示。在其他关键字中,键入以下行:
SCF=紧密测试 Pop=MK iop(6/33=2) iop(6/42=6) opt
注意:在这里,GaussView 会自动将 Hartree Fock (HF) 设置为泛函,并将基设置为....
结果
为了说明协议的实施并评估结果,将考虑以下分析。通过为修饰的氨基酸残基分配新参数生成的数据集是基于电子结构的优化构建的,这些电子结构支持部分 RESP 负载。 图 9 显示了使用参数分配优化的一个氨基酸残基的结构构象。
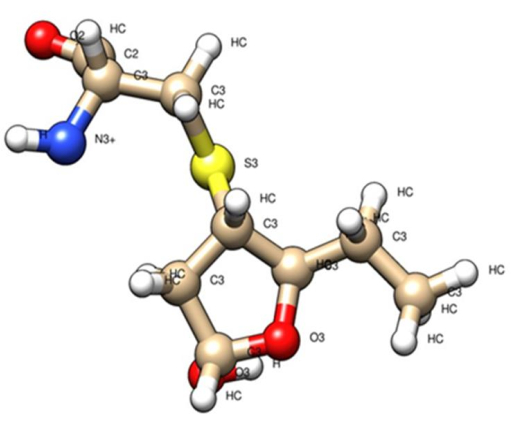
图 9.......
讨论
开发 AMBER 参数化方案的关键步骤之一是对用脂质过氧化衍生物修饰的新氨基酸残基进行量子优化,因为与 AMBER 前室中的最小化和分配 RESP 电荷的方式相关的能量可变性。为此 ,使用Hartree-Fock (HF/6-31G) 和半经验密度泛函理论 (DFT;B3LYP/6-31G 和 M062X/6-31G) 的建立来评估对载荷分配的响应。因此,HF 函数呈现出更好的性能/计算成本比,将此作为协议的前一步考虑在内。周 et al.1.......
披露声明
作者没有什么可披露的。
致谢
这项工作得到了科学部、Tecnología e Innovación (Minciencias) 和卡塔赫纳大学 (Colombia) 的研究资助代码 1107-844-67943 的支持,以支持 2021 年研究小组和 Acta 017-2022。
....材料
| Name | Company | Catalog Number | Comments |
| AmberTools16 or Upper | The Amber Project | Amber is a suite of biomolecular simulation programs | |
| Gaussian 09 or Upper | Gaussian Inc | Draw and optimize structures | |
| Linux Ubuntu | GNU/Linux | Platform for AmberTools | |
| NVIDIA GPUs GTX 1080 or Upper | Nvidia | Compatible with PMEMD |
参考文献
- Cornell, W. D., et al. A second generation force field for the simulation of proteins, nucleic acids, and organic molecules. J Am Chem Soc. 117 (19), 5179-5197 (1995).
- Wang, J., Wolf, R. M., Caldwell, J. W., Kollman, P. A., Case, D. A.
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。