需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
单动物、单管 RNA 提取,通过 qRT-PCR 比较缓步动物 Hypsibius 示例中的相对转录水平
摘要
这项工作提出了一种快速 RNA 提取和转录水平比较方法,用于分析缓步动物 Hypsibius exemplaris 中的基因表达。使用物理裂解,这种高通量方法需要单个缓步动物作为起始材料,并产生用于定量逆转录聚合酶链反应 (qRT-PCR) 的 cDNA。
摘要
缓步动物 Hypsibius exemplaris 是一种新兴的模式生物,以其在极端环境中生存的能力而闻名。为了探索这种极端耐受性的分子机制和遗传基础,许多研究依赖于 RNA 测序 (RNA-seq),该测序可以对从大型群体到个体动物的种群进行。随后使用逆转录聚合酶链反应 (RT-PCR) 和 RNA 干扰 (RNAi) 分别用于确认 RNA-seq 结果并评估候选基因的遗传要求。此类研究需要一种高效、准确且经济实惠的 RNA 提取方法,并通过定量 RT-PCR (qRT-PCR) 测量相对转录水平。本研究提出了一种高效的单缓步动物、单管 RNA 提取方法 (STST),该方法不仅可靠地从单个缓步动物中分离 RNA,而且还减少了每次提取所需的时间和成本。这种 RNA 提取方法产生的 cDNA 量可用于通过定量 PCR (qRT-PCR) 扩增和检测多个转录本。该方法通过分析编码两种热休克调节蛋白(热休克蛋白 70 β2 (HSP70 β2) 和热休克蛋白 90α (HSP90α) 的基因表达的动态变化来验证,从而可以使用 qRT-PCR 评估它们在热暴露个体中的相对表达水平。STST 有效地补充了现有的批量和单个缓步动物 RNA 提取方法,允许通过 qRT-PCR 快速且经济地检查单个缓步动物转录水平。
引言
缓步动物是一种小型多细胞动物,以其能够在对大多数其他生命形式致命的极端条件下生存而闻名1。例如,这些动物可以在对人类致命的电离辐射剂量的近 1000 倍中存活 2,3,4,5,6,7,8,9,10,几乎完全干燥 11,12,13,14,15,在没有添加的情况下冻结冷冻保护剂 16,17,18,甚至在干燥状态下,甚至是太空的真空 19,20。由于它们在极端环境中独特的生存能力,这些动物已成为理解复杂多细胞生物体极端耐受性的基础模型 1,21,22,23。
直到最近,这些非凡动物的稳定基因作,包括转基因和种系基因修饰,仍然难以捉摸24,25。因此,大多数揭示极端耐受分子机制的实验都是通过 RNA 测序的转录分析进行的。在各种极端条件下,缓步动物存在许多有价值且信息丰富的 RNA 测序数据集,范围从辐射8、9、26、27、28、热应激29、冷冻应激12 和干燥27、30、31、32、33.其中一些研究利用了大量 RNA 提取和纯化方法来阐明我们对极端耐受性的分子理解。然而,从许多动物中批量提取 RNA 转录本会阻止分析个体之间基因表达的变异,从而错过更精细数据集的潜在丰富性。重要的是,这些研究通常分析动物的异质种群,包括在环境压力源中存活的动物和不能存活的动物。因此,这些研究因平均来自多种且可能截然不同的反应状态的表达数据而感到困惑。为了解决这个问题,Arakawa 等人,201634 开发了一种优雅的低起始量 RNA-seq 管道,该管道应用 RNA 提取试剂盒,然后使用单个 34,35,36 或多个 30,37,38 只动物作为输入进行线性 PCR 扩增步骤。这些研究是我们理解缓步动物极端耐受性22 的基础。有趣的是,该方案也已应用于使用 7 只动物作为起始材料的 qRT-PCR24。
在大多数模式生物中,通过 RNA-seq 确定了潜在靶标,然后进行 qRT-PCR 以确认 RNA-seq 识别的转录变化,并以高分辨率方式评估候选基因的表达时间进程。为了测试已鉴定基因的功能,此类研究通常随后进行 RNAi 介导的分子靶标敲低39,40 和极端耐受性能力分析12,41。通常通过 qRT-PCR 直接监测转录本丰度的降低来确认每种 RNAi 敲低的功效。然而,RNAi 在缓步动物中是一个劳动密集型过程,因为每个 dsRNA 都必须通过个体的手动显微注射来递送39,40。由于该策略的低通量性质,适用于从单只动物进行 qRT-PCR 的快速、低成本 RNA 提取方法对于缓步动物研究将非常有价值。尽管之前已经开发了从单个缓步动物中提取 RNA 的方法,但这些方案并未将其提取与 qRT-PCR 相结合,而是依赖于基于光密度的方法 12,40,41。在这些挑战的推动下,我们试图开发一种方案,该方案能够在数量和质量上可靠地产生可用于单个 H. exemplaris 的 qRT-PCR。
STST 改编自为秀丽隐杆线虫 42 开发的单动物 RNA 提取方案,针对 H. exemplaris 进行了优化。提取方法包括六个快速冻融步骤,物理破坏角质层,允许 RNA 提取和随后的 cDNA 合成。如Boothby等人,201843 年,与大量 RNA 提取方法相比,STST 方法将提取时间缩短了 24 倍以上,与单个缓步动物 RNA 提取试剂盒相比,提取时间缩短了 30%,如 Arakawa 等人,201634 年所述。此外,与 RNA 提取试剂盒制备相比,样品-实验者相互作用的次数从 5 次减少到仅 1 次,从而降低了外源性核糖核酸酶污染的风险。在查询高表达基因时,STST 方法可为每个缓步动物产生足够的 cDNA 进行 25 次定量 RT-PCR 反应,每次反应只需 25 μL cDNA 总体积中的 1 μL。然而,对于丰度较低的转录本,需要根据经验确定模板浓度。
通过研究编码热休克蛋白 90α (HSP90α) 和热休克蛋白 70β2 (HSP70β2) 的基因在 35°C 下响应短期热休克 20 分钟的差异表达,评价 STST 方法分析基因表达动态变化的有效性。大多数真核生物中的 HSP70β2 和 HSP90α 在短期热休克暴露(20 分钟)后迅速上调42。对 H. exemplaris 的分析表明,从单个热处理的缓步动物中提取的 HSP70β2 和 HSP90α 编码 RNA 在短期热暴露后表达均显示出统计学上的显着增加。这些发现表明 STST 方案可用于分析个体动物基因表达随时间的动态变化。
STST 提取方法应通过促进快速和廉价的 RNA 提取以及随后通过 qRT-PCR 比较转录水平来补充现有的实验方法,例如 RNA-seq。与单独光密度相比,该方法对于更定量地评估手动注射个体中 RNAi 的效率和外显率也很有价值。最后,由于它们相似的表皮结构和物理特性,这种方法很可能也对分析其他缓步动物物种的基因表达有效44。
研究方案
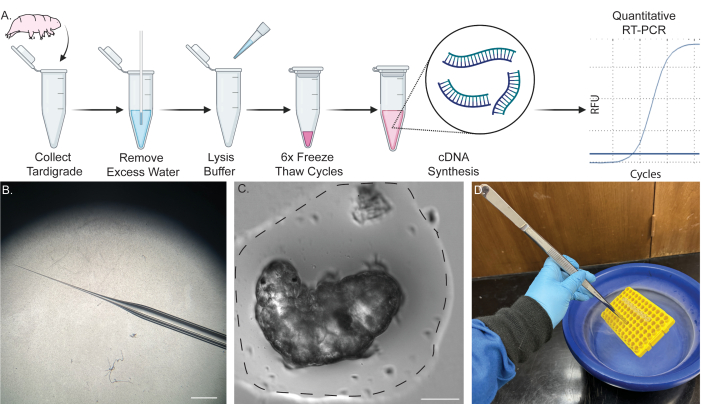
图 1:从单个缓步动物中提取 RNA 的单管管道。 (A) 示意图显示了从单个缓步动物中提取 RNA 的方案,包括六个冻融循环和随后的 cDNA 合成。样本随后可用于 RT-PCR 和 qRT-PCR。(B) 用于去除水分的微量移液器锥度的图像。比例尺:2 毫米。(C) 缓步动物在少量水中(虚线)的明场图像。成功提取需要去除大部分水分,并防止裂解缓冲液稀释。比例尺:50 μm。(D) 图像显示使用长镊子将样品浸入液氮中,以快速安全地冻融样品。一些内容是在 BioRender 中创建的。Kirk, M. (2022) BioRender.com/d93s511 请单击此处查看此图的较大版本。
注意:图 1A 显示了该过程的示意图。有关缓步动物和藻类培养程序的详细信息,请参阅以前发布的报告 45,46,47。
1. 泉水的杀菌
- 将 2 L 泉水从 5 加仑水壶中倒入 5 L 矿泉中(具体情况见 材料表 )到 2 L 高压灭菌器安全玻璃瓶中。
- 将盖子盖在高压灭菌器安全瓶上,并用少量高压灭菌胶带密封。不要拧紧瓶子;将盖子放在上面。
- 在湿循环中将泉水高压灭菌 50 分钟,无干燥步骤。
- 让水达到室温 (RT),并在 RT 中储存之前将盖子密封。
2. 玻璃微量移液器拉动(带移液器拉拔器)
- 将玻璃微量移液器(外径:1 毫米,内径:0.58 毫米,长度:10 厘米)固定在微量移液器拉拔器上。避免接触加热丝,因为这会改变移液器形状并损坏加热丝。
- 根据经验确定每个细丝和移液器拉拔器的移液器拉力。但是,作为优化的起点,请使用 78 °C 和 182.2 g 的拉力步长。
- 让细丝加热和重力,将玻璃微量移液器分成两个带有尖头的玻璃微量移液器(图 1B)。
- 将这些拉出的玻璃微量移液器存放在带有蜡或粘土的 100 mm 密闭培养皿中,以将它们固定到位并防止锋利的尖端破裂。
3. 玻璃微量移液器拉动(不带移液器拉拔器)
- 以较低的设置点燃本生灯或其他受控火焰源。
- 取每只手一端的玻璃微量移液器。
- 将玻璃微量移液器的中心放在火焰上,直到玻璃开始熔化。然后,迅速将两端拉开。这将产生两个非常精致、锋利的尖端。
- 用一对无菌细镊子轻轻折断尖端。
- 将这些拉出的玻璃微量移液器存放在带有蜡或粘土的 100 mm 密闭培养皿中,以将它们固定到位并防止锋利的尖端破裂。
4. RNA 提取
- 在冷冻安全容器中获取 0.5 L 液氮。
注意:液氮是低温的,如果接触皮肤或眼睛可能会导致灼伤。处理时,请使用防护服、防溅护目镜、丁腈手套、冷冻手套、实验室外套和露趾鞋。在运输液体之前,请确定容器对液氮安全。对于此步骤,也可以使用乙醇干冰浴。 - 制作 cDNA 合成预混液:10 μL 溶液,含有 1 μL 随机六聚体引物、2 μL DNase、4 μL 5x RT 缓冲液、1 μL 酶混合物、1 μL H2O 和 1 μL 10 mM dNTP。将此溶液储存在冰上。
- 制备缓步动物裂解缓冲液(5 mM Tris (pH = 8)、0.5% (v/v) 去污剂 1、0.5% (v/v) 去污剂 2、0.25 mM EDTA,溶于无菌无核酸酶水中)。
注意:该溶液可在工作台上存放 6 个月。但是,请保持无菌并避免潜在的 RNAse 污染源。 - 分装足够的裂解缓冲液用于提取(2 μL/缓步生物)。
- 将 RNAse 抑制剂添加到缓步动物裂解缓冲液中,最终浓度为 4 U/μL。
- 在台式离心机上以 2000 x g 的速度在 RT 下以 2000 x g 的速度涡旋并旋转溶液 5 秒,然后将溶液储存在冰上。
- 使用无菌过滤嘴 P1000 移液器从培养物中去除实验所需的尽可能多的缓步动物,并将它们放入无菌的 35 mm 培养皿中。
注意:任何数量的缓步动物都可以以这种方式处理。通常,每个条件处理三只缓步动物进行提取。 - 使用 1 mL 高压灭菌的无菌泉水和无菌过滤头的 P1000 移液器清洗缓步动物 3 次。缓慢地上下吹打它们有助于去除藻类污染物。
- 使用 25 倍至 50 倍放大倍率的解剖显微镜,使用无菌滤嘴吸头的 P10 移液器将单个缓步动物从这种洗涤的培养物转移到新的无菌 35 mm 培养皿中。
- 使用无菌过滤喷嘴 P200 移液器在 100 μL 无菌无核酸酶水中洗涤单个缓步动物。
注:此洗涤步骤用于进一步去除污染物,包括核糖核酸酶。 - 使用无菌过滤喷嘴的 P10 移液器,将洗涤后的缓步动物转移到干净、无菌的 PCR 管底部,在 1-2 μL 无菌无核酸酶水中,小心确保缓步动物不会粘在尖端的侧面。
- 在解剖显微镜下以 25 倍放大倍率观察缓步动物。
- 为了便于去除水分,请在试管外轻轻折断拉出的玻璃微量移液器的尖端。确保孔足够大,可以拉起水,但不能拉起缓步动物。
- 使用拉动的玻璃微量移液器的毛细管作用,除去水,直到动物被直径约为两个缓步动物长度的小气泡包围。
- 通过解剖镜监测除水过程,以确保水位合适并且缓步动物保持水分。
注意: 图 1C 提供了要去除多少水的示例。这是一个关键的步骤。一个小水泡会围绕缓步动物以防止其变干,但应尽可能多地去除多余的水,以防止裂解缓冲液稀释。有关剩余水位的示例,请参阅 图 1C。 - 除去水后,立即在试管底部加入 2 μL 缓步动物裂解缓冲液,短暂涡旋,然后在台式离心机上以 2000 x g 的 RT 离心试管 5 秒。
- 立即将含有缓步动物的样品放入 PCR 管架中,并确保它们被架子紧紧固定。
- 使用一对长而粗的镊子夹住架子,然后将装有样品的架子轻轻浸入液氮中,直至完全冻结(图 1D)。
- 从液氮中取出架子,并立即将其放在冰上。让样品解冻(需要 ~45 秒到 1 分钟)。每 15 秒将样品从冰中取出并目视检查,以监测样品。样品明显透明后,继续下一步。
- 再重复步骤 4.18-4.19 五次。最大裂解和提取总共需要 6 次冻融循环(图 2A、B)。
- 冻融完成后,将样品置于冰上并立即进行下一步。此时不要冷冻样品进行储存,因为这会减少可用于 cDNA 制备的 RNA。
5. cDNA 合成
- 将 2 μL cDNA 合成预混液添加到含有缓步石裂解物的 PCR 管中。短暂轻弹试管,并在 RT 下以 2000 x g 的速度用台式离心机旋转 5 秒,然后在冰上更换样品。
- 将样品放入热循环仪中,在 25 °C 下孵育 10 分钟以退火引物,在 55 °C 下孵育 30 分钟以进行逆转录,最后,在 85 °C 下热灭活酶 5 分钟。
- 孵育后,立即将试管置于冰上,加入 21 μL 无菌无核酸酶水,将样品稀释至 25 μL 总体积。对于低拷贝数转录本,根据经验确定改变此稀释步骤。
6. qPCR 检测
- 使用从大量缓步动物制备的总 RNA 确定引物组的退火温度,例如,Boothby,201843 中提出的批量提取方法。
- 在运行 qRT-PCR 之前,运行 PCR 温度梯度以确定最佳退火温度(有关本方案中使用的所有 PCR 设置,请参阅 表 1 和 表 2)。
- 在冰上解冻一管指示剂染料超级混合物并避光隔离。将 96 孔 qPCR 板置于冰上,并在所需孔数中放入 5 μL 超级混合物、2 μL 水、1 μL 每种引物 (10 μM) 和 1 μL cDNA 产物。
- 用板密封密封 PCR 板,并使用适合引物组的退火温度运行 qRT-PCR(对于本文中使用的所有 qRT-PCR 设置,请参阅 表 3)。
7. 定量和结果解释
- 将结果定量地与一个或多个对照管家基因进行比较,预计其表达在施加的条件下是恒定的。在这项研究中,使用了肌动蛋白基因。
- 获取每个孔的 Ct 值或循环阈值,并将其与对照管家基因反应的 Ct 值进行比较。使用以下公式计算基因表达的倍数变化:
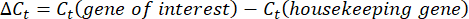
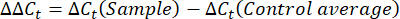
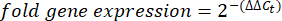
注:将每个转录本和缓步生物的倍数基因表达绘制为 2-(ΔΔCt)48。 - 要从 Ct 值获得转录数的粗略估计值,请使用以下公式:
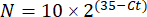
其中 N 是转录本的数量,2 是假设的 PCR 效率或每个 PCR 循环荧光增加的倍数48。
结果
单缓步动物 RNA 提取的开发和优化
采用 Ly等人,201542 的方案在缓步动物中提取 RNA,STST 系统经过优化以最大限度地提高制备的数量和质量(图 1A)。对肌动蛋白转录本进行 RT-PCR,通过扩增跨越外显子 1 和 2 的 527 bp 区域来定量转录本产量(这些引物的序列见 表 1)。用 ImageJ/FIJI 将预期肌动蛋白条带的光...
讨论
本研究提出了一种用于单缓步 qRT-PCR 的 RNA 提取的有效方法。将 STST 方法与现有的单个缓步动物 RNA 提取试剂盒直接进行比较,发现 STST RNA 提取产生的肌动蛋白 RNA 转录本量高出 >200 倍,将每个样品的成本降低到不到 1 美元,并将提取所需的时间缩短了 30%。为了将 STST 应用于相关的生物学问题,我们评估了短期热休克反应表达谱。我们发现 HSP70β2 和 HSP90α 的转录物,正如...
披露声明
作者声明没有利益冲突需要披露。
致谢
我们要感谢 NIH Ruth Kirschstein 奖学金 # 5F32AG081056-02 和 Errett Fisher 博士后奖学金,它支持 Molly J. Kirk 博士,Crowe 家庭奖学金,它支持徐超明,以及加州大学圣巴巴拉分校学术参议院助学金,以及 NIH #R01GM143771 和 #2R01HD081266 助学金,支持这些研究工作。作者还感谢加利福尼亚纳米系统研究所内的生物纳米结构实验室的使用,该实验室得到了加利福尼亚大学圣巴巴拉分校和加利福尼亚大学校长办公室的支持。
材料
| Name | Company | Catalog Number | Comments |
| 10 µL Premium Barrier Tips Low Binding, Racked, Sterile | Genesee Scientific | 23-401 | Refered to as Sterile Filter-Tipped P 10 Pipette Tips |
| 1000 µL Premium Pipet Tips, Low Binding, Racked, Sterile | Genesee Scientific | 23-165RS | Refered to as Sterile Filter-Tipped P 1000 Pipette Tips |
| 200 µL Premium Barrier Tips Low Binding, Racked, Sterile | Genesee Scientific | 23-412 | Refered to as Sterile Filter-Tipped P 200 Pipette Tips |
| 4 Star Straight Strong Medium Point Tweezer | Excelta | 00-SA-DC | Refered to as Long forceps |
| 96-Well PCR Rack with Lid Assorted, 5 Racks/Unit | Genesee Scientific | 27-202A | Refered to as PCR Rack |
| Andwin Scientific 3M LEAD FREE AUTOCLAVE TAPE 1" | Thermo Fisher Scientific | NC0802040 | Refered to as Autoclave Tape |
| Autoclave Tape | Thermo Fisher Scientific | AB1170 | Refered to as PCR Plate Seals |
| Benchling v8 | Benchling | N/A | Refered to as Benchling |
| BioRadHard-Shell 96-Well PCR Plate | BioRad | HSS9641 | Refered to as PCR Plate |
| BULWARK FR Lab Coat: | Grainger | 26CF64 | Refered to as Lab Coat |
| C1000 Touch Bio-rad Thermocycler | BioRad | 1851148 | Refered to as Thermocycler |
| C1000 Touch Bio-rad Thermocycler with CFX Optics Module | BioRad | 1845097 | Refered to as qPCR thermocycler |
| Chloroccoccum hypnosporum. | Carolina | 152091 | Refered to as Algae |
| Corning PYREX Reusable Media Storage Bottles | Thermo Fisher Scientific | 06-414-1E | Refered to as 2 L Autoclave-safe Glass Bottle |
| Daigger & Company Vortex-Genie 2 Laboratory Mixer | Thermo Fisher Scientific | 3030A | Refered to as Vortexer |
| Direct-zol Micro Prep | Zymo Research | R2060 | Refered to as RNA extraction kit |
| Dumont 5 Biology Tweezers | Fine Science Tools | 11254-20 | Refered to as Fine Forceps |
| EDTA | Fisher Scientific | S311-500 | Refered to as EDTA |
| FIJI v 2.14.0/1.54f | ImageJ, | N/A | Refered to as FIJI/ImageJ |
| Filament for pippette Puller | Tritech Research | PC-10H | Refered to as Filament |
| Fisherbrand Economy Impact Goggles | Fisher Scientific | 19-181-501 | Refered to as Splash Goggles |
| Glass Micropipette O.D. 1mm ID 0.58, Length 10 cm | TriTech Research | GD-1 | Reffered to as glass micropipette |
| Hypsibius exemplaris Z151 Strain | Carolina | 133960 | Refered to as Tardigrades or H. exemplaris |
| Liquid Nitrogen Dewar 1 L | Agar Scientific | AGB7475 | Refered to as Cryo-safe container |
| Maxima H Minus First Strand cDNA Synthesis Kit | Thermo Fisher Scientific | K1651 | Refered to as cDNA Synthesis Master Mix |
| Narishige Dual-Stage Glass Micropipette Puller | Tritech Research | PC-10 | Refered to as micropipette puller |
| Nitrile Gloves | Fisher Scientific | 17-000-314 | Refered to as Nitrile Gloves |
| PETRI DISH, PS, 35/10 mm, WITH VENTS | Grenier | 627102 | Refered to as 35 mm Petri dish |
| PIPETMAN P10, 1–10 µL, Metal Ejector | Gilson | F144055M | Refered to as P 10 Pipette |
| PIPETMAN P1000, 100–1000 µL, Metal Ejector | Gilson | F144059M | Refered to as P 1000 Pipette |
| PIPETMAN P200, 20–200 µL, Metal Ejector | Gilson | F144058M | Refered to as P 200 Pipette |
| Pound This 4-Color Modeling Clay | American Science Surplus | 96517P001 | Refered to as Clay |
| Prism v10.0 | GraphPad | N/A | Refered to a Prism |
| RNAse-Free, 8 Strip 0.2 mL PCR Tubes with caps | Invitrogen | AM12230 | Refered to as Sterile PCR Tube |
| RNasin Ribonuclease Inhibitor | Promega | N2111 | Refered to as RNAse inhibitor |
| Spring water | Nestle Pure Life | 44221229 | Refered to as Spring Water |
| SsoAdvanced Universal SYBR Green Supermix | BIO RAD | 1725271 | Refered to as Indicator Dye Super mix |
| Stereo-Microscope System w/optics and illumination | TriTech Research | SMT1 | Refered to as Dissecting Microscope |
| Supertek Scientific Tirrill Burners | Thermo Fisher Scientific | S09572B | Refered to as Bunsen Burner |
| Table Top Centrifuge | Qualitron | DW-41-115-NEW | Refered to as Table Top Centrifuge |
| Tempshield Cryo-Gloves | Fisher Scientific | 11-394-305 | Refered to as Cryo Gloves |
| Thermo Scientific Nunc Petri Dishes | Thermo Fisher Scientific | 08-757-099 | Refered to as 100 mm Petri dish |
| Tris base | Fisher Scientific | T395-500 | Refered to as Tris or Tris Base |
| Triton X-100 | Fluka | 93443 | Refered to as Detergent 1 |
| TWEEN 20 | Sigma aldrich | P1379-500 | Refered to as Detergent 2 |
| Water - PCR/RT-PCR certified, nuclease-free | Growcells | PCPW-0500 | Refered to as Sterile Nuclease Free Water |
参考文献
- Møbjerg, N., Neves, R. C. New insights into survival strategies of tardigrades. Comp Biochem Physiol Part A Mol Integr Physiol. 254, 110890 (2021).
- Jönsson, K. I., Harms-Ringdahl, M., Torudd, J. Radiation tolerance in the eutardigrade Richtersius coronifer. Int J Radiat Biol. 81 (9), 649-656 (2005).
- Horikawa, D. D., et al. Radiation tolerance in the tardigrade Milnesium tardigradum. Int J Radiat Biol. 82 (12), 843-848 (2006).
- Bruckbauer, S. T., Cox, M. M. Experimental evolution of extremophile resistance to ionizing radiation. Trends Genet. 37 (9), 830-845 (2021).
- Jönsson, K. I., Hygum, T. L., Andersen, K. N., Clausen, L. K. B., Møbjerg, N. Tolerance to gamma radiation in the marine heterotardigrade, Echiniscoides sigismundi. PLoS One. 11 (12), e0168884 (2016).
- Jönsson, K. I. Radiation tolerance in tardigrades: Current knowledge and potential applications in medicine. Cancers (Basel). 11 (9), 1333 (2019).
- Yoshida, Y., et al. RNA sequencing data for gamma radiation response in the extremotolerant tardigrade Ramazzottius varieornatus. Data Brief. 36, 107111 (2021).
- Clark-Hachtel, C. M., et al. The tardigrade Hypsibius exemplaris dramatically upregulates DNA repair pathway genes in response to ionizing radiation. Curr Biol. 34 (9), 1819-1830.e6 (2024).
- Anoud, M., et al. Comparative transcriptomics reveal a novel tardigrade specific DNA binding protein induced in response to ionizing radiation. Elife. 13, RP92621 (2024).
- Jönsson, K. I., Schill, R. O. Induction of Hsp70 by desiccation, ionising radiation and heat-shock in the eutardigrade Richtersius coronifer. Comp Biochem Physiol B Biochem Mol Biol. 146 (4), 456-460 (2007).
- Boothby, T. C. Desiccation of Hypsibius exemplaris. Cold Spring Harb Protoc. 2018 (11), 871-873 (2018).
- Boothby, T. C., et al. Tardigrades use intrinsically disordered proteins to survive desiccation. Mol Cell. 65 (6), 975-984.e5 (2017).
- Horikawa, D. D., Higashi, S. Desiccation tolerance of the tardigrade Milnesium tardigradum collected in Sapporo, Japan, and Bogor, Indonesia. Zoolog Sci. 21 (8), 813-816 (2004).
- Halberg, K. A., Jørgensen, A., Møbjerg, N. Desiccation tolerance in the tardigrade Richtersius coronifer relies on muscle mediated structural reorganization. PLoS One. 8 (12), e3330 (2013).
- Sørensen-Hygum, T. L., Stuart, R. M., Jørgensen, A., Møbjerg, N. Modelling extreme desiccation tolerance in a marine tardigrade. Sci Rep. 8 (1), 11495 (2018).
- Lyons, A. M., Roberts, K. T., Williams, C. M. Survival of tardigrades (Hypsibius exemplaris) to subzero temperatures depends on exposure intensity, duration, and ice-nucleation - as shown by large-scale mortality dye-based assays. bioRxiv. , (2024).
- Møbjerg, A., et al. Extreme freeze-tolerance in cryophilic tardigrades relies on controlled ice formation but does not involve significant change in transcription. Comp Biochem Physiol Part A Mol Integr Physiol. 271, 111245 (2022).
- Tsujimoto, M., Imura, S., Kanda, H. Recovery and reproduction of an Antarctic tardigrade retrieved from a moss sample frozen for over 30 years. Cryobiology. 72 (1), 78-81 (2016).
- Jönsson, K. I. Tardigrades as a potential model organism in space research. Astrobiology. 7 (5), 757-766 (2007).
- Jönsson, K. I., Rabbow, E., Schill, R. O., Harms-Ringdahl, M., Rettberg, P. Tardigrades survive exposure to space in low Earth orbit. Curr Biol. 18 (17), R729-R731 (2008).
- Kasianchuk, N., Rzymski, P., Kaczmarek, &. #. 3. 2. 1. ;. The biomedical potential of tardigrade proteins: A review. Biomed Pharmacother. 158, 113983 (2023).
- Arakawa, K. Examples of extreme survival: Tardigrade genomics and molecular anhydrobiology. Annu Rev Anim Biosci. 10 (1), 519-542 (2022).
- Hvidepil, L. K. B., Møbjerg, N. New insights into osmobiosis and chemobiosis in tardigrades. Front Physiol. 14, 1274522 (2023).
- Tanaka, S., Aoki, K., Arakawa, K. In vivo expression vector derived from anhydrobiotic tardigrade genome enables live imaging in Eutardigrada. Proc Natl Acad Sci U S A. 120 (5), e2216739120 (2023).
- Kondo, K., Tanaka, A., Kunieda, T. Single-step generation of homozygous knockout/knock-in individuals in an extremotolerant parthenogenetic tardigrade using DIPA-CRISPR. PloS Genet. 20 (6), e1011298 (2024).
- Yoshida, Y., Hirayama, A., Arakawa, K. Transcriptome analysis of the tardigrade Hypsibius exemplaris exposed to the DNA-damaging agent bleomycin. bioRxiv. , (2024).
- Yoshida, Y., et al. Time-series transcriptomic screening of factors contributing to the cross-tolerance to UV radiation and anhydrobiosis in tardigrades. BMC Genomics. 23 (1), 405 (2022).
- Yoshida, Y., et al. RNA sequencing data for gamma radiation response in the extremotolerant tardigrade Ramazzottius varieornatus. Data Brief. 36, 107111 (2021).
- Neves, R. C., et al. Differential expression profiling of heat stressed tardigrades reveals major shift in the transcriptome. Comp Biochem Physiol Part A Mol Integr Physiol. 267, 111143 (2022).
- Yoshida, Y., et al. Comparative genomics of the tardigrades Hypsibius dujardini and Ramazzottius varieornatus. PLoS Biol. 15 (7), e2002266 (2017).
- Wang, C., Grohme, M. A., Mali, B., Schill, R. O., Frohme, M. Towards decrypting cryptobiosis - analyzing anhydrobiosis in the tardigrade Milnesium tardigradum using transcriptome sequencing. PLoS One. 9 (3), e92663 (2014).
- Mali, B., et al. Transcriptome survey of the anhydrobiotic tardigrade Milnesium tardigradum in comparison with Hypsibius dujardini and Richtersius coronifer. BMC Genomics. 11 (1), 168 (2010).
- Förster, F., et al. Transcriptome analysis in tardigrade species reveals specific molecular pathways for stress adaptations. Bioinform Biol Insights. 6, 69-96 (2012).
- Arakawa, K., Yoshida, Y., Tomita, M. Genome sequencing of a single tardigrade Hypsibius dujardini individual. Sci Data. 3 (1), 160063 (2016).
- Arakawa, K. Transcriptome assembly of Richtersius coronifer with annotated BLAST result against Ramazzottius varieornatus. Figshare. Dataset. , (2019).
- Yoshida, Y., Konno, S., Nishino, R., Murai, Y., Tomita, M., Arakawa, K. Ultralow input genome sequencing library preparation from a single tardigrade specimen. J Vis Exp. (137), (2018).
- Murai, Y., et al. Multiomics study of a heterotardigrade, Echiniscus testudo, suggests convergent evolution of anhydrobiosis-related proteins in Tardigrada. bioRxiv. , (2020).
- Yoshida, Y., Sugiura, K., Tomita, M., Matsumoto, M., Arakawa, K. Comparison of the transcriptomes of two tardigrades with different hatching coordination. BMC Dev Biol. 19 (1), 24 (2019).
- Tenlen, J. R. Microinjection of dsRNA in tardigrades. Cold Spring Harb Protoc. 2018 (11), (2018).
- Tenlen, J. R., McCaskill, S., Goldstein, B. RNA interference can be used to disrupt gene function in tardigrades. Dev Genes Evol. 223 (3), 171-181 (2013).
- Giovannini, I., et al. Production of reactive oxygen species and involvement of bioprotectants during anhydrobiosis in the tardigrade Paramacrobiotus spatialis. Sci Rep. 12 (1), 15888 (2022).
- Ly, K., Reid, S. J., Snell, R. G. Rapid RNA analysis of individual Caenorhabditis elegans. MethodsX. 2, 59-63 (2015).
- Boothby, T. C. Total RNA extraction from tardigrades. Cold Spring Harb Protoc. 2018 (11), 905-907 (2018).
- Czerneková, M., Vinopal, S. The tardigrade cuticle. Limnol Rev. 21 (3), 127-146 (2021).
- McNuff, R. Laboratory culture of Hypsibius exemplaris. Cold Spring Harb Protoc. 2018 (11), 867-870 (2018).
- Gabriel, W. N., et al. The tardigrade Hypsibius dujardini, a new model for studying the evolution of development. Dev Biol. 312 (2), 545-559 (2007).
- Ruiz-Villalba, A., Ruijter, J. M., van den Hoff, M. J. B. Use and misuse of cq in qPCR data analysis and reporting. Life (Basel). 11 (6), 508 (2021).
- Antonov, J., et al. Reliable gene expression measurements from degraded RNA by quantitative real-time PCR depend on short amplicons and a proper normalization. Lab Invest. 85 (8), 1040-1050 (2005).
- Toussaint, J., et al. Improvement of the clinical applicability of the genomic grade index through a qRT-PCR test performed on frozen and formalin-fixed paraffin-embedded tissues. BMC Genomics. 10, 424 (2009).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。