需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
完整和再生蝾螈肢体软骨的原子力显微镜测量
* 这些作者具有相同的贡献
摘要
在该协议中,我们展示了如何为原子力显微镜 (AFM) 准备蝾螈组织,并在完整和再生的肢体软骨中进行压痕测量。
摘要
机械力为发育组织中的正常细胞功能和模式形成提供了重要信号,它们在胚胎发生和发病过程中的作用已得到广泛研究。相比之下,对动物再生过程中的这些信号知之甚少。
蝾螈是研究再生的重要模式生物,因为它能够在受伤后完全恢复许多器官和组织,包括缺失的软骨和骨骼。由于其作为脊椎动物身体中主要支撑组织的关键作用,在再生过程中恢复骨骼功能需要恢复缺失的结构及其机械性能。该协议描述了一种用于原子力显微镜 (AFM) 处理蝾螈肢体样品的方法,这是在高空间分辨率下探测细胞和组织力学特性的金标准。
利用蝾螈的再生能力,本研究测量了体内平衡和肢体再生的两个阶段:组织组织溶解和软骨凝结期间肢体软骨的刚度。我们表明 AFM 是深入了解动态组织重组和再生过程中发生的机械变化的宝贵工具。
引言
骨骼,尤其是软骨和骨骼,为脊椎动物身体的软组织提供主要的机械支撑。因此,骨骼系统中的任何损伤都可能极大地损害功能甚至生存。在人类中,骨折是最常见的创伤性损伤之一1,其中大多数会在几周内修复,但其中 5%-10% 会延迟愈合或从未完全恢复 2,3。此外,人类无法从广泛的骨骼或软骨流失中恢复过来 4,5。然而,一些蝾螈可以再生各种身体结构,包括完整的肢体6 (full limbs),这使它们成为研究骨骼再生的理想模型。
蝾螈 (Ambystoma mexicanum) 是一种蝾螈,其肢体再生已被广泛研究。这个过程发生在四个主要的连续但重叠的阶段:1) 伤口愈合,2) 炎症/组织溶解,3) 胚层形成,以及 4) 胚层生长/分化(7,8 中已论述)。截肢后,与损伤部位接壤的角质形成细胞迅速迁移,闭合伤口并形成伤口上皮 (WE)。在随后的炎症和组织溶解过程中,病原体被清除,碎片和受损细胞被清除,截肢表面下的细胞外基质 (ECM) 被重塑9。组织组织裂解对于肢体再生的发生至关重要10,其中蛋白水解酶的分泌不仅对整体 ECM 重塑至关重要,而且对于释放产生胚层的细胞和释放隔离在 ECM 本身中的生物活性分子也至关重要8。事实上,在许多再生环境和模式生物中的研究表明,ECM 在组织分解过程中的独特材料特性能够诱导去分化过程或引导细胞向损伤部位迁移(11 中已论述)。此外,在组织溶解的晚期钙化组织的再吸收已被证明是正确整合新形成的肢体骨骼元件的关键12。在组织溶解阶段之后,胚芽在伤口上皮 (WE) 下形成,作为由去分化成熟组织细胞或常驻干细胞产生的未分化、多谱系祖细胞的积累。Blastema 细胞增殖并分化成所有缺失的细胞类型。最后,发生肢体形态发生,骨骼组织通过源自 periskeletal 细胞和转分化真皮成纤维细胞的软骨生成器的浓缩再生 13,14,15。
尽管已经确定了许多调节细胞身份和 ECM 组成变化的生化线索 10,13,14,16,17,18,但肢体再生不同阶段的组织力学特性及其对再生的影响在很大程度上仍未得到探索。许多研究表明,细胞在多种情况下感知并整合调节其命运和行为的机械线索(19,20 年综述)。因此,用组织力学测量来补充我们对肢体再生的细胞和分子知识将大大提高我们对这些过程的理解。
已经开发了不同的技术,允许对生物样品进行机械表征和力操纵21。在这些技术中,原子力显微镜 (AFM) 已成为机械生物学的黄金标准,其中通过使用超灵敏的力传感器 AFM 悬臂22 压痕,以高空间分辨率探测生物样品的粘弹性。由于该技术需要与样品直接接触,因此通常会生成组织切片,这在某些情况下可能具有挑战性。因此,需要针对每个特定样品调整和优化制备条件,使其能够尽可能接近生理条件,并产生最少的伪影23。该协议描述了如何使用 AFM 测量蝾螈肢体的组织刚度,重点关注完整条件下的软骨组织,同时进行组织溶解和软骨凝结阶段(图 1 和 图 2)。这种方法也可以扩展到其他组织类型的测量。
研究方案
蝾螈 (Ambystoma mexicanum) 是在德累斯顿理工大学 (TUD) 德累斯顿再生疗法中心 (CRTD) 的蝾螈设施中生长的。饲养条件的完整描述可以在24 中找到。简而言之,将房间保持在 20-22 °C,昼夜循环为 12/12 小时。所有处理和外科手术均按照当地伦理委员会的指导方针进行,并已获得德国萨克森州州长的批准。
这项研究在所有实验中使用白色 (d/d) 蝾螈,这是一种天然存在的突变菌株,缺乏体色素沉着(很少或没有黑素细胞和黄细胞),虹膜细胞仅在眼睛的虹膜中。本研究使用了从鼻子到尾巴(5-7 个月大)长 8-15 厘米的蝾螈,没有性别特异性偏倚。
1. 准备工作
- 准备 10% (w/v) 苯佐卡因原液,用于蝾螈的麻醉和安乐死(见下文)。为此,将 50 g 苯佐卡因与 100% 乙醇混合在一个容量瓶中,直到达到 500 mL 的限制。
- 制备 0.03% (w/v) 苯佐卡因储备液。
- 对于 1 L,将 50 mL 的 10x Tris 缓冲盐水 (TBS) 与 30 mL 的 10% (w/v) 苯佐卡因和 5 mL 的 4000% (w/v) Holtfreter 溶液与 915 mL 蒸馏水混合,并与磁力搅拌棒混合过夜。
- 对于 1 L 10x TBS 溶液,将 24.2 g Trizma 碱和 90 g NaCl 与 990 mL 去离子水混合。与磁力搅拌棒充分混合。然后,加入大约 10 mL 的浓 HCl(12 M 或 37%)并调节 pH 值 8。
- 对于 1 L 4000% (w/v) Holtfreter 溶液,将 158.4 g NaCl、11.13 g MgSO4·7H2O、5.36 g CaCl2·2H2O 和 2.88 g KCl 与去离子水混合至 1 L。
- 通过以 1:3 的比例稀释 0.03% (w/v) 储备溶液,在储罐水中制备 0.01% (w/v) 工作苯佐卡因稀释液用于麻醉。
- 对于 1 L 溶液,使用 333 mL 苯佐卡因储备液和 666 mL 储水箱水。
- 制备 5 mg/mL 酒石酸布托啡诺储备液用于镇痛。为此,将 100 毫克布托啡诺 (+)-酒石酸盐与 20 毫升超纯水混合。将等分试样储存在 4 °C。
- 截肢当天,新鲜制备 0.5 mg/L 酒石酸布托啡诺工作溶液,通过在储罐水中以 1:10.000 的比例稀释储备溶液,以减轻外科手术后的动物疼痛。
- 对于长度小于 8 cm 的动物,将 20 μL 储备液混合到 200 mL 储水箱水中。
- 对于体型较大的动物,将 60 μL 储备液混合到 600 mL 储水箱水中。
- 制备无菌两栖动物磷酸盐缓冲盐水 (APBS),一种 80% (v/v) PBS 溶液,并通过将 800 mL DPBS 与 200 mL 去离子水混合,将其保持在室温 (RT) 下。
- 制备 1 mg/mL 胰岛素储备溶液。为此,将 250 mg 胰岛素粉与 25 mL HCl 0.1 M 混合,然后缓慢搅拌直至溶解。搅拌时,加入 225 mL APBS,直到溶液澄清。无菌过滤,并在 -20 °C 下储存 4 mL 等分试样。
- 准备无菌培养基(所有% (v / v):62.5%L15培养基,10%热灭活FBS,1%青霉素/链霉素,1%胰岛素,1%L-谷氨酰胺)并保持在4°C直至使用当天。在实验当天,使用前平衡至 RT。
- 对于 400 mL 培养基,使用 250 mL L15 培养基、40 mL 热灭活 FBS、4 mL 青霉素/链霉素、4 mL 胰岛素溶液(来自步骤 1.7)、4 mL L-谷氨酰胺和 98 mL 去离子无菌水。混合所有组分后,在无菌条件下准备无菌过滤器。准备 15 mL 等分试样。
- 在 APBS 溶液中制备 2.5% 和 3% (w/v) 低熔点 (lmp) -琼脂糖,并在 70 °C 下加热以完全溶解琼脂糖。在 1.5 mL 试管中制备等分试样,并在 4 °C 下储存直至使用当天。
- 对于 20 mL 的 2.5% (w/v) lmp-琼脂糖,在 50 mL 试管中混合 0.5 g lmp-琼脂糖与 20 mL APBS,并在 70 °C 的水浴中加热,直至琼脂糖完全熔化。解决方案必须变得清晰透明。在仍然温暖的情况下准备等分试样。
- 对于 20 mL 的 3% (w/v) lmp-琼脂糖,将 0.6 g lmp-琼脂糖与 20 mL APBS 混合,然后按上述方法进行。
- (可选):对于 20 mL 的 1% (w/v) lmp-琼脂糖,将 0.2 g lmp-琼脂糖与 20 mL APBS 混合,然后按上述方法进行。
注:熔解时间取决于 lmp-琼脂糖的浓度,范围约为 15-40 分钟。
- 对于截肢和组织安装,请使用明场立体镜。
- 组装直径为 100 mm 的塑料培养皿、镊子、手术刀和手术剪刀,用于截肢和组织采集。
- 准备直径为 35 毫米的塑料培养皿用于压痕测量。
- 准备 ~1 厘米长、直径为 ~1 厘米的圆柱体。为此,在本生灯火焰下加热纸板切割机,然后用加热的刀片切割 15 mL 的试管。
- 切下 1 厘米2 平方的小封口膜并储存。
- 通过在冰箱中放置至少 1 小时,在 -20 °C 下制备金属块或冷块。
- 准备塑料巴斯德移液器。
- (可选):制备固定剂 MEMFa 溶液(3-(N-吗啉代)丙磺酸 [MOPS] 0.1 M pH 7.4,乙二醇-双(2-氨基乙醚)-N,N,N′,N′-四乙酸 [EGTA] 2 mM,MgSO4·7H2O 1 mM,3.7% 甲醛)。为此,加入 10.465 g MOPS、0.123 g MgSO4·7H2O、2 mL 0.5 M EDTA pH 8.0,然后加水至 45 mL。添加 5 mL 甲醛 37% (w/v) 至总体积为 50 mL。
2. 试剂
- 有关用于这项工作的试剂,请参阅 材料表 ,但也可以使用其他商业提供商。
3. 蝾螈截肢和肢体再生
- 在所有实验程序之前,在储水箱水中稀释的 0.01% (w/v) 苯佐卡因中麻醉动物(步骤 1.3)20 分钟,确保动物被深度麻醉并且对触觉刺激没有反应。
- 从麻醉中取出蝾螈,将其放在 100 毫米培养皿的顶部,培养皿中装有用含麻醉水润湿的纸组织。将肢体垂直于身体轴线,并将板放在立体镜下方以获得更好的可视化效果。
注意:这里使用了带有紧凑型鹅颈灯的立体镜。 - 用锋利的无菌手术刀截肢,紧邻 zeugopodial 区域(桡骨/尺骨)的钙化区域远端(图 1A)。
注意:遵循 3R 动物福利原则,建议使用最初因截肢而切除的肢体作为完整的对照。 - 将动物留在培养皿上,用浸有苯佐卡因水的加湿纸巾覆盖 15 分钟,以允许血液凝固和伤口闭合。
- 将动物放回装有新鲜饮用水和镇痛药的水箱中(酒石酸布托啡诺,0.5 mg/L,从步骤 1.5 开始)。
- 术后镇痛治疗后 24 小时,将动物移至含有新鲜保水的原始水箱中。
- 让动物再生直到所需的感兴趣阶段。
注:在 5 个月大动物的截肢日收集完整期和截肢后 5 天 (dpa) 的组织溶解期分析的肢体。在 21 dpa 时测量 7 个月龄动物的软骨凝结阶段。
4. 用于测量的组织安装和加工
- 在热模块上将含有 2.5% 或 3% (w/v) lmp-琼脂糖(来自步骤 1.9)的 1.5 mL 试管在 70 °C 下加热,直到琼脂糖完全熔化。在 37 °C 下将试管更换为不同的加热块,并在使用前让温度平衡。
注:在这里,2.5% lmp 琼脂糖用于测量再生组织,3% 用于完整组织。 - 用一个封口膜片(步骤 1.14)覆盖 1 厘米长的圆柱体(来自步骤 1.13)的一侧,使其底部完全密封。
- 将 15 mL 等分试样的培养基平衡至 RT(从步骤 1.8 开始)
- 通过将动物浸入含麻醉剂的水(0.01% (w/v) 苯佐卡因在储罐水中稀释)至少 20 分钟来麻醉动物。
- 使用允许定量测量的软件在立体镜下对肢体进行成像。测量感兴趣结构的长度,并计算在到达感兴趣部分之前需要从肢体远端去除多少组织。
- 对于再生肢体,用手术刀和/或手术剪刀在肘部水平切割并从肢体上解剖出多余的组织来收集它们。将组织留在 APBS 溶液中,同时准备下一步。确保切口与下臂轴线横向 (90°) 以形成均匀的表面。对于完整的肢体,通过切开腕部区域来移开手。
- 通过将动物暴露于致死剂量的麻醉剂(0.1% 苯佐卡因)中至少 20 分钟来对动物实施安乐死。为此,添加所需体积的 10% (w/v) 苯佐卡因溶液以达到 0.1% (w/v) 浓度。
- 如果动物在 100 mL 的 0.01% (w/v) 苯佐卡因溶液中麻醉,则添加 900 μL 的 10% (w/v) 苯佐卡因溶液。
- 将肢体浸入 APBS 溶液中冲洗肢体。
- 确保巴斯德移液器和加热块(用琼脂糖等分试样稳定在 37 °C 下)靠近工作站。从 -20 °C 冰箱中取出冷块,将圆柱体放在上面,封口膜覆盖的一端朝下。
- 抓住解剖的肢体,用薄纸轻轻去除多余的液体。将肢体放在干净的板上,将熔化的 lmp-琼脂糖添加到顶部,然后在琼脂糖中短暂移动肢体,以从皮肤表面置换任何剩余的 APBS。
- 快速工作,将肢体放在圆柱体内,确保其垂直定向,感兴趣区域朝上。
- 用镊子轻轻握住肢体的同时,在圆筒内加入 lmp-琼脂糖,直到组织完全覆盖。在琼脂糖凝固之前轻轻取下镊子。
- 从冷块中取出圆筒,让琼脂糖在 RT 下完全固化约 30 秒。
- 在 RT 下,立即将含有组织的琼脂糖块与无菌培养基等分试样和 APBS 一起带到振动切片机室。
- 从圆柱体底部取下封口膜,并用氰基丙烯酸酯胶将含有组织的琼脂糖连接到振动切片机载物台上。确保琼脂糖和样品都粘在载物台上。
- 将载物台浸入 APBS 中进行切片。
注意:完整的组织包括较硬的骨骼/软骨,而再生组织较软。因此,振动切片机参数必须相应地调整。本研究使用以下参数:完整组织(振幅 1.2 mm,速度 0.1 mm/s)和再生组织(振幅 0.9 mm,速度 0.4 mm/s)。 - 开始以小步骤(例如,以 100 μm 步骤)切片琼脂糖,直到到达组织尖端。然后,将组织块切片,直到去除组织的远端部分(在步骤 4.5 中计算)。这样,将很容易获得感兴趣区域的横向横截面。
注意:去除的组织切片将包含与用 AFM 探测的表面紧邻的表面,并且可以作为组织结构的参考。因此,它可用于直接互补分析或固定用于后续染色(参见第 6 节)。 - 用剃须刀片小心地从振动切片机载物台上去除含有组织的块,并去除所有胶水痕迹。立即用手术组织胶水将块粘在 35 mm 塑料培养皿上,并在 RT 下加入约 2 mL 培养基,以确保组织完全覆盖。
注:琼脂糖块中暴露的组织表面是要探测的表面。
5. 使用 AFM 进行测量
- 至少在测量前 1 天,准备用于测量的悬臂。
注意:在这项工作中,用聚苯乙烯珠(直径 20 μm)对无尖端硅悬臂进行功能化,并且在用于压痕测量之前,将珠子-悬臂粘合至少加强 1 天。- 使用环氧树脂胶将珠子连接到悬臂上,加工时间为 5 分钟。
- 将悬臂以最少的接触快速降低到涂有胶水的载玻片上,以便在其末端形成一小滴胶水,然后立即使其与合适的胶珠接触。
- 保持悬臂珠触点 ~10 分钟,然后将带有束缚珠的悬臂从表面提起。
- 测量前校准悬臂。
注意:在每组实验之前,使用 AFM 软件的内置程序通过热噪声方法校准修改后的悬臂。 - 将带有组织块(来自步骤 4.18)和培养基的培养皿插入 AFM 的培养皿支架中,并在明场显微镜下获得概览图像(图 1B)。
注意:对于压痕测试,使用在正置光学显微镜顶部配备电动载物台的装置。安装 AFM 头并将悬臂定位在组织上,并将悬臂靠近表面。 - 选择感兴趣的特定区域并记录力 - 距离曲线数组(图 2A)。
注:使用相对力设定点 2-25 nN,以达到不同刚度的组织 ~1-4 μm 的可比压痕深度,接近和回缩速度为 7.5 μm/s,z 长度为 50 μm,网格尺寸为 70 μm x 70 μm,具有 3 x 3 点。 - 对于每个探测区域,获取明场显微镜图像,以将获得的值与肢体切片中的每个特定区域相关联。
- 对于所有样品,每种组织类型至少探测 3 个不同的区域。
注意:在所有情况下,本研究测量了软骨中心的 4 个区域和软骨区域外围的 3 个区域,并将组织在显微镜下最多保存 1 小时。 - 压痕测量完成后,丢弃或固定组织以进行进一步分析。
注:测量结束后,将样品置于含有 MEMFa 溶液的 2 mL 试管中,并在 4 °C 下固定过夜,从而固定样品。使用 MEMFa(步骤 1.17),但也可以使用 PBS 中的 4% (w/v) 甲醛溶液。
6. (可选)处理相邻组织切片
- 如果相邻的组织切片用于后续染色,请在切片后立即将它们放入含有固定剂的 2 mL 小管中,并在 4 °C 下固定过夜。
- 为了通过肌动蛋白细胞骨架和细胞核的标记来揭示组织结构,用 PBS 洗涤 3 次,每次 5 分钟以去除固定剂,并用含有 Alexa Fluor 488 偶联鬼笔环肽 (1:250) 和 Hoechst 33258 (1:10000) 的溶液染色在 PBS 中,在室温下在摇动平台上放置 1 小时。
- 接下来,使用1%(w / v)lmp-琼脂糖在37°C稳定的APBS中将染色样品固定在玻璃底培养皿的顶部(来自步骤1.9)。确保感兴趣的表面朝向玻璃。琼脂糖凝固后,用 PBS 覆盖样品以防止脱水。
注意:完整和组织裂解样品用倒置共聚焦显微镜(10 倍放大倍率和 8 μm 光学切片)成像。 图 1C 中的图像是 8 个光学切片的最大投影。
- 如果相邻组织切片用于需要新鲜组织的直接下游分析(如 RNA 提取、与活染料孵育等),请确保快速工作并使用细胞培养基(从步骤 1.8 开始)以确保尽可能高的组织完整性。
7. 数据分析与展示
- 为了计算表观杨氏模量,假设泊松比为 0.5,使用 JPK/Bruker 数据处理软件对球形压头的 Hertz/Sneddon 模型(方程 1)分析力-距离曲线。
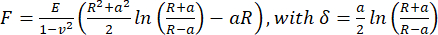 (方程 1)
(方程 1)
其中 R:压头半径,E:弹性模量, ν:泊松比,a:压头和样品之间圆形接触区域的半径,δ:压痕深度。 - 对于粘弹性分析,在 PyJibe 0.15.0 中分析力-压痕曲线的接近部分,扩展名是"使用 Kelvin-Voigt-Maxwell (KVM) 校正粘弹性的赫兹模型"(由 Paul Müller 撰写,https://github.com/AFM-analysis/PyJibe)25。拟合函数基于 Abuhattum 等人描述的模型26,该模型集成了 Kelvin-Voigt-Maxwell 模型单元。
- 对力压痕曲线进行预处理,以使用带有线(基线)的分段拟合和接近零件的多项式拟合函数来估计接触点。
- 将力-压痕曲线拟合到"使用 Kelvin-Voigt-Maxwell (KVM) 模型校正粘弹性的赫兹模型"。从拟合中,获得未松弛的杨氏模量、表观杨氏模量和表观粘度。该模型还给出了 Maxwell 松弛和压痕时间。
注意:在几条分析的力压痕曲线(特别是完整组织)中,麦克斯韦弛豫时间明显大于或小于压痕时间,这分别表明相当弹性的行为或 Kelvin-Voigt 机械行为。
- 将所有测量值导出到电子表格中,并计算每种组织类型和样品的中位表观杨氏模量。
- 使用适当的软件绘制和统计分析数据。
注意:此处使用了 GraphPad Prism,在描述结果时,该研究是指每个样品的中位表观杨氏模量的平均 ± SD(如图 2D-F 所示)。 - 通过使用斐济最大投影功能投影共聚焦图像来显示光学切片。调整每个单独通道的亮度和对比度,以实现荧光染色的最佳可视化。
- 使用适当的软件生成图窗面板。
注意:Affinity Designer 用于生成面板, 图 1A 中的模型是使用 Affinity Designer 绘制的。
结果
使用上述方案,我们测量了软骨蝾螈肢体组织在稳态("完整")条件下、早期软骨组织溶解和晚期软骨凝结阶段的表观杨氏模量(图 1A)。我们还探测了不同区域骨骼元件的机械性能,包括它们的中心和外围,如描绘悬臂位置的图像所示(图 1B)。为了显示组织结构并将其与刚度测量相关联,将横向组织切片或进行测量的组织?...
讨论
在这里,我们展示了一种用 AFM 测量蝾螈肢体软骨刚度的技术。然而,这种方法也可以扩展到探测其他组织类型。成功进行 AFM 测量的关键步骤是样品制备,事实证明,这对于蝾螈样品来说尤其具有挑战性。我们发现,探测仍嵌入琼脂糖块中的组织表面是保持组织完整性的最佳方法。这是因为蝾螈皮肤在表皮表面分泌高水平的粘液,从而阻止切片组织切片稳定地嵌入琼脂?...
披露声明
作者声明没有利益冲突
致谢
我们感谢 Sandoval-Guzmán 实验室的所有成员在这项工作开发过程中的持续支持和陪伴。我们还感谢 Anja Wagner、Beate Grühl 和 Judith Konantz 对蝾螈护理的奉献。我们还感谢 Paul Müller 为 AFM 数据分析提供代码。这项工作得到了德累斯顿工业大学 CMCB 技术平台的光学显微镜设施的支持。AT 是德国癌症援助组织 (Deutsche Krebshilfe) 资助的德累斯顿 P2 早期职业中心 Mildred Scheel 的研究员。RA 由 Deutsche Forschungsgemeinschaft(DFG,德国研究基金会)的临时 PI 职位 (Eigene Stelle) – AI 214/1-1 资助。
材料
| Name | Company | Catalog Number | Comments |
| Affinity Designer | Affinity | version 1.10.4 | For figure assembling |
| Agarose Low Melt | Roth | 6351.1 | For sample preparation |
| Alexa Fluor 488 Phalloidin | Invitrogen | A12379 | To stain tissue |
| Axiozoom | Zeiss | To image samplea under the AFM | |
| Benzocaine | Sigma-Aldrich | E1501 | To anesthetize the animals |
| Butorphanol (+)-tartrate salt | Sigma-Aldrich | B9156 | As analgesic |
| Cantilever | NanoWorld | Arrow TL1 | For AFM indentation measurements |
| Cellhesion 200 setup equipped with a motorstage | JPK/Bruker | For AFM indentation measurements | |
| CellSense Entry | For imaging in Stereoscope Olympus UC90 | ||
| Dulbecco’s Phosphate Buffered Saline (DPBS, 1x) | Gibco | 14190-144 | To clean samples and section under vibratome |
| FIJI (ImageJ2) | https://imagej.net/software/fiji | version 2.9.0/1.53t | For image processing |
| GraphPad Prism | GraphPad Software | (version 8.4.3) | To graph and statistically analyze the data |
| Heat-inactivated FBS | Gibco | 10270-106 | For cell culture medium |
| Histoacryl glue (2-Butyl-Cyanoacrylate) | Braun | To glue sample to petri dishes | |
| Hoechst 33258 | Abcam | ab228550 | To stain tissue |
| Insulin | Sigma-Aldrich | I5500 | For cell culture medium |
| Inverted confocal microscope | Zeiss | 780 LSM | To image tissue sections |
| Inverted confocal microscope | Zeiss | 980 LSM | To image tissue sections |
| JPK/Bruker data processing software | JPK/Bruker | SPM 6.4 | To analyze force-distance curves |
| L15 medium (Leibovitz) | Sigma | L1518 | For cell culture medium |
| L-Glutamine | Gibco | 25030-024 | For cell culture medium |
| Penicillin/Streptomycin | Gibco | 15140-122 | For cell culture medium |
| polystyrene beads ( 20 µm diameter); ) | microParticles | For AFM indentation measurements | |
| Pyjibe | written by Paul Müller https://github.com/AFM-analysis/PyJibe | 0.15.0 | For viscoelastic analysis |
| Stereoscope Olympus SX10 | Olympus | SX10 | For limb amputations and tissue mounting |
| Stereoscope Olympus UC90 | Olympus | UC90 | For imaging |
| Vibratome Leica | Leica | VT 1200S | For tissue sectioning |
参考文献
- Mills, L. A., Aitken, S. A., Simpson, A. H. R. W. The risk of non-union per fracture: current myths and revised figures from a population of over 4 million adults. Acta Orthop. 88 (4), 434-439 (2017).
- Calori, G. M., et al. Non-unions. Clin Cases Miner Bone Metab. 14 (2), 186-188 (2017).
- Nandra, R., Grover, L., Porter, K. Fracture non-union epidemiology and treatment. Trauma. 18 (1), 3-11 (2016).
- Nauth, A., Schemitsch, E., Norris, B., Nollin, Z., Watson, J. T. Critical-size bone defects: Is there a consensus for diagnosis and treatment. J Orthop Trauma. 32 (3 Suppl), S7-S11 (2018).
- Schemitsch, E. H. Size matters: Defining critical in bone defect size. J Orthop Trauma. 31 (10), S20-S22 (2017).
- Joven, A., Elewa, A., Simon, A. Model systems for regeneration: Salamanders. Development (Cambridge). 146 (14), 0 (2019).
- Aires, R., Keeley, S. D., Sandoval-Guzmán, T. Basics of Self-Regeneration. Cell Engineering and Regeneration. , (2020).
- Stocum, D. L. Mechanisms of urodele limb regeneration. Regeneration. 4 (4), 159-200 (2017).
- Bassat, E., Tanaka, E. M. The cellular and signaling dynamics of salamander limb regeneration. Curr Opin Cell Biol. 73, 117-123 (2021).
- Vinarsky, V., Atkinson, D. L., Stevenson, T. J., Keating, M. T., Odelberg, S. J. Normal newt limb regeneration requires matrix metalloproteinase function. Dev Biol. 279 (1), 86-98 (2005).
- Godwin, J., Kuraitis, D., Rosenthal, N. Extracellular matrix considerations for scar-free repair and regeneration: Insights from regenerative diversity among vertebrates. International J Biochem Cell Biol. 56, 47-55 (2014).
- Riquelme-Guzmán, C., et al. Osteoclast-mediated resorption primes the skeleton for successful integration during axolotl limb regeneration. ELife. 11, e79966 (2022).
- Gerber, T., et al. Single-cell analysis uncovers convergence of cell identities during axolotl limb regeneration. Science. 362 (6413), aaq0681 (2018).
- Kragl, M., et al. Cells keep a memory of their tissue origin during axolotl limb regeneration. Nature. 460 (7251), 60-65 (2009).
- Currie, J. D., et al. Live Imaging of Axolotl Digit Regeneration Reveals Spatiotemporal Choreography of Diverse Connective Tissue Progenitor Pools. Dev Cell. 39 (4), 411-423 (2016).
- Qin, T., et al. Single-cell RNA-seq reveals novel mitochondria-related musculoskeletal cell populations during adult axolotl limb regeneration process. Cell Death Differ. 28 (3), 1110-1125 (2021).
- Sandoval-Guzman, T., et al. Fundamental differences in dedifferentiation and stem cell recruitment during skeletal muscle regeneration in two salamander species. Cell Stem Cell. 14, 174-187 (2014).
- Currie, J. D., et al. The Prrx1limb enhancer marks an adult subpopulation of injury-responsive dermal fibroblasts. Biol Open. 8, bio043711 (2019).
- Wagh, K., Ishikawa, M., Garcia, D. A., Stavreva, D. A., Upadhyaya, A., Hager, G. L. Mechanical regulation of transcription: Recent advances. Trends Cell Biol. 31 (6), 457-472 (2021).
- d'Angelo, M., et al. The role of stiffness in cell reprogramming: A potential role for biomaterials in inducing tissue regeneration. Cells. 8 (9), 1036 (2019).
- Akhtar, R., Sherratt, M. J., Cruickshank, J. K., Derby, B. Characterizing the elastic properties of tissues. Mater Today (Kidlington). 14 (3), 96-105 (2011).
- Krieg, M., et al. Atomic force microscopy-based mechanobiology. Nat Rev Phys. 1 (1), 41-57 (2019).
- El Kirat, K., Burton, I., Dupres, V., Dufrene, Y. F. Sample preparation procedures for biological atomic force microscopy. J Microsc. 218 (3), 199-207 (2005).
- Riquelme-Guzmán, C., et al. Postembryonic development and aging of the appendicular skeleton in Ambystoma mexicanum. Dev Dyn. 251 (6), 1015-1034 (2021).
- López-Alonso, J., Eroles, M., Janel, S., et al. PyFMLab: Open-source software for atomic force microscopy microrheology data analysis [version 2; peer review: 2 approved]. Open Res Europe 2024. 3 (187), (2024).
- Abuhattum, S., Mokbel, D., Müller, P., Soteriou, D., Guck, J., Aland, S. An explicit model to extract viscoelastic properties of cells from AFM force-indentation curves. iScience. 25 (4), 104016 (2022).
- Glorieux, L., et al. In-depth analysis of the pancreatic extracellular matrix during development for next-generation tissue engineering. Int J Mol Sci. 24 (12), 10268 (2023).
- Hiratsuka, S., et al. Power-law stress and creep relaxations of single cells measured by colloidal probe atomic force microscopy. Jpn J Appl Phys. 48 (8 PART 3), 08JB17 (2009).
- Johnson, K. L., Kendall, K., Roberts, A. D. Surface energy and the contact of elastic solids. Proc R Soc Lond A. 324, 301-313 (1971).
- Kondiboyina, V., Duerr, T. J., Monaghan, J. R., Shefelbine, S. J. Material properties in regenerating axolotl limbs using inverse finite element analysis. J Mech Behav Biomed Mater. 150, 106341 (2024).
- Calve, S., Simon, H. Biochemical and mechanical environment cooperatively regulate skeletal muscle regeneration. FASEB J. 26 (6), 2538-2545 (2012).
- Calve, S., Simon, H. G. Extracellular control of limb regeneration. IUTAM Symposium on Cellular, Molecular and Tissue Mechanics. IUTAM Bookseries. 16, 257-266 (2010).
- Kaufmann, R. A., Kozin, S. H., Barnes, A., Kalluri, P. Changes in strain distribution along the radius and ulna with loading and interosseous membrane section. J Hand Surg Am. 27 (1), 93-97 (2002).
- Martin, A. R., et al. Measurement of distal forearm bone mineral density: Can different forearm segments be used interchangeably. J Clin Densitom. 2 (4), 381-387 (1999).
- Riquelme-Guzmán, C., et al. In vivo assessment of mechanical properties during axolotl development and regeneration using confocal Brillouin microscopy. Open Biol. 12 (6), 220078 (2022).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。