Method Article
通过 Yeast Surface Display 进行蛋白质工程
摘要
该方案描述了进行酵母表面展示选择活动以富集与目标抗原结合的蛋白质变体的基本步骤。
摘要
蛋白质工程能够改善给定蛋白质的现有功能或产生新功能。蛋白质工程领域使用最广泛和用途最广泛的工具之一是酵母表面显示,其中随机化蛋白质库在酵母表面表达。表型(例如,酵母展示的蛋白质与目标抗原的结合)和基因型(编码蛋白质变体的质粒)的联系使得选择该文库以获得所需的特性并随后对富集的变体进行测序。通过将磁珠选择与流式细胞术分选相结合,可以选择和富集与靶抗原结合增强的蛋白质变体。值得注意的是,除了亲和力成熟之外,还可以在没有任何初始结合亲和力的情况下实现与靶标的结合。在这里,我们提供了一个分步协议,该协议涵盖了酵母表面展示活动的所有基本部分,并给出了典型酵母表面展示结果的示例。我们证明酵母表面展示是一种广泛适用且可靠的方法,可以在任何可以使用流式细胞术的分子生物学实验室中建立。
引言
酵母表面显示是蛋白质工程领域的关键技术之一。它能够选择具有所需特性(例如提高亲和力或稳定性)的蛋白质变体。它于 1997 年首次推出1,是除噬菌体显示 2,3、核糖体显示4 和哺乳动物细胞显示 5,6,7 之外最常用的显示技术之一。目标蛋白 (POI) 通过融合锚定蛋白质来显示在酵母细胞表面。有一系列不同的锚蛋白可供选择,最常见的是,POI 与酵母凝集素交配蛋白 Aga2p 的 C 端融合 1,8。此外,POI 的两侧通常有两个标签,例如血凝素标签 (HA-tag) 和 c-myc 标签,它们可以通过使用荧光标记的抗体和流式细胞术来检测显示水平(图 1A)。典型的酵母选择活动包括磁珠选择和流式细胞术分选的组合。磁珠选择能够处理大量细胞并富集与靶抗原结合的蛋白质变体,并且亲和力也很低,因为与抗原负载珠子的多价相互作用会导致亲和力效应,因此可以防止低亲和力变体的丢失(图 1B)。流式细胞术分析和选择的优势在于可视化所显示的 POI 变体与标记抗原的结合。因此,可以对结合群体进行分选和培养,从而在几个分选轮次中富集具有所需特征的蛋白质变体。此外,可以进行额外轮次的随机诱变以进一步增加多样性,从而有可能找到有助于蛋白质亲和力和/或稳定性的其他突变。
酵母表面显示具有某些优势,例如 (a) 真核表达机制,能够进行氧化蛋白质折叠以及真核翻译后修饰(如 N-糖基化),(b) 由于检测到蛋白质两侧的两个肽标签而使表达正常化,(c) 通过流式细胞术目视检查选择进度(例如,结合细胞的百分比和结合强度)和 (d) 分析单个蛋白质突变体的可能性酵母(例如,分析热稳定性和亲和力),为费力的蛋白质表达和纯化提供了一种节省时间的替代方案9。事实上,酵母表面显示的蛋白质的亲和力(KD 值)和稳定性(T50 值)都与使用生物物理方法获得的数据和可溶性蛋白显示出良好的相关性 9,10,11,12。酵母表面显示已用于各种蛋白质的工程设计,例如抗体片段13、14、15、16、第 10个 III 型纤连蛋白结构域17、18、rcSso7d19、20 或结节蛋白21.同样,已经进行了广泛的研究,通过改变随机位置和氨基酸密码子的使用来优化酵母文库设计 17,22,23。酵母表面显示已被证明在稳定性 14,15,24,25、亲和力 18、26、27、酶活性28、29,30,31 和蛋白质表达 32 的工程设计方面取得了成功.此外,更复杂的应用,如存在或不存在小分子的条件结合,是使用酵母表面显示20 完成的。
在该方案中,我们描述了具有酵母表面显示的选择活动的所有基本步骤,并以 G4 文库(基于第 10个 III 型纤连蛋白结构域,Fn3)为例,针对抗原人视黄醇结合蛋白 4 (hRBP4) 在小分子 A112020 存在下进行选择。进行这种选择是为了产生蛋白质-蛋白质相互作用,该相互作用依赖于可用作分子开关的小分子。值得注意的是,虽然可以使用酵母表面显示的替代方法,但典型的酵母选择通常旨在与靶抗原结合,而之前没有任何结合亲和力。我们涵盖了酵母选择活动的所有步骤,包括酵母文库的培养、磁珠选择、流式细胞术分选以及通过易错 PCR (epPCR) 进行亲和成熟。因此,该方案补充了以前的酵母表面展示方案33,34,并可用作任何给定酵母文库和所选靶抗原的酵母表面展示选择的基础(图 1)。
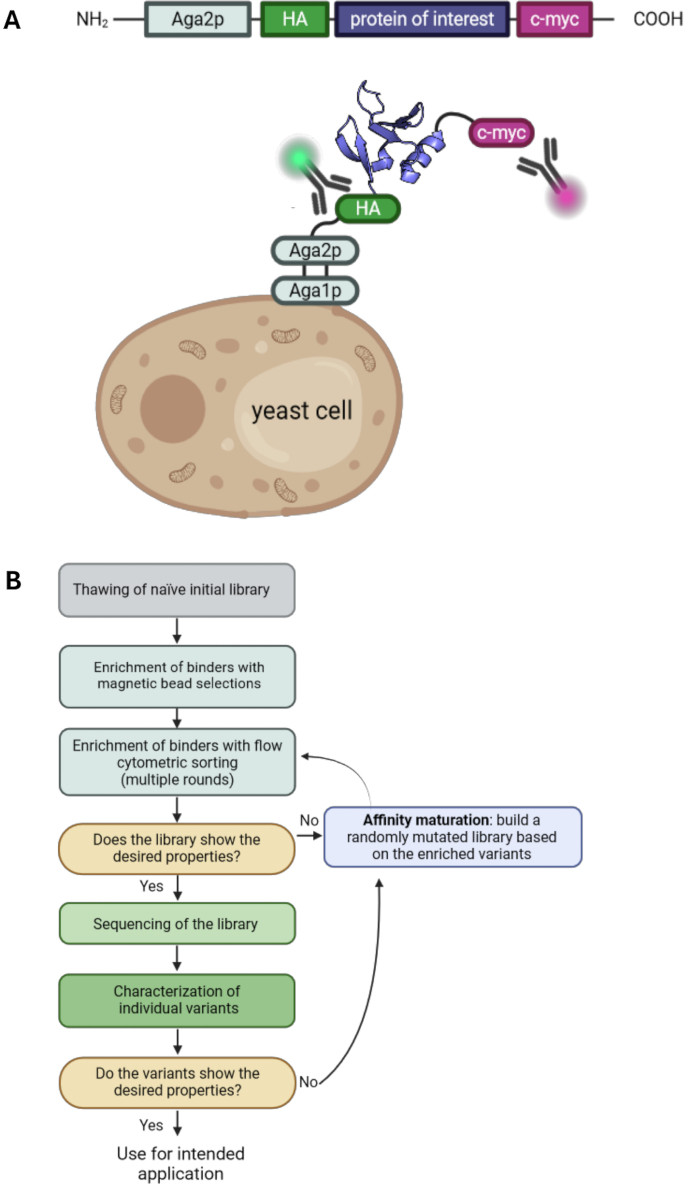
图 1:酵母表面显示的原理和酵母表面显示选择的典型工作流程。 (A) POI 被克隆到酵母表面展示载体中,两侧通常有一个 N 端 HA 和一个 C 端 c-myc 标签。该构建体与酵母交配蛋白 Aga2p 融合,以便在表面显示。所描述的蛋白质是来自 PDB ID:6QBA20 的工程结合物"RS3"。(B) 说明酵母表面显示选择活动的典型工作流程的流程图,该活动通过磁珠选择和流式细胞术分选以及 epPCR 进行亲和成熟,将蛋白质变体的富集与所需特性相结合。 请单击此处查看此图的较大版本。
研究方案
1. 酵母文库的解冻和培养
- 在 RT 下解冻足够等分的冷冻酵母文库,并立即在 SD-CAA 中稀释(见 表 1)至 OD600 约为 1(相当于 1 x 107 个细胞/mL)。
注意:确保将库的多样性覆盖至少 10 倍。因此,如果文库大小占 2.5 x 108 个克隆(这是本例中使用的 G4 文库的多样性),则在 250 mL SD-CAA 中接种至少 2.5 x 109 个细胞,以达到 OD600 为 1。该方案也可以从液体培养物(用文库新鲜转化的酵母细胞)开始。 - 评估活细胞的数量。
- 从该酵母悬浮液中取出 100 μL 等分试样(OD600 为 1),并在微量离心管中用 900 μL SD-CAA 培养基进行 1:10 稀释系列。
- 准备高达 10-5 的稀释液,并在 SD-CAA 板上接种 100 μL 稀释液 10-3、10-4 和 10-5 (表 1)。将板在 30 °C 下孵育 2-3 天。确保解冻后活细胞的数量至少超过文库多样性的 10 倍。
- 同时,继续使用剩余的细胞悬液,并在玻璃瓶中将含有 G4 文库的酵母培养物在 30 °C 下摇动 (180 rpm) 过夜。过夜孵育后,继续第 2 部分(酵母表面蛋白质表达的诱导)。
- 孵育 2-3 天后,通过目视检查对 SD-CAA 平板上的菌落进行计数。在计算解冻后活细胞的最终数量时,请考虑稀释因子和铺板(即 100 μL)与原始培养体积(即 250 mL)的比率。
活细胞/mL = 平板上的菌落数 × 稀释因子
活细胞(总)=(活细胞/mL)×培养体积
| 培养基/缓冲液 | 元件 | 浓度 [g/L] | 评论/描述 | |||
| SD-CAA 型 | D-葡萄糖 | 20 | 将所有培养基成分溶解在 1000 mL ddH2O 和带有一次性 0.22 μm 无菌过滤器的无菌滤液中。 | |||
| 酵母氮碱 | 6.7 | |||||
| 卡斯米诺酸 | 5 | |||||
| Citric acid monohydrate (柠檬酸一水合物) | 7.4 | |||||
| 柠檬酸三钠二水合物 | 10.83 | |||||
| SG-CAA 公司 | D-半乳糖 | 20 | 将所有培养基成分溶解在 1000 mL ddH2O 和带有一次性 0.22 μm 无菌过滤器的无菌滤液中。 | |||
| D-葡萄糖 | 2 | |||||
| 酵母氮碱 | 6.7 | |||||
| 酪蛋白氨基酸 | 5 | |||||
| 磷酸氢二钠七水合物 | 10.2 | |||||
| 磷酸二氢钠一水合物 | 8.56 | |||||
| SD-CAA 板 | 山梨醇 | 182 | 将山梨醇、磷酸氢二钠七水合物、磷酸二氢钠一水合物和琼脂溶于 900 mL ddH2O 中并高压灭菌。将剩余组分溶解并无菌过滤在 100 mL ddH2O 中,并在高压灭菌培养基温热时添加。 | |||
| 磷酸氢二钠七水合物 | 10.2 | |||||
| 磷酸二氢钠一水合物 | 7.44 | |||||
| 琼脂 | 15 | |||||
| D-葡萄糖 | 20 | |||||
| 酵母氮碱 | 6.7 | |||||
| 酪蛋白氨基酸 | 5 | |||||
| YPD | 蛋白胨 | 20 | 用一次性 0.22 μm 无菌过滤器制备 10x D-葡萄糖原液 (200 g/L) 和无菌滤液。将蛋白胨和酵母提取物溶解在 900 mL ddH2O 中并高压灭菌。当温热时,加入 100 mL 10x D-葡萄糖。 | |||
| 酵母提取物 | 10 | |||||
| D-葡萄糖 | 20 | |||||
| YPD 板 | 蛋白胨 | 20 | 用一次性 0.22 μm 无菌过滤器制备 10x D-葡萄糖原液 (200 g/L) 和无菌滤液。将蛋白胨、酵母提取物和琼脂溶解在 900 mL ddH2O 中并高压灭菌。当温热时,加入 100 mL 10x D-葡萄糖。 | |||
| 酵母提取物 | 10 | |||||
| D-葡萄糖 | 20 | |||||
| 琼脂 | 15 | |||||
| PBSA | BSA | 1 | 将 BSA 溶解在 PBS 中,并使用一次性 0.22 μm 无菌过滤器进行无菌滤液。 | |||
表 1:培养基和缓冲液组成。
2. 在酵母表面诱导蛋白质表达
- 过夜孵育后,酵母培养物通常达到 2-20 之间的 OD600 。用光度计测量 OD600 以确定酵母培养物的密度。
注意:OD 测量值仅在 0.1-1 范围内呈线性。因此,在 SD-CAA 中进行稀释(通常为 1:20 和 1:50)以确定 OD600。- 在 SD-CAA 中将细胞稀释至 OD600 为 1,并确保覆盖多样性 10 倍(即,使用 2.5 x 109 个细胞稀释 G4 文库)。在 30 °C 下继续孵育,同时振荡以使细胞达到指数生长期。
- 从过夜培养物中制备酵母文库(在含有 15% 甘油的 SD-CAA 中)的甘油原液(覆盖多样性 50x)。为此,离心所需数量的酵母细胞(2000 x g ,3 分钟,4 °C),弃去上清液,并将细胞沉淀重悬于 1 mL 补充有 15% 甘油的 SD-CAA 培养基中。立即将甘油原液在 -80 °C 下冷冻。
注:如果酵母培养物在冷冻前生长至固定相,酵母细胞的活力会增加。在以后的选择轮次中,当文库的多样性较低时,通常使用 700 μL 酵母培养物并将其与 300 μL 补充有 50% 甘油的 SD-CAA 混合就足够了。在这里,酵母培养物在固定相中通常显示 OD600 值为 15,但实际值可能取决于所使用的光度计。因此,可以通过重复的 OD600 测量来确定固定相。如果 OD600 值在 2 小时的孵育期内没有增加,则达到稳定相。
- 稀释酵母培养物后 4-6 小时(典型的 OD600 值在 4-6 之间),以 2000 x g 离心适当体积的细胞以覆盖多样性 10 倍 5 分钟,并弃去上清液。
- 将细胞沉淀重悬于 SG-CAA 中(见 表 1)以产生 OD600 为 1,并将其转移到新的玻璃瓶中。
注:在含半乳糖的培养基中重悬会通过半乳糖诱导启动子在表面诱导蛋白质的表达。 - 将酵母培养物在 20 °C 下孵育过夜,同时以 180 rpm 振荡以诱导 POI 的表面表达。过夜诱导后,酵母培养物通常会达到 2-5 之间的 OD600 值;如果诱导温度升高到 37 °C,则预期 OD600 值为 4-10。
注:将温度降低到 20°C 会减慢细胞生长,并且通常会产生更好的显示水平。然而,在 37 °C 下诱导可能有助于增加对稳定突变体的选择压力 35。
3. 酵母文库的第一轮微珠选择(阳性选择)
注:标准微珠选择程序包括 6 个步骤(表 2)。
| 日 | 步 | |
| 0 | 过夜培养 | |
| 1 | 在酵母细胞表面诱导蛋白质表达 | |
| 2 | 第一个微珠选择和 1 个阳性选择 | |
| 3 | 去除磁珠、传代、在酵母细胞表面诱导蛋白质表达和冷冻文库 | |
| 4 | 第二次微珠选择,包括 3 个阴性和 1 个阳性选择 | |
| 5 | 去除珠子并冻结文库 | |
表 2:酵母文库磁珠选择的典型时间表。
- 珠子的制备
- 为第一次珠子选择准备珠子(参见 材料表):对于每个试管,将 10 μL 生物素结合剂磁珠(4 ×10 5 个珠子/μL)重悬于 990 μL PBSA(表 1)中进行洗涤,将试管放在磁架上 2 分钟,盖子打开。小心去除上清液。用 1 mL PBSA 重复洗涤步骤。
注:如果需要多管具有相同生物素化抗原的微珠,则可以在此步骤中将它们制备到一个试管中,以产生均匀的抗原微珠溶液。在这种情况下,下一步需要相应地增加抗原的量。 - 在 1.5 mL 微量离心管中,将珠子重悬于总体积为 1 mL PBSA 和 6.7-33 pmole 生物素化抗原(此处:6.86 μL hRBP4,浓度为 0.125 mg/mL)中。
注意:虽然 6.7 pmole 的抗原就足够了,但如果抗原没有限制性,则应使用 33 pmole。如果对与小分子药物或配体结合的抗原进行选择,在这种情况下,该药物/配体在选择过程中必须存在,并且应在此步骤中添加(此处:5 μM A1120)。 - 在 4 °C 下孵育 2 小时,轻轻摇动或旋转。
注意:可以在此步骤中制备细胞。 - 孵育后,加入细胞之前,将试管放在磁力架上 2 分钟,并打开盖子。如上所述,去除上清液并用 1 mL PBSA 洗涤载有抗原的珠子。重复洗涤步骤。
注:如果选择了与药物或配体结合的抗原,则洗涤缓冲液中应含有过量的该药物或配体(此处:5 μM A1120)。 - 将加载抗原的磁珠重悬于 50 μL PBSA 中(在药物或配体存在下,如果适用)。
注:如果为多个试管制备了相同的抗原负载磁珠,请相应地调整体积(对于 G4 文库,此处的最终体积为 100 μL,可产生两个试管的磁珠)。
- 为第一次珠子选择准备珠子(参见 材料表):对于每个试管,将 10 μL 生物素结合剂磁珠(4 ×10 5 个珠子/μL)重悬于 990 μL PBSA(表 1)中进行洗涤,将试管放在磁架上 2 分钟,盖子打开。小心去除上清液。用 1 mL PBSA 重复洗涤步骤。
- 酵母细胞的制备
- 如上所述,测量诱导酵母培养物的 OD600 。
- 沉淀足够的细胞以覆盖文库多样性的 10 倍(2000 x g ,10 分钟,4 °C)。对于 G4 文库,使用 2.5 x 109 个细胞进行离心。去除上清液并用 10 mL PBSA 洗涤细胞。
- 再次离心(2000 x g,10 分钟,4 °C),并在适当体积的 PBSA 中将细胞重悬至 1.25 x 109 个细胞/mL 的浓度(此处:2 mL),然后转移到微量离心管中。
注意:PBSA 的体积取决于细胞的数量。 - 离心(2000 x g,3 分钟,4 °C),并重悬于 950 μL PBSA 中。
- 酵母细胞与载有抗原的磁珠一起孵育
- 将 50 μL 载抗原珠子添加到 950 μL 细胞悬液中,并在 4 °C 下孵育 2 小时,同时摇动或旋转。
- 孵育后,将细胞抗原珠溶液放在磁力架上,盖上盖子。将液体从盖子移液到试管中,以对整个酵母培养物进行磁性选择。等待 2 分钟,然后小心丢弃未结合的细胞。立即用 1 mL PBSA 洗涤。
注意:如果在存在药物/配体的情况下进行选择 - 在本例中为 A1120 - 这需要存在于 PBSA 中。 - 用 PBSA 洗涤步骤后,将细胞快速重悬于 1 mL SD-CAA 中,以防止它们变干。
- 加入 200 mL SD-CAA 并如上所述进行连续稀释。在 SD-CAA 板上铺板 100 μL,并在 30 °C 下孵育 2-3 天,以确定新的最大多样性。在这里,第一次磁珠选择后 G4 文库的多样性为 1.25 x106 个细胞。
注:由于原始文库在此选择轮中被过采样了 10 倍,因此可以合理地假设大多数克隆在选择后将在富集池中多次出现。因此,实际多样性会更低,这个数字应该被视为最大值。通常,在第一轮选择中,多样性至少减少了 100 倍。将剩余的培养物 (200 mL) 在 30 °C 下振荡 (180 rpm) 孵育至少 16 小时。通过将细胞置于 4 °C 孵育过夜后,可以暂停该方案。
4. 在下一轮磁珠选择之前去除磁珠并进行培养
- 如上所述测量 OD600 。沉淀足够的细胞以覆盖多样性至少 10 倍(2000 x g,3 分钟,RT)。弃去上清液,将细胞重悬于 1 mL SD-CAA 中。
- 转移到微量离心管中,打开盖子放在磁架上 2 分钟。将未结合的细胞转移到新的微量离心管中,并重复分离过程。
- 在 100 mL SD-CAA 中稀释未结合的细胞,并在 30 °C 下以 180 rpm 振荡孵育,直到达到 OD600 的 3-4。
注:如果在去除磁珠之前 OD600 低于 4,则可以在去除磁珠之前直接诱导细胞,无需孵育。 - 当达到 OD600 为 3-4 时,离心足够数量的细胞以覆盖多样性至少 10 倍(2000 x g,3 分钟,RT),并在 SG-CAA 中重悬至 OD600 为 1 以诱导蛋白质表达,并在 20 °C 下孵育,同时以 180 rpm 振荡过夜。
- 当剩余的 SD-CAA 酵母培养物达到静止生长期(OD600 高于 15)时,沉淀足够的细胞以覆盖多样性 50x(2000 x g,3 分钟,RT)。通过离心(2000 x g ,3 分钟)冷冻细胞,然后将细胞沉淀重悬于含有 15% 甘油的 SD-CAA 中并储存在 -80 °C。
5. 第二轮磁珠选择,包括 3 个阴性和 1 个阳性选择
- 如上所述准备细胞和抗原珠。为阴性选择准备裸珠溶液(不添加抗原)。
注意:由于进行了 3 次阴性选择,因此可以一起为所有三轮制备磁珠。 - 洗涤后,将抗原珠重悬于 50 μL PBSA 中,并将裸珠重悬于 150 μL PBSA 中(如果它们已准备好所有三种阴性选择)。
- 对于第一次阴性选择,将 50 μL 洗涤的裸珠添加到 PBSA 中的 950 μL 洗涤细胞中,并在 4 °C 下孵育 1.5 小时。
- 孵育后,将含有裸珠细胞悬液的试管放在磁力架上,盖上盖子。将盖子中的任何液体移液到试管中并等待 2 分钟。将未结合的细胞转移到新鲜的微量离心管中,并加入 50 μL 洗涤的裸珠。
- 对另外两轮负选区重复该过程。同时,为阳性选择准备抗原负载的珠子。
- 3 轮阴性选择后,向细胞中加入 50 μL 载有抗原的珠子溶液,并在 4 °C 下孵育 2 小时。
- 将含有抗原负载珠子的细胞放在磁力架上,打开盖子,然后将盖子中的任何液体移液到试管中。等待 2 分钟,然后丢弃未结合的细胞。
- 按照第一次抗原珠选择所述执行所有剩余步骤。与第一次阳性选择(步骤 3.3.2)的唯一区别是洗涤步骤的数量:而不是用 PBSA 洗涤一次,而是用 PBSA 洗涤珠细胞悬液两次,以增加选择的严格性。
注意:通常,多样性会再次减少 10 倍到 100 倍。第二次磁珠选择后 G4 文库的最大多样性为 3.45 x 105 个细胞。如上所述,由于库的过度采样,实际分集预计会更低。
6. 通过流式细胞术分选选择文库
- 在 SG-CAA 中诱导表面表达过夜后,如上所述测量 OD600 。沉淀足够的细胞以覆盖 10 倍的多样性(2000 x g,5 分钟,RT)。丢弃上清液。
- 将沉淀重悬于 PBSA 中,并将其转移到微量离心管中。每个试管使用 3 x 107 个细胞进行染色。根据需要准备任意数量的试管,具体取决于多样性。准备一根对照管,用于无抗原染色。
注:例如,如果应分选 30 x 106 个细胞,则沉淀 300 x 106 个细胞以获得可见的细胞沉淀并防止后续步骤中的细胞损失。重悬于 5 mL PBSA 中,并将 3 至 4 份 500 μL 的等分试样转移至微量离心管中。准备的细胞大约是应分选数量的 3 倍,因为在染色过程中会丢失一小部分细胞。如果在存在药物/配体的情况下进行筛选,则仅制备含有抗原但不含药物/配体的额外对照。 - 再次离心(2000 x g,5 分钟,RT),将沉淀重悬于 200 μL 含有抗原的 PBSA(hRBP4 与 5 μM A1120)中,并在 4 °C 下孵育 1 小时。
注意:从此时开始,所有步骤都应在冰上和冷试剂下进行。为防止抗体或抗原耗竭,与试管中酵母展示的蛋白质总数相比,所有试剂的添加量应过量。否则,由于抗原和/或抗体的可用性有限,可能存在限制酵母展示变体结合的风险。通常,每个酵母细胞使用 5 x 104 个分子进行计算,并建议添加至少 10 倍的化学测量过量的抗原和抗体。如果抗原非常有限,则过量可以减少到 5 倍。更详细的解释,包括避免配体耗竭的计算示例,在之前的方案9 中提供。 - 孵育后,离心细胞(2000 x g,5 分钟,4 °C)并用 1 mL PBSA 洗涤(如果在选择过程中存在药物/配体 - 在本例中为 A1120 - 这需要存在于洗涤缓冲液中)。
- 将细胞重悬于 100 μL 含有用于显示染色和检测结合抗原的抗体的冷 PBSA 中,并在 4 °C 下孵育 30 分钟。
- 使用抗 c-myc 抗体(最终稀释度 1:100)检测全长显示的 POI。使用抗 HA 抗体(最终稀释度 1:50)检测显示的蛋白质总量。使用 Penta-His 抗体(最终稀释度 1:20)或链霉亲和素(最终稀释度 1:200)选择与分别含有 6x HIS 标签或生物素的可溶性抗原结合的酵母细胞。
- 在分选轮次之间交替使用检测试剂,以防止与二级试剂结合的蛋白质变体富集。
注:可以使用不同的荧光基团组合,但 Alexa Fluor 488 和 Alexa Fluor 647 是不错的选择,因为它们在流式细胞术中不需要补偿。为防止抗体耗竭,与试管中酵母展示的蛋白质总数相比,应以化学计量过量的方式添加抗体。通常,计算基于每个酵母细胞 5 x 104 个分子。前面的协议9 中提供了更详细的解释。
- 孵育后,离心细胞(2000 x g,5 分钟,4 °C)并向沉淀中加入 1 mL PBSA(含有药物/配体,对于 hRBP4 选择的 G4 文库,5 μM 的 A1120)并再次离心。去除大部分上清液,仅保留 20-30 μL,以防止沉淀变干。
- 仅在分选前将沉淀重悬于冷 PBSA 中,并分选显示具有所需特性的 Fn3 分子的酵母细胞(即,在 A1120 存在下与 hRBP4 结合)。
注意:门控策略如图 2A 所示。 - 将细胞直接分选到 SD-CAA 培养基中,分选后加入更多的 SD-CAA 培养基,并在 30 °C 下以 180 rpm 振荡孵育。
注:如果分选后体积显著增加,应进行离心步骤(2000 x g,5 分钟,RT)以除去多余的鞘液,并将细胞重悬于新鲜的 SD-CAA 培养基中。记下分选到新试管中的细胞数,因为这成为下一轮分选的新最大多样性。如上所述,由于库的过度采样,库的实际多样性会更低。 - 重复流式细胞术分选几轮,以查看酵母文库中结合蛋白变体的富集(图 2B)。
注:在分析流式细胞术实验中并排比较文库可能很有用。在以后的选择轮次中,降低抗原的浓度可能有助于区分低亲和力和高亲和力结合蛋白,并专门对在低浓度下对靶标显示较高结合信号的蛋白质变体进行分选(图 2C)。在以后的筛选轮次中,还可以进行分选以提高热稳定性。为此,如第 7 节所述,通过 epPCR 对文库进行随机诱变。通过诱导、OD600 的测定和细胞洗涤来制备细胞,类似于其他流式细胞术分选轮次。将酵母细胞置于冰上的 PBSA 中 10 分钟,然后在热循环仪中孵育 10 分钟,以便在高温下进行热休克。然后,将它们再次置于冰上 10 分钟,然后继续进行染色方案。稳定的变体将抵抗热变性,因此在热孵育后显示出维持的结合。根据野生型蛋白的初始热稳定性,可以测试不同的温度进行热休克测试。通常,40-80 °C 的温度范围是合适的。然而,高于 48 °C,酵母细胞的活力将急剧下降。因此,如步骤 7.1 所述,有必要在对热休克酵母文库进行分类后提取质粒。在这种情况下,在进行下一轮分选36 之前,需要用富集的变体新鲜转化 EBY100 细胞。提高热稳定性的更详细方案可在其他地方找到37。如果显示的蛋白质的变性是可逆的,则此方法将不起作用。或者,在分选前一天在 37 °C 而不是 20 °C 下诱导蛋白质表达,并分选仍然显示高效全长显示的蛋白质变体。
7. 使用 epPCR 进行亲和成熟以引入随机突变
注:使用 epPCR 的亲和成熟可以在第一轮流式细胞术分选之前或在流式细胞术分选轮之间进行。为了在 A1120 存在下选择含有 hRBP4 的 G4 文库,在第一轮流式细胞术分选之前进行亲和成熟。这也取决于微珠选择后的文库大小以及流式细胞术可检测到的结合信号。特别是,在流式细胞术实验中,微珠选择后的亲和力不足以获得信号的情况下(因为抗原在洗涤步骤中会快速解离),epPCR 可以产生改进的变异,随后可以通过流式细胞术进行检测和选择。
- 使用市售质粒小量制备 II 试剂盒(参见 材料表)从富集的酵母文库中进行酵母小量制备 DNA,并进行以下修改:
- 测量过夜培养物的细胞密度,早上稀释至 OD600 为 0.2,并在 30 °C 下振荡孵育。
- 当 OD600 达到 0.6 时,通过以 600 x g 离心细胞 2 分钟来收获 3 mL 培养物。
- 向沉淀中加入 200 μL 溶液 1。加入 6 μL 酶解酶。通过轻弹或轻度涡旋重悬沉淀,并在 37 °C 下孵育 3 小时。
- 加入 200 μL 溶液 2 并轻轻混合。加入 400 μL 溶液 3 并轻轻混匀。
- 以最大速度离心 3 分钟。
- 将上清液转移到离心 I 柱中,以最大速度离心 30 秒,然后弃去流出液。
- 用 550 μL 含乙醇的洗涤缓冲液洗涤,并以最大速度离心 2 分钟。
- 将色谱柱转移到干净的微量离心管中,并以最大速度离心 2 分钟以去除残留的洗涤缓冲液。
- 将色谱柱转移到新的微量离心管中,加入 10 μL 无核酸酶的水,并以最大速度旋转 1 分钟以洗脱 DNA。
- 进行初始 PCR 以扩增用于 epPCR 的模板。
- 使用酵母表面展示载体上插入片段外侧退火的引物,通过 PCR 扩增 DNA。
注:由于在酵母小量制备过程中会与目标质粒一起洗脱大量基因组酵母 DNA,因此建议此步骤以获得干净的 DNA 片段,用作 epPCR 的模板。 - 对于 pCTCON2,使用引物 fwd 5'- GGCTCTGGTGGAGGCGGTAGCGGAGGCGGAG
GGTCGGCTAGC 和修订版 5'- CTATTACAAGTCC
TCTTCAGAAATAAGCTTTTGTTCGGATCC 具有 表 3 中描述的反应组分和 表 4 中描述的循环条件。 - 用 PCR 和 DNA 纯化试剂盒纯化 PCR。在 20 μL 无核酸酶的 H2O 中洗脱。
- 使用酵母表面展示载体上插入片段外侧退火的引物,通过 PCR 扩增 DNA。
- 进行 epPCR 以引入随机突变。
- 使用具有指定成分(表 5)和条件(表 6)的核苷酸类似物进行 epPCR。
- 在 120 V 的制备型 1% 琼脂糖凝胶上运行整个 epPCR 产物 45 分钟。包括一个标记物,并在 epPCR 产物和分子量标准之间留出一个泳道。切除与预期插入片段大小相对应的所需条带,使用凝胶提取试剂盒纯化,并测量 DNA 浓度。
- 通过 PCR 扩增随机化的 DNA。
- 用 Q5 聚合酶扩增 50 ng DNA。在指定条件下(表 8),为每个电穿孔(表 7)设置 2 x 100 μL PCR 反应。使用与第一次 PCR 相同的引物。
注:电穿孔到酵母中需要大量的 DNA。 - 在 1% 琼脂糖凝胶上分析 5 μL PCR 产物,在 120 V 下运行 45 分钟。
- 用 Q5 聚合酶扩增 50 ng DNA。在指定条件下(表 8),为每个电穿孔(表 7)设置 2 x 100 μL PCR 反应。使用与第一次 PCR 相同的引物。
- 用乙醇沉淀纯化扩增的 DNA。
- 将 2 x 100 μL 混合在一起。加入 20 μL(体积的 10%)无菌过滤的 3 M 乙酸钠,pH 值为 5.2。
注意:该缓冲液可以提前制备并在 RT 中储存几个月。 - 加入 440 μL 100% 乙醇(或至少 2 倍体积),并在室温下孵育 2 分钟。
- 在 RT 下以 20,000 x g 离心 5 分钟。去除上清液,加入 500 μL 70% 乙醇并短暂混合。
- 在 RT 下以 20,000 x g 离心 5 分钟。去除上清液,加入 500 μL 100% 乙醇并短暂混合。
- 在 RT 下以 20,000 x g 离心 5 分钟。去除上清液,让 DNA 沉淀在层流罩下干燥,直到所有乙醇蒸发。
注意:使用 48 °C 的加热块可加快此过程。DNA 沉淀可能并非始终可见。 - 将干燥的 DNA 沉淀溶解在 10 μL 无核酸酶的 H2O 中。
注:DNA 应以尽可能小的体积溶解。不要测量 DNA 浓度。
- 将 2 x 100 μL 混合在一起。加入 20 μL(体积的 10%)无菌过滤的 3 M 乙酸钠,pH 值为 5.2。
| 体积 [μL] | 最终浓度 | |
| 5x Q5 增强剂 | 10 | 1 倍 |
| 5x Q5 缓冲液 | 10 | 1 倍 |
| 引物 fwd 10 μM | 2.5 | 0.5 微米 |
| 引物转速 10 μM | 2.5 | 0.5 微米 |
| dNTP 10 毫米 | 1 | 200 微米 |
| Q5 聚合酶 | 0.5 | 20 U/mL |
| 来自酵母小量制备的 DNA | 10 | |
| 无核酸酶 H2O | 13.5 |
表 3:从分离的酵母小量制备中扩增 POI 基因的第 1 步 PCR 的条件。
| 步 | 温度 | 时间 |
| 初始变性 | 98 摄氏度 | 30 秒 |
| 25 次循环 | 98 摄氏度 | 10 秒 |
| 72 摄氏度 | 30 秒 | |
| 72 摄氏度 | 30 秒 | |
| 最终扩展 | 72 摄氏度 | 2 分钟 |
| 拿 | 4 °C |
表 4:从分离的酵母小量制备中扩增 POI 基因的第 1步 PCR 的循环条件。
| 体积 [μL] | 最终浓度 | |
| 无核酸酶 H2O | 最多 50 个 | |
| 10x Thermopol 缓冲液 | 5 | 1 倍 |
| Primer_fwd (10 μM) | 2.5 | 0.5 微米 |
| Primer_rev (10 μM) | 2.5 | 0.5 微米 |
| dNTP (10 毫米) | 1 | 200 微米 |
| 8-氧代-dGTP (100 μM) | 1 | 2 微 M |
| dPTP (100 μM) | 1 | 2 微 M |
| 来自第一次 PCR 的 PCR 产物 | 二十 | 50 纳克 |
| Taq DNA 聚合酶 | 0.5 | 0.05 U/微升 |
表 5:使用第 1 步 PCR 扩增 POI DNA 后进行的 epPCR 的条件。
| 步 | 温度 | 时间 |
| 初始变性 | 94 摄氏度 | 30 秒 |
| 15 次循环 | 94 摄氏度 | 45 秒 |
| 60 °摄氏度 | 30 秒 | |
| 72 摄氏度 | 1 分钟 | |
| 最终扩展 | 72 摄氏度 | 10 分钟 |
| 拿 | 4 °C |
表 6:epPCR 的循环条件。
| 体积 [μL] | 最终浓度 | |
| 5x Q5 增强剂 | 20 | 1 倍 |
| 5x Q5 缓冲液 | 20 | 1 倍 |
| 引物 fwd 10 μM | 5 | 0.5 微米 |
| 引物转速 10 μM | 5 | 0.5 微米 |
| dNTP 10 毫米 | 1 | 200 微米 |
| Q5 聚合酶 | 1 | 20 U/mL |
| 50 ng DNA | 二十 | |
| ddH20 | 最高 100 |
表 7:EBY100 细胞电穿孔前用于扩增 epPCR 产物的第2 步 PCR 的条件。
| 步 | 温度 | 时间 |
| 初始变性 | 98 摄氏度 | 30 秒 |
| 25 次循环 | 98 摄氏度 | 10 秒 |
| 72 摄氏度 | 30 秒 | |
| 72 摄氏度 | 30 秒 | |
| 最终扩展 | 72 摄氏度 | 2 分钟 |
| 拿 | 4 °C |
表 8:用于扩增 epPCR 产物的第 2步 PCR 的循环条件。
8. 用于电穿孔的酵母展示载体的线性化
- 线性化 pCTCON2 酵母表面展示载体 (Addgene 质粒 #41843)。
- 进行大规模酶解,并将该酶解线性化载体用于所有进一步的电穿孔。
- 在 37 °C 下用 Sal I 消化 2 x 200 μg pCTCON2 质粒 DNA 24 小时,以进行初始线性化(表 9)。
- 在 37 °C 下用 NheI 和 BamHI 消化 24 小时以切除插入片段(表 10)。
- 最后,向试管中加入 5 μL 每种酶(Sal I、NheI、BamHI),并在 37 °C 下再次孵育 24 小时。
- 在制备型琼脂糖凝胶上运行消化的载体。切除条带并用凝胶提取试剂盒纯化。在 20 μL 中洗脱载体,测量 DNA 浓度并将其调节至约 2 μg/μL。
注:长时间消化质粒可确保插入片段的充分切割,并导致电穿孔后载体的背景减少。
| 脱氧核糖核酸 | 200 微克 |
| 10 倍 CutSmartBuffer | 50 微升 |
| 盐 I-HF (NEB) | 30 μL (60 U) |
| 端头H 2O | 高达 500 μL |
表 9:酵母表面大规模消化的第一步条件显示载体 pCTCON2。
| pCTCON2(Sal I 消化) | 500 微升 |
| 10 倍 CutSmartBuffer | 37.5 微升 |
| NheI-HF (NEB) | 15 微升 (30 U) |
| BamHI-HF (NEB) | 15 微升 (30 U) |
| 端头H 2O | 高达 875 μL |
表 10:酵母表面大规模消化第二步的条件显示载体 pCTCON2。
9. 使用随机 DNA 和线性化载体对 EBY100 进行电穿孔
- 通过在电穿孔前 3 天从甘油原液中敲击来制备 EBY100 酵母细胞(ATCC,参见 材料表)。由于未转化的 EBY100 细胞不含酵母表面显示载体,因此使用全培养基 YPD 板(表 1)。在 30 °C 下孵育。
- 电穿孔前一天,将一个 EBY100 菌落接种到 30 mL 新鲜制备的 YPD 培养基中(表 1)。在 30 °C 下孵育过夜,同时以 180 rpm 振荡。
注意:YPD 培养基的保存时间不应超过 1 个月。 - 第二天早上,在 100 mL YPD 培养基中将 EBY100 过夜培养物稀释至 OD600 为 0.2,并在 30 °C 下孵育,同时以 180 rpm 振荡。
注:50 mL 的培养体积足以进行 2 次电穿孔。通常,制备 100 mL 以备有足够的细胞用于对照(仅细胞和细胞加载体)。 - 当 OD600 达到 1.3-1.5 时,将细胞沉淀在 50 mL 管中,以 2000 x g 的速度沉淀 3 分钟,然后去除上清液。
- 将沉淀重悬于 25 mL 的 100 mM 无菌过滤乙酸锂中(参见 材料表)。加入新鲜制备的无菌过滤 DTT (1 M)(参见 材料表)至终浓度为 10 mM。
注:醋酸锂体积应为 YPD 原始体积的一半。如有必要,可以缩小或扩大规模。乙酸锂 (100 mM) 可以提前制备并在室温下储存几个月,但 1 M DTT 原液应在使用前准备好并保存在冰上。 - 将细胞在 30 °C 下孵育,同时摇动 10 分钟。
- 以 2000 x g 离心 3 分钟,弃去上清液,并将细胞置于冰上。
注:以下所有步骤均应在冰上并使用冷却的试剂、比色皿和微量离心管进行。 - 将沉淀重悬于 25 mL 凉爽无菌 H2O 中,以 2000 x g 离心 3 分钟。
注意: 使用的 H2O 体积应为原始 YPD 体积的一半。 - 重悬于 350 μL 无菌 H2O 中。
注:重悬后的总体积应为 500 μL。如果需要,添加更多的 H2O。 - 平行制备 DNA 文库:将含有乙醇纯化插入片段的微量离心管置于冰上,并加入 4 μg 线性化 pCTCON2 载体。
- 向 DNA 中加入 250 μL 细胞,并剪碎轻轻混合。转移到预冷的 2 mm 电穿孔比色皿中,并保持在冰上直至电穿孔。
注:添加对照(仅含 DNA 的细胞和仅含线性化载体且不含插入片段的细胞)以确定电穿孔是否成功。 - 使用 2 mm 电穿孔比色皿(参见 材料表)在以下条件下进行电穿孔:方波协议、单脉冲、500 V、15 ms 脉冲持续时间。
注意:典型的下垂为 3%- 9%。可以使用其他电穿孔仪,但所示条件针对本研究中使用的仪器进行了优化。 - 用 1 mL 预热的 YPD 拯救细胞,转移到预热的微量离心管中,并在 30 °C 下不摇晃孵育 1 小时。
注:将加热块放在电穿孔仪旁边,以确保回收管保持在适当的温度。 - 在 SD-CAA 平板上接种 100 μL 10-3、10-4、10-5 和 10-6 稀释的细胞(见 表 1),并允许在 30 °C 下转化体生长。
注意:理论文库多样性是通过计算回原始培养体积的转化体数量来获得的,这里为 1 mL。通常,该转化方案产生大约 10个 7-10 8 个转化细胞。 - 将剩余的细胞以 2000 x g 沉淀 3 分钟,重悬于 200 mL SD-CAA 中,并在 30 °C 下以 180 rpm 振荡孵育过夜。含有 epPCR 随机文库的转化细胞可以在 SD-CAA 中传代,并于第二天在 SG-CAA 中诱导表面表达,或储存在 4 °C 直至进一步使用。确定多样性后,在 epPCR 后冷冻文库的甘油储备液。由于在第二天,一小部分细胞仍然是在转化过程中携带的未转化细胞,而没有获得质粒,因此建议在第一个传代步骤中传代至少 20 倍的文库多样性,以补偿这种影响。
10. 几轮选择后对酵母文库进行测序
- 如上所述,通过进行酵母小量制备来提取酵母 DNA。
- 用分离的 DNA 电穿孔商业大 肠杆菌 10-β 电感受态细胞(参见 材料表)。
- 将电穿孔比色皿(1 mm,参见 材料表)和微量离心管置于冰上。在 37 °C 下用 950 μL 生长培养基预热微量离心管,以回收电穿孔后的细胞。
- 在冰上解冻细胞,轻轻弹动试管混合。将 50 μL 细胞转移到冷冻的微量离心管中。加入 1 μL 酵母小量制备 DNA。
- 小心地将细胞 DNA 混合物转移到冷冻的电穿孔比色皿中。
注意:避免气泡并确保细胞沉积在比色皿底部。 - 使用以下条件使用电穿孔设备进行电穿孔:2.0 kV;200 Ω;25 μF 的
注意:典型的时间常数为 4.8-5.1 毫秒。可以使用其他电穿孔仪,但所示条件针对本研究中使用的仪器进行了优化。 - 立即向比色皿中加入 950 μL 37 °C 预热的生长培养基(参见 材料表),轻轻混合,然后转移回温热的微量离心管中。
注:将加热块放在电穿孔仪旁边,以确保回收管保持在适当的温度。 - 在 37 °C 和 300 rpm 下孵育 40 分钟。将 10 μL、100 μL 和其余细胞(离心后)接种到预热的选择性 LB 板上(见 表 1)。将板在 37 °C 下孵育过夜。
注:对于 pCTCON2 载体,氨苄青霉素用于抗生素选择。 - 用 LB 琼脂和所需的抗生素(这里使用氨苄青霉素)准备复制板:在 96 孔板的每个孔中吸取 200 μL 含有 100 μg/mL 氨苄青霉素的 LB 琼脂,并使其固化。
- 制备一个 96 孔测序板,每孔含 50 μL LB 和 100 μg/mL 氨苄青霉素。
- 从板中挑选一个菌落,首先接种复制板和测序板的同一孔。
注:复制板可确保通过接种液体培养物和进行小量制备来轻松获得所需的质粒。 - 将复制板在 37 °C 下孵育过夜,然后在 4 °C 下储存。
- 将测序板在 37 °C 下孵育 3-4 小时,同时摇动 (180 rpm),用塑料或铝箔覆盖。
- 密封测序板并将其与 500 μL 10 μM pCTCON2 测序引物 (5'-CGTTTGTCAGTAATTGCGGTTCTC) 一起送去测序。
- 为了分析序列,使用合适的程序将获得的 DNA 序列翻译成氨基酸序列,例如 EMBOSS Transeq38。接下来,使用多序列比对 (MUSCLE)39 或类似的比对工具将变异序列彼此比对,并与亲本野生型蛋白比对。
注意:通过对不同选择轮次的文库进行测序(例如,在较早的一轮选择之后,以及在以较低抗原浓度进行额外轮次选择以富集高亲和力变体之后),可以识别不同的变体。这在很大程度上取决于分选轮次期间应用的门的严格程度,因此,库的多样性。
结果
针对与小分子药物 A1120 结合的抗原 hRBP4 选择 G4 文库。按照方法 6 中的说明对用于流式细胞术分选的文库进行染色,应用的门控策略如图 2A 所示。初始设门包括基于细胞形态的所有细胞,第二个设门(FSC-Width 的直方图)显示了严格的设门策略,用于选择单个细胞并去除细胞聚集体。第三个也是最后一个门显示了蛋白质变体(x 轴)与抗原结合(y 轴)的显示。对显示信号和结合信号的酵母细胞进行分类。重要的是,分选门以严格的方式设置,以高结合信号富集结合结构域,从而获得高亲和力。这种严格的选择产生了在整个选择活动中特异性结合靶抗原的展示酵母细胞的富集(图 2B)。在后来的流式细胞术分选轮次中,抗原浓度降低了 10 倍(从 100 nM 降至 10 nM)。因此,整体结合信号降低,只有具有高亲和力的结合剂仍然可以检测和排序(图 2C)。
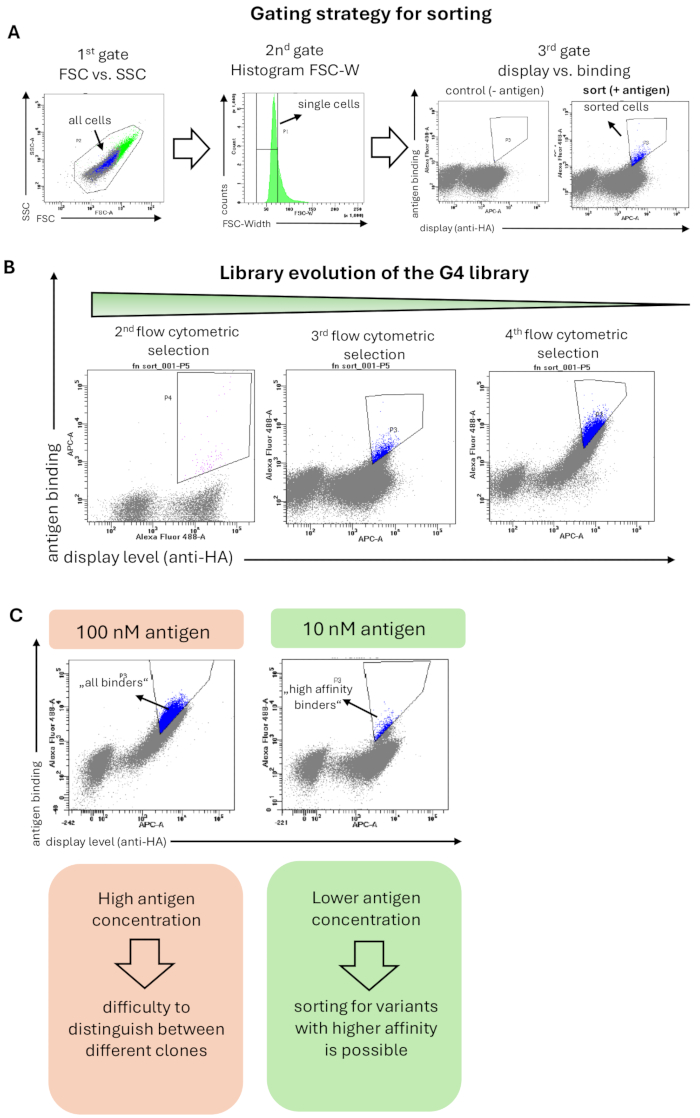
图 2:酵母表面显示基于 Fn3 的 G4 文库选择与抗原结合的代表性结果(在 A1120 存在下为 hRBP4)。 (A) 酵母文库分选的一般设门策略。第一个关卡(FSC 与 SSC)是选择所有酵母细胞并排除散射事件;第二个门 (FSC-W 的直方图) 旨在去除细胞聚集体并仅选择单个酵母细胞。第三个门绘制表面显示水平(检测 HA 或 c-myc 标签)与与抗原结合(此处 hRBP4 在 5 μM A1120 存在下,由抗 His 抗体检测)。文库还仅用二抗(无抗原)染色,预计不会发生抗原结合。排序的单元格以蓝色突出显示。(B) G4 文库在 3 轮流式细胞术分选中的演变。每轮选择都可以观察到结合群体的富集。(C) 使用较低的抗原浓度可以选择对靶抗原具有较高亲和力的蛋白质变体。当抗原浓度(此处为 hRBP4)降低 10 倍后,出现不同的对角线,表明存在具有较高(分选细胞,蓝色)或较低亲和力的克隆。 请单击此处查看此图的较大版本。
讨论
酵母表面显示已发展成为蛋白质工程中使用的关键方法之一。虽然它通常用于亲和力 1,18,40,41、表达/稳定性 24、27、42、43 和活性28,44 的工程设计,但进一步用于表位定位45,46 或表征酵母细胞表面的单个突变体9也是可能的。在该协议中,我们提供了开始酵母表面展示选择活动的基本步骤,包括用磁珠和流式细胞术分选以及通过 epPCR 实现酵母文库的多样化以进行亲和成熟。
传统酵母表面显示选择的一个基本要求是提供足够质量的可溶性蛋白质。从具有高纯度和明确寡聚状态(即单体蛋白应仅以单体形式存在)的充分折叠靶蛋白开始,为选择具有高亲和力与靶抗原结合的蛋白质变体提供了最高的成功率。难以表达的靶蛋白的另一种选择是基于细胞的选择,它提供了一种合理的策略来规避这一限制47。然而,酵母表面展示具有许多优势,例如可以直接在酵母表面表征所得蛋白质变体,而无需进行费力且耗时的克隆、以可溶性形式表达和蛋白质纯化。变体的亲和力和稳定性都可以直接在酵母表面进行分析9。
在该方案中,我们展示了如何在小分子 A1120 存在下选择蛋白质变体的 G4 文库,更具体地说是人纤连蛋白的第 10个 III 型结构域与抗原 hRBP4 结合。微珠选择和流式细胞术分选相结合,产生了变体的富集,这表明在整个选择轮次中与靶抗原的结合增加(图 2B)。我们表明,使用较低浓度的抗原可以选择高亲和力蛋白质变体(图 2C)。通常,通过酵母显示选择可以实现的亲和力在纳摩尔甚至皮摩尔范围内18。最终的亲和力取决于靶抗原、选择轮数和亲和力成熟度、使用的结合支架以及应用的门控策略。本协议未涵盖单个蛋白质变体的表征,但在我们之前的工作9 中进行了详细解释。尽管酵母展示最初用于抗体片段(如 scFvs 1,40)的工程设计,但该方法也已广泛用于非基于抗体的蛋白质10。
总而言之,酵母表面展示是一种强大的蛋白质工程工具,能够生成具有新颖或改进特性的蛋白质变体,例如与几乎任何靶蛋白结合和/或提高稳定性。
披露声明
M.W.T. 获得 Miltenyi Biotec 的资助。所有作者都是通过使用酵母表面显示开发的技术和工程蛋白的专利申请的发明人。
致谢
这项工作得到了奥地利科学基金(FWF 项目 W1224 - 蛋白质生物分子技术博士课程 - BioToP 和 FWF 项目 ESP 465-B)、奥地利联邦数字和经济事务部、奥地利国家研究、技术和发展基金会到基督教多普勒研究协会(下一代 CAR T 细胞的基督教多普勒实验室)的支持, 以及通过向 St. Anna Children's Cancer Research Institute(奥地利维也纳)的私人捐款。E.S. 是奥地利科学院圣安娜儿童癌症研究所 DOC 奖学金的获得者。人物是使用 BioRender.com 创建的。
材料
| Name | Company | Catalog Number | Comments |
| 10-beta electrocompetent E. coli | NEB | C3020K | |
| Agar-Agar, Kobe I | Carl Roth | 5210.5 | |
| Ampicillin sodium salt | Carl Roth | K029.2 | |
| Anti-c-myc antibody, clone 9E10, AF488 | Invitrogen | MA1-980-A488 (Thermo Fisher) | |
| Anti-c-myc antibody, clone 9E10, AF647 | Invitrogen | MA1-980-A647 (Thermo Fisher) | |
| Anti-HA antibody, clone 16B12, AF488 | BioLegend | 901509 (Biozym) | |
| Anti-HA antibody, clone 16B12, AF647 | BioLegend | 682404 (Biozym) | |
| BamHI-HF | NEB | R3136S | |
| Bovine serum albumin, cold ethanol fraction | Sigma-Aldrich | A4503 | |
| Citric acid monohydrate | Sigma-Aldrich | C1909 | |
| D-Galactose | Carl Roth | 4987.2 | |
| D-Glucose | Sigma-Aldrich | G8270 | |
| Difco yeast nitrogen base | Becton Dickinson (BD) | 291940 | |
| Di-Sodium hydrogen phosphate heptahydrate | Carl Roth | X987.3 | |
| DL-Dithiothreitol | Sigma-Aldrich | D0632 | |
| D-Sorbitol | Carl Roth | 6213.1 | |
| Dulbecco’s phosphate buffered saline (10x) | Thermo Scientific | 14190169 | |
| Dynabeads Biotin Binder | Invitrogen | 11047 (Fisher Scientific) | |
| DynaMag-2 Magnet | Thermo Fisher | 12321D | |
| EBY100 | ATCC | MYA-4941 | |
| Electroporation cuvette 1 mm (for E.coli) | VWR | 732-1135 | |
| Electroporation cuvette 2 mm (for yeast) | VWR | 732-1136 | |
| Ethanol absolute | MERCK | 1070172511 | |
| GeneMorph II Random Mutagenesis Kit | Agilent Technologies | 200550 | |
| Gibco Bacto Casamino Acids | Becton Dickinson (BD) | 223120 | |
| Glycerol | AppliChem | 131339.1211 | |
| LE agarose | Biozym | 840004 | |
| Lithium acetate dihydrate | Sigma-Aldrich | L4158 | |
| Monarch DNA Gel Extraction Kit | NEB | T1020S | |
| Monarch PCR & DNA Cleanup Kit | NEB | T1030S | |
| Multifuge 1S-R | Heraeus | ||
| NheI-HF | NEB | R3131S | |
| Outgrowth medium | NEB | B9035S | |
| pCTCON2 | Addgene | #41843 | |
| Penicillin G sodium salt | Sigma-Aldrich | P3032 | |
| Penta-His antibody, AF488 | Qiagen | 35310 | |
| Penta-His antibody, AF647 | Qiagen | 35370 | |
| Peptone ex casein tryptically digested | Carl Roth | 8986.3 | |
| Q5 High-Fidelity DNA Polymerase | NEB | M0491S | |
| SaII-HF | NEB | R3138S | |
| Sodium acetate | Sigma-Aldrich | S8750-1KG | |
| Sodium chloride | Carl Roth | 3957.2 | |
| Sodium dihydrogen phosphate monohydrate | Carl Roth | K300.2 | |
| Steritop threaded bottle top filter | MERCK | S2GPT01RE | |
| Streptavidin, AF488 | Invitrogen | S32354 (Thermo Fisher) | |
| Streptavidin, AF647 | Invitrogen | S32357 (Thermo Fisher) | |
| Streptomycin sulfate | Sigma-Aldrich | S6501 | |
| Tri-Sodium citrate dihydrate | Carl Roth | 4088.1 | |
| UV-Vis spectrophotometer | Agilent | 8453 | |
| Yeast extract, micro-granulated | Carl Roth | 2904.4 | |
| Zymoprep Yeast Plasmid Miniprep II | Zymo Research | D2004 |
参考文献
- Boder, E. T., Wittrup, K. D. Yeast surface display for screening combinatorial polypeptide libraries. Nat Biotechnol. 15 (6), 553-557 (1997).
- Scott, J. K., Smith, G. P. Searching for peptide ligands with an epitope library. Science. 249 (4967), 386-390 (1990).
- Smith, G. P. Filamentous fusion phage: novel expression vectors that display cloned antigens on the virion surface. Science. 228 (4705), 1315-1317 (1985).
- Schaffitzel, C., Hanes, J., Jermutus, L., Pluckthun, A. Ribosome display: an in vitro method for selection and evolution of antibodies from libraries. J Immunol Methods. 231 (1-2), 119-135 (1999).
- Ho, M., Nagata, S., Pastan, I. Isolation of anti-CD22 Fv with high affinity by Fv display on human cells. Proc Natl Acad Sci U S A. 103 (25), 9637-9642 (2006).
- Wagner, A., et al. Identification of activating mutations in the transmembrane and extracellular domains of EGFR. Biochemistry. 61 (19), 2049-2062 (2022).
- Wagner, A., et al. PhosphoFlowSeq - A high-throughput kinase activity assay for screening drug resistance mutations in EGFR. J Mol Biol. 433 (22), 167210(2021).
- Cherf, G. M., Cochran, J. R. Applications of yeast surface display for protein engineering. Methods Mol Biol. 1319, 155-175 (2015).
- Zajc, C. U., Teufl, M., Traxlmayr, M. W. Affinity and stability analysis of yeast displayed proteins. Methods Mol Biol. 2491, 155-173 (2022).
- Gai, S. A., Wittrup, K. D. Yeast surface display for protein engineering and characterization. Curr Opin Struct Biol. 17 (4), 467-473 (2007).
- Orr, B. A., Carr, L. M., Wittrup, K. D., Roy, E. J., Kranz, D. M. Rapid method for measuring ScFv thermal stability by yeast surface display. Biotechnol Prog. 19 (2), 631-638 (2003).
- Teufl, M., Zajc, C. U., Traxlmayr, M. W. Engineering strategies to overcome the stability-function trade-off in proteins. ACS Synth Biol. 11 (3), 1030-1039 (2022).
- Wozniak-Knopp, G., et al. Introducing antigen-binding sites in structural loops of immunoglobulin constant domains: Fc fragments with engineered HER2/neu-binding sites and antibody properties. Protein Eng Des Sel. 23 (4), 289-297 (2010).
- Traxlmayr, M. W., et al. Directed evolution of stabilized IgG1-Fc scaffolds by application of strong heat shock to libraries displayed on yeast. Biochim Biophys Acta. 1824 (4), 542-549 (2012).
- Traxlmayr, M. W., et al. Directed evolution of Her2/neu-binding IgG1-Fc for improved stability and resistance to aggregation by using yeast surface display. Protein Eng Des Sel. 26 (4), 255-265 (2013).
- Hasenhindl, C., et al. Stability assessment on a library scale: a rapid method for the evaluation of the commutability and insertion of residues in C-terminal loops of the CH3 domains of IgG1-Fc. Protein Eng Des Sel. 26 (10), 675-682 (2013).
- Koide, A., Wojcik, J., Gilbreth, R. N., Hoey, R. J., Koide, S. Teaching an old scaffold new tricks: monobodies constructed using alternative surfaces of the FN3 scaffold. J Mol Biol. 415 (2), 393-405 (2012).
- Hackel, B. J., Kapila, A., Wittrup, K. D. Picomolar affinity fibronectin domains engineered utilizing loop length diversity, recursive mutagenesis, and loop shuffling. J Mol Biol. 381 (5), 1238-1252 (2008).
- Traxlmayr, M. W., et al. Strong enrichment of aromatic residues in binding sites from a charge-neutralized hyperthermostable Sso7d scaffold library. J Biol Chem. 291 (43), 22496-22508 (2016).
- Zajc, C. U., et al. A conformation-specific ON-switch for controlling CAR T cells with an orally available drug. Proc Natl Acad Sci U S A. 117 (26), 14926-14935 (2020).
- Moore, S. J., Cochran, J. R. Engineering knottins as novel binding agents. Methods Enzymol. 503, 223-251 (2012).
- Hackel, B. J., Ackerman, M. E., Howland, S. W., Wittrup, K. D. Stability and CDR composition biases enrich binder functionality landscapes. J Mol Biol. 401 (1), 84-96 (2010).
- Hackel, B. J., Wittrup, K. D. The full amino acid repertoire is superior to serine/tyrosine for selection of high affinity immunoglobulin G binders from the fibronectin scaffold. Protein Eng Des Sel. 23 (4), 211-219 (2010).
- Laurent, E., et al. Directed evolution of stabilized monomeric CD19 for monovalent CAR interaction studies and monitoring of CAR-T cell patients. ACS Synth Biol. 10 (5), 1184-1198 (2021).
- Julian, M. C., et al. Co-evolution of affinity and stability of grafted amyloid-motif domain antibodies. Protein Eng Des Sel. 28 (10), 339-350 (2015).
- Cochran, J. R., Kim, Y. S., Lippow, S. M., Rao, B., Wittrup, K. D. Improved mutants from directed evolution are biased to orthologous substitutions. Protein Eng Des Sel. 19 (6), 245-253 (2006).
- Kieke, M. C., et al. High affinity T cell receptors from yeast display libraries block T cell activation by superantigens. J Mol Biol. 307 (5), 1305-1315 (2001).
- Lipovsek, D., et al. Selection of horseradish peroxidase variants with enhanced enantioselectivity by yeast surface display. Chem Biol. 14 (10), 1176-1185 (2007).
- Fushimi, T., et al. Mutant firefly luciferases with improved specific activity and dATP discrimination constructed by yeast cell surface engineering. Appl Microbiol Biotechnol. 97 (9), 4003-4011 (2013).
- Lam, S. S., et al. Directed evolution of APEX2 for electron microscopy and proximity labeling. Nat Methods. 12 (1), 51-54 (2015).
- Park, S. W., Lee, D. S., Kim, Y. S. Engineering a U-box of E3 ligase E4B through yeast surface display-based functional screening generates a variant with enhanced ubiquitin ligase activity. Biochem Biophys Res Commun. 612, 147-153 (2022).
- Schutz, M., et al. Directed evolution of G protein-coupled receptors in yeast for higher functional production in eukaryotic expression hosts. Sci Rep. 6, 21508(2016).
- Angelini, A., et al. Protein engineering and selection using yeast surface display. Methods Mol Biol. 1319, 3-36 (2015).
- Chen, T. F., de Picciotto, S., Hackel, B. J., Wittrup, K. D. Engineering fibronectin-based binding proteins by yeast surface display. Methods Enzymol. 523, 303-326 (2013).
- Shusta, E. V., Holler, P. D., Kieke, M. C., Kranz, D. M., Wittrup, K. D. Directed evolution of a stable scaffold for T-cell receptor engineering. Nat Biotechnol. 18 (7), 754-759 (2000).
- Traxlmayr, M. W., Obinger, C. Directed evolution of proteins for increased stability and expression using yeast display. Arch Biochem Biophys. 526 (2), 174-180 (2012).
- Traxlmayr, M. W., Shusta, E. V. Directed evolution of protein thermal stability using yeast surface display. Methods Mol Biol. 1575, 45-65 (2017).
- Madeira, F., et al. The EMBL-EBI Job Dispatcher sequence analysis tools framework in 2024. Nucleic Acids Res. 52 (W1), W521-W525 (2024).
- Edgar, R. C. MUSCLE: multiple sequence alignment with high accuracy and high throughput. Nucleic Acids Res. 32 (5), 1792-1797 (2004).
- Chao, G., et al. Isolating and engineering human antibodies using yeast surface display. Nat Protoc. 1 (2), 755-768 (2006).
- VanAntwerp, J. J., Wittrup, K. D. Fine affinity discrimination by yeast surface display and flow cytometry. Biotechnol Prog. 16 (1), 31-37 (2000).
- McConnell, A. D., et al. A general approach to antibody thermostabilization. MAbs. 6 (5), 1274-1282 (2014).
- Pavoor, T. V., Wheasler, J. A., Kamat, V., Shusta, E. V. An enhanced approach for engineering thermally stable proteins using yeast display. Protein Eng Des Sel. 25 (10), 625-630 (2012).
- Chen, I., Dorr, B. M., Liu, D. R. A general strategy for the evolution of bond-forming enzymes using yeast display. Proc Natl Acad Sci U S A. 108 (28), 11399-11404 (2011).
- Traxlmayr, M. W., et al. Construction of a stability landscape of the CH3 domain of human IgG1 by combining directed evolution with high throughput sequencing. J Mol Biol. 423 (3), 397-412 (2012).
- Chao, G., Cochran, J. R., Wittrup, K. D. Fine epitope mapping of anti-epidermal growth factor receptor antibodies through random mutagenesis and yeast surface display. J Mol Biol. 342 (2), 539-550 (2004).
- Stern, L. A., et al. Cellular-based selections aid yeast-display discovery of genuine cell-binding ligands: Targeting oncology vascular biomarker CD276. ACS Comb Sci. 21 (3), 207-222 (2019).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。