Method Article
CIRCLE-Seq 用于研究脱靶基因编辑
摘要
CRISPR 等技术的一个重大障碍是可能破坏重要基因的脱靶事件。"通过测序进行切割效应的 体外 报告循环化"(CIRCLE-seq) 是一种旨在识别意外切割位点的技术。该方法以高灵敏度且无偏差的方式绘制 CRISPR-Cas9 的全基因组活性。
摘要
通过 测序体外报告 切割效应的循环化 (CIRCLE-seq) 是一种新技术,用于通过对 CRISPR-Cas9 切割的 DNA 进行靶向测序来公正地鉴定 CRISPR-Cas9 的意外切割位点。该方案涉及基因组 DNA (gDNA) 的环化,随后用 Cas9 蛋白和目标向导 RNA (gRNA) 处理。处理后,纯化裂解的 DNA 并制备为 Illumina 测序的文库。测序过程生成双端读长,提供每个切割位点的全面数据。与其他 体外 方法相比,CIRCLE-seq 具有多项优势,包括测序深度要求最低、背景低和 Cas9 裂解 gDNA 的高富集度。这些优势增强了识别预期和非预期切割事件的灵敏度。本研究提供了一个全面的分步程序,用于使用 CIRCLE-seq 检查 CRISPR-Cas9 的脱靶活性。例如,通过在 AAVS1 基因座修饰期间绘制 CRISPR-Cas9 的全基因组意外切割位点来验证该协议。整个 CIRCLE-seq 过程可在两周内完成,为细胞生长、DNA 纯化、文库制备和 Illumina 测序留出足够的时间。将测序数据输入到 CIRCLE-seq 流程中有助于简化切割位点的解释和分析。
引言
基因组工程在过去 20 年中取得了重大进展,一个重要的里程碑是在 2012 年发现了成簇的规则间隔短回文重复序列 (CRISPR)-Cas91。利用细菌 DNA 核酸内切酶的可编程特性,CRISPR-Cas9 技术能够精确靶向和修饰几乎任何 DNA 序列。自成立以来,该系统已经过优化,仅依靠 Cas9 核酸内切酶和向导 RNA (gRNA) 来编辑特定的基因组区域。CRISPR-Cas9 作为治愈疗法的潜力已在针对各种疾病的临床试验中得到证明,例如 Leber 先天性黑朦、转甲状腺素蛋白淀粉样变性和镰状细胞性贫血等 2,3,4。
CRISPR-Cas9 诱导双链断裂 (DSB),这通常通过以下两种机制之一解决:如果模板 DNA 可用,则容易出错的非同源末端连接 (NHEJ) 或更精确的同源定向修复 (HDR)。CRISPR-Cas9 导致 NHEJ 相关插入和缺失 (插入缺失) 的趋势,以及意外基因组位点的切割,限制了其在临床环境中的应用 5,6,7,8,9,10。此外,意外的基因组修饰可以产生隐蔽的剪接位点、无义或错义突变、诱导染色体裂网或赋予细胞致癌潜力——这些结果已在多项基因组编辑试验中观察到11、12、13、14、15.总之,准确识别 CRISPR-Cas9 的脱靶活性对其临床应用至关重要,尤其是在可能改变数十亿个细胞的全身基因疗法中。
可以采用多种方法来识别 CRISPR-Cas9 脱靶切割位点,包括全基因组双链断裂无偏鉴定 (GUIDE)-seq16,它使用双链寡脱氧核苷酸标记活细胞中的 DSB。然而,对这种方法的批评是,假阳性可能来自随机 DSB 或 PCR 伪影,必须通过排除与目标位点相似性差的捕获位点来丢弃。基于整合酶缺陷慢病毒载体 (IDLV) 的方法灵敏度较低,并且可能会遗漏许多脱靶位点17。其他原位方法,如 DSBCapture、BLESS 和 BLISS 18,19,20 涉及固定细胞并直接标记 DSB,但它们受到对即时 DSB 捕获和外源 DNA 缺失的依赖性的限制。Digenome-seq21(一种体外方法)和通过测序选择性富集和鉴定标记基因组 DNA 末端 (SITE-seq)22 都提供了测序解决方案,但分别在背景噪声和单端分析方面存在局限性。通过测序发现原位 Cas 脱靶 (Discover-Seq)23 可通过 MRE11 结合对 Cas9 活性进行体内和原位鉴定,但仅检测样品制备时存在的 DSB24。最后,CRISPR 编辑推理 (ICE) 使用生物信息学方法,使用 Sanger 数据25 稳健地分析 CRISPR 编辑。
本文介绍了通过 测序进行体外 切割效应报告的循环化 (CIRCLE-seq) 的详细程序 :一种体外技术,可灵敏、公正地绘制具有目标 gRNA 的复合物中 Cas9 核酸酶的全基因组脱靶活性26。这种方法从培养目标细胞和分离 DNA 开始,然后通过聚焦超声进行随机剪切,然后进行核酸外切酶和连接酶处理。该过程最终产生环状双链 DNA 分子,然后通过质粒安全的 DNase 处理进行纯化。然后,该环状 DNA 暴露于 Cas9-gRNA 复合物中,Cas9-gRNA 复合物在有意和无意的切割位点进行切割,留下暴露的 DNA 末端,作为 Illumina 接头连接的底物。该过程产生一个多样化的基因组 DNA (gDNA) 文库,其中包含每个核酸酶诱导的 DSB 的两端,确保每个读数都具有每个切割位点所需的所有信息。这允许使用测序覆盖度要求较低的 Illumina 测序,使 CIRCLE-seq 与上述其他类似方法区分开来。需要注意的是,虽然 CIRCLE-seq 作为 体外 方法确实比其他方案具有更高的脱靶灵敏度,但由于不存在其他方法(如 GUIDE-seq16)中存在的表观遗传景观,因此代价是假阳性率更高。此外,DSB DNA 修复及其相关机制不存在于 CIRCLE-seq 中,消除了插入缺失或本来会被观察到的适当修复。
除了描述执行 CIRCLE-seq 的分步方案外,该方案还通过识别 AAVS1 基因座修饰期间发生的 CRISPR-Cas9 全基因组意外切割位点来验证,例如。该方案易于遵循,提供了从诱导多能干细胞 (iPSC) 培养和 gDNA 分离到 gDNA 环化、Cas9-gRNA 切割、文库制备、测序和管道分析的详细说明。鉴于测序覆盖度要求较低,CIRCLE-seq 可供任何能够进行新一代测序的实验室使用。
研究方案
材料 表中列出了用于本研究的试剂、耗材和设备的详细信息。
1. 细胞培养(5 天)
- 在整个方案中包括一个阴性对照。准备足够的细胞来额外制备 25 μg gDNA(每个样品 ~2.0e7 个细胞)。
- 根据既定方案培养 iPSC27.收集细胞并将它们重悬于 10 mL 的 PBS 中。吸取 6 μL 细胞样品,并以 1:1 的比例用台盼蓝重悬。使用自动细胞计数仪对样品进行计数。
- 每管分装 2 x 107 个细胞,然后在 25 °C(室温 (RT))下以 300 x g 离心 3 分钟。这对于多个重复来说已经足够了。移液并丢弃上清液。
2. 基因组 DNA 分离(1 天)
- 按照制造商的说明,使用市售的 DNA 纯化试剂盒分离 gDNA:
- 将 200 μL PBS 添加到含有细胞沉淀物的 15 mL 锥形管中并重悬。然后,将 3 mL 细胞裂解缓冲液和 15 μL 蛋白酶 K 移液到试管中。将试管倒置 25 次以充分混合。将试管置于设置为 55 °C 和 150 rpm 的水浴振荡器中 3 小时,或过夜以获得最佳 DNA 产量。
- 移液管加入 15 μL RNase A,倒置 25 次。置于 37 °C 的水浴中 1 小时。
- 将样品在冰上冷却 5 分钟。然后,加入 1 mL 蛋白质沉淀溶液,高速涡旋 20 秒,并在室温下以 2000 x g 离心 10 分钟。蛋白质应在试管底部形成可见的致密沉淀。如果看不到沉淀,请将样品在冰上再孵育 5 分钟,然后再次离心。
- 将 3 mL 100% 异丙醇添加到新的 15 mL 锥形管中。小心地将步骤 2.1.3 中的上清液移液到试管中。将试管倒置 50 次以混合,然后在 2000 x g 和室温下离心 3 分钟。在不干扰 DNA 沉淀的情况下,使用连接到真空阱的巴斯德移液器小心吸出上清液,并在干净、不起毛的湿巾上倒置试管。
- 将 3 mL 70% (v/v) 乙醇移液到 DNA 沉淀上,倒置 10 次洗涤。在 RT 下以 2,000 x g 离心 3 分钟。然后,小心地倒出上清液。
- 保持试管打开,让所得 DNA 沉淀干燥 30 分钟,确保所有乙醇已完全蒸发。移液加入 50 μL DNA 水合溶液,轻轻移液充分混合。
- 将样品置于 65 °C 的水浴振荡器中 1 小时,溶解 DNA,然后将样品在 RT 中放置过夜。将样品在 RT 和 2,000 x g 下离心 1 分钟,然后使用 dsDNA BR 检测试剂盒和相关试管用荧光计定量分离的 DNA。
3. gRNA 的制备(7 天)
- 从商业来源订购感兴趣的合成 gRNA(参见 材料表)。该方案也与 crRNA/tracrRNA 兼容。
4. gRNA 体外 切割试验
注:此处使用了 AAVS1 基因中的靶标。为了靶向其他感兴趣的基因,设计引物(表 1)以扩增靶标区域,并用定制引物替换以下步骤中的引物。
- 准备 PCR 反应:混合 25 μL Phusion 热启动 Flex 2x 预混液(终浓度 1x)、0.5 μL AAVS1 F 引物(终浓度 0.1 μM)、0.5 μL AAVS1 R 引物(终浓度 0.1 μM)、5 μL gDNA(100 ng、20 ng/μL,来自步骤 2.1.6)和 19 μL 无核酸酶 H2O(总体积: 50 μL)。
- 使用以下热循环仪参数:变性:98 °C 2 分钟(1 个循环),变性:98 °C 10 秒(10 个循环),退火:72-62 °C(-1 °C/循环)15 秒(10 个循环),延伸:72 °C 30 秒(10 个循环),变性,98 °C 10 秒(30 个循环)。退火:65 °C 持续 15 秒(30 个周期),延伸:72 °C 持续 30 秒(30 个周期),最终延伸:72 °C 持续 5 分钟(1 个周期),保持:4 °C 无限期。
- 使用 AMPure XP 微珠纯化 PCR 反应产物。首先,将 1.8 倍体积或 90 μL 的 XP 磁珠移液到 PCR 产物中。移液 10 次以充分混合。将混合物在 RT 下放置 5 分钟孵育。
- 使用磁架,将 PCR 反应板放在磁力架上 3 分钟,将珠子与溶液分离。吸出澄清的溶液并丢弃。向磁珠中加入 200 μL 80% 乙醇 (v/v),孵育 30 秒,然后去除乙醇。重复此洗涤步骤两次以确保完全去除乙醇。
- 将板放在磁力架上,让样品自然干燥 3 分钟。从磁力架上取下板,加入 40 μL TE 缓冲液,pH 值为 8.0。通过上下吹打 10 次来混合。让样品在 RT 下静置 2 分钟。
- 将 PCR 反应板放在磁力架上再放置一分钟。1 分钟后,将上清液转移到新板中。使用分光光度计测量纯化的 PCR 产量,并按照制造商的说明,使用带有光学管联管盖的光学联管联排以及高灵敏度 D1000 ScreenTape 和高灵敏度 D1000 试剂(分子量标准和缓冲液)在 TapeStation 上进行分析。将制备的样品在 -20 °C 下储存长达数月。
- 将 Cas9 核酸酶蛋白稀释至 1 μM,如下所示:混合 2 μL 10x Cas9 缓冲液(终浓度 1x)、1 μL Cas9 核酸酶、 化脓性链球菌 (终浓度 1 μM)和 17 μL 不含核酸酶的 H2O,总体积为 20 μL。
- 执行无 RNase 程序以防止 gRNA 降解。在 H2O 中将 gRNA(从步骤 3.1 开始)稀释至 3 μM,至总体积为 10 μL。
注:使用以下公式估计 gRNA 的分子量:ssRNA 的分子量 (g/mol) = (ssRNA 的长度 (nt) x 321.47 g/mol) + 18.02 g/mol。作为参考,3 μM 的 104 nt 长 gRNA 约为 100 ng/μL。
5. DNA 剪切 (3 h)
- 首先将控制臂归位,准备 ME220。然后,用纯化的去离子 H2O 填充储液槽。在控制站笔记本电脑上,访问 Water Works 并单击 Fill。将温度调节至 4.5 °C。
- 将 25 μg gDNA 转移到微管(微管-130 AFA 纤维螺帽)中。然后,用 1x TE 将试管填充至总体积为 130 μL。使用以下条件将 DNA 剪切至约 300 bp 的平均长度:将 Duration 设置为 10 s; 峰值功率 达到 70; 占空比 % 至 20; Cycles / Burst 至 50;所有这些都会自动将 Avg Power 设置为 14.0。
6. 纯化剪切的基因组 DNA (1 h)
- 将剪切的基因组 DNA 分成两份,每份 65 μL。按照步骤 4.3-4.6 中概述的程序,使用 1.8 倍体积的 XP 微珠 (117 μL) 进行纯化。将上清液转移到新的 PCR 板中,并使用分光光度计测量数量。
- 根据制造商的说明,在 TapeStation 上运行 1 μL 洗脱的剪切 gDNA,以确保 gDNA 被剪切成约 300 bp 的广泛分布。如果需要,将剪切的 gDNA 在 -20 °C 下储存长达数月。
7. CIRCLE-seq 文库的准备(3 天)
- 发夹适配器退火
- 在 1x TE 中重悬 oSQT1288(表 1),发夹接头,至终浓度为 100 μM。
- 按如下方式进行接头退火:混合 40 μL oSQT1288(终浓度 40 μM)、10 μL 10x STE(终浓度 1x)和 50 μL 无核酸酶 H2O,总体积为 100 μL。
- 使用以下退火参数:95 °C 持续 5 分钟,-1 °C 每分钟 70 次循环,无限期保持在 4 °C。
- 执行 End repair 。使用无 PCR 的 HTP 文库制备试剂盒并制备末端修复预混液。
- 混合 8 μL 无核酸酶 H2O、7 μL 10x 末端修复缓冲液(终浓度 1x)和 5 μL 末端修复酶混合物(终体积 20 μL 总末端修复预混液)。
- 将 20 μL 末端修复预混液移液到步骤 4.3-4.6 中剪切的 gDNA 样品中。将 20 μL 末端修复预混液与 50 μL 剪切 gDNA 混合,最终体积为 70 μL。
- 将混合物置于 20 °C 的热循环仪中 30 分钟,然后在 4 °C 下无限期保持。
- 加入 1.7 倍体积或 120 μL 的 XP 微珠,并按照步骤 4.3-4.6 中的纯化步骤进行作。用 42 μL TE (pH 值 8.0) 洗脱。确保珠子保留在溶液中以备下一步使用。
- 执行 A 尾。使用 HTP 文库制备(无 PCR)试剂盒制备 A 尾预混液。
- 混合 5 μL 10x A-tailing 缓冲液(终浓度 1x)和 3 μL A-tailing 酶(8 μL A-tailing Master Mix 的总终体积)。
- 将 8 μL 的 A -tailing Master Mix 移液到每个含有步骤 7.2.4 中珠子的 DNA 样品中,将 8 μL 的 A-tailing Master Mix 混合到 42 μL 含有珠子的末端修复的 DNA 中(最终总体积为 50 μL)。置于 30 °C 的热循环仪中 30 分钟。在 4 °C 下无限期保持。
- 将 1.8 体积或 90 μL 的 PEG/NaCl SPRI 溶液(HTP 文库制备试剂盒的一种组分(无 PCR;96 次反应))移液到 A 尾 DNA 中。根据步骤 4.3-4.6 纯化 A 尾 DNA。在 30 μL TE (pH 8.0) 中洗脱 A 尾 DNA。将珠子保存在溶液中以备下一步使用。
- 进行接头连接。使用 HTP 文库制备(PCR-free)试剂盒,制备接头连接预混液。
- 混合步骤 7.1.3 中的 10 μL 5x 连接缓冲液(终浓度 1x)、5 μL DNA 连接酶和 5 μL 退火发夹接头 (40 μM)。确保总接头连接预混液的最终浓度为 4 μM,总浓度为 20 μL。
- 将 20 μL 接头连接预混液移液到每个洗脱的 DNA 样本中,该样本含有步骤 7.3.3 中的珠子(每个样本的总最终体积为 50 μL)。
- 置于 20 °C 的热循环仪中 1 小时。在 4 °C 下无限期保持。
- 将 1x 体积或 50 μL 的 PEG/NaCl SPRI 溶液转移到接头连接的 DNA 中,并根据步骤 4.3-4.6 进行纯化。用 30 μL TE(pH 值为 8.0)洗脱,然后将上清液倒入新的半裙边 PCR 板中。使用 dsDNA BR 检测合并并定量 DNA。如果需要,将纯化的接头连接的 DNA 在 -20 °C 下储存长达 1 个月。
- 制备 Lambda 核酸外切酶/核酸外切酶 I(大肠杆菌) 预混液(用于去除两端未连接接头的单链或双链 DNA)。
- 从步骤 7.4.4 中取出 1 μg 接头连接的 DNA,将其稀释至 40 μL。混合 5 μL 10x 核酸外切酶 I 反应缓冲液(终浓度 1x)、4 μL λ核酸外切酶(终浓度 0.4 U/μL)和 1 μL 核酸外切酶 I(大肠杆菌)(终浓度 0.4 U/μL),得到总体积为 10 μL 的 Lambda 核酸外切酶/核酸外切酶 I 预混液。
- 将 10 μL Lambda 核酸外切酶/核酸外切酶 I 预混液移液到 40 μL (1 μg) 接头连接的 DNA(总体积为 50 μL)中。置于热循环仪中,37°C放置1小时,然后在75°C放置10分钟。在 4 °C 下无限期保持。
- 将 1.8 倍体积或 90 μL XP 珠子移液到 Lambda 核酸外切酶/核酸外切酶 I 处理的 DNA 中。根据步骤 4.3-4.6 中的说明进行纯化。在 40 μL TE 中洗脱,pH 值为 8.0。确保磁珠保留在溶液中,用于下一个酶促步骤。
- 用 USER 酶和 T4 多核苷酸激酶 (PNK) 处理。制备 USER/T4 PNK 预混液(释放 4 bp 突出端和制备后续连接反应所需的即用型 DNA 末端)。
- 混合 5 μL 10x T4 DNA 连接酶缓冲液(终浓度 1x)、3 μL USER 酶(终浓度 0.05 U/μL)和 2 μL T4 PNK(终浓度 0.4 U/μL),使 USER/PNK 预混液总体积为 10 μL。
- 将 10 μL 的 USER Enzyme/T4 PNK Master Mix 移液到 40 μL 含有步骤 7.5.3 中珠子的 lambda 和核酸外切酶 I 处理的 DNA 标本中,总体积为 50 μL。置于 37 °C 的热循环仪中 1 小时。在 4 °C 下无限期保持。
- 将 1.8 倍体积或 90 μL 的 PEG/NaCl SPRI 溶液移液到用 USER/T4 PNK 处理的 DNA 中,并根据步骤 4.3-4.6 进行纯化。在 35 μL TE 中洗脱,pH 值为 8.0。将上清液倒入新的半裙边 PCR 板中。使用 dsDNA HS 检测合并定量 DNA。
- 进行分子内环化。制备循环预混液。
- 混合 8 μL 无核酸酶 H2O、10 μL 10x T4 DNA 连接酶缓冲液(终浓度 1x)和 2 μL T4 DNA 连接酶(终浓度 8 U/μL),使环化预混液总体积为 20 μL。
- 将 20 μL 循环预混液移液到 500 ng 步骤 7.6.3 中用 USER/PNK 处理的 DNA 中。在 80 μL 中稀释 500 ng 经 USER/PNK 处理的 DNA,然后加入 20 μL 环化预混液(总体积为 100 μL)。在热循环仪中于 16 °C 孵育 16 小时(过夜)。
- 向环化 DNA 中加入 1x 体积或 100 μL 的 XP 珠子,并根据步骤 4.3-4.6 进行纯化。在 38 μL TE 中洗脱,pH 值为 8.0。将上清液倒入新的半裙边 PCR 板中。
- 用质粒安全的 ATP 依赖性 DNase 处理。制备质粒安全的 ATP 依赖性 DNase 预混液(降解残留线性 DNA 所需)。
- 混合 5 μL 10x 质粒安全反应缓冲液(终浓度 1x)、2 μL ATP(终浓度 1 mM)和 5 μL 质粒安全 ATP 依赖性 DNase(终浓度 1 U/μL),质粒安全预混液总体积为 12 μL。
- 从步骤 7.7.3 中移液 12 μL ATP 依赖性 DNase 预混液到 38 μL 环化 DNA 中(总体积为 50 μL)。在热循环仪中于 37 °C 孵育 1 小时,然后在 70 °C 下孵育 30 分钟。在 4 °C 下无限期保持。
- 将 1 倍体积或 50 μL 的 XP 珠子移液到用质粒安全的 ATP 依赖性 DNase 处理的 DNA 中,并根据步骤 4.3-4.6 进行纯化。在 15 μL TE (pH 8.0) 中洗脱。将上清液倒入新的半裙边 PCR 板中。
- 合并 DNA 并使用 dsDNA HS 检测进行定量。如果需要,可将环状 DNA 在 -20 °C 下储存长达数月。
8. 在 体外 切割酶纯化的环状 gDNA (2 h)
- 使用 Cas9:gRNA 复合物进行 体外 切割。制备 体外 Cleavage Master Mix。混合 5 μL 10x Cas9 缓冲液(终浓度 1x)、4.5 μL 化脓性链球菌 Cas9(终浓度 90 nM)和 1.5 μL gRNA(终浓度 90 nM),使切割预混液总体积为 11 μL。
- 将切割预混液在 RT 下保持 10 分钟以形成 Cas9:gRNA RNP 复合物。
- 从步骤 7.8.3 中稀释 125 ng 质粒安全 DNase 处理的 DNA,至最终体积为 39 μL。然后,将 11 μL 切割预混液添加到 39 μL 质粒安全型 DNase 处理的 DNA 中,总体积为 50 μL。
注:在此步骤中包括一个阴性对照样本,该样本包含与 Cas9 缓冲液混合的环状 DNA,不含 Cas9:gRNA 复合物。 - 在热循环仪中于 37 °C 孵育 1 小时。 无限期保持在 4 °C。向 体外切割的 DNA 中加入 50 μL(1 倍体积)的 XP 珠子,并按照步骤 4.3-4.6 纯化 DNA。在 42 μL TE 缓冲液(pH 值为 8.0)中洗脱。确保珠子留在溶液中以备下一步使用。
9. 下一代测序文库的制备(4 - 6 小时)
- 执行 A 尾。准备 A 尾预混液。
- 混合 5 μL 10x A-tailing 缓冲液(终浓度 1x)和 3 μL A-tailing 酶(A-tailing 预混液的总体积为 8 μL)。
- 将 8 μL 的 A 型预混液移液到 42 μL 含有步骤 8.4 中珠子的洗脱 DNA 样品中(总体积为 50 μL)。置于热循环仪中,在 30 °C 下放置 30 分钟。 在 4 °C 下无限期保持。
- 将 1.8 倍体积的 PEG/NaCl SPRI 溶液或 90 μL 移液到 A 尾 DNA 中,并根据步骤 4.3-4.6 纯化 DNA。在 25 μL TE 中洗脱,pH 值为 8.0。确保将珠子保存在溶液中,以便进行下一步作。
- 进行接头连接。准备接头连接预混液。
注:必须准备 NEB 接头的一次性等分试样,以防止冻融诱导的 3' T' 水解导致接头二聚体的形成。- 混合 10 μL 5x 连接缓冲液(终浓度 1x)、5 μL DNA 连接酶和 10 μL 用于测序的接头(终浓度为 3 μM),总共 25 μL。
- 将 25 μL 接头连接预混液移液到 25 μL 含有步骤 9.1.3 中珠子的 A 尾 DNA 样品中。置于热循环仪中,在 20 °C 下放置 1 小时。 在 4 °C 下无限期保持。
- 将 1x 体积或 50 μL 的 PEG/NaCl SPRI 溶液移液到接头连接的 DNA 中,并根据步骤 4.3-4.6 纯化 DNA。在 47 μL TE 中洗脱,pH 值为 8.0。确保磁珠保留在溶液中,用于下一个酶促步骤。
- 用 USER 酶进行处理(在尿嘧啶残基处产生单个核苷酸间隙)。
- 将 3 μL USER 酶(包含在 Dual Index Primers Kit 中)添加到包含步骤 9.2.3 中珠子的接头连接的 DNA 标本中。在 37 °C 孵育 15 分钟。
- 向 USER 酶处理的 DNA 中加入 35 μL(0.7 倍体积)的 PEG/NaCl SPRI 溶液,并根据步骤 4.3-4.6 进行纯化。在 20 μL TE 缓冲液(pH 值为 8.0)中洗脱。将上清液转移到新的半裙边 PCR 板中,并使用 dsDNA HS 测定法测量 DNA 浓度。预期浓度应约为 2-5 ng/μL。
- (可选)在进行下一步之前,可以使用 PippinHT 进行 DNA 大小选择。使用大小范围为 250-850 bp 的 1.5% PippinHT 盒。所得样品可直接用于下一步的 PCR。
- 进行 PCR 以添加条形码
注:确保为每个样品选择的引物序列组合是唯一的。如果可能,每个样品都应具有唯一的 i5 和 i7 条形码。- 制备 PCR 预混液以添加双索引条形码。混合 5 μL 无核酸酶的 H2O、25 μL 2x 热启动预混液(终浓度 1x)、5 μL i5 引物(终浓度 1 μM)和 5 μL i7 引物(终浓度 1 μM)(总预混液体积为 40 μL)。
- 将 40 μL PCR 预混液移液到 10 μL 纯化的 DNA 中,该纯化 DNA 经过步骤 9.3.2 中的 USER 酶处理(约 20 ng)(总体积为 50 μL)。
- 选择以下 PCR 热循环条件:变性:98 °C 45 秒,1 个循环,变性:98 °C 15 秒,20 个循环,退火:65 °C 30 秒,20 个循环,延伸:72 °C 30 秒,20 个循环,最终延伸:72 °C 1 分钟,1 个循环,保持:4 °C 无限期。
- 向 PCR 产物中加入 0.7 倍体积或 35 μL XP 珠子,并根据步骤 4.3-4.6 进行纯化。在 30 μL TE 中洗脱,pH 值为 8.0。将上清液倒入新的半裙边 PCR 板中。如有需要,可将环化 DNA 在 -20°C 下储存长达数月。
注意:在 Tapestation 上运行 PCR 样品以控制文库的质量并评估接头二聚体的形成。如果检测到接头二聚体,请重复步骤 9.5.4。
10. 通过微滴式数字 PCR 定量 CIRCLE-seq 文库 (dd_PCR) (6 h)
注:也可以使用 qPCR、Tapestation 或类似方法进行定量。
- 从文库中的 5 μL DNA(PCR 步骤 9.5.4)开始,与 45 μL 无核酸酶的 TE 充分混合,然后以 50 μL 体积对每个样品进行 1:10 的连续稀释,稀释度范围为 10-1 至 10-8 。
- 设置 dd_PCR Master Mix 储备液。混合 11 μL 2x dd_PCR探针混合物(终浓度 1x)、0.055 μL 探针 oSQT1310(终浓度 250 nM)、0.055 μL 探针 oSQT1311(终浓度 250 nM)、0.099 μL 引物 oSQT1274(终浓度 450 nM)、0.099 μL 引物 oSQT1275(终浓度 450 nM)和 6.292 μL 无核酸酶 H2O, dd_PCR 预混液总体积为 17.6 μL。为所有样品制备预混液,以确保体积足以进行准确移液。
- 测定三种最低稀释度(10-6、10-7 和 10-8),一式两份(在 96 孔板中)。必须使用非模板对照 (NTC)。
- 按如下方式向每个样品中移取 17.6 μL dd_PCR Master Mix:将 17.6 μL 与 4.4 μL 样品混合(向孔中加入不含核酸酶的 H2O,含 NTC),总体积为 22 μL。密封板,然后在室温下以 2000 x g 离心 1 分钟。
- 执行液滴生成、热循环和分析。使用 Droplet Reader PCR 系统,将 DG8 小柱(8 孔)转移到小柱支架中。在卡式瓶的油行中,为探针分配 70 μL 液滴发生油。
注:从步骤 10.1.2 中取出 20 μL 样品。并将其添加到 8 孔小柱的样品行中,用 DG8 橡胶垫圈覆盖小柱,将其放入液滴发生器中,然后关闭它以开始该过程(自动)。完成后,取出小柱,将 40 μL 从 8 孔小柱的液滴行中移入半裙边 96 孔 PCR 板中,确保缓慢移液。- 将加热块放入 PX1 PCR 板密封机中。打开后,封口机将开始加热至 180 °C。将铝箔热封放在板上,确保红线在顶部。将板放入 PX1 中,然后按 Seal。
- 选择以下热循环仪条件:酶活化:95 °C 10 分钟,1 个循环,变性:94 °C 30 秒,40 个循环,退火/延伸:60 °C 1 分钟,40 个循环,酶失活:98 °C 10 分钟,1 个循环,保持:4 °C 无限期。
- 在微滴检测仪上,打开兼容的软件并选择要读取的孔。选择 ABS 作为实验类型,并为探针 dd_PCR Supermix 。为目标 1 选择 Ch1 Unknown ,为目标 2 选择 Ch2 Unknown 。选择 Apply(应用),然后选择 OK(确定)。将板放入微液检测仪中。对于染料组,选择 FAM/HEX ,然后单击 Run。
- 分析 dd_PCR 结果。使用阴性对照作为参考,对双阳性液滴群体进行门控。计算重复值的平均值,然后乘以稀释因子和dd_PCR的 5 倍稀释因子。
注:每微升总拷贝数计算如下:每微升总拷贝数 = 稀释因子×平均值 5 ×,其中"平均值"表示 Ch1 和 Ch2 的平均定量值。 - 将所有样品合并为一个等摩尔浓度的文库。1x 混合文库应包含约 4.5 x 109 个分子,总体积为 5 μL。
11. 下一代测序
- 将样品提交给外部机构进行测序,确保记录正确的接头序列。
12. CIRCLE-seq 数据分析 (1 - 3 小时)
- 安装 Python 版本 2.7、Burrows-Wheeler Aligner (BWA) 和 SAMtools。从 http://hgdownload.cse.ucsc.edu/goldenPath/hg38/bigZips/hg38.fa.gz 下载参考基因组(例如,hg38)。
注意:如果目标物种的基因组不可用,CIRCLE-seq 计算工作流程可以在不依赖参考的模式下运行。在这种情况下,可以跳过此步骤。 - 使用以下命令下载并安装 CIRCLE-seq 管道:(1) git clone https://github.com/tsailabSJ/circleseq.git,(2) cd circleseq,(3) pip install -r requirements.txt。
- 创建 YAML 格式 (.yaml) 的清单文件。下面是一个示例清单,可与 CIRCLE-seq 软件中提供的示例数据集一起使用,以测试工作流程。
注意:(1) 参考基因组:data/input/CIRCLEseq_test_genome.fa;(2) analysis_folder:数据/输出;(3) BWA:BWA;(4) samtools:samtools;(5) read_threshold:;(6) window_size:;(7) mapq_threshold:<值>;(8) start_threshold:<值>;(9) gap_threshold:;(10) mismatch_threshold:;(11) merged_analysis:对;(12) 样品:U2OS_EMX1;(13) 目标:GAGTCCGAGCAGAAGAAGAAGAANGG;(14) read1: 数据/输入/EMX1.r1.fastq.gz;(15) read2: 数据/输入/EMX1.r2.fastq.gz;(16) controlread1: 数据/输入/EMX1_control.r1.fastq.gz;(17) controlread2: 数据/输入/EMX1_control.r2.fastq.gz;(18) 描述: U2OS.使用了以下清单值:read_threshold:4、window_size:3、mapq_threshold:50、start_threshold:1、gap_threshold:3、mismatch_threshold:6 - 定义参考基因组 FASTA 文件、用于分析的输出目录以及 BWA 和 SAMtools 命令的路径。定义核酸酶切割样品和对照样品的靶序列和解多路 FASTQ 文件的路径。通过将多个实验包含在单个清单文件中,可以在批处理模式下同时处理多个实验。
- 对于基于标准引用的分析,执行以下命令:(1) python /path/to/circleseq.py all - manifest; (2) /path/to/manifest.yaml。
- 或者,对于标准的非基于引用的分析,请执行以下命令:(1) Python /path/to/circleseq.py reference-free - manifest;(2) /path/to/manifest.yaml 中。
- 执行完整管道时,在为该特定步骤指定的不同 output_folder 中查找每个步骤的输出结果。
结果
在这里,CIRCLE-seq 用于研究复合物中核酸酶诱导的 Cas9 切割位点,其中 gRNA 旨在使用从诱导多能干细胞 (iPSC) 中分离的 DNA 靶向腺相关病毒整合位点 1 (AAVS1)。该 gRNA 之前在我们的出版物27 中进行了描述。从 iPSC 中分离出约 25 μg gDNA,通过聚焦超声剪切,并使用 AMPure XP 微珠纯化选择大小,以产生约 300 bps 的片段。从这 25 μg DNA 中,成功环化约 2-5 ng DNA 用于 体外 Cas9:gRNA 切割。整个过程如图 1 所示。
在使用我们的计算工作流程进行 CIRCLE-seq 程序和分析之后, 图 2A 显示了所有检测到的脱靶和脱靶切割位点的可视化。CIRCLE-seq 管道还提供"合并读数",通过 R 统计软件进行分析,以产生曼哈顿图,显示检测到的核酸酶诱导的切割位点沿每条染色体映射(图 2B)。
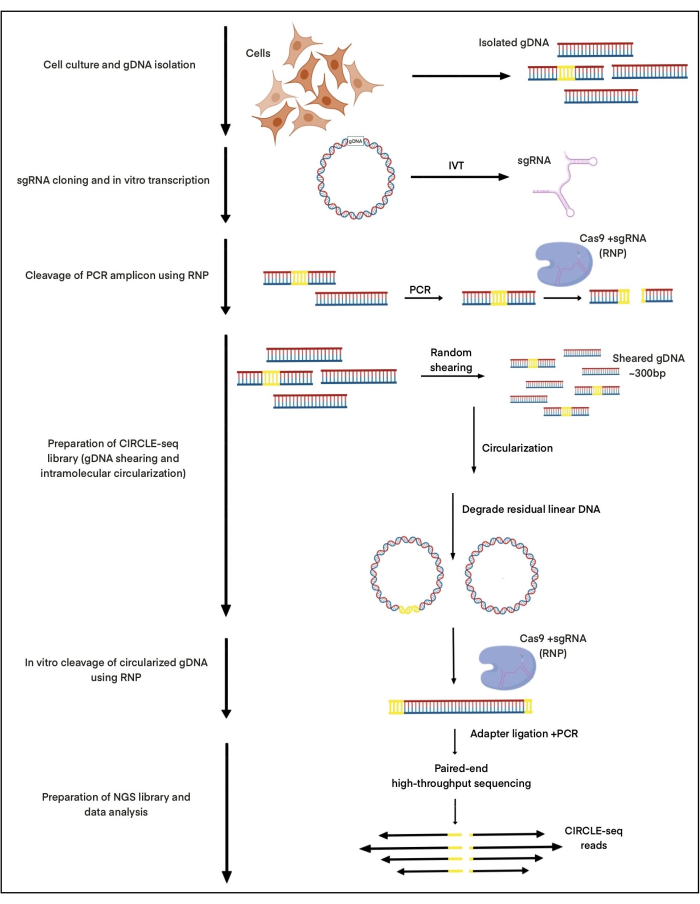
图 1:CIRCLE-seq 工作流程示意图。 指出了协议的主要步骤。 请单击此处查看此图的较大版本。
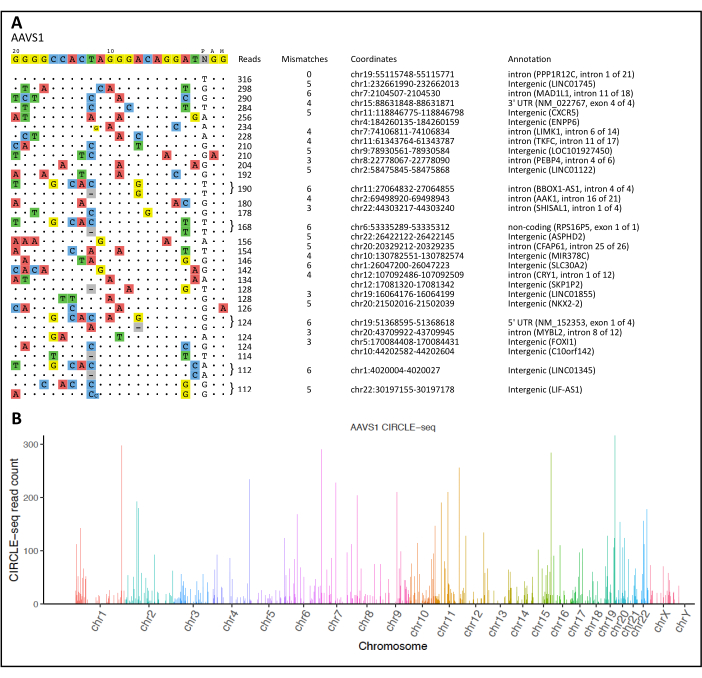
图 2:CIRCLE-seq 可视化和曼哈顿图。 (A) 脱靶位点与 AAVS1 基因座的预期靶标的比对。目标序列显示在顶部,其中非目标按读取计数降序排列。原始靶序列的差异由彩色核苷酸显示。显示了 AAVS1 位点的主要意外切割位点的样本。(B) 曼哈顿图说明了检测到的 AAVS1 基因座的意外切割位点。条形高度表示每个染色体位置的读取计数。 请单击此处查看此图的较大版本。
| 底漆 | 序列 (5'-3') | 评论/描述 |
| AAVS1 单向导 RNA (sgRNA) | GGGGCCACUAGGGACAGGAU | 用于 AAVS1 位点的荧光蛋白敲入 |
| AAVS1 正向引物 | GCTCTGGGCGGAGGAATATG | 用于 gRNA 体外切割试验 |
| AAVS1 反向引物 | ATTCCCAGGGCCGGTTAATG | 用于 gRNA 体外切割试验 |
| oSQT1288 | /5Phos/CGGTGGACCGATGATC /ideoxyU/ATCGGTCCACCGaT | CIRCLE-seq 发夹适配器 |
| oSQT1274 | AATGATACGGCGACCACCGAG | TruSeq F1 |
| oSQT1275 | CAAGCAGAAGACGGCATACGAGAT | TruSeqF2 系列 |
| oSQT1310 | /56-FAM/CCTACACGA/ZEN/CGCTCTTCCGATCT/3IABkFQ/ | TruSeq 探针 |
| oSQT1311 | /5HEX/TCGGAAGAG/ZEN/CACACGTCTGAACT/3IABkFQ/ | TruSeq 探针 |
表 1:用于 AAVS1 基因座 CIRCLE-seq 分析的 gRNA 和引物序列。
讨论
在这里,CIRCLE-seq 被证明是一种无偏倚且高度敏感的技术,用于识别整个基因组中核酸酶诱导的 DSB,这些 DSB 是由于靶向 iPSC 来源的 gDNA 中的 AAVS1 基因座而产生的。iPSC 中的 AAVS1 位点是众所周知的安全港位点,通常用作使用 CRISPR-Cas9 的外源基因的整合位点28。我们最近的报告通过将组成型表达的 EGFP 报告基因进行 CRISPR 介导的整合到 AAVS1 位点来研究 EGFP 标记的 iPSC 的潜力,由于 EGFP 在整个细胞谱系中持续存在,因此能够标记和跟踪 iPSC 和分化的 iPSC27。该 iPSC 系可在 体内 用于评估移植后 iPSC 衍生细胞的生物分布。由于该细胞系已经过 CRISPR 修饰,也用于测试 iPSC 的临床应用,因此必须了解并询问潜在的 AAVS1 脱靶位点,以确保安全性和有效性,使其成为测试 CIRCLE-seq 的理想位点。
Butterfield 等人之前发表的研究27 与这项研究之间的一个显着区别是使用修饰的 gRNA 来靶向 AAVS1 基因座。gRNA 可以设计为提高基因组编辑的准确性24。引导序列是影响脱靶和脱靶效率的最关键因素。因此,将所选 gRNA 与其他几种指南进行比较测试,发现具有优异的保真度。此外,一篇关于重编程人成纤维细胞的方法文章证实了这一发现,即含有修饰核碱基的合成加帽 mRNA 受益于抗病毒反应的低激活29,30。虽然低免疫原性在体外检测中可能无关紧要,但当最终目标是开发可用于活细胞的临床相关疗法时,它就变得至关重要。
与类似方法相比,CIRCLE-seq 具有许多优势。例如,Digenome-seq 使用 ~4 亿个读数对核酸酶切割和未切割的 gDNA 进行测序21。这会导致高背景,因此很难过滤掉低频的真实切割位点。由于核酸酶切割的 gDNA 富集,CIRCLE-seq 仅使用 ~3-5 百万个读数,导致低背景。此外,Digenome-seq 和类似方法 SITE-seq 依赖于对单个核酸酶切割的 DNA 末端进行测序。相比之下,CIRCLE-seq 读数包括切割位点的两端,无需参考 21,22,26 即可识别脱靶位点。
CIRCLE-seq 的一个优点是与依赖细胞培养的方法(如 GUIDE-seq)相比,其灵敏度更高。当比较这两种方法时,CIRCLE-seq 能够捕获 GUIDE-seq 检测到的所有脱靶位点,并发现 GUIDE-seq 遗漏的其他意外切割位点。然而,一个显着的区别是 GUIDE-seq 可能受到表观遗传景观的阻碍,而 CIRCLE-seq 可以访问整个基因组。
作为一种 体外 检测,CIRCLE-seq 存在几个局限性,其中第一个是检测假阳性。表观遗传学会阻碍 体内某些位点的核酸酶活性,而超声处理可在 体外消除这些障碍,从而在细胞环境中通常可能无法接近的位置进行脱靶活性。此外,Cas9 在该 体外 测定中以高浓度存在,允许进行体内不可能的 切割。该测定还需要相对大量的起始 gDNA,根据可用资源,这可能会否定该方案的使用。最后,由于当前下一代测序技术的限制,一些脱靶位点可能无法检测到。
最近的一项研究使用了 计算机模拟 方法,其算法确定了许多相关参数,用于比较不同的核酸酶表征方法,包括 CIRCLE-seq 和 GUIDE-seq31。其中两个相关参数是 "cut-site enrichment" 和 "% false positives"。有趣的是,CIRCLE-seq 的假阳性率计算为 88%,但其切割位点富集比其他 体外 方法高出 10 倍。对每种方法的比较分析表明,GUIDE-seq 表现最佳,因为它表现出最高的靶向特异性,只有中等的假阳性率32。这并没有使 CIRCLE-seq 无效,而是暗示了结合使用 CIRCLE-seq 和 GUIDE-seq 的可能性,用 GUIDE-seq 验证 CIRCLE-seq 的发现,因为前者具有更高的灵敏度,而后者是一种基于细胞的方法,具有高切割位点富集。数据还表明,基于扩增子的下一代测序 (NGS) 应该是在潜在候选位点鉴定真正脱靶修饰的首选方法31。该数据表明了一种潜在的策略,即使用 CIRCLE-seq,然后是 GUIDE-seq,然后是基于扩增子的 NGS 来检查脱靶效应。
披露声明
作者没有什么可披露的。
致谢
对美国国立卫生研究院 (R01AR078551 和 T32AR007411)、奥地利营养不良性大疱性表皮松解症研究协会 (DEBRA)、盖茨 Grubstake 基金和盖茨前沿基金提供的资金支持表示最深切的感谢。
材料
| Name | Company | Catalog Number | Comments |
| 0.2-mL Thin-walled Tubes and Flat Caps | ThermoFisher Scientific | AB1114 | |
| 1.5% PippinHT cassette | Sage Science | HTC1510 | |
| 10 mL Serological Pipettes | FisherScientific | 12567603 | |
| 15 mL Conical Tube | FisherScientific | 339651 | |
| 150 x 25 mm Tissue Culture Dish | FisherScientific | 877224 | |
| 25 mL Reagent Reservoir | FisherScientific | 2138127C | |
| 2x Kapa KiFi HotStart Ready Mix | Kapa Biosystems | KK2602 | |
| 5 mL Serological Pipettes | FisherScientific | 170355 | |
| 50 mL Conical Tube | FisherScientific | 339653 | |
| 50x TAE Electrophoresis Buffer | ThermoFisher Scientific | B49 | |
| 6 Well Cell Culture Plate | Corning | 3516 | |
| Agencourt AMPure XP Reagent, 60 mL | Beckman Coulter | A63881 | |
| Benchtop Microcentrifuge | Eppendorf | 5400002 | |
| BsaI-HF | New England BioLabs | ||
| Buffer QX1 | Qiagen | 20912 | |
| Cas9 nuclease, Streptococcus Pyogenes | New England BioLabs | M0386M | |
| CIRCLE-seq Library Preparation and NGS | |||
| Corning Matrigel hESC-Qualified Matrix | Corning | 354277 | Extracellular matrix (ECM) for culturing iPSCs |
| ddPCR SuperMix for Probes | Bio-Rad | 1863010 | |
| DG8 Cartridge Holder | Bio-Rad | 1863051 | |
| DG8 Cartridges | Bio-Rad | 1864008 | |
| DG8 Gaskets | Bio-Rad | 1863009 | |
| DPBS, no calcium, no magnesium | ThermoFisher Scientific | 14190144 | Made by Invitrogen |
| Droplet Generation Oil for Probes | Bio-Rad | 1863005 | |
| Droplet Reader Oil | Bio-Rad | 1863004 | |
| EDTA (0.5 M) | ThermoFisher Scientific | 15575020 | |
| EDTA (0.5 M), pH 8.0, RNase-free | ThermoFisher Scientific | AM9260G | Made by Invitrogen |
| Eppendorf ThermoMixer | Eppendorf | 5384000020 | |
| Equipment | |||
| Ethyl Alcohol, Pure | Sigma-Aldrich | E7023 | |
| Exonuclease I | New England BioLabs | M0293L | E. coli |
| Filter Unit | FisherScientific | FB0875713 | |
| Filtered Sterile Pipette Tips | |||
| Focused Ultrasonicator | Covaris | ME220 | |
| Genomic DNA Isolation | |||
| Genomic DNA Shearing | |||
| Gentra Puregene Cell Core Kit | Qiagen | 158043 | |
| gRNAs | Synthego | ||
| HCl | ThermoFisher Scientific | A144500 | |
| Heracell VIOS Tri-gas Humidified Tissue Culture Incubator | ThermoFisher Scientific | 51030411 | Need for culturing and expanding iPSCs (37 °C/5% CO2/5% O2) |
| High Sensitivity D1000 DNA ScreenTape | Agilent | 5067-5584 | |
| High Sensitivity D1000 Reagents | Agilent | 5067-5585 | |
| HTP Library Preparation Kit | Kapa Biosystems | KK8235 | |
| HyClone Antibiotic Antimycotic Solution | Cytiva | SV30079.01 | |
| IDTE pH 8.0 (1x TE Solution) | Integrated DNA Technologies | 11050204 | |
| Inverted Microscope | Need for imaging iPS colonies in bright-field and fluorescent channels | ||
| iPSC Culture | |||
| Isopropanol | Sigma-Aldrich | 190764 | |
| Lambda Exonuclease | New England BioLabs | M0262L | |
| Loading Tips, 10 pack | Agilent | 5067-5599 | |
| Magnum FLX Enhanced Universal Magnet Plate | Alpaqua | A00400 | |
| Microcentrifuge Tube | Axygen | 31104051 | |
| Microtube AFA Fiber Pre-Slit Snap-Cap | Covaris | 520045 | |
| mTeSR-1 5x Supplement | StemCell Technology | 85852 | |
| mTeSR-1 Basal Medium (400 mL) | StemCell Technology | 85851 | Media for maintaining iPSC in culture |
| Nanodrop 8000 Spectrophotometer | ThermoFisher Scientific | ND-8000-GL | |
| NEBNext Multiplex Oligos for Illumina | New England BioLabs | E7600S | Dual Index Primers Set 1 |
| Optical tube strip caps, 8x strip | Agilent | 401425 | |
| Optical tube strips, 8x strip | Agilent | 401428 | |
| Other Reagents | |||
| PCR Plate Sealer | Bio-Rad | PX1 | Model Number PX1 |
| PEG/NaCl SPRI Solution | Kapa Biosystems | ||
| Phusion Hot Start Flex 2x Master Mix | New England BioLabs | M0536L | |
| Pierceable Foil Heat Seal | Bio-Rad | 1814040 | |
| Plasmid-Safe ATP-dependent DNase | Epicentre | E3110K | |
| Primers, Adapters and Probes | IDT | Sequences are listed in Table 1 | |
| Proteinase K | Qiagen | 19131 | |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | |
| QIAxcel Gel Analysis System | Qiagen | 9001941 | |
| Qubit Assay Tubes | ThermoFisher Scientific | Q32856 | |
| Qubit dsDNA BR Assay Kit | ThermoFisher Scientific | Q32853 | |
| Qubit dsDNA BR Assay Kit | ThermoFisher Scientific | Q32853 | |
| Qubit dsDNA HS Assay Kit | ThermoFisher Scientific | Q32854 | |
| Qubit Fluorometer | ThermoFisher Scientific | Q33226 | |
| QX200 Droplet Digital PCR System | Bio-Rad | 1864001 | Contain a QX200 droplet generator and a QX200 droplet reader |
| RNase A | Qiagen | 19101 | |
| Semi-skirted PCR Plate | ThermoFisher Scientific | 14230244 | |
| SeqPlaque GTG Agarose | Lonza | 50110 | |
| SYBR Safe DNA Gel Stain | ThermoFisher Scientific | S33102 | |
| T4 DNA Ligase | New England BioLabs | M0202L | |
| T4 Polynucleotide Kinase (PNK) | New England BioLabs | M0201L | |
| T-75 Flasks | FisherScientific | 7202000 | |
| Tapestation 4150 | Agilent | G2992AA | |
| Thermocycler with programmable temperature-stepping functionality | Bio-Rad C1000 Touch | ||
| Tris base | ThermoFisher Scientific | BP1521 | |
| Twin.tec PCR Plate | Eppendorf | E951020346 | 96 wells, semi-skirted, green |
| UltraPure DNase/RNase-Free Distilled Water | ThermoFisher Scientific | 10977015 | |
| USER Enzyme | New England BioLabs | M5505L |
参考文献
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Frangoul, H., et al. CRISPR-Cas9 gene editing for sickle cell disease and beta-thalassemia. N Engl J Med. 384 (3), 252-260 (2021).
- Ledford, H. CRISPR treatment inserted directly into the body for first time. Nature. 579 (7798), 185 (2020).
- Gillmore, J. D., et al. CRISPR-Cas9 in vivo gene editing for transthyretin amyloidosis. N Engl J Med. 385 (6), 493-502 (2021).
- Fu, Y., et al. High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells. Nat Biotechnol. 31 (9), 822-826 (2013).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol. 31 (9), 827-832 (2013).
- Anzalone, A. V., et al. Search-and-replace genome editing without double-strand breaks or donor DNA. Nature. 576 (7785), 149-157 (2019).
- Hong, S. A., et al. Therapeutic base editing and prime editing of COL7A1 mutations in recessive dystrophic epidermolysis bullosa. Mol Ther. 30 (8), 2664-2679 (2022).
- Zhang, G., et al. Enhancement of prime editing via xrRNA motif-joined pegRNA. Nat Commun. 13 (1), 1856 (2022).
- Lee, J., et al. Prime editing with genuine Cas9 nickases minimizes unwanted indels. Nat Commun. 14 (1), 1786 (2023).
- Howe, S. J., et al. Insertional mutagenesis combined with acquired somatic mutations causes leukemogenesis following gene therapy of SCID-X1 patients. J Clin Invest. 118 (9), 3143-3150 (2008).
- Hacein-Bey-Abina, S., et al. Insertional oncogenesis in 4 patients after retrovirus-mediated gene therapy of SCID-X1. J Clin Invest. 118 (9), 3132-3142 (2008).
- Has, C., Kiritsi, D. Therapies for inherited skin fragility disorders. Exp Dermatol. 24 (5), 325-331 (2015).
- Murnane, J. P., Yezzi, M. J., Young, B. R. Recombination events during integration of transfected DNA into normal human cells. Nucleic Acids Res. 18 (9), 2733-2738 (1990).
- Hsieh, M. M., et al. Myelodysplastic syndrome unrelated to lentiviral vector in a patient treated with gene therapy for sickle cell disease. Blood Advances. 4 (9), 2058-2063 (2020).
- Tsai, S. Q., et al. GUIDE-seq enables genome-wide profiling of off-target cleavage by CRISPR-Cas nucleases. Nat Biotechnol. 33 (2), 187-197 (2015).
- Wang, X., et al. Unbiased detection of off-target cleavage by CRISPR-Cas9 and TALENs using integrase-defective lentiviral vectors. Nat Biotechnol. 33 (2), 175-178 (2015).
- Lensing, S. V., et al. DSBCapture: In situ capture and sequencing of DNA breaks. Nat Methods. 13 (10), 855-857 (2016).
- Crosetto, N., et al. Nucleotide-resolution DNA double-strand break mapping by next-generation sequencing. Nat Methods. 10 (4), 361-365 (2013).
- Yan, W. X., et al. BLISS is a versatile and quantitative method for genome-wide profiling of DNA double-strand breaks. Nat Commun. 8, 15058 (2017).
- Kim, D., et al. Digenome-seq: Genome-wide profiling of CRISPR-Cas9 off-target effects in human cells. Nat Methods. 12 (3), 237-243 (2015).
- Cameron, P., et al. Mapping the genomic landscape of CRISPR-Cas9 cleavage. Nat Methods. 14 (6), 600-606 (2017).
- Wienert, B., Wyman, S. K., Yeh, C. D., Conklin, B. R., Corn, J. E. CRISPR off-target detection with DISCOVER-seq. Nat Protoc. 15 (5), 1775-1799 (2020).
- Guo, C., Ma, X., Gao, F., Guo, Y. Off-target effects in CRISPR/Cas9 gene editing. Front Bioeng Biotechnol. 11, 1143157 (2023).
- Conant, D., et al. Inference of CRISPR edits from Sanger trace data. CRISPR J. 5 (1), 123-130 (2022).
- Lazzarotto, C. R., et al. Defining CRISPR-Cas9 genome-wide nuclease activities with CIRCLE-seq. Nat Protoc. 13 (11), 2615-2642 (2018).
- Butterfield, K. T., McGrath, P. S., Han, C. M., Kogut, I., Bilousova, G. Generation of an induced pluripotent stem cell line with the constitutive EGFP reporter. Methods Mol Biol. 2155, 11-21 (2020).
- Hayashi, H., Kubo, Y., Izumida, M., Matsuyama, T. Efficient viral delivery of Cas9 into human safe harbor. Sci Rep. 10 (1), 21474 (2020).
- Kogut, I., et al. High-efficiency RNA-based reprogramming of human primary fibroblasts. Nat Commun. 9 (1), 745 (2018).
- Kariko, K., Buckstein, M., Ni, H., Weissman, D. Suppression of RNA recognition by Toll-like receptors: The impact of nucleoside modification and the evolutionary origin of RNA. Immunity. 23 (2), 165-175 (2005).
- Bao, X. R., Pan, Y., Lee, C. M., Davis, T. H., Bao, G. Tools for experimental and computational analyses of off-target editing by programmable nucleases. Nat Protoc. 16 (1), 10-26 (2021).
- Cromer, M. K., et al. Comparative analysis of CRISPR off-target discovery tools following ex vivo editing of CD34(+) hematopoietic stem and progenitor cells. Mol Ther. 31 (4), 1074-1087 (2023).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。