Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Laserfreies Hydroxylradikalprotein-Footprinting zur Durchführung einer Strukturanalyse höherer Ordnung von Proteinen
In diesem Artikel
Zusammenfassung
Dieses Protokoll stellt eine Methode zur Verwendung von Inline-Radikaldosimetrie und einer Plasmalichtquelle vor, um Flash-Oxidationsprotein-Footprinting durchzuführen. Diese Methode ersetzt den gefährlichen UV-Laser, um die Reproduzierbarkeit der schnellen photochemischen Oxidation von Proteinstudien zu vereinfachen und zu verbessern.
Zusammenfassung
Hydroxyl Radical Protein Footprinting (HRPF) ist eine aufstrebende und vielversprechende Strukturanalysetechnik höherer Ordnung, die Informationen über Veränderungen der Proteinstruktur, Protein-Protein-Interaktionen oder Protein-Liganden-Interaktionen liefert. HRPF verwendet Hydroxylradikale(▪OH), um die lösungsmittelzugängliche Oberfläche eines Proteins irreversibel zu kennzeichnen. Die inhärente Komplexität, die Kosten und die Gefährlichkeit der HrPF-Durchführung haben die breite Akzeptanz in der Biopharmazie erheblich eingeschränkt. Zu diesen Faktoren gehören: 1) die Verwendung komplizierter, gefährlicher und teurer Laser, die erhebliche Sicherheitsvorkehrungen erfordern; und 2) die Irreproduzierbarkeit von HRPF, die durch Hintergrundabfangen von ▪OH verursacht wird, die vergleichende Studien einschränken. Diese Veröffentlichung enthält ein Protokoll für den Betrieb eines laserfreien HRPF-Systems. Dieses laserfreie HRPF-System nutzt eine hochenergetische Hochdruck-Plasma-Lichtquellen-Blitzoxidationstechnologie mit Inline-Radikaldosimetrie. Die Plasmalichtquelle ist sicherer, einfacher zu bedienen und effizienter bei der Erzeugung von Hydroxylradikalen als laserbasierte HRPF-Systeme, und das Inline-Radikaldosimeter erhöht die Reproduzierbarkeit von Studien. In Kombination adressiert und überwindet das laserfreie HRPF-System die genannten Mängel und Grenzen laserbasierter Techniken.
Einleitung
Proteinkonformation und die damit verbundene Struktur höherer Ordnung (HOS) sind die Hauptdeterminanten der richtigen biologischen Funktion und des abweichenden Verhaltens1. Gleiches gilt für Biopharmazeutika, deren Struktur und funktionelle Aktivität von verschiedenen Aspekten ihrer Produktion und Umgebung abhängt. Biopharmazeutische Veränderungen in HOS wurden mit unerwünschten Arzneimittelwirkungen (ADR) in Verbindung gebracht, die auf unerwünschte Pharmakologie und immunologisches Ansprechen des Patienten zurückzuführen sind2,3. Das Auftreten von ADRs hat die biopharmazeutische Industrie auf die entscheidende Rolle aufmerksam gemacht, die protein HOS für die Sicherheit und Wirksamkeit von Biotherapeutika spielt, und sie haben den Bedarf an neuen und verbesserten HOS-Analysen festgestellt4.
Hydroxyl Radical Protein Footprinting (HRPF) ist eine vielversprechende Technik, um die Veränderung des Proteins HOS zu verfolgen. HRPF beinhaltet die irreversible Markierung der Außenseite eines Proteins mit ▪OH, gefolgt von einer Massenspektrometrie (MS) -Analyse, um die lösungsmittelzugängliche Oberfläche des Proteins5,6,7zu identifizieren . HRPF wurde erfolgreich eingesetzt, um Defekte im Protein HOS und seine Funktion8,9, charakterisieren die HOS von monoklonalen Antikörpern (mAb)10,11,12,13, bestimmen die Bindung Kd eines Liganden14und vieles mehr15,16,17,18,19. Eine gängige Methode zur Erzeugung der ▪OH für HRPF ist die schnelle photochemische Oxidation von Proteinen (FPOP), bei der hochenergetische, schnelle UV-Laser verwendet werden,um ▪ OH aus der Photolyse vonH2O2herzustellen. In den meisten Fällen verwendet FPOP teure Excimer-Laser mit gefährlichem Gas (KrF), die erhebliche Sicherheitsvorkehrungen erfordern, um Atem- und Augenverletzungen zu vermeiden20. Um Inhalationsgefahren zu vermeiden, haben andere Frequenzvervierfachte Neodym-Yttrium-Aluminium-Granat-Laser (Nd: YAG)21verwendet, die den Einsatz von giftigen Gasen eliminieren, aber immer noch teuer sind, erhebliche Betriebskenntnisse erfordern und umfangreiche Streulichtsteuerungen erfordern, um Benutzer vor Augenverletzungen zu schützen.
Obwohl mit HRPF umfangreiche Informationen gewonnen werden können, wurde eine breite Akzeptanz in der Biopharmazie nicht erreicht. Zwei Hindernisse für die begrenzte HRPF-Einführung sind: 1) die Verwendung gefährlicher und teurer Laser, die erhebliche Sicherheitsvorkehrungen erfordern20; und 2) die Unreproduzierbarkeit von HRPF, die durch Hintergrundabfangen von ▪OH verursacht wird, die vergleichende Studieneinschränken 22. Um den Lasereinsatz zu ersetzen, wurde eine Hochgeschwindigkeits-Plasmablitz-Photolyseeinheit mit hoher Energie entwickelt, um FPOP auf einfache Weise sicher durchzuführen. Um die Irreproduzierbarkeit von HRPF-Experimenten zu verbessern, wird eine Echtzeit-Radikaldosimetrie implementiert.
Die Praxis der HRPF wurde durch die Irreproduzierbarkeit eingeschränkt, die auf die Hintergrundabfängerung von ▪OH22zurückzuführen ist. Während ▪OH ausgezeichnete Sonden der Proteintopographie sind, reagieren sie auch mit vielen Bestandteilen, die in Präparaten gefunden werden, so dass es notwendig ist, die effektive Konzentration von Radikalen zu messen, die zur Oxidation eines Zielproteins zur Verfügung stehen. Variationen in der Puffervorbereitung, Wasserstoffperoxidkonzentration, Ligandeneigenschaften oder Photolyse können zu Oxidationsunterschieden zwischen Kontroll- und Experimentellengruppen führen, die in HOS-Differentialstudien zu Mehrdeutigkeiten führen. Die Hinzufügung der Echtzeit-Radikaldosimetrie ermöglicht die Anpassung des Effekts ▪OH-Last und erhöht somit die Zuverlässigkeit und Reproduzierbarkeit während eines HRPF-Experiments. Die Verwendung der Radikaldosimetrie in FPOP wurde an anderer Stelle23,24,25beschrieben und in einer kürzlich erschienenen Veröffentlichung26ausführlich diskutiert. Hier beschreiben wir die Verwendung eines neuartigen Flash-Photolysesystems und einer Echtzeitdosimetrie zur Markierung von Apo-Myoglobin (aMb) von Pferden und vergleichen die Niveaus der Peptidoxidation in einem FPOP-Experiment mit denen, die bei verwendung eines Excimer-Lasers erhalten wurden.
Protokoll
1. Einbau des Kapillarrohrs
- Mit einem Kieselsäurespaltenstein 250 μm Innendurchmesser (ID) Silica-Kapillare auf 27 Zoll spalten. Überprüfen Sie die Kapillarenden auf einen sauberen, geraden Schnitt.
- Erstellen Sie zwei Fenster mit einer Länge von etwa 15 mm, indem Sie die Polyimidbeschichtung verbrennen. Ausgehend vom "unteren Ende" ist das erste Photolysefenster 90 mm vom "unteren Ende" und das zweite Dosimeterfenster 225 mm vom "unteren Ende" entfernt.
HINWEIS: Sobald die Beschichtung weggebrannt ist, ist die Kapillare sehr zerbrechlich. - Schrauben Sie die Mutter und die Ferrule an Port 5 ab und setzen Sie das "untere Ende" der Kapillare direkt hinter dem konischen Ende der Ferrule ein (Abbildung 1A).
- Entfernen Sie auf dem Photolysemodul die Photolyse-Zellkappe, indem Sie sie gerade herausziehen, und entfernen Sie dann die magnetisch montierte Metallmaske, die die Kapillare an Ort und Stelle hält.
ACHTUNG: Im Inneren der Photolyse-Zellkappe befindet sich ein gekrümmter Spiegel, lassen Sie nichts den Spiegel berühren. - Öffnen Sie auf dem Dosimetermodul die Dosimeterzelle, indem Sie auf die Lasche auf der linken Seite drücken und die Dosimeterzelle nach rechts öffnen. Entfernen Sie die magnetisch angebrachten Clips, die die Kapillare an Ort und Stelle halten.
- Legen Sie die Kapillare in den gerillten Kanal in der Basis der Photolysezelle. Zentrieren Sie das Kapillarfenster mit dem Photolysezellenfenster. Während Sie die Kapillare mit einer Hand in Position halten, fügen Sie die magnetische Maske hinzu, um die Kapillare an Ort und Stelle zu halten. Setzen Sie die Photolysekappe wieder in Position.
- Legen Sie die Kapillare in den gerillten Kanal in der Basis der Dosimeterzelle. Zentrieren Sie das zweite Kapillarfenster auf die kleine Öffnung in der Mitte der Dosimeter-Zellbasis. Während Sie die Kapillare mit einer Hand in Position halten, platzieren Sie die beiden magnetischen Clips in Position, um die Kapillare an Ort und Stelle zu halten. Schließen Sie die Dosimeterzelle, bis sie auf Geschlossen klickt.
- Führen Sie die Kapillare durch die Rändelmutter auf den Kapillarlift des Produktkollektors ein (Abbildung 1B). Verlängern Sie die Kapillare bis knapp über den Boden der Durchstechflasche.
HINWEIS: Es ist wichtig, dass die Kapillare den Boden der Durchstechflasche erreicht, um sicherzustellen, dass die Kapillare während eines Experiments in die Abschrecklösung eingetaucht ist.
2. Einbau einer Injektionsschleife
- Verwenden Sie Teflonschläuche mit einem Außendurchmesser von 1/16" und einem Innendurchmesser von 0,015" (381 μm). Folgen Sie Gleichung 1, um die Länge des Schlauches zu berechnen, der für das gewünschte Volumen benötigt wird, indem Sie Gleichung 1 verwenden.
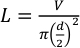
Dabei ist L die Länge des Schlauches in Millimetern, V das gewünschte Volumen in Mikrolitern und d der Rohrinnendurchmesser in Millimetern. Für ein Injektionsvolumen von 25 μL und einen Innendurchmesser von 381 μm muss die Schlauchlänge ca. 219,3 mm beträgen.
HINWEIS: Für Volumina unter 20 μL verwenden Sie PTFE-Schläuche mit einem Innendurchmesser von 0,01". - Schneiden Sie den Teflonschlauch mit einem Rohrschneider auf die erforderliche Länge. Überprüfen Sie die Enden des Rohres auf einen sauberen, geraden Schnitt.
- Setzen Sie ein Ende der neuen Injektionsschlaufe durch eine der Muttern ein und legen Sie eine neue Ferrule auf das Ende des Rohrs. Halten Sie die Ferrule und die Mutter an Ort und Stelle und führen Sie das Rohr in Port 3 des Einspritzventils ein, bis es im Ventil auf den Boden fällt. Halten Sie das Röhrchen fest und ziehen Sie den Mutterfinger fest. Ziehen Sie die Mutter mit einem Schraubenschlüssel 1/4 Umdrehung weiter an. Entfernen und überprüfen Sie die Baugruppe.
HINWEIS: Wenn die Ferrule nicht in Position fixiert ist, installieren Sie sie neu und ziehen Sie sie 1/8 weiter an. Wiederholen Sie den Vorgang mit dem anderen Ende der Schleife. - Sobald beide Enden eine Mutter und eine feste Ferrule haben, schrauben Sie ein Ende lose an Port 3 und das andere Ende an Port 6 (Abbildung 1C). Sobald Sie in Position sind, ziehen Sie beide Seiten fest, um den Finger fest zu machen, dann drehen Sie sich 1/4 mit einem Schraubenschlüssel am Finger vorbei.
3. Initialisieren Sie das Photolysesystem
- Schalten Sie die Photolysemodule in der folgenden Reihenfolge ein: (1) Fluidikmodul (2) Photolysemodul (3) Dosimetriemodul (4) Produktkollektor und schließlich (5) den Systemcomputer und starten Sie die Steuerungssoftware
HINWEIS: Lassen Sie dem Dosimeter-Modul mindestens eine halbe Stunde Zeit, um sich von einem Kaltstart aufzuwärmen. - Den Schlauch vollständig von der Position "Ventil" an der Spritzenpumpe (Abbildung 1D) in 10 ml Puffer tauchen. Legen Sie den Schlauch aus der Position "Abfall" (Abbildung 1D) und den Schlauch von Port 2 auf den Spritzenanschluss (Abbildung 1C) in einen leeren Behälter, um Abfälle zu sammeln.
HINWEIS: Verwenden Sie den Puffer, in dem Ihr Protein suspendiert wird. Ein empfohlener Puffer ist 10 mM NaPO4. - Legen Sie auf dem Produktkollektorkarussell 1,5 ml Mikrozentrifugenröhrchen an die mit H und 6 gekennzeichnete Position.
- Wenn Sie eine 250 μm ID-Kapillare verwenden, stellen Sie den Lastdurchfluss auf 500 μL/min, den Verarbeitungsdurchfluss auf 7,5 μL/min und den Abfalldurchfluss auf 500 μL/min ein.
- Berechnen Sie die Blitzverzögerung, die Produktzeit und die Verschwendungszeit für die Verwendung des halbautomatischen Modus mit Gleichung 2.
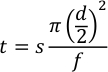
Dabei ist t die Zeit in Minuten, s der Kapillarabstand in Millimetern, d der Innendurchmesser der Kapillare in Millimetern und f die Durchflussrate in μL/min.- Für die Blitzverzögerung beträgt der Abstand vom "unteren Ende" der Kapillare zum ersten Fenster, das etwa 90 mm betragen sollte. Mit 250 μm ID-Kapillare und einer Verarbeitungsdurchflussrate von 7,5 μL/min gelangt die Probe in etwa 35 Sekunden zum Photolysefenster. Lassen Sie zwei Blitze bei 1 Hz auftreten, bevor die Samples ankommen, also stellen Sie die Blitzverzögerung auf 33 Sekunden ein.
- Geben Sie für die Produktzeit die Zeit ein, die die injizierte Lösung benötigen wird, um vom Injektionsventil zum Ende der Kapillare zu fließen. Für eine 27" lange 250 μm ID-Kapillare stellen Sie die Produktzeit auf 4,5 Minuten ein.
- Geben Sie für die Verschwendungszeit die Zeit ein, die das Gesamtvolumen der injizierten Lösung benötigen wird, um gesammelt zu werden. Zu diesem Zeitpunkt wechselt der Produktsammler von der Produktposition zur Abfalldurchstechflasche. Bei einem Injektionsvolumen von 25 μL und einem Durchfluss von 7,5 μL/min wird die Verschwendungszeit auf 7,8 Minuten eingestellt.
- Spülen Sie die Injektionsschleife fünfmal aus, indem Sie 25 μL HPLC-Wasser in den Injektionsanschluss injizieren, wobei das Einspritzventil auf die Lastposition eingestellt ist.
- Drehen Sie das Einspritzventil manuell in die Einspritzposition, um den Rest des Systems zu spülen. Wählen Sie in der Steuerungssoftware Prozess (Aus), um mit dem Fließen von Wasser zu beginnen. Heben Sie während des Fließens den Kapillarlift des Produktkollektors an, indem Sie unter dem Produktkollektor Nach oben auswählen, damit Sie das Ende der Kapillare sehen können. Fließen Sie Wasser durch die Kapillare, bis sich ein Tröpfchen bildet.
HINWEIS: Wenn sich kein Tröpfchen bildet, überprüfen Sie das Injektorventil auf Lecks. Wenn es ein Leck gibt, lösen Sie die Mutter, setzen Sie die Kapillare wieder ein und schrauben Sie sie wieder an.
4. Bestimmen Sie die tatsächliche ▪OH-Ausbeute, um radikalische Aufrascheffekte aus dem Puffer zu testen.
- Starten Sie in der Steuerungssoftware den Ablauf, indem Sie Prozess (Aus) auswählen. Stellen Sie auf der Registerkarte Einstellungen die Blitzspannung auf 0 V ein. Wählen Sie auf der Registerkarte Manuelle Steuerung im Abschnitt Dosimeterdaten die Option Daten starten + AutoZeroaus.
- Wählen Sie die Position für die Produktfläschchen (H) und die Abfallfläschchen (6).
- Wählen Sie Bereit,drehen Sie das Einspritzventil manuell in die Lastposition herunter und injizieren Sie 25 μL 1 mM Adenin mit 100 mMH2O2 in den Einspritzanschluss. Nach der Injektion drehen Sie das Einspritzventil manuell in die Einspritzposition.
HINWEIS: Dadurch wird das System automatisch veranlasst, den Durchfluss zu starten, die Plasmaquelle einzuschalten und die Dosimetriedaten zu erfassen. - Fahren Sie die Spannung hoch, indem Sie zur Registerkarte Einstellungen navigieren und die Blitzspannung ändern. Wiederholen Sie die Schritte 4.2 und 4.3 mit 500 V, 750 V, 1000 V und 1250 V. Führen Sie das Ablesen der Adeninabsorption bei jeder Spannung in Triplikaten durch.
- Wählen Sie die Registerkarte Berechnungen, um die durchschnittliche Absorption jeder Probe zu bestimmen. Klicken Sie zunächst auf Auswählen, und wählen Sie dann manuell den Anfang und das Ende der Spitzenabsorption aus. Geben Sie im verfügbaren Bereich eine Beschreibung des Beispiels ein. Wiederholen Sie diesen Vorgang für alle erfassten Daten.
HINWEIS: Blasen können sich bilden, die zu einem Anstieg des Dosimeterwerts führen. Wenn Sie Daten auswählen, um die durchschnittliche Absorption zu bestimmen, lassen Sie Bereiche mit Blasen weg. - Kopieren und einfügen Sie Daten in Excel, um die durchschnittliche Änderung der Adeninabsorption für jede Spannung zu berechnen und so die effektive ▪OH-Ausbeute zu bestimmen.
- Wiederholen Sie die Schritte 4.1-4.6, wenn mehrere Probenbedingungen (unterschiedliche Puffer/Additive) verwendet werden, um die effektive ▪OH-Ausbeute für jede Bedingung zu normalisieren.
5. Modifikation des Proteins, um Veränderungen in der Struktur höherer Ordnung zu erkennen.
- Mischen Sie 4 mM Adenin mit 20 μM Protein in einem Eins-zu-Eins-Verhältnis, um eine Lösung mit 2 mM Adenin und 10 μM Protein herzustellen.
- Machen Sie die Abschrecklösung mit 0,3 mg / ml Katalase, um überschüssiges Wasserstoffperoxid abzubauen, und 35 mM Methioninamid, um verbleibende Reste abzuschrecken. Aliquot 25 μL Abschrecklösung in eine 200 μL Mikroröhrchen, so dass ein gleiches Volumen an Abschreckung und das markierte Produkt gemischt wird.
- H2O2auf200 mM verdünnen und auf Eis halten.
- Starten Sie auf der Registerkarte "Steuerungssoftware" die Blitzspannung bei 0 V, um eine Hintergrundoxidation zu bestimmen.
- Wählen Sie auf der Registerkarte Manuelle Steuerung die Option Startdaten + AutoZero, gefolgt von Prozess (Aus),dann Bereit, und drehen Sie schließlich das Einspritzventil auf die Lastposition herunter.
- Legen Sie eine Abschrecklösung in Position 1 auf das Produktsammelkarussell. Ändern Sie in der Systemsteuerungssoftware die Produktfläschchen in 1.
- Unmittelbar vor der Injektion werden 12,5 μL der Adenin- und Proteinmischung mit 12,5 μLH2O2gemischt, um eine Endkonzentration von 1 mM Adenin, 5 μM Protein und 100 mMH2O2zu erzielen. Zum Mischen vorsichtig auf und ab pipetten, schnell herunterdrehen und innerhalb von 10 Sekunden nach dem Mischen 25 μL mit dem Injektionsanschluss injizieren.
- Schalten Sie das Einspritzventil in die Injektionsposition und warten Sie, während die Probe verarbeitet wird.
- Wiederholen Sie die Erfassung mit 500 V, 750 V, 1000 V und 1250 V. Führen Sie jede Spannungsmessung in dreifacher Ausführung durch.
- Berechnen Sie die durchschnittliche Absorption wie in Schritt 4.5 beschrieben. Kopieren Sie alle Daten und fügen Sie sie in Excel ein.
6. Fahren Sie das System herunter
- Nachdem alle Proben entnommen wurden, spülen Sie den Spritzenanschluss und die Probenschleife aus, indem Sie das Injektionsventil auf die Lastposition herunterstellen und fünfmal 25 μL HPLC-Wasser einspritzen.
- Drehen Sie das Einspritzventil in die Einspritzposition, um den Rest des Systems mit HPLC-Wasser zu spülen.
- Stoppen Sie den Durchfluss, schließen Sie die Systemsteuerungssoftware und schalten Sie die Module aus, beginnend mit dem Produktkollektor, dem Dosimetermodul, dem Photolysemodul und schließlich dem Fluidikmodul.
7. Probenvorbereitung und Flüssigkeitschromatographie-Massenspektrometrie
- Denaturieren Sie das Protein, indem Sie Proben bei 80 °C für 20 min in Gegenwart von 50 mM Tris und 1 mM CaCl2inkubieren. Kühlen Sie die Proben auf Raumtemperatur ab und fügen Sie 1:20 Trypsin zu Protein hinzu. Verdauen Sie das Protein über Nacht bei 37 °C mit Probenmischung. Am nächsten Morgen beenden Sie den Trypsinaufschluss, indem Sie die Proben 10 Minuten lang auf 95 °C erhitzen.
- Detektieren Sie Peptide mit einem hochauflösenden LC-MS/MS-System, das mit einem UPLC-System verbunden ist.
- Die Probe zuerst auf die Fallensäule laden (300 μm ID X 5 mm 100 Å Porengröße, 5 μm Partikelgröße) und bei 5,0 μL/ml für 3 min mit Wasser gewaschen, das 2% Lösungsmittel B (Acetonitril und 0,1% Ameisensäure) enthält.
- Trennen Sie die Peptide auf einer C18-Nanokolumne (0,75 mm x 150 mm, 2 μm Partikelgröße, 100 Å Porengröße) bei einer Durchflussrate von 300 nL/min mit einem Gradienten zwischen Lösungsmittel A (Wasser mit 0,1% Ameisensäure) und Lösungsmittel B. Der Gradient für die Pepteidelution besteht aus einem linearen Anstieg von 2 auf 35% B über 22 min, der über 5 min auf 95% B hochgefahren und für 3 min gehalten wird, um die Säule zu waschen, und dann über 3 min auf 2% B zurückgeführt und für 9 min gehalten wird, um die Säule wieder auszugleichen.
- Erfassen Sie die Daten im positiven Ionenmodus. Stellen Sie die Sprühspannung auf 2400 V und die Temperatur des Ionenübertragungsrohrs auf 300 °C ein.
- Erfassen Sie die vollständigen MS-Scans von 250-2000 m / z, gefolgt von acht nachfolgenden datenabhängigen MS / MS-Scans auf den acht häufigsten Peptidionen. Verwenden Sie kollisionsinduzierte Dissoziation bei 35% normalisierter Energie, um die Peptide zu fragmentieren.
- Identifizieren Sie alle unveränderten Peptide, die aus der MS/MS-Analyse mit einer verfügbaren Proteinanalysesoftware nachgewiesen wurden, anhand der erforderlichen FASTA-Datei, die die Proteinsequenz und das relevante proteolytische Enzym enthält.
- Suchen und quantifizieren Sie modifizierte Peptide mit einer HRPF-Datenverarbeitungssoftware. Das Ausmaß der Oxidation für jedes identifizierte Peptid wird berechnet, indem die summierte chromatographische Peakfläche eines modifizierten Peptids durch die gesamte chromatographische Peakfläche dieses Peptids dividiert wird, die mit Gleichung 3 modifiziert und unverändert wurde.
P = [I(einzeln oxidiert) X 1 + I (doppelt oxidiert) X 2 + I(dreifach oxidiert) X 3 + .../[Iunoxidiert + I(einzeln oxidiert) + I(doppelt oxidiert) + I(dreifach oxidiert) ...]
wobei P die durchschnittliche Anzahl von Oxidationsereignissen pro Peptidmolekül bezeichnet und I die Spitzenfläche des unoxidierten Peptids (Iunoxidiert) und des Peptids mit n Oxidationsereignissen darstellt.
8. Für eine Differenzstudie wiederholen Sie die Schritte 5-7 unter der zweiten Bedingung.
HINWEIS: Um die Reproduzierbarkeit zu bestätigen, werden zwei biologische Replikate zusätzlich zu technischen Triplikaten für jede Bedingung empfohlen.
Ergebnisse
Die Hochdruck-Plasmaquelle in Verbindung mit Echtzeit-Dosimetrie ermöglicht eine bessere Kontrolle ▪OH-Ausbeute, um Veränderungen in der Proteinstruktur höherer Ordnung genauer zu beobachten. Die Zugabe von Adenin ermöglicht ein effektives Echtzeit-Radikaldosimeter. Bei der Oxidation verliert Adenin die UV-Absorption bei 265 nm (Abbildung 2A). Die Änderung der Adeninabsorption steht in direktem Zusammenhang mit der Konzentration der für HRPF verfügbaren Radikale und biete...
Diskussion
Es gibt mehrere kritische Schritte, um die korrekte Markierung von Proteinen während eines HRPF-Experiments sicherzustellen. Zunächst werden eine geeignete Durchflussrate und eine geeignete Quellblitzrate ausgewählt, um sicherzustellen, dass jeder Bolus der Probe einmal bestrahlt wird. Dies stellt sicher, dass das Protein einem einzigen Bolus neu gebildeter ▪OH ausgesetzt wird. Sobald ein Protein oxidiert ist, kann die Proteinstruktur höherer Ordnung verändert werden. Um sicher zu sein, dass die native ...
Offenlegungen
E.E.C., J.S.S. und S.R.W. legen eine bedeutende finanzielle Beteiligung an GenNext Technologies, Inc. bekannt, einem Unternehmen in der Frühphase, das Technologien für die Proteinstrukturanalyse höherer Ordnung kommerzialisieren möchte. Die zur Verfügung gestellten repräsentativen Daten wurden von S.K.M. überprüft, die keinen finanziellen Interessenkonflikt hat.
Danksagungen
Diese Arbeit wurde vom National Institute of General Medical Sciences (R43GM125420 und R44GM125420) finanziert.
Materialien
| Name | Company | Catalog Number | Comments |
| 15 mL Conical Centrifuge Tubes | Fisher Scientific | 14-959-53A | any brand is sufficient |
| 50 µL SGE Gastight Syringes | Fisher Scientific | SG-00723 | |
| Acclaim PepMap 100 C18 nanocolumn (0.75 mm X 150 mm, 2 µm) | Thermo Scientific | ||
| Acetonitrile with 0.1% Formic Acid (v/v), LC/MS Grade | Fisher Scientific | LS120-500 | |
| Apomyoglobin | Sigma-Aldrich | ||
| Catalase | Sigma-Aldrich | C9322 | |
| Centrifuge | Eppendorf | 022625501 | |
| Delicate Task Wipers | Fisher Scientific | 06-666A | |
| Hydrogen Peroxide | Fisher Scientific | H325-100 | any 30% hydrogen peroxide is sufficient |
| Methionine amide | Chem-Impex | 03109 | |
| Microcentrifuge | Thermo Scientific | 75002436 | |
| Orbitrap Fusion Lumos Tribrid Mass Spectrometer | Thermo Scientific | Orbitrap Fusion Lumos Tribrid Mass Spectrometer | other high resolution instruments (e.g. Q exactive Orbitrap or Orbitrap Fusion) can be used |
| Pierce Trypsin Protease, MS Grade | Thermo Scientific | 90058 | |
| Polymicro Cleaving Stone, 1" x 1" x 1/32” | Molex | 1068680064 | any capillary tubing cutter is sufficient |
| UPLC | Thermo Scientific | ||
| Water with 0.1% Formic Acid (v/v), LC/MS Grade | Fisher Scientific | LS118-500 | |
| Water, LC/MS Grade | Fisher Scientific | W6-4 |
Referenzen
- Nagarkar, R. P., Murphy, B. M., Yu, X., Manning, M. C., Al-Azzam, W. A. Characterization of protein higher order structure using vibrational circular dichroism spectroscopy. Current Pharmaceutical Biotechnology. 14 (2), 199-208 (2013).
- Giezen, T. J., Schneider, C. K. Safety assessment of biosimilars in Europe: a regulatory perspective. Generics and Biosimilars Initiative Journal. , 1-8 (2014).
- Giezen, T. J., Mantel-Teeuwisse, A. K., Strauss, S. Safety-related regulatory actions for biologicals approved in the United States and the Europena Union. Journal of the American Medical Society. 300 (16), 1887-1896 (2008).
- Gabrielson, J. P., Weiss, W. F. Technical decision-making with higher order structure data: starting a new dialogue. Journal of Pharmaceutical Sciences. 104 (1), 1240-1245 (2015).
- Brenowitz, M., Erie, D. A., Chance, M. R. Catching RNA polymerase in the act of binding: intermediates in transcription illuminated by synchrotron footprinting. Proceedings of the National Academy of Sciences U S A. 102 (13), 4659-4660 (2005).
- Guan, J. Q., Takamoto, K., Almo, S. C., Reisler, E., Chance, M. R. Structure and dynamics of the actin filament. Biochemistry. 44 (9), 3166-3175 (2005).
- Hambly, D. M., Gross, M. L. Laser flash photochemical oxidataion to locate heme binding and conformataional changes in myoglobin. International Journal of Mass Spectrometry. 259 (2007), 124-129 (2007).
- Li, K. S., Shi, L., Gross, M. L. Mass Spectrometry-Based Fast Photochemical Oxidation of Proteins (FPOP) for Higher Order Structure Characterization. Accounts of Chemical Research. 51 (3), 736-744 (2018).
- Watson, C., Sharp, J. S. Conformational Analysis of Therapeutic Proteins by Hydroxyl Radical Protein Footprinting. American Association of Pharmaceutical Scientists Journal. 14 (2), 206-217 (2012).
- Deperalta, G., et al. Structural analysis of a therapeutic monoclonal antibody dimer by hydroxyl radical footprinting. mAbs. 5 (1), 86-101 (2013).
- Jones, L. M., et al. Complementary MS methods assist conformational characterization of antibodies with altered S-S bonding networks. Journal of American Society of Mass Spectrometry. 24 (6), 835-845 (2013).
- Storek, K. M., et al. Monoclonal antibody targeting the β-barrel assembly machine of Escherichia coli is bactericidal. Proceedings of the National Academy of Sciences. , (2018).
- Vij, R., et al. A targeted boost-and-sort immunization strategy using Escherichia coli BamA identifies rare growth inhibitory antibodies. Scientific Reports. 8 (1), 7136 (2018).
- Liu, X. R., Zhang, M. M., Rempel, D. L., Gross, M. L. A Single Approach Reveals the Composite Conformational Changes, Order of Binding, and Affinities for Calcium Binding to Calmodulin. Analytical Chemistry. 91 (9), 5508-5512 (2019).
- Lu, Y., et al. Fast Photochemical Oxidation of Proteins Maps the Topology of Intrinsic Membrane Proteins: Light-Harvesting Complex 2 in a Nanodisc. Analytical Chemistry. 88 (17), 8827-8834 (2016).
- Marty, M., Zhang, H., Cui, W., Gross, M., Sligar, S. Interpretation and Deconvolution of Nanodisc Native Mass Spectra. Journal of American Society of Mass Spectrometry. 25, (2013).
- Johnson, D. T., Di Stefano, L. H., Jones, L. M. Fast photochemical oxidation of proteins(FPOP): A powerful mass spectrometry based structural proteomics tool. Journal of Biological Chemistry. , (2019).
- Chea, E. E., Jones, L. M. Analyzing the structure of macromolecules in their native cellular environment using hydroxyl radical footprinting. Analyst. 143 (4), 798-807 (2018).
- Aprahamian, M. L., Chea, E. E., Jones, L. M., Lindert, S. Rosetta Protein Structure Prediction from Hydroxyl Radical Protein Footprinting Mass Spectrometry Data. Analytical chemistry. 90 (12), 7721-7729 (2018).
- Linde. . Linde Specialty Gases of North America. , (2009).
- Aye, T. T., Low, T. Y., Sze, S. K. Nanosecond laser-induced photochemical oxidation method for protein surface mapping with mass spectrometry. Analytical Chemistry. 77 (18), 5814-5822 (2005).
- Niu, B., Zhang, H., Giblin, D., Rempel, D. L., Gross, M. L. Dosimetry determines the initial OH radical concentration in fast photochemical oxidation of proteins (FPOP). Journal of American Society of Mass Spectrometry. 26 (5), 843-846 (2015).
- Misra, S. K., Orlando, R., Weinberger, S. R., Sharp, J. S. Compensated Hydroxyl Radical Protein Footprinting Measures Buffer and Excipient Effects on Conformation and Aggregation in an Adalimumab Biosimilar. American Association of Pharmaceutical Scientists Journal. 21 (5), 87 (2019).
- Olson, L. J., Misra, S. K., Ishihara, M., Battaile, K. P., Grant, O. C., Sood, A., Woods, R. J., Kim, J. P., Tiemeyer, M., Ren, G., Sharp, J. S., Dahms, N. M. Allosteric regulation of lysosomal enzyme recognition by the cation-independent mannose 6-phosphate receptor. Communications Biology. 3 (1), 498 (2020).
- Sharp, J. S., Misra, S. K., Persoff, J. J., Egan, R. W., Weinberger, S. R. Real Time Normalization of Fast Photochemical Oxidation of Proteins Experiments by Inline Adenine Radical Dosimetry. Analytical Chemistry. 90 (21), 12625-12630 (2018).
- Misra, S. K., Sharp, J. S. Enabling Real-Time Compensation in Fast Photochemical Oxidations of Proteins for the Determination of Protein Topography Changes. Journal of Visualized Experiments. 163, (2020).
- Hambly, D. M., Gross, M. L. Laser flash photolysis of hydrogen peroxide to oxidize protein solvent-accessible residues on the microsecond timescale. Journal of American Society of Mass Spectrometry. 16 (12), 2057-2063 (2005).
- Liu, X. R., Zhang, M. M., Gross, M. L. Mass Spectrometry-Based Protein Footprinting for Higher-Order Structure Analysis: Fundamentals and Applications. Chemical Reviews. 120 (10), 4355 (2020).
- Jones, L. M., Sperry, B. J., Carroll, A. J., Gross, M. L. Fast photochemical oxidation of proteins for epitope mapping. Analytical chemistry. 83 (20), 7657-7661 (2011).
- Li, J., et al. Mapping the Energetic Epitope of an Antibody/Interleukin-23 Interaction with Hydrogen/Deuterium Exchange, Fast Photochemical Oxidation of Proteins Mass Spectrometry, and Alanine Shave Mutagenesis. Analytical chemistry. 89 (4), 2250-2258 (2017).
- Liu, X. R., Zhang, M. M., Rempel, D. L., Gross, M. L. A Single Approach Reveals the Composite Conformational Changes, Order of Binding, and Affinities for Calcium Binding to Calmodulin. Analytical Chemistry. 91 (9), 5508-5512 (2019).
- Kiselar, J. G., Janmey, P. A., Almo, S. C., Chance, M. R. Structural analysis of gelsolin using synchrotron protein footprinting. Molecular and Cellular Proteomics. 2 (10), 1120-1132 (2003).
- Chea, E. E., Deredge, D. J., Jones, L. M. Insights on the Conformational Ensemble of Cyt C Reveal a Compact State during Peroxidase Activity. Biophysical Journal. 118 (1), 128-137 (2020).
- Poor, T. A., et al. Probing the paramyxovirus fusion (F) protein-refolding event from pre- to postfusion by oxidative footprinting. Proceedings of the National Academy of Sciences U S A. 111 (25), 2596-2605 (2014).
- Roush, A. E., Riaz, M., Misra, S. K., Weinberger, S. R., Sharp, J. S. Intrinsic Buffer Hydroxyl Radical Dosimetry Using Tris(hydroxymethyl)aminomethane. Journal of American Society of Mass Spectrometry. 31 (2), 169-172 (2020).
- Everett, E. A., Falick, A. M., Reich, N. O. Identification of a critical cysteine in EcoRI DNA methyltransferase by mass spectrometry. Journal of Biological Chemistry. 265 (29), 17713-17719 (1990).
- Sanderson, R. J., Mosbaugh, D. W. Identification of specific carboxyl groups on uracil-DNA glycosylase inhibitor protein that are required for activity. Journal of Biological Chemistry. 271 (46), 29170-29181 (1996).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten