In vitro On-Chip-Modell eines fötalen menschlichen Gefäßes zur Untersuchung der Entwicklungsmechanobiologie
In diesem Artikel
Zusammenfassung
Hier wird ein einfacher Arbeitsablauf zur Unterscheidung von Endothelzellen von humanen pluripotenten Stammzellen beschrieben, gefolgt von einem detaillierten Protokoll für ihre mechanische Stimulation. Dies ermöglicht die Untersuchung der Entwicklungsmechanobiologie von Endothelzellen. Dieser Ansatz ist kompatibel mit nachgeschalteten Assays von lebenden Zellen, die nach mechanischer Stimulation aus dem Kulturchip entnommen wurden.
Zusammenfassung
Das Herz ist das erste Organ, das während der Entwicklung funktionell aufgebaut wird und so schon sehr früh in der Schwangerschaft den Blutkreislauf in Gang setzt. Neben dem Transport von Sauerstoff und Nährstoffen, um das Wachstum des Fötus zu gewährleisten, steuert die fetale Zirkulation viele wichtige Entwicklungsereignisse, die innerhalb der Endothelschicht stattfinden, durch mechanische Hinweise. Biomechanische Signale induzieren strukturelle Veränderungen der Blutgefäße, legen eine arteriovenöse Spezifizierung fest und steuern die Entwicklung hämatopoetischer Stammzellen. Die Unzugänglichkeit der sich entwickelnden Gewebe schränkt das Verständnis der Rolle der Zirkulation in der frühen menschlichen Entwicklung ein. Daher sind In-vitro-Modelle zentrale Werkzeuge für die Untersuchung der Mechanobiologie von Gefäßen. In dieser Arbeit wird ein Protokoll zur Differenzierung von Endothelzellen aus humanen induzierten pluripotenten Stammzellen und deren anschließende Aussaat in ein fluidisches Gerät beschrieben, um ihre Reaktion auf mechanische Signale zu untersuchen. Dieser Ansatz ermöglicht die Langzeitkultur von Endothelzellen unter mechanischer Stimulation, gefolgt von der Entnahme der Endothelzellen zur phänotypischen und funktionellen Charakterisierung. Das hier etablierte In-vitro-Modell wird dazu beitragen, die intrazellulären molekularen Mechanismen aufzuklären, die die durch mechanische Signale vermittelte Signalübertragung übertragen, die letztendlich die Gefäßentwicklung während des menschlichen Fötuslebens orchestrieren.
Einleitung
Während der Embryonalentwicklung ist das Herz das erste Organ, das die Funktionalität1 entwickelt, mit nachweisbaren Kontraktionen ab dem frühesten Stadium der Endokardtubenbildung2. Die Durchblutung steuert zusammen mit den mechanischen Reizen, die durch den Blutfluss innerhalb des Gefäßes vermittelt werden, viele entscheidende Aspekte der frühen Entwicklung. Vor der Etablierung des fetalen Kreislaufs ist das Gefäßsystem in einem primären Kapillarplexus organisiert; Bei der Herzfunktion reorganisiert sich dieser Plexus in venöse und arterielle Gefäße3. Die Rolle mechanischer Signale bei der arteriovenösen Spezifizierung spiegelt sich in der pan-endothelialen Expression von arteriellen und venösen Markern vor der Initiierung des Blutflusses wider4.
Hämodynamische Kräfte steuern nicht nur die Entwicklung des Gefäßsystems selbst, sondern spielen auch eine grundlegende Rolle bei der Steuerung der Blutzellbildung. Hämatopoetische Stamm- und Vorläuferzellen (HSPCs) entstehen aus spezialisierten Endothelzellen, die als hämogenes Endothel 5,6,7,8 bezeichnet werden und in verschiedenen anatomischen Regionen der Embryonen ausschließlich im frühen Entwicklungsstadium vorhanden sind. Herzdefiziente Modelle haben zusammen mit In-vitro-Modellen gezeigt, dass mechanische Signale die HSPC-Produktion aus dem hämogeren Endothel instruieren und erhöhen 9,10,11,12,13,14.
Es wurde gezeigt, dass verschiedene Arten der Flussdynamik den Zellzyklus15 differentiell steuern, von dem bekannt ist, dass er sowohl für das hämogene Endothel 16,17 als auch für die arterielle Zellspezifikation18 wichtig ist. Insgesamt sind mechanische Signale entscheidende Determinanten der Zellidentität und -funktion während der Entwicklung. Neuartige In-vitro-Fluidik-Geräte ermöglichen es uns, die Einschränkungen zu überwinden, die mit der Untersuchung der Entwicklungsmechanobiologie während der menschlichen Blutentwicklung in vivo verbunden sind.
Das übergeordnete Ziel des Protokolls in diesem Manuskript ist es, Schritt für Schritt die experimentelle Pipeline zu beschreiben, um die Wirkung von Scherstress auf menschliche Endothelzellen zu untersuchen, die in vitro aus humanen induzierten pluripotenten Stammzellen (hiPSCs) gewonnen werden. Dieses Protokoll enthält detaillierte Anweisungen zur Differenzierung von hiPS-Zellen in Endothelzellen und deren anschließender Aussaat in fluidische Chips für das Stimulationsprotokoll. Damit können verschiedene in vitro gewonnene Endothelzellen auf ihre Fähigkeit getestet werden, die Scherspannung zu spüren, indem ihre Orientierung als Reaktion auf die Strömung analysiert wird. Dies wird es anderen Laboren ermöglichen, Fragen über die Reaktion auf Scherspannung und deren funktionelle Auswirkungen auf verschiedene Endothelzellidentitäten zu beantworten.
Protokoll
HINWEIS: Alle Zellkulturtechniken müssen unter sterilen Bedingungen in einer Laminar-Flow-Haube durchgeführt werden, und die Zellen müssen bei 37 °C in einer humifizierten Atmosphäre mit 5 % CO2 inkubiert werden. Anweisungen für alle Zytokinpräparate sowohl für die Erhaltungstherapie (rhbFGF) als auch für das Differenzierungsprotokoll (rhBMP4, rhVEGF, rhbFGF, rhIL6, rhFLT3L, rhIGF1, rhIL11, rhSCF, rhEPO, rhTPO, rhIL3) finden Sie in der Zusatztabelle S1.
1. Kultivierung von hiPSCs - Auftauen, Pflegen und Einfrieren von Zellen
- Herstellung von Erhaltungsmedium, Wachstumsfaktoren und anderen Reagenzien
- Bereiten Sie das Nährmedium vor, indem Sie das gesamte serumfreie Medium hESCs, 36 ml 25 % Rinderserumalbumin (BSA) und 1 ml 55 mM β-Mercaptoethanol zu Dulbeccos Modified Eagle Medium/F12 (DMEM/F-12) Basalmedium hinzufügen (siehe Materialtabelle).
- Resuspendieren Sie 1 mg Rho-Kinase-Inhibitor (iRock) in 1 ml DMSO, stellen Sie Aliquoten von 50 μl her und lagern Sie sie bei -20 °C.
HINWEIS: Diese Aliquots können 1 Jahr lang bei -20 °C aufbewahrt werden. Nach dem Auftauen sind sie 1 Woche bei 4 °C haltbar. - Die Vitronektin-Lösung (VTN-N) wird vor der Lagerung bei -80 °C auf Eis und aliquoten 60 μl pro Durchstechflasche aufgetaut. Kurz vor dem Beschichten der Platten verdünnen Sie den 60-μl-Stamm in 6 ml phosphatgepufferter Kochsalzlösung (DPBS) von Dulbecco. die Endkonzentration von 5 μg/ml.
- Auftauen von hiPSC-Zelllinien
ANMERKUNG: Die humane pluripotente Stammzelllinie SFCi55 wurde zuvor intern entwickelt und in großem Umfang für die Differenzierung in verschiedene Zelltypen und verschiedene embryonale Linien verwendet 19,20,21,22.- Eine Vertiefung einer 6-Well-Platte mit 1 ml VTN-N-Lösung für 1 h bei 37 °C beschichten.
HINWEIS: Nach der Inkubation können beschichtete Platten bis zu 1 Woche bei 4 °C gelagert werden. - Die VTN-N-Lösung wird mit einer Aspirationspipette abgesaugt und 1 ml vorgewärmtes Nährmedium mit 20 ng/ml rhbFGF hinzugefügt (Ergänzungstabelle S1).
- Tauen Sie das Fläschchen mit den hiPS-Zellen schnell in einem Wasserbad auf und überführen Sie die Zelle in 5 ml vorgewärmtes Kulturmedium.
- Schleudern Sie die Zellen für 3 Minuten bei 300 × g bei Raumtemperatur.
- Resuspendieren Sie das Zellpellet in 0,5 ml Kulturmedium, ergänzt mit 20 ng/ml rhbFGF.
- Überführen Sie die Zellen in eine beschichtete Vertiefung, die bereits 1 ml Medium enthält.
- Geben Sie 5 μl iRock in die Vertiefungen mit den Zellen in insgesamt 1,5 ml Medium.
- Kultivieren Sie die Zellen im Inkubator, wechseln Sie das Medium täglich während der Woche und füttern Sie die Zellen doppelt, indem Sie den Zellen das Doppelte des normalen Mediumvolumens hinzufügen, um die Fütterung über das Wochenende zu gewährleisten.
HINWEIS: Die Zellen werden nur 24 Stunden lang in Gegenwart von iRock gezüchtet.
- Eine Vertiefung einer 6-Well-Platte mit 1 ml VTN-N-Lösung für 1 h bei 37 °C beschichten.
- Wartung und Weiterleitung von hiPSCs
- Wechseln Sie das Medium täglich mit frischem, vorgewärmtem Nährmedium, das mit 20 ng/ml rhbFGF ergänzt wird.
- Passieren Sie die Zellen, wenn sie eine Konfluenz von etwa 80 % erreicht haben, in der Regel zweimal pro Woche.
- Um die Zellen zu passieren, wird eine Platte mit VTN-N beschichtet, wie zuvor in den Schritten 1.2.1 und 1.2.2 beschrieben.
- Saugen Sie das Medium aus den Vertiefungen mit Zellen an und waschen Sie sie mit DPBS.
- Das DPBS absaugen und 1 ml Dissoziationsreagenz (siehe Materialtabelle) zugeben und 1 Minute lang inkubieren.
- Das Dissoziationsreagenz wird abgesaugt und weitere 3 Minuten inkubiert. Klopfen Sie auf jeder Seite 10 Mal fest auf die Platte.
HINWEIS: Der Dissoziationsschritt muss möglicherweise zelltypspezifisch in Bezug auf die Inkubationszeit und das Klopfverfahren optimiert werden. - Geben Sie 1 ml Kulturmedium zu den Zellen und waschen Sie es mit einer Pasteur-Pipette einmal mit dem Medium, um sicherzustellen, dass die meisten Kolonien gesammelt werden.
- Geben Sie 150 μl der Zellsuspension in jede Vertiefung, um ein Durchgangsverhältnis von 1 Vertiefung in 6 zu erhalten.
HINWEIS: Unmittelbar nach dem Auftauen einer neuen Durchstechflasche ist es besser, die Zellen in einem niedrigeren Verhältnis wie 1:1 oder 1:2 für ein oder zwei Durchgänge zu passieren, damit sie eine stetige Wachstumsphase erreichen können, bevor sie in einem Verhältnis von 1:6 passieren. - Kultivieren Sie die Zellen im Inkubator, wechseln Sie das Medium täglich während der Woche und füttern Sie die Zellen einmal am Wochenende.
- Einfrieren von hiPS-Zelllinien
HINWEIS: Frieren Sie die Zellen in den ersten beiden Durchgängen nach dem Auftauen ein, um sicherzustellen, dass eine konstante Charge gefrorener Fläschchen mit niedriger Passage erhalten bleibt, um die Kultur zu starten. Frieren Sie die Zellen ein, wenn sie eine Konfluenz von ca. 80 % erreichen.- Wechseln Sie das Medium zu frischem, vorgewärmtem Nährmedium, das mit 20 ng/ml rhbFGF und 5 μl iRock ergänzt wird, und inkubieren Sie mindestens 1 h.
- Trennen Sie die Zellen wie in den Schritten 1.3.2.2 bis 1.3.2.5 beschrieben.
- Die abgelösten Zellen werden in einem 15-ml-Zentrifugenröhrchen mit 5 ml Kulturmedium gesammelt.
- 3 min bei 300 × g bei Raumtemperatur zentrifugieren.
- Saugen Sie den Überstand an und fügen Sie 1 ml Kryokonservierungslösung hinzu (siehe Materialtabelle).
- Pipettieren Sie die Zellen mit einer Pasteurpipette vorsichtig auf und ab, um sie in der Kryokonservierungslösung zu mischen.
HINWEIS: Vermeiden Sie übermäßiges Pipettieren, da dies zu einer Dissoziation der Zellcluster führen kann. - Teilen Sie die Zellsuspension in zwei Kryokonservierungsfläschchen mit je 0,5 ml auf.
- Die Kryokonservierungsfläschchen werden in einen Kryokonservierungsbehälter überführt, der auf 4 °C vorgekühlt ist.
- Den Behälter mit den Zellen für 24 Stunden in einen Gefrierschrank bei -80 °C stellen, bevor die Fläschchen zur Langzeitlagerung in flüssigen Stickstoff überführt werden.
2. Differenzierung von hiPS-Zellen zu Endothelzellen
- Herstellung von Differenzierungsmedium, Zytokinen und Wachstumsfaktoren
- Serumfreies Differenzierungsmedium (SFD) gemäß Tabelle 1 herstellen. Verwenden Sie dieses Medium von Tag 0 bis Tag 5 der Differenzierung.
- Bereiten Sie serumfreies Medium für CD34+ -Zellen (SFM-34) vor, indem Sie 34 Nährstoffpräparate und 5 ml L-Glutamin-Präparat zu dem 34 SFM Basalmedium hinzufügen (siehe Materialtabelle). Verwenden Sie dieses Medium ab dem 6. Tag der Differenzierung.
- Resuspendieren Sie 1 mg CHIR99021 in 716 μl DMSO, um eine 3 mM Lösung zu erhalten. Bei Raumtemperatur inkubieren, bis es vollständig resuspendiert ist; bei Bedarf schnell auf 37 °C aufwärmen. Stellen Sie 20 μl Aliquots her und lagern Sie diese bis zu 6 Monate bei -20 °C. Sofort nach dem Auftauen verwenden und nicht wieder einfrieren oder lagern.
- Resuspendieren Sie die Zytokine gemäß den Anweisungen in der Ergänzenden Tabelle S1. Lagern Sie alle Zytokine bei -80 °C.
- Differenzierung von Endothelzellen
ANMERKUNG: Für jeden Tag der Differenzierung sind 18 ml (3 ml Medium/Well) vorgewärmtes SFD-Medium gemäß den Zytokinmischungen vorzubereiten, die in Tabelle 2.- Tag 0 - Bildung von Embryoidkörpern (EBs)
- Bereiten Sie 18 ml SFD-Medium mit Mix 1 Zytokin gemäß Tabelle 2 für jede 6-Well-Platte (3 ml/Well) vor.
- Geben Sie 2 ml vorgewärmtes SFD-Medium mit Mix 1 Zytokin in jede Vertiefung einer zellabweisenden 6-Well-Platte (siehe Materialtabelle).
- Um EBs zu bilden, führen Sie die in den Schritten 1.3.2.2-1.3.2.4 beschriebenen Schritte aus.
HINWEIS: Stellen Sie sicher, dass die hiPSCs zu 70-80 % konfluierend sind, um die Differenzierung zu starten. - Geben Sie 1 ml vorgewärmtes SFD-Medium mit Mix 1 Zytokinen in jede Vertiefung der abgelösten Zellcluster.
- Verwenden Sie eine Pasteurpipette, um die Zellcluster vorsichtig in eine einzige Vertiefung mit zellabweisender Vertiefung für die EB-Bildung im Verhältnis 1:1 zu übertragen.
- Nachdem Sie die Platte in den Inkubator gelegt haben, bewegen Sie sie nach vorne und hinten, nach rechts und links, um die EBs gleichmäßig in der Vertiefung zu verteilen.
- Tag 1 - Mediumwechsel zu den EBs
HINWEIS: Dieser Schritt ist nur notwendig, wenn am Tag 1 der Differenzierung neben den EBs viele Einzelzellen in Suspension vorhanden sind.- Bereiten Sie 18 ml SFD-Medium mit Mix 1 Zytokin gemäß Tabelle 2 für jede 6-Well-Platte (3 ml/Well) vor.
- Schwenken Sie die Platte mit den EBs, um sie in die Mitte zu bewegen, und sammeln Sie sie mit einer Pasteur-Pipette in ein 15-ml-Zentrifugenröhrchen.
HINWEIS: Wenn die EBs wie in Fäden verklumpt aussehen, trennen Sie sie, indem Sie sie mit einem P1000 auf und ab pipettieren, bevor Sie sie in das 15-ml-Zentrifugenröhrchen sammeln. - Warten Sie 5-10 Minuten, bis sich die EBs am Boden des Röhrchens abgesetzt haben.
HINWEIS: Wenn die EBs zu klein sind, zentrifugieren Sie sie 5 Minuten lang bei 100 × g , damit sie sich absetzen. - Waschen Sie die zellabweisenden Platten mit sterilem Wasser oder DPBS, um einzelne Zellen oder Ablagerungen zu entfernen.
- Saugen Sie den Überstand vorsichtig und langsam von den EBs ab, ohne sie zu lösen.
- Geben Sie 2 ml SFD mit gemischten 1 Zytokinen in jede Vertiefung der zellabweisenden Platten.
- Resuspendieren Sie die EBs mit 1 ml SFD-Medium und mischen Sie 1 Zytokine für jede Ausgangsvertiefung - für eine 6-Well-Platte fügen Sie 6 ml Medium hinzu.
- Übertragen Sie die EBs in 1 ml Volumen pro Well, die bereits 2 mL SFD-Medium enthält, auf die zellabweisenden Platten.
- Nachdem Sie die Platte in den Inkubator gelegt haben, bewegen Sie sie nach vorne und hinten, nach rechts und links, um die EBs gleichmäßig in der Vertiefung zu verteilen.
- Tag 2 - Hinzufügen von CHIR99021
- Schwenken Sie die EBs in die Mitte der Platte und fügen Sie CHIR99021 gemäß Tabelle 2 an der Seite der Vertiefung hinzu, um einen direkten Kontakt mit den Zellen zu vermeiden.
HINWEIS: Wenn das Medium an Tag 1 nicht gewechselt wurde, ersetzen Sie das gesamte Medium, anstatt CHIR allein hinzuzufügen. Bereiten Sie 18 ml SFD-Medium mit Mix 2 gemäß Tabelle 2 für jede 6-Well-Platte (3 ml/Well) vor. - Nachdem Sie die Platte in den Inkubator gelegt haben, bewegen Sie sie nach vorne und hinten, nach rechts und links, um die EBs gleichmäßig in der Vertiefung zu verteilen.
- Schwenken Sie die EBs in die Mitte der Platte und fügen Sie CHIR99021 gemäß Tabelle 2 an der Seite der Vertiefung hinzu, um einen direkten Kontakt mit den Zellen zu vermeiden.
- Tag 3 - Mediumwechsel der EBs und Zugabe von Mix 3 Zytokinen
- Bereiten Sie 18 ml SFD-Medium mit Mix 3 Zytokinen gemäß Tabelle 2 für jede 6-Well-Platte (3 ml/Well) vor.
- Sammeln Sie die EBs wie in den Schritten 2.2.2.2-2.2.2.4 beschrieben.
- Geben Sie vorgewärmte 2 ml SFD-Medium mit Mix 3 Zytokinen zu den zellabweisenden Platten.
- Saugen Sie den Überstand vorsichtig von den EBs ab. Fügen Sie 1 ml/Well SFD mit Mix 3 Zytokinen hinzu.
- Verteilen Sie die EBs wie in den Schritten 2.2.2.8-2.2.2.9 beschrieben auf die Vertiefungen.
- Tag 6 - Mediumwechsel für SFM-34 und Zugabe von Mix 4 Zytokinen
- Bereiten Sie 18 ml SFD-Medium mit Mix 4 Zytokinen gemäß Tabelle 2 für jede 6-Well-Platte (3 ml/Well) vor.
- Sammeln Sie die EBs wie in den Schritten 2.2.2.2-2.2.2.4 beschrieben.
- Geben Sie 2 ml vorgewärmtes SFM-34-Medium mit Mix 4 Zytokinen zu den zellabweisenden Platten.
- Saugen Sie den Überstand vorsichtig von den EBs ab. Fügen Sie 1 ml/Well SFM-34 mit Mix 4 Zytokinen hinzu.
- Verteilen Sie die EBs wie in den Schritten 2.2.2.8-2.2.2.9 beschrieben auf die Vertiefungen.
- Tag 0 - Bildung von Embryoidkörpern (EBs)
3. Isolierung von CD34+ -Zellen und Seeding in den Chip
HINWEIS: CD34+ -Zellen werden über einen positiven Isolationsansatz mit einem CD34-Mikrokügelchen-Kit (siehe Materialtabelle) isoliert, das CD34-Mikrokügelchen enthält, die mit monoklonalen Mausantikörpern, antihumanen CD34-Antikörpern und FcR-Blockierungsreagenz (humanes IgG) konjugiert sind. Es ist wichtig, die Effizienz der Säulenisolierung zu validieren, indem die Zellen vor und nach der Isolierung für die Durchflusszytometrie gefärbt werden. Unten wird angegeben, wann Zellen für diese Analyse entnommen werden müssen.
- Bereiten Sie Materialien und Reagenzien vor.
- Bereiten Sie den Waschpuffer vor, indem Sie 5 ml 5%ige BSA-Lösung und 200 μl EDTA 0,5 M bis 45 ml DPBS hinzufügen, um PBS + 0,5 % BSA + 2 mM EDTA zu erhalten. Für jede Isolierung frisch zubereiten, filtern und bis zur Verwendung im Kühlschrank aufbewahren.
- Beschichten Sie fluidische Chips mit Lamininlösung, die durch Verdünnung des rhLaminin 521 1:50 in DPBS Ca 2+ Mg2+ hergestellt wurde. Beschichten Sie jeden Chip mit dem für den verwendeten Chip geeigneten Volumen und inkubieren Sie ihn vor der Aussaat 2 h lang im Inkubator.
HINWEIS: Andere Matrices können für die Beschichtung verwendet werden und sollten für den spezifischen Zelltyp/das spezifische Experiment getestet werden. - Bereiten Sie Mix 4 SFM-34-Medium vor, indem Sie 18 ml SFM-34-Medium mit Mix-4-Zytokinen gemäß Tabelle 2 ergänzen und die Mischung in ein 50-ml-Röhrchen im Inkubator geben, wobei der Deckel leicht abgeschraubt ist, um den Gasaustausch zu erleichtern.
- Legen Sie die ausgewählten Perfusionssets und alle anderen zu verwendenden Schläuche zum Entgasen in den Inkubator.
- Tag 8 - Dissoziation von EBs und CD34+ Isolierung
- Sammeln Sie die EBs wie in den Schritten 2.2.2.2-2.2.2.5 beschrieben.
- Fügen Sie 1 ml Zelldissoziationsreagenz pro Ausgangsvertiefung der gesammelten EBs hinzu (wenn 6 Vertiefungen entnommen wurden, fügen Sie 6 ml hinzu).
- 1 ml der EB-Suspension im Zelldissoziationsreagenz in jede Vertiefung der zellabweisenden Platte zurückgeben.
- 10 Minuten im Inkubator inkubieren.
- Pipettieren Sie die EBs vorsichtig mit einem P1000 auf und ab gegen die Vertiefung, nicht mehr als 10 Mal.
- Wiederholen Sie die Schritte 3.2.4-3.2.5 insgesamt 3x.
HINWEIS: Wenn die EBs schwer zu dissoziieren sind, wiederholen Sie die obigen Schritte insgesamt 4x. - Fügen Sie 5 ml Waschpuffer für jede Vertiefung dissoziierter EBs hinzu.
- Sammeln Sie die Zellen in ein 50-ml-Zentrifugenröhrchen, indem Sie sie durch ein 40-μm-Sieb führen. Nehmen Sie 10 μl der Zellsuspension, um die Zellen zu zählen.
HINWEIS: Um die Wirksamkeit der Isolierung zu testen, werden 105 Zellen/Röhrchen in zwei verschiedene Röhrchen (ungefärbte Kontrolle und vorsortierte Testprobe) überführt, die später für die Durchflusszytometrie verwendet werden (wie in den Schritten 4.3.9-4.3.13 beschrieben). Für eine 6-Well-Platte sollten nach der Filtration ~106 Zellen gesammelt werden. - Schleudern Sie die Zellen für 10 Minuten bei 300 × g herunter.
- Resuspendieren Sie die Zellen in 300 μl Waschpuffer und pipettieren Sie sie einige Male vorsichtig, um sicherzustellen, dass keine Klumpen vorhanden sind. Befolgen Sie weiterhin das Protokoll des Herstellers (siehe Materialtabelle).
- CD34+ Zell-Seeding in fluidische Chips
HINWEIS: Der im Protokoll verwendete Fluidik-Chip hat eine Kanalhöhe von 0,6 mm und eine Länge von 50 mm, was einer Gesamtwachstumsfläche von 2,5cm2 entspricht (ergänzende Abbildung S1). Diese Art von Chip wird mit einem Gesamtvolumen der Zellsuspension von 150 μl ausgesät. Es können verschiedene Chips verwendet werden, und das Volumen der Aussaat und die Zelldichte sollten entsprechend der Wachstumsfläche angepasst werden. Je nach verwendeter Zelllinie und deren Wachstum kann eine zusätzliche Optimierung erforderlich sein.- Resuspendieren Sie die isolierten CD34+ -Zellen in 300 μl vorgewärmtem SFM-34-Medium mit Mix-4-Zytokinen.
- Nehmen Sie 10 μl der Zellsuspension und zählen Sie die Zellen.
- Resuspendierung von 2,5 × 105 Zellen in einem Endvolumen von 150 μl ergänztem SFM-34; 0,5 μl iRock hinzufügen.
HINWEIS: Um die Wirksamkeit der Isolierung zu testen, werden 105 Zellen/Röhrchen in ein Röhrchen (nachsortierte Testprobe) überführt, das später für die Durchflusszytometrie verwendet wird (wie in den Schritten 4.3.9-4.3.13 beschrieben). - Saugen Sie das Laminin langsam aus den Fluidik-Chips ab, indem Sie die Spitze eines P200 in den Behälter am Rand des Kanals stecken.
HINWEIS: Wenn die Flüssigkeit schwer aufzufangen ist, heben Sie langsam eine Seite des Chips an, damit sich die Flüssigkeit in den gegenüberliegenden Behälter bewegt. - Geben Sie die Zellsuspension gleichmäßig in den Kanal, um sicherzustellen, dass sich keine Blasen bilden.
HINWEIS: Führen Sie die Schritte 3.3.4-3.3.5 schnell, aber vorsichtig durch, um ein Austrocknen des Laminins und die Bildung von Blasen in den Kanälen des Chips zu vermeiden. Wenn sich Blasen bilden, heben Sie eine Seite des Chips an und klopfen Sie vorsichtig auf den Objektträger, um die Blasen zu mobilisieren. Wenn sie das Reservoir erreichen, steigen sie an die Luftschnittstelle und sollten nicht in der Lage sein, den Kanal zu mieten. - Übertragen Sie den Chip in den Inkubator und lassen Sie ihn über Nacht stehen, damit die Zellen vollständig mit dem Kanal verbunden sind und länglich aussehen.
- Wenn die Zellen vollständig angebunden sind, saugen Sie das Medium wie in Schritt 3.3.4 an und ersetzen Sie es durch 200 μl Zytokin-ergänztes SFM-34.
- Tauschen Sie das Medium von nun an täglich aus, bis die Zellen eine 90%-100%ige Konfluenz erreicht haben.
4. Anwendung des kontinuierlichen Flusses auf Endothelzellen - Aorta-on-a-Chip
- Bereiten Sie Materialien und Reagenzien vor.
- Bereiten Sie Mix 4 SFM-34-Medium vor, indem Sie 18 ml SFM-34-Medium mit Mix-4-Zytokinen gemäß Tabelle 2 ergänzen und in ein 50-ml-Röhrchen im Inkubator geben, wobei der Deckel leicht abgeschraubt ist, um den Gasaustausch zu erleichtern.
- Legen Sie die ausgewählten Perfusionssets und alle Schläuche, die für den fluidischen Aufbau verwendet werden sollen, in den Inkubator, um sie zu entgasen.
- Montage von Fluidiksystemen
- Installieren Sie das Perfusionsset gemäß dem Protokoll des Herstellers in das Gerät.
HINWEIS: Denken Sie daran, Klemmen im System zu verwenden. Wenn für diesen Schritt Schiebeklemmen verwendet werden, müssen diese vor dem Anschluss an den Chip auf den Schlauch geschoben werden. - Bringen Sie einen neuen Fluidik-Chip an und fügen Sie das mit Zytokinen angereicherte SFM-34-Medium hinzu, wobei Sie sicherstellen müssen, dass beide Reservoirs unter sterilen Bedingungen in der Haube gefüllt sind.
- Führen Sie das Blasenentfernungsprogramm und den Kalibrierungsschritt durch.
- Entnehmen Sie die Fluidikeinheit mit dem angeschlossenen Set aus dem Inkubator und setzen Sie sie in die Haube um. Nehmen Sie auch die Chips mit den Zellen aus dem Inkubator.
- Klemmen Sie den Schlauch auf beiden Seiten des Testchips ein.
- Entfernen Sie den Schlauch vom Testchip.
HINWEIS: Vergewissern Sie sich, dass keine Blasen im Luer-Anschluss am Ende des Schlauchs vorhanden sind. Wenn Blasen sichtbar sind, saugen Sie diese vorsichtig mit einer P200-Pipette ab und fügen Sie bei Bedarf weiteres Medium hinzu, um sicherzustellen, dass der Konnektor mit Medium gefüllt ist. - Verbinden Sie den Chip, der die Zellen enthält, mit dem Schlauch.
- Entfernen oder öffnen Sie die Klemmen.
- Übertragen Sie das System in den Inkubator und schließen Sie die Luftpumpe an die Fluidikeinheit an.
- Starten Sie das vorgewählte Programm mit der pumpenspezifischen Software (ergänzende Abbildung S2) mit der in Tabelle 3 beschriebenen allmählichen Erhöhung der Schubspannung.
HINWEIS: Abhängig von dem spezifischen Experiment, das benötigt wird, muss das Stimulationsprogramm möglicherweise optimiert werden. Hier wird ein allmählicher Anstieg der Scherspannung beschrieben, der zu einem Endwert von 5 dyn/cm2 führt, der die Scherspannung nachahmt, die berechnet wurde, um die Wand der dorsalen Aorta zu Beginn der fetalen Zirkulation zu erfahren 9. Unabhängig von der endgültigen Scherspannung, die angewendet wird, ist es notwendig, die Spannung im Laufe der Zeit allmählich zu erhöhen, damit sich die Zellen an die Kraft anpassen können, ohne dass sich die Zellen vom Chip lösen. Wenn das gewählte Stimulationsprotokoll länger als 3 Tage dauert, sollten Zytokine in das System aufgefüllt werden, indem 1 ml SFM-34 mit den Mix-4-Zytokinen hinzugefügt werden, die normalerweise in 18 ml zugegeben würden. Dazu wird das Pumpenprogramm schnell pausiert und je 500 μL supplementiertes Medium in jede der beiden Spritzen des Fluidik-Sets gegeben.
- Installieren Sie das Perfusionsset gemäß dem Protokoll des Herstellers in das Gerät.
- Zellentnahme zur Analyse
- Den Dissoziationspuffer im Wasserbad vorwärmen.
- Nehmen Sie die Fluidikeinheit aus dem Inkubator und setzen Sie sie in die Haube ein.
- Klemmen Sie den Schlauch, der den Chip flankiert, auf beiden Seiten ein und entfernen Sie den Schlauch aus den Reservoirs auf dem Chip.
- Entfernen Sie das Medium vorsichtig vom Chip und ersetzen Sie es durch DPBS Ca 2+ Mg2+ , um die Zellen zu waschen.
HINWEIS: Dieser Schritt des Waschens mit PBS kann übersprungen werden, wenn sich die Zellen zu lösen beginnen. - 150 μl Dissoziationspuffer vorsichtig zugeben und 3 min bei 37 °C inkubieren.
HINWEIS: Prüfen Sie unter dem Mikroskop, ob die Zellen einzeln und abgetrennt sind. Andernfalls weitere 2 Minuten inkubieren. Es ist wichtig, dass die Zellen vor dem Absaugen des Mediums vollständig vom Kanal gelöst werden, da der Chip es nicht zulässt, die Zellablösung durch Pipettieren zu unterstützen. Andere Lösungen können verwendet werden, um die Zellen abzutrennen, wie z. B. Trypsin oder EDTA-basierte Puffer. - Sammeln Sie den Dissoziationspuffer, der die Zellen enthält, aus einem Reservoir und geben Sie ihn in ein 15-ml-Zentrifugenröhrchen und waschen Sie den Kanal einmal mit DPBS, um alle abgelösten Zellen zu sammeln.
- Geben Sie 1 ml Waschpuffer in das 15-ml-Röhrchen mit den Zellen und nehmen Sie 10 μl, um die Zellen zu zählen.
- Teilen Sie die Zellsuspension in Durchflusszytometrieröhrchen auf, um 10bis 5 Zellen pro Reagenzglas zu erhalten.
- Schleudern Sie die Röhrchen 5 min bei 300 × g.
- Bereiten Sie die Färbelösung so vor, dass sie 50 μl für jedes zu färbende Reagenzglas enthält. CD34 PerCP-efluor710 oder CD34-PE in einer Verdünnung von 1:100 bzw. 1:200 zugeben.
- Die Zellen werden in 50 μl Färbelösung resuspendiert und 30 Minuten lang bei 4 °C inkubiert.
- Waschen Sie die Zellen durch Zugabe von 2 ml Waschpuffer und schleudern Sie sie 5 Minuten lang bei 300 × g.
- Resuspendieren Sie die Pellets in 100 μl Färbelösung und erfassen Sie die Daten mit einem Durchflusszytometer.
HINWEIS: Die Zellen können auch direkt im Chip für die RNA-Extraktion mit 150 μl RNA-Lysepuffer lysiert oder für die Bildgebung mit 4% Paraformaldehyd in DPBS für 10 Minuten bei Raumtemperatur fixiert werden.
- Analyse der Zellorientierung
- Analysieren Sie die Bilder, um Änderungen der Zellorientierung mit FIJI23 zu quantifizieren (ergänzende Abbildung S3).
- Öffnen Sie den ROI-Manager (Region of Interest) über den Menüpunkt Analysieren | Werkzeuge | ROI-Manager-Menü .
- Zeichnen Sie die Zellenkonturen manuell mit dem Polygonauswahlwerkzeug , und fügen Sie sie dem ROI-Manager hinzu, indem Sie auf Hinzufügen klicken oder die Tastenkombination STRG+T verwenden.
- Messen Sie die Ausrichtung der einzelnen ROIs, indem Sie im Menü Analysieren | Menü "Maße festlegen" .
- Wenden Sie die Messung auf alle ROIs an, indem Sie Mehr... Multi-Measure-Befehl im ROI-Manager.
HINWEIS: In diesem Schritt wird eine Ellipse an jede ROI angepasst und eine Tabelle generiert, die die Länge der Haupt- und Nebenellipsenachsen sowie den Winkel enthält. - Exportieren Sie die Tabelle in eine CSV-Datei, um sie zum Plotten in andere Software zu importieren.
HINWEIS: Das Skript, das für die Plots verwendet wird, ist unter https://gist.github.com/nicolaromano/708b3231d730ee7f70763a7cf885 verfügbar.
0ddc.
- Analysieren Sie die Bilder, um Änderungen der Zellorientierung mit FIJI23 zu quantifizieren (ergänzende Abbildung S3).
Ergebnisse
Wir beschreiben hier ein Protokoll zur Differenzierung und Mechanostimulation von Endothelzellen, die von hiPSCs abgeleitet sind, das es ermöglicht, ihre Reaktion auf mechanische Signale zu untersuchen (Abbildung 1). Dieses Protokoll führt zur Produktion von funktionell mechanosensitiven Endothelzellen. Wir liefern hier repräsentative Ergebnisse und beschreiben den erwarteten Phänotyp, um zu beurteilen, wie die Zellen während der Differenzierung auf die Zytokinstimulation reagieren.
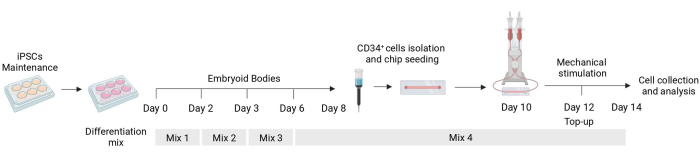
Abbildung 1: Schematische Darstellung des Differenzierungs- und mechanischen Stimulationsprotokolls. Schematische Darstellung des Differenzierungsprotokolls mit dem Timing der verschiedenen Mischungen von Zytokinen, der CD34+ -Zellisolierung, dem fluidischen Chip-Seeding und der abschließenden Analyse der mechanisch stimulierten Zellen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Kultur von hiPS-Zellen
Es ist wichtig, das Protokoll von hiPSCs zu starten, die unter Selbsterneuerungsbedingungen korrekt wachsen. Ein guter Indikator für die Qualität der Kultur ist die Geschwindigkeit ihres Wachstums. Nach dem Auftauen benötigen die Zellen möglicherweise 2-3 Wochen, um die richtige Wachstumsphase zu erreichen, die eine gute Differenzierung gewährleistet. Wenn die Zellen zweimal pro Woche im Verhältnis 1:6 durchlaufen werden können, um fast vollständige Konfluenz zu erreichen, ist dies der Zeitpunkt, an dem sie bereit sind, am selben Tag differenziert zu werden, an dem sie durchlaufen werden müssen.
Differenzierung von hiPS-Zellen in Endothelzellen
Der erste Schritt der Differenzierung, der in der Bildung von Embryoidkörpern (EBs) besteht, ist zelllinienabhängig und muss möglicherweise für die jeweilige Zelllinie optimiert werden. Die in den Protokollschritten 1.3.2.2 bis 1.3.2.4 beschriebene Dissoziation kann modifiziert werden, indem entweder die Inkubation mit dem Dissoziationsreagenz und die anschließende Dissoziation mit der Pasteurpipette reduziert oder verlängert werden. Des Weiteren können für diesen Schritt neben der physikalischen Dissoziation der Kolonien mit einem Schneidewerkzeug oder einer P100-Pipettenspitze auch andere Dissoziationsreagenzien verwendet werden. EBs von guter Qualität zeigen am 2. Tag der Differenzierung eine definierte Kante und erscheinen klar und hell, wenn sie mit einem Mikroskop beobachtet werden; Dunklere Bereiche können auf den Zelltod innerhalb der EBs hinweisen (Abbildung 2).
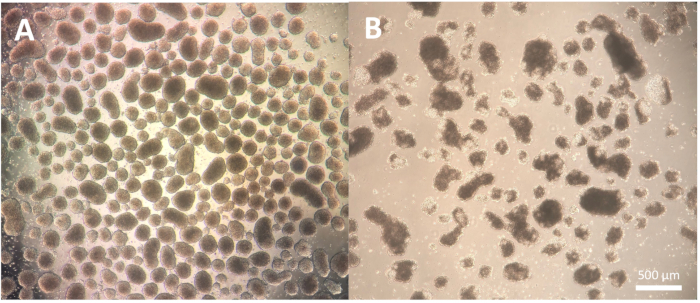
Abbildung 2: Morphologie der Embryoidkörper. (A) Tag 2 Embryoidkörper mit gut definierten äußeren Rändern und gleichbleibender Größe. (B) Tag 2 Embryoidkörper von schlechter Qualität, die einen ausgedehnten Zelltod aufweisen, der zu einer Disaggregation der Struktur führt. Maßstabsleiste = 500 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
An Tag 2 hemmt die Zugabe von CHIR99021 zu den EBs das GSK-3-Protein, was zur Aktivierung des Wnt-Signalwegs führt. Verschiedene Zelllinien reagieren unterschiedlich auf die CHIR-Behandlung, und dies sollte durch Quantifizierung der Anzahl der CD34+ -Zellen, die an Tag 8 erhalten wurden, unter Verwendung verschiedener Konzentrationen getestet werden (Abbildung 3).
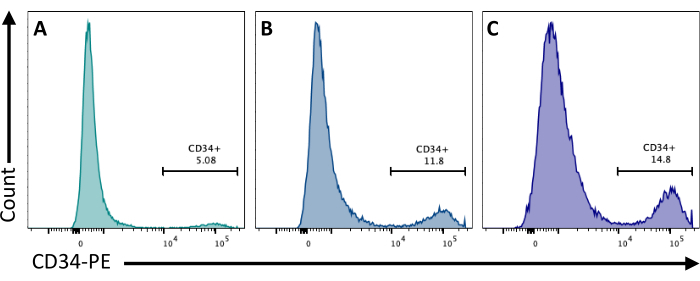
Abbildung 3: Differenzierung von Endothelzellen mit verschiedenen CHIR-Behandlungen. Quantifizierung der Endothelzellbindung durch Durchflusszytometrie am Tag 8 der Differenzierung durch CD34-Membranexpression nach CHIR-Behandlung an Tag 2 bei (A) 3 μM, (B) 5 μM und (C) 7 μM. Die Durchflusszytometriedaten wurden mit Fünf-Laser-Zytometern und spezieller Software gewonnen (siehe Materialtabelle). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
CD34+ Zellisolierung
Es ist wichtig zu validieren, dass die CD34+-Anreicherung mit den magnetischen Kügelchen nach der Elution der Säule mindestens 80% CD34+ liefert. Um eine ausreichende Reinheit zu gewährleisten, kann ein Aliquot von Zellen, die aus der magnetischen Isolierung gewonnen wurden, durch Durchflusszytometrie analysiert werden, wobei sichergestellt wird, dass ein anderer Antikörperklon als der für die magnetische Anreicherung verwendete verwendet wird. Hier wurde der 4H11-Klon verwendet und nach der Anreicherung wurde eine Reinheit von ~85% erreicht (Abbildung 4).
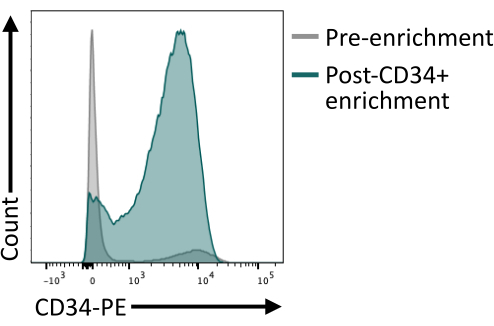
Abbildung 4: Membranexpression von CD34 vor und nach Anreicherung durch magnetische Sortierung. Tag 8 dissoziierte Embryoidkörper (grau) und Zellen nach magnetischer Anreicherung (grün) wurden auf CD34-Expression gefärbt und mittels Durchflusszytometrie analysiert, wobei eine erfolgreiche Anreicherung nach der Sortierung gezeigt wurde. Die Daten der Durchflusszytometrie wurden mit Hilfe von Fünf-Laser-Zytometern und spezieller Software gewonnen (siehe Materialtabelle). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Aussaat von Zellen in den fluidischen Kanal
Bei der Aussaat der Zellen im fluidischen Kanal ist es entscheidend, die Adhäsion und Proliferation der Endothelzellen zu verfolgen. Nach der Aussaat benötigen die Zellen ~5 Stunden, um vollständig am Kanal zu haften (Abbildung 5A). In dieser Phase kann auch eine alternative Beschichtungslösung getestet werden, um die Haftung zu verbessern. Um zu bestätigen, dass die getesteten Zellen mechanosensitiv sind und somit in der Lage sind, auf mechanische Stimulation zu reagieren, kann die Zellorientierung über die Zeit getestet werden. Die Zellen vor der Stimulation zeigen eine zufällige Orientierung (Abbildung 5A und Abbildung 5C) und orientieren sich parallel zur Richtung des Flusses (Abbildung 5B, C). Das hier beschriebene Protokoll ermöglicht die Entnahme der Zellen aus dem Kanal, um eine nachgeschaltete Analyse, z. B. Durchflusszytometrie, zur Untersuchung ihres Membranimmunphänotyps durchzuführen und die endotheliale Identität der stimulierten Zellen bereitzustellen (Abbildung 5D,E).
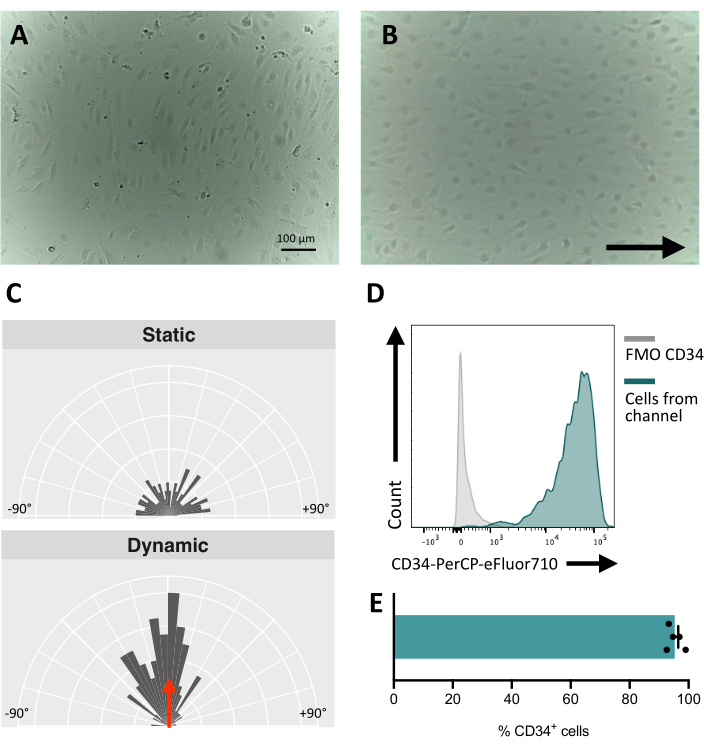
Abbildung 5: Mechanoreaktionsfähigkeit von hiPSCs-abgeleiteten Endothelzellen. (A) Konfluente Schicht isolierter CD34+ -Zellen 48 h nach der Aussaat. (B) Reorientierte Schicht von Endothelzellen 3 Tage unter dynamischer Kultur. (C) Orientierungsanalyse der Endothelzellen nach 5 Tagen dynamischer Kultur. (D) CD34-Expressionsprofil von Zellen, die 5 Tage lang unter Fluss kultiviert wurden. (E) Prozentsatz der CD34+ -Zellen der Zellpopulation, die aus dem fluidischen Kanal gewonnen wurden. Die Bilder wurden mit einem inversen Inkubatormikroskop aufgenommen; Die Daten der Durchflusszytometrie wurden mit Fünf-Laser-Zytometern und einer speziellen Software gewonnen (siehe Materialtabelle). Maßstabsbalken = 100 μm (A,B). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Reagenzien | Bestandskonzentration | Hinzugefügtes Volumen | Endkonzentration |
| Iscove's Modified Dulbecco's Medium (IMDM) | - | 333 ml | - |
| Schinken F-12 Nährstoffmischung (F-12) | - | 167 ml | - |
| N-2 Zuschlag (100x) | 100 x | 5 ml | 1-fach |
| B-27 Zuschlag (50x) | 50 x | 10 ml | 1-fach |
| Ascorbinsäure | 10 mg/ml | 1,25 ml | 25 μg/ml |
| α-Monothioglycerin (MTG) | 11,5 Mio. | 19,5 μl | 448,5 μM |
| Humanes Serumalbumin | 100 mg/ml | 2,5 ml | 0,5 mg/ml |
| Holo-Transferrin | 100 mg/ml | 0,75 ml | 150 μg/ml |
Tabelle 1: Zusammensetzung und Rezeptur für 500 ml serumfreies Differenzierungsmedium (SFD).
| Tage der Differenzierung | Zytokin-Mischung | Zytokin | Endkonzentration |
| Tag 0 - 2 | Mischung 1 | BMP4-KARTON | 20 ng/ml |
| Tag 2 | Mischung 2 | CHIR99021 | 7 μM |
| Ab Tag 3 | 3 und 4 mischen | VEGF | 15 ng/ml |
| bFGF | 5 ng/ml | ||
| Ab dem 6. Tag | Mischung 4 | IL6-KARTON | 10 ng/ml |
| FLT3L | 10 ng/ml | ||
| IGF1-KARTON | 25 ng/ml | ||
| IL11-KARTON | 5 ng/ml | ||
| SCF | 50 ng/ml | ||
| EPO | 3 U/ml | ||
| TPO | 30 ng/ml | ||
| IL3-KARTON | 30 ng/ml |
Tabelle 2: Mischungen von Zytokinen, die für die Differenzierung von Endothelzellen verwendet werden, Tage, an denen sie dem SFD-Medium zugesetzt werden, und Endkonzentration.
| Schubspannung (dyn/cm2) | Zeit (h) |
| 0.5 | 1 |
| 1 | 1 |
| 1.5 | 1 |
| 2 | 1 |
| 2.5 | 1 |
| 3 | 1 |
| 3.5 | 1 |
| 4 | 1 |
| 4.5 | 1 |
| 5 | Bis zum Ende des Experiments |
Tabelle 3: Schubspannungswerte für die dynamische Kultur und die Dauer ihrer Anwendung.
Ergänzende Abbildung S1: Geometrie und Abmessungen des Chips und der Schläuche, die für dieses Protokoll verwendet werden. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung S2: Schritt-für-Schritt-Anleitung für die Software zur Steuerung der Luftpumpe mit einer Beschreibung der einzelnen Schritte. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung S3: Leitfaden für die Orientierungsanalyse mit FIJI mit der Zeichnung der Zellform, der elliptischen Anpassung und der endgültigen Messung. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Tabelle S1: Einheitsgröße, Resuspensionsvolumen und Stammkonzentrationen für Zytokine, die im Differenzierungsprotokoll verwendet werden. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Das Protokoll, das wir hier beschreiben, ermöglicht die Generierung mechanosensitiver Endothelzellen aus humanen pluripotenten Stammzellen und die Untersuchung ihrer Reaktion auf mechanische Stimulation, die durch kontrollierten Scherstress vermittelt wird. Dieses Protokoll basiert vollständig auf Zytokinen und ist vollständig kompatibel mit GMP-Reagenzien für eine mögliche Translation in die Produktion von Zellen für die Zelltherapie.
Die Ableitung von hiPS-Zellen bietet Wissenschaftlern ein instrumentelles Modell für die frühen Stadien der Embryonalentwicklung, das es ermöglicht, Prozesse zu untersuchen, die sonst in vivo nur schwer zu untersuchen sind 24. Tatsächlich werden menschliche embryonale Gewebe, die für die Forschung zur Verfügung stehen, von Embryonen gesammelt, denen die Durchblutung fehlt, und dies könnte einen erheblichen Einfluss auf die molekulare Signatur haben, die durch mechanische Signale gesteuert wird. Der hier beschriebene Ansatz ermöglicht Live-Bildgebung und Echtzeit-Untersuchung der Zellantwort auf Scherspannung. Die Kombination von hiPS-Zellen mit Fluidik bietet ein Studienmodell, das die begrenzte Verfügbarkeit und Unzugänglichkeit des sich entwickelnden fetalen Gewebes überwindet, wenn die Initiierung der Zirkulation die Etablierung des Herz-Kreislauf- und Blutsystems umgestaltet und steuert 3,9,10,25.
Eine Einschränkung des Protokolls besteht darin, dass die aus diesem Protokoll abgeleiteten Endothelzellen möglicherweise nicht die verschiedenen Identitäten verschiedener Endothelzellen widerspiegeln, die in den sich entwickelnden Geweben vorhanden sind. Um diese Einschränkung zu überwinden, könnte eine spezifische Kombination von Zytokinen während des Differenzierungsprozesses vor der fluidischen Stimulation erforderlich sein, um die gewünschte Identität oder den gewebespezifischen Phänotyp zu erhalten26. Die Isolierung von endothelialen Untergruppen kann mit einem verfeinerten Immunphänotyp während des Isolierungsschritts erreicht werden. Dieses Protokoll isoliert Endothelzellen nur auf der Grundlage der Expression von CD34 und ermöglicht so eine Säulenisolierung anstelle der fluoreszenzaktivierten Zellsortierung (FACS). Dadurch werden der Zelltod und das Risiko einer Kontamination reduziert. Darüber hinaus wurde dieses Protokoll speziell entwickelt, um die Rolle der Scherspannung zu untersuchen, die durch laminare Strömung vermittelt wird. Alternative strömungstechnische Ansätze müssen eingesetzt werden, um die Auswirkungen anderer mechanischer Signale wie Dehnung oder Kompression oder anderer Arten von Strömungen wie gestörter oder gestörter Strömung zu untersuchen.
Wir haben bereits gezeigt, dass iPSC-abgeleitete Endothelzellen die heterogenen arteriovenösen zellulären Identitäten27 nachahmen, ähnlich denen, die in der fetalen dorsalen Aorta beobachtet wurden28,29,30. Dies ist von besonderer Bedeutung im Zusammenhang mit der Gefäßentwicklung und der zellulären Spezifizierung, von der bekannt ist, dass sie durch den Blutkreislauf gesteuert wird. Studien in verschiedenen Modellen zeigten, dass mangelnde Durchblutung zu einer veränderten arteriovenösen Spezifikation führt11,14,31. Die Mechanismen, die mechanische Signale mit der Zellspezifikation verbinden, sind noch unbekannt, und die hier beschriebene Pipeline ermöglicht verfeinerte funktionelle Studien, die in vivo nicht getestet werden konnten.
Diese Pipeline beschreibt die Produktion und Stimulation von Endothelzellen, die aus hiPSCs gewonnen werden, unter Verwendung kommerziell erhältlicher fluidischer Kanäle, wodurch die Notwendigkeit eines Gießens der Bauelemente wie bei den weit verbreiteten Polydimethylsiloxan-Bauelementen (PDMS) vermieden wird12. Darüber hinaus macht die Verwendung von PDMS-Chips die Entnahme der stimulierten Zellen besonders schwierig, während mit diesem Protokoll die Zellen leicht aus dem Kanal entnommen werden können. Dies verbessert die analytische Aussagekraft erheblich und ermöglicht nachfolgende Analysen wie Proteom- und Transkriptomanalysen, Durchflusszytometrie und funktionelle Assays, die möglicherweise weitere Kultur- oder In-vivo-Assays erfordern.
Offenlegungen
Die Autoren haben keine Interessenkonflikte offenzulegen.
Danksagungen
Diese Arbeit wurde durch den Research Advanced Grant 2021 der European Hematology Association, den Global Research Award 2021 der American Society of Hematology und den Internal Strategy Support Fund ISSF3 unterstützt, der vom Welcome Trust und der University of Edinburgh finanziert wird. Wir danken Fiona Rossi von der Flow Cytometry Facility für die Unterstützung bei der Durchflusszytometrie-Analyse. Zum Zwecke des Open Access hat der Autor eine Creative Commons Attribution (CC BY)-Lizenz auf jede vom Autor akzeptierte Manuskriptversion angewendet, die aus dieser Einreichung hervorgeht.
Materialien
| Name | Company | Catalog Number | Comments |
| 0.6 Luer uncoated slide | ibidi | IB-80186 | |
| 25% BSA | Life Technologies | A10008-01 | |
| 6-well plates | Greiner Bio-one | 657160 | |
| Accutase | Life Technologies | A1110501 | Cited as Dissociation reagent |
| Ascorbic acid | Merck | A4544-100G | |
| Aspiration pipette | Sardtedt | 86.1252.011 | |
| B27 supplement | Life Technologies | 17504044 | Cited as Neuronal cell culture supplement (50x) |
| BD FACS DIVA | BD Biosciences | Version 8.0.1 | Cited as flow cytometry software |
| BD LSR Fortessa 5 Laser | BD Biosciences | ||
| bFGF | Life Technologies | PHG0021 | |
| CD34 Microbead kit | Miltenyil Biotec | 30-046-702 | |
| CD34 PE clone 4H11 | Invitrogen | 12-0349-42 | |
| CD34 PerCP-eFluor 710 clone 4H11 | Invitrogen | 44-0349-42 | |
| Cellstar cell-repellent surface 6-well plates | Greiner Bio-one | 657970 | Cited as cell-repellent plate |
| CHIR99021 | Cayman Chemicals | 13122-1mg-CAY | |
| Cryostor CS10 cell cryopreservation | Merck | C2874-100ML | Cited as Cryopreservation solution |
| Dimethyl Sulfoxide | VWR | 200-664-3 | Cited as DMSO |
| DMEM/F-12 | Life Technologies | 10565-018 | |
| DPB Ca2+ Mg2+ | Life Technologies | 14080055 | |
| DPBS | Life Technologies | 14200075 | |
| EASY Strainer 40 μm | Greiner Bio-one | 542040 | |
| EDTA | Life technologies | 15575020 | |
| FcR Blocking Reagent | Miltenyil Biotec | 130-059-901 | |
| Fiji | Version 1.53c | ||
| Flow Jo | Version 10.7.1 | Cited as flow cytometry sanalysis oftware | |
| FLT3L | Peprotech | 300-19-10uG | |
| Fluidic unit | ibidi | 10903 | |
| GlutaMax | Life Technologies | 35050038 | Cited as L-glutamine supplement |
| Ham F-12 | Life Technologies | 11765054 | |
| Holo-transferrin | Merk | T0665-500MG | |
| Human Serum Albumin | Fujifilm UK LTD | 9988 | |
| Ibidi Pump system | ibidi | 10902 | Cited as Pump system |
| IMDM | Life Technologies | 12440053 | |
| Inverted microscope | ioLight/Thisle Scientific | IOL-IO-INVERT | Cited as inverted in-incubator microscope |
| Lyophilised BSA | Merck | A2153-100G | |
| MiniMACS Separator | Miltenyil Biotec | 130-042-102 | Cited as Magnetic separator |
| MS Columns | Miltenyil Biotec | 130-042-201 | Cited as Magnetic column |
| MTG | Merck | M6145-25ML | |
| N2 supplement | Life Technologies | 17502048 | |
| Notebook for pump system | ibidi | 10908 | |
| Paraformaldehyde 37-41% | Fisher Chemicals | F/1501/PB15 | |
| Pastette | Greiner Bio-one | 612398 | |
| Pen/Strep | Gibco | 15070063 | |
| Perfusion Set YELLOW/GREEN: 50 cm, ID 1.6 mm, 10 mL reservoirs | Ibidi | IB-10964 | Cited as Perfusion set |
| Polystyrene Round Bottom Tubes | Falcon | 352008 | Cited as Flow cytometry tubes |
| Prism 9 | Verison 9.4.0 | ||
| Pump control software | ibidi | version 1.6.1 | Cited as Pump software |
| ReLeSR | Stem cell tecchonologies | 5872 | Cited as Detaching solution |
| rhBMP4 | R&D | 314-BP-010 | |
| rhEPO | R&D | 287-TC-500 | |
| rhIGF1 | Peprotech | 100-11-100uG | |
| rhIL11 | Peprotech | 200-11-10uG | |
| rhIL3 | Peprotech | 200-03-10uG | |
| rhIL6 | R&D | 206-IL-010 | |
| rhLaminin-521 | Life technologies | A29248 | Cited as Laminin |
| rhSCF | Life Technologies | PHC2111 | |
| rhTPO | R&D | 288-TPN-025 | |
| rhVEGF | R&D | 293-VE-010 | |
| RLT Lysis Buffer | Qiagen | 79216 | |
| Serial Connector for µ-Slides: Sterile, Sterile | ibidi | IB-10830 | |
| StemPro-CD34 SFM media | Life Technologies | 10639011 | Cited as Serum-Free media for CD34+ cells (SFM-34) |
| StemPro-CD34 Nutrient Supplement | Life Technologies | 10641-025 | Cited as 34 nutrient supplement |
| StemPro hESC SFM | Life Technologies | A1000701 | Cited as Culture media |
| StemPro supplement | Life Technologies | A10006-01 | |
| Vitronectin (VTN-N) recombinant human protein, truncated | Invitrogen | A31804 | |
| Y-27632 dihydrochloride | Tocris | 1254 | Cited as iRock |
| β-Mercaptoethanol | Gibco | 21985023 |
Referenzen
- Copp, A. J. Death before birth: clues from gene knockouts and mutations. Trends in Genetics. 11 (3), 87-93 (1995).
- Ji, R. P., et al. Onset of cardiac function during early mouse embryogenesis coincides with entry of primitive erythroblasts into the embryo proper. Circulation Research. 92 (2), 133-135 (2003).
- Peacock, H. M., Daems, M., Jones, E. A. V. Hemodynamic control of endothelial cell fates in development. Cardiac and Vascular Biology. 8, 127-166 (2021).
- Chong, D. C., Koo, Y., Xu, K., Fu, S., Cleaver, O. Stepwise arteriovenous fate acquisition during mammalian vasculogenesis. Developmental Dynamics. 240 (9), 2153-2165 (2011).
- Jaffredo, T., Gautier, R., Eichmann, A., Dieterlen-Lièvre, F. Intraaortic hemopoietic cells are derived from endothelial cells during ontogeny. Development. 125 (22), 4575-4583 (1998).
- Zovein, A. C., et al. Fate Tracing reveals the endothelial origin of hematopoietic stem cells. Cell Stem Cell. 3 (6), 625-636 (2008).
- Bertrand, J. Y., et al. Haematopoietic stem cells derive directly from aortic endothelium during development. Nature. 464 (7285), 108-111 (2010).
- Boisset, J. C., et al. In vivo imaging of haematopoietic cells emerging from the mouse aortic endothelium. Nature. 464 (7285), 116-120 (2010).
- Adamo, L., et al. Biomechanical forces promote embryonic haematopoiesis. Nature. 459 (7250), 1131-1135 (2009).
- Diaz, M. F., et al. Biomechanical forces promote blood development through prostaglandin E2 and the cAMP-PKA signaling axis. Journal of Experimental Medicine. 212 (5), 665-680 (2015).
- North, T. E., et al. Hematopoietic stem cell development is dependent on blood flow. Cell. 137 (4), 736-748 (2009).
- Lundin, V., et al. YAP regulates hematopoietic stem cell formation in response to the biomechanical forces of blood flow. Developmental Cell. 52 (4), 446.e5-460.e5 (2020).
- Li, J., et al. Mimicry of embryonic circulation enhances the hoxa hemogenic niche and human blood development. Cell Reports. 40 (11), 111339 (2022).
- Azzoni, E., et al. The onset of circulation triggers a metabolic switch required for endothelial to hematopoietic transition. Cell Reports. 37 (11), 110103 (2021).
- Li, Y. S. J., Haga, J. H., Chien, S. Molecular basis of the effects of shear stress on vascular endothelial cells. Journal of Biomechanics. 38 (10), 1949-1971 (2005).
- Batsivari, A., et al. Understanding hematopoietic stem cell development through functional correlation of their proliferative status with the intra-aortic cluster architecture. Stem Cell Reports. 8 (6), 1549-1562 (2017).
- Canu, G., et al. Analysis of endothelial-to-haematopoietic transition at the single cell level identifies cell cycle regulation as a driver of differentiation. Genome Biology. 21 (1), 157 (2020).
- Luo, W., et al. Arterialization requires the timely suppression of cell growth. Nature. 589 (7842), 437-441 (2020).
- Yang, C. -. T., et al. Activation of KLF1 enhances the differentiation and maturation of red blood cells from human pluripotent stem cells. Stem Cells. 35 (4), 886-897 (2017).
- Lopez-Yrigoyen, M., et al. A human iPSC line capable of differentiating into functional macrophages expressing ZsGreen: A tool for the study and in vivo tracking of therapeutic cells. Philosophical Transactions of the Royal Society B: Biological Sciences. 373 (1750), 20170219 (2018).
- Lopez-Yrigoyen, M., et al. Production and characterization of human macrophages from pluripotent stem cells. Journal of Visualized Experiments. 2020 (158), (2020).
- Fidanza, A., et al. Single cell analyses and machine learning define hematopoietic progenitor and HSC-like cells derived from human PSCs. Blood. 136 (25), 2893-2904 (2020).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Takahashi, K., et al. Induction of pluripotent stem cells from adult human fibroblasts by defined factors. Cell. 131 (5), 861-872 (2007).
- North, T. E., et al. Hematopoietic stem cell development is dependent on blood flow. Cell. 137 (4), 736-748 (2009).
- Nguyen, J., Lin, Y. Y., Gerecht, S. The next generation of endothelial differentiation: Tissue-specific ECs. Cell Stem Cell. 28 (7), 1188-1204 (2021).
- Petazzi, P., et al. Arterial cells support the development of human hematopoietic progenitors in vitro via secretion of IGFBP2. bioRxiv. , (2022).
- Crosse, E. I., et al. Multi-layered spatial transcriptomics identify secretory factors promoting human hematopoietic stem cell development. Cell Stem Cell. 27 (5), 822 (2020).
- Calvanese, V., et al. Mapping human haematopoietic stem cells from haemogenic endothelium to birth. Nature. 604 (7906), 534-540 (2022).
- Zeng, Y., et al. Tracing the first hematopoietic stem cell generation in human embryo by single-cell RNA sequencing. Cell Research. 29 (11), 881-894 (2019).
- Hwa, J. J., et al. Abnormal arterial-venous fusions and fate specification in mouse embryos lacking blood flow. Scientific Reports. 7 (1), 11965 (2017).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten