Verwendung von Lebendzell-STED-Bildgebung zur Visualisierung der mitochondrialen Innenmembran-Ultrastruktur in neuronalen Zellmodellen
In diesem Artikel
Zusammenfassung
Dieses Protokoll stellt einen Arbeitsablauf für die Vermehrung, Differenzierung und Färbung von kultivierten SH-SY5Y-Zellen und primären Hippocampusneuronen der Ratte für die Visualisierung und Analyse der mitochondrialen Ultrastruktur unter Verwendung der stimulierten Emissionsverarmungsmikroskopie (STED) dar.
Zusammenfassung
Mitochondrien spielen viele wesentliche Rollen in der Zelle, darunter die Energieproduktion, die Regulierung derCa2+ -Homöostase, die Lipidbiosynthese und die Produktion von reaktiven Sauerstoffspezies (ROS). Diese Mitochondrien-vermittelten Prozesse übernehmen spezialisierte Rollen in Neuronen, koordinieren den aeroben Stoffwechsel, um den hohen Energiebedarf dieser Zellen zu decken, modulieren die Ca2+ -Signalgebung, liefern Lipide für das Wachstum und die Regeneration von Axonen und stimmen die ROS-Produktion auf die neuronale Entwicklung und Funktion ab. Die mitochondriale Dysfunktion ist daher ein zentraler Treiber bei neurodegenerativen Erkrankungen. Struktur und Funktion der Mitochondrien sind untrennbar miteinander verbunden. Die morphologisch komplexe innere Membran mit strukturellen Infolds, die Cristae genannt werden, beherbergt viele molekulare Systeme, die die charakteristischen Prozesse des Mitochondriums ausführen. Die architektonischen Merkmale der inneren Membran sind ultrastrukturell und daher zu klein, um mit herkömmlicher beugungsbegrenzter aufgelöster Mikroskopie sichtbar gemacht zu werden. Daher stammen die meisten Erkenntnisse über die mitochondriale Ultrastruktur aus der Elektronenmikroskopie an fixierten Proben. Neue Technologien in der superauflösenden Fluoreszenzmikroskopie bieten jedoch jetzt eine Auflösung von bis zu zehn Nanometern und ermöglichen die Visualisierung ultrastruktureller Merkmale in lebenden Zellen. Die hochauflösende Bildgebung bietet daher eine noch nie dagewesene Möglichkeit, feine Details der mitochondrialen Struktur, der nanoskaligen Proteinverteilungen und der Cristae-Dynamik direkt abzubilden, was grundlegende neue Erkenntnisse liefert, die Mitochondrien mit der menschlichen Gesundheit und Krankheit in Verbindung bringen. Dieses Protokoll stellt die Verwendung von hochauflösender Mikroskopie mit stimulierter Emissionsverarmung (STED) vor, um die mitochondriale Ultrastruktur von lebenden menschlichen Neuroblastomzellen und primären Rattenneuronen zu visualisieren. Dieses Verfahren ist in fünf Abschnitte unterteilt: (1) Wachstum und Differenzierung der SH-SY5Y-Zelllinie, (2) Isolierung, Plattierung und Wachstum von primären Hippocampusneuronen der Ratte, (3) Verfahren zur Färbung von Zellen für die lebende STED-Bildgebung, (4) Verfahren für STED-Experimente mit lebenden Zellen unter Verwendung eines STED-Mikroskops als Referenz und (5) Anleitung zur Segmentierung und Bildverarbeitung anhand von Beispielen zur Messung und Quantifizierung morphologischer Merkmale der inneren Membran.
Einleitung
Mitochondrien sind eukaryotische Organellen endosymbiontischen Ursprungs, die für die Regulierung mehrerer zellulärer Schlüsselprozesse verantwortlich sind, darunter der intermediäre Stoffwechsel und die ATP-Produktion, die Ionenhomöostase, die Lipidbiosynthese und der programmierte Zelltod (Apoptose). Diese Organellen sind topologisch komplex und enthalten ein Doppelmembransystem, das mehrere Unterkompartimente1 etabliert (Abbildung 1A). Die äußere mitochondriale Membran (OMM) interagiert mit dem Zytosol und stellt direkte interorganelle Kontakte her 2,3. Die innere mitochondriale Membran (IMM) ist eine energiesparende Membran, die Ionengradienten aufrechterhält, die hauptsächlich als elektrisches Membranpotential (ΔΨm) gespeichert sind, um die ATP-Synthese und andere energieintensive Prozesse anzutreiben 4,5. Die IMM ist weiter unterteilt in die innere Grenzmembran (IBM), die eng an die OMM angrenzt, und hervorstehende Strukturen, die als Cristae bezeichnet werden und von der Cristae-Membran (CM) gebunden werden. Diese Membran grenzt das innerste Matrixkompartiment vom intrakristallalen Raum (ICS) und dem Intermembranraum (IMS) ab.
Mitochondrien haben eine dynamische Morphologie, die auf kontinuierlichen und ausgewogenen Spaltungs- und Fusionsprozessen basiert, die von Mechanoenzymen der Dynamin-Superfamilie gesteuert werden6. Die Fusion ermöglicht eine erhöhte Konnektivität und Bildung von retikulären Netzwerken, während die Spaltung zu einer Fragmentierung der Mitochondrien führt und die Entfernung beschädigter Mitochondrien durch Mitophagie ermöglicht7. Die mitochondriale Morphologie variiert je nach Gewebetyp8 und Entwicklungsstadium9 und wird so reguliert, dass sich die Zellen an Faktoren wie Energiebedarf10,11 und Stressoren12 anpassen können. Morphometrische Standardmerkmale von Mitochondrien, wie z. B. das Ausmaß der Netzwerkbildung (vernetzt vs. fragmentiert), der Umfang, die Fläche, das Volumen, die Länge (Seitenverhältnis), die Rundheit und der Grad der Verzweigung, können durch optische Standardmikroskopie gemessen und quantifiziert werden, da die Größe dieser Merkmale größer ist als die Beugungsgrenze des Lichts (~200 nm)13.
Die Cristae-Architektur definiert die innere Struktur der Mitochondrien (Abbildung 1B). Die Vielfalt der Cristae-Morphologien kann grob in flach (lamellär oder diskoidal) oder tubulär-vesikulär eingeteilt werden14. Alle Cristae sind an der IBM durch röhrenförmige oder schlitzartige Strukturen verbunden, die als Cristae Junctions (CJs) bezeichnet werden und dazu dienen können, das IMS vom ICS und das IBM vom CM15 zu trennen. Die Morphologie der Cristae wird durch Schlüsselproteinkomplexe des IMM reguliert, einschließlich (1) der mitochondrialen Kontaktstelle und des Cristae-Organisationssystems (MICOS), das sich an CJs befindet und die IMM-OMM-Kontakte16 stabilisiert, (2) der GTPase der Optikusatrophie 1 (OPA1), die den Umbau der Cristae 17,18,19 reguliert, und (3) der F1FO ATP-Synthase, die stabilisierende oligomere Anordnungen an den Cristae-Spitzen (CTs) bildet20, 21. Anmelden Darüber hinaus ist das IMM mit den nicht-zweischichtigen Phospholipiden Phosphatidylethanolamin und Cardiolipin angereichert, die das stark gekrümmte IMM22 stabilisieren. Cristae sind auch dynamisch und zeigen morphologische Veränderungen unter verschiedenen Bedingungen, wie z. B. verschiedenen Stoffwechselzuständen 23,24, mit unterschiedlichen respiratorischen Substraten 25, unter Hunger und oxidativem Stress 26,27, mit Apoptose 28,29 und mit Alterung 30. Kürzlich wurde gezeigt, dass Cristae größere Umbauereignisse auf einer Zeitskala von Sekunden durchlaufen können, was ihre dynamische Natur unterstreicht31. Mehrere Merkmale von Cristae können quantifiziert werden, einschließlich der Abmessungen von Strukturen innerhalb einzelner Cristae (z. B. CJ-Breite, Crista-Länge und -Breite) und Parameter, die einzelne Crista mit anderen Strukturen in Beziehung setzen (z. B. Intra-Cristae-Abstand und Cristae-Einfallswinkel relativ zum OMM)32. Diese quantifizierbaren Cristae-Parameter zeigen eine direkte Korrelation mit der Funktion. Zum Beispiel steht das Ausmaß der mitochondrialen ATP-Produktion in einem positiven Zusammenhang mit der Häufigkeit von Cristae, quantifiziert als Cristae-Dichte oder Cristae-Anzahl, die auf ein anderes Merkmal normiert ist (z. B. Cristae pro OMM-Fläche)33,34,35. Da die IMM-Morphologie durch nanoskalige Merkmale definiert ist, umfasst sie eine mitochondriale Ultrastruktur, die bildgebende Verfahren erfordert, die eine Auflösung bieten, die größer als die Lichtbeugungsgrenze ist. Wie im Folgenden beschrieben, umfassen solche Techniken die Elektronenmikroskopie und die superauflösende Mikroskopie (Nanoskopie).
Die Nerven- und Gliazellen des zentralen Nervensystems (ZNS) sind in besonderem Maße auf die Funktion der Mitochondrien angewiesen. Im Durchschnitt macht das Gehirn nur 2 % des gesamten Körpergewichts aus, verwertet aber 25 % der gesamten Körperglukose und ist für 20 % des Sauerstoffverbrauchs des Körpers verantwortlich, was es anfällig für Beeinträchtigungen des Energiestoffwechsels macht36. Progressive neurodegenerative Erkrankungen (NDs), einschließlich der Alzheimer-Krankheit (AD), der amyotrophen Lateralsklerose (ALS), der Huntington-Krankheit (HD), der Multiplen Sklerose (MS) und der Parkinson-Krankheit (PD), gehören zu den bisher am besten untersuchten Pathologien, wobei die Forschungsbemühungen vom Verständnis der molekularen Grundlagen dieser Krankheiten bis hin zur Suche nach potenzieller therapeutischer Prävention und Interventionen reichen. NDs sind mit erhöhtem oxidativem Stress verbunden, der zum Teil auf reaktive Sauerstoffspezies (ROS) zurückzuführen ist, die von der mitochondrialen Elektronentransportkette (ETC) erzeugt werden37, sowie auf eine veränderte mitochondriale Kalziumverarbeitung38 und den mitochondrialen Lipidstoffwechsel39. Diese physiologischen Veränderungen werden von festgestellten Defekten in der mitochondrialen Morphologie begleitet, die mit AD 40,41,42,43,44, ALS45,46, HD47,48,49, MS 50 und PD 51,52,53 assoziiert sind . Diese strukturellen und funktionellen Defekte können durch komplexe Ursache-Wirkungs-Beziehungen gekoppelt werden. Da beispielsweise die Cristae-Morphologie die OXPHOS-Enzyme54 stabilisiert, werden mitochondriale ROS nicht nur vom ETC erzeugt, sondern schädigen auch die Infrastruktur, in der sich die ETC befindet, und fördern einen Feed-Forward-ROS-Zyklus, der die Anfälligkeit für oxidative Schäden erhöht. Darüber hinaus wurde gezeigt, dass die Desorganisation von Cristae Prozesse wie die Freisetzung von mitochondrialer DNA (mtDNA) und Entzündungswege auslöst, die mit Autoimmun-, Stoffwechsel- und altersbedingten Erkrankungen verbunden sind55. Daher ist die Analyse der mitochondrialen Struktur der Schlüssel zu einem vollständigen Verständnis von NDs und ihren molekularen Grundlagen.
Gängige Methoden zur Betrachtung von Cristae, darunter die Transmissionselektronenmikroskopie, die Elektronentomographie und die Kryo-Elektronentomographie (Kryo-ET) sowie die Röntgentomographie, insbesondere die Kryo-Soft-Röntgentomographie, haben wichtige Erkenntnisse zutage gefördert und arbeiten mit einer Vielzahl von Probentypen 56,57,58,59,60. Trotz der jüngsten Fortschritte bei der besseren Beobachtung der Organellen-Ultrastruktur sind diese Methoden immer noch mit dem Vorbehalt verbunden, dass sie eine Probenfixierung erfordern und daher die Echtzeitdynamik von Cristae nicht direkt erfassen können. Die hochauflösende Fluoreszenzmikroskopie, insbesondere in Form der strukturierten Beleuchtungsmikroskopie (SIM), der stochastischen optischen Rekonstruktionsmikroskopie (STORM), der photoaktivierten Lokalisierungsmikroskopie (PALM), der Expansionsmikroskopie (ExM) und der Stimulated Emission Depletion (STED)-Mikroskopie, ist zu einer beliebten Methode geworden, um Strukturen zu betrachten, die eine Auflösung unterhalb der Beugungsgrenze erfordern, die klassische Methoden der optischen Mikroskopie einschränkt. Wenn ExM in Verbindung mit einer anderen Super-Resolution-Technik verwendet wird, sind die Ergebnisse beeindruckend, aber die Probe muss in einem Gel61 fixiert und gefärbt werden. Im Vergleich dazu wurden SIM, PALM/STORM und STED alle erfolgreich mit lebenden Proben eingesetzt, und neue und vielversprechende Farbstoffe, die das IMM im Allgemeinen färben, bieten einen neuartigen und einfachen Ansatz für die Live-Bildgebung der Dynamik von Mitochondria cristae 62,63,64,65,66. Jüngste Fortschritte bei Lebendfarbstoffen für die STED-Bildgebung haben die Helligkeit und Photostabilität der Farbstoffe verbessert, und diese Farbstoffe zielen mit einem höheren Grad an Spezifität als ihre Vorgänger auf das IMM ab. Diese Entwicklungen ermöglichen die Sammlung von Langzeit-Zeitraffer- und Z-Stack-Experimenten mit hochauflösender Bildgebung und öffnen die Tür zu einer besseren Analyse der mitochondrialen Ultrastruktur und Dynamik in lebenden Zellen.
In dieser Arbeit werden Protokolle für die Bildgebung von lebenden Zellen von undifferenzierten und differenzierten SH-SY5Y-Zellen bereitgestellt, die mit dem Farbstoff PKmito Orange (PKMO) unter Verwendung von STED63 gefärbt wurden. Die SH-SY5Y-Zelllinie ist ein dreimal subkloniertes Derivat der elterlichen Zelllinie SK-N-SH, das aus einer Knochenmarkbiopsie des metastasierten Neuroblastoms67,68,69,70 gewonnen wurde. Diese Zelllinie ist ein häufig verwendetes In-vitro-Modell in der ND-Forschung, insbesondere bei Krankheiten wie AD, HD und PD, bei denen eine mitochondriale Dysfunktion stark beteiligt ist 10,43,71,72,73. Die Fähigkeit, SH-SY5Y-Zellen durch Manipulation von Kulturmedien in Zellen mit einem neuronenähnlichen Phänotyp zu differenzieren, hat sich als geeignetes Modell für die neurowissenschaftliche Forschung erwiesen, ohne auf primäre neuronale Zellen angewiesen zu sein10,74. In diesem Protokoll wurde dem Zellkulturmedium Retinsäure (RA) zugesetzt, um die Differenzierung von SH-SY5Y-Zellen zu induzieren. RA ist ein Vitamin-A-Derivat und reguliert nachweislich den Zellzyklus und fördert die Expression von Transkriptionsfaktoren, die die neuronale Differenzierung regulieren75. Ein Protokoll für die Kultivierung und Bildgebung von lebenden Zellen von Neuronen, die aus dem Hippocampus der Ratte isoliert wurden, wird ebenfalls bereitgestellt. Es wurde gezeigt, dass der Hippocampus von der mitochondrialen Degeneration betroffen ist und zusammen mit dem Kortex eine wichtige Rolle beim Altern und ND spielt 76,77,78,79,80.
Protokoll
1. Vermehrung und Differenzierung von SH-SY5Y-Zellen
- Vorbereitung von Medien für Zellwachstum und -erhalt
- Bereiten Sie ein vollständiges, glukosereiches Dulbecco's Modified Eagle's Medium (DMEM, 4,5 g/l D-Glukose, 4 mM L-Glutamin, 110 mg/l Natriumpyruvat) zu, ergänzt mit einem letzten 1%igen (v/v) Antibiotikum-Antimykotikum (10.000 Einheiten/ml Penicillin, 10.000 μg/ml Streptomycin und 25 μg/ml Amphotericin B) und unterschiedlichen Mengen an fötalem Kälberserum (FBS) (siehe Materialtabelle). Die FBS-Mengen in den Differenzierungsmedien variieren zwischen den endgültigen 10 %, 5 % oder 2 % (v/v) FBS.
- Wartung der Zellen
- Die Zellen werden in DMEM mit 10 % (v/v) FBS ergänzt und bei 37 °C und 5 % CO2 platziert, dann zur Differenzierung in DMEM mit 5 % (v/v) FBS eingesät. Gefrorene Zellvorräte wurden in FBS mit 10% (v/v) Dimethylsulfoxid (DMSO) bei 1-2 x 107 Zellen/ml gelagert.
- Herstellung von Retinsäure (RA)
- 7,51 mg all-trans-RA (siehe Materialtabelle) werden in 5 ml frisch zubereitetem 95%igem Ethanol gelöst, um 5 mM Stammlösung zu erhalten. Die Konzentration mit einer Absorption von 350 nm (ɛ = 44.300 M-1 cm-1), die aus dem Produktinformationsblatt des Protokolls81 des Herstellers entnommen wurde, wird unter Verwendung einer Verdünnung der Stammlösung bei 5 μM in Ethanol überprüft. 5 mM Brühe lichtgeschützt bei 4 °C bis zu 6 Wochen lagern.
- Herstellung von Poly-D-Lysin für die Deckglasbeschichtung
HINWEIS: Das Poly-D-Lysin (PDL)-Produktprotokoll, das im Abschnitt "Dokumente und Downloads" auf der Herstellerseite82 zu finden ist, enthält Informationen für die Beschichtung einer Vielzahl von Kulturgefäßen.- Dieses Protokoll umfasst Volumina, die auf einem 2-Well-Kammergefäß mit einer Fläche von 4cm2 pro Well mit sterilen #1.5 Borosilikat-Deckglasböden basieren (siehe Materialtabelle). Verdünnen Sie die PDL-Stammlösung zweifach auf 50 μg/ml mit PBS von Dulbecco (DPBS; kein Kalzium, kein Magnesium).
HINWEIS: #1.5 oder #1.5H Deckglas sind beides akzeptable Dicken, die für die Bildqualität unerlässlich sind. Andere Dicken führen zu sphärischen Aberrationen und sollten vermieden werden.
- Dieses Protokoll umfasst Volumina, die auf einem 2-Well-Kammergefäß mit einer Fläche von 4cm2 pro Well mit sterilen #1.5 Borosilikat-Deckglasböden basieren (siehe Materialtabelle). Verdünnen Sie die PDL-Stammlösung zweifach auf 50 μg/ml mit PBS von Dulbecco (DPBS; kein Kalzium, kein Magnesium).
- Deckglasbeschichtung mit PDL
HINWEIS: Deckgläser können zur weiteren Sterilisation 10-15 Minuten lang in einer Biosicherheitswerkbank ultraviolettem (UV) Licht ausgesetzt werden.- Tragen Sie 1,2 ml 50 μg/ml PDL-Lösung auf jede Vertiefung steriler Deckgläser in einem Zellkulturschrank auf und inkubieren Sie 1 Stunde lang bei Raumtemperatur. Entfernen Sie die PDL-Lösung und spülen Sie sie dreimal mit 3,6 ml destilliertem Wasser ab. Nach Beendigung der letzten Wäsche die beschichtete Kammer 2 h an der Luft trocknen lassen, bevor sie abgespült und sofort verwendet oder in einem luftdichten Behälter bei 4 °C bis zu 2 Wochen gelagert wird.
HINWEIS: Spülen Sie die Deckgläser gründlich aus, da überschüssiges PDL für die Zellen giftig sein kann.
- Tragen Sie 1,2 ml 50 μg/ml PDL-Lösung auf jede Vertiefung steriler Deckgläser in einem Zellkulturschrank auf und inkubieren Sie 1 Stunde lang bei Raumtemperatur. Entfernen Sie die PDL-Lösung und spülen Sie sie dreimal mit 3,6 ml destilliertem Wasser ab. Nach Beendigung der letzten Wäsche die beschichtete Kammer 2 h an der Luft trocknen lassen, bevor sie abgespült und sofort verwendet oder in einem luftdichten Behälter bei 4 °C bis zu 2 Wochen gelagert wird.
- Differenzierung von SH-SY5Y-Zellen mit RA
HINWEIS: Verwenden Sie keine Zellen oberhalb von Passage 15. Die Zellen werden mit 80%-90% Konfluenz durchgelassen. Die Differenzierungsverfahren unterscheiden sich, folgen aber ähnlichen Schritten. Eine zusätzliche Differenzierung von Neuroblastomen zu reifen Neuronen wird durch eine weitere Behandlung mit dem Brain-derived Neurotrophic Factor (BDNF)68,83,84,85 erreicht, wurde aber in diesem Protokoll nicht durchgeführt.
OPTIONAL: Zellen mindestens 24 Stunden vor der Aussaat auf Deckglas aufstellen. Um Zellen aus gefrorenen Vorräten vorzubereiten, tauen Sie schnell 1 ml gefrorene Durchstechflasche mit Zellen auf und fügen Sie 9 ml vorgewärmtes Medium hinzu, das mit 10 % FBS ergänzt ist, schleudern Sie dann 10 Minuten lang (bei Raumtemperatur) bei 350 x g herunter und verwerfen Sie den Überstand, um DMSO zu entfernen. Resuspendieren Sie das Zellpellet in 5 ml vorgewärmtem Medium und Saatzellen in einem T-25-Kolben. Sobald die Zellen eine Konfluenz von 80 % bis 90 % erreicht haben, lassen Sie die Zellen durch, indem Sie sie zählen und gegebenenfalls zur Differenzierung aussäen.- Tag 0: Samenzellen.
- Säen Sie die Zellen auf ein gekammertes Deckglas aus gefrorenen Vorräten oder einen Arbeitskolben. Verwenden Sie eine Aussaatdichte von 1,5 x 104 Zellen/cm2.
HINWEIS: Für eine einzelne Vertiefung in einem Standard-2-Well-Deckglas mit einer Kulturfläche von 4cm2 werden 6,0 x 104 Zellen benötigt. Zellen, die undifferenziert bleiben, sollten mit DMEM ausgesät werden, das mit 10 % (v/v) FBS ergänzt wird, und Zellen, die differenziert werden, sollten mit DMEM ausgesät werden, das mit 5 % (v/v) FBS ergänzt wird.
- Säen Sie die Zellen auf ein gekammertes Deckglas aus gefrorenen Vorräten oder einen Arbeitskolben. Verwenden Sie eine Aussaatdichte von 1,5 x 104 Zellen/cm2.
- Tag 1: Beginn der RA-Differenzierungsbehandlung.
- Bereiten Sie DMEM vor, das mit 5 % (v/v) FBS, 1 % (v/v) Antibiotikum-Antimykotikum und einer Endkonzentration von 10 μM RA oder Ethanol desselben Additivvolumens ergänzt wird, um als Vehikelkontrolle für dieses Differenzierungsverfahren zu dienen. Entfernen Sie das Medium aus dem für die Aussaat verwendeten Kammerglas, spülen Sie es mit 1x PBS ab und geben Sie das neue DMEM in die Vertiefungen.
- Tag 3: Ersetzen Sie Medien durch frische, RA- oder ethanolhaltige Medien.
- Entfernen Sie das Medium von Tag 1 und ersetzen Sie es durch frisches Medium, das mit 2 % (v/v) FBS, 1 % (v/v) Antibiotika-Antimykotikum und entweder 10 μM RA oder 95 % Ethanol desselben Additivvolumens ergänzt ist, um als Vehikelkontrolle für dieses Differenzierungsverfahren zu dienen. Entfernen Sie das Medium aus dem für die Aussaat verwendeten Kammerglas, spülen Sie es mit 1x PBS ab und geben Sie neue Medien in die Vertiefungen.
- Tag 6: Führen Sie die Bildgebung der Zellen durch.
ANMERKUNG: Die Zelldifferenzierungszeiten variieren je nach Protokoll, aber eine sechstägige Exposition gegenüber RA ist ausreichend, um einen neuronenähnlichen Phänotyp in SH-SY5Y-Zellen zu induzieren86.- Führen Sie eine Live-Bildgebung durch, mit Details in den Abschnitten 3 und 4 (Abbildung 2).
- Tag 0: Samenzellen.
2. Primäre Neuronenkultur im Hippocampus der Ratte
- Materialvorbereitung für die Isolierung von Neuronen im Hippocampus der Ratte.
- Bereiten Sie frisches, ergänztes DMEM zu.
- Filtersterilisieren Sie eine Mischung aus DMEM (hoher Glukosegehalt, kein Natriumpyruvat), ergänzt mit 10 % (v/v) hitzeinaktiviertem FBS, 1 % (v/v) Natriumpyruvatlösung und 1 % (v/v) Penicillin-Streptomycin (10.000 U/ml). Bis zu 2 Wochen bei 4 °C lagern.
- Bereiten Sie frisches, ergänztes Neuronenwachstumsmedium vor.
- Filtersterilisieren Sie eine Mischung aus Neuronenwachstumsmedien, die mit 2 % (v/v) B27-Ergänzung, 0,25 % (v/v) Glutamin-Präparat und 1 % (v/v) Penicillin-Streptomycin ergänzt wurden (siehe Materialtabelle). Bis zu 2 Wochen bei 4 °C lagern.
- Bereiten Sie frisches, ergänztes DMEM zu.
- Bereiten Sie die primäre Neuronenkultur des Hippocampus vor.
- Vorbereiten der primären Hippocampus-Neuronenkultur im Anschluss an die zuvor veröffentlichte Arbeit87 und aus dem Produktprotokoll auf der Website des Herstellers, aus dem der präparierte E18-Ratten-Hippocampus88 gewonnen wird (siehe Materialtabelle). Dieses Protokoll führt zu einer überwiegend neuronalen Population mit <2% Astrozyten.
HINWEIS: Die Hibernate-Medien, mit denen dieses Gewebe versendet wird, werden für zukünftige Schritte in diesem Protokoll verwendet. Werfen Sie es nicht weg. - Bereiten Sie Materialien und Medien für die Gewebedissoziation vor.
- Flammen Sie eine Pasteurpipette ab, um den Durchmesser der Öffnung zu verringern, und bewahren Sie sie in Folie auf, um eine Kontamination bis zur Verwendung zu vermeiden. Das vorbereitete DMEM, 1X Hank's Buffered Salt Solution (HBSS) und das Neuronenwachstumsmedium auf 37 °C vorwärmen. Geben Sie 2 Flocken DNAase mit einer sterilen Pinzette in ein konisches 15-ml-Röhrchen.
- Führen Sie eine Gewebedissoziation durch.
- Entfernen Sie so viel wie möglich von dem überwinternden Medium, in dem der präparierte E18-Ratten-Hippocampus aufbewahrt wird, bevor Sie das Gewebe in das konische 15-ml-Röhrchen mit DNAase geben und kurz bei 37 °C inkubieren. Fügen Sie 900 μl 1X HBSS gefolgt von 100 μl 0,5% Trypsin hinzu. Inkubieren Sie das Gewebe bei 37 °C für 15 Minuten.
HINWEIS: PDL-beschichtete Platten können aus dem Lager genommen und in einen Inkubator gelegt werden, bis sie während dieser Inkubationszeit verwendet werden.
- Entfernen Sie so viel wie möglich von dem überwinternden Medium, in dem der präparierte E18-Ratten-Hippocampus aufbewahrt wird, bevor Sie das Gewebe in das konische 15-ml-Röhrchen mit DNAase geben und kurz bei 37 °C inkubieren. Fügen Sie 900 μl 1X HBSS gefolgt von 100 μl 0,5% Trypsin hinzu. Inkubieren Sie das Gewebe bei 37 °C für 15 Minuten.
- Führen Sie eine Gewebehomogenisierung und Zellzählung durch.
- Nach der Inkubation mit Trypsin wird das Medium entfernt und 1 ml vorgewärmte Hibernat-Media-DNAase aus dem vorherigen Schritt in das Gewebe gegeben und mit der Pasteur-Pipette homogenisiert. Das Medium sieht undurchsichtig aus und wird dann allmählich klar, wenn die Homogenisierung fortschreitet.
- Geben Sie dissoziierte Neuronen in ein neues Röhrchen mit 4 ml vorgewärmtem DMEM und zählen Sie dann die Zellen mit einem Zellzähler.
- Führen Sie die Zellplattierung und das Wachstum von Primärzellen durch.
- Plattenzellen mit einer Dichte von ca. 1,65 x 104 Zellen/cm2 in DMEM. Für ein 2-Well-Kammer-Deckglas (4 cm 2) werden 65.000 – 70.000 Zellen pro Well mit2 mL DMEM ausgesät. Die Zellen werden 2 h lang bei 37 °C und 5 %CO2 inkubiert, bevor sie auf Adhärenz geprüft werden. Sobald die Zellen anfangen zu haften, entfernen Sie 1 ml des Mediums und ersetzen Sie es durch das gleiche Volumen des Ruhezustands, und rühren Sie dann vorsichtig um, um es zu mischen. Sobald das Medium gemischt ist, wiederholen Sie diesen Vorgang und entfernen Sie die Hälfte des vorhandenen Mediums und ersetzen Sie es durch das gleiche Volumen des Neuronenwachstumsmediums, dann mischen Sie es vorsichtig.
HINWEIS: Der Tag der Beschichtung gilt als Day in vitro (DIV) 0, und die Zellen sind bereit für die Abbildung bei DIV 7 (Abbildung 2).
- Plattenzellen mit einer Dichte von ca. 1,65 x 104 Zellen/cm2 in DMEM. Für ein 2-Well-Kammer-Deckglas (4 cm 2) werden 65.000 – 70.000 Zellen pro Well mit2 mL DMEM ausgesät. Die Zellen werden 2 h lang bei 37 °C und 5 %CO2 inkubiert, bevor sie auf Adhärenz geprüft werden. Sobald die Zellen anfangen zu haften, entfernen Sie 1 ml des Mediums und ersetzen Sie es durch das gleiche Volumen des Ruhezustands, und rühren Sie dann vorsichtig um, um es zu mischen. Sobald das Medium gemischt ist, wiederholen Sie diesen Vorgang und entfernen Sie die Hälfte des vorhandenen Mediums und ersetzen Sie es durch das gleiche Volumen des Neuronenwachstumsmediums, dann mischen Sie es vorsichtig.
- Vorbereiten der primären Hippocampus-Neuronenkultur im Anschluss an die zuvor veröffentlichte Arbeit87 und aus dem Produktprotokoll auf der Website des Herstellers, aus dem der präparierte E18-Ratten-Hippocampus88 gewonnen wird (siehe Materialtabelle). Dieses Protokoll führt zu einer überwiegend neuronalen Population mit <2% Astrozyten.
3. Vorbereitung von Zellen für die Bildgebung lebender Zellen
HINWEIS: Zelltypen und Herkunft (d. h. kultivierte und primäre Zellen) können sich in den Anforderungen an die Färbung unterscheiden. Weitere Informationen finden Sie in den veröffentlichten Berichten62,63.
- Zubereitung von PKmito Orange
HINWEIS: Andere Farbstoffe, die im Allgemeinen das IMM färben, wurden mit64,65,66 beschrieben und sind im Handel erhältlich. PKMO ist das einzige, das in diesem Protokoll verwendet wird.- Resuspendieren Sie das Pulver PKMO (siehe Materialtabelle) in DMSO gemäß den Anweisungen des Herstellers89. Saugen Sie das Medium aus den Zellen ab und waschen Sie es in vorgewärmten phenolroten freien Medien. Bereiten Sie einen Vorrat an PKMO in vorgewärmtem, phenolrotfreiem DMEM vor, das je nach Differenzierungszustand mit 2 % (v/v) FBS oder 10 % (v/v) ergänzt wird, HEPES auf eine Endkonzentration von 20 mM und 1 % (v/v) antibiotisch-antimykotisch, bevor Sie die Zellen gemäß den Anweisungen des Herstellers färben. Diese Formulierung ohne PKMO ist das Bildgebungsmedium für lebende Zellen.
- Zellfärbung mit PKMO
- Die Zellen werden mit dem Farbstoff bei 37 °C und 5 % CO2 30 min lang inkubiert. Die Zellen werden dreimal mit lebenden Bildgebungsmedien gewaschen und für die letzte Wäsche 30 Minuten lang bei 37 °C und 5 % CO2 inkubiert.
- Fügen Sie frische, vorgewärmte Live-Cell-Imaging-Medien hinzu. Die Zellen sind nun bereit für die Bildgebung.
HINWEIS: Akute Behandlungen (z. B. Medikamente und/oder Stressoren) werden, wenn sie angewendet werden, vor der Live-Bildgebung hinzugefügt. Weitere Informationen finden Sie im Abschnitt "Diskussion" und in der ergänzenden Abbildung 1.
4. Abbildung lebender Zellen mittels STED-Mikroskopie
HINWEIS: Dieses Protokoll verwendet ein STED-System, das auf einem inversen Mikroskop basiert, wobei das System in der Materialtabelle angegeben ist. Dieses System ist mit gepulsten Anregungslasern (561 nm Laser mit Nennleistung ~300 μW) und einem gepulsten 775 nm STED-Verarmungslaser (Nennleistung 1,2 W), einem stufenlos einstellbaren Galvano-Scanner und einem 615/20 nm filterbasierten Avalanche-Photodiodendetektor (APD) ausgestattet. Hier kommt eine 100x/1,40 Ölimmersionslinse für STED zum Einsatz. Für die Bildaufnahme wird die Lightbox-Software verwendet. Alle bereitgestellten Details beziehen sich direkt auf diese Software und das System-Setup.
- Allgemeine Richtlinien und Schritte für die Bildgebung
- Verwenden Sie einen Stage-Top-Inkubator oder eine Klimakammer, um die Lebensfähigkeit der Zellen aufrechtzuerhalten, aber auch kurzfristige Experimente bei Raumtemperatur sind akzeptabel. Diese Schritte sind spezifisch für das oben beschriebene STED-Setup.
- Wählen Sie die Laser- und Filtersets für die Bildgebung aus.
- Verwenden Sie Parameter für einen orangefarbenen Farbstoff, indem Sie den/die bei der Färbung verwendete(n) Farbstoff(e) aus der Farbstoffliste auswählen oder denjenigen mit spektralen Eigenschaften, der/die dem/den verwendeten Farbstoff(en) am nächsten kommen. Aktivieren Sie diese, indem Sie auf sie doppelklicken oder sie in die Beispielliste ziehen, wo "Farbstoff(e) hierher ziehen" angezeigt wird.
- Wählen Sie einen Bereich aus, der abgebildet werden soll.
- Erstellen Sie in einer Übersicht eine Region of Interest (ROI) um ein Mitochondrium von Interesse, indem Sie die rechteckige ROI-Schaltfläche auswählen und klicken und ziehen, um die Region zu formen. Die ROI kann mithilfe der ROI-Ecken oder gekrümmten Kanten, die angezeigt werden, wenn Sie mit der Maus über eine Ecke fahren, in der Größe geändert und gedreht werden.
HINWEIS: Eine Zusammenfassung der vorgeschlagenen Bildgebungsparameter finden Sie in Tabelle 1. Diese Parameter wurden empirisch unter Verwendung derjenigen angepasst, die zuvor für diesen STED-Aufbau und die Farbstoffkombination63 berichtet wurden.
- Erstellen Sie in einer Übersicht eine Region of Interest (ROI) um ein Mitochondrium von Interesse, indem Sie die rechteckige ROI-Schaltfläche auswählen und klicken und ziehen, um die Region zu formen. Die ROI kann mithilfe der ROI-Ecken oder gekrümmten Kanten, die angezeigt werden, wenn Sie mit der Maus über eine Ecke fahren, in der Größe geändert und gedreht werden.
- Gating einrichten.
- Wählen Sie neben dem Menü " Allgemein " das Menü " Verknüpfung" aus, oder klicken Sie und halten Sie die Maustaste gedrückt, um das Menü zur Ansicht hinzuzufügen. Es wird empfohlen, das STED-Detektor-Gating auf 1-1,05 bis 7,8-7,85 ns einzustellen, wie hier dargestellt. Die Anschnittzeiten können variieren und von 1-1,05 bis 7-7,05 ns betragen.
- Passen Sie die Intensität entsprechend der Probe an.
HINWEIS: Im Allgemeinen ist die für STED verwendete Anregungsleistung 2-3 mal so hoch wie die für konfokale Leistung, so dass eine Probe, die 5 % Anregungslaserleistung für konfokal benötigt, während der STED-Erfassung 10 % bis 15 % Anregungslaserleistung verwenden kann.- Stellen Sie den Anregungslaser für STED auf 15%-20% und den STED-Verarmungslaser auf 20%-25% mit 10 Linienakkumulationen ein. Verwenden Sie eine Pixelverweilzeit von 4 μs bei einer Pixelgröße von 20-25 nm. Die Lochblende wurde bei 1,0 AE für kultivierte Zellen und 0,7 AE für primäre Ratten-Hippocampuszellen gehalten, um die optischen Schnitte in den dichter gepackten Mitochondrien zu verbessern.
HINWEIS: Sowohl konfokale als auch STED-Bilder können zum direkten Vergleich aufgenommen werden (Abbildung 3A, B, Abbildung 4A) oder es können nur STED-Bilder aufgenommen werden.
- Stellen Sie den Anregungslaser für STED auf 15%-20% und den STED-Verarmungslaser auf 20%-25% mit 10 Linienakkumulationen ein. Verwenden Sie eine Pixelverweilzeit von 4 μs bei einer Pixelgröße von 20-25 nm. Die Lochblende wurde bei 1,0 AE für kultivierte Zellen und 0,7 AE für primäre Ratten-Hippocampuszellen gehalten, um die optischen Schnitte in den dichter gepackten Mitochondrien zu verbessern.
- Zusatzinformationen für Zeitreihen/Z-Reihen
- Wählen Sie die Zeitreihe aus.
- Wählen Sie das Dropdown-Menü Zeit aus. Definieren Sie die Anzahl der Iterationen (hier 5) und das Zeitintervall (hier 25 oder 30 s), die für einen Zeitraffer gewünscht sind (Abbildung 3C, D, Abbildung 4B).
HINWEIS: Wenn das Intervall kürzer als die Erfassungszeit ist, werden die Iterationen ohne Verzögerung fortgesetzt. Bei der Durchführung eines Zeitraffers wird dringend empfohlen, eine perfekte Fokuseinheit oder eine ähnliche Fokusverfolgung zu aktivieren, um eine mögliche Drift zu vermeiden.
- Wählen Sie das Dropdown-Menü Zeit aus. Definieren Sie die Anzahl der Iterationen (hier 5) und das Zeitintervall (hier 25 oder 30 s), die für einen Zeitraffer gewünscht sind (Abbildung 3C, D, Abbildung 4B).
- Stellen Sie den Lautstärkebereich ein.
- Stellen Sie den Z-Lautstärkebereich wie gewünscht ein, indem Sie die Option Volume aktivieren und die beiden Enden des Bereichs anpassen. Die in diesem Protokoll für die 2D-Bildgebung verwendeten Schrittweiten betrugen 150-200 nm. Die empfohlene Schrittweite in Bezug auf die Nyquist-Probenahme, die für die Dekonvolution von Roh-STED erforderlich ist, kann mit den Online-Werkzeugen90 berechnet werden.
HINWEIS: Die Leistung des STED-Verarmungslasers und die Anzahl der abgebildeten Ebenen können das Photobleichen des Farbstoffs und die Lichtexposition gegenüber der Zelle auf ein schädliches Niveau erhöhen. Prüfen Sie nach dem Erwerb auf Anzeichen von Phototoxizität und Lichtschäden.
- Stellen Sie den Z-Lautstärkebereich wie gewünscht ein, indem Sie die Option Volume aktivieren und die beiden Enden des Bereichs anpassen. Die in diesem Protokoll für die 2D-Bildgebung verwendeten Schrittweiten betrugen 150-200 nm. Die empfohlene Schrittweite in Bezug auf die Nyquist-Probenahme, die für die Dekonvolution von Roh-STED erforderlich ist, kann mit den Online-Werkzeugen90 berechnet werden.
- Wählen Sie die Zeitreihe aus.
5. Verarbeitungs- und Analysewerkzeuge für die mitochondriale Ultrastruktur
HINWEIS: Die Bildverarbeitung (d. h. die Dekonvolution) ist optional, wird aber in der Regel bei der Erstellung und Analyse von STED-Bildern für die Veröffentlichung verwendet. Die Dekonvolution zur Verbesserung des Kontrasts und zur Reduzierung des Rauschens wird dringend empfohlen, um die einzelnen Cristae optimal zu segmentieren, wie unten beschrieben (Abbildung 2).
- STED-Bild-Dekonvolution
HINWEIS: Die Software, die in diesem Protokoll für die Dekonvolution verwendet wird, ist in der Materialtabelle angegeben.- Stellen Sie die mikroskopischen Parameter des Bildes ein.
HINWEIS: Stellen Sie sicher, dass die Metadaten des Mikroskops korrekt in die mikroskopischen Parameter des Bildes eingegeben werden. Dazu gehört auch die Montage des mittleren Brechungsindex; Immersionsmedium; Pixelgröße; Anregungs-, Emissions- und Verarmungswellenlängen; und alle anderen relevanten Informationen. Vorlagen mit diesen Parametern können zur Wiederverwendung gespeichert werden. - Dekonvolutieren Sie STED-Rohbilder mit dem Software-Algorithmus.
- Der Zugriff auf automatisierte Dekonvolutionsalgorithmen in Dekonvolutionssoftware ermöglicht eine manuelle Dekonvolutionsbildverarbeitung. Wählen Sie die Schaltfläche Express aus, und stellen Sie den Entfaltungstyp auf Schnell, Standard, Aggressiv oder Konservativ ein, um unterschiedliche Faltungsleistungen zu erzielen. Es werden repräsentative Bilder mit Express-Dekonvolution mit aggressiven Einstellungen gezeigt (Abbildung 3, Abbildung 4 und Abbildung 5).
- Speichern Sie die Bilder aus der Dekonvolutionssoftware im ICS2-Format.
- Führen Sie für die manuelle Dekonvolution die folgenden Schritte aus.
- Kurz gesagt, wenn Sie die manuelle Dekonvolution durchführen, speichern Sie die Vorlagen des Dekonvolutionsassistenten aus Gründen der Konsistenz und haben Sie die Möglichkeit, beim Starten des Assistenten eine Vorlage zu laden. Verwenden Sie PSF-Informationen (Measured Point Spread Function), wenn diese mit Erfassungseinstellungen und -parametern generiert wurden.
- Schneiden Sie das STED-Rohbild bei Bedarf zu, bevor Sie das Bild von der Dekonvolutionssoftware automatisch stabilisieren lassen. Add-ons zu Dekonvolutions-Softwarepaketen ermöglichen die gezielte Kompensation möglicher Abbildungsartefakte wie thermische Drift und chromatische Aberrationen.
- Generieren Sie als Nächstes ein logarithmisches Histogramm, um die Hintergrundsubtraktion manuell oder automatisch durchzuführen. Wählen Sie den klassischen CMLE-Dekonvolutionsalgorithmus (Maximum Likelihood Estimation) aus.
HINWEIS: Für die Dekonvolution sind die wichtigsten Werte, die angepasst werden müssen, der Schwellenwert für das Signal-Rausch-Verhältnis, die Anzahl der Iterationen und der Qualitätsschwellenwert. Diese Werte können angepasst werden, und die Dekonvolution kann in der Vorschau angezeigt werden, um die optimalen Einstellungen zu bestimmen.
- Stellen Sie die mikroskopischen Parameter des Bildes ein.
- Segmentierung und Partikelanalyse
HINWEIS: Dieses Protokoll verwendet FIJI (Is Just ImageJ), eine Open-Source-Software (siehe Material-Tabelle), für die Segmentierung und Analyse. Für diese Zwecke steht andere vergleichbare Software zur Verfügung, darunter CellProfiler, Icy, Ilastik und QuPath.- Bereiten Sie Bilder für die Segmentierung vor.
- Öffnen Sie die .obf-RAW-STED-Bilder oder .ics2-Dateien aus der Dekonvolutionssoftware, indem Sie zu Datei → Öffnen gehen oder auf die Dateien klicken und sie in die ImageJ-Symbolleiste ziehen. Von hier aus kann jede Verarbeitung, die die Segmentierung von Bildern erleichtert, vor der Segmentierung durchgeführt werden.
- Um die Änderungen konsistent zu halten, zeichnen Sie Funktionen mit Plugins → Makros auf → Zeichnen Sie Tastaturkurzbefehle auf und fügen Sie sie in ein neues Makro ein, und zwar aus Plug-ins → Neues →Makro. Stellen Sie sicher, dass Sie das zu verarbeitende Bild auswählen, bevor Sie das Makro ausführen.
HINWEIS: Zu den allgemein akzeptierten Änderungen für die Quantifizierung von Größe und Form gehören das Zuschneiden, die Projektion eines Z-Stapels und die Hintergrundsubtraktion mit deaktivierter Glättung. Änderungen sollten konsistent zwischen den Bildern innerhalb eines Datensatzes durchgeführt und gemeldet werden.
- Anpassen der Einstellungen für die trainierbare Weka-Segmentierung
HINWEIS: Zusätzliche Details mit Schritt-für-Schritt-Anleitungen für die Verwendung des halbautomatischen Segmentierungstools und nachgeschaltete Analysen für Mitochondrien wurden veröffentlicht91.- Öffnen Sie die entfalteten STED-Bilder im Plugin Trainable Weka Segmentation (TWS)92 , das sich unter Plugins → Segmentation → Trainable Weka Segmentation befindet. Wählen Sie in den Segmentierungseinstellungen die Funktionen Gaußscher Weichzeichner, Membranprojektionen und Sobel-Filter aus. Der Standardwert für die Membrandicke ist 1 und der Standardwert für die Membranpatchgröße ist 19.
- Beschriften Sie entweder Klasse 1 oder 2 als "Cristae" und die andere als "Hintergrund" (Abbildung 2). Modelle können auch mit der Schaltfläche Klassifikator speichern gespeichert werden. Wählen Sie den Klassifikator laden aus, um diese Einstellungen für andere Bilder wiederzuverwenden.
- Führen Sie TWS-Klassenablaufverfolgungen durch.
- Verwenden Sie das Linienwerkzeug oder andere Formen, um einige der Kristen oder den Hintergrund hervorzuheben. Zumindest einige Hintergrundauswahlen sollten Leerzeichen zwischen den Cristae enthalten. Ziehen Sie eine Linie über die Struktur, die Sie einer der beiden Klassen zuweisen möchten, und wählen Sie dann auf der rechten Seite die Schaltfläche Hinzufügen zu für Cristae oder Hintergrund aus. Doppelklicken Sie auf eine Ablaufverfolgung, um die Struktur aus dieser Beschriftung zu entfernen.
- Führen Sie ein TWS-Klassifizierungstraining durch.
- Wählen Sie auf der linken Seite die Schaltfläche Klassifikator trainieren aus, um eine Karte basierend auf den dem Plugin zur Verfügung gestellten Informationen zu generieren. Das Overlay von segmentierten Klassen kann mit der Schaltfläche Toggle Overlay ein- und ausgeschaltet werden, und die Deckkraft des Overlays kann in den Einstellungen angepasst werden. Der Klassifikator kann mit zusätzlichen Labels neu trainiert werden. Wenn Sie zufrieden sind, wählen Sie die Schaltfläche Wahrscheinlichkeit abrufen aus.
- Messen Sie die Partikel.
- Verwenden Sie die Cristae-Wahrscheinlichkeitskarte, um einen Schwellenwert für das Bild zu erstellen, um eine Maske zu generieren, und gehen Sie dann zu Analysieren → Partikel analysieren. Im Allgemeinen kann der Standardschwellenwerttyp verwendet und der Bereich angepasst werden, um sicherzustellen, dass alle Kriterien berücksichtigt werden. Die Messungen, die durch die Analyse von Partikeln bereitgestellt werden, können mit Analysieren → Messungen festlegen angepasst werden.
HINWEIS: Beispiele für Größen- und Formparameter wie Fläche, Umfang, Kreisförmigkeit und Seitenverhältnis der Cristae werden gemessen und basierend auf den ausgewählten Messungen angezeigt (Abbildung 2, Abbildung 5A, Ergänzende Tabelle 1). - OPTIONAL: Wählen Sie das STED-Rohbild mit den gleichen Abmessungen wie das entfaltete Bild aus, wenden Sie die ROIs aus dem Manager an und wählen Sie dann im ROI-Manager Messen aus, um Intensitätswerte zu erhalten.
- Verwenden Sie die Cristae-Wahrscheinlichkeitskarte, um einen Schwellenwert für das Bild zu erstellen, um eine Maske zu generieren, und gehen Sie dann zu Analysieren → Partikel analysieren. Im Allgemeinen kann der Standardschwellenwerttyp verwendet und der Bereich angepasst werden, um sicherzustellen, dass alle Kriterien berücksichtigt werden. Die Messungen, die durch die Analyse von Partikeln bereitgestellt werden, können mit Analysieren → Messungen festlegen angepasst werden.
- Abrufen von Liniendiagrammen.
- Aus den entfalteten STED-Bildern wurden Liniendiagramme generiert. Zeichnen Sie eine mehrpunktige Linie, passen Sie die Linienstärke auf mehrere Pixel Breite an, um das Rauschen zu mitteln, und verbiegen Sie die Linie so, dass sie zu den Mitochondrien passt (Abbildung 2, Abbildung 5B). Das resultierende Liniendiagramm wird verwendet, um Peak-to-Peak-Entfernungen zu messen, um die Periodizität und Verteilung von Cristae über einen bestimmten Bereich zu melden.
ANMERKUNG: In diesem Zusammenhang kann die Cristae-Dichte als die Anzahl unabhängiger Cristae innerhalb eines bestimmten Bereichs angegeben werden, die durch Messung des äußeren Teils der Mitochondrien bestimmt wird. Der mitochondriale Bereich kann bestimmt werden, indem ein Filter auf das dekonvolvierte oder rohe STED-Bild angewendet wird, um eine Maske zu erzeugen. Stellen Sie sicher, dass die Maske genau zum Umriss der Mitochondrien passt, bevor Sie den Bereich messen.
- Aus den entfalteten STED-Bildern wurden Liniendiagramme generiert. Zeichnen Sie eine mehrpunktige Linie, passen Sie die Linienstärke auf mehrere Pixel Breite an, um das Rauschen zu mitteln, und verbiegen Sie die Linie so, dass sie zu den Mitochondrien passt (Abbildung 2, Abbildung 5B). Das resultierende Liniendiagramm wird verwendet, um Peak-to-Peak-Entfernungen zu messen, um die Periodizität und Verteilung von Cristae über einen bestimmten Bereich zu melden.
- Bereiten Sie Bilder für die Segmentierung vor.
Repräsentative Ergebnisse
Dieses Protokoll beschreibt die Zellwachstumsbedingungen für kultivierte und primäre Zellen mit Schwerpunkt auf der STED-Bildgebung von lebenden Zellen und der anschließenden Analyse von mitochondrialen Cristae. Projektionen, die mit ImageJ von Mitochondrien aus undifferenzierten SH-SY5Y- (Abbildung 3A) und RA-differenzierten SH-SY5Y-Zellen (Abbildung 3B) erstellt wurden, können als Z-Stapel mit herkömmlichen konfokalen und STED-Zellen gesammelt werden, und die STED-Rohbilder können dann entfaltet werden. Es können auch Zeitrafferaufnahmen durchgeführt und anschließend entfaltet werden (Abbildung 3C,D). Unter Verwendung leicht unterschiedlicher Bildgebungsparameter für primäre Hippocampusneuronen der Ratte (Tabelle 1) können konfokale und rohe STED-Bilder als Z-Stapel aufgenommen und die rohen STED-Bilder entfaltet werden (Abbildung 4A). Zeitraffer-Aufnahmen von Mitochondrien aus primären Neuronen sind ebenfalls möglich (Abbildung 4B). Im Allgemeinen sollten die Zeitrafferbilder in der Lage sein, mitochondriale dynamische Ereignisse zu zeigen.
Wenn rohe STED- und dekonvolvierte STED-Z-Stack-Projektionen aus den für die Segmentierung verwendeten Proben konsistent erscheinen, werden quantitative Messungen durchgeführt. Das TWS-Plugin verwendet das dekonvolvierte STED-Bild, um zu segmentieren, um eine Wahrscheinlichkeitsmaske zu erstellen, die dann verwendet wird, um eine binäre Maske der Cristae zu erstellen, um Größen- und Formparameter zu erhalten (Abbildung 5A). Die Regionen aus dieser Maske werden im ROI-Manager gespeichert und können bei Bedarf auf das STED-Rohbild angewendet werden, um Unterschiede in der relativen Intensität zu messen. Die dekonvolvierten STED-Projektionen können auch verwendet werden, um die Periodizität und Dichte der Kristen in einem bestimmten Gebiet zu bestimmen (Abbildung 5B).
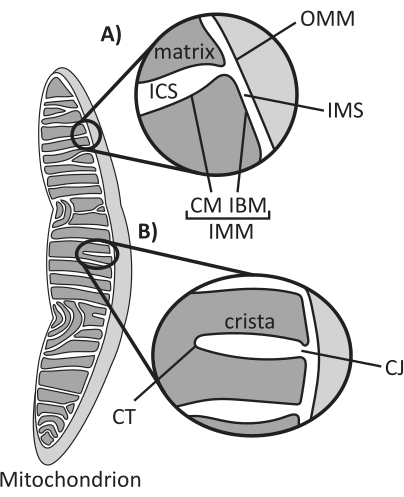
Abbildung 1: Mitochondriale Morphologie. Mitochondrien haben ein Zwei-Membran-System, das verschiedene Subkompartimente definiert (A). Cristae sind Einfaltungen der inneren Membran mit definierten Merkmalen (B). Abkürzungen: OMM, äußere Mitochondrienmembran; ICS, intrakristalliner Raum; IMS, Intermembranraum; CM, Cristae-Membran; IBM, innere Begrenzungsmembran; IMM, innere Mitochondrienmembran; CT, Cristae-Spitze; CJ, Cristae-Kreuzung. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
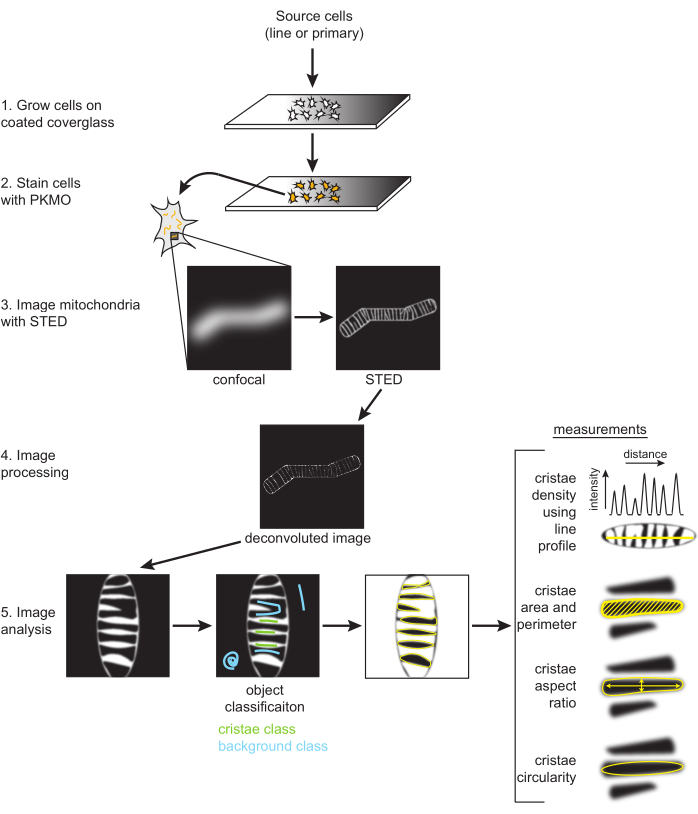
Abbildung 2: Schematische Darstellung des Arbeitsablaufs. SH-SY5Y-Zellen oder primäre Hippocampus-Neuronen der Ratte werden auf einem PDL-beschichteten Deckglas gezüchtet. SH-SY5Y-Zellen werden parallel gezüchtet, um über einen Zeitraum von sechs Tagen undifferenziert zu bleiben oder einer RA-Differenzierung unterzogen zu werden. Primäre Hippocampus-Neuronen der Ratte wurden auf einem PDL-beschichteten Deckglas gezüchtet, nachdem sie sieben Tage lang aus Hippocampus-Abschnitten isoliert worden waren. Sobald die Zellen für die Bildgebung bereit waren, wurden sie mit PKMO gefärbt und mit STED abgebildet. Die rohen STED-Bilder werden dann entfaltet, und die entfalteten Bilder werden in Fidschi verarbeitet, um Größen- und Formmessungen wie Cristae-Dichte, Fläche, Umfang, Zirkularität und Seitenverhältnis zu erhalten. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
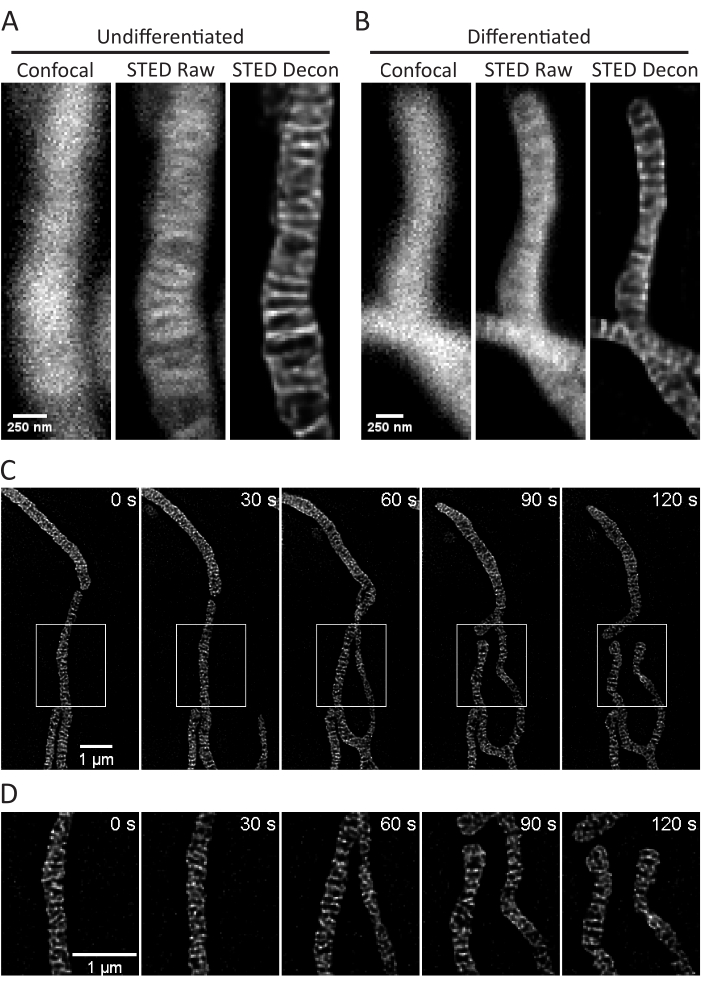
Abbildung 3: Bildgebung von Mitochondrien in SH-SY5Y-Zellen. Repräsentative konfokale (links), rohe STED (Mitte) und Huygens-dekonvolvierte STED-Bild-Z-Stapel-Projektionen (rechts) von Mitochondrien aus nicht-differenzierten (A) und RA-differenzierten (B) SH-SY5Y-Zellen mit PKMO-Färbung sind zu sehen. Ein Zeitraffer mit 30-s-Intervallen und 5 Iterationen von RA-differenzierten SH-SY5Y-Zellen wird gezeigt (C) mit ausgewählten Regionen (weiße Kästchen), die unter Verwendung skalierter Bilder dieser Regionen ohne Interpolation erweitert (D) werden. Maßstabsleisten: A,B, 250 nm; C,D, 1 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
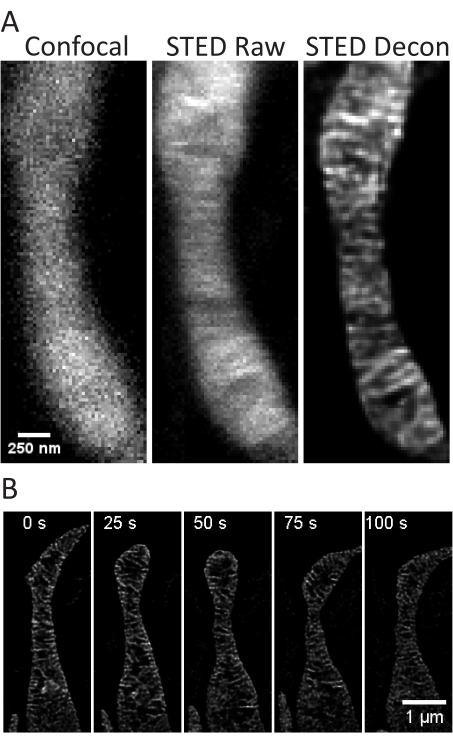
Abbildung 4: Bildgebung von Mitochondrien in primären Hippocampusneuronen der Ratte. Repräsentative konfokale (links), rohe STED (Mitte) und Huygens-dekonvolvierte STED-Bild-Z-Stack-Projektionen von Mitochondrien aus primären Hippocampusneuronen der Ratte sind dargestellt (A). Es wird ein Zeitraffer mit 25-Sekunden-Intervallen und 5 Iterationen von Mitochondrien in diesen Neuronen gezeigt (B). Maßstabsleisten: A, 250 nm; B , 1 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
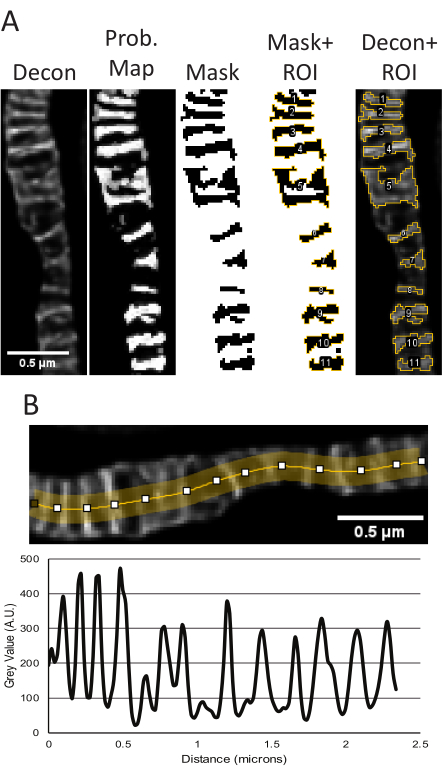
Abbildung 5: Verarbeitung von dekonvolvierten STED-Bildern in ImageJ. Die repräsentative Verwendung des Trainable Weka Segmentation Plugins zur Messung von Cristae Größe und Form wird gezeigt (A). Von links nach rechts werden die folgenden Bilder gezeigt: das dekonvolvierte STED-Bild, die Wahrscheinlichkeitskarte basierend auf der Segmentierung aus dem TWS-Plugin, die Maske von der Schwellenwertbestimmung in Fidschi unter Verwendung der Wahrscheinlichkeitskarte als Eingabe, die Maske mit den umrissenen ROIs und die ROIs, die über das ursprüngliche dekonvolvierte STED-Bild gelegt sind. Die resultierenden Flächen-, Umfangs-, Kreis- und Aspektverhältnismessungen, die diesen Objekten entsprechen, sind in der Zusatztabelle 1 zu finden. Es wird ein Liniendiagramm gezeigt, das das dekonvolvierte STED-Bild zur Messung von Spitze-zu-Spitze-Entfernungen als Auslesung für die Cristae-Dichte verwendet (B). Maßstabsleisten: 0,5 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Pixelgröße (nm) | Verweilzeit (μs) | Linie gem. | 561 nm Anregung während der STED-Erfassung (%) | 775 nm STED-Verarmungsleistung (%) | Schrittweite (nm) | Lochblende (AU) | Zeitraffer-Intervall(e) | Zeitraffer-Iterationen | |
| Undifferenzierter SH-SY5Y | 20 | 4 | 10 | 15 | 20 | 200 | 1.0 | 30 | 5 |
| RA-Differenziert SH-SY5Y | 20-25 | 4 | 10-12 | 15 | 20-22 | 150-200 | 1.0 | 30 | 5 |
| Primäre Neuronen | 20-25 | 4 | 10 | 10 | 25 | 200 | 0.7 | 30 | 5 |
| HINWEIS: Die Pixelgröße kann je nach Bildgebungsanforderungen und der Absicht, Bilder zu entflechten, variieren. Für die Entfaltung ist eine ordnungsgemäße Probenahme erforderlich. Die Pixelgrößen für STED-Rohbilder ohne Dekonvolution können bis zu 30 nm betragen. | |||||||||
Tabelle 1: Zusammenfassung der STED-Erfassungsparameter. Die Einstellungen, die für die 2D-STED-Bildgebung für jeden Zelltyp, undifferenziertes SH-SY5Y, RA-differenziertes SH-SY5Y und primäre Hippocampusneuronen der Ratte verwendet werden, werden angezeigt. Für alle Zeitraffer wurden 5 Iterationen mit unterschiedlichen Intervallen basierend auf der ROI-Größe durchgeführt.
Ergänzende Abbildung 1: Bildgebung von SH-SY5Y-Zellen mit Amyloid-β (Aβ)-Addition. Repräsentative konfokale (links), rohe STED- (Mitte) und dekonvolvierte STED-Bilder (rechts) von RA-differenzierten SH-SY5Y-Zellen mit PKMO-Färbung (oben) und Aβ-HiLyte647 (unten) sind zu sehen (A). Gezeigt werden zusammengeführte Z-Stack-Projektionen von rohem PKMO STED (grün) mit rohem Aβ STED (magenta) (B) oder dekonvolviertem PKMO STED (grün) mit dekonvolviertem Aβ STED (magenta) (C). Maßstabsleisten: 0,5 μm. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Tabelle 1: Größen- und Formmaße von segmentierten Cristae. Die Größen- und Formmaße der Fläche (μm2), des Umfangs (μm), der Zirkularität und des Seitenverhältnisses, die den in Abbildung 5A skizzierten Objekten aus segmentierten Mitochondrien entsprechen, sind dargestellt. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Tabelle 2: Zusammenfassung der Erfassungsparameter mit Amyloid-β-Proben. Die Einstellungen, die für die 2D-STED-Bildgebung von PKMO und Aβ-HiLyte647 in undifferenzierten und RA-differenzierten SH-SY5Y-Zellen verwendet werden, werden angezeigt. Konfokal von Aβ-HiLyte647 kann allein verwendet werden, da es keine spezifische Struktur gibt, die aufgelöst werden muss. STED-Bilder von Aβ-HiLyte647 sind hier für kleinere Partikelgrößen zu sehen. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzungsdatei 1: Amyloid-β-Behandlungsprotokoll. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Dieses Protokoll präsentiert die Verwendung der humanen Neuroblastom-Zelllinie SH-SY5Y und primärer Hippocampus-Neuronen der Ratte mit dem neuartigen IMM-gerichteten PKMO-Farbstoff für die STED-Bildgebung von lebenden Zellen. Aufgrund der Neuartigkeit von PKMO gibt es derzeit nur wenige Publikationen, die diesen Farbstoff für die Live-STED-Bildgebung verwenden. Die Verwendung dieser Zelltypen für die STED-Bildgebung stellt eine Herausforderung dar, insbesondere weil neuronale Zellen engere Mitochondrien aufweisen. Eine Einschränkung dieses Protokolls ist der verwendete PKMO-Farbstoff, da er für Zellen toxisch sein kann. Verschiedene Zellen und Zelllinien reagieren unterschiedlich auf den Farbstoff, daher können Anpassungen der Farbstoffkonzentration und der Inkubationszeit erforderlich sein, um die Ergebnisse für ein starkes Signal zu optimieren, ohne die Zellen zu schädigen. Eine vorgeschlagene Lösung besteht darin, die Konzentration zu senken und die Färbezeitzu verlängern 63; Dies kann jedoch zu einer schlechteren Färbung führen, ohne die Lebensfähigkeit der Zellen zu erhöhen.
Ähnlich wie PKMO weist auch der kommerzielle Farbstoff Live Orange mito (Table of Materials) eine gewisse Zelltoxizität auf. Dieser Farbstoff wurde für eine Vielzahl von kultivierten Zellen verwendet, war aber nicht in der Lage, eine vergleichbare Färbung in RA-differenzierten SH-SY5Y-Zellen mit den gleichen Parametern wie ihre undifferenzierten Gegenstücke erfolgreich zu zeigen (unsere unveröffentlichten Beobachtungen). Zugängliche Färbeprotokolle können jedoch für diese Sonde und ausgewählte Zelltypen optimiert werden. Mit diesem Farbstoff wurden Detektor-Gating-Zeiten von 1-1,05 bis 7-7,05 ns verwendet, wobei alle anderen Parameter in Tabelle 1 gleich blieben. Im Allgemeinen lieferte die Färbung von Zellen mit 200-250 nM Live Orange Mito für 45 min vergleichbare Ergebnisse wie die PKMO-Ergebnisse. Eine Färbung mit höherer Konzentration für kürzere Zeit oder eine Färbung mit niedrigerer Konzentration für die gleiche Zeit oder etwas länger kann zu anderen Ergebnissen führen und für andere Zelltypen oder Wachstumsbedingungen günstig sein.
Die Bildgebung primärer Hippocampus-Neuronen der Ratte unterscheidet sich von immortalisierten Zellen durch die Art der Axon- und Dendritenprojektionen sowie durch die mitochondriale Verteilung zum Zeitpunkt der Bildgebung. Eine Schwierigkeit in diesem Teil des Protokolls besteht darin, dass die Aussaatdichte bestimmt, ob die Primärkulturen in der Lage sind, zu haften und gesund zu wachsen, und bei höheren Dichten neigen die Projektionen dazu, um DIV 10 zu wachsen. Daher stammen die Mitochondrien, die von diesen primären Neuronen abgebildet werden, wahrscheinlich aus dem Zellkörper und nicht aus den Projektionen. Erfolgreiches Wachstum aus einer geringeren Ausgangszelldichte führt jedoch zu besseren Bildgebungsergebnissen zu späteren Wachstumszeiten. Der Schlüssel liegt darin, einen niedrigen Hintergrund und unscharfes Licht zu gewährleisten, um den besten Kontrast für STED zu erzielen. Um Bedenken hinsichtlich der Zellpopulation auszuräumen, verhindert die Kultivierung primärer Hippocampuszellen in B27-ergänzten Neuronenwachstumsmedien das Wachstum von Gliazellen, und die Quelle berichtet, dass <5 % der Zellen Astrozyten sind und das Fehlen von NbActiv1-Präparat in den Wachstumsmedien die Anzahl der Astrozyten in Kulturen auf <2 % reduziert87. Sowohl für kultivierte SH-SY5Y-Zellen als auch für primäre Neuronen des Rattenhippocampus trägt die PDL-Beschichtung, die für das Wachstum verwendet wird, zur Hintergrundtrübung in Bildern bei. Mit den in (Tabelle 1) angegebenen Einstellungen wird ein ausreichendes Signal-Rausch-Verhältnis erreicht, und die Dekonvolution entfernt den größten Teil des beobachteten Hintergrunds.
Zusätzlich zu der hier behandelten Bildgebung ist es auch möglich, Zellen vor oder während der Bildgebung zu behandeln oder zu belasten. Zum Beispiel induziert die Zugabe von tert-Butylhydrogenperoxid (tBHP) oxidativen Stress, und es ist möglich, Veränderungen in den Mitochondrien im Laufe der Zeit nach der Zugabe zu überwachen. Die Zugabe von Amyloid-β (Aβ) mit einer fluoreszierenden Markierung ermöglicht die Überwachung der Verteilung dieses Peptids in Bezug auf die Mitochondrien sowie die mitochondriale Struktur im Laufe der Zeit. Die Gesundheit der Mitochondrien wurde stark mit Alzheimer in Verbindung gebracht und es wird allgemein angenommen, dass sie eine Rolle bei der Aβ-Toxizität spielt 43,71,72. Insbesondere beeinflusst der Differenzierungsstatus von SH-SY5Y-Zellen die Lokalisierung von Aβ-Proteinvorläufern (AβPP)85, und Experimente mit AβPP sollten sorgfältig konstruiert werden.
Als Beispiel dafür, wie dieses Protokoll angepasst werden kann, wird gezeigt, dass die fluoreszierende Variante Aβ(1-42)-HiLyte 647 15 min vor der Bildgebung zu PKMO-gefärbten Zellen hinzugefügt werden kann (Ergänzende Abbildung 1). Die Bildgebungsparameter sind ähnlich (Ergänzende Tabelle 2), mit dem Hauptunterschied, dass bei der Abbildung schmalerer Mitochondrien eine kleinere Lochblende benötigt wird. Die Bildgebung von Aβ-HiLyte647 mit STED erfordert eine geringere Gesamtanregung (6%-8%) und STED-Verarmung (10%-12%) Laserleistung und weniger Akkumulationen (sechs). Das Detektor-Gating wird ebenfalls von 0,1 auf 10 ns erweitert. Obwohl die STED-Auflösung von Aβ nicht erforderlich ist, waren das gesamte Signal-Rausch-Verhältnis und die Aβ-Partikelgröße von rohem STED besser als die der konfokalen Bilder, und eine anschließende Dekonvolution kann ebenfalls durchgeführt werden. Das Sammeln von STED-Bildern und das Dekonvoltieren von rohen STED-Z-Stack-Projektionen von Aβ erscheint besonders nützlich, wenn es mit rohen STED- oder dekonvolvierten STED-Bildern der PKMO-Färbung zusammengeführt wird (Ergänzende Abbildung 1B,C). Beide Kanäle wurden in einem einzigen Frame-Schritt erfasst. Messungen der zeitabhängigen Lokalisation, ähnlich den in Abbildung 2 aufgeführten und gegebenenfalls in Abbildung 5 dargestellten, und der Unterschiede in der Cristae-Architektur können nach einer Spannungsbehandlung oder anderen Ergänzungen durchgeführt werden.
Andere mögliche Methoden zur dualen Markierung von Mitochondrien in lebenden Zellen, über die hier nicht berichtet wird, die aber von anderen berichtet wurden, umfassen die Verwendung von SNAP-markierten Proteinen93, Halo-markierten Proteinen und die Verwendung anderer zellpermeabler Farbstoffe mit generischen Zielen, wie z. B. mtDNA63. Bemerkenswert ist, dass die Markierungsstrategie von SNAP- und Halo-Tagging die resultierende Fluoreszenzsignalintensität und Langlebigkeit bei der Bildgebungbeeinflusst 94. Während dieses Protokoll mehrere Beispiele für Analysen enthält, die auf segmentierte Mitochondrien angewendet werden können, gibt es viele andere Analysen, die Softwarepakete an diesen Bildern durchführen können.
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Primäre Hippocampus-Neuronen der Ratte wurden von Dr. George Lykotrafitis und Shiju Gu vom Biomedical Engineering Department der University of Connecticut (Storrs, CT, USA) geliefert. Das Abberior STED-Instrument, das in der Advanced Light Microscopy Facility im Center for Open Research Resources and Equipment untergebracht ist, wurde mit einem NIH-Stipendium erworben S10OD023618 an Christopher O'Connell vergeben. Diese Forschung wurde durch ein NIH-Stipendium finanziert R01AG065879 an Nathan N. Alder vergeben.
Materialien
| Name | Company | Catalog Number | Comments |
| 0.05% Trypsin-EDTA | Gibco | 25300054 | |
| 0.4% Trypan blue | Invitrogen | T10282 | |
| 0.5% Trypsin-EDTA, no phenol red | Gibco | 15400054 | |
| 100 X antibiotic-antimycotic | Gibco | 15240062 | |
| 100 X/1.40 UPlanSApo oil immersion lens | Olympus | Equipped in Olympus IX83 microscope for STED setup described in Section 4 | |
| All-trans-retinoic acid | Sigma | R2625 | |
| Amyloid-β (1-42, HiLyte Fluor647, 0.1 mg) | AnaSpec | AS-64161 | Other fluorescent conjugates available |
| B27 supplement (50 X), serum free | Gibco | 17504044 | |
| Cell Counter (Countess II FL) | Life Technologies | AMQAF1000 | |
| Centrifuge | Eppendorf | 5804-R | |
| Counter slides | Invitrogen | C10283 | |
| Conical tubes (15 mL) | Thermo Fisher Scientific | 339650 | |
| Cuvettes (Quartz Cells) | Starna Cells, Inc. | 9-Q-10 | Used with Spectrometer as described in Section 1.3 |
| DMEM (high glucose with sodium pyruvate) | Gibco | 11995073 | Used for SH-SY5Y cell materials as described in Section 1 |
| DMEM (high glucose no sodium pyruvate) | Gibco | 11965092 | Used for primary cell materials as described in Section 2 |
| DMEM (phenol red-free) | Gibco | 31053028 | Used for imaging as described in Section 3 |
| DMSO | Sigma | D8418 | |
| DNAase I from bovine pancreas | Sigma | DN25 | Used for primary cell materials as described in Section 2.2.1 and 2.2.2 |
| DPBS (no calcium, no magnesium) | Gibco | 14190144 | |
| E18 Rat Hippocampus | Transnetyx Tissue | SDEHP | |
| Ethanol (200 proof) | Fisher Bioreagents | BP28184 | |
| Fetal bovine serum (FBS), not heat-inactivated | Gibco | 26140079 | For cultured cells, in Section 1 |
| Fetal bovine serum (heat inactivated) | Gibco | 10082147 | For primary cell culture, Section 2 |
| Filter sterilization unit (0.1 µm, 500 mL) | Thermo Fisher Scientific | 5660010 | |
| FIJI (Is Just ImageJ) and Trainable Weka Segmentation (TWS) plug-in | -- | -- | Free, open-source image analysis software that includes plug-ins including Trainable Weka Segmentation described in Section 5; TWS plug-in from ref. 90 of the main text |
| GlutaMAX supplement (100 X) | Gibco | 35050061 | Glutamine supplement used for primary cell materials described in Section 2.1.2 |
| Hausser Scientific bright-Line and Hy-Lite Counting Chambers | Hausser Scientific | 267110 | |
| HBSS (no calcium, no magnesium) | Gibco | 14170120 | Used for primary cell materials described in Section 2.2.1 and 2.2.2 |
| HEPES | Gibco | 15630080 | |
| Huygens Professional deconvolution software (V. 20.10) | Scientific Volume Imaging (SVI) | -- | The deconvolution software used in this protocol and described in Section 5 |
| IX83 inverted microscope with Continuous Autofocus | Olympus | -- | This paper uses a STED Infinity Line system built around an Olympus IX83 inverted microscope, described in Section 4 |
| Lightbox software (V. 16.3.16118) | Abberior | -- | Vendor software used for STED image acquisition, described in Section 4 |
| Live Orange Mito dye | Abberior | LVORANGE-0146-30NMOL | Live cell imaging IMM-targeting dye described in Discussion |
| Neurobasal media | Gibco | 21103049 | Used for primary cell materials referred to in Section 2.1.2 |
| Nunc Lab-Tek II 2-well chambered coverglass | Nunc | 155379 | Can purchase a variety of chambers but make sure the coverglass is #1.5 |
| Pasteur Pipets (Fisherbrand) | Thermo Fisher Scientific | 22183632 | |
| Penicillin-Streptomycin (10,000 U/mL) | Gibco | 15140122 | |
| PKmito Orange dye | Spirochrome | SC053 | |
| Poly-D-lysine | Gibco | A3890401 | |
| SH-SY5Y Cell line | ATCC | CRL2266 | |
| Sodium pyruvate (100 mM) | Gibco | 11360070 | Used for primary cell materials described in Section 2 |
| Spectrometer (GENESYS 180 UV-Vis) | Thermo Fisher Scientific | 840309000 | |
| STED Expert Line microscope | Abberior | -- | STED setup can be customized, but at time of purchase instrument was considered Abberior’s Expert Line; brief description of setup is available in Section 4 of protocol |
| T25 flask (TC-treated, filter cap) | Thermo Fisher Scientific | 156367 | Other culture vessels and sizes available |
Referenzen
- Iovine, J. C., Claypool, S. M., Alder, N. N. Mitochondrial compartmentalization: emerging themes in structure and function. Trends in Biochemical Sciences. 46 (11), 902-917 (2021).
- Gupta, A., Becker, T. Mechanisms and pathways of mitochondrial outer membrane protein biogenesis. Biochimica et Biophysica Acta (BBA) - Bioenergetics. 1862 (1), 148323 (2021).
- Gordaliza-Alaguero, I., Cantó, C., Zorzano, A. Metabolic implications of organelle-mitochondria communication. EMBO Reports. 20 (9), e47928 (2019).
- Klecker, T., Westermann, B. Pathways shaping the mitochondrial inner membrane. Open Biology. 11 (12), 210238 (2021).
- Navarro, A., Boveris, A. The mitochondrial energy transduction system and the aging process. American Journal of Physiology-Cell Physiology. 292 (2), C670-C686 (2007).
- Yu, R., Lendahl, U., Nistér, M., Zhao, J. Regulation of mammalian mitochondrial dynamics: opportunities and challenges. Frontiers in Endocrinology. 11, 374 (2020).
- Horn, A., Raavicharla, S., Shah, S., Cox, D., Jaiswal, J. K. Mitochondrial fragmentation enables localized signaling required for cell repair. The Journal of Cell Biology. 219 (5), e201909154 (2020).
- Glancy, B., Kim, Y., Katti, P., Willingham, T. B. The functional impact of mitochondrial structure across subcellular scales. Frontiers in Physiology. 11, 541040 (2020).
- Bahat, A., et al. MTCH2-mediated mitochondrial fusion drives exit from naïve pluripotency in embryonic stem cells. Nature Communications. 9 (1), 5132 (2018).
- Detmer, S. A., Chan, D. C. Functions and dysfunctions of mitochondrial dynamics. Nature Reviews Molecular Cell Biology. 8, 870-879 (2007).
- Bertholet, A. M., et al. Mitochondrial fusion/fission dynamics in neurodegeneration and neuronal plasticity. Neurobiology of Disease. 90, 3-19 (2016).
- Zemirli, N., Morel, E., Molino, D. Mitochondrial dynamics in basal and stressful conditions. International Journal of Molecular Sciences. 19 (2), 564 (2018).
- Harwig, M. C., et al. Methods for imaging mammalian mitochondrial morphology: a prospective on mitograph. Analytical Biochemistry. 552, 81-99 (2018).
- Pánek, T., Eliáš, M., Vancová, M., Lukeš, J., Hashimi, H. Returning to the fold for lessons in mitochondrial crista diversity and evolution. Current Biology. 30 (10), R575-R588 (2020).
- Gottschalk, B., Madreiter-Sokolowski, C. T., Graier, W. F. Cristae junction as a fundamental switchboard for mitochondrial ion signaling and bioenergetics. Cell Calcium. 101, 102517 (2022).
- Khosravi, S., Harner, M. E. The MICOS complex, a structural element of mitochondria with versatile functions. Biological Chemistry. 401 (6-7), 765-778 (2020).
- Frezza, C., et al. OPA1 controls apoptotic cristae remodeling independently from mitochondrial fusion. Cell. 126 (1), 177-189 (2006).
- Meeusen, S., et al. Mitochondrial inner-membrane fusion and crista maintenance requires the dynamin-related GTPase Mgm1. Cell. 127 (2), 383-395 (2006).
- Patten, D. A., et al. OPA1-dependent cristae modulation is essential for cellular adaptation to metabolic demand. The EMBO Journal. 33 (22), 2676-2691 (2014).
- Paumard, P., et al. The ATP synthase is involved in generating mitochondrial cristae morphology. The EMBO Journal. 21 (3), 221-230 (2002).
- Strauss, M., Hofhaus, G., Schröder, R. R., Kühlbrandt, W. Dimer ribbons of ATP synthase shape the inner mitochondrial membrane. The EMBO Journal. 27 (7), 1154-1160 (2008).
- Basu Ball, W., Neff, J. K., Gohil, V. M. The role of nonbilayer phospholipids in mitochondrial structure and function. FEBS Letters. 592 (8), 1273-1290 (2018).
- Hackenbrock, C. R. Ultrastructural bases for metabolically linked mechanical activity in mitochondria. I. Reversible ultrastructural changes with change in metabolic steady state in isolated liver mitochondria. The Journal of Cell Biology. 30 (2), 269-297 (1966).
- Dlasková, A., et al. Mitochondrial cristae narrowing upon higher 2-oxoglutarate load. Biochimica et Biophysica Acta (BBA) - Bioenergetics. 1860 (8), 659-678 (2019).
- Pérez-Hernández, C. A., et al. Mitochondrial ultrastructure and activity are differentially regulated by glycolysis-, krebs cycle-, and microbiota-derived metabolites in monocytes. Biology. 11 (8), 1132 (2022).
- Mannella, C. A. Structural diversity of mitochondria: functional implications. Annals of the New York Academy of Sciences. 1147, 171-179 (2008).
- Plecitá-Hlavatá, L., Ježek, P. Integration of superoxide formation and cristae morphology for mitochondrial redox signaling. The International Journal of Biochemistry & Cell Biology. 80, 31-50 (2016).
- Scorrano, L., et al. A distinct pathway remodels mitochondrial cristae and mobilizes cytochrome c during apoptosis. Developmental Cell. 2 (1), 55-67 (2002).
- Heath-Engel, H. M., Shore, G. C. Mitochondrial membrane dynamics, cristae remodelling and apoptosis. Biochimica et Biophysica Acta (BBA) - Molecular Cell Research. 1763 (5-6), 549-560 (2006).
- Brandt, T., et al. Changes of mitochondrial ultrastructure and function during ageing in mice and Drosophilia. eLife. 6, e24662 (2017).
- Kondadi, A. K., et al. Cristae undergo continuous cycles of membrane remodelling in a MICOS-dependent manner. EMBO Reports. 21, e49776 (2020).
- Quintana-Cabrera, R., Mehrotra, A., Rigoni, G., Soriano, M. E. Who and how in the regulation of mitochondrial cristae shape and function. Biochemical and Biophysical Research Communications. 500 (1), 94-101 (2018).
- Nielsen, J., et al. Plasticity in mitochondrial cristae density allows metabolic capacity modulation in human skeletal muscle: Enlarged mitochondrial cristae density in athletes. The Journal of Physiology. 595 (9), 2839-2847 (2017).
- Afzal, N., Lederer, W. J., Jafri, M. S., Mannella, C. A. Effect of crista morphology on mitochondrial ATP output: A computational study. Current Research in Physiology. 4, 163-176 (2021).
- Heine, K. B., Parry, H. A., Hood, W. R. How does density of the inner mitochondrial membrane influence mitochondrial performance. American Journal of Physiology-Regulatory, Integrative and Comparative Physiology. 324 (2), R242-R248 (2023).
- Wang, W., Zhao, F., Ma, X., Perry, G., Zhu, X. Mitochondria dysfunction in the pathogenesis of Alzheimer's disease: recent advances. Molecular Neurodegeneration. 15, 30 (2020).
- Singh, A., Kukreti, R., Saso, L., Kukreti, S. Oxidative stress: a key modulator in neurodegenerative diseases. Molecules. 24, 1583 (2019).
- Pchitskaya, E., Popugaeva, E., Bezprozvanny, I. Calcium signaling and molecular mechanisms underlying neurodegenerative diseases. Cell Calcium. 70, 87-94 (2018).
- Estes, R. E., Lin, B., Khera, A., Davis, M. Y. Lipid metabolism influence on neurodegenerative disease progression: is the vehicle as important as the cargo. Frontiers in Molecular Neuroscience. 14, 788695 (2021).
- Calkins, M. J., Manczak, M., Mao, P., Shirendeb, U., Reddy, P. H. Impaired mitochondrial biogenesis, defective axonal transport of mitochondria, abnormal mitochondrial dynamics and synaptic degeneration in a mouse model of Alzheimer's disease. Human Molecular Genetics. 20 (23), 4515-4529 (2011).
- Petersen, C. A. H., et al. The amyloid beta-peptide is imported into mitochondria via the TOM import machinery and localized to mitochondrial cristae. Proceedings of the National Academy of Sciences of the United States of America. 105 (35), 13145-13150 (2008).
- Gan, X., et al. Inhibition of ERK-DLP1 signaling and mitochondrial division alleviates mitochondrial dysfunction in Alzheimer's disease cybrid cell. Biochimica et Biophysica Acta (BBA) - Molecular Basis of Disease. 1842 (2), 220-231 (2014).
- Baloyannis, S. J., Costa, V., Michmizos, D. Mitochondrial alterations Alzheimer's disease. American Journal of Alzheimer's Disease & Other Dementias. 19 (2), 89-93 (2004).
- Tillement, L., Lecanu, L., Papadopoulos, V. Alzheimer's disease: Effects of β-amyloid on mitochondria. Mitochondrion. 11 (1), 13-21 (2011).
- Choi, S. Y., et al. C9ORF72-ALS/FTD-associated poly(GR) binds Atp5a1 and compromises mitochondrial function in vivo. Nature Neuroscience. 22 (6), 851-862 (2019).
- Smith, E. F., Shaw, P. J., De Vos, K. J. The role of mitochondria in amyotrophic lateral sclerosis. Neuroscience Letters. 710, 132933 (2019).
- Costa, V., et al. Mitochondrial fission and cristae disruption increase the response of cell models of Huntington's disease to apoptotic stimuli. EMBO Molecular Medicine. 2 (12), 490-503 (2010).
- Costa, V., Scorrano, L. Shaping the role of mitochondria in the pathogenesis of Huntington's disease: Mitochondrial and Huntington's disease. The EMBO Journal. 31 (8), 1853-1864 (2012).
- Vanisova, M., et al. Mitochondrial organization and structure are compromised in fibroblasts from patients with Huntington's disease. Ultrastructural Pathology. 46 (5), 462-475 (2022).
- de Barcelos, I. P., Troxell, R. M., Graves, J. S. Mitochondrial dysfunction and multiple sclerosis. Biology. 8 (2), 37 (2019).
- Park, J., et al. Mitochondrial dysfunction in Drosophila PINK1 mutants is complemented by parkin. Nature. 441, 1157-1161 (2006).
- Meng, H., et al. Loss of Parkinson's disease-associated protein CHCHD2 affects mitochondrial crista structure and destabilizes cytochrome c. Nature Communications. 8, 15500 (2017).
- Lu, L., et al. CHCHD2 maintains mitochondrial contact site and cristae organizing system stability and protects against mitochondrial dysfunction in an experimental model of Parkinson's disease. Chinese Medical Journal. 135 (13), 1588-1596 (2022).
- Cogliati, S., et al. Mitochondrial Cristae shape determines respiratory chain supercomplexes assembly and respiratory efficiency. Cell. 155 (1), 160-171 (2013).
- He, B., et al. Mitochondrial cristae architecture protects against mtDNA release and inflammation. Cell Reports. 41 (10), 111774 (2022).
- Polo, C. C., et al. Three-dimensional imaging of mitochondrial cristae complexity using cryo-soft X-ray tomography. Scientific Reports. 10, 21045 (2020).
- Rybka, V., et al. Transmission electron microscopy study of mitochondria in aging brain synapses. Antioxidants. 8 (6), 171 (2019).
- Mannella, C. A. Structure and dynamics of the mitochondrial inner membrane cristae. Biochimica et Biophysica Acta (BBA) - Molecular Cell Research. 1763 (5-6), 542-548 (2006).
- Fry, M. Y., et al. In situ architecture of Opa1-dependent mitochondrial cristae remodeling. biorxiv. , (2023).
- Barad, B. A., Medina, M., Fuentes, D., Wiseman, R. L., Grotjahn, D. A. Quantifying organellar ultrastructure in cryo-electron tomography using a surface morphometrics pipeline. The Journal of Cell Biology. 222 (4), 202204093 (2023).
- Kunz, T. C., Götz, R., Gao, S., Sauer, M., Kozjak-Pavlovic, V. Using expansion microscopy to visualize and characterize the morphology of mitochondrial cristae. Frontiers in Cell and Developmental Biology. 8, 617 (2020).
- Yang, Z., et al. Cyclooctatetraene-conjugated cyanine mitochondrial probes minimize phototoxicity in fluorescence and nanoscopic imaging. Chemical Science. 11 (32), 8506-8516 (2020).
- Liu, T., et al. Multi-color live-cell STED nanoscopy of mitochondria with a gentle inner membrane stain. Proceedings of the National Academy of Sciences. 119 (52), e2215799119 (2022).
- Yang, X., et al. Mitochondrial dynamics quantitatively revealed by STED nanoscopy with an enhanced squaraine variant probe. Nature Communications. 11, 3699 (2020).
- Zheng, S., et al. Long-term, super-resolution HIDE imaging of the inner mitochondrial membrane in live cells with a cell-permeant lipid probe. biorxiv. , (2022).
- Wang, C., et al. A photostable fluorescent marker for the superresolution live imaging of the dynamic structure of the mitochondrial cristae. Proceedings of the National Academy of Sciences of the United States of America. 116 (32), 15817-15822 (2019).
- Feles, S., et al. Streamlining culture conditions for the neuroblastoma cell line sh-sy5y: a prerequisite for functional studies. Methods and Protocols. 5 (4), 58 (2022).
- Shipley, M. M., Mangold, C. A., Szpara, M. L. Differentiation of the SH-SY5Y human neuroblastoma cell line. Journal of Visualized Experiments. (108), e53193 (2016).
- Kovalevich, J., Langford, D., Amini, S., White, M. K. Considerations for the use of SH-SY5Y neuroblastoma cells in neurobiology. Neuronal Cell Culture. 1078, 9-21 (2013).
- Biedler, J. L., Helson, L., Spengler, B. A. Morphology and growth, tumorigenicity, and cytogenetics of human neuroblastoma cells in continuous culture. Cancer Research. 33 (11), 2643-2652 (1973).
- Swerdlow, R. H. Mitochondria and mitochondrial cascades in alzheimer's disease. Journal of Alzheimer's Disease. 62 (3), 1403-1416 (2018).
- Wang, W., Zhao, F., Ma, X., Perry, G., Zhu, X. Mitochondria dysfunction in the pathogenesis of Alzheimer's disease: recent advances. Molecular Neurodegeneration. 15, 30 (2020).
- Reddy, P. H. Mitochondrial dysfunction in aging and alzheimer's disease: strategies to protect neurons. Antioxidants & Redox Signaling. 9 (10), 1647-1658 (2007).
- Horn, A., Raavicharla, S., Shah, S., Cox, D., Jaiswal, J. K. Mitochondrial fragmentation enables localized signaling required for cell repair. Journal of Cell Biology. 219 (5), e201909154 (2020).
- Korecka, J. A., et al. Phenotypic characterization of retinoic acid differentiated SH-SY5Y cells by transcriptional profiling. PLoS ONE. 8 (5), e63862 (2013).
- Baghel, M. S., Thakur, M. K. Vdac1 downregulation causes mitochondrial disintegration leading to hippocampal neurodegeneration in scopolamine-induced amnesic mice. Molecular Neurobiology. 56 (3), 1707-1718 (2019).
- Jiang, S., et al. Mfn2 ablation causes an oxidative stress response and eventual neuronal death in the hippocampus and cortex. Molecular Neurodegeneration. 13 (1), 5 (2018).
- Mu, Y., Gage, F. H. Adult hippocampal neurogenesis and its role in Alzheimer's disease. Molecular Neurodegeneration. 6, 85 (2011).
- Rao, Y. L., et al. Hippocampus and its involvement in Alzheimer's disease: a review. 3 Biotech. 12 (2), 55 (2022).
- Weerasinghe-Mudiyanselage, P. D. E., Ang, M. J., Kang, S., Kim, J. -. S., Moon, C. Structural Plasticity of the hippocampus in neurodegenerative diseases. International Journal of Molecular Sciences. 23 (6), 3349 (2022).
- . Poly-D-Lysine Available from: https://www.thermofisher.com/order/catalog/product/A3890401 (2023)
- Dravid, A., Raos, B., Svirskis, D., O'Carroll, S. J. Optimised techniques for high-throughput screening of differentiated SH-SY5Y cells and application for neurite outgrowth assays. Scientific Reports. 11, 23935 (2021).
- Hromadkova, L., et al. Brain-derived neurotrophic factor (BDNF) promotes molecular polarization and differentiation of immature neuroblastoma cells into definitive neurons. Biochimica et Biophysica Acta (BBA) - Molecular Cell Research. 1867 (9), 118737 (2020).
- Riegerová, P., et al. Expression and Localization of AβPP in SH-SY5Y cells depends on differentiation state. Journal of Alzheimer's Disease. 82 (2), 485-491 (2021).
- Hoffmann, L. F., et al. Neural regeneration research model to be explored: SH-SY5Y human neuroblastoma cells. Neural Regeneration Research. 18 (6), 1265-1266 (2022).
- Abiraman, K., Tzingounis, A. V., Lykotrafitis, G. K. Ca 2 channel localization and regulation in the axon initial segment. The FASEB Journal. 32 (4), 1794-1805 (2018).
- E18 Rat Hippocampus. Transnetyx Tissue Available from: https://tissue.transnetyx.com/E18-Rat-Hippocampus_4 (2023)
- Kmito ORANGE - Probe for live cell imaging of mitochondria. Spirochrome Available from: https://spirochrome.com/product/pkmito_orange/ (2023)
- Nyquist Calculator. Scientific Volume Imaging Available from: https://svi.nl/Nyquist-Calculator (2023)
- Segawa, M., et al. Quantification of cristae architecture reveals time-dependent characteristics of individual mitochondria. Life Science Alliance. 3 (7), e2019000620 (2020).
- Arganda-Carreras, I., et al. Trainable Weka Segmentation: a machine learning tool for microscopy pixel classification. Bioinformatics. 33 (15), 2424-2426 (2017).
- Stephan, T., Roesch, A., Riedel, D., Jakobs, S. Live-cell STED nanoscopy of mitochondrial cristae. Scientific Reports. 9, 12419 (2019).
- Erdmann, R. S., et al. Labeling strategies matter for super-resolution microscopy: A comparison between HaloTags and SNAP-tags. Cell Chemical Biology. 26 (4), 584-592 (2019).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten