Immunhistochemische Multiplex-Analyse der räumlichen Immunzelllandschaft der Tumormikroumgebung
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt detailliert, wie die Immunzellcharakterisierung der Tumormikroumgebung mittels Multiplex-Immunhistochemie durchgeführt wird.
Zusammenfassung
Die Immunzelllandschaft der Tumormikroumgebung enthält potenziell Informationen für die Entdeckung von prognostischen und prädiktiven Biomarkern. Die Multiplex-Immunhistochemie ist ein wertvolles Werkzeug, um verschiedene Arten von Immunzellen in Tumorgeweben unter Beibehaltung ihrer räumlichen Informationen zu visualisieren und zu identifizieren. Hier stellen wir detaillierte Protokolle zur Analyse von Lymphozyten-, myeloischen und dendritischen Zellpopulationen in Gewebeschnitten zur Verfügung. Angefangen beim Schneiden von formalinfixierten, in Paraffin eingebetteten Schnitten, über automatische Multiplex-Färbeverfahren auf einer automatisierten Plattform, das Scannen der Objektträger auf einem multispektralen Bildgebungsmikroskop bis hin zur Analyse der Bilder mit einem eigens entwickelten maschinellen Lernalgorithmus ImmuNet. Diese Protokolle können auf eine Vielzahl von Tumorproben angewendet werden, indem einfach Tumormarker ausgetauscht werden, um Immunzellen in verschiedenen Kompartimenten der Probe (Tumor versus invasiver Rand) zu analysieren und die Nearest-Neighbor-Analyse anzuwenden. Diese Analyse ist nicht nur auf Tumorproben beschränkt, sondern kann auch auf andere (nicht-)pathogene Gewebe angewendet werden. Durch Verbesserungen der Ausstattung und des Arbeitsablaufs in den letzten Jahren konnten die Durchlaufzeiten deutlich verkürzt werden, was die zukünftige Anwendung dieses Verfahrens in der Diagnostik erleichtert.
Einleitung
Immunzellen spielen eine entscheidende Rolle beim Schutz vor Krankheitserregern wie Viren und Bakterien, aber auch vor Krebszellen1. Daher ist das Immunsystem in der Tumormikroumgebung (TME) vielversprechend für die Entdeckung prognostischer und prädiktiver Biomarker2. Immunzellinfiltrate wurden mit der Prognose bei verschiedenen Krebsarten korreliert, obwohl dies in der klinischen Versorgung noch nicht implementiert wurde 3,4. Bei den meisten Tumorarten sind eine hohe Anzahl von zytotoxischen T-Zellen und T-Helfer-1-Zellen und/oder eine geringe Anzahl von regulatorischen T-Zellen mit guten Prognosen verbunden. Es gibt Bestrebungen, einen sogenannten "Immunoscore" in das TNM-Staging von Darmkrebs einzubauen und daraus ein TNM-I-Staging zu machen 5,6. Der Immunoscore ergibt sich aus der Gesamtzahl der T-Zellen (nachgewiesen mit CD3) und zytotoxischen T-Zellen (nachgewiesen mit CD8) in zwei verschiedenen Tumorregionen: dem Tumorkern und dem invasiven Rand (IM) von Tumoren. Es wurde vorgeschlagen, dass der Immunoscore auch bei anderen Krebsarten wie Melanom, Lungenkrebs und Brustkrebs von prognostischem Wert ist 6,7,8,9. Darüber hinaus können Immunzellinfiltrate auch mit dem Ansprechen auf eine Checkpoint-Blockade-Immuntherapie korrelieren10. Diese prädiktiven Biomarker müssen jedoch in prospektiven Studien validiert werden, bevor sie routinemäßig in der klinischen Praxis eingesetzt werden können. Darüber hinaus wurde auch vorgeschlagen, dass ein einzelner Biomarker für eine aussagekräftige Vorhersage nicht ausreicht11. Daher wurde die Erstellung einer vollständigen Karte einer Patientenprobe durch die Kombination verschiedener Biomarker als umfassenderer prädiktiver Biomarker in einem sogenannten "Krebsimmunogramm" vorgeschlagen12.
Unter den Methoden zur Untersuchung von Immunzellen im Rahmen der TME ist die Immunhistochemie (IHC) die älteste und bekannteste Technik, die routinemäßig für diagnostische Tests bei verschiedenen Krankheiten, insbesondere Krebs, eingesetzt wird13. Diese Technik war lange Zeit auf die Verwendung eines oder nur einiger weniger Marker14 beschränkt und wurde daher in Forschungsumgebungen von anderen Techniken wie der Durchflusszytometrie und dem Genexpressionsprofiling (GEP) verdrängt. Die in der Routinediagnostik und -forschung typischerweise verwendeten formalinfixierten und paraffineingebetteten (FFPE) Tumorgewebe sind jedoch nicht (optimal) für die Durchflusszytometrie und GEP geeignet. Obwohl GEP und Durchflusszytometrie viele Einblicke in den Phänotyp und die Funktion von Zellen bieten, ist der Mangel an räumlichen Informationen ein großer Nachteil. Daher könnten Heterogenitäten innerhalb einer Probe, wie z. B. Unterschiede zwischen Immunzell-infiltrierten und Immunzell-ausgeschlossenen Bereichen eines Tumors, unentdeckt bleiben15. Für die Multiplex-Analyse von FFPE-Geweben wurden neuartige Plattformen entwickelt, wie z. B. Multiplex-IHC, bildgebende Massenzytometrie und CO-Detection by indEXing (CODEX), die zum gleichzeitigen Nachweis mehrerer Marker innerhalb eines Gewebeschnittsverwendet werden können 16. Immunzellen in der TME werden umfassend untersucht, um die besten Biomarker für die Immuntherapie zu finden. Multiplex-Techniken und automatisierte Bildanalyse stellen jedoch ihre eigenen Hürden dar.
Unser Labor verfügt über umfangreiche Erfahrung in der Multiplex-IHC-Färbung mit der Opal/Tyramid-Signalverstärkungsmethode (TSA) und hat diese auf einer IHC-Plattform automatisiert (siehe Materialtabelle)17,18,19,20,21,22,23,24,25,26,27,28, 29,30,31. Wir haben Immunzell-Panels für den Nachweis verschiedener Untergruppen von Lymphozyten, myeloischen Zellen und dendritischen Zellen (DCs) optimiert. Gewebe, die dichte Immunzellbereiche enthalten - für Lymphozyten oder komplexe Zellmorphologien (d. h. myeloische Zellen und DCs) - sind besonders schwierig zu analysieren, da die Gefahr besteht, dass die Anzahl der vorhandenen Immunzellen über- oder unterschätzt wird. Um dieses Problem zu lösen, wurde von unserer Gruppe32 die Analysesoftware ImmuNet entwickelt, die die Qualität der Erkennung dieser verschiedenen Arten von Immunzellen immens verbesserte. Ein detailliertes Protokoll von der Gewinnung des FFPE-Materials bis zur Analyse der Immunzelldichten in verschiedenen Gewebekompartimenten und Abständen zwischen Immunzelltypen wird hier beschrieben.
Dieses Protokoll beschreibt, wie die Multiplex-IHC-Panels am Radboud University Medical Center seit der Implementierung des digitalen Pathologie-Imagers im Jahr 2022 durchgeführt werden. Die beschriebenen Multiplex-IHC-Panels können für verschiedene Karzinome (z.B. Lunge, Prostata, Darm, Blase, Brust) unter Verwendung eines Pan-Cytokeratin-Antikörpers als Tumormarker oder für Melanome unter Verwendung von Melanozyten-assoziierten Antikörpern als Tumormarker verwendet werden. Diese Multiplex-IHC-Protokolle wurden in Bezug auf die Primärantikörperkonzentration, die Fluorophor-Kombinationen und die Reihenfolge des Färbeverfahrens sorgfältig optimiert. Wir und andere haben die Multiplex-IHC-Panel-Optimierung bereits beschrieben 17,33,34,35. Multiplex-IHC-Panels können angepasst werden, aber die beschriebenen Analyse-Pipelines müssen evaluiert und möglicherweise entsprechend angepasst oder neu trainiert werden. Die beschriebenen siebenfarbigen Multiplex-IHC-Protokolle verwenden die Opalfluorophore Opal480, Opal520, Opal570, Opal620, Opal690, Opal780 und 4',6-Diamidino-2-phenylindol (DAPI), so dass mit "Multispectral One Touch ImmunoFluorescence" (MOTiF) ein einfaches Entmischen und schnelles Scannen auf dem Imager ermöglicht wird. Neunfarbiges Färben und Scannen wird in diesem Protokoll nicht beschrieben, da dies eine noch feinere Abstimmung des Versuchsaufbaus und einen anderen Scanmodus auf dem Imager erfordert, der den abstimmbaren Flüssigkristallfilter verwendet.
Protokoll
Patientenmaterial, das für dieses Protokoll gezeigt wird, war Teil einer zuvor durchgeführten Studie und wurde von der lokalen medizinischen Ethikkommission von Radboudumc in Übereinstimmung mit der niederländischen Gesetzgebung offiziell als von der medizinisch-ethischen Genehmigung ausgenommen eingestuft (Aktenzeichen 2017-3164)30.
1. Entnahme von FFPE-Material, Auswahl der Blöcke und Vorbereitung der Proben
- Rufen Sie FFPE-Block-Identifikatoren aus Patientenakten über behandelnde Ärzte oder Pathologen ab. Erkundigen Sie sich bei den örtlichen Vorschriften, ob eine ethische Genehmigung erforderlich ist.
- Fordern Sie FFPE-Blöcke beim örtlichen Pathologiearchiv oder bei externen Krankenhäusern an.
HINWEIS: Es ist auch möglich, dass Tumormaterial oder eine Biopsie für eine bestimmte Studie entnommen wird. Dies kann bei kleinen klinischen Versuchen oder Tierversuchen der Fall sein. In diesen Fällen kann die Aufbereitung der Gewebeprobe in der Verantwortung des Forschers liegen. - Wenn mehrere FFPE-Blöcke verfügbar sind, wählen Sie den repräsentativsten FFPE-Block aus, der lebensfähiges Tumorgewebe enthält, vorzugsweise mit umgebendem Stromagewebe, indem Sie die mit Hämatoxylin und Eosin (HE) gefärbten Objektträger beurteilen (Abbildung 1).
HINWEIS: Es wird empfohlen, für diese Auswahl ein Gutachten einzuholen (z. B. einen Pathologen). Es ist möglich, dass Hochschulen für die Bewertung des Inhalts eines FFPE-Blocks nicht zur Verfügung stehen und neue für die Auswahl erstellt werden müssen. Eine Beschreibung finden Sie in Abschnitt 2. - Schneiden Sie FFPE-Bänder mit einer Dicke von 4 μm auf ein Mikrotom.
HINWEIS: Die Dicke kann zwischen 1 μm und 6 μm liegen, ohne dass die Flecken spürbar werden. 4 μm ist jedoch der Standard. - Montieren Sie die Proben auf Objektträgern in einer Position, die für die Fluidik des Autostainers günstig ist (Abbildung 2A-C), und zwar mit einer der unten beschriebenen Methoden:
- Legen Sie die Abschnitte auf die Oberfläche von destilliertem 40 °C heißem Wasser in ein Wasserbad, um sie auszudehnen und mit einem Glasobjektträger aufzunehmen.
ODER
Legen Sie Objektträger auf eine 40 °C heiße Heizplatte und achten Sie darauf, dass die Stelle, an der das Profil auf der Schiene montiert werden soll, mit einem Tropfen destilliertem Wasser bedeckt ist. Legen Sie den Abschnitt mit einer Pinzette auf diesen Tropfen und lassen Sie ihn sich ausdehnen. Nehmen Sie destilliertes Wasser mit einem Papiertuch auf und entfernen Sie überschüssiges Wasser, indem Sie auf die Rutsche klopfen.
HINWEIS: Wenn Gewebeschnitte zu nahe an der Beschriftung des Objektträgers platziert werden, führt dies zu einer suboptimalen Färbung (Abbildung 2D,E). Wir neigen dazu, 6-10 Objektträger pro Probe zu montieren, um die verschiedenen Multiplex-IHC-Panels durchzuführen und ein Backup zu haben.
- Legen Sie die Abschnitte auf die Oberfläche von destilliertem 40 °C heißem Wasser in ein Wasserbad, um sie auszudehnen und mit einem Glasobjektträger aufzunehmen.
- Lassen Sie die montierten Objektträger 1 h bei 56 °C oder über Nacht bei 37 °C trocknen.
- Verwenden Sie für das Experiment die montierten Objektträger oder lagern Sie diese in Boxen bei 4 °C.
HINWEIS: Nach unseren bisherigen Erfahrungen können diese gerahmten Objektträger jahrelang gelagert werden, bevor eine Multiplex-IHC-Färbung durchgeführt wird.
2. Erzeugung von Hämatoxylin- und Eosin-gefärbten Objektträgern
HINWEIS: Alle folgenden Schritte in Abschnitt 2 sind in einem Abzug durchzuführen.
- Objektträger in Xylol entparaffinieren (2 x 5 min).
- Rehydrieren Sie in Ethanol (99,6 % 1 x 5 min; 95 % 1 x 5 min; 70 % 1 x 2 min). Alternativ können Sie die Objektträger 3x in 99,6% Ethanol tauchen.
- Waschen Sie die Objektträger in destilliertem Wasser (2 min).
- Färben Sie die Zellkerne mit Hämatoxylin (10 min).
- Waschen Sie die Objektträger mit destilliertem H2O (5 min).
- Färben Sie die Dias mit Eosin (5 min).
- Dehydrieren Sie die Objektträger, indem Sie 3x in 99,6% Ethanol tauchen.
- Tauchen Sie die Objektträger 2x in Xylol.
- Geben Sie ein paar Tropfen Eindeckmedium hinzu und verschließen Sie es mit einem Deckglas.
- Lassen Sie die Rutschen aushärten und nehmen Sie die Folien aus dem Abzug, wenn alle Chemikalien verdunstet sind.
3. Durchführung von Monoplex- und Multiplex-IHC im Autostainer
- Berechnen Sie, wie viel Reagenz in Abhängigkeit von der Anzahl der zu färbenden Proben benötigt wird.
HINWEIS: Pro Lauf hat der Autostainer eine Kapazität von 30 Objektträgern und benötigt ~18 Stunden, um das Multiplex-IHC-Protokoll mit sechs Antikörpern abzuschließen. Wenn mehr Objektträger gebeizt werden müssen, können jede Nacht der (Arbeits-)Woche mehrere Chargen eingelegt werden. 4 Nächte mit 30 Rutschen = 120 Rutschen pro Woche.- Bereiten Sie zu Beginn der Woche alle notwendigen Reagenzien vor. Das Autostainer-System dosiert 150 μl Reagenz pro Objektträger. Verwenden Sie die 6-ml-Titrationsbehälter für Antikörper- und Opal-Reagenzien und die 30-ml-Behälter für das Blockierungsreagenz und die sekundäre Antikörper-Meerrettich-Peroxidase.
HINWEIS: Die 6-ml-Behälter verfügen über praktische Einsätze, die bei Bedarf leicht herausgenommen und ausgetauscht werden können. Bei Reagenzienberechnungen muss man das Totvolumen von 1,6 mL bzw. 300 μL für den 30 mL Behälter bzw. 6 mL Titrationsbehälter berücksichtigen. - Verdünnen Sie alle Opalfluorophore und Digoxigenin (DIG) 1:100 in dem mitgelieferten Verdünnungsmittel; Opal780 1:25 im Antikörperverdünnungsmittel verdünnen. Verdünnen Sie alle Primärantikörper in Antikörperverdünnungsmittel mit den in der Zusatzdatei 1 angegebenen Verdünnungen.
- Bereiten Sie zu Beginn der Woche alle notwendigen Reagenzien vor. Das Autostainer-System dosiert 150 μl Reagenz pro Objektträger. Verwenden Sie die 6-ml-Titrationsbehälter für Antikörper- und Opal-Reagenzien und die 30-ml-Behälter für das Blockierungsreagenz und die sekundäre Antikörper-Meerrettich-Peroxidase.
- Um diesem Protokoll zu folgen, führen Sie Monoplex IHC (Supplemental File 2) auf Objektträgern durch, die sowohl Mandelkontrollgewebe als auch andere (Tumor-)Gewebetypen von Interesse enthalten, bevor Sie mit dem eigentlichen Multiplex-IHC-Experiment beginnen, um sicherzustellen, dass alle Reagenzien gut vorbereitet sind.
HINWEIS: Monoplex IHC dauert ~3,5 h und kann vor Ende des Tages auf Signalmuster und -intensität überprüft werden. Sind bestimmte Signale zu schwach (Abbildung 3), können Anpassungen an den Reagenzien vorgenommen werden. - Für die Autofluoreszenzkorrektur bereiten Sie einen Objektträger mit (Tumor-)Gewebe vor, das autofluoreszierende Strukturen wie Blut und Kollagen enthält. Bereiten Sie diesen Objektträger gleichzeitig mit Monoplex-IHC-Objektträgern vor, jedoch mit einem Blockierungsreagenz, das den Antikörper und die Opal-Reagenzien ersetzt (Ergänzende Datei 3).
HINWEIS: Prinzipiell kann ein solcher Objektträger für die multispektrale Bildgebung wiederverwendet werden, bis die Autofluoreszenzkorrektur nicht mehr optimal ist. Bei stark autofluoreszierenden Geweben wie Gehirn und Leber ist es jedoch ratsam, dieses Gewebe für die Autofluoreszenzkorrektur zu verwenden. - Laden Sie bei jedem Multiplex-IHC-Lauf 29 Proben mit einem Kontrollgewebeobjektträger in das Autostainer-System, um die Leistung jedes Multiplex-IHC-Laufs zu überprüfen.
- Multiplex-IHC-Protokolle von der Website des Autostainers unter dem Reiter Downloads herunterladen und an jedes kundenspezifische Multiplex-IHC-Panel36 anpassen. Für Multiplex-IHC siehe Ergänzende Datei 4 für das Protokoll und für kundenspezifische Multiplex-IHC-Panels siehe Ergänzende Datei 1.
- Nehmen Sie nach Abschluss des Färbeprotokolls die Objektträger aus dem Autostainer und legen Sie sie in einen Behälter mit Waschpuffer.
- Um eine Kontamination des Autostainer-Systems mit DAPI zu vermeiden, da die Proben bereits in sehr geringen Konzentrationen gefärbt sind, tragen Sie DAPI manuell auf, bevor Sie die Objektträger mit Deckgläsern abdecken. Geben Sie zwei Tropfen DAPI pro ml Waschpuffer hinzu und inkubieren Sie 5 Minuten lang bei Raumtemperatur im Dunkeln.
HINWEIS: Beim Erstellen von Spektralbibliotheken ist es wichtig, dass keine DAPI in den Proben gefärbt sind. Ein Tropfen DAPI pro mL Waschpuffer und eine 10-minütige Inkubation bei RT sind ebenfalls möglich. - Waschen Sie die Objektträger 3x mit Waschpuffer.
- Legen Sie die Objektträger auf Küchenpapier und klopfen Sie den überschüssigen Waschpuffer von den Objektträgern.
- Pipettieren Sie ein paar Tropfen Einbettmedium auf das Gewebe.
- Legen Sie ein Deckglas vorsichtig auf das Eindeckmedium, um das Objektträger schräg abzudecken und Luftblasen zu vermeiden.
- Entfernen Sie überschüssiges Eindeckmedium und Luftblasen, indem Sie mit einer Pinzette oder einer sauberen Pipettenspitze vorsichtig auf das Glasdeckglas drücken.
- Lassen Sie die Objektträger ~24 h lang ungestört, bevor das Eindeckmedium erstarrt, entweder horizontal auf einem Mikroskopie-Objektträger oder laden Sie sie direkt in das Mikroskop für die Bildgebung.
- Nachdem das Eindeckmedium verfestigt wurde oder nachdem die Objektträger abgebildet wurden, lagern Sie die Objektträger in Mikroskopierboxen bei 4 °C.
4. Bildgebung mit dem Digital Pathology Imager und Annotation von Scandateien
- Schalten Sie den Imager ein, indem Sie den Netzschalter auf der rechten Seite des Geräts drücken. Starten Sie die Software nach mindestens 20 s.
HINWEIS: Warten Sie 20 s, bis die Hardware korrekt gestartet wird. - Legen Sie die Dias in die Kassetten pro vier Dias ein.
- Optional: Tragen Sie die Folien in eine .csv Datei ein, für die eine Vorlage heruntergeladen werden kann (Ergänzungsdatei 5). Um die .csv Datei in das Programm zu laden, speichern Sie sie unter C:\Users\Public\Akoya\VectraPolaris\States.
HINWEIS: Es können maximal 20 Kassetten oder 80 Dias gleichzeitig eingelegt werden.
- Optional: Tragen Sie die Folien in eine .csv Datei ein, für die eine Vorlage heruntergeladen werden kann (Ergänzungsdatei 5). Um die .csv Datei in das Programm zu laden, speichern Sie sie unter C:\Users\Public\Akoya\VectraPolaris\States.
- Referenz-Einstellungen
- Öffnen Sie Check Dashboard aus dem Hauptmenü.
HINWEIS: Eine Kassette mit Referenzdias wird vom Hersteller mitgeliefert und kann optional dauerhaft im Steckplatz 20 aufbewahrt werden. - Setzen Sie die Hellfeldreferenzen einmal pro Woche auf der bereitgestellten Folie nach Herstellerangaben (dauert einige Minuten).
- Stellen Sie die Fluoreszenzreferenzen auf dem mitgelieferten Objektträger einmal pro Monat gemäß den Anweisungen des Herstellers ein (dauert mehr als 1 h).
- Öffnen Sie Check Dashboard aus dem Hauptmenü.
- Erstellen oder Anpassen des Protokolls
- Gehen Sie zurück zum Hauptmenü und klicken Sie auf Protokoll bearbeiten , um ein Protokoll zu erstellen.
- Klicken Sie auf Neu... und wählen Sie Fluoreszenz als Bildgebungsmodus, Multispektraler Dia-Scan und Opal Polaris 5, 6 und 7 Farbe unter der Option Färbung .
- Geben Sie dem Protokoll unter Protokollname einen Namen, und speichern Sie es unter einer Studie, indem Sie eine Studie aus Verfügbare Studien auswählen, oder erstellen Sie eine Studie unter Neue Studie erstellen | Name der Studie.
- Wählen Sie abschließend Protokoll erstellen aus.
- Verwenden Sie für diese Art des Scannens nur die Einstellungen für multispektrale Objektträgerscans im linken Fenster. Ignorieren Sie das Fenster auf der rechten Seite Multispektrale Feldeinstellungen.
- Scannen Sie die Dias in verschiedenen Vergrößerungen. Um diesem Protokoll zu folgen, scannen Sie mit 20-facher Vergrößerung, indem Sie die Pixelauflösung bei 0,50 μm (20x) belassen.
- Legen Sie die Belichtungszeiten fest, indem Sie Scan-Belichtungen auswählen.
- Laden Sie die Kassette, in der sich die Dias befinden, indem Sie unter der Option Ladungsträger den richtigen Schlitz auswählen.
- Um die Navigation durch die Folien zu erleichtern, wählen Sie Übersicht erstellen , um ein Übersichtsbild des Trägers mit den Folien zu erhalten, nachdem der Träger geladen wurde. Um dies automatisch ein- oder auszuschalten, klicken Sie auf das Zahnradsymbol oben rechts, gehen Sie zu Einstellungen... und aktivieren Sie die Option ein oder aus unter Navigation Übersichtsbild , um den Bildträger beim Laden automatisch für interaktive Aufgaben zu aktivieren.
- Stellen Sie die Belichtungszeiten pro Filter auf den entsprechenden Monoplex-IHC-gefärbten Objektträgern ein, indem Sie Scan-Belichtungen einstellen auswählen und verschiedene Stellen mit einem positiven Signal finden. Fokussieren Sie manuell oder verwenden Sie den Autofokus und wählen Sie Autoexponieren , nachdem Sie zum kompatiblen Filter für dieses Signal gewechselt haben. Wählen Sie die niedrigste Belichtungszeit, um eine Überbelichtung zu vermeiden, und machen Sie Schnappschüsse jeder Folie als Referenz, nachdem alle Belichtungszeiten eingestellt wurden (Abbildung 3).
HINWEIS: Ignorieren Sie die Option " Feldbelichtungen festlegen" für diese Art des Scannens. - Stellen Sie die Belichtungszeiten auf einem Multiplex-Dias ein, indem Sie alle Filter an einigen Stellen mit positivem Signal überprüfen. Reduzieren Sie die niedrigste Belichtungszeit um 10 %, um eine Überbelichtung zu vermeiden, und machen Sie einige Schnappschüsse, nachdem alle Belichtungszeiten eingestellt sind.
- Machen Sie Schnappschüsse des ungefärbten Objektträgers für die Autofluoreszenzkompensation, indem Sie den Proben-AF-Filter zur Navigation verwenden (Abbildung 3H).
HINWEIS: Stellen mit Erythrozyten und Kollagenstrukturen sind von Interesse. Die Belichtungszeit des Opal480-Filters muss bei stark autofluoreszierenden Bereichen möglicherweise verkürzt werden. Wenn das Opal480-Signal stark genug ist, sollte es aufgrund der Implementierung des proprietären Sample AF-Filters dennoch gut von den autofluoreszierenden Strukturen getrennt werden (siehe Abschnitt 6). - Beurteilen Sie die Qualität der Färbung und Bildgebung mit der Software (siehe Abschnitte 5 und 6; Abbildung 4, Ergänzende Akte 6: Ergänzende Abbildung S1 und Ergänzende Abbildung S2).
- Wählen Sie die Schaltfläche Speichern... , um sicherzustellen, dass das Protokoll und die angepassten Belichtungszeiten im Protokoll gespeichert werden.
HINWEIS: Wenn das Protokoll bereits gespeichert ist, gibt die Software bisher keine zusätzliche Benachrichtigung über nicht gespeicherte Anpassungen.
- Automatisches Scannen von Dias
- Gehen Sie zurück zum Hauptmenü und klicken Sie auf Folien scannen , um die Folien zu scannen.
- Geben Sie die Foliennamen/IDs und die entsprechenden Aufgaben und Protokolle manuell unter Aufgaben konfigurieren ein oder automatisch aus der zuvor erstellten .csv Datei mit Load Setup.
- Klicken Sie auf Scannen , um den Scanvorgang zu starten.
- Warten Sie, bis ein Fenster angezeigt wird, um die Scan-Einrichtung zu speichern. Klicken Sie auf Speichern , um die Standardeinstellungen zu verwenden und den Scanvorgang zu starten.
HINWEIS: Das Scannen mit dieser Methode dauert ~10-20 Minuten pro Objektträger. Abhängig von der Anzahl der Objektträger kann das Scannen bis zu einem ganzen Tag dauern. - Überprüfen Sie, ob das Scannen der Objektträger für alle Objektträger erfolgreich war, indem Sie nach Fehlermeldungen suchen. Um festzustellen, ob der Scanvorgang erfolgreich war, suchen Sie nach einer gespeicherten Akoya-Datei für den gesamten Objektträger (.qptiff) des Scans und des gesamten Gewebes im Scan.
5. Annotation von Daten mit dem Slide Viewer
- Gehen Sie zurück zum Hauptmenü und klicken Sie auf Phenochart starten , um den Folienbetrachter zu öffnen.
- Wenn die Scandateien nicht direkt sichtbar sind, legen Sie ihren Speicherort fest, indem Sie zuerst auf das Zahnradsymbol in der oberen rechten Ecke klicken, gehen Sie zu Browserspeicherort ändern... und wählen Sie nach dem Zufallsprinzip eine der .qptiff-Dateien des gewünschten Datensatzes aus.
HINWEIS: Die Daten werden standardmäßig unter D:\Data\VectraPolaris gespeichert. - Laden Sie eine Folie, indem Sie sie auswählen und in der oberen rechten Ecke auf Laden klicken oder indem Sie darauf doppelklicken.
- Melden Sie sich an, indem Sie auf die Schaltfläche "Anmelden" in der oberen rechten Ecke klicken.
HINWEIS: Der Benutzername kann nur aus den Initialen oder dem Namen bestehen und wird verwendet, um nachzuverfolgen, wer welche Anmerkungen gemacht hat. - Um das Mischen aufzuheben, klicken Sie oben auf die Schaltfläche Mischen aufheben und wählen Sie die Option Opal + AF .
HINWEIS: Dies ist nützlich, um einen Teil des autofluoreszierenden Signals in der Nähe des Opal 480-Kanals zu entfernen, aber nicht alle. - Um einen Algorithmus für die Stapelverarbeitung der Daten zu generieren, wählen Sie repräsentative Bilder mit dem Stempel mit der Option für inForm-Projekte 1 x 1-Bilder (Bildgröße: 928 μm x 696 μm) aus.
HINWEIS: Einige repräsentative Stempel mit Tumor-, Stroma-, Hintergrund- und verschiedenen Arten von Immunzellen werden im gesamten Datensatz ausgewählt, um am Ende ~20-30 Bilder zu erhalten. - Je nachdem, was im Gewebe analysiert werden muss, wählen Sie mit der ROI-Option einen Bereich von Interesse aus und wählen Sie für inForm Batch. Löschen Sie manuell Bilder, die nicht analysiert werden müssen, z. B. Bilder, die zu weit vom Tumor entfernt oder im Hintergrund sind.
HINWEIS: Wir neigen dazu, einen ROI um den gesamten Tumor herum zu zeichnen und ein zusätzliches Bild außerhalb der Tumorregion auszuwählen, um einen IM von ~0,5 mm analysieren zu können.
Wenn der erzielte ROI relativ klein ist, besteht der ROI aus 2-9 zusammengeführten 20x-Bildern. Da dies von uns nicht bevorzugt wird, stempeln Sie das gewünschte Gewebe (ausgewählt für inForm Batch) manuell, um dies zu umgehen. - Wenn Sie mit dem Kommentieren fertig sind, lassen Sie die Anmerkungen automatisch speichern und laden Sie die nächste Folie.
- Überprüfen Sie während des Anmerkungsvorgangs, ob die Folien korrekt gescannt wurden.
- Wenn eine .qptiff-Datei fehlt oder ein Objektträger nicht erfolgreich gescannt wurde, überprüfen Sie, ob Gewebe auf dem Objektträger vorhanden ist, reinigen Sie den Objektträger mit 70 % Ethanol und scannen Sie erneut.
- Wenn das Gewebe nicht vollständig gescannt wird und dadurch eine potenziell wichtige (Tumor-)Region fehlt, oder wenn die Abtastung der wichtigen Region unscharf war, reinigen Sie den Objektträger mit 70%igem Ethanol und scannen Sie erneut.
HINWEIS: In beiden Fällen kann es auch hilfreich sein, das Gewebe mit einem Marker auf dem Deckglas zu umgeben, um dem System zu helfen, das Gewebe zu lokalisieren und den Scanvorgang erneut zu versuchen (Ergänzende Datei 6: Ergänzende Abbildung S3). In unseren Händen funktionierte ein dünner roter Marker besser als ein dicker schwarzer Marker.
- Sobald das Scannen und die Anmerkungen aller Proben abgeschlossen sind, sichern Sie die Daten, indem Sie sie auf einem anderen Computer oder einer externen Festplatte speichern.
6. Spektrale Entmischung
- Öffnen Sie die inForm Automated Image Analysis Software.
- Laden Sie die Bilder über Datei | Bild öffnen; Wählen Sie .qptiff-Dateien aus. Lassen Sie die Stempel, die in Schritt 5.6 als inForm-Projekte markiert wurden, in das Projekt laden.
- Laden Sie die .qptiff-Dateien, die für die Autofluoreszenzkompensation abgebildet wurden.
- Um die Autofluoreszenz zu kompensieren, verwenden Sie das Werkzeug Autofluoreszenz auf dem Bild auswählen , um eine Linie auf dem Bild von der ungefärbten Folie durch verschiedene Arten von Strukturen zu ziehen, die autofluoreszierend sind, wie z. B. Erythrozyten und Kollagen.
- Weisen Sie im Abschnitt "Marker und Farben bearbeiten..." Markernamen zu, die dem Opalfluorophor entsprechen, und passen Sie die Farbe an die gewünschte Farbe an.
- Um die Fluorophore zu entmischen, wählen Sie in der unteren linken Ecke die Option Alle vorbereiten aus.
- Gehen Sie die Bilder durch und prüfen Sie, ob alle Signale auf den Bildern sichtbar sind und ob die Entmischung gut verlaufen ist. Wählen Sie das Augapfelsymbol aus, um alle Marker nacheinander ein- und auszuschalten und die Qualität zu überprüfen.
- Trainieren Sie optional die Algorithmen für die Gewebesegmentierung, die Zellsegmentierung und die Phänotypisierung.
- Gehen Sie zur Registerkarte Exportieren und erstellen Sie ein neues leeres Exportverzeichnis, indem Sie auf die Schaltfläche Durchsuchen... unter dem Exportverzeichnis klicken.
- Wählen Sie unter den zu exportierenden Bildern die Option Composite-Image und Component-Images (TIFF mit mehreren Bildern) aus.
- Wählen Sie Datei | Speichern | Projekt, um den Algorithmus an einem bestimmten Ort zu speichern.
- Gehen Sie vertikal auf der linken Seite zur Registerkarte Stapelanalyse , um die Stapelverarbeitung von Objektträgern durchzuführen.
- Wählen Sie unter den Exportoptionen die Option Separate Verzeichnisse für jedes Element erstellen aus.
- Um Folien für die Analyse hinzuzufügen, wählen Sie unter der Schaltfläche Folien hinzufügen... .qptiff-Dateien aus und laden Sie sie in die Batch-Analyse.
- Wählen Sie Ausführen aus, um die Stapelverarbeitung von Folien zu starten.
7. ROI-Zeichnung
- Erstellen Sie einen Ordner, der nur die Komponentendateien aus Abschnitt 6 enthält, aber behalten Sie die hierarchische Ordnerstruktur bei (Komponentendateien befinden sich in Ordnern, die nach Beispiel/Folie benannt sind).
- Öffnen Sie die QuPath Software für den gesamten Diabetrachter.
- Klicken Sie links auf Projekt erstellen und wählen Sie einen neuen leeren Ordner mit einem geeigneten Namen aus.
- Klicken Sie auf Automatisieren und wählen Sie Skript-Editor anzeigen aus.
- Kopieren Sie das Skript, das in der Zusatzdatei 7 verfügbar ist, und fügen Sie es ein. Ändern Sie in Zeile 34 den Speicherort der Folienordner mit allen Komponentendateien (der Ordner, der in Schritt 7.1 erstellt wurde.
- Wählen Sie Ausführen und zurückkehren, wenn das Stapelheften von Folien abgeschlossen ist (am nächsten Tag oder später), um fortzufahren.
- Ziehen Sie die generierten .ome.tif Dateien in das QuPath-Projekt und speichern Sie sie als Projekt.
- Wenn automatisch ein neues Fenster angezeigt wird, wählen Sie Bildtyp festlegen | Fluoreszenz und klicken Sie auf Importieren.
- Beobachten Sie im Menü auf der linken Seite die Liste der Proben. Doppelklicken Sie auf eine Taste, um das Beispiel zu öffnen (Abbildung 5A).
- Um die Intensität der Kanäle anzupassen und sie besser sichtbar zu machen, klicken Sie auf das Kontrastsymbol.
- Wählen Sie alle Kanäle aus und klicken Sie auf Zurücksetzen.
- Schalten Sie die Autofluoreszenz aus.
- Um mit der Erstellung eines ROI für den Tumor zu beginnen, klicken Sie auf das Kontrastsymbol und wählen Sie Graustufen anzeigen. Wählen Sie den Tumormarkerkanal aus und passen Sie die Intensität an, um ihn optimal sichtbar zu machen (Abbildung 5B).
- Klicken Sie auf das Pinselwerkzeug , um einen ungefähren ROI für den Tumor zu zeichnen.
- Klicken Sie bei der Auswahl des Zauberstab-Werkzeugs außerhalb des ROI, während Sie die Alt-Taste drücken, um den ROI von außen zu glätten (Abbildung 5C).
- Führen Sie getrennte Tumorstücke mit dem gleichen ROI zusammen.
- Geben Sie dem ROI einen passenden Namen, z. B. Tumor , indem Sie mit der rechten Maustaste auf die Anmerkung in der Liste auf der linken Seite klicken. Wählen Sie Eigenschaften festlegen und geben Sie den Namen ein.
- Um einen ROI für den IM zu erstellen, erweitern Sie den vorhandenen ROI aus der Tumorregion, indem Sie Folgendes auswählen: Objekte | Anmerkungen... | Erweitern Sie Anmerkungen.
- Wählen Sie die Größe des Erweiterungsradius aus, und wählen Sie "Innenraum entfernen " und "Auf übergeordnetes Element beschränken" aus (Abbildung 5D).
- Klicken Sie auf das Kontrastsymbol, wählen Sie den Autofluoreszenzkanal aus und passen Sie die Intensität an, um ihn optimal sichtbar zu machen.
- Klicken Sie auf den Zauberstab und passen Sie den ROI an, während Sie die Alt-Taste drücken, um den ROI von außen zu glätten und alle Hintergründe zu entfernen, die nicht Teil dieses ROI sein sollten.
- Geben Sie dem ROI einen passenden Namen, z. B. invasiver Rand oder IM , indem Sie mit der rechten Maustaste auf die Anmerkung in der Liste auf der linken Seite klicken, Eigenschaften festlegen auswählen, den Namen eingeben und optional die Farbe in Grün ändern.
- Speichern Sie die Anmerkungen: Datei | Objekte exportieren | Alle Objekte exportieren, und klicken Sie mit der Standardauswahl bei Als FeatureCollection exportieren auf OK, und speichern Sie sie an einem bevorzugten Speicherort.
8. Nachweis von Immunzellen
- Da ImmuNet Komponentendaten (Mehrkanal-TIFF-Dateien) sowohl für das Training als auch für die Inferenz verwendet, sollten Sie die Anmerkungen in Trainings- und Validierungssätze aufteilen. Um das Modell zu trainieren, führen Sie die in der Readme-Datei des Repositorys beschriebenen Schritte aus, und ersetzen Sie das Beispieldataset und die Anmerkungen durch die gewünschten Daten. Versorgen Sie das Modell neben verschiedenen Immunzellen mit Negativbeispielen, indem Sie Hintergrundanmerkungen an Stellen vornehmen, die nicht als Zelle von Interesse erkannt werden sollten: Tumorzellen, andere Zellen oder "keine Zellen" (Strukturen, die mit Zellen von Interesse verwechselt werden könnten); siehe die ImmuNet-Publikationfür Details 32.
- Stellen Sie mithilfe der Validierungsanmerkungen sicher, dass die Leistung zufriedenstellend ist. Betrachten Sie die Fehlerrate pro Anmerkungstyp – ein Anteil der Validierungsanmerkungen, die das Modell nicht erkannt hat – die einfachste Auswertungsmetrik. Bewerten Sie die Leistung in Bezug auf Fehlalarme, indem Sie einige vollständig kommentierte ROIs erstellen und die F-Scores berechnen.
- Zusätzlich zur quantitativen Bewertung sollten Sie die Vorhersage visuell überprüfen, um ein qualitatives Gefühl für die Fehler zu erhalten, die das Modell tendenziell macht (Abbildung 6, Ergänzende Datei 6: Ergänzende Abbildung S4 und Ergänzende Abbildung S5). Wenn die Modellleistung als unzureichend eingestuft wird, visualisieren Sie die Vorhersage für einige Kacheln, wie im Repository beschrieben, und prüfen Sie, welche Standorte am fehleranfälligsten sind. Nehmen Sie an solchen Stellen weitere Anmerkungen vor, und führen Sie das Training und die Auswertung des Modells erneut durch.
- Wenn die Zielleistung erreicht ist, führen Sie den Rückschluss für den gesamten Datensatz aus, wie im Abschnitt Rückschluss für den gesamten Datensatz der Repository-Readme-Datei beschrieben. Verwenden Sie die erhaltenen .csv Dateien mit der Modellvorhersage als Eingabe für die Datenanalyse (schreiben Sie dafür ein Python- oder R-Skript).
9. Vorhersage-Phänotypisierung und Datenanalyse
HINWEIS: In diesem Abschnitt geben wir ein Beispiel für eine einfache Datenanalyse für eine einzelne Melanomprobe, die mit dem Lymphozyten-Panel gefärbt wurde, das die mit ImmuNet identifizierten Positionen von Immunzellen (Abschnitt 8) und die mit QuPath abgegrenzten ROIs (Abschnitt 7) kombiniert. Die Analyse wurde in R 4.1.1 durchgeführt (ein Skript wird als Ergänzungsdatei 8 bereitgestellt). Das Skript benötigt die Pakete: plyr 1.8.8, dplyr 1.0.8, tidyr 1.2.0, sf 1.0-7, ggplot2 3.4.0, RANN 2.6.1 und RColorBrewer 1.1-2, die mit dem Befehl install.packages() installiert werden können. Als Eingabe wird eine .csv Datei mit der ImmuNet-Vorhersage einer Stichprobe und eine Datei mit ROIs verwendet, die aus QuPath exportiert wurden. Die Schritte 9.1-9.6 beschreiben die Analyse einer einzelnen Probe, die im bereitgestellten Skript durchgeführt wird, und die Abschnitte 9.7-9.9 beschreiben Optionen für die Analyse mehrerer Proben.
- Nachdem Sie die Vorhersage von ImmuNet in R geladen haben, bestimmen Sie die Schwellenwerte für die vorhergesagte Markerexpression, indem Sie die Marker, die die Phänotypen definieren, gegeneinander darstellen und die Schwellenwerte auswählen, die die Populationen am besten voneinander trennen.
HINWEIS: Die für die gegebene Stichprobe verwendete Gating-Strategie ist in Abbildung 7B dargestellt. Gating-Strategien für die myeloischen und dendritischen Zellpanels sind in der ergänzenden Datei 6 dargestellt: Ergänzende Abbildung S6 und ergänzende Abbildung S7. - Nachdem Sie die Schwellenwerte bestimmt haben, verwenden Sie sie, um jeder ImmuNet-Vorhersage einen Phänotyp zuzuweisen, der in einem Panel definiert ist. Bei einigen Vorhersagen ist zu beachten, dass keiner der vorhergesagten Marker über dem Schwellenwert liegt oder dass die Kombination von Markern, die nach dem Schwellenwert exprimiert werden, inkonsistent sein kann (z. B. CD3+ CD20+ Vorhersagen in den Lymphozyten-Panels). Wenn in Schritt 8.3 eine gute Modellleistung erreicht wird, ist der Anteil solcher Vorhersagen gering. Filtern Sie diese vor der Analyse heraus.
- Um die ROIs für den Tumor und seinen invasiven Rand von bis zu 100 μm, der in QuPath gezeichnet wurde, separat zu analysieren, laden Sie die entsprechenden GeoJSON-Dateien in R und bestimmen Sie für jede Vorhersage den ROI, in den die Vorhersage fällt.
- Für eine Plausibilitätsprüfung und als Teil der explorativen Datenanalyse visualisieren Sie die in einer Probe gefundenen Immunzellen getrennt in den entsprechenden ROIs zusammen mit den ROI-Grenzen (Abbildung 7A).
- Berechnen Sie nun die Dichten verschiedener Immunzellen separat für jeden ROI. Die in der gegebenen Probe gefundenen Dichten sind in Tabelle 1 dargestellt.
- Wenn mehrere Proben verfügbar sind, visualisieren Sie die Verteilung der Zelldichten. Logarithmisch transformieren Sie die Dichtewerte, um normalverteilte Werte zu erreichen.
HINWEIS: Wenn die Anzahl bestimmter Phänotypen 0 ist, können diese nicht logarithmisch transformiert werden, was zu fehlenden Werten führt. Um dieses Problem zu lösen, kann die LaPlacian-Glättung angewendet werden, indem zuerst 0,5 zu allen Zellzahlen addiert wird, bevor sie durch die Oberfläche dividiert wird. - Analysieren Sie die Dichtewerte, und stellen Sie sie mit der Software Ihrer Wahl dar (Abbildung 8).
- Beibehaltene Positionen von Zellen ermöglichen eine räumliche Analyse. Suchen Sie beispielsweise für jede erkannte Immunzelle einen nächsten Nachbarn und berechnen Sie dann für jeden Phänotyp den Prozentsatz der Fälle, in denen die verschiedenen Phänotypen als nächster Nachbar auftreten.
HINWEIS: Da die Anzahl der in dieser Probe gefundenen natürlichen Killerzellen (NK) sehr gering war, haben wir sie von dieser Analyse ausgeschlossen. Die erhaltenen Ergebnisse für Tumor- und IM-ROIs sind in Abbildung 9 dargestellt.
Ergebnisse
FFPE-Blöcke, die Tumorgewebe enthalten, wurden auf der Grundlage von pathologischen Berichten und HE-gefärbten Objektträgern ausgewählt. Wenn mehrere Tumorläsionen des Patienten reseziert werden und/oder Tumorproben groß sind, werden diese auf mehrere FFPE-Blöcke aufgeteilt. Wir bevorzugen es, Immunzellen sowohl im Tumorkompartiment als auch am sogenannten invasiven Rand (IM) des Tumors zu analysieren. Das IM ist nicht-krebsartiges Stromagewebe, das an den Tumor angrenzt. Wenn also mehrere FFPE-Blöcke für eine Tumorprobe verfügbar sind, werden die FFPE-Blöcke ausgewählt, die beide Gewebetypen enthalten. Wie auf den HE-gefärbten Objektträgern zu sehen ist, enthielt ein FFPE-Block Tumorgewebe und Stromagewebe, das an den Tumor angrenzte (Abbildung 1A). Ein anderer FFPE-Block aus demselben Tumor enthielt viel weniger umgebendes Stromagewebe (Abbildung 1B). Für einige Gewebeproben gibt es jedoch keine Wahl in FFPE-Blöcken oder der IM ist in keinem der FFPE-Blöcke vorhanden. Dies ist häufig bei (Nadel-)Biopsien der Fall, die bei der Dateninterpretation im Auge behalten werden müssen.
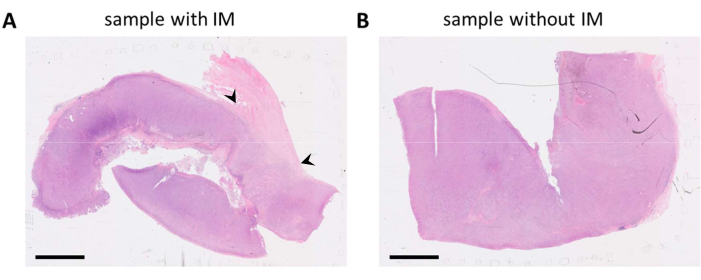
Abbildung 1: HE-gefärbte Objektträger einer Melanomtumorprobe. (A) Ein Beispiel für eine Tumorprobe mit stromalem Gewebe neben dem Tumor (IM) in der oberen rechten Ecke der Probe (gekennzeichnet durch schwarze Pfeilspitzen). (B) Eine weitere Probe aus derselben Tumorläsion mit wenig bis gar keinem Stromagewebe in der Probe. Maßstabsleisten = 5 mm. Abkürzungen: HE = Hämatoxylin und Eosin; IM = invasiver Rand. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Die Multiplex-IHC-Färbung mit einem vorgeschlagenen siebenfarbigen Panel (Supplemental File 4) kann entweder manuell in einem 3-tägigen Färbeprozess (unter Berücksichtigung der normalen Arbeitszeiten) oder über Nacht in einem Autostainer durchgeführt werden. Bei der Verwendung des Autostainers müssen die Abschnitte an einer bestimmten Stelle auf dem Objektträger montiert werden, die eine optimale Fluidik des Systems ermöglicht (Abbildung 2A). Wenn die Abschnitte korrekt auf Objektträgern montiert sind (Abbildung 2B), werden sie gleichmäßig gebeizt (Abbildung 2C). Werden Schnitte nicht optimal auf dem Objektträger montiert (Abbildung 2D), ergibt sich oft ein suboptimales Färbebild (Abbildung 2E), da die Fluidik des Autostainers das (vollständige) Gewebe nicht erreicht. Dies kann der Fall sein, wenn die Proben sehr groß sind oder wenn gemountete Objektträger von jemandem bereitgestellt werden, der sich dieses Problems nicht bewusst ist. In diesen Fällen sollte nur der gut gefärbte Teil des Objektträgers für die Analyse ausgewählt werden. Eine weitere Möglichkeit für diese Art von Proben könnte darin bestehen, sie manuell zu färben, um die Flüssigkeiten optimal zu verteilen.
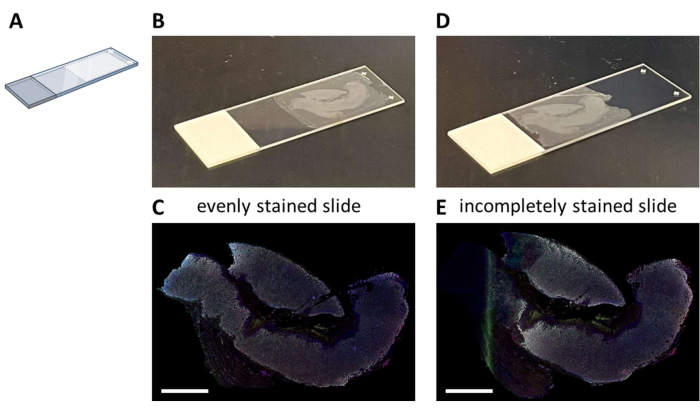
Abbildung 2: Montage des FFPE-Profils auf dem Objektträger und Aufprall. (A) Schematische Darstellung der Montagestelle auf dem Objektträger für eine optimale Färbung auf dem Autostainer. (B) Beispiel für eine korrekt montierte Rutsche. (C) Korrekt montierte Objektträger führen zu einem gleichmäßig gefärbten Gewebeschnitt. (D) Beispiel für eine suboptimale montierte Folie. (E) Suboptimale montierte Objektträger können zu einem unvollständigen gefärbten Gewebeschnitt führen, wie auf der linken Seite dieses Bildes zu sehen ist. Maßstabsleisten = 5 mm. Abkürzung: FFPE = formalin-fixed und paraffin-embedded. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Wenn große Multiplex-IHC-Experimente in mehreren Färberunden durchgeführt werden und große Mengen an Lösungen hergestellt werden müssen, ist es am besten, diese Reagenzien zuerst in einem Monoplex-IHC-Lauf zu testen, bevor Sie mit dem Multiplex-IHC fortfahren. Die Monoplex IHC wird mit dem digitalen Pathologie-Imager auf erwartete Färbemuster überprüft und die Belichtungszeiten werden mit den entsprechenden Filtern auf den Kontrollobjektträgern eingestellt (Abbildung 3A-H). Mandelgewebe wird als Positivkontrolle für die meisten Immunzellmarker verwendet. Da die DAPI-Expositionszeit in Mandelkontrollgewebe immer höher ist als in anderen Geweben (Abbildung 3G), muss die DAPI-Expositionszeit für den zu untersuchenden Gewebetyp eingestellt werden. Die regulären Belichtungszeiten bei dieser Art der Abtastung liegen je nach Fluorophor und Filter zwischen 1 ms und 30 ms (Abbildung 3I). Wenn eine Monoplex-IHC diese Zahlen überschreitet oder das Färbemuster nicht so klar wie erwartet ist, sollte die Antikörperlösung angepasst oder ausgetauscht werden. In dem hier gezeigten Beispiel haben wir uns entschieden, die Konzentration von FOXP3 zu erhöhen (Abbildung 3C und Abbildung 3I), um die Intensität mehr im Bereich der anderen Marker zu halten. Die Autofluoreszenz kann auch in anderen Geweben stärker sein als in Mandelkontrollgewebe. In unserer Einstellung liegt die Belichtungszeit für den Sample-AF-Filter zwischen 25 ms und 50 ms (Abbildung 3H,I).

Abbildung 3: Einstellung der Expositionszeiten an Monoplex-IHC und ungefärbten Kontrollproben. (A) CD20 - Opal 480 Signal im Mandelkontrollgewebe. (B) CD3 - Opal 520 Signal im Mandelkontrollgewebe. (C) FOXP3 - Opal 570-Signal im Mandelkontrollgewebe. (D) CD56 - Opal 620 Signal im Mandelkontrollgewebe (E) CD8 - Opal 690 Signal im Mandelkontrollgewebe. (F) Tumormarker - Opal 780-Signal im Mandelkontrollgewebe. (G) Das DAPI-Signal im Mandelkontrollgewebe ist oft schwächer als der interessierende Gewebetyp. (H) Autofluoreszenz - Proben-AF-Signal im Tumorkontrollgewebe. (I) Screenshot der Belichtungszeiten vor dem Einstellen mit 10% und der Überprüfung auf Multiplex-IHC-gefärbten Objektträgern. Maßstabsleisten = 100 μm. Abkürzungen: AF = Autofluoreszenz; IHC = Immunhistochemie; DAPI = 4'6-Diamidino-2-phenylindol. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Nachdem die Multiplex-IHC durchgeführt wurde, werden die Belichtungszeiten über die Monoplex-IHC-Einstellungen angepasst, indem Sie einige Multiplex-IHC-Dias überprüfen und die Option "Automatisch belichten" auswählen. Bei dieser Art des Scannens gibt es keine Option zum Sättigungsschutz und daher ist es äußerst wichtig, die Belichtung nicht zu hoch einzustellen, um eine Überbelichtung zu vermeiden. Eine Überbelichtung erschwert die spektrale Entmischung der Fluorophore. Wir legen oft keine Belichtungszeiten fest, die über die Belichtungszeiten hinausgehen, die auf der Monoplex-IHC basieren, und wir verkürzen die Belichtungszeiten nur für Marker, die in der Multiplex-IHC stärker sind (Abbildung 3G und Abbildung 4A). Durch die automatische Belichtung an verschiedenen Stellen auf wenigen Dias kann beobachtet werden, dass die Belichtungszeiten einiger weniger Filter noch zu hoch sind. Diese müssen auf die niedrigste Zahl eingestellt werden, die bei Verwendung der Einstellung für die automatische Belichtung beobachtet wird, und weitere 10 % des Wertes abziehen, um eine Überbelichtung an anderen unsichtbaren Stellen zu verhindern (Abbildung 4A). Mit dieser Methode können die Belichtungszeiten für bestimmte Filter kürzer sein als bei Monoplex-IHC. Bei einem erfolgreichen Multiplex-IHC-Experiment sollten jedoch alle Marker zumindest auf dem Kontrollobjektträger beobachtbar sein (Abbildung 4B-H, Ergänzende Datei 6: Ergänzende Abbildung S1 und Ergänzende Abbildung S2). Beachten Sie, dass bestimmte Marker möglicherweise nicht in jeder Probe vorhanden sind. Durch die Beifügung eines Kontrollobjektträgers, der mindestens einen Mandelschnitt enthält, kann eine erfolgreiche Färbung aller Marker der Standardplatten und der Signalstärke überprüft werden.
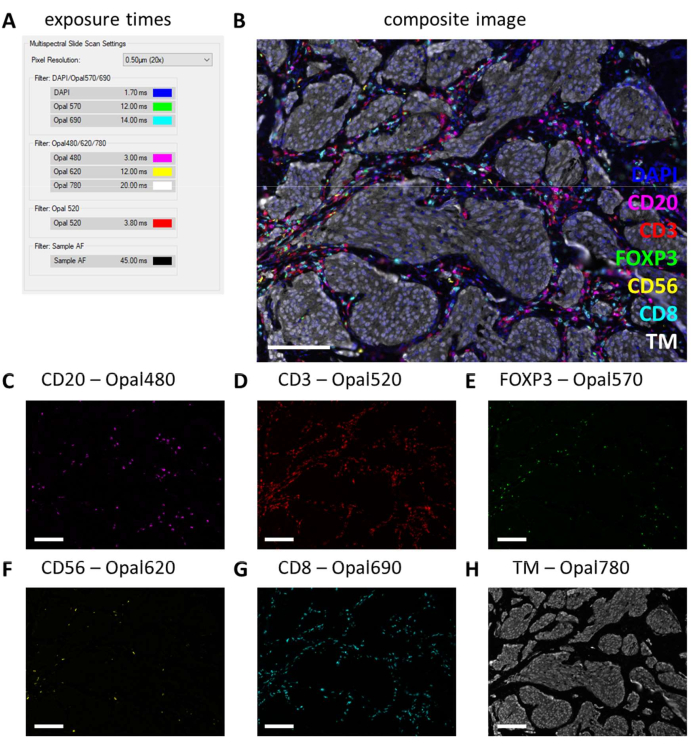
Abbildung 4: Beispiel für einen erfolgreich gefärbten Schnitt mit dem Lymphozytenpanel in einer Melanomtumorprobe. (A) Expositionszeiten, die zur Aufzeichnung dieser Multiplex-IHC-Probe verwendet wurden. (B) Zusammengesetztes Bild eines Multiplex-IHC-Lymphozyten-Panels im Tumorgewebe. (C) CD20 - Opal 480 Signal in Magenta. (D) CD3 - Opal 520 Signal in rot. (E) FOXP3 - Opal 570 Signal in grün. (F) CD56 - Opal 620 Signal in gelb. (G) CD8 - Opal 690 Signal in Cyan. (H) TM - Opal 780 in weiß. Maßstabsleisten = 100 μm. Abkürzung: TM = Tumormarker; IHC = Immunhistochemie. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Multiplex-IHC-Dias werden vom digitalen Imager vollständig gescannt. Kacheln für die nachfolgende Analyse werden in der Folienbetrachtung ausgewählt. Wenn jedoch spezifischere Regionen analysiert werden müssen, wie z. B. Tumor versus IM, können diese Regionen von Interesse (ROIs) mit QuPath gezeichnet werden. Nachdem die Stapelverarbeitung der Kacheln, die in der Folienanzeige ausgewählt sind, abgeschlossen ist, werden die Komponentendateien wieder zusammengeführt (Abbildung 5A und ergänzende Datei 7). Mit Hilfe des Tumormarkerkanals (Abbildung 5B) und des Zauberstab-Tools in QuPath kann der Tumorumriss nachgezeichnet werden, um den "Tumor-ROI" zu bilden (Abbildung 5C). Als nächstes kann der Tumor-ROI um einen bestimmten Abstand, in diesem Fall 500 μm, erweitert werden, um einen "invasiven Rand-ROI" zu erzeugen (Abbildung 5D). Jeder unerwünschte Hintergrund (Nicht-Gewebe) wird mit dem Zauberstab aus diesem ROI entfernt, indem das Autofluoreszenzsignal betrachtet wird (Abbildung 5E). Sowohl der Tumor-ROI als auch der IM-ROI werden zur weiteren Verarbeitung als GeoJSON-Datei gespeichert (Abbildung 5F).
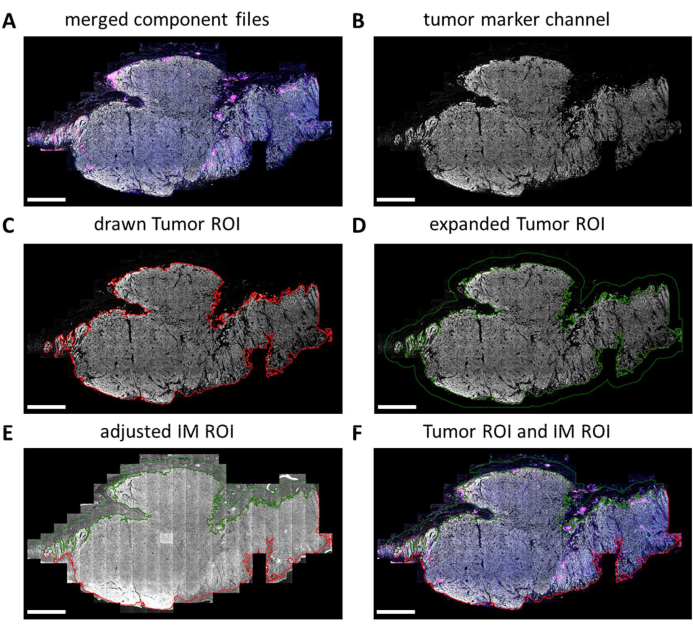
Abbildung 5: Ziehprozess des Tumor-ROI und des invasiven Rand-ROI in QuPath. (A) Zusammengeführte Komponentendateien. (B) Graustufenbild, das nur den Tumormarkerkanal zeigt. (C) Der Tumor-ROI wird um das Tumormarkersignal herum gezeichnet. (D) Ein neuer ROI wird erzielt, indem der Tumor-ROI um 100-500 μm erweitert wird, um den IM-ROI zu bilden. (E) Der IM-ROI wird so angepasst, dass nur Stromagewebe berücksichtigt wird, indem Hintergrundgewebe (negatives Signal) und andere große Gewebestrukturen wie Fett, Blutgefäße und Haarfollikel ausgeschlossen werden. (F) Der resultierende Tumor-ROI und IM-ROI werden gespeichert und in GeoJSON-Dateien exportiert, um die Regionen weiter zu verarbeiten. Der Tumor-ROI wird mit einer roten Umrandung und der IM-ROI mit einer grünen Kontur angezeigt. Maßstabsleisten = 2 mm. Abkürzungen: ROI = Region of Interest; IM = invasiver Rand; GeoJSON = Geografische JavaScript-Objektnotation. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
ImmuNet-Netzwerke können zum Nachweis von Immunzellen genutzt werden. Für das Lymphozyten-Panel kann das experimentelle Kompositbild (Abbildung 6A) visuell mit den von der Software erkannten Immunzellen verglichen werden (Abbildung 6B). Ähnliche visuelle Vergleiche können für das myeloische Panel (Supplemental File 6: Supplemental Abbildung S4) und das dendritische Zellpanel (Supplemental File 6: Supplemental Abbildung S5) durchgeführt werden.
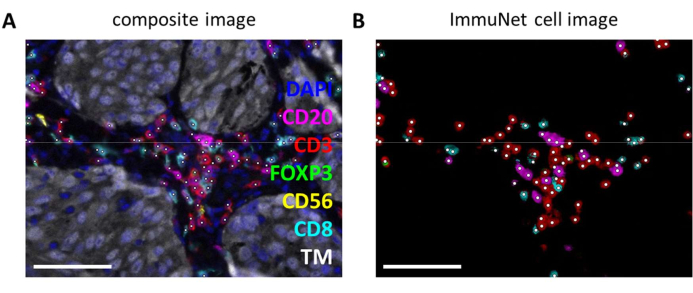
Abbildung 6: Lymphozyten, die von ImmuNet erkannt wurden. (A) Zusammengesetztes Bild von Abbildung 4B , das von ImmuNet erkannte Zellen mit weißen Punkten zeigt. (B) Zellen, die von ImmuNet erkannt und anschließend detektiert wurden. Maßstabsleisten = 50 μm. Abkürzung: TM = Tumormarker. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Immunzellen, die von ImmuNet erkannt und in .csv Format gespeichert werden, können zur weiteren Analyse in jede Programmiersprache importiert werden. Wir führten eine räumliche Visualisierung und ein Gating in R durch (Supplemental File 8). Die detektierten Zellen können dann räumlich visualisiert werden (Abbildung 7A, Ergänzungsdatei 6: Ergänzende Abbildung S6 und Ergänzende Abbildung S7). Ein Gating auf Pseudomarker-Expression kann durchgeführt werden, um die einzelnen Immunzellen zu phänotypisieren (Abbildung 7B).
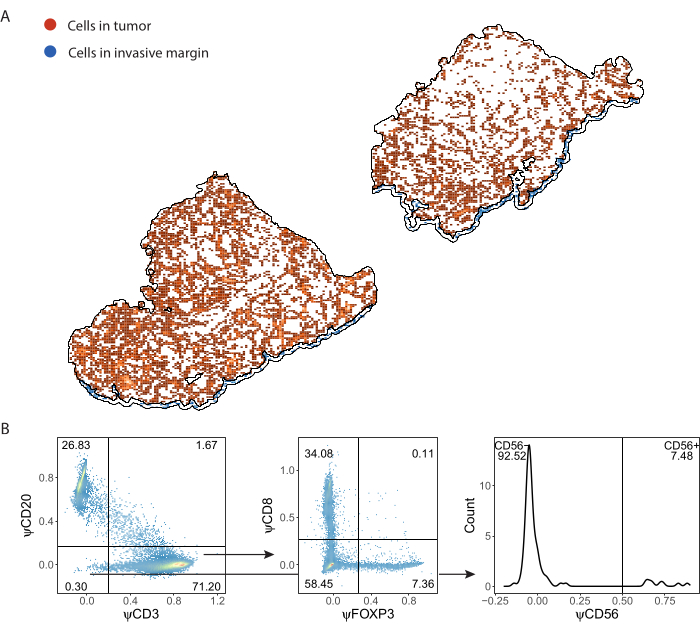
Abbildung 7: Gating-Strategie des Lymphozyten-Panels. (A) Immunzellen, die in Tumor- und invasiven Randregionen von Interesse nachgewiesen wurden, die mit QuPath abgegrenzt wurden. (B) Gating aller mit ImmuNet nachgewiesenen Zellen aus Teil A. Lymphozyten werden zunächst auf CD20+ B-Zellen und CD3+ T-Zellen fixiert. CD3+ T-Zellen sind weiterhin für die CD8- und FOXP3-Expression gesteuert. Die CD20-CD3-Population ist für CD56+ natürliche Killerzellen gesperrt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Wenn Phänotypen der vorhergesagten Zellen mit Gating bestimmt werden, können Zelldichten verschiedener Phänotypen innerhalb unterschiedlicher ROIs berechnet werden. Dies wird berechnet, indem die Gesamtzahl der Zellen pro Phänotyp durch die Oberfläche des ROI dividiert wird (Tabelle 1, Abbildung 8 und ergänzende Datei 8). Dabei werden B-Zellen als CD3-CD20+, T-Helferzellen als CD3+CD20-CD8-FoxP3-, regulatorische T-Zellen als CD3+CD20-CD8-FoxP3+, zytotoxische T-Zellen als CD3+CD20-CD8+FoxP3- und NK-Zellen als CD3-CD20-CD56+ definiert.
| Phänotyp | Dichte im Tumor (Zellen/mm 2) | Dichte in IM (Zellen/mm2) |
| B-Zelle | 185.74 | 145.62 |
| T-Zelle des Helfers | 301.46 | 157.51 |
| Regulatorische T-Zellen | 38.53 | 19.53 |
| Zytotoxische T-Zellen | 185.35 | 83.21 |
| NK-Zelle | 0.18 | 0 |
Tabelle 1: Dichten von Phänotypen in ROIs. Dichten von Zellen verschiedener Phänotypen, die in einer einzigen Melanomprobe gefunden wurden, die mit dem Lymphozytenpanel gefärbt wurde. Die Dichten werden separat in Tumor- und IM-ROIs berechnet. Abkürzungen: IM = invasiver Rand; ROI = Region von Interesse.
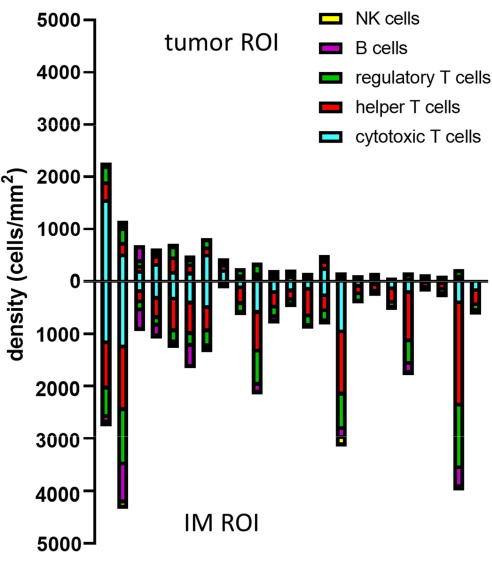
Abbildung 8: Beispiel für eine Datenanalyse für mehrere Proben. Dichteanalyse verschiedener Lymphozytenphänotypen im Tumor und IM von 23 primären Melanomtumoren. Abkürzungen: IM = invasiver Rand; ROI = Region von Interesse. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Um mehr in die räumlichen Informationen dieser Immunzellen einzutauchen, ist es auch möglich, Abstände zwischen identifizierten Phänotypen oder Prozentsätze von Phänotypen der nächsten Nachbarn in einer Probe zu bestimmen (Abbildung 9).
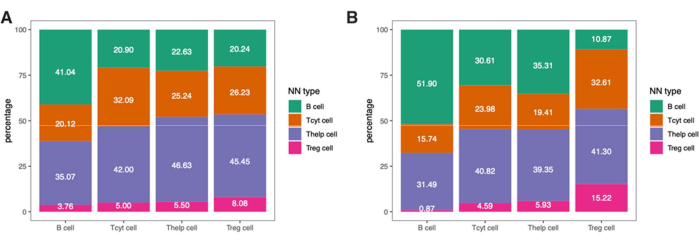
Abbildung 9: Beispiel für eine Nearest-Neighbor-Analyse für eine einzelne Stichprobe. Prozentsatz der Phänotypen des nächsten Nachbarn für verschiedene Zelltypen in (A) Tumor- und (B) IM-ROIs, die in einer einzigen Melanomprobe gefunden wurden, die mit dem Lymphozytenpanel gefärbt wurde. Abkürzungen: IM = invasiver Rand; ROI = Region von Interesse. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergänzende Datei 1: Multiplex IHC mit einer Zusammenfassung der Protokollspezifikationen. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 2: Autostainer-Protokoll für Monoplex. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 3: Autostainer-Protokoll zur Autofluoreszenzkompensation. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 4: Autostainer-Protokoll für die Multiplex-Immunhistochemie. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 5: Vorlage .csv Datei. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 6: Myeloische und dendritische Zell-Panels in einer Melanom-Gewebeprobe; Markierung von Objektträgern im Falle eines Scan-Versagens; myeloische und dendritische Zellen, die von ImmuNet erkannt wurden; Gating-Strategien von myeloischen und dendritischen Zell-Panels. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 7: QuPath-Stitch-Skript. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 8: Skript zur Datenanalyse. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Die räumliche Analyse der TME ist eine gefragte Technik, um mehr über das Kompartiment der Immunzellen zu erfahren und neue prognostische und prädiktive Biomarker zu entdecken, insbesondere im Bereich der Immunonkologie16. Zu diesem Zweck werden viele verschiedene Techniken entwickelt, die den Nachweis von Proteinen, mRNA-Transkripten oder einer Kombination aus beidem beinhalten, mit Schätzungen von bis zu 100-1.000 Zielen. Ein höheres Multiplexing geht jedoch auf Kosten von weniger Experimenten mit hohem Durchsatz, höheren experimentellen Kosten und technischen Herausforderungen, und oft kann nur ein kleiner Teil der TME analysiert werden. Multiplex IHC mit der TSA-basierten Methode, die wir hier beschreiben, detektiert sechs verschiedene Marker + DAPI gleichzeitig, ist relativ kostengünstig durchzuführen und ganze Gewebeschnitte werden in weniger als 20 Minuten abgebildet und können vollständig analysiert werden. Diese Technik ist durch die Automatisierung des Färbeverfahrens weniger komplex geworden. Verbesserungen am Multispektralmikroskop, zu denen auch das Hinzufügen von zwei zusätzlichen Filtern gehört, haben die spektrale Entmischung und die Scanzeiten enorm verbessert. Es ist möglich, bis zu acht verschiedene Marker + DAPI gleichzeitig zu erkennen. Durch die Erweiterung des Multiplexings um mehr Marker verschwinden die oben genannten Vorteile jedoch, da die spektrale Entmischung schwieriger wird und die Scanzeiten für ganze Objektträger erheblich zunehmen. Es werden Anstrengungen unternommen, um die Multiplex-IHC zwischen verschiedenen Institutionen zu standardisieren, um die Implementierung im diagnostischen Umfeld zu erleichtern. Für diese Standardisierung von Multiplex-IHC empfehlen wir den Anwendern, sich an das leichter zugängliche Protokoll mit sechs verschiedenen Markern + DAPI zu halten. Nichtsdestotrotz ist noch einiges an technischem Know-how notwendig und die nachgelagerte Analyse kann eine Herausforderung darstellen, für die wir Methoden entwickelt haben, die in diesem Protokoll beschrieben werden.
Die Standardisierung beginnt mit der Entwicklung von Multiplex-IHC-Panels. Die Bedeutung der Auswahl von Primärantikörpern zum Nachweis bestimmter Proteinziele wurde bereits vor17 Jahren betont. Unsere Multiplex-IHC-Panels werden meist mit primären Antikörperklonen entwickelt, die auch in unserer Diagnostikabteilung für IHC verwendet und validiert werden. Im Fall des dendritischen Zellmultiplex-IHC-Panels wurden die meisten Antikörper jedoch nicht im diagnostischen Setting verwendet (van der Hoorn et al., Manuskript in der Einreichung). Um die Spezifität zu gewährleisten und Chargenunterschiede zu minimieren, haben wir uns für die Verwendung von monoklonalen Antikörpern anstelle von polyklonalen Antikörpern entschieden und die meisten Antikörper auch mit transfizierten Zelllinien und Primärzellen validiert. Im Laufe der Jahre wurden verschiedene Versionen von Multiplex-IHC-Paneelen in zahlreichen Studien mit dem Vectra 3-System 18,21,23,24,25,26,27,28,29,30,31,32 verwendet . Um diese Multiplex-IHC-Panels optimal auf dem PhenoImager HT-System zu implementieren, mussten einige Anpassungen in den Kombinationen von primären Antikörpern und Fluorophoren vorgenommen werden. Um von einer besseren spektralen Entmischung und schnelleren Scanzeiten ganzer Gewebeschnitte zu profitieren, ist die Implementierung der neuesten Opal480- und Opal780-Fluorophore und die Vermeidung der Verwendung von Opal540- und Opal650-Fluorophoren in siebenfarbigen Multiplex-IHC-Panels erforderlich. Die Scanzeiten sind je nach Größe des Gewebeschnitts ~3-10 Mal schneller. Die Anpassung des Multiplex-IHC-Panels war recht einfach zu bewerkstelligen, aber einige Überlegungen müssen im Auge behalten werden. Das Fluoreszenzspektrum von Opal480 überlappt sich stark mit dem Autofluoreszenzspektrum und stört daher die spektrale Entmischung von Erythrozyten und anderen autofluoreszierenden Strukturen. Die Verwendung einer erhöhten Konzentration des primären Antikörpers in Kombination mit Opal480 löste dieses Problem in den meisten Fällen. Die Implementierung des proprietären Sample AF-Filters auf dem PhenoImager HT erleichtert die Entmischung von Opal480 und Autofluoreszenz. Es ist jedoch am besten, einen Primärantikörper zu verwenden, der bei Verwendung mit Opal480 ein klares Signal liefert, so dass sein Signal höher ist als die Autofluoreszenz.
Auch wenn diese Multiplex-IHC-Panels etabliert sind, muss eine Variation von Charge zu Charge berücksichtigt werden. Durch die Durchführung von Monoplex-IHC-Kontrollen vor Beginn des vollständigen Multiplex-IHC-Experiments beobachteten wir manchmal, dass primäre Antikörper von Experiment zu Experiment entweder stärker oder schwächer abschnitten. Die Gründe dafür können Pipettierfehler, suboptimale Lagerbedingungen für Reagenzien und die Haltbarkeit sein. Wir haben dieses Problem gelöst, indem wir die primäre Antikörperlösung auf der Grundlage unserer Erfahrungen angepasst haben. Auch wenn keine der oben genannten Anpassungen vorgenommen werden musste, ist es bei jedem Multiplex-IHC-Batch-Experiment wichtig, die Belichtungszeiten auf Basis von Monoplex-IHC-gefärbten Kontrollobjektträgern einzustellen.
Da sich unsere Forschung ursprünglich auf verschiedene Arten von Karzinomen und Melanomen konzentrierte, mussten Multiplex-IHC-Panels mit minimalen Anpassungen zwischen Tumorarten austauschbar sein. Daher haben wir immer mehrere (Tumor-)Gewebetypen in den Optimierungsprozess einbezogen und beobachtet, dass die Verdünnungen für primäre Antikörper für Immunzellmarker zwischen verschiedenen Tumorarten ähnlich gehalten werden können. Für die Detektion von Tumorgewebe zwischen Karzinomen und Melanomen werden jedoch unterschiedliche Tumormarker benötigt. Dementsprechend wurde der Tumormarker immer so optimiert, dass er am Ende eines jeden Multiplex-IHC-Panels arbeitet und wird derzeit immer in Verbindung mit Opal780 verwendet, das zufälligerweise auch bei einem Multiplex-IHC-Färbeverfahren am letzten Fluorophor sein muss. Durch die konsequente Verwendung des Tumormarkers am Ende des Multiplex-IHC können diese Multiplex-IHC-Panels leicht gegen andere Tumorarten wie das Glioblastom (d.h. GFAP) und das Hodgkin-Lymphom (d.h. CD30) ausgetauscht werden. Für das Angiosarkom verwendeten wir dieses Lymphozyten-Multiplex-IHC-Panel mit dem Erythroblast transformation-specific-related Gen (ERG) als Tumormarker mit nur zwei Optimierungsexperimenten25. Die Optimierung umfasste die Titration des ERG-Primärantikörpers und das Testen des Multiplex-IHC-Panels mit ERG am Ende.
Weitere Anpassungen an diesen Multiplex-IHC-Panels können auch vorgenommen werden, indem ein bestimmter Immunzellmarker gegen einen anderen Immun- oder Funktionsmarker ausgetauscht wird. Jede Änderung erfordert eine Optimierung. Das Protokoll zur Optimierung konnte wie zuvor beschrieben befolgt werden17. Bestimmte Änderungen an den vorgeschlagenen Multiplex-IHC-Panels werden die von uns erstellten ImmuNet-Algorithmen beeinträchtigen. Es müssen ausreichend Daten generiert werden und es muss Zeit aufgewendet werden, um diese Änderungen in den Algorithmus zu implementieren (mindestens 750 Annotationen für jeden neuen Marker und/oder Zellphänotyp und 150 Annotationen für die Validierung zuvor trainierter Marker). Die hier vorgestellten Panels enthalten keine funktionellen Marker, obwohl die Implementierung von Immun-Checkpoint-Markern wie PD-1 und PD-L1 in Multiplex-IHC-Panels in unserem Labor durchgeführt wird. Die Analyse von Markern, die in negativen und positiven Signalen weniger binär sind, hat sich jedoch als schwieriger erwiesen und ist ein Bereich aktiver Forschung in unserer Gruppe.
Die Anzahl der Marker, die gleichzeitig mit Multiplex-IHC beurteilt werden können, ist im Vergleich zu anderen neuartigen Techniken begrenzt. Dies kann zwar umgangen werden, indem verschiedene Paneele auf aufeinanderfolgenden Schichten eines FFPE-Blocks analysiert werden, aber es wird schwierig sein, diese Schichten räumlich zu vergleichen. Ausrichtung und gefaltete Artefakte sind nach der Vorbereitung der Folie wahrscheinlich nicht mehr dieselben. Nichtsdestotrotz ist die Multiplex-IHC leicht zugänglich, was sie zu einem attraktiven Werkzeug für mehr Institutionen und Forscher macht und daher besser für die zukünftige Implementierung in einem diagnostischen Umfeld geeignet ist. Mit der Standardisierung von Multiplex-IHC-Immunzellpanels für mehrere Tumorarten und nachgelagerten Analysepipelines konnten mehr Erkenntnisse über die Unterschiede in der TME zwischen Patienten und Tumorarten gewonnen werden. Dies kann zum Beispiel zu mehr Erkenntnissen über die Rolle der TME bei der Reaktion auf bestimmte Behandlungen gegen Tumore führen. Dies kann sogar zu neuen Biomarkern führen, um Faktoren wie das Ansprechen auf die Behandlung und das erwartete Überleben vorherzusagen. Insgesamt kann dies dazu führen, dass die Multiplex-IHC zu einem klinischen Instrument wird, das die klinische Entscheidungsfindung in einem personalisierten Medizinansatz unterstützt. Zugegebenermaßen sollten wahrscheinlich mehr Schritte des Analyseverfahrens automatisiert und standardisiert werden, damit es für den Einsatz in einer täglichen diagnostischen Umgebung machbar ist, so dass es sich bisher hauptsächlich um eine futuristische Perspektive handelt.
Die Analyse mehrerer Marker auf einem einzigen Objektträger kann trotz ihrer technischen Herausforderungen ein sehr leistungsfähiges Werkzeug sein. Mit standardisierten experimentellen Protokollen und einer robusten Analysemethode, wie wir hier mit ImmuNet beschrieben haben, ist die Quantifizierung mehrerer Marker informativer als die klassische IHC, während die Multiplex-IHC im Vergleich zu neuartigen experimentellen Methoden mit höherem Plex einen relativ hohen Durchsatz aufweist.
Offenlegungen
Die Autoren haben keine Interessenkonflikte offenzulegen.
Danksagungen
Der PhenoImager HT wurde mit Mitteln des Radboud University Medical Center und des Radboud Technology Center for Microscopy erworben. CF wird finanziell unterstützt durch ein Stipendium der Dutch Cancer Society (10673) und ein ERC Adv Grant ARTimmune (834618). JT wird finanziell unterstützt durch einen NWO Vidi Zuschuss (VI.Vidi.192.084). Die Autoren danken Eric van Dinther und Ankur Ankan für ihre Unterstützung bei der Erstellung von Workflows zum Speichern von Multiplex-IHC-Daten, und Bengt Phung wird für Anweisungen zur Implementierung von Multiplex-IHC-Daten in QuPath für die ROI-Zeichnung gedankt.
Materialien
| Name | Company | Catalog Number | Comments |
| anti-CD14 | Cell Marque | 114R-16 | section 3, clone EPR3653 |
| anti-CD163 | Cell Marque | 163M-15 | section 3, clone MRQ-26 |
| anti-CD19 | Abcam | ab134114 | section 3, clone EPR5906 |
| anti-CD1c (BDCA1) | Thermo Scientific | TA505411 | section 3, clone OTI2F4 |
| anti-CD20 | Thermo Scientific | MS-340-S | section 3, clone L26 |
| anti-CD3 | Thermo Scientific | RM-9107 | section 3, clone sp7 |
| anti-CD303/BDCA2 | Dendritics via Enzo Lifesciences/Axxora | DDX0043 | section 3, clone 124B3.13 |
| anti-CD56 | Cell Marque | 156R-94 | section 3, clone MRQ-42 |
| anti-CD66b | BD Biosciences | 555723 | section 3, clone G10F5 |
| anti-CD68 | Dako Agilent | M087601 | section 3, clone PG-M1 |
| anti-CD8 | Dako Agilent | M7103 | section 3, clone C8/144B |
| anti-Foxp3 | Thermo Scientific | 14-4777 | section 3, clone 236A/E7 |
| anti-Gp100 | Dako Agilent | M063401 | section 3, clone HMB45 |
| anti-HLA-DR, DP, DQ | Santa Cruz | sc-53302 | section 3, clone CR3/43 |
| anti-MART-1 | Thermo Scientific | MS-799 | section 3, clone A103 |
| anti-pan cytokeratin | Abcam | ab86734 | section 3, clone AE1/AE3 + 5D3 |
| anti-SOX10 | Sigma Aldrich | 383R | section 3, clone EP268 |
| anti-Tyrosinase | Sanbio | MONX10591 | section 3, clone T311 |
| anti-XCR1 | Cell Signaling Technologies via Bioké | 44665S | section 3, clone D2F8T |
| antibody diluent | Akoya BioSciences | SKU ARD1001EA | section 3, from Opal 7-Color Automation IHC Kit 50 slide (can optionally also be replaced by TBST with 10% BSA) |
| Bond Aspirating Probe | Leica Biosciences | S21.0605 | section 3 |
| Bond Aspirating Probe Cleaning | Leica Biosciences | CS9100 | section 3 |
| Bond Dewax Solution | Leica Biosciences | AR9222 | section 3 |
| Bond Objectglas label + print lint | Leica Biosciences | S21.4564.A | section 3 |
| Bond Research Detection System 2 | Leica Biosciences | DS9777 | section 3 |
| Bond RX autostainer | Leica Biosciences | - | section 3, automated platform |
| Bond TM Epitope Retrieval 1 - 1 L | Leica Biosciences | AR9961 | section 3 |
| Bond TM Epitope Retrieval 2 - 1 L | Leica Biosciences | AR9640 | section 3 |
| Bond TM Wash Solution 10x - 1 L | Leica Biosciences | AR9590 | section 3 |
| BOND Universal Covertile | Leica Biosciences | S21.4611 | section 3 |
| Bond(TM) Titration Kit | Leica Biosciences | OPT9049 | section 3 |
| Coverslip 24 x 32 mm #1 (0.13-0.16 mm) | Fisher Scientific | 15717592 | section 2 |
| coverslip 24 x 50 mm | VWR | 631-0146 | section 2 |
| DAPI Fluoromount-G | VWR | 0100-20 | section 3, whenever monoplex slides need to be quickly checked, not for official analysis, then DAPI is stained seperately for better results |
| Eosine | section 2, home made | ||
| Ethanol 99.5% | VWR | 4099.9005 | section 2 |
| Fluoromount-G | VWR | 0100-01 | section 3 |
| haematoxyline | - | section 2, home made | |
| ImmuNet | - | immune cell detection and phenotyping pipeline | |
| inForm software 2.4.10 | Akoya BioSciences | - | section 4 & 6 |
| OPAL 480 reagent pack | Akoya BioSciences | FP1500001KT | section 3 |
| OPAL 520 reagent pack | Akoya BioSciences | FP1487001KT | section 3 |
| OPAL 570 reagent pack | Akoya BioSciences | FP1488001KT | section 3 |
| OPAL 620 reagent pack | Akoya BioSciences | FP1495001KT | section 3 |
| OPAL 690 reagent pack | Akoya BioSciences | FP1497001KT | section 3 |
| OPAL 780 reagent pack | Akoya BioSciences | FP1501001KT | section 3 |
| Opal 7-Color Automation IHC Kit 50 slide | Akoya BioSciences | NEL821001KT | section 3 |
| PhenoChart 1.1.0 | Akoya BioSciences | - | section 5 |
| PhenoImagerHT | Akoya BioSciences | CLS143455 | section 4, digital pathology imager with slide viewer and imaging software (formerly known as Vectra Polaris) |
| Quick-D mounting medium | Klinipath | 7280 | section 2 |
| QuPath 0.3.2 | whole slide image analysis software platform | ||
| R 4.1.1 | |||
| Slide boxes | VWR | 631-0737 | section 1 |
| SuperFrost Plus | Thermo Scientific through VWR | 631-9483 | section 1 |
| Vectra Polaris software 1.0.13 | Akoya BioSciences | - | section 4 |
| Xylene | VWR | 4055-9005 | section 2 |
Referenzen
- Dunn, G. P., Bruce, A. T., Ikeda, H., Old, L. J., Schreiber, R. D. Cancer immunoediting: from immunosurveillance to tumor escape. Nature Immunology. 3 (11), 991-998 (2002).
- van der Woude, L. L., Gorris, M. A. J., Halilovic, A., Figdor, C. G., de Vries, I. J. M. Migrating into the tumor: a roadmap for T cells. Trends in Cancer. 3 (11), 797-808 (2017).
- Fridman, W. H., Pages, F., Sautes-Fridman, C., Galon, J. The immune contexture in human tumours: impact on clinical outcome. Nature Reviews. Cancer. 12 (4), 298-306 (2012).
- Fridman, W. H., et al. The immune microenvironment of human tumors: general significance and clinical impact. Cancer Microenvironment. 6 (2), 117-122 (2013).
- Pages, F., et al. International validation of the consensus Immunoscore for the classification of colon cancer: a prognostic and accuracy study. Lancet. 391 (10135), 2128-2139 (2018).
- Angell, H. K., Bruni, D., Barrett, J. C., Herbst, R., Galon, J. The Immunoscore: colon cancer and beyond. Clinical Cancer Research. 26 (2), 332-339 (2020).
- Angell, H., Galon, J. From the immune contexture to the Immunoscore: the role of prognostic and predictive immune markers in cancer. Current Opinion in Immunology. 25 (2), 261-267 (2013).
- Galon, J., et al. Cancer classification using the Immunoscore: a worldwide task force. Journal of Translational Medicine. 10, 205 (2012).
- Galon, J., et al. World-wide Immunoscore Task Force: meeting report from the 34;Melanoma Bridge", Napoli, November 30th-December 3rd, 2016. Journal of Translational Medicine. 15 (1), 212 (2017).
- Tumeh, P. C., et al. PD-1 blockade induces responses by inhibiting adaptive immune resistance. Nature. 515 (7528), 568-571 (2014).
- Creemers, J. H. A., et al. A tipping point in cancer-immune dynamics leads to divergent immunotherapy responses and hampers biomarker discovery. Journal for Immunotherapy of Cancer. 9 (5), e002032 (2021).
- Blank, C. U., Haanen, J. B., Ribas, A., Schumacher, T. N. CANCER IMMUNOLOGY. The "cancer immunogram". Science. 352 (6286), 658-660 (2016).
- Teruya-Feldstein, J. The immunohistochemistry laboratory looking at molecules and preparing for tomorrow. Archives of Pathology & Laboratory Medicine. 134 (11), 1659-1665 (2010).
- Ramos-Vara, J. A., Miller, M. A., et al. When tissue antigens and antibodies get along: revisiting the technical aspects of immunohistochemistry--the red, brown, and blue technique. Veterinary Pathology. 51 (1), 42-87 (2014).
- Hegde, P. S., Karanikas, V., Evers, S. The where, the when, and the how of immune monitoring for cancer immunotherapies in the era of checkpoint inhibition. Clinical Cancer Research. 22 (8), 1865-1874 (2016).
- Parra, E. Novel platforms of multiplexed immunofluorescence for study of paraffin tumor tissues. Journal of Cancer Treatment and Diagnosis. 2 (1), 43-53 (2018).
- Gorris, M. A. J., et al. Eight-color multiplex immunohistochemistry for simultaneous detection of multiple immune checkpoint molecules within the tumor microenvironment. Journal of Immunology. 200 (1), 347-354 (2018).
- Roelofsen, T., et al. Spontaneous regression of ovarian carcinoma after septic peritonitis; a unique case report. Frontiers in Oncology. 8, 562 (2018).
- van den Brand, D., et al. Peptide-mediated delivery of therapeutic mRNA in ovarian cancer. European Journal of Pharmaceutics and Biopharmaceutics. 141, 180-190 (2019).
- van den Brand, D., et al. EpCAM-binding DARPins for targeted photodynamic therapy of ovarian cancer. Cancers. 12 (7), 1762 (2020).
- Di Blasio, S., et al. The tumour microenvironment shapes dendritic cell plasticity in a human organotypic melanoma culture. Nature Communications. 11 (1), 2749 (2020).
- van Beek, J. J. P., et al. Human pDCs are superior to cDC2s in attracting cytolytic lymphocytes in melanoma patients receiving DC vaccination. Cell Reports. 30 (4), 1027-1038 (2020).
- Rodriguez-Rosales, Y. A., et al. Immunomodulatory aged neutrophils are augmented in blood and skin of psoriasis patients. Journal of Allergy and Clinical Immunology. 148 (4), 1030-1040 (2021).
- Hoeijmakers, Y. M., et al. Immune cell composition in the endometrium of patients with a complete molar pregnancy: Effects on outcome. Gynecologic Oncology. 160 (2), 450-456 (2021).
- van Ravensteijn, S. G., et al. Immunological and genomic analysis reveals clinically relevant distinctions between angiosarcoma subgroups. Cancers. 14 (23), 5938 (2022).
- van der Woude, L. L., et al. Tumor microenvironment shows an immunological abscopal effect in patients with NSCLC treated with pembrolizumab-radiotherapy combination. Journal for Immunotherapy of Cancer. 10 (10), e005248 (2022).
- Graham Martinez, C., et al. The immune microenvironment landscape shows treatment-specific differences in rectal cancer patients. Frontiers in Immunology. 13, 1011498 (2022).
- Cortenbach, K. R. G., et al. Topography of immune cell infiltration in different stages of coronary atherosclerosis revealed by multiplex immunohistochemistry. International Journal of Cardiology. Heart & Vasculature. 44, 101111 (2023).
- van Wilpe, S., et al. Homologous recombination repair deficient prostate cancer represents an immunologically distinct subtype. Oncoimmunology. 11 (1), 2094133 (2022).
- Gorris, M. A. J., et al. Paired primary and metastatic lesions of patients with ipilimumab-treated melanoma: high variation in lymphocyte infiltration and HLA-ABC expression whereas tumor mutational load is similar and correlates with clinical outcome. Journal for Immunotherapy of Cancer. 10 (5), e004329 (2022).
- van Wilpe, S., et al. Intratumoral T cell depletion following neoadjuvant chemotherapy in patients with muscle-invasive bladder cancer is associated with poor clinical outcome. Cancer Immunology, Immunotherapy. 72 (1), 137-149 (2023).
- Sultan, S., Gorris, M. A. J., Buytenhuijs, F., Lvan de Woude, L. A Segmentation-free machine learning architecture for immune land-scape phenotyping in solid tumors by multichannel imaging. bioRxiv. , (2021).
- Parra, E. R., et al. Immuno-profiling and cellular spatial analysis using five immune oncology multiplex immunofluorescence panels for paraffin tumor tissue. Scientific Reports. 11 (1), 8511 (2021).
- Parra, E. R., et al. Validation of multiplex immunofluorescence panels using multispectral microscopy for immune-profiling of formalin-fixed and paraffin-embedded human tumor tissues. Scientific Reports. 7 (1), 13380 (2017).
- Sun, Z., Nyberg, R., Wu, Y., Bernard, B., Redmond, W. L. Developing an enhanced 7-color multiplex IHC protocol to dissect immune infiltration in human cancers. PLoS One. 16 (2), e0247238 (2021).
- LeicaBiosystems. BOND RX Fully Automated Research Stainer Protocols. , (2022).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten