Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Abschätzung der strukturellen Empfindlichkeit von intrinsisch ungeordneten Regionen als Reaktion auf hyperosmotischen Stress in lebenden Zellen mit FRET
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Intrinsisch ungeordnete Regionen (IDRs) sind flexible Proteindomänen, die ihre Konformation als Reaktion auf Umweltveränderungen verändern. Der Ensemble-Fluoreszenz-Resonanz-Energietransfer (FRET) kann Proteindimensionen unter verschiedenen Bedingungen abschätzen. Wir präsentieren einen FRET-Ansatz zur Bewertung der strukturellen IDR-Sensitivität in lebenden Saccharomyces cerevisiae-Zellen unter hyperosmotischem Stress.
Zusammenfassung
Intrinsisch ungeordnete Regionen (IDRs) sind Proteindomänen, die an entscheidenden zellulären Prozessen beteiligt sind. Unter Stressbedingungen ändern sich die physikalisch-chemischen Eigenschaften der zellulären Umgebung, was sich direkt auf das Konformationsensemble der IDRs auswirkt. IDRs reagieren von Natur aus empfindlich auf Umweltstörungen. Die Untersuchung, wie die physikalisch-chemischen Eigenschaften der Zelle das Konformationsensemble von IDRs regulieren, ist für das Verständnis der Umweltkontrolle ihrer Funktion unerlässlich. Hier beschreiben wir eine schrittweise Methode zur Messung der strukturellen Sensitivität von IDRs in lebenden Saccharomyces cerevisiae-Zellen als Reaktion auf hyperosmotische Stressbedingungen. Wir stellen die Verwendung des Ensemble-Fluoreszenz-Resonanz-Energietransfers (FRET) vor, um abzuschätzen, wie sich die globalen Dimensionen von IDRs während eines fortschreitenden Anstiegs des hyperosmotischen Stresses ändern, der Zellen mit einem beliebigen Osmolyten auferlegt wird. Darüber hinaus stellen wir ein Skript zur Verarbeitung von Fluoreszenzmessungen und zum Vergleich der strukturellen Empfindlichkeit für verschiedene IDRs zur Verfügung. Durch diese Methode können Forscher wertvolle Einblicke in die Konformationsänderungen gewinnen, die IDRs im komplexen intrazellulären Milieu bei wechselnden Umgebungen erfahren.
Einleitung
Intrinsisch ungeordnete Regionen (IDRs) sind kritische Komponenten in zellulären Prozessen1. In Kombination mit strukturierten Domänen sind IDRs essentiell für Proteinfunktionen. Die Aminosäurezusammensetzung von IDRs ist verzerrt und wird hauptsächlich durch geladene, hydrophile und kleine Reste dargestellt. Aufgrund dieser Eigenschaft werden IDRs als Domänen mit geringer Komplexität betrachtet 2,3. Zahlreiche IDRs haben Aufmerksamkeit erregt, vor allem, weil diese Regionen eine entscheidende Rolle bei pathologischen Zuständen spielen, insbesondere bei neurodegenerativen Erkrankungen. Solche Krankheiten sind durch Selbstorganisation und anschließende extrazelluläre oder intrazelluläre Ablagerung von IDRs in Neuronen gekennzeichnet4. Beispiele für solche IDRs sind Amyloid-β (Aβ) bei der Alzheimer-Krankheit, Huntingtin (HTT) bei der Huntington-Krankheit und das TAR-DNA-bindende Protein-43 (TDP-43) und fusioniert im Sarkom (FUS) bei amyotropher Lateralsklerose und frontotemporaler Demenz4. Die Untersuchung der strukturellen Umlagerungen von IDRs im Zusammenhang mit Krankheiten wurde durch spektroskopische Methoden, einschließlich des Fluoreszenz-Resonanz-Energietransfers (FRET), erheblich verbessert.
Die hydrophile und erweiterte Natur von IDRs macht sie extrem empfindlich gegenüber Änderungen der physikalisch-chemischen Eigenschaften der Lösungsumgebung5. Der Grad, in dem das Konformationsensemble von IDRs durch die Umwelt modifiziert wird, wird als strukturelle Sensitivität bezeichnet 5,6,7. Zur Untersuchung der Konformation und Dynamik von IDRs können verschiedene Techniken verwendet werden, darunter Zirkulardichroismus (CD) und Röntgenkleinwinkelstreuung (SAXS)8,9. Leider benötigen CD und SAXS große Mengen an gereinigten Proteinen, so dass sie für Studien in Zellen nicht geeignet sind. Im Gegensatz dazu ist FRET eine Technik, die die Fluoreszenzintensität von zwei fluoreszierenden Molekülen misst, die spezifisch eine IDR markieren, was bedeutet, dass sie in komplexen Gemischen wie lebenden Zellen überwacht werden können10. Die dynamische Messung der strukturellen Empfindlichkeit von IDRs in lebenden Zellen ist notwendig, um zu verstehen, wie die Umgebung die Konformation und Funktion des ungeordneten Proteoms reguliert.
FRET ist eine leistungsstarke Methode zur Quantifizierung der strukturellen Empfindlichkeit von IDRs sowie von globulären und Multidomänenproteinen in lebenden Zellen. Die Methode erfordert ein Konstrukt, das aus einem IDR von Interesse besteht, der zwischen zwei fluoreszierenden Proteinen (FP) eingebettet ist, einem sogenannten FRET-Paar. Für dieses Protokoll empfehlen wir die Verwendung von mCerulean3 als Spender-FP und Citrin als Akzeptor-FP aufgrund ihres großen Dynamikbereichs im Vergleich zu anderen FPs, die in einer früheren Studie über die Sensitivität von IDRs berichtet wurden6. FRET wurde zuvor genutzt, um die strukturelle Empfindlichkeit einer pflanzlichen IDR in verschiedenen zellulären Kontexten zu messen6. Darüber hinaus wurde diese Technik verwendet, um die Gesamtproteindimensionen von IDRs von verschiedenen Forschungsgruppen sowohl in vitro als auch in vivo zu charakterisieren 5,11.
Hier beschreiben wir die Ensemble-FRET-Methode zur Untersuchung der strukturellen Empfindlichkeit von IDRs in lebenden Hefezellen (Saccharomyces cerevisiae). Wir zeigen repräsentative Ergebnisse, die auf einer Pflanzen-IDR namens AtLEA4-5 basieren. AtLEA4-5 ist in Lösung ungeordnet, faltet sich aber zu α-Helix, wenn in vitro makromolekulares Crowding induziert wird 12. AtLEA4-5 ist ein gutes Referenzmodell für diese Methode, da es relativ klein (158 Rückstände), ungeordnet und empfindlich gegenüber Umweltstörungen ist, wie in silico und in vitro berichtet 6,12. Die hier vorgestellte Methode kann für Hochdurchsatzansätze skaliert werden, da Hefezellen leicht zu züchten sind und die Behandlung in kleinen Volumina angewendet wird. Darüber hinaus können kleine Modifikationen des Protokolls auf andere zelluläre Systeme wie Bakterien und Pflanzenzellen angewendet werden6. Das Protokoll kann in jedem molekularbiologischen Labor durchgeführt werden, das Zugang zu einem Mikroplatten-Reader mit Fluoreszenzmodus hat, ein Gerät, das in den meisten Forschungseinrichtungen verfügbar ist.
Protokoll
1. Plasmid-Konstrukt
- Amplifizieren Sie den offenen Leserahmen (ORF), der für die gewünschte IDR kodiert, durch Polymerase-Kettenreaktion (PCR). Schließen Sie das Stoppcodon nicht ein, da der ORF von den Genen flankiert wird, die für die fluoreszierenden Proteine kodieren. Für die Amplifikation werden Primer mit den Restriktionsstellen SacI (5') und BglII (3') entworfen.
HINWEIS: Für den Abschnitt "Repräsentative Ergebnisse" haben wir AtLEA4-5 als ausgewählten IDR verwendet. Wir haben den ORF von AtLEA4-5 aus dem pTrc99A-AtLEA4-5-Plasmid12 amplifiziert. - Das ORF-PCR-Produkt wird durch konsekutive Restriktion mit den Enzymen SacI und BglII13 verdaut.
- Beziehen Sie das kommerzielle Plasmid pDRFLIP38-AtLEA4-5 (#178189) von Addgene (https://www.addgene.org/178189/).
- Führen Sie eine aufeinanderfolgende Restriktion unter Verwendung von Sac-I-und Bgl-II-Enzymen durch, um das AtLEA4-5-ORF aus dem pDRFLIP38-AtLEA4-5-Plasmid zu entfernen, wobei ein offenes Plasmid verbleibt, das das FRET-Paar13 enthält.
- Die verdauten DNA-Fragmente werden durch Gelrückgewinnung aus einer Agarose-Gelelektrophorese14 gereinigt. Ligate die eingeschränkten Fragmente mit der DNA-Ligase15.
- Die Klonierungsreaktion wird in kompetente Escherichia coli-Zellen (DH5α oder verwandte Stämme) umgewandelt und in Luria-Bertani (LB)-Platten mit 50 μg/ml Ampicillin16 selektiert. Über Nacht (ON) bei 37 °C anbauen.
- Isolieren und züchten Sie mindestens fünf transformierte Kolonien in einer neuen Platte und einem neuen Genotyp durch PCR17. Aufreinigung der Plasmid-DNA aus einer positiv transformierten Kolonie mit Standard-Miniprep-Methoden18.
- Überprüfen Sie die Extraktion und Integrität der Plasmid-DNA mit Hilfe der Agarose-Gelelektrophorese19. Überprüfen Sie die korrekte Klonierung des Konstrukts mit der Sanger-Sequenzierung20.
2. Plasmidexpression in Hefezellen
HINWEIS: Verwenden Sie aseptische Standardtechniken, um die folgenden Schritte auszuführen. Verwenden Sie eine Kultur-Laminar-Flow-Haube oder ein Feuerzeug.
- Beimpfen Sie einen Streifen S. cerevisiae BJ5465 (ATCC: 208289) in 3 ml Hefe-Pepton-Dextrose-Medium (YPD) und wachsen Sie bei 30 °C und 200 U/min weiter.
HINWEIS: BJ5465 ist ein Protease-defizienter (CH1) Stamm, der für die Arbeit mit IDRs empfohlen wird. Andere S. cerevisiae-Stämme können getestet und verwendet werden. - Zentrifugieren Sie 1 ml der über Nacht gesättigten (OD600 ~ 3-4) Hefekultur bei 14.000 x g für 1 Minute. Entfernen Sie den Überstand vorsichtig durch Pipettieren.
- Resuspendieren Sie das Pellet in 1 ml Tris-EDTA (TE)-Puffer (pH 7,5), indem Sie das Röhrchen vorsichtig schnippen. Bei 14.000 x g 1 min zentrifugieren und den Überstand vorsichtig entfernen.
- Resuspendieren Sie das Pellet in 500 μl Lazy Bones-Puffer (40 % w/v PEG 3.350; 100 mM Lithiumacetat; 10 mM Tris-HCl; 1 mM EDTA; pH 7,5) durch Pipettieren. 25 μl gekochte (100 °C für 5 Minuten, gefolgt von 5 Minuten Abkühlen auf Eis) Lachsspermien-DNA (2 mg/ml) zu den resuspendierten Hefezellen geben.
- Fügen Sie der Mischung 100 ng der Plasmid-DNA hinzu und wirbeln Sie die Mischung 1 Minute lang ein, um eine gründliche Mischung zu gewährleisten. Lassen Sie die Mischung 1-2 h bei Raumtemperatur stehen.
- Die Hefemischung bei 42 °C 12 min hitzeschocken, dann weitere 12 min auf Eis abkühlen lassen. Das Röhrchen wird 1 Minute lang bei 14 000 x g zentrifugiert und der Überstand vorsichtig entfernt.
- Resuspendieren Sie das Pellet in 1 ml TE-Puffer (pH 7,5). Platte 100 μl der transformierten Zellen auf Hefe-Synthetik-Dropout-Medium ohne Uracil (SD-ura) Platten ergänzt mit 15 g/L Agar.
- Inkubieren Sie die Platten 2-3 Tage lang bei 30 °C (Abbildung 1). Streifen Sie mindestens fünf Hefe-Transformanten in einen neuen Teller und wachsen Sie weiter.
3. Validierung von Hefe-Transformanten
- Wählen Sie einen kleinen Teil jedes Transformantenkandidaten aus, indem Sie 5 μl 20 mM NaOH vorsichtig abkratzen und in einzelne PCR-Röhrchen resuspendieren.
- Die Probe wird 10 Minuten lang in einem Thermocycler auf 99 °C erhitzt, um die Zellen zu lysieren und die DNA in die Lösung freizusetzen. Dieser Schritt ist entscheidend für die Vorbereitung des DNA-Templates für die PCR.
- 1 μl der gekochten Probe wird in ein separates PCR-Reaktionsgemisch überführt, das die entsprechenden Primer und PCR-Reagenzien für die Genotypisierung enthält. Wir haben die folgenden Primer verwendet: Forward Primer: 5'-AAATATACCCCAGCCTCGATCTAGA-3'. Umgekehrte Grundierung: 5'-GTAATACGACTCACTATAGGGCG-3'.
- Richten Sie eine PCR-Reaktion ein und validieren Sie das Vorhandensein des richtigen Plasmids mithilfe der Agarose-Gelelektrophorese. Wählen Sie einen Transformanten für weitere Experimente aus.
4. Vorbereitung der Hefezellkultur für den FRET-Assay
- Beimpfen Sie einen Streifen des ausgewählten Hefetransformanten in 3 ml flüssiges 1x SD-Ura-Medium.
Anmerkungen: Führen Sie diesen Vorgang in einer Laminar-Flow-Haube und sterilem Material durch. - Wachsen Sie bei 30 °C, 200 U/min für mindestens 12 Stunden, um die Sättigung zu erreichen. Messen Sie den OD600. Das Wachstum sollte zwischen OD600 = 1,0-2,0 liegen.
HINWEIS: Wenn OD600 niedriger als 1,0 ist, geben Sie den Zellen mehr Inkubationszeit zum Wachsen. Wenn OD600 2.0 überschreitet, fahren Sie nicht fort und starten Sie mit Schritt 4.1 neu.
5. Hefezellvorbereitung für FRET-Assay
- Sammeln Sie 2 ml der Nachthefekultur und zentrifugieren Sie sie bei 14.000 x g bei Raumtemperatur. Entfernen Sie den Überstand vorsichtig durch Pipettieren.
- Resuspendieren Sie das Pellet in 1 ml 50 mM 2-(N-Morpholino) Ethansulfonsäure (MES)-Puffer (pH 6).
- Bei Raumtemperatur bei 14.000 x g zentrifugieren. Entfernen Sie den Überstand.
- Wiederholen Sie die Schritte 5.2 und 5.3. Hefezellen in 2 ml MES-Puffer, pH 6, resuspendieren und das Volumen in ein Reagenzreservoir gießen.
6. Einrichten der Fluoreszenzmessungen
HINWEIS: Es wird davon ausgegangen, dass der vorliegende Scan in einem Mikroplatten-Reader mit Fluoreszenzmodus durchgeführt wird.
- Stellen Sie das Gerät für die Grundeinstellungen wie folgt ein: Messtyp: Fluoreszenzintensitätsspektrum (FI); Name der Mikroplatte: Greiner 96 F-Boden.
- Stellen Sie das Gerät für die Optikeinstellungen wie folgt ein: Nein. Anzahl der Wellenlängen-Scanpunkte: 91; Anregungswellenlänge: 433 nm; Anregungsbandbreite: 10 nm; Emissionswellenlänge: 460 - 550; Schrittweite: 1 nm; Emissionsbandbreite: 10 nm; Brennhöhe durch Autofokus; Brennhöhe: 5 mm; Für die Verstärkung verwendete Wellenlänge: 490 nm.
- Stellen Sie das Gerät für allgemeine Einstellungen wie folgt ein: Einschwingzeit: 0,1 s; Leserichtung: unidirektional, horizontal von links nach rechts, von oben nach unten.
HINWEIS: Die manuell eingegebene Verstärkung muss für jede Messung mit dem Well angepasst werden, von dem erwartet wird, dass es die höchsten Fluoreszenzintensitätsstufen während des gesamten Wellenlängenscans anzeigt.
7. Herstellung hypertonischer Lösungen und Fluoreszenzmessungen
- Bereiten Sie Lösungen von 0 M, 0,2 M, 0,4 M, 0,6 M, 0,8 M, 1 M, 1,5 M und 2 M NaCl in einer 96-Well-Schwarzplatte mit klarem Boden vor. Das endgültige Volumen in jeder Vertiefung beträgt 200 μl (150 μl Osmolytlösung + 50 μl Hefezellsuspension).
HINWEIS: Andere Osmolyte können verwendet werden, um hyperosmotischen Stress zu induzieren. Alle Lösungen müssen in 50 mM MES-Puffer pH 6 hergestellt werden. - Laden Sie 150 μl 50 mM MES-Puffer, pH 6 in die ersten drei Vertiefungen der Reihe A (A1, A2, A3) und fügen Sie in den nachfolgenden Vertiefungen 150 μl der entsprechenden Lösung in aufsteigender Reihenfolge von links nach rechts hinzu (Abbildung 2). Um alle vorgeschlagenen Konzentrationen zu prüfen, wird die Beladung in Reihe B fortgesetzt.
HINWEIS: Diese Einstellung berücksichtigt die Messungen von 3 technischen Replikaten für jede Konzentration. - Verwenden Sie eine 12-Kanal-Mikropipette, um 50 μl gewaschene Hefezellen in jede Vertiefung zu übertragen. Hefezellen neigen dazu, sich auf dem Boden des Reservoirs zu sedimentieren. Stellen Sie sicher, dass Sie die Hefesuspension gründlich resuspendieren, indem Sie auf und ab pipettieren, bevor Sie die Zellen übertragen.
- Wägen Sie die Zellen in die 96-Well-Platte mit den verschiedenen Lösungen und mischen Sie sie gut, indem Sie mindestens 4x auf und ab pipettieren.
- Messen Sie die Emissionsspektren der Fluoreszenzintensität sofort für die gewünschten Wells in einem Mikroplatten-Reader mit den Spezifikationseinstellungen von Schritt 6. Wiederholen Sie das Verfahren für jede IDR, die im FRET-Assay getestet werden soll.
- Optionaler Schritt. Falls verfügbar, messen Sie ein reines Spenderkonstrukt (pDRFLIP38-mCerulean3) gemäß den hier beschriebenen Schritten.
HINWEIS: Wir empfehlen, diesen Schritt durchzuführen, um die FRET-Effizienz jeder Bedingung zu berechnen. Wenn das Lesegerät diesen Schritt nicht ausführen kann, kann FRET mit der FRET-Verhältnis-Methode berechnet werden. Beide Methoden für FRET-Berechnungen werden in den Schritten 8 und 9 erläutert. - Führen Sie für jedes Konstrukt mindestens drei Replikationen an drei unabhängigen Tagen durch.
8. Datenverarbeitung mit der FRET-Effizienzmethode
HINWEIS: Für Leser mit Kenntnissen der Programmiersprache R stellen wir eine Reihe von R-Skripts zur Verfügung, um die in diesem Abschnitt beschriebene Datenverarbeitung durchzuführen. Skripte finden Sie unter https://github.com/Kaz-bits/cuevaslab-procotols/tree/main/FRET. Befolgen Sie die Anweisungen in der README-Datei.
- Exportieren Sie Daten als .xlsx Dateien aus dem Mikroplatten-Reader.
- Normalisieren Sie die Fluoreszenzintensitätswerte bei jeder Scanwellenlänge auf den isosbestischen Punkt des verwendeten FRET-Paares.
HINWEIS: Dieser Schritt ist notwendig, um die Spektren aller getesteten Bedingungen zu vergleichen. Der isosbestische Punkt des mCerulean3- und Citrin-FRET-Paares beträgt 515 nm. Ermitteln Sie beispielsweise das Verhältnis der Fluoreszenzintensitätswerte von 460 nm/515 nm, 461 nm/515 nm, 462 nm/515 nm usw. - Berechnen Sie den Mittelwert für jedes technische Replikat, sobald die Fluoreszenzwerte normalisiert sind. Insgesamt sollte es für jeden Zustand von hyperosmotischem Stress eine Säule geben.
- Visualisieren Sie alle Fluoreszenzintensitätsspektren im selben Diagramm (Abbildung 3). Jedes Spektrum sollte eine Kombination der Fluoreszenzemissionsspektren einzelner mCerulean3 und Citrin aufweisen. In seltenen Fällen, in denen der FRET-Wirkungsgrad 0 beträgt, wird nur das Donorfluoreszenzemissionsspektrum beobachtet, oder wenn der FRET-Wirkungsgrad 1 beträgt, wird nur die Akzeptorfluoreszenz beobachtet. Der Fluoreszenzemissionspeak für mCerulean3 (Donor) beträgt 475 nm und der Fluoreszenzemissionspeak für Citrin (Akzeptor) 525 nm.
- Stellen Sie fest, ob die Fluoreszenzmessung eine typische FRET-Änderung als Reaktion auf hyperosmotischen Stress zeigt. Wenn die Konformation des IDR empfindlich auf die Behandlung reagiert, spiegelt sich dies als Änderung der FRET-Effizienz wider. Eine typische Änderung der FRET-Effizienz koppelt eine Abnahme der Fluoreszenzintensität des Donors mit einer Erhöhung der Fluoreszenzintensität des Akzeptors oder umgekehrt.
- Quantifizieren Sie die FRET-Effizienz (EFRET). Erhalten Sie die normalisierten Spender-Peakwerte (475 nm/515 nm) für das IDR-Konstrukt und das Nur-Donor-Konstrukt für jeden Zustand hyperosmotischer Belastung. Verwenden Sie die mittleren normalisierten Spitzenwerte der technischen Replikate. Führen Sie diesen Vorgang für jedes der drei unabhängigen Replikate aus. Berechnen Sie EFRET mit der folgenden Formel:
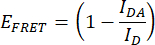
Dabei istI DA das Verhältnis 475 nm/515 nm aus dem IDR-Konstrukt (FRET-Paar-Konstrukt) und ID das Verhältnis 475 nm/515 nm aus dem Nur-Donor-Konstrukt. - Vergleichen Sie die FRET-Wirkungsgrade. Normalisieren Sie dieE-FRET-Werte jeder hyperosmotischen Spannungsbedingung auf dieE-FRET-Werte der Nicht-Stressbedingung (z. B. 0 M NaCl). Vergleiche mit einem Boxplot oder einem Diagramm mit glatten Kurven. Führen Sie eine einfache ANOVA gefolgt von einem statistischen Post-hoc-Tukey-Test durch (p-Werte: *p < 0,05, **p < 0,01, ***p < 0,001, ****p < 0,0001).
9. Datenverarbeitung nach der FRET-Ratio-Methode
HINWEIS: Wenn keine reinen Spendermessungen erfasst werden können, führen Sie die FRET-Verhältnis-Methode durch. Diese Methode vergleicht das FRET-Verhältnis über die verschiedenen hyperosmotischen Stressbedingungen. Die Schritte 7.1 bis 7.5 sollten vor den Schritten dieses Abschnitts ausgeführt werden, um ein typisches FRET-Verhalten zu validieren. Für Leser mit Kenntnissen der Programmiersprache R stellen wir eine Reihe von R-Skripts zur Verfügung, um die in diesem Abschnitt beschriebene Datenverarbeitung durchzuführen. Skripte finden Sie unter https://github.com/Kaz-bits/cuevaslab-procotols/tree/main/FRET. Befolgen Sie die Anweisungen in der README-Datei.
- Extrahieren Sie Daten bei 475 nm und 525 nm aus der xlsx-Datei, die zuvor vom Mikroplatten-Reader exportiert wurde. Diese Werte entsprechen der Fluoreszenzemissionsspitze für mCerulean3 (Donor, DxDm) und der Fluoreszenzemissionsspitze für Citrin (Akzeptor, DxAm) bei Anregung des Donorfluorophors (433 nm).
- Normalisieren Sie die Fluoreszenzintensitätswerte bei 475 nm und 525 nm auf den isosbestischen Punkt des verwendeten FRET-Paares. Der isosbestische Punkt des mCerulean3- und Citrin-FRET-Paares beträgt 515 nm. Erhalten Sie das FRET-Verhältnis (DxAm/DxDm).
- Vergleichen Sie FRET-Verhältnisse. Normalisieren Sie die DxAm/DxDm-Werte jeder hyperosmotischen Stressbedingung auf den Mittelwert DxAm/DxDm der Nicht-Stressbedingung (z. B. 0 M NaCl). Vergleiche mit einem Boxplot oder einem Diagramm mit glatten Kurven (Abbildung 4 und Abbildung 5). Führen Sie eine einfache ANOVA gefolgt von einem statistischen Post-hoc-Tukey-Test durch (p-Werte: *p < 0,05, **p < 0,01, ***p < 0,001, ****p < 0,0001).
Ergebnisse
Nach der Transformation von Hefezellen mit dem pDRFLIP38-AtLEA4-5-Plasmid wurde die Fluoreszenz der positiven Transformanten mit einem Blaulicht-Transilluminator und einem Filter beobachtet (Abbildung 1). Die Vorbereitung der verschiedenen Lösungen zur Induktion von hyperosmotischem Stress ist zeitaufwändig, daher empfehlen wir, der 96-Well-Vorlage in Abbildung 2 zu folgen. Unmittelbar nach der hyperosmotischen Stressbehandlung mit unterschiedlichen Konzentrat...
Diskussion
Die hier vorgestellte Methode bietet eine Möglichkeit, Einblicke zu gewinnen, wie die globalen Dimensionen des Ensembles von IDRs Umweltstörungen wahrnehmen und darauf reagieren. Diese Methode beruht auf einem genetisch kodierten Konstrukt und benötigt keine zusätzlichen Komponenten außer einer plasmidstabilen Expression in Hefezellen, wodurch sie für potenzielle Anwendungen in anderen Zelltypen angepasst werden kann. Darüber hinaus ist es vielseitig einsetzbar, um andere physikalisch-chemische Störungen zu erfor...
Offenlegungen
Die Autoren erklären keine Interessenkonflikte.
Danksagungen
Wir danken den Mitgliedern des Cuevas-Velazquez-Labors für die kritische Überprüfung des Manuskripts. Diese Arbeit wurde unterstützt durch das Programa de Apoyo a Proyectos de Investigación e Innovación Tecnológica, die Dirección General de Asuntos del Personal Académico, die Universidad Nacional Autónoma de México (UNAM-PAPIIT) Projektnummer IA203422; Consejo Nacional de Humanidades, Ciencias y Tecnología (CONAHCYT), Projektnummer 252952; und Programa de Apoyo a la Investigación y el Posgrado, Facultad de Química, Universidad Nacional Autónoma de México, Grant 5000-9182. CET (CVU 1083636) und CAPD (CVU 1269643) würdigen CONAHCYT für ihr M.Sc-Stipendium.
Materialien
| Name | Company | Catalog Number | Comments |
| 96-well plate | Greiner Bio-One | 655096 | |
| Agar | Sigma-Aldrich | 5040 | |
| BglII | New England BioLabs | R0144S | |
| BJ5465 cells | American Type Culture Collection | 208289 | |
| Buffer MES 50 mM | Sigma-Aldrich | M8250 | |
| Buffer Tris-HCl 10 mM | Invitrogen | 15506017 | |
| EDTA 1 mM | Merck | 108452 | |
| Falcon tubes | Corning | 352057 | |
| LB media | Sigma-Aldrich | L2897 | |
| Lithium acetate 0.1 M | Sigma-Aldrich | L6883 | |
| Low Melt Agarose | GOLDBIO | A-204-25 | |
| Microcentrifuge | eppendorf | 5452000010 | |
| Miniprep kit | ZymoPure | D4210 | |
| NaOH 0.02 M | Merck | 106462 | |
| PEG 3,350 40% | Sigma-Aldrich | 1546547 | |
| plasmid pDRFLIP38-AtLEA4-5 | addgene | 178189 | |
| Plate reader | BMG LABTECH | CLARIOstar Plus | |
| SacI | New England BioLabs | R3156S | |
| Salmon sperm DNA 2 mg/mL | Thermo Fisher Scientific | 15632011 | |
| SD-Ura | Sigma-Aldrich | Y1501 | |
| Sodium cloride | Sigma-Aldrich | S9888 | |
| Taq polymesare | Promega | M5123 | |
| Transiluminator | Accuris instruments | E4000 | |
| UV-Visible spectrophotometer | Thermo Fisher Scientific | Biomate3 | |
| YPD media | Sigma-Aldrich | Y1500 |
Referenzen
- Wright, P. E., Dyson, H. J. Intrinsically disordered proteins in cellular signalling and regulation. Nat Rev Mol Cell Biol. 16 (1), 18-29 (2015).
- Covarrubias, A. A., Romero-Pérez, P. S., Cuevas-Velazquez, C. L., Rendón-Luna, D. F. The functional diversity of structural disorder in plant proteins. Arch Biochem Biophys. 680, 108229 (2019).
- Ahmed, S. S., et al. Characterization of intrinsically disordered regions in proteins informed by human genetic diversity. PLoS Comput Biol. 18 (3), e1009911 (2022).
- Birol, M., Melo, A. M. Untangling the conformational polymorphism of disordered proteins associated with neurodegeneration at the single-molecule level. Front Mol Neurosci. 12, 309 (2019).
- Moses, D., et al. Revealing the hidden sensitivity of intrinsically disordered proteins to their chemical environment. J Phys Chem Lett. 11 (23), 10131-10136 (2020).
- Cuevas-Velazquez, C. L., et al. Intrinsically disordered protein biosensor tracks the physical-chemical effects of osmotic stress on cells. Nat Commun. 12 (1), 5438 (2021).
- Holehouse, A. S., Sukenik, S. Controlling structural bias in intrinsically disordered proteins using solution space scanning. J Chem Theory Comput. 16 (3), 1794-1805 (2020).
- Martin, E. W., Hopkins, J. B., Mittag, T. Small-angle X-ray scattering experiments of monodisperse intrinsically disordered protein samples close to the solubility limit. Methods Enzymol. 646, 185-222 (2021).
- Miles, A. J., Drew, E. D., Wallace, B. A. DichroIDP: a method for analyses of intrinsically disordered proteins using circular dichroism spectroscopy. Commun Biol. 6 (1), 823 (2023).
- Kaminski, C. F., Rees, E. J., Schierle, G. S. K. A quantitative protocol for intensity-based live cell FRET imaging. Method Mol Biol. 1076, 445-454 (2014).
- Moses, D., et al. Structural biases in disordered proteins are prevalent in the cell. bioRxiv. , (2022).
- Cuevas-Velazquez, C. L., Saab-Rincón, G., Reyes, J. L., Covarrubias, A. A. The Unstructured N-terminal Region of Arabidopsis Group 4 Late Embryogenesis Abundant (LEA) Proteins Is Required for Folding and for Chaperone-like Activity under Water Deficit. J Biol Chem. 291 (20), 10893-10903 (2016).
- JoVE Science Education Database. . Restriction enzyme digests. , (2023).
- JoVE Science Education Database. . Gel purification. , (2023).
- JoVE Science Education Database. . DNA ligation reactions. , (2023).
- JoVE Science Education Database. . Bacterial transformation using heat Ssock and competent cells. , (2023).
- JoVE Science Education Database. . PCR: Principle, instrumentation, and applications. , (2023).
- JoVE Science Education Database. . Plasmid purification. , (2023).
- JoVE Science Education Database. . DNA gel electrophoresis. , (2023).
- JoVE Core Molecular Biology. Sanger/chain termination sequencing using dideoxynucleotides - Concept Available from: https://app.jove.com/science-education/v/12020/sanger-sequencing (2023)
- Theillet, F. X., et al. Physicochemical properties of cells and their effects on intrinsically disordered proteins (IDPs). Chem Rev. 114 (13), 6661-6714 (2014).
- Brutscher, B., et al. NMR methods for the study of instrinsically disordered proteins structure, dynamics, and interactions: General overview and practical guidelines. Adv Exp Med Biol. 870, 49-122 (2015).
- Metskas, L. A., Rhoades, E. Single-molecule FRET of intrinsically disordered proteins. Annu Rev Phys Chem. 71, 391-414 (2020).
- Roebroek, T., et al. Simultaneous readout of multiple FRET pairs using photochromism. Nat Commun. 12 (1), 2005 (2021).
- Algar, W. R., Hildebrandt, N., Vogel, S. S., Medintz, I. L. FRET as a biomolecular research tool - understanding its potential while avoiding pitfalls. Nat Methods. 16 (9), 815-829 (2019).
- Lyon, A. S., Peeples, W. B., Rosen, M. K. A framework for understanding the functions of biomolecular condensates across scales. Nat Rev Mol Cell Biol. 22 (3), 215-235 (2021).
- Belott, C., Janis, B., Menze, M. A. Liquid-liquid phase separation promotes animal desiccation tolerance. Proc Natl Acad Sci U S A. 117 (44), 27676-27684 (2020).
- Miermont, A., et al. Severe osmotic compression triggers a slowdown of intracellular signaling, which can be explained by molecular crowding. Proc Natl Acad Sci U S A. 110 (14), 5725-5730 (2013).
- Saito, H., Posas, F. Response to hyperosmotic stress. Genetics. 192 (2), 289-318 (2012).
- Selenko, P., Wagner, G. Looking into live cells with in-cell NMR spectroscopy. J Struct Biol. 158 (2), 244-253 (2007).
- Cattani, J., Subramaniam, V., Drescher, M. Room-temperature in-cell EPR spectroscopy: alpha-Synuclein disease variants remain intrinsically disordered in the cell. Phys Chem Chem Phys. 19 (28), 18147-18151 (2017).
- Beveridge, R., Chappuis, Q., Macphee, C., Barran, P. Mass spectrometry methods for intrinsically disordered proteins. Analyst. 138 (1), 32-42 (2013).
- Beveridge, R., et al. Ion mobility mass spectrometry uncovers the impact of the patterning of oppositely charged residues on the conformational distributions of intrinsically disordered proteins. J Am Chem Soc. 141 (12), 4908-4918 (2019).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten